e5ea5658a75cec5c45782573d852b51d.ppt
- Количество слайдов: 35

Variations de nombre de copies dans le génome humain Richard Redon rr 2@sanger. ac. uk
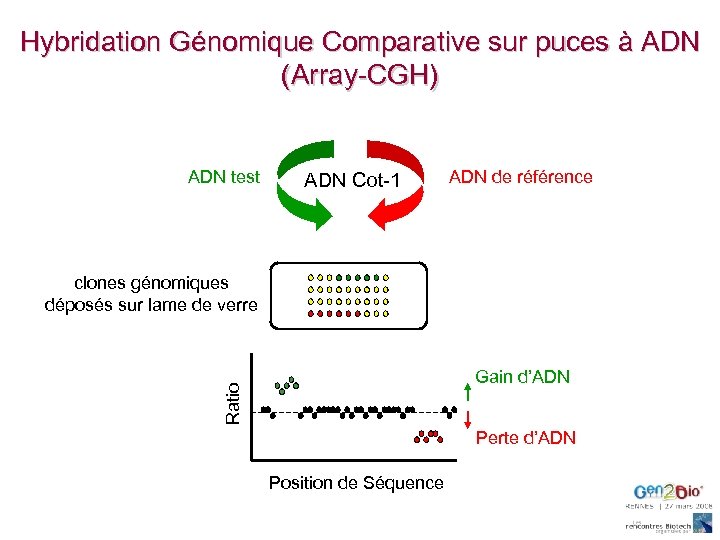
Hybridation Génomique Comparative sur puces à ADN (Array-CGH) ADN test ADN Cot-1 ADN de référence clones génomiques déposés sur lame de verre Ratio Gain d’ADN Perte d’ADN Position de Séquence

La puce 1 -Mb : couverture de l’ensemble du génome avec une résolution moyenne d’un clone par Mb 3523 BAC ou PAC déposés en double Clones validés par séquençage des extrémités Fiegler et al. (2003) Genes Chromosomes Cancer 36: 361 -374

50 patients 12 avec un déséquilibre 6 délétions et 1 duplication sont de novo 1 délétion et 4 duplications sont héritées

Identification de nouveaux syndromes (“Genomic disorders”)
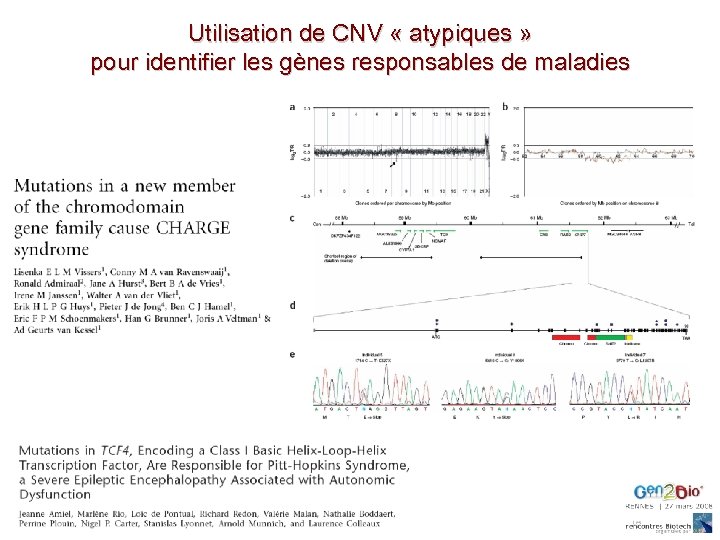
Utilisation de CNV « atypiques » pour identifier les gènes responsables de maladies

Les CNV à l’origine de maladies à caractère… • dominant CNV absents dans la population non atteinte • récessif CNV présents dans la population non atteinte • complexe

2004 2005 Nov. 2006
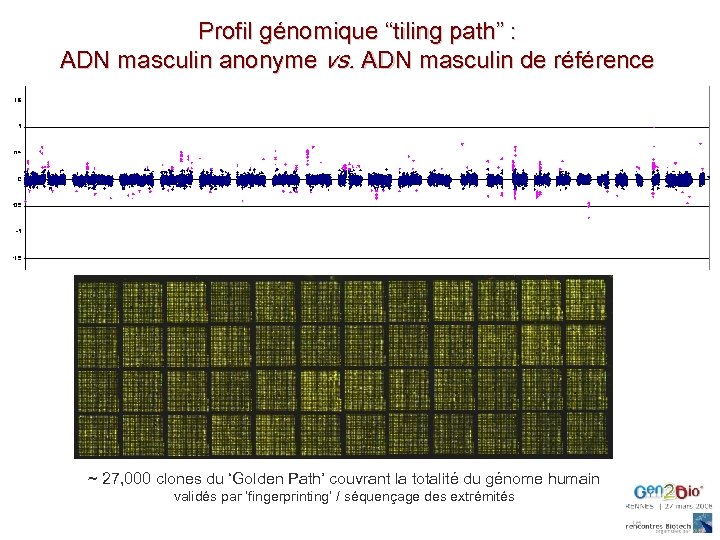
Profil génomique “tiling path” : ADN masculin anonyme vs. ADN masculin de référence ~ 27, 000 clones du ‘Golden Path’ couvrant la totalité du génome humain validés par ‘fingerprinting’ / séquençage des extrémités
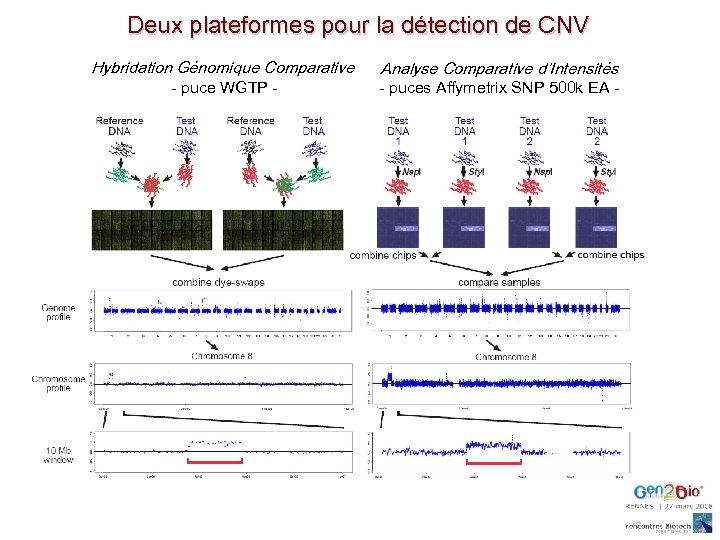
Deux plateformes pour la détection de CNV Hybridation Génomique Comparative - puce WGTP - Analyse Comparative d’Intensités - puces Affymetrix SNP 500 k EA -

Détection de CNV pour 1447 régions le long du génome
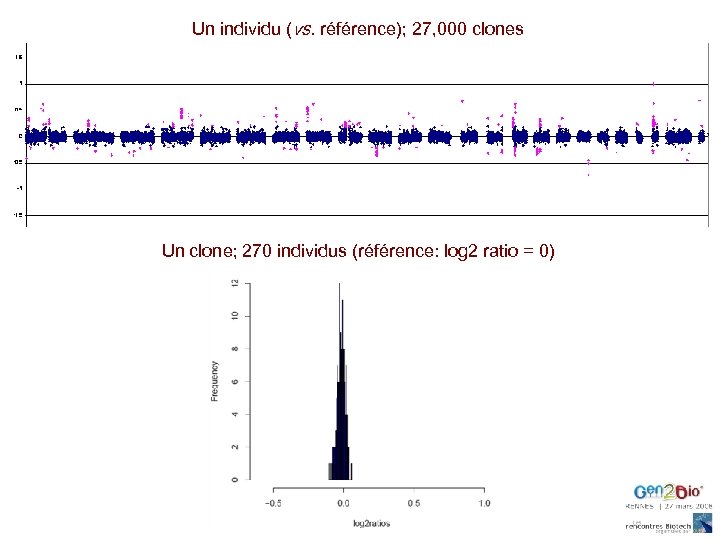
Un individu (vs. référence); 27, 000 clones Un clone; 270 individus (référence: log 2 ratio = 0)
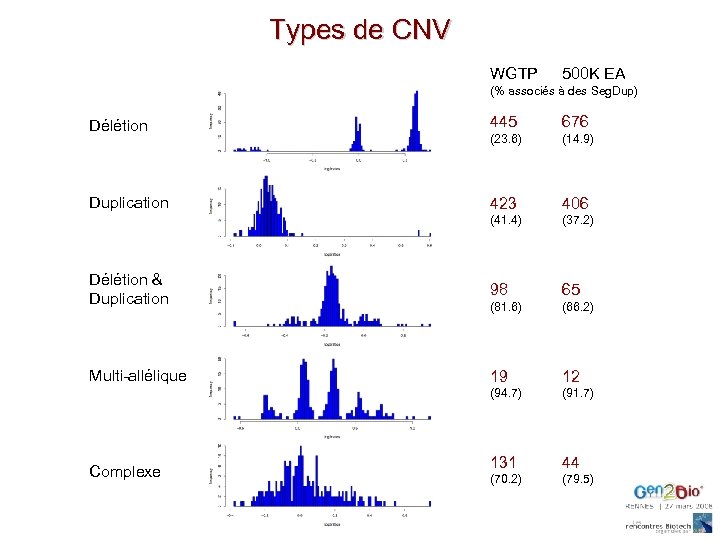
Types de CNV WGTP 500 K EA Délétion 445 676 Duplication 423 406 Délétion & Duplication 98 65 Multi-allélique 19 12 131 44 (% associés à des Seg. Dup) Complexe (23. 6) (41. 4) (81. 6) (94. 7) (70. 2) (14. 9) (37. 2) (66. 2) (91. 7) (79. 5)
CNV et phénotypes CNV causant une pathologie Variations de nombre de copies: - prédisposant à des maladies ? - causant des phénotypes plus légers ? « Polymorphismes » de nombre de copies 0 2 4 Taille en Mb 6 8 10
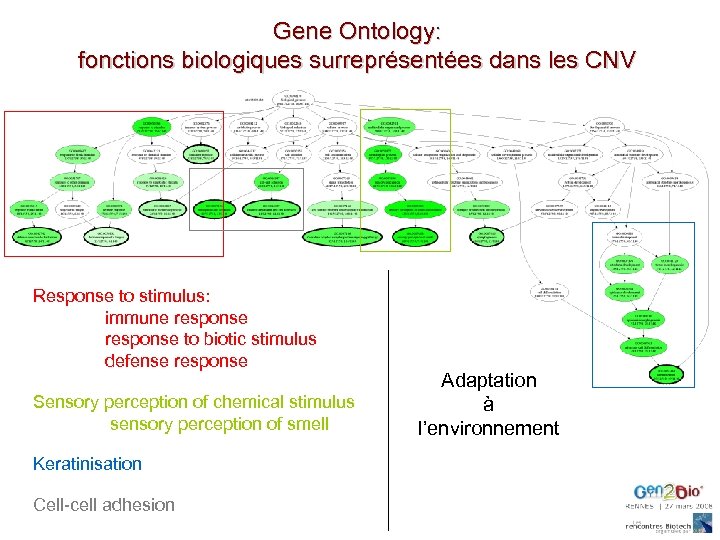
Gene Ontology: fonctions biologiques surreprésentées dans les CNV Response to stimulus: immune response to biotic stimulus defense response Sensory perception of chemical stimulus sensory perception of smell Keratinisation Cell-cell adhesion Adaptation à l’environnement
immune response (GO: 0006955)
Différenciation des Populations YRI: CHB+JPT CEU: CHB+JTP CEU: YRI J Clin Endocrinol Metab 91(2): 687 -693 Large Differences in Testosterone Excretion in Korean and Swedish Men Are Strongly Associated with a UDP-Glucuronosyl Transferase 2 B 17 Polymorphism UGT 2 B 17 VST=(VT-VS)/VT Jenny Jakobsson, Lena Ekström, Nobuo Inotsume, Mats Garle, Mattias Lorentzon, Claes Ohlsson, Hyung-Keun Roh, Kjell Carlström and Anders Rane CCL 3 L 1

Nouvelles avancées sur les CNV Plus de populations 410 individus de 20 différentes ethnies sur puce WGTP

Populations CEPH analysées sur puces WGTP Population Origine géographique Total Adygei Russie (Caucase) 15 Bantu N. E. Kenya 12 Pygmées Biaka Brahui CEU** Druze Français Chinois Han * Karitiana Maya Pygmées Mbuti République Centrafricaine Pakistan Utah, US Israël (Carmel) France (Différentes régions) Beijing, Chine Brésil Mexique Rép. Démocratique du Congo 25 23 27 29 27 23 21 23 14 Miaozu Mozabite NAN Melanesian Papuan 17 Pima San Sindhi Yakut Yoruba** Chine Algérie (Mzab) Bougainville Nouvelle Guinée 10 30 19 Mexique Namibie Pakistan Sibérie Nigeria 7 7 24 22 24 Excepté: * Hap. Map plaque I ** Hap. Map plaque II Référence: NA 10851 20 populations TOTAL = 410 individus
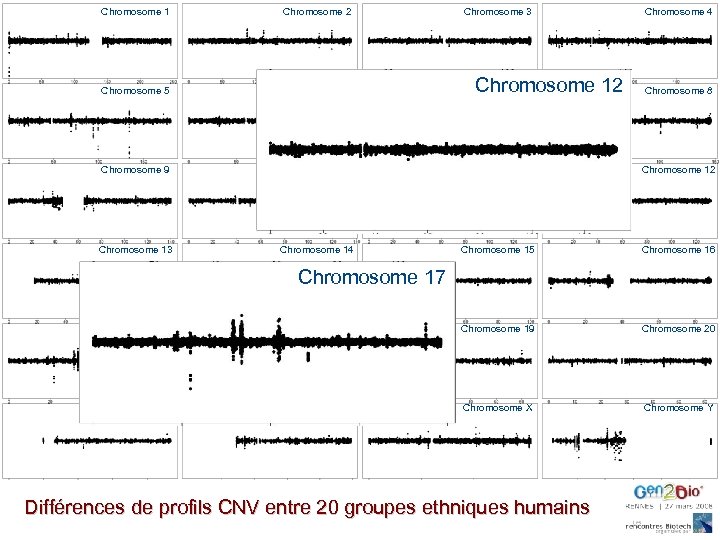
Chromosome 1 Chromosome 2 Chromosome 3 Chromosome 5 Chromosome 6 Chromosome 7 Chromosome 8 Chromosome 9 Chromosome 10 Chromosome 11 Chromosome 12 Chromosome 13 Chromosome 14 Chromosome 15 Chromosome 16 Chromosome 12 Chromosome 4 Chromosome 17 Chromosome 18 Chromosome 19 Chromosome 20 Chromosome 21 Chromosome 22 Chromosome X Chromosome Y Différences de profils CNV entre 20 groupes ethniques humains

Nouvelles avancées sur les CNV Plus de populations 410 individus de 20 différentes ethnies sur puce WGTP Plus d’espèces Différences de nombres de copies et CNV chez les grands singes

Recherche des Différences de Nombre de Copies entre les Grands Singes n= 5 Comparaisons sur puces WGTP (humaines) ORANG-OUTAN n= 4 GORILLE Référence (NA 10851) n= 4 CHIMPANZE HOMME Fengtang Yang Chris Tyler-Smith n= 3 BONOBO
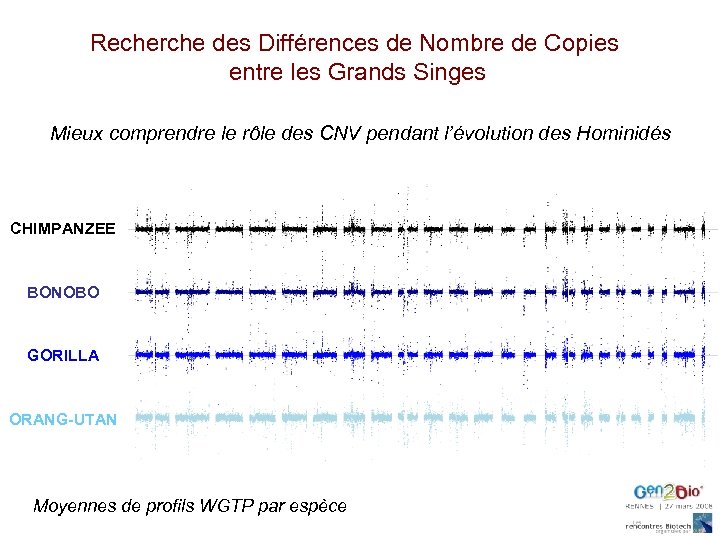
Recherche des Différences de Nombre de Copies entre les Grands Singes Mieux comprendre le rôle des CNV pendant l’évolution des Hominidés CHIMPANZEE BONOBO GORILLA ORANG-UTAN Moyennes de profils WGTP par espèce
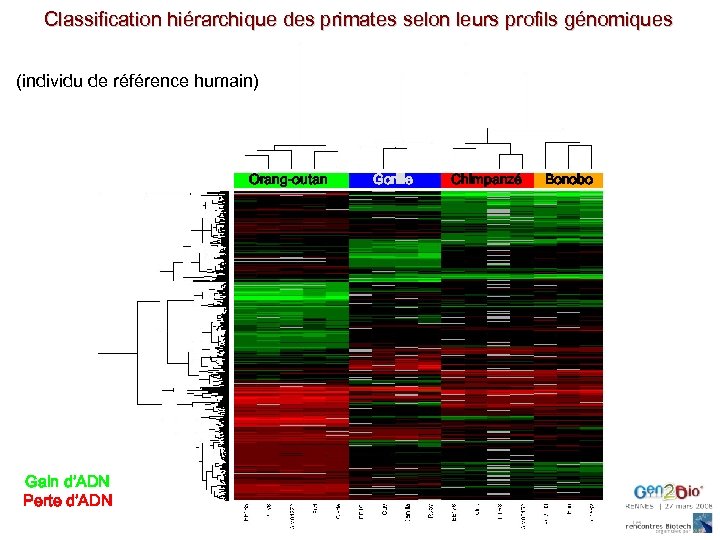
Classification hiérarchique des primates selon leurs profils génomiques (individu de référence humain) Orang-outan Gain d’ADN Perte d’ADN Gorille Chimpanzé Bonobo

Variations de Nombre de Copies chez le Chimpanzé dans le contexte du Génome Humain CNV chez 30 chimpanzés CNV chez 30 humains Différences de nombre de copies entre homme et chimpanzé 29 d’Afrique Occidentale 1 d’Afrique Orientale 10 Yoruba (Hap. Map) 10 Pygmées Biaka 10 Pygmées Mbuti référence: Clint référence: NA 10851 PJ Perry WGTP 29 k
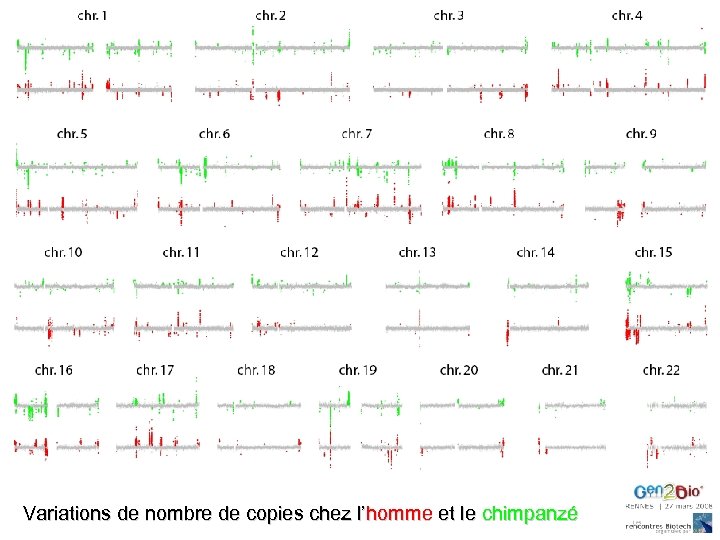
Variations de nombre de copies chez l’homme et le chimpanzé

Nouvelles avancées sur les CNV Plus de populations 410 individus de 20 différentes ethnies sur puce WGTP Plus d’espèces Différences de nombres de copies et CNV chez les grands singes Meilleure résolution De 27, 000 clones à 42 M oligos (20 puces 2. 1 M Nimble. Gen)
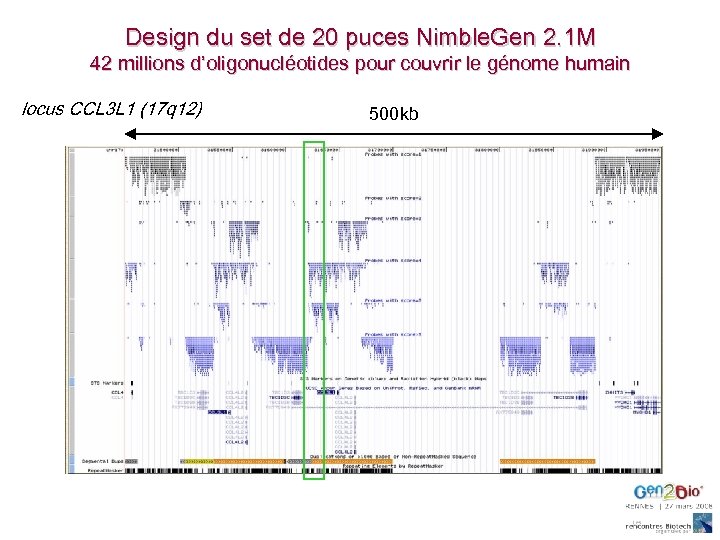
Design du set de 20 puces Nimble. Gen 2. 1 M 42 millions d’oligonucléotides pour couvrir le génome humain locus CCL 3 L 1 (17 q 12) 500 kb
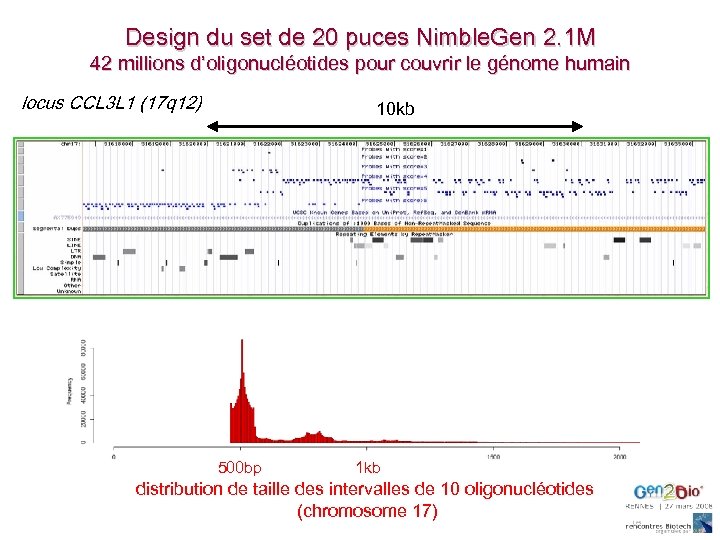
Design du set de 20 puces Nimble. Gen 2. 1 M 42 millions d’oligonucléotides pour couvrir le génome humain locus CCL 3 L 1 (17 q 12) 10 kb 500 bp 1 kb distribution de taille des intervalles de 10 oligonucléotides (chromosome 17)
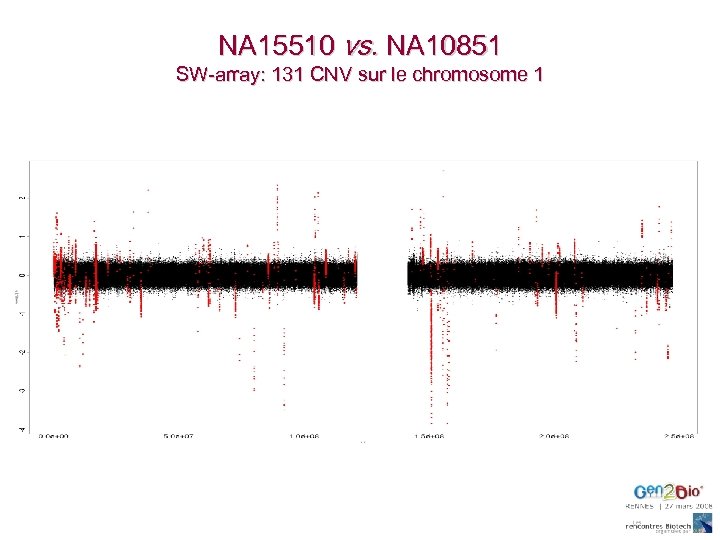
NA 15510 vs. NA 10851 SW-array: 131 CNV sur le chromosome 1
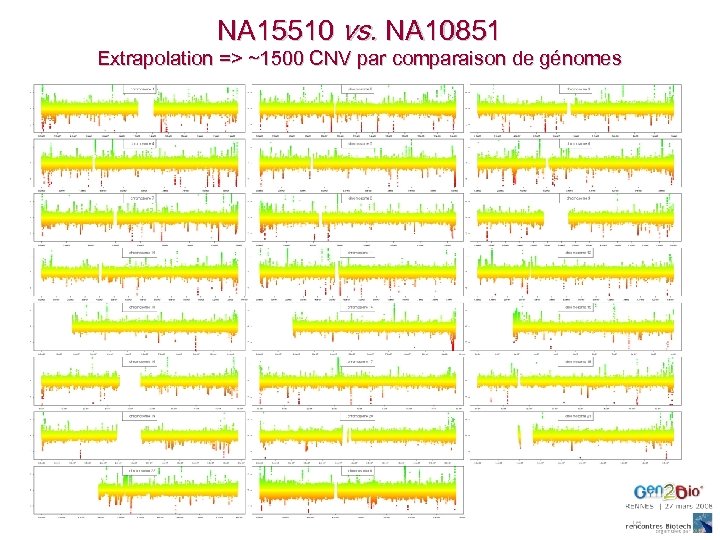
NA 15510 vs. NA 10851 Extrapolation => ~1500 CNV par comparaison de génomes
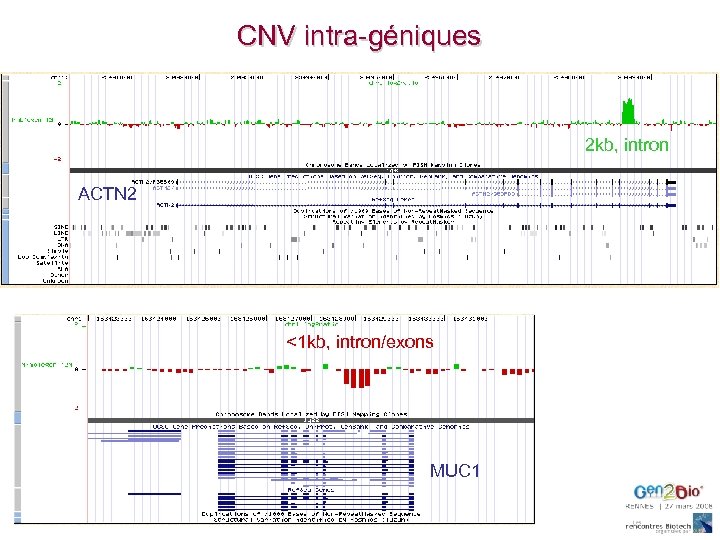
CNV intra-géniques 2 kb, intron ACTN 2 <1 kb, intron/exons MUC 1
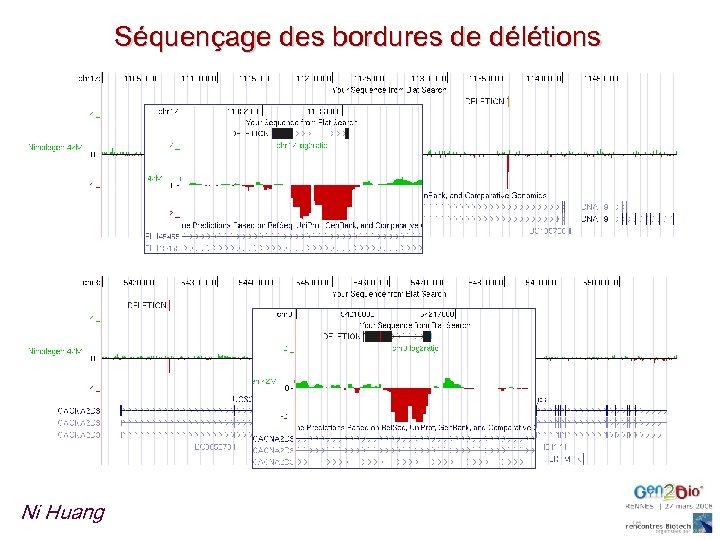
Séquençage des bordures de délétions Ni Huang

Nouvelles avancées sur les CNV Plus de populations 410 individus de 20 différentes ethnies sur puce WGTP Plus d’espèces Différences de nombres de copies et CNV chez les grands singes Meilleure résolution 40 individus sur Nimble. Gen 42 M (20 CEU + 20 Yoruba) Nouvelles technologies de séquençage: méthodes en développement Etudes d’association WTCCC 2 - étude pilote: vers une plateforme CGH dédiée au génotypage des CNV

Genome Structural Variation Consortium Lars Feuk Steve Scherer Toronto Charles Lee Boston Team 70 Nigel Carter (Heike Fiegler) (Dimitrios Kalaitzopoulos) Diane Rigler Team 16 Stephen Montgomery Manolis Dermitzakis Microarray Facility Peter Ellis Cordelia Langford The CNV project Nigel Carter Matthew Hurles Chris Tyler-Smith (Dan Andrews) Chris Barnes Don Conrad Tomas Fitzgerald Ni Huang (Lyndal Montgomery) Nelo Onyiah (Armand Valsesia) Klaudia Walter Fengtang Yujun Zhang Merci ! Arizona State University PJ Perry A Stone
e5ea5658a75cec5c45782573d852b51d.ppt