L_2[1].ppt
- Количество слайдов: 73
 Тема: Вирівнювання послідовностей
Тема: Вирівнювання послідовностей
 План: n Типи вирівнювань послідовностей; n Методи вирівнювання; n Алгоритми розпізнавання доменів у білкових структурах.
План: n Типи вирівнювань послідовностей; n Методи вирівнювання; n Алгоритми розпізнавання доменів у білкових структурах.
 Література: n Игнасимуту С. Основы биоинформатики. – М. – Ижевск: НИЦ «Регулярная и хаотическая динамика» , Институт компьютерных исследований, 2007. – 320 с. n Гриценко В. І. , Котова А. Б. , Вовк М. І. та ін. Інформаційні технології в біології та медицині. Навчальний посібник. – Київ: Наукова думка, 2007. – 381 с. n David W. Moumt. «Bioinformatics» , 2006. – 565 p.
Література: n Игнасимуту С. Основы биоинформатики. – М. – Ижевск: НИЦ «Регулярная и хаотическая динамика» , Институт компьютерных исследований, 2007. – 320 с. n Гриценко В. І. , Котова А. Б. , Вовк М. І. та ін. Інформаційні технології в біології та медицині. Навчальний посібник. – Київ: Наукова думка, 2007. – 381 с. n David W. Moumt. «Bioinformatics» , 2006. – 565 p.
 Інформаційні ресурси: n http: //www. bioinformatix. ru n http: //www. matbio. org n http: //bioinformatics. ru
Інформаційні ресурси: n http: //www. bioinformatix. ru n http: //www. matbio. org n http: //bioinformatics. ru
 Попарне вирівнювання augaguucucuaaagcuccagaagaggcucgcagccuccgugcugcgaugcggcaagaag aaggucugguuggaucccaaugaaaucaacgagaucgcuaacacaaacucgcgucagaac Множинне вирівнювання
Попарне вирівнювання augaguucucuaaagcuccagaagaggcucgcagccuccgugcugcgaugcggcaagaag aaggucugguuggaucccaaugaaaucaacgagaucgcuaacacaaacucgcgucagaac Множинне вирівнювання
 Поділ амінокислот Category Amino Acid Кислотиаміди Asp (D) Glu(E) Asn (N) Gln (Q) Основи His (H) Lys (K) Arg (R) Ароматичні Phe (F) Tyr (Y) Trp (W) Гідрофільні Ala (A) Cys (C) Gly (G) Pro (P) Ser (S) Thr (T) Гідрофобні Ile (I) Leu (L) Met (M) Val (V)
Поділ амінокислот Category Amino Acid Кислотиаміди Asp (D) Glu(E) Asn (N) Gln (Q) Основи His (H) Lys (K) Arg (R) Ароматичні Phe (F) Tyr (Y) Trp (W) Гідрофільні Ala (A) Cys (C) Gly (G) Pro (P) Ser (S) Thr (T) Гідрофобні Ile (I) Leu (L) Met (M) Val (V)
 Типи вирівнювань: n Глобальне n Локальное
Типи вирівнювань: n Глобальне n Локальное
 Попарне вирівнювання – вставка проміжків AKWTNLK----WAKV-ADVAGH-G AK-TNVKAKLPWGKVGAHVAGEYG - вставкавидалення проміжка - видовження проміжка
Попарне вирівнювання – вставка проміжків AKWTNLK----WAKV-ADVAGH-G AK-TNVKAKLPWGKVGAHVAGEYG - вставкавидалення проміжка - видовження проміжка
 Для аналізу порівняння обраховують: n Відсоток ідентичності; n Відсоток схожості.
Для аналізу порівняння обраховують: n Відсоток ідентичності; n Відсоток схожості.
 Попарне вирівнювання Людський гемоглобін (HH): VLSPADKTNVKAAWGKVGAHAGYEG Міоглобін кашалота (SWM): VLSEGEWQLVLHVWAKVEADVAGHG
Попарне вирівнювання Людський гемоглобін (HH): VLSPADKTNVKAAWGKVGAHAGYEG Міоглобін кашалота (SWM): VLSEGEWQLVLHVWAKVEADVAGHG
 Попарне вирівнювання - ідентичність (HH) VLSPADKTNVKAAWGKVGAHAGYEG ||| | | (SWM) VLSEGEWQLVLHVWAKVEADVAGHG Відсоток ідентичності: 36 %
Попарне вирівнювання - ідентичність (HH) VLSPADKTNVKAAWGKVGAHAGYEG ||| | | (SWM) VLSEGEWQLVLHVWAKVEADVAGHG Відсоток ідентичності: 36 %
 Попарне вирівнювання - схожість (HH) VLSPADKTNVKAAWGKVGAHAGYEG |||. | | | (SWM) VLSEGEWQLVLHVWAKVEADVAGHG Відсоток схожості: 40 % Відсоток ідентичності: 36 %
Попарне вирівнювання - схожість (HH) VLSPADKTNVKAAWGKVGAHAGYEG |||. | | | (SWM) VLSEGEWQLVLHVWAKVEADVAGHG Відсоток схожості: 40 % Відсоток ідентичності: 36 %
 Попарне вирівнювання – вставка проміжків (gaps) (HH) VLSPADKTNVKAAWGKVGAH-AGYEG . (SWM) VLSEGEWQLVLHVWAKVEADVAGH-G n Gaps: 2 n Відсоток схожості: 56 n Відсоток ідентичності: 44
Попарне вирівнювання – вставка проміжків (gaps) (HH) VLSPADKTNVKAAWGKVGAH-AGYEG . (SWM) VLSEGEWQLVLHVWAKVEADVAGH-G n Gaps: 2 n Відсоток схожості: 56 n Відсоток ідентичності: 44
 Попарне вирівнювання - підрахунок Фінальна оцінка вирівнювання – це сума сум позитивних балів і штрафних балів: + Кількість ідентичних + Кількість схожих - Кількість вставлених проміжків - Кількість видовжених проміжків Оцінка вирівнювання
Попарне вирівнювання - підрахунок Фінальна оцінка вирівнювання – це сума сум позитивних балів і штрафних балів: + Кількість ідентичних + Кількість схожих - Кількість вставлених проміжків - Кількість видовжених проміжків Оцінка вирівнювання
 Які задачі розв'язує вирівнювання? n Нуклеотиди n Вивчення еволюційних зв'язків. n Пошук генів, доменів, сигналів … n Білки n Вивчення еволюційних зв'язків. n Класифікація білкових сімейств за функцією або структурою. n Ідентифікація загальних доменів за функцією або структурою.
Які задачі розв'язує вирівнювання? n Нуклеотиди n Вивчення еволюційних зв'язків. n Пошук генів, доменів, сигналів … n Білки n Вивчення еволюційних зв'язків. n Класифікація білкових сімейств за функцією або структурою. n Ідентифікація загальних доменів за функцією або структурою.
 Що зображено? Номер стовпця вирівнювання Назва послідовності Концервативний залишок Функціонально концервативна позиція Номер останнього в ряді залишка З ЦІЄЇ ПОСЛІДОВНОСТІ
Що зображено? Номер стовпця вирівнювання Назва послідовності Концервативний залишок Функціонально концервативна позиція Номер останнього в ряді залишка З ЦІЄЇ ПОСЛІДОВНОСТІ
 Види матриць: n Матриці ТМ відсотка очкових утацій, В ( т м PAM). n Матриці БЛОЗАМ (блокових замін).
Види матриць: n Матриці ТМ відсотка очкових утацій, В ( т м PAM). n Матриці БЛОЗАМ (блокових замін).
 ВТМ еволюційної дистанції (PAM)
ВТМ еволюційної дистанції (PAM)
 Матриця БЛОЗАМ.
Матриця БЛОЗАМ.
 Матриця БЛОЗАМ
Матриця БЛОЗАМ
 Методи для попарного вирівнювання: n Точковий графік; n Динамічне програмування; n Метод “слів”, або k-кортежів.
Методи для попарного вирівнювання: n Точковий графік; n Динамічне програмування; n Метод “слів”, або k-кортежів.
 Точковий графік
Точковий графік
 Точкова матриця (співпадіння між коротким і повним іменами)
Точкова матриця (співпадіння між коротким і повним іменами)
 Точкова матриця (співпадіння повторюваної послідовності з самою собою (ABRACADA BRACADABRA)
Точкова матриця (співпадіння повторюваної послідовності з самою собою (ABRACADA BRACADABRA)
 Точкова матриця (співпадіння паліндромних послідовностей)
Точкова матриця (співпадіння паліндромних послідовностей)
 Точкова матриця (напрям руху для вирівнювання послідовностей)
Точкова матриця (напрям руху для вирівнювання послідовностей)
 Динамічне програмування Послідовність Фібоначчі, числа Фібоначчі — числова послідовність задана рекурентним співвідношенням другого порядку
Динамічне програмування Послідовність Фібоначчі, числа Фібоначчі — числова послідовність задана рекурентним співвідношенням другого порядку
 Окремі операції редагування включають в себе: Заміна bj на ai відображається записом (ai, bj); Делеція ai з послідовності А відображається записом (ai, Ø); Делеція bj з послідовності В відображається записом (Ø, bj).
Окремі операції редагування включають в себе: Заміна bj на ai відображається записом (ai, bj); Делеція ai з послідовності А відображається записом (ai, Ø); Делеція bj з послідовності В відображається записом (Ø, bj).
 Функція ваги d: d(ai, bj) = ціна мутації у вирівнюванні, де позиція і послідовності А відповідає позиції j послідовності В, і мутація проводить заміну ai↔bj, d(ai, Ø) або d(Ø, bj) = ціна делеції або вставки.
Функція ваги d: d(ai, bj) = ціна мутації у вирівнюванні, де позиція і послідовності А відповідає позиції j послідовності В, і мутація проводить заміну ai↔bj, d(ai, Ø) або d(Ø, bj) = ціна делеції або вставки.
 Відстань мінімальної ваги між послідовностями А і В: D(A, B)=min∑d(x, y) A→B де х, у Є А+ і мінімум береться з усієї послідовності операції редагування, які перетворюють А і В в загальну послідовність.
Відстань мінімальної ваги між послідовностями А і В: D(A, B)=min∑d(x, y) A→B де х, у Є А+ і мінімум береться з усієї послідовності операції редагування, які перетворюють А і В в загальну послідовність.
 Значення D(i, j) відповідає перетворенню першопочаткових послідовностей Аi=а 1 а 2…аi і Вj=b 1 b 2… bj в загальну послідовність шляхом L операцій редагування Sk, k=1, …, L, які можуть бути розглянуті в порядку зростання позицій в рядках.
Значення D(i, j) відповідає перетворенню першопочаткових послідовностей Аi=а 1 а 2…аi і Вj=b 1 b 2… bj в загальну послідовність шляхом L операцій редагування Sk, k=1, …, L, які можуть бути розглянуті в порядку зростання позицій в рядках.
 Відміна останньої операції редагування: Утворена в результаті вкорочена послідовність операцій редагування Sk, k=1, …, L-1, являє собою послідовність операцій конвертування підрядка Аi і підрядка Вj в загальний результат.
Відміна останньої операції редагування: Утворена в результаті вкорочена послідовність операцій редагування Sk, k=1, …, L-1, являє собою послідовність операцій конвертування підрядка Аi і підрядка Вj в загальний результат.
 Відповідність між індивідуальними операціями редагування і кроками між суміжними комірками матриці: (i-1, j-1) → (i, j) відповідає заміні аi → bj. (i-1, j) → (i, j) відповідає делеції аi з А. (i, j-1) → (i, j) відповідає вставці bj в А в позицію i.
Відповідність між індивідуальними операціями редагування і кроками між суміжними комірками матриці: (i-1, j-1) → (i, j) відповідає заміні аi → bj. (i-1, j) → (i, j) відповідає делеції аi з А. (i, j-1) → (i, j) відповідає вставці bj в А в позицію i.
 Послідовності операцій редагування відповідають сходинчастому шляху по матриці (io, jo) = (0, 0) → (i 1, j 1) → … (n, m) де 0 ≤ ik+1 -ik ≤ 1, (для 0 ≤ k ≤ n-1), 0 ≤ jk+1 - jk ≤ 1, (для 0 ≤ k ≤ m -1).
Послідовності операцій редагування відповідають сходинчастому шляху по матриці (io, jo) = (0, 0) → (i 1, j 1) → … (n, m) де 0 ≤ ik+1 -ik ≤ 1, (для 0 ≤ k ≤ n-1), 0 ≤ jk+1 - jk ≤ 1, (для 0 ≤ k ≤ m -1).
 Отже, кінцевий алгоритм: Обрахувати матрицю D розміром (m+1)x(n+1), застосовуючи: 1) початкові умови на верхній ряд і ліву колонку і D(i, 0)=∑d(ak, Ø) k=0 j D(0, j)=∑d(Ø, bk) k=0
Отже, кінцевий алгоритм: Обрахувати матрицю D розміром (m+1)x(n+1), застосовуючи: 1) початкові умови на верхній ряд і ліву колонку і D(i, 0)=∑d(ak, Ø) k=0 j D(0, j)=∑d(Ø, bk) k=0
 2) рекурентні співвідношення: D(i, j)= min {D(i-1, j)+d(ai, Ø), D(i-1, j-1)+d(ai, bj), D(i, j-1)+d(Ø, bj)} де i=1, …, n; j=1, …, m.
2) рекурентні співвідношення: D(i, j)= min {D(i-1, j)+d(ai, Ø), D(i-1, j-1)+d(ai, bj), D(i, j-1)+d(Ø, bj)} де i=1, …, n; j=1, …, m.
 Це означає, що ми враховуємо всі три можливих кроки для D(i, j): Операція Загальна ціна Вставка пропуску в послідовність А D(i-1, j)+d(ai, Ø) Заміна ai↔bj D(i-1, j-1)+d(ai, bj) Вставка пропуску в послідовність В D(i, j-1)+d(Ø, bj)
Це означає, що ми враховуємо всі три можливих кроки для D(i, j): Операція Загальна ціна Вставка пропуску в послідовність А D(i-1, j)+d(ai, Ø) Заміна ai↔bj D(i-1, j-1)+d(ai, bj) Вставка пропуску в послідовність В D(i, j-1)+d(Ø, bj)
 Динамічне програмування для редакційної відстані (червоні стрілки відповідають вставкам і видаленням) di, j di+1, j di, j+1 di+1, j+1
Динамічне програмування для редакційної відстані (червоні стрілки відповідають вставкам і видаленням) di, j di+1, j di, j+1 di+1, j+1
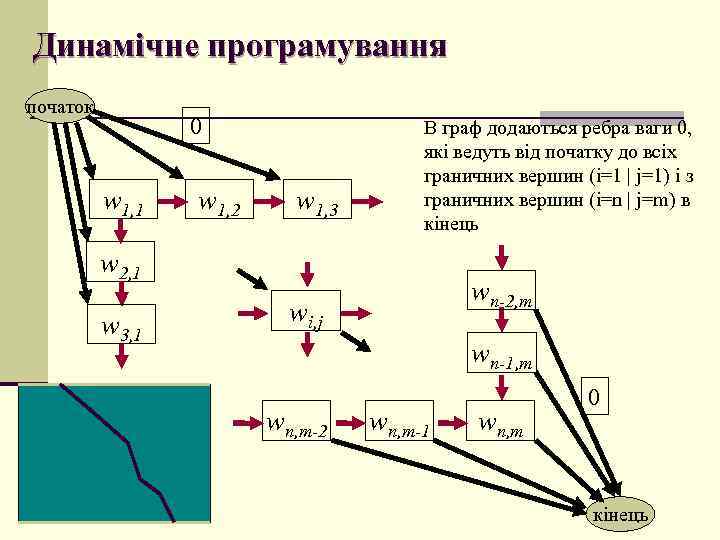 Динамічне програмування початок 0 w 1, 1 w 1, 2 w 1, 3 В граф додаються ребра ваги 0, які ведуть від початку до всіх граничних вершин (i=1 | j=1) і з граничних вершин (i=n | j=m) в кінець w 2, 1 w 3, 1 wn-2, m wi, j wn-1, m wn, m-2 wn, m-1 wn, m 0 кінець
Динамічне програмування початок 0 w 1, 1 w 1, 2 w 1, 3 В граф додаються ребра ваги 0, які ведуть від початку до всіх граничних вершин (i=1 | j=1) і з граничних вершин (i=n | j=m) в кінець w 2, 1 w 3, 1 wn-2, m wi, j wn-1, m wn, m-2 wn, m-1 wn, m 0 кінець
 Алгоритм Нідлмана-Вунша
Алгоритм Нідлмана-Вунша

 Шлях в графі:
Шлях в графі:
 Схематичне зображення співставлених структур 1 Білок 1 2 3 4 5 6 Сα атоми 3 2 1 6 7 10 7 8 11 8 12 9 Білок 2 Відповідність між Сα атомами двох співставлених структур, основане на близькості в просторі
Схематичне зображення співставлених структур 1 Білок 1 2 3 4 5 6 Сα атоми 3 2 1 6 7 10 7 8 11 8 12 9 Білок 2 Відповідність між Сα атомами двох співставлених структур, основане на близькості в просторі
 1 1 2 2 3 3 4 4 5 5 6 6 7 8 9 7 Стрілки як на попередньому 8 слайді 10 11 12 Вставка трьох залишків
1 1 2 2 3 3 4 4 5 5 6 6 7 8 9 7 Стрілки як на попередньому 8 слайді 10 11 12 Вставка трьох залишків
 Динамічне програмування (алгоритм Сміта-Уотермана)
Динамічне програмування (алгоритм Сміта-Уотермана)
 Алгоритм Сміта-Уотермана
Алгоритм Сміта-Уотермана

 Значимість вирівнювань: 1. Z-score=0 – схожість послідовностей випадкова; Z-score≥ 5 – наявна значимість вирівнювання. 2. P-value; E-value.
Значимість вирівнювань: 1. Z-score=0 – схожість послідовностей випадкова; Z-score≥ 5 – наявна значимість вирівнювання. 2. P-value; E-value.
 Значимість вирівнювань: P-value: P≤ 10 -100 – точне співпадіння; P між 10 -100 – 10 -50 – послідовності майже ідентичні (наприклад, алелі чи поліморфізми); P між 10 -50 – 10 -10 – близькоспоріднені послідовності (гомологія очевидна); P між 10 -5 – 10 -1 – зазвичай далекоспоріднені послідовності; P>10 -1 – відповідність незначна.
Значимість вирівнювань: P-value: P≤ 10 -100 – точне співпадіння; P між 10 -100 – 10 -50 – послідовності майже ідентичні (наприклад, алелі чи поліморфізми); P між 10 -50 – 10 -10 – близькоспоріднені послідовності (гомологія очевидна); P між 10 -5 – 10 -1 – зазвичай далекоспоріднені послідовності; P>10 -1 – відповідність незначна.
 Значимість вирівнювань: E-value B-перетворюючий показних; P-система підрахунку; K-розмір області пошуку; S-показник схожості послідовності. m- довжина досліджуваної послідовності; n-довжина послідовності з бази даних.
Значимість вирівнювань: E-value B-перетворюючий показних; P-система підрахунку; K-розмір області пошуку; S-показник схожості послідовності. m- довжина досліджуваної послідовності; n-довжина послідовності з бази даних.
 Значимість вирівнювань: Е-value: Е≤ 0, 02 – послідовності є гомологічними; Е між 0, 02 і 1 – гомологія не очевидна; Е>1 – випадкове співпадіння.
Значимість вирівнювань: Е-value: Е≤ 0, 02 – послідовності є гомологічними; Е між 0, 02 і 1 – гомологія не очевидна; Е>1 – випадкове співпадіння.
 Значимість вирівнювань: де х – вага, K і λ – параметри пов´язані з розташуванням максимуму і шириною розподілу.
Значимість вирівнювань: де х – вага, K і λ – параметри пов´язані з розташуванням максимуму і шириною розподілу.
 Алгоритм BLAST (Basic Local Alignment Search Tool) Потребує параметр W (довжина слова). Білки W = 3 n ДНК W = 11. n
Алгоритм BLAST (Basic Local Alignment Search Tool) Потребує параметр W (довжина слова). Білки W = 3 n ДНК W = 11. n
 Алгоритм BLAST (крок 1) 1. Пошук ідентичнихподібних ділянок 2. Спроба «видовжити» ці ділянки наскільки можливо (тобто поки score росте) В результаті: High-scoring Segment Pairs (HSPs) THEFIRSTLINIHAVEADREAMESIRPATRICKREAD INVIEIAMDEADMEATTNAMHEWASNINETEEN
Алгоритм BLAST (крок 1) 1. Пошук ідентичнихподібних ділянок 2. Спроба «видовжити» ці ділянки наскільки можливо (тобто поки score росте) В результаті: High-scoring Segment Pairs (HSPs) THEFIRSTLINIHAVEADREAMESIRPATRICKREAD INVIEIAMDEADMEATTNAMHEWASNINETEEN
 Алгоритм BLAST (крок 2) Спроба зєднати сусідні HSPs шляхом вирівнювання послідовностей між ними: THEFIRSTLINIHAVEADREA____M_ESIRPATRICKREAD INVIEIAMDEADMEATTNAMHEW___ASNINETEEN
Алгоритм BLAST (крок 2) Спроба зєднати сусідні HSPs шляхом вирівнювання послідовностей між ними: THEFIRSTLINIHAVEADREA____M_ESIRPATRICKREAD INVIEIAMDEADMEATTNAMHEW___ASNINETEEN
 Методи для множинного вирівнювання: n Профілі; n Блоки; n Індикатори; n Закриті марківські моделі.
Методи для множинного вирівнювання: n Профілі; n Блоки; n Індикатори; n Закриті марківські моделі.
 Множинне вирівнювання Колір Тип залишку Амінокислоти жовтий Маленькі неполярні залишки Gly, Ala, Ser, Thr зелений Гідрофобні Cys, Val, Ile, Leu, Pro, Phe, Tyr, Met, Trp фіолетовий Полярні Asn, Gln, His червоний Негативно заряджені Asp, Glu синій Позитивно заряджені Lys, Arg
Множинне вирівнювання Колір Тип залишку Амінокислоти жовтий Маленькі неполярні залишки Gly, Ala, Ser, Thr зелений Гідрофобні Cys, Val, Ile, Leu, Pro, Phe, Tyr, Met, Trp фіолетовий Полярні Asn, Gln, His червоний Негативно заряджені Asp, Glu синій Позитивно заряджені Lys, Arg
 Множинне вирівнювання (тиоредоксин у E. Coli та інших гомологів)
Множинне вирівнювання (тиоредоксин у E. Coli та інших гомологів)
 Профіль (Profile) Профіль містить інформацію про символи в кожному стовпці вирівнювання. A T C T T G T A A C T T C T 1 2 3 4 5 6 A 1 0. 67 0 0 . . T 0 0. 33 1 1 . . C 0 0 . . G 0 0 . .
Профіль (Profile) Профіль містить інформацію про символи в кожному стовпці вирівнювання. A T C T T G T A A C T T C T 1 2 3 4 5 6 A 1 0. 67 0 0 . . T 0 0. 33 1 1 . . C 0 0 . . G 0 0 . .
 Profile Різний профіль A A C T T G C A A C T T G T A A G T C G T A A C T T G T C A C T T C T A A C T T C T 1 2 3 4 5 6 A 0. 66 1 0 0 . . A 1 1 0 0 . . T 0 0 0 1 . . C 0. 33 0 0. 66 0 . . C 0 0 1 0 . . G 0 0 0. 33 0 . . G 0 0 . .
Profile Різний профіль A A C T T G C A A C T T G T A A G T C G T A A C T T G T C A C T T C T A A C T T C T 1 2 3 4 5 6 A 0. 66 1 0 0 . . A 1 1 0 0 . . T 0 0 0 1 . . C 0. 33 0 0. 66 0 . . C 0 0 1 0 . . G 0 0 0. 33 0 . . G 0 0 . .
 n Ітерація
n Ітерація
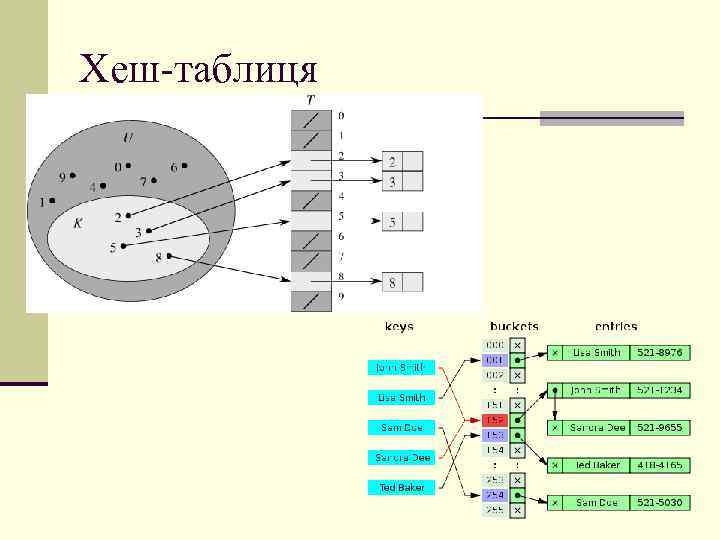 Хеш-таблиця
Хеш-таблиця
 Приховані марківські моделі HMM (Hidden Markov Models) Рис. 1. Структура Прихованої марківської моделі. Кожній позиції у множинному вирівнюванні HMM відповідають стану співставлення (m) і делеції (d). Стани вставки (і) з’являються між позиціями залишків, на початку і в кінці.
Приховані марківські моделі HMM (Hidden Markov Models) Рис. 1. Структура Прихованої марківської моделі. Кожній позиції у множинному вирівнюванні HMM відповідають стану співставлення (m) і делеції (d). Стани вставки (і) з’являються між позиціями залишків, на початку і в кінці.
 Дві дослідницькі групи, що спеціалізуються на біологічному застосуванні НММ, мають Web-сервери і пропонують свої програми:
Дві дослідницькі групи, що спеціалізуються на біологічному застосуванні НММ, мають Web-сервери і пропонують свої програми:
 Алгоритми на основі компактності доменів: n САРОБ (Синтаксичний аналізатор розгорнутих одиниць білка) – намагається максимізувати взаємодію в межах кожної структурної одиниці (домену) і при цьому мінімізувати взаємодію між самими одиницями. n Домак – багатократно розділяє білок на дві довільні частини і обраховує “величину розщеплення” за кількістю контактів при кожному довільному розділенні. n Детектив – основана на допущенні про те, що кожний домен повинен містити гідрофобне ядро.
Алгоритми на основі компактності доменів: n САРОБ (Синтаксичний аналізатор розгорнутих одиниць білка) – намагається максимізувати взаємодію в межах кожної структурної одиниці (домену) і при цьому мінімізувати взаємодію між самими одиницями. n Домак – багатократно розділяє білок на дві довільні частини і обраховує “величину розщеплення” за кількістю контактів при кожному довільному розділенні. n Детектив – основана на допущенні про те, що кожний домен повинен містити гідрофобне ядро.
 Алгоритм “Струдл” Використовує графову евристику Кернігана-Ліня і розбиває білок на групи залишків, які показують мінімальну міжгрупову взаємодію. Граф визначає зв'язки між вузлами і представлений матрицею. Починаючи з відповідного розбиття, “Струдл” мінімізує функцію вартості, яка залежить від взаємодій між вузлами. Для цього алгоритм виконує перестановку пар вузлів до тих пір, поки не буде отримано оптимального розбиття. Міжвузлові взаємодії визначаються за вирівняною діаграмою Вороного.
Алгоритм “Струдл” Використовує графову евристику Кернігана-Ліня і розбиває білок на групи залишків, які показують мінімальну міжгрупову взаємодію. Граф визначає зв'язки між вузлами і представлений матрицею. Починаючи з відповідного розбиття, “Струдл” мінімізує функцію вартості, яка залежить від взаємодій між вузлами. Для цього алгоритм виконує перестановку пар вузлів до тих пір, поки не буде отримано оптимального розбиття. Міжвузлові взаємодії визначаються за вирівняною діаграмою Вороного.
 ДЯКУЮ ЗА УВАГУ!!!
ДЯКУЮ ЗА УВАГУ!!!
 Динамічне програмування
Динамічне програмування




 Blast n Blast – це сімейство програм: Blast. N, Blast. P, Blast. X, t. Blast. N Query: Белок Database: n n ДНК Белок Blast. N - ДНК vs ДНК Blast. P – білок vs білок Blast. X - translated ДНК vs білок t. Blast. N - білок vs translated ДНК
Blast n Blast – це сімейство програм: Blast. N, Blast. P, Blast. X, t. Blast. N Query: Белок Database: n n ДНК Белок Blast. N - ДНК vs ДНК Blast. P – білок vs білок Blast. X - translated ДНК vs білок t. Blast. N - білок vs translated ДНК


