lecture 4.ppt
- Количество слайдов: 79
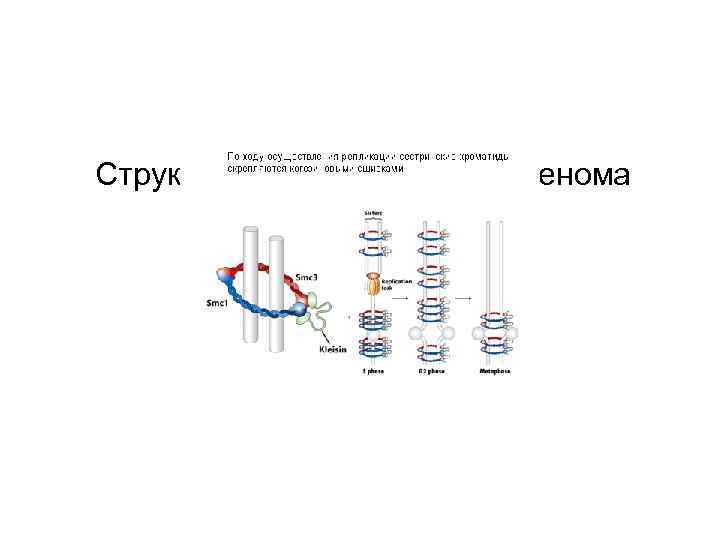 Структура эукариотического генома
Структура эукариотического генома
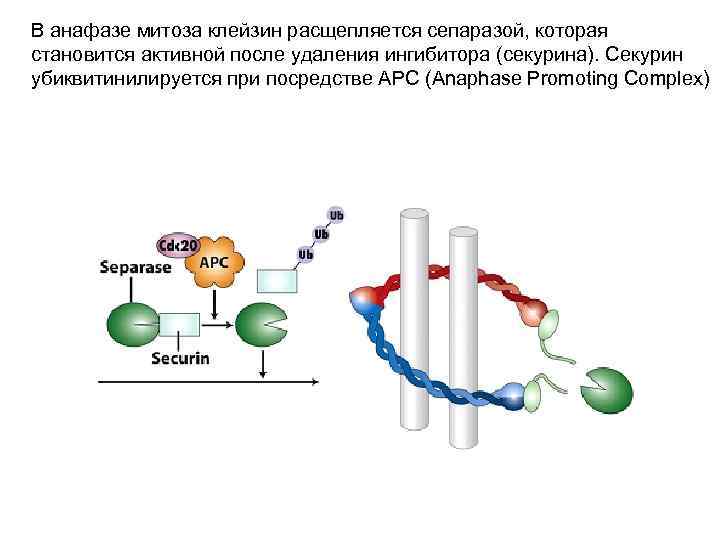 В анафазе митоза клейзин расщепляется сепаразой, которая становится активной после удаления ингибитора (секурина). Секурин убиквитинилируется при посредстве APC (Anaphase Promoting Complex)
В анафазе митоза клейзин расщепляется сепаразой, которая становится активной после удаления ингибитора (секурина). Секурин убиквитинилируется при посредстве APC (Anaphase Promoting Complex)
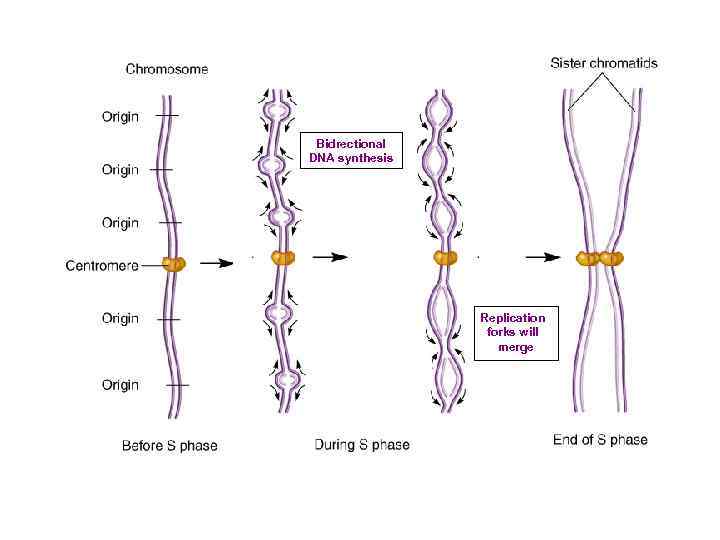 Bidrectional DNA synthesis Replication forks will merge
Bidrectional DNA synthesis Replication forks will merge
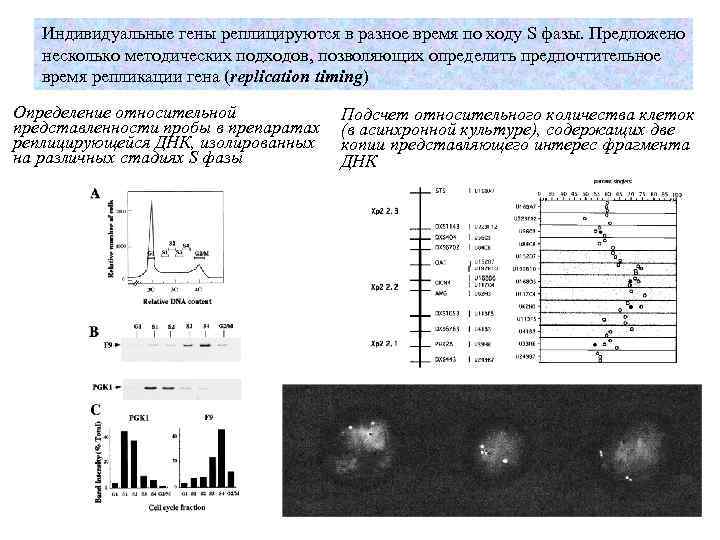 Индивидуальные гены реплицируются в разное время по ходу S фазы. Предложено несколько методических подходов, позволяющих определить предпочтительное время репликации гена (replication timing) Определение относительной представленности пробы в препаратах реплицирующейся ДНК, изолированных на различных стадиях S фазы Подсчет относительного количества клеток (в асинхронной культуре), содержащих две копии представляющего интерес фрагмента ДНК
Индивидуальные гены реплицируются в разное время по ходу S фазы. Предложено несколько методических подходов, позволяющих определить предпочтительное время репликации гена (replication timing) Определение относительной представленности пробы в препаратах реплицирующейся ДНК, изолированных на различных стадиях S фазы Подсчет относительного количества клеток (в асинхронной культуре), содержащих две копии представляющего интерес фрагмента ДНК
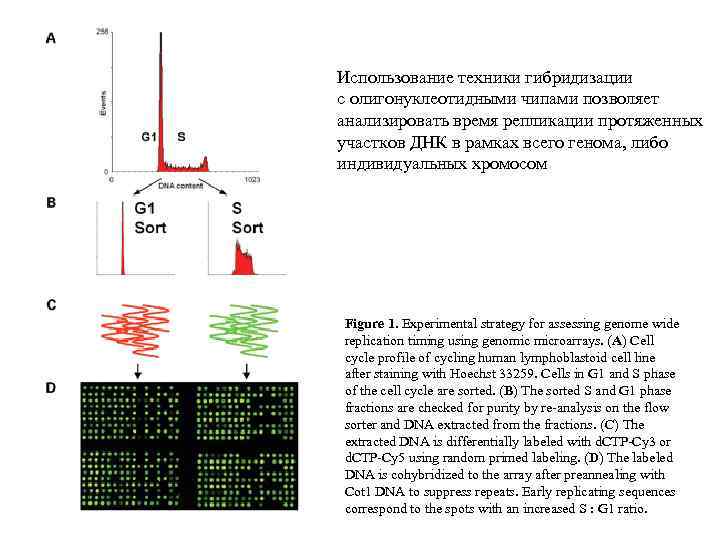 Использование техники гибридизации с олигонуклеотидными чипами позволяет анализировать время репликации протяженных участков ДНК в рамках всего генома, либо индивидуальных хромосом Figure 1. Experimental strategy for assessing genome wide replication timing using genomic microarrays. (A) Cell cycle profile of cycling human lymphoblastoid cell line after staining with Hoechst 33259. Cells in G 1 and S phase of the cell cycle are sorted. (B) The sorted S and G 1 phase fractions are checked for purity by re-analysis on the flow sorter and DNA extracted from the fractions. (C) The extracted DNA is differentially labeled with d. CTP-Cy 3 or d. CTP-Cy 5 using random primed labeling. (D) The labeled DNA is cohybridized to the array after preannealing with Cot 1 DNA to suppress repeats. Early replicating sequences correspond to the spots with an increased S : G 1 ratio.
Использование техники гибридизации с олигонуклеотидными чипами позволяет анализировать время репликации протяженных участков ДНК в рамках всего генома, либо индивидуальных хромосом Figure 1. Experimental strategy for assessing genome wide replication timing using genomic microarrays. (A) Cell cycle profile of cycling human lymphoblastoid cell line after staining with Hoechst 33259. Cells in G 1 and S phase of the cell cycle are sorted. (B) The sorted S and G 1 phase fractions are checked for purity by re-analysis on the flow sorter and DNA extracted from the fractions. (C) The extracted DNA is differentially labeled with d. CTP-Cy 3 or d. CTP-Cy 5 using random primed labeling. (D) The labeled DNA is cohybridized to the array after preannealing with Cot 1 DNA to suppress repeats. Early replicating sequences correspond to the spots with an increased S : G 1 ratio.
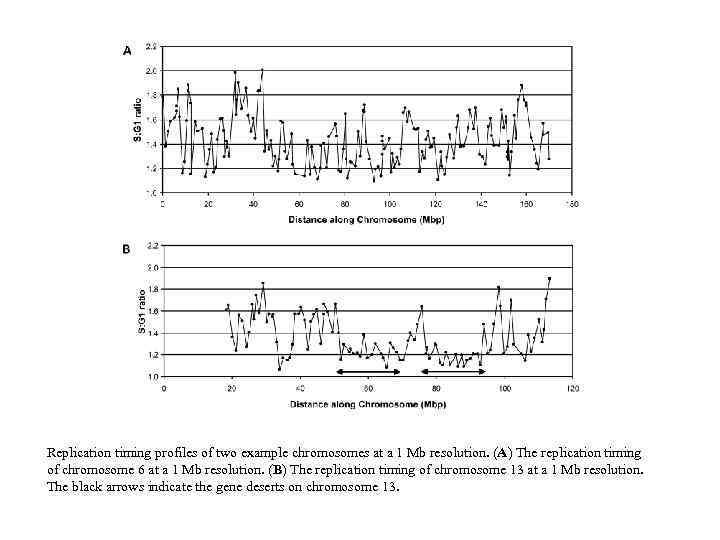 Replication timing profiles of two example chromosomes at a 1 Mb resolution. (A) The replication timing of chromosome 6 at a 1 Mb resolution. (B) The replication timing of chromosome 13 at a 1 Mb resolution. The black arrows indicate the gene deserts on chromosome 13.
Replication timing profiles of two example chromosomes at a 1 Mb resolution. (A) The replication timing of chromosome 6 at a 1 Mb resolution. (B) The replication timing of chromosome 13 at a 1 Mb resolution. The black arrows indicate the gene deserts on chromosome 13.
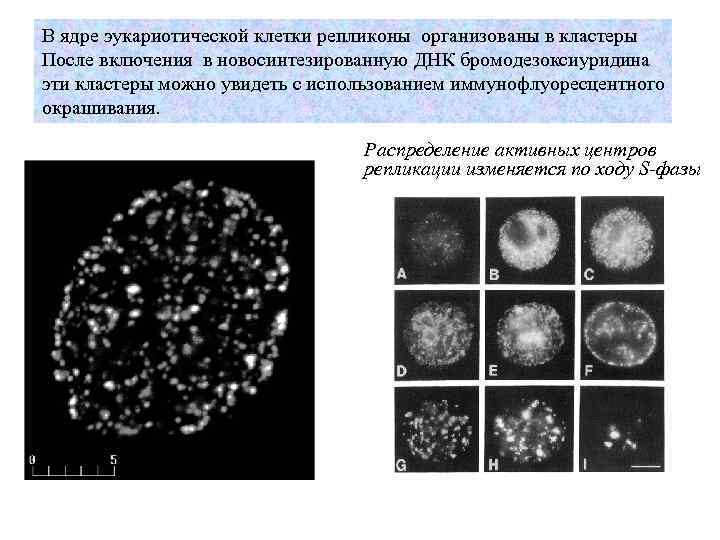 В ядре эукариотической клетки репликоны организованы в кластеры После включения в новосинтезированную ДНК бромодезоксиуридина эти кластеры можно увидеть с использованием иммунофлуоресцентного окрашивания. Распределение активных центров репликации изменяется по ходу S-фазы
В ядре эукариотической клетки репликоны организованы в кластеры После включения в новосинтезированную ДНК бромодезоксиуридина эти кластеры можно увидеть с использованием иммунофлуоресцентного окрашивания. Распределение активных центров репликации изменяется по ходу S-фазы
 В начале S-фазы в клетках присутствует несколько сотен фокусов репликации
В начале S-фазы в клетках присутствует несколько сотен фокусов репликации
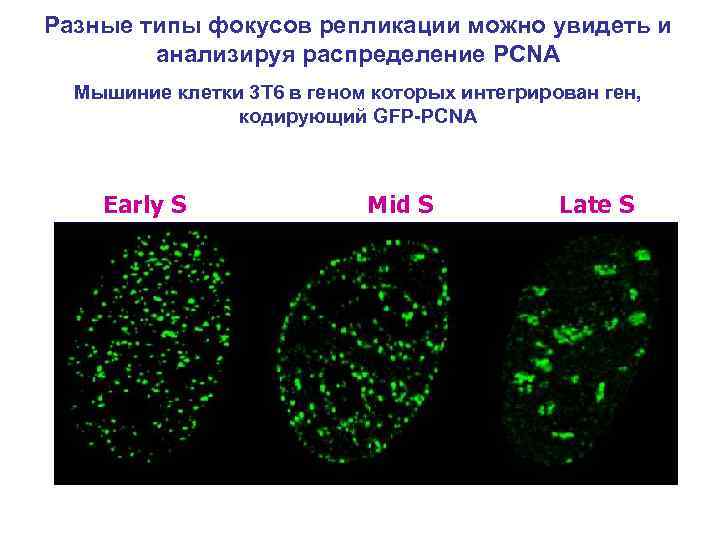 Разные типы фокусов репликации можно увидеть и анализируя распределение PCNA Мышиние клетки 3 Т 6 в геном которых интегрирован ген, кодирующий GFP-PCNA Early S Mid S Late S
Разные типы фокусов репликации можно увидеть и анализируя распределение PCNA Мышиние клетки 3 Т 6 в геном которых интегрирован ген, кодирующий GFP-PCNA Early S Mid S Late S
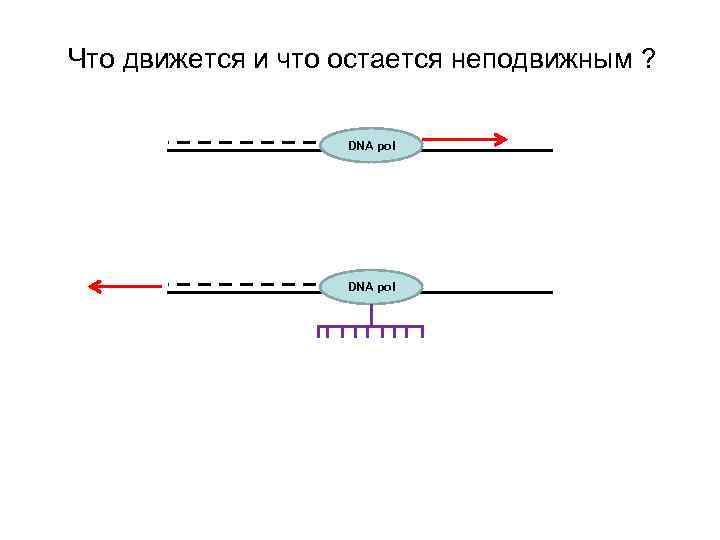 Что движется и что остается неподвижным ? DNA pol
Что движется и что остается неподвижным ? DNA pol
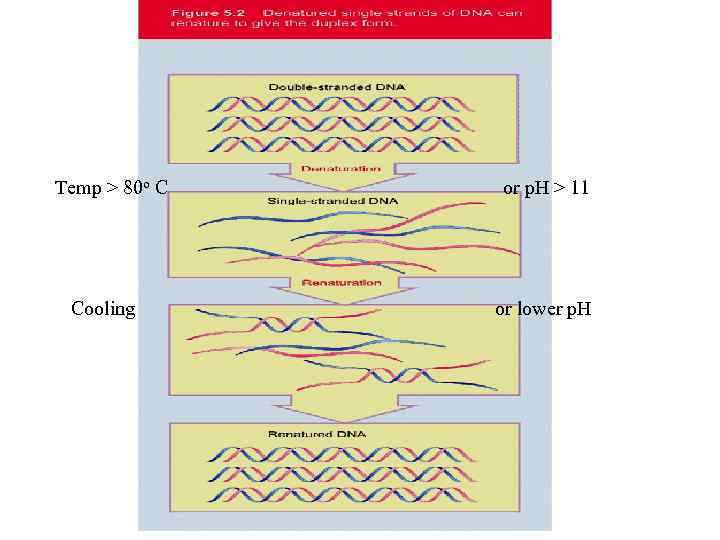 Temp > 80 o C Cooling or p. H > 11 or lower p. H
Temp > 80 o C Cooling or p. H > 11 or lower p. H
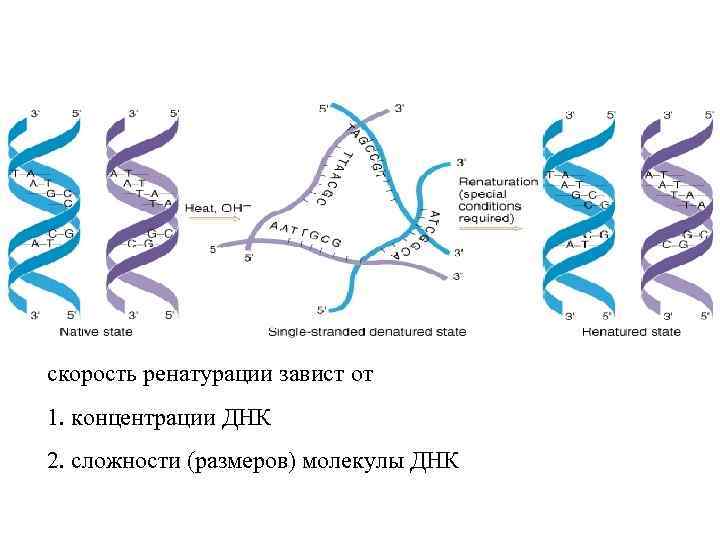 скорость ренатурации завист от 1. концентрации ДНК 2. сложности (размеров) молекулы ДНК
скорость ренатурации завист от 1. концентрации ДНК 2. сложности (размеров) молекулы ДНК
 Критическими параметрами, определяющими полноту ренатурации являются концентрация ДНК и время инкубации (C T) 0 При изучении кинетики ренатурации ДНК дробят до фрагментов размером 100 -200 пн 1 2 Если взять в реакцию одинаковое количество ДНК, то фактическая концентрация «красного» фрагмента в препарате 2 будет в 10 раз меньше, чем в препарате 1. Это позволяет использовать анализ кинетики ренатурации для оценки размеров ДНК
Критическими параметрами, определяющими полноту ренатурации являются концентрация ДНК и время инкубации (C T) 0 При изучении кинетики ренатурации ДНК дробят до фрагментов размером 100 -200 пн 1 2 Если взять в реакцию одинаковое количество ДНК, то фактическая концентрация «красного» фрагмента в препарате 2 будет в 10 раз меньше, чем в препарате 1. Это позволяет использовать анализ кинетики ренатурации для оценки размеров ДНК
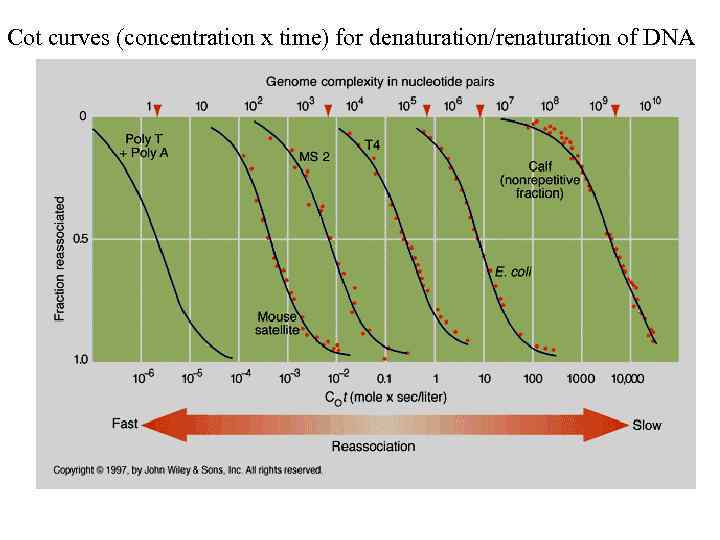 Cot curves (concentration x time) for denaturation/renaturation of DNA
Cot curves (concentration x time) for denaturation/renaturation of DNA
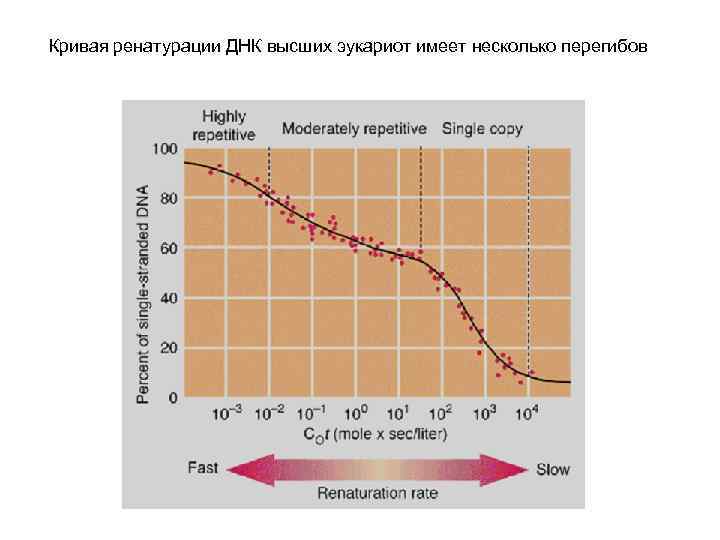 Кривая ренатурации ДНК высших эукариот имеет несколько перегибов
Кривая ренатурации ДНК высших эукариот имеет несколько перегибов
 Наличие перегибов в кривой ренатурации ДНК высших эукариот свидетельствует о том, что она состоит из уникальных и повторяющихся последовательностей На основании степени повторенности выделяют три основных класса последовательностей: 1. Уникальные последовательности: 1 - 10 копий на гаплоидный геном (50 -60% генома млекопитающих) 2. умеренно повторяющиеся последовательности: 10 - 105 копий на гаплоидный геном (25 -40% генома млекопитающих) 3. Высокоповторяющиеся последовательности: более 105 копий на гаплоидный геном (10 -15% генома млекопитающих)
Наличие перегибов в кривой ренатурации ДНК высших эукариот свидетельствует о том, что она состоит из уникальных и повторяющихся последовательностей На основании степени повторенности выделяют три основных класса последовательностей: 1. Уникальные последовательности: 1 - 10 копий на гаплоидный геном (50 -60% генома млекопитающих) 2. умеренно повторяющиеся последовательности: 10 - 105 копий на гаплоидный геном (25 -40% генома млекопитающих) 3. Высокоповторяющиеся последовательности: более 105 копий на гаплоидный геном (10 -15% генома млекопитающих)
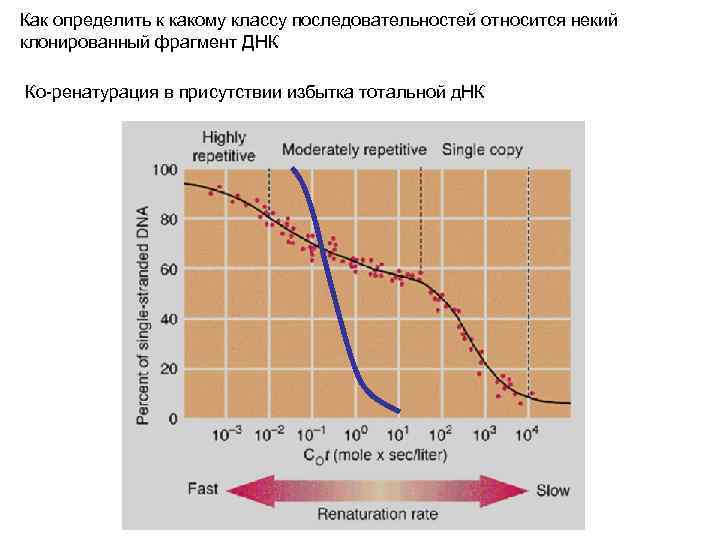 Как определить к какому классу последовательностей относится некий клонированный фрагмент ДНК Ко-ренатурация в присутствии избытка тотальной д. НК
Как определить к какому классу последовательностей относится некий клонированный фрагмент ДНК Ко-ренатурация в присутствии избытка тотальной д. НК
 Пять классов повторяющихся последовательностей 1. Перемежающиеся повторы (Interspersed repeats) 2. Процессированные псевдогены 3. Повторы, состоящие из простых последовательностей (минисателлиты и микросателлиты) 4. Сегментные дупликации 5. Блоки тандемных повторов (сателлиты)
Пять классов повторяющихся последовательностей 1. Перемежающиеся повторы (Interspersed repeats) 2. Процессированные псевдогены 3. Повторы, состоящие из простых последовательностей (минисателлиты и микросателлиты) 4. Сегментные дупликации 5. Блоки тандемных повторов (сателлиты)
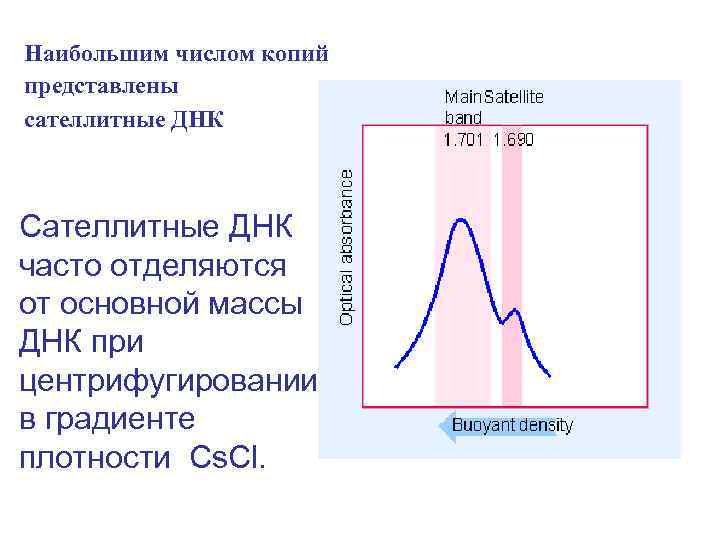 Наибольшим числом копий представлены сателлитные ДНК Сателлитные ДНК часто отделяются от основной массы ДНК при центрифугировании в градиенте плотности Cs. Cl.
Наибольшим числом копий представлены сателлитные ДНК Сателлитные ДНК часто отделяются от основной массы ДНК при центрифугировании в градиенте плотности Cs. Cl.
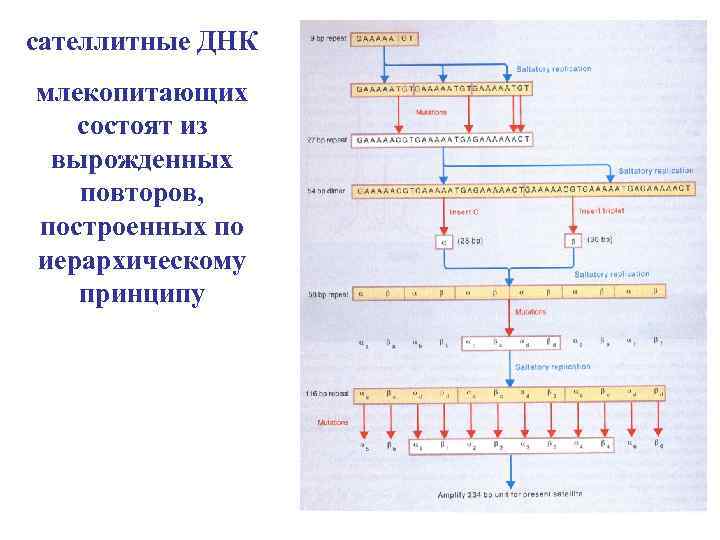 сателлитные ДНК млекопитающих состоят из вырожденных повторов, построенных по иерархическому принципу
сателлитные ДНК млекопитающих состоят из вырожденных повторов, построенных по иерархическому принципу
 Сателлитные ДНК – Размер области, содержащей повторяющиеся блоки составляет от 100 Kb до 1 Mb и более. – Альфоидный сателлит располагается в центромерных участках всех хромосом человека. Размер повторяющейся единицы 171 bp. – В ДНК каждой из хромосм человека альфоидный сателлит составляет 3 -5%
Сателлитные ДНК – Размер области, содержащей повторяющиеся блоки составляет от 100 Kb до 1 Mb и более. – Альфоидный сателлит располагается в центромерных участках всех хромосом человека. Размер повторяющейся единицы 171 bp. – В ДНК каждой из хромосм человека альфоидный сателлит составляет 3 -5%
 МИНИСАТЕЛЛИТЫ • Протяженность варьирует от 1 kb to 20 kb. • Размер повторяющейся единицы от ~10 до 500 bp. • Количество повторов может варьировать у разных особей (полиморфизм на основании которого возможено осуществление ДНК фингерпринтинга. • Теломерные последовательности также относят к числу минисателлитов (у человека протяженность ~ 15 kb, повторяющаяся единица GGGTTA.
МИНИСАТЕЛЛИТЫ • Протяженность варьирует от 1 kb to 20 kb. • Размер повторяющейся единицы от ~10 до 500 bp. • Количество повторов может варьировать у разных особей (полиморфизм на основании которого возможено осуществление ДНК фингерпринтинга. • Теломерные последовательности также относят к числу минисателлитов (у человека протяженность ~ 15 kb, повторяющаяся единица GGGTTA.
 Микросателлиты также называют короткими тандемными повторами (short tandem repeats (STR)) Длина повторяющейся единицы 2 -8 bp, протяженность блока повторяющихся единиц не более 150 bp. Протяженность блоков различных микросателлитов варьирует у разных особей (основа для ДНК фингерпринтинга) Примеры микросателлитов: (A)n, (CGG)n Из динуклеотидных повторов чаще всего встречаются AC, AT, AG Экспансия микросателлитов может быть причиной возникновения различных заболеваний, связанных с нарушением функций генов
Микросателлиты также называют короткими тандемными повторами (short tandem repeats (STR)) Длина повторяющейся единицы 2 -8 bp, протяженность блока повторяющихся единиц не более 150 bp. Протяженность блоков различных микросателлитов варьирует у разных особей (основа для ДНК фингерпринтинга) Примеры микросателлитов: (A)n, (CGG)n Из динуклеотидных повторов чаще всего встречаются AC, AT, AG Экспансия микросателлитов может быть причиной возникновения различных заболеваний, связанных с нарушением функций генов
 Сегментные дупликации Блоки от 1 до 300 Kb (чаще 10 -50 Kb), которые дуплицированы на одной хромосоме или на разных хромосомах. Могут иметь до 99% сходства по нуклеотидным последовательностям По некоторым оценкам у человека сегментные дупликации составляют ~3, 5 -5% от гаплоидного генома
Сегментные дупликации Блоки от 1 до 300 Kb (чаще 10 -50 Kb), которые дуплицированы на одной хромосоме или на разных хромосомах. Могут иметь до 99% сходства по нуклеотидным последовательностям По некоторым оценкам у человека сегментные дупликации составляют ~3, 5 -5% от гаплоидного генома
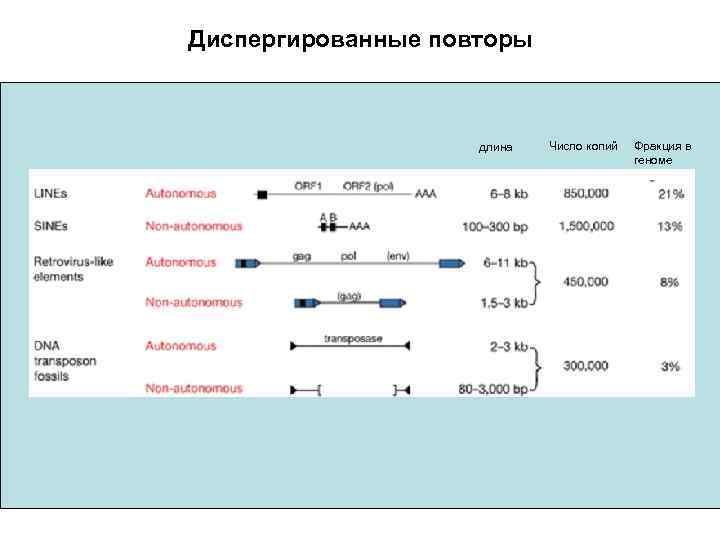 Диспергированные повторы ~45% генома человека. Сюда входят: длина Число копий Ретроэлементы различных типов Long-terminal repeat transposons (LTRs; RNA-mediated) Long interspersed elements (LINEs); Short interspersed elements (SINEs) ДНК транспозоны (~3% от генома человека) Фракция в геноме
Диспергированные повторы ~45% генома человека. Сюда входят: длина Число копий Ретроэлементы различных типов Long-terminal repeat transposons (LTRs; RNA-mediated) Long interspersed elements (LINEs); Short interspersed elements (SINEs) ДНК транспозоны (~3% от генома человека) Фракция в геноме
 • В геноме человека есть несколько семейств LINE элементов; наиболее представлено семейство L 1. В геноме человека присутствует от 60, 000 to 100, 000 L 1 элементов. ORF 1 кодирует белок p 40, функции которого не известны. ORF 2 кодирует обратную транскриптазу, которая необходима для перемещения мобильных элементов. Красным цветом обозначены прямые повторы, которые есть у всех мобильных элементов.
• В геноме человека есть несколько семейств LINE элементов; наиболее представлено семейство L 1. В геноме человека присутствует от 60, 000 to 100, 000 L 1 элементов. ORF 1 кодирует белок p 40, функции которого не известны. ORF 2 кодирует обратную транскриптазу, которая необходима для перемещения мобильных элементов. Красным цветом обозначены прямые повторы, которые есть у всех мобильных элементов.
 SINEs Short Interspersed Nuclear Elements Длина ~ 300 bp Не содержат функциональных генов (LINE элементы с делетированной внутренней частью) У человека наиболее известо семейство Alu элементов (от 700. 000 до 1. 000 Alu элементов на гаплоидный геном)
SINEs Short Interspersed Nuclear Elements Длина ~ 300 bp Не содержат функциональных генов (LINE элементы с делетированной внутренней частью) У человека наиболее известо семейство Alu элементов (от 700. 000 до 1. 000 Alu элементов на гаплоидный геном)
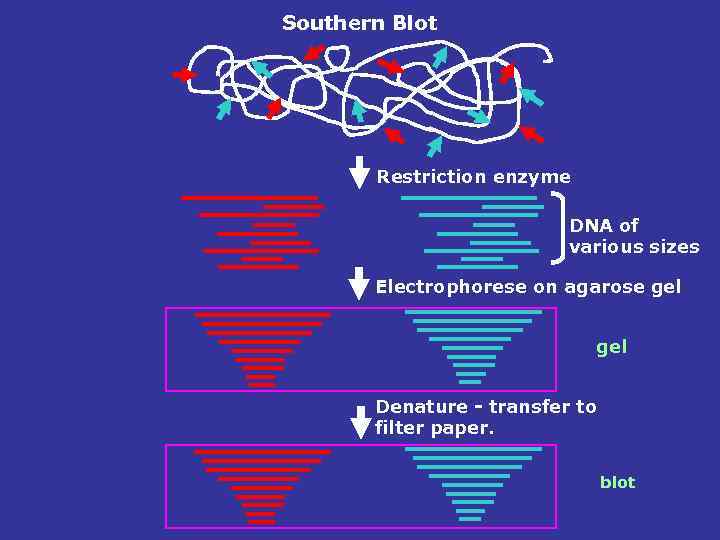 Southern Blot Restriction enzyme DNA of various sizes Electrophorese on agarose gel Denature - transfer to filter paper. blot
Southern Blot Restriction enzyme DNA of various sizes Electrophorese on agarose gel Denature - transfer to filter paper. blot
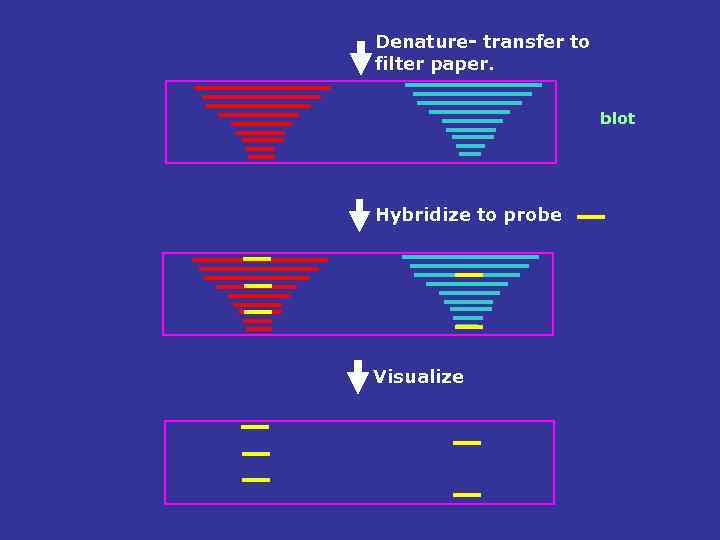 Denature- transfer to filter paper. blot Hybridize to probe Visualize
Denature- transfer to filter paper. blot Hybridize to probe Visualize
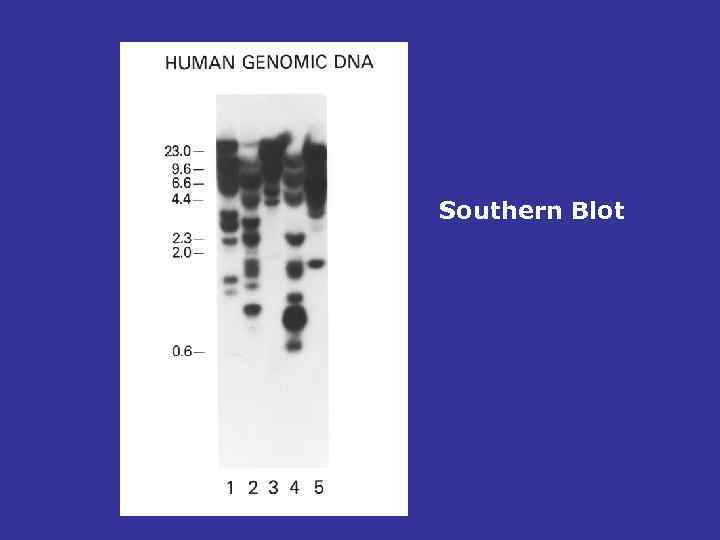 Southern Blot
Southern Blot
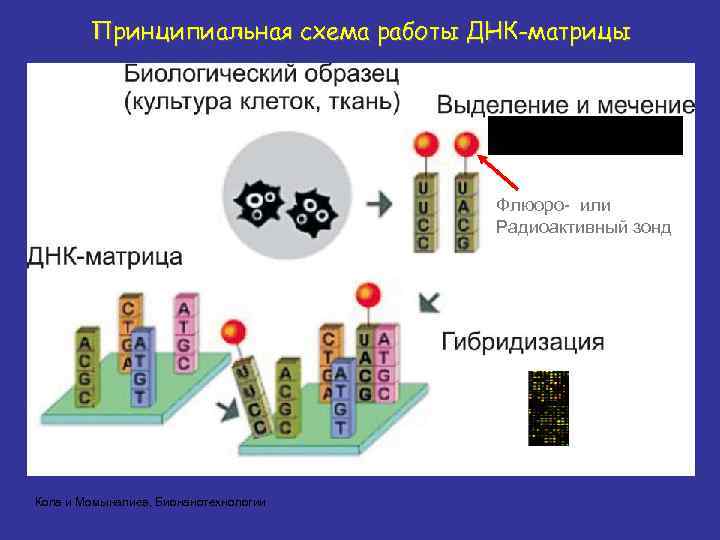 Принципиальная схема работы ДНК-матрицы геномной ДНК Флюоро- или Радиоактивный зонд Копа и Момыналиев, Бионанотехнологии
Принципиальная схема работы ДНК-матрицы геномной ДНК Флюоро- или Радиоактивный зонд Копа и Момыналиев, Бионанотехнологии
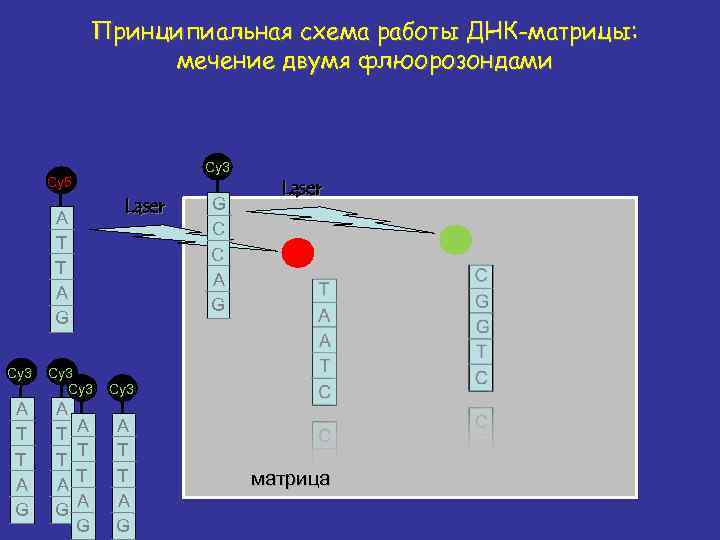 Принципиальная схема работы ДНК-матрицы: мечение двумя флюорозондами Cy 3 Cy 5 Laser A T T A G Cy 3 A T T A G G C C A G Laser Cy 3 A T T A G матрица
Принципиальная схема работы ДНК-матрицы: мечение двумя флюорозондами Cy 3 Cy 5 Laser A T T A G Cy 3 A T T A G G C C A G Laser Cy 3 A T T A G матрица
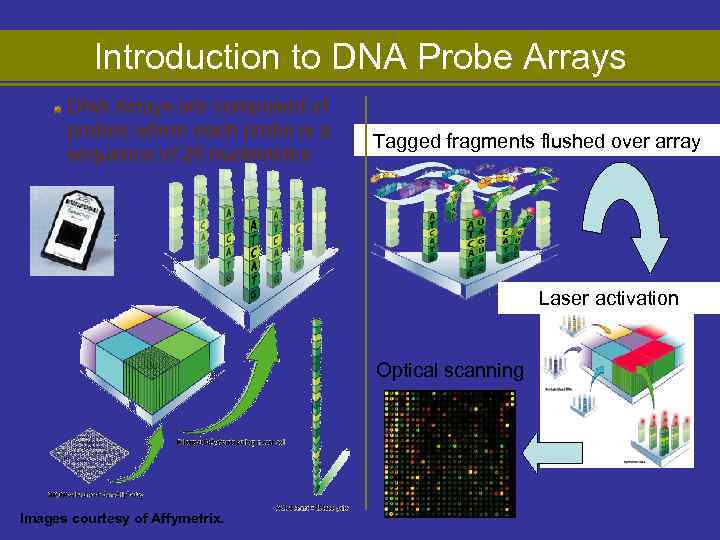 Introduction to DNA Probe Arrays DNA Arrays are composed of probes where each probe is a sequence of 25 nucleotides Tagged fragments flushed over array Laser activation Optical scanning Images courtesy of Affymetrix.
Introduction to DNA Probe Arrays DNA Arrays are composed of probes where each probe is a sequence of 25 nucleotides Tagged fragments flushed over array Laser activation Optical scanning Images courtesy of Affymetrix.
 Microarray types The main types of gene expression microarrays: • Short oligonucleotide arrays (Affymetrix) • c. DNA or spotted arrays (Brown lab) • Long oligonucleotide arrays (Agilent Inkjet) • Fiber-optic arrays • . . .
Microarray types The main types of gene expression microarrays: • Short oligonucleotide arrays (Affymetrix) • c. DNA or spotted arrays (Brown lab) • Long oligonucleotide arrays (Agilent Inkjet) • Fiber-optic arrays • . . .
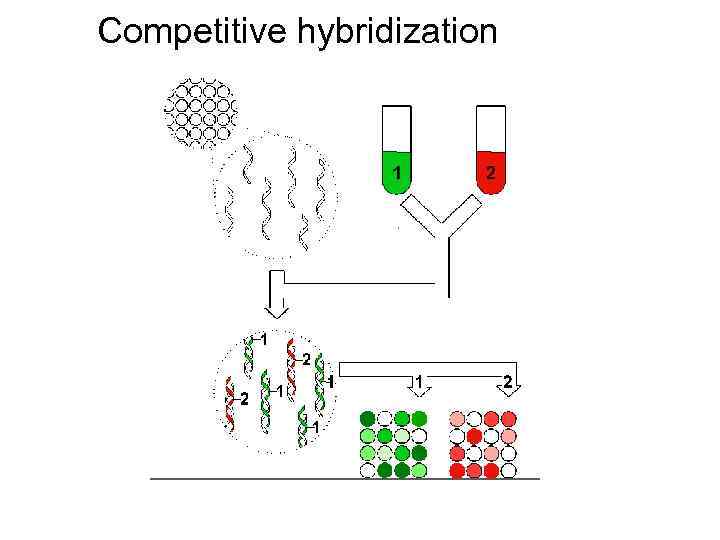 Competitive hybridization
Competitive hybridization
 Microarray image data mouse heart versus liver hybridization
Microarray image data mouse heart versus liver hybridization
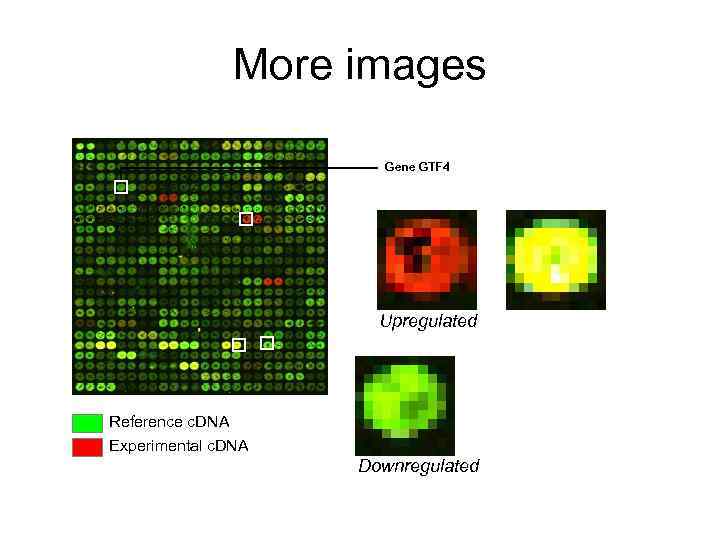 More images Gene GTF 4 Upregulated Reference c. DNA Experimental c. DNA Downregulated
More images Gene GTF 4 Upregulated Reference c. DNA Experimental c. DNA Downregulated
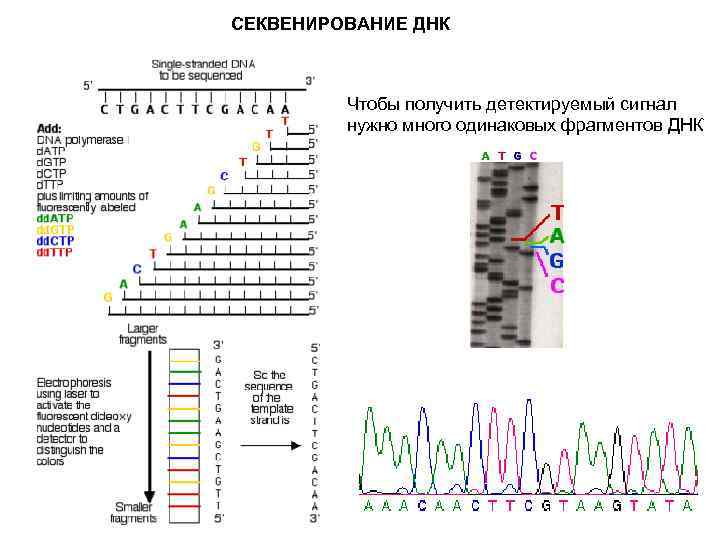 СЕКВЕНИРОВАНИЕ ДНК Чтобы получить детектируемый сигнал нужно много одинаковых фрагментов ДНК
СЕКВЕНИРОВАНИЕ ДНК Чтобы получить детектируемый сигнал нужно много одинаковых фрагментов ДНК


 проект «Геном человека» начало – 1990 предварительная версия – 2000 (публикация 2001) полная версия – 2003 сейчас чаще всего используют сборку 2006 г 8% (центромерные участки) не секвенировано цена проекта 2, 7 миллиарда долларов ~3, 000, 000 п. н. ~20, 000 генов, кодирующих белки (~2% of the genome) некодирующие последовательности (~95% of the genome) сейчас – индивидуальная геномика проект 1000 геномов (завершен) цена ресеквенирования генома человека ~30000 долларов (с покрытием >10) время ресеквенирования – несколько дней задача – $ 1000, 1 день
проект «Геном человека» начало – 1990 предварительная версия – 2000 (публикация 2001) полная версия – 2003 сейчас чаще всего используют сборку 2006 г 8% (центромерные участки) не секвенировано цена проекта 2, 7 миллиарда долларов ~3, 000, 000 п. н. ~20, 000 генов, кодирующих белки (~2% of the genome) некодирующие последовательности (~95% of the genome) сейчас – индивидуальная геномика проект 1000 геномов (завершен) цена ресеквенирования генома человека ~30000 долларов (с покрытием >10) время ресеквенирования – несколько дней задача – $ 1000, 1 день
 ПАРАЛЛЕЛЬНОЕ СЕКВЕНИРОВАНИЕ: метод 454 Life Sciences • Геном фрагментируется • К фрагментам ДНК присоединятся адаптеры для связывания, амплификации и идентификации
ПАРАЛЛЕЛЬНОЕ СЕКВЕНИРОВАНИЕ: метод 454 Life Sciences • Геном фрагментируется • К фрагментам ДНК присоединятся адаптеры для связывания, амплификации и идентификации
 метод 454: амплификация ДНК • Каждый фрагмент связывается с уникальной микросферой (~28 μm di. ) • Мкрогранулы помещают в водно-масляную эмульсию, содержащую все для амплификации (ПЦР) • Результат: in ~106 копий на каждой грануле
метод 454: амплификация ДНК • Каждый фрагмент связывается с уникальной микросферой (~28 μm di. ) • Мкрогранулы помещают в водно-масляную эмульсию, содержащую все для амплификации (ПЦР) • Результат: in ~106 копий на каждой грануле
 Только одна микрогранула в лунке
Только одна микрогранула в лунке
 • • Феременты для пирофосфатного секвенирования ииммобилизованы на бусинках меньшего размера
• • Феременты для пирофосфатного секвенирования ииммобилизованы на бусинках меньшего размера
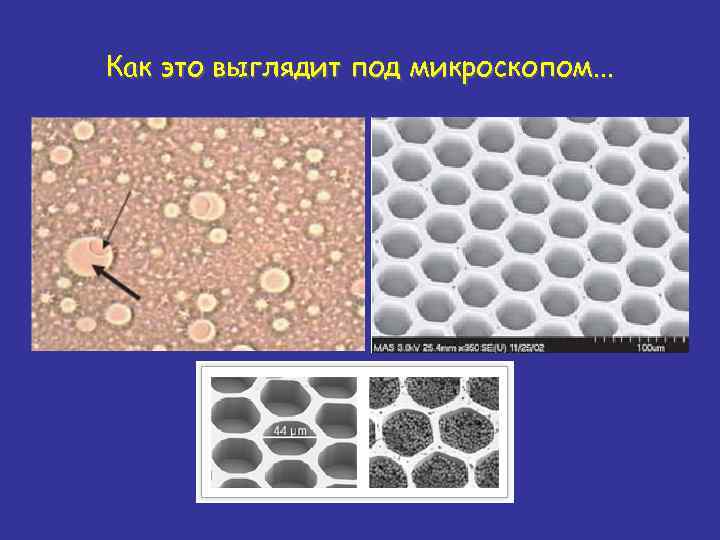 Как это выглядит под микроскопом. . .
Как это выглядит под микроскопом. . .
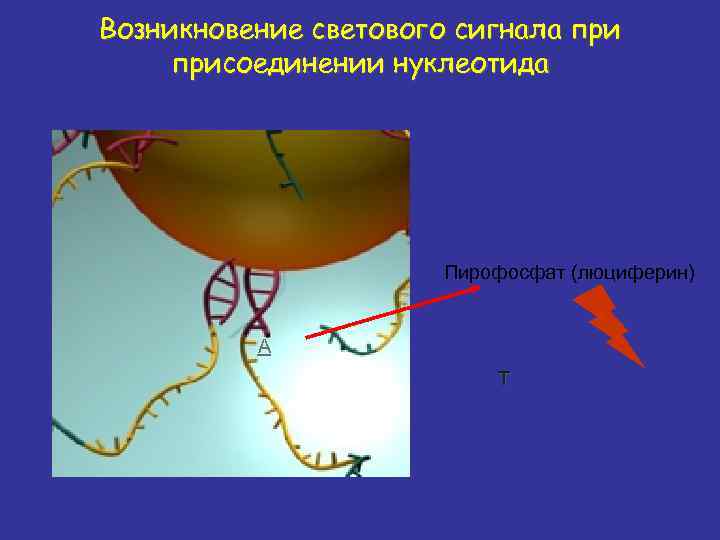 Возникновение светового сигнала присоединении нуклеотида Пирофосфат (люциферин) А Т
Возникновение светового сигнала присоединении нуклеотида Пирофосфат (люциферин) А Т
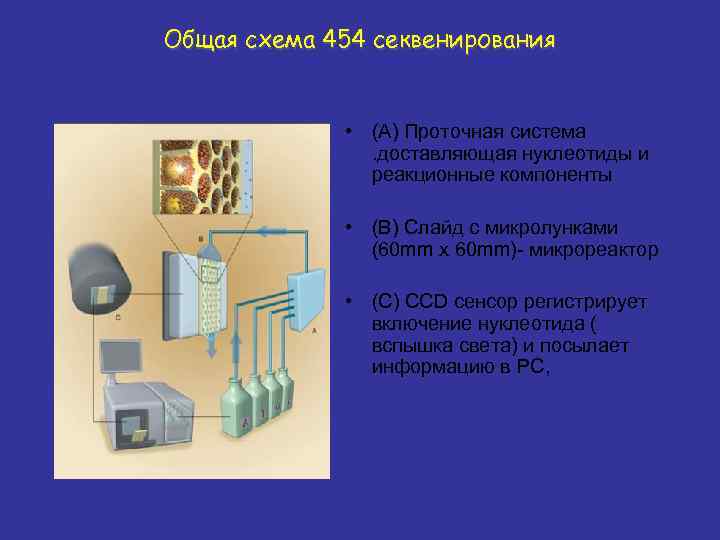 Общая схема 454 секвенирования • (A) Проточная система . доставляющая нуклеотиды и реакционные компоненты • (B) Слайд с микролунками (60 mm x 60 mm)- микрореактор • (C) CCD сенсор регистрирует включение нуклеотида ( вспышка света) и посылает информацию в РС,
Общая схема 454 секвенирования • (A) Проточная система . доставляющая нуклеотиды и реакционные компоненты • (B) Слайд с микролунками (60 mm x 60 mm)- микрореактор • (C) CCD сенсор регистрирует включение нуклеотида ( вспышка света) и посылает информацию в РС,

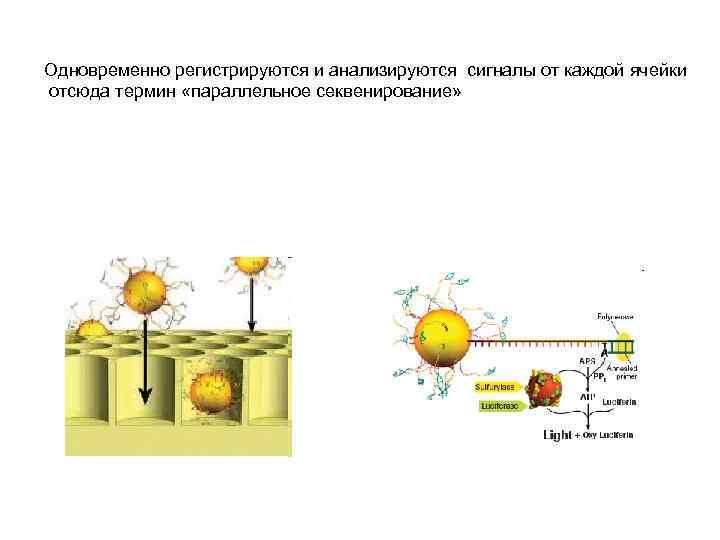 Одновременно регистрируются и анализируются сигналы от каждой ячейки отсюда термин «параллельное секвенирование»
Одновременно регистрируются и анализируются сигналы от каждой ячейки отсюда термин «параллельное секвенирование»
 Весь геном Mycoplasma genitalium (582, 970 оснований) был прочтён с точностью 99, 4 % за один раз!
Весь геном Mycoplasma genitalium (582, 970 оснований) был прочтён с точностью 99, 4 % за один раз!
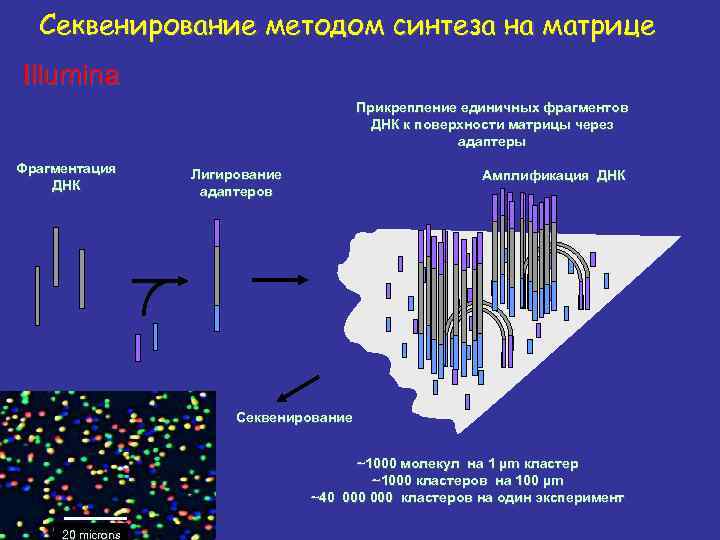 Секвенирование методом синтеза на матрице Illumina Прикрепление единичных фрагментов ДНК к поверхности матрицы через адаптеры Фрагментация ДНК Лигирование адаптеров Амплификация ДНК Секвенирование ~1000 молекул на 1 µm кластер ~1000 кластеров на 100 µm ~40 000 кластеров на один эксперимент 20 microns
Секвенирование методом синтеза на матрице Illumina Прикрепление единичных фрагментов ДНК к поверхности матрицы через адаптеры Фрагментация ДНК Лигирование адаптеров Амплификация ДНК Секвенирование ~1000 молекул на 1 µm кластер ~1000 кластеров на 100 µm ~40 000 кластеров на один эксперимент 20 microns
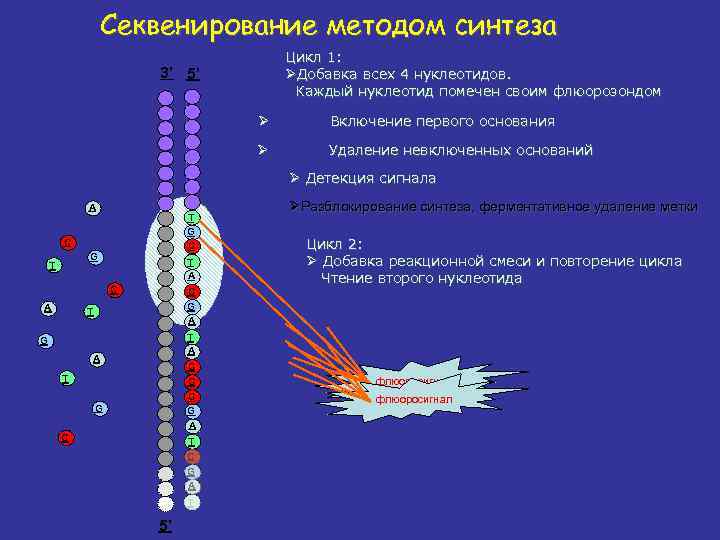 Секвенирование методом синтеза Цикл 1: ØДобавка всех 4 нуклеотидов. Каждый нуклеотид помечен своим флюорозондом 3’ 5’ Ø Включение первого основания Ø Удаление невключенных оснований Ø Детекция сигнала A T G C C G T T A C ØРазблокирование синтеза, ферментативное удаление метки Цикл 2: Ø Добавка реакционной смеси и повторение цикла Чтение второго нуклеотида G A T T A G A C T C C G A G C флюоросигнал T C G A T 5’
Секвенирование методом синтеза Цикл 1: ØДобавка всех 4 нуклеотидов. Каждый нуклеотид помечен своим флюорозондом 3’ 5’ Ø Включение первого основания Ø Удаление невключенных оснований Ø Детекция сигнала A T G C C G T T A C ØРазблокирование синтеза, ферментативное удаление метки Цикл 2: Ø Добавка реакционной смеси и повторение цикла Чтение второго нуклеотида G A T T A G A C T C C G A G C флюоросигнал T C G A T 5’
 С матрицы считывается тип включенного основания в каждый кластер Около 1 миллиона оснований определяется за один цикл синтеза T G C T A C G A T … 1 2 3 4 5 6 7 8 T T T T G T … Последовательность ДНК определяется путем анализа всей совокупности данных, снятых с матрицы 9
С матрицы считывается тип включенного основания в каждый кластер Около 1 миллиона оснований определяется за один цикл синтеза T G C T A C G A T … 1 2 3 4 5 6 7 8 T T T T G T … Последовательность ДНК определяется путем анализа всей совокупности данных, снятых с матрицы 9
 Illumina секвенировала индивидуальный геном африканского мужчины за несколько недель, достигнув скорости прочтения до 3 миллионов оснований за цикл (февраль 2008)
Illumina секвенировала индивидуальный геном африканского мужчины за несколько недель, достигнув скорости прочтения до 3 миллионов оснований за цикл (февраль 2008)
 РЕПАРАЦИЯ ДНК
РЕПАРАЦИЯ ДНК
 Причины появления повреждений в ДНК • Ошибки репликации • Повреждения ДНК эндогенными агентами Гидролиз (депуринизация, дезаминирование) • Повреждения ДНК экзогенными агентами облучение повреждение химическими агентами (например, алкилирование) • Репликация «через повреждения» с использованием полимераз, отличающихся низкой точностью копирования
Причины появления повреждений в ДНК • Ошибки репликации • Повреждения ДНК эндогенными агентами Гидролиз (депуринизация, дезаминирование) • Повреждения ДНК экзогенными агентами облучение повреждение химическими агентами (например, алкилирование) • Репликация «через повреждения» с использованием полимераз, отличающихся низкой точностью копирования
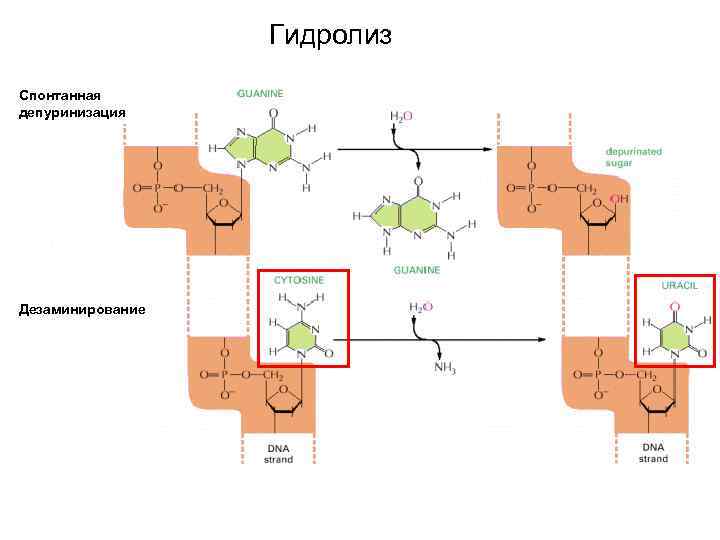 Гидролиз Спонтанная депуринизация Дезаминирование
Гидролиз Спонтанная депуринизация Дезаминирование
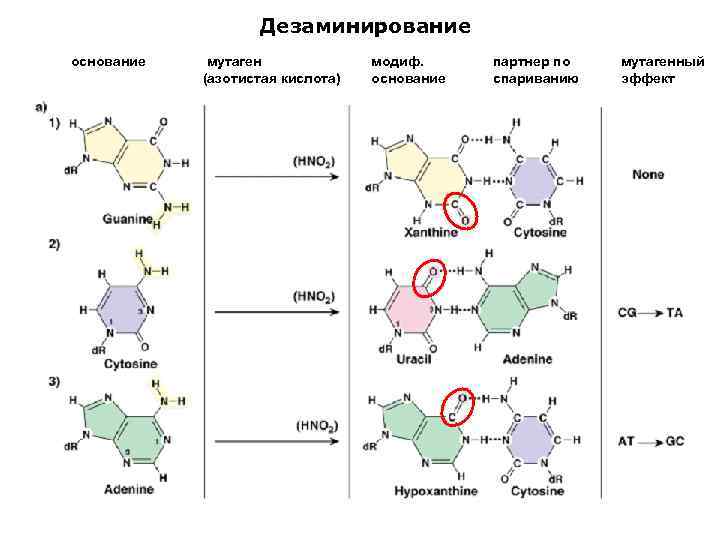 Дезаминирование основание мутаген (азотистая кислота) модиф. основание партнер по спариванию мутагенный эффект
Дезаминирование основание мутаген (азотистая кислота) модиф. основание партнер по спариванию мутагенный эффект
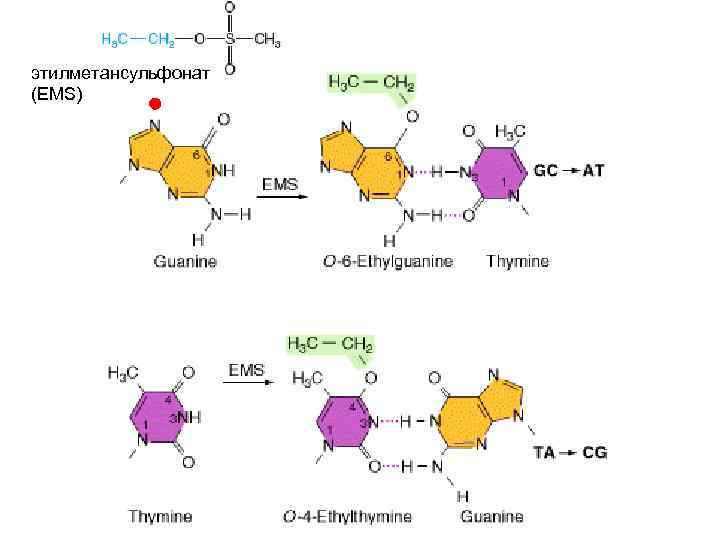 этилметансульфонат (EMS)
этилметансульфонат (EMS)
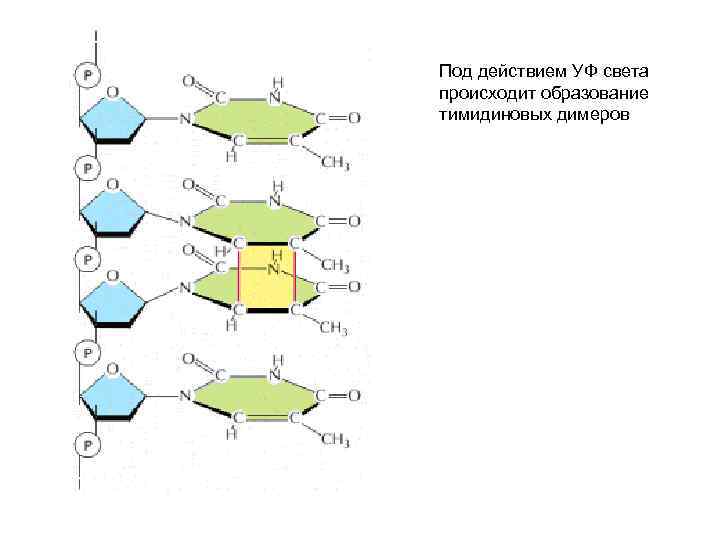 Под действием УФ света происходит образование тимидиновых димеров
Под действием УФ света происходит образование тимидиновых димеров
 Cтратегии коррекции повреждений • Ошибки репликации: • исправление ошибок ДНК полимеразой (3’-5’ экзонуклеаза), • репарация неспаренных оснований (mismatch repair) • Повреждение ДНК эндогенными и экзогенными агентами: • • Прямое удаление повреждений Эксцизия оснований (base excision repair) Эксцизия нуклеотидов (nucleotide excision repair) Рекомбинация и черезблоковый синтез особыми полимеразами (не удаляет ошибок но позволяет продолжить репликацию)
Cтратегии коррекции повреждений • Ошибки репликации: • исправление ошибок ДНК полимеразой (3’-5’ экзонуклеаза), • репарация неспаренных оснований (mismatch repair) • Повреждение ДНК эндогенными и экзогенными агентами: • • Прямое удаление повреждений Эксцизия оснований (base excision repair) Эксцизия нуклеотидов (nucleotide excision repair) Рекомбинация и черезблоковый синтез особыми полимеразами (не удаляет ошибок но позволяет продолжить репликацию)
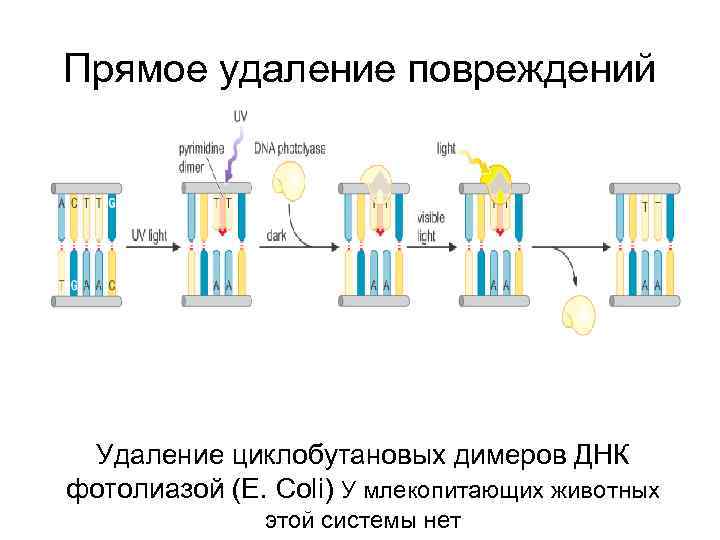 Прямое удаление повреждений Удаление циклобутановых димеров ДНК фотолиазой (E. Coli) У млекопитающих животных этой системы нет
Прямое удаление повреждений Удаление циклобутановых димеров ДНК фотолиазой (E. Coli) У млекопитающих животных этой системы нет
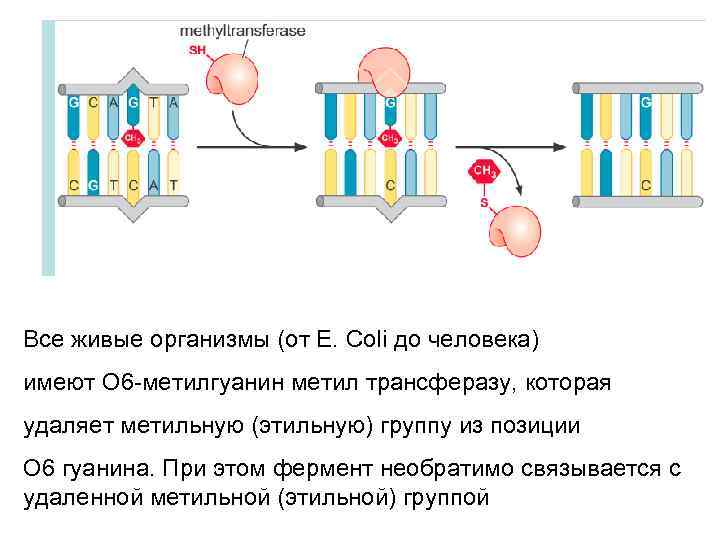 Все живые организмы (от E. Coli до человека) имеют О 6 -метилгуанин метил трансферазу, которая удаляет метильную (этильную) группу из позиции О 6 гуанина. При этом фермент необратимо связывается с удаленной метильной (этильной) группой
Все живые организмы (от E. Coli до человека) имеют О 6 -метилгуанин метил трансферазу, которая удаляет метильную (этильную) группу из позиции О 6 гуанина. При этом фермент необратимо связывается с удаленной метильной (этильной) группой
 Другие повреждения удаляются с помощью более сложных механизмов Эксцизия оснований (base excision repair) удаляется один поврежденный нуклеотид Эксцизия нуклеотидов (nucleotide excision repair) удаляется достаточно протяженный олигонуклеотид (12 -13 н. у E. Coli и 24 -32 н. у эукариотических организмов)
Другие повреждения удаляются с помощью более сложных механизмов Эксцизия оснований (base excision repair) удаляется один поврежденный нуклеотид Эксцизия нуклеотидов (nucleotide excision repair) удаляется достаточно протяженный олигонуклеотид (12 -13 н. у E. Coli и 24 -32 н. у эукариотических организмов)
 Base excision repair (BER) прокариоты и эукариоты • Основной путь удаления модифицированных (в том числе окисленных) оснований и включенного по ошибке урацила • Различные ДНК-гликозилазы адресно узнают поврежденные основания и удаляют их, разрезая гликозидную связь. При этом возникает AP (апуриновый/апиримидиновый) сайт. • В клетке существует несколько АР эндонуклеаз, которые разрезают фосфодиэфирный остов ДНК рядом с AP сайтом • AP nucleotide удаляется экзонуклеазой/дезоксирибофосфодиэстеразой и «брешь» застраивается ДНК полимеразой
Base excision repair (BER) прокариоты и эукариоты • Основной путь удаления модифицированных (в том числе окисленных) оснований и включенного по ошибке урацила • Различные ДНК-гликозилазы адресно узнают поврежденные основания и удаляют их, разрезая гликозидную связь. При этом возникает AP (апуриновый/апиримидиновый) сайт. • В клетке существует несколько АР эндонуклеаз, которые разрезают фосфодиэфирный остов ДНК рядом с AP сайтом • AP nucleotide удаляется экзонуклеазой/дезоксирибофосфодиэстеразой и «брешь» застраивается ДНК полимеразой
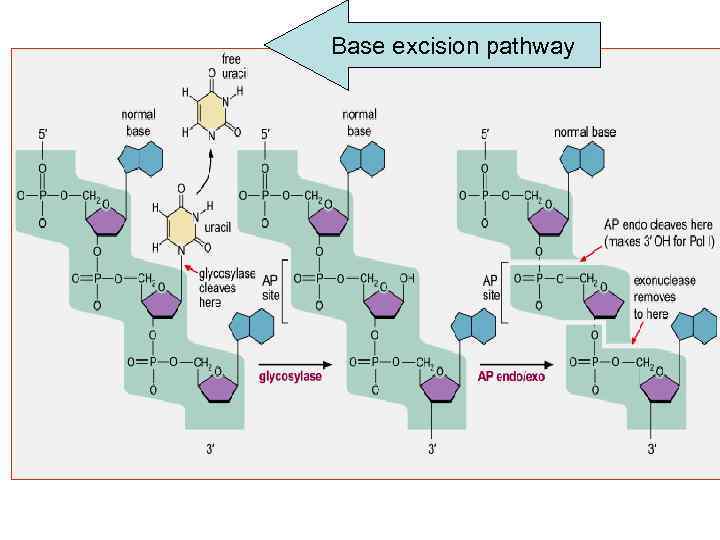 Base excision pathway
Base excision pathway
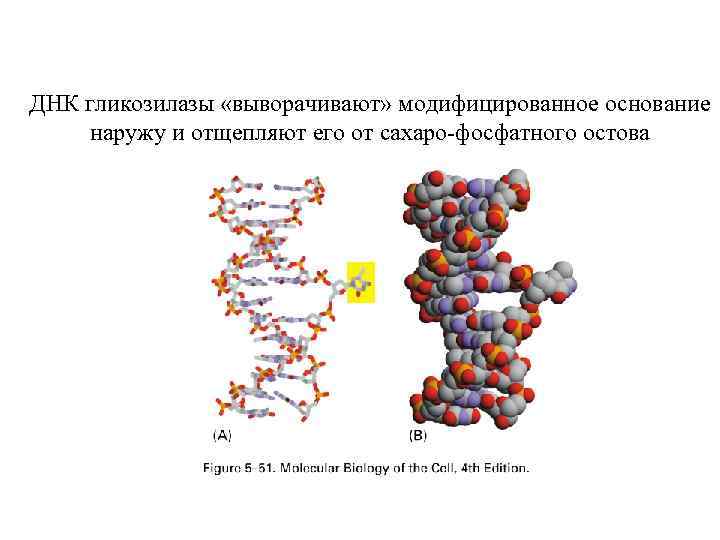 ДНК гликозилазы «выворачивают» модифицированное основание наружу и отщепляют его от сахаро-фосфатного остова
ДНК гликозилазы «выворачивают» модифицированное основание наружу и отщепляют его от сахаро-фосфатного остова
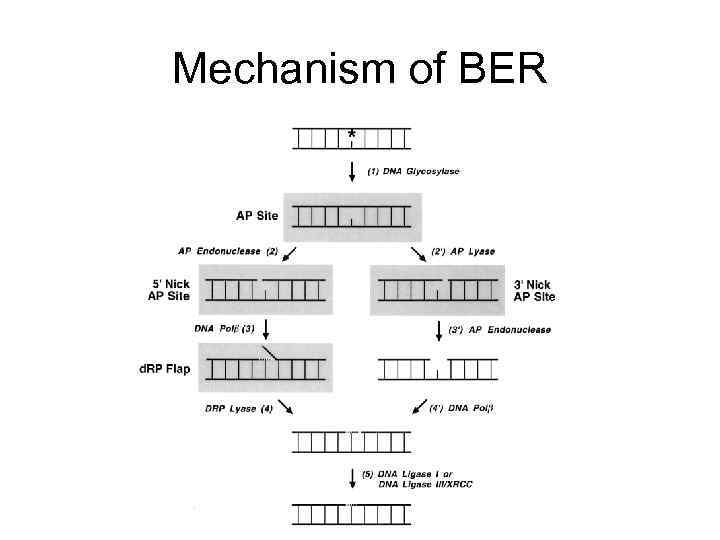 Mechanism of BER
Mechanism of BER
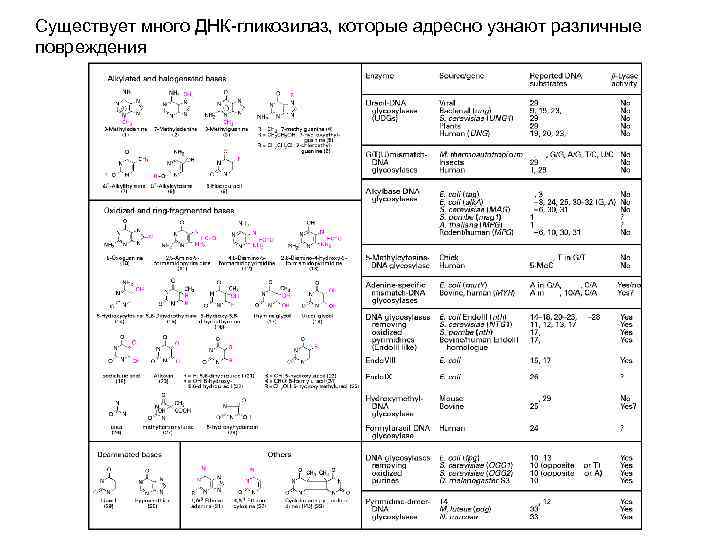 Существует много ДНК-гликозилаз, которые адресно узнают различные повреждения
Существует много ДНК-гликозилаз, которые адресно узнают различные повреждения
 ДНК-гликозизазы, присутствующие в клетках человека DNA glycosylase Specific for MBD 4 Uracil MPG Ethenoadenine, hypoxanthine, 3 methyladenine NTH 1 Cytosine glycol, dihydrouracil, formamidopyrimidine, thymine glycol OGG 1 Formamidopyrimidine, 8 -oxoguanine SMUG 1 Uracil TDG Ethenocytosine, uracil UNG Uracil, 5 -hydroxyuracil
ДНК-гликозизазы, присутствующие в клетках человека DNA glycosylase Specific for MBD 4 Uracil MPG Ethenoadenine, hypoxanthine, 3 methyladenine NTH 1 Cytosine glycol, dihydrouracil, formamidopyrimidine, thymine glycol OGG 1 Formamidopyrimidine, 8 -oxoguanine SMUG 1 Uracil TDG Ethenocytosine, uracil UNG Uracil, 5 -hydroxyuracil
 Основные типы повреждений, которые удаляются посредством BER (все эти повреждения не блокируют репликацию) • Окисленные основания, в том числе 8 -oкси-G, который спаривается с А, вызывая GC --> TA трансверсии • Дезоксиурацил • Различные продукты алкилирования оснований (например, 3 -me. A) • Спонтанно возникающие апуриновые сайты
Основные типы повреждений, которые удаляются посредством BER (все эти повреждения не блокируют репликацию) • Окисленные основания, в том числе 8 -oкси-G, который спаривается с А, вызывая GC --> TA трансверсии • Дезоксиурацил • Различные продукты алкилирования оснований (например, 3 -me. A) • Спонтанно возникающие апуриновые сайты
 Эксцизия нуклеотидов (nucleotide excision repair) прокариоты и эукриоты Этот механизм используется для коррекции «серьезных» повреждений, которые блокируют репликацию (у человека таковыми являются, в частности, тимидиновые димеры). 1. Особые белки узнают поврежденные участки ДНК и привлекают специальные нуклеазы, которые вносят разрывы перед повреждением и после него. 2. Фрагмент ДНК, содержащий повреждение, удаляется, и образовавшаяся брешь застраивается ДНК полимеразой
Эксцизия нуклеотидов (nucleotide excision repair) прокариоты и эукриоты Этот механизм используется для коррекции «серьезных» повреждений, которые блокируют репликацию (у человека таковыми являются, в частности, тимидиновые димеры). 1. Особые белки узнают поврежденные участки ДНК и привлекают специальные нуклеазы, которые вносят разрывы перед повреждением и после него. 2. Фрагмент ДНК, содержащий повреждение, удаляется, и образовавшаяся брешь застраивается ДНК полимеразой
 У E. Coli четыре белка участвуют в эксцизии нуклеотидов: Uvr. A, Uvr. B, Uvr. C, Uvr. D 1. Uvr. A и Uvr. B сканируют ДНК и выявляют поврежденные места 2. После выявления поврежденного участка Uvr. A освобождается из комплекса, а Uvr. B вызывает локальную денатурацию поврежденного участка и привлекает Uvr. C. 3. Комплекс Uvr. BС вносит однонитевые разрывы с 5’- и 3’- конца от повреждения 4. DNA хеликаза Uvr. D обеспечивает удаление из дуплекса фрагмента ДНК, содержащего повреждение. 5. DNA Pol I застраивает брешь и лигаза «зашивает» однонитевой разрыв.
У E. Coli четыре белка участвуют в эксцизии нуклеотидов: Uvr. A, Uvr. B, Uvr. C, Uvr. D 1. Uvr. A и Uvr. B сканируют ДНК и выявляют поврежденные места 2. После выявления поврежденного участка Uvr. A освобождается из комплекса, а Uvr. B вызывает локальную денатурацию поврежденного участка и привлекает Uvr. C. 3. Комплекс Uvr. BС вносит однонитевые разрывы с 5’- и 3’- конца от повреждения 4. DNA хеликаза Uvr. D обеспечивает удаление из дуплекса фрагмента ДНК, содержащего повреждение. 5. DNA Pol I застраивает брешь и лигаза «зашивает» однонитевой разрыв.
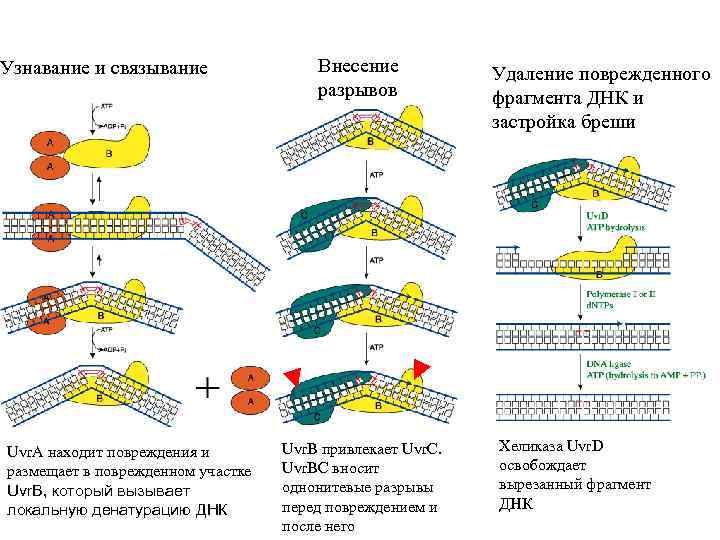 Узнавание и связывание Uvr. A находит повреждения и размещает в поврежденном участке Uvr. B, который вызывает локальную денатурацию ДНК Внесение разрывов Uvr. B привлекает Uvr. C. Uvr. BC вносит однонитевые разрывы перед повреждением и после него Удаление поврежденного фрагмента ДНК и застройка бреши Хеликаза Uvr. D освобождает вырезанный фрагмент ДНК
Узнавание и связывание Uvr. A находит повреждения и размещает в поврежденном участке Uvr. B, который вызывает локальную денатурацию ДНК Внесение разрывов Uvr. B привлекает Uvr. C. Uvr. BC вносит однонитевые разрывы перед повреждением и после него Удаление поврежденного фрагмента ДНК и застройка бреши Хеликаза Uvr. D освобождает вырезанный фрагмент ДНК
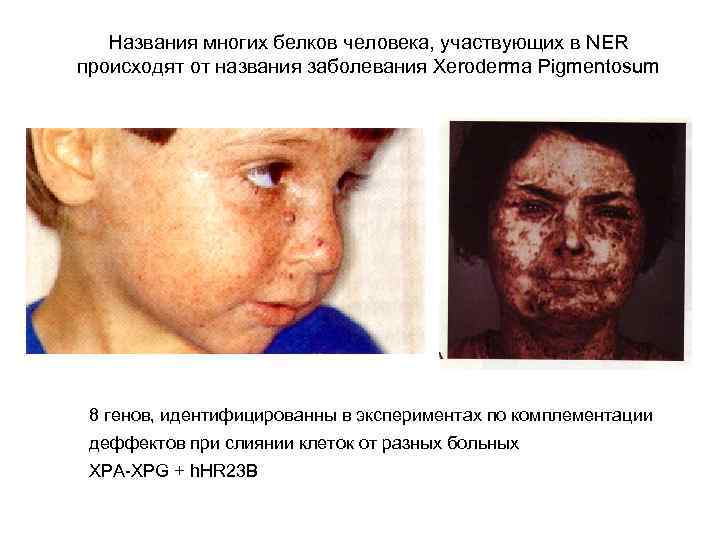 Названия многих белков человека, участвующих в NER происходят от названия заболевания Xeroderma Pigmentosum 8 генов, идентифицированны в экспериментах по комплементации деффектов при слиянии клеток от разных больных XPA-XPG + h. HR 23 B
Названия многих белков человека, участвующих в NER происходят от названия заболевания Xeroderma Pigmentosum 8 генов, идентифицированны в экспериментах по комплементации деффектов при слиянии клеток от разных больных XPA-XPG + h. HR 23 B
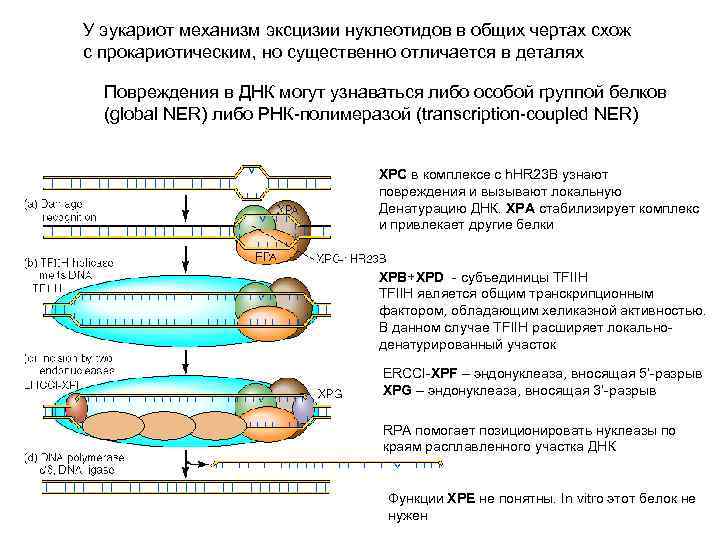 У эукариот механизм эксцизии нуклеотидов в общих чертах схож с прокариотическим, но существенно отличается в деталях Повреждения в ДНК могут узнаваться либо особой группой белков (global NER) либо РНК-полимеразой (transcription-coupled NER) XPC в комплексе с h. HR 23 B узнают повреждения и вызывают локальную Денатурацию ДНК. XPA стабилизирует комплекс и привлекает другие белки XPB+XPD - субъединицы TFIIH является общим транскрипционным фактором, обладающим хеликазной активностью. В данном случае TFIIH расширяет локальноденатурированный участок ERCCI-XPF – эндонуклеаза, вносящая 5’-разрыв XPG – эндонуклеаза, вносящая 3’-разрыв RPA помогает позиционировать нуклеазы по краям расплавленного участка ДНК Функции XPE не понятны. In vitro этот белок не нужен
У эукариот механизм эксцизии нуклеотидов в общих чертах схож с прокариотическим, но существенно отличается в деталях Повреждения в ДНК могут узнаваться либо особой группой белков (global NER) либо РНК-полимеразой (transcription-coupled NER) XPC в комплексе с h. HR 23 B узнают повреждения и вызывают локальную Денатурацию ДНК. XPA стабилизирует комплекс и привлекает другие белки XPB+XPD - субъединицы TFIIH является общим транскрипционным фактором, обладающим хеликазной активностью. В данном случае TFIIH расширяет локальноденатурированный участок ERCCI-XPF – эндонуклеаза, вносящая 5’-разрыв XPG – эндонуклеаза, вносящая 3’-разрыв RPA помогает позиционировать нуклеазы по краям расплавленного участка ДНК Функции XPE не понятны. In vitro этот белок не нужен
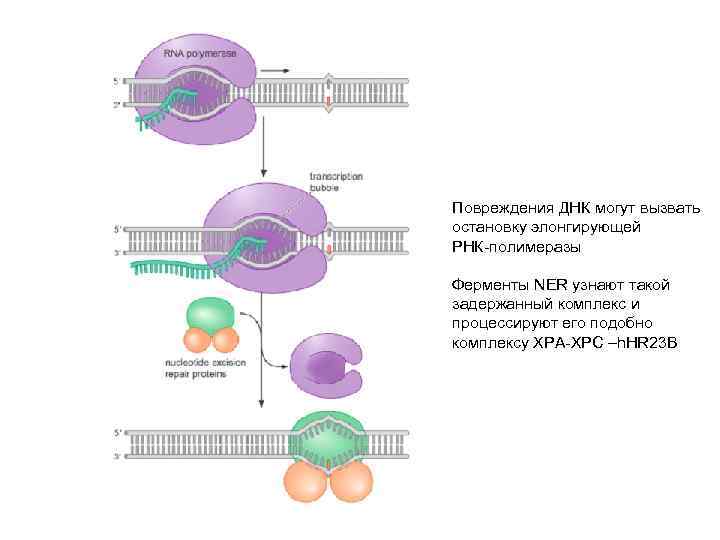 Повреждения ДНК могут вызвать остановку элонгирующей РНК-полимеразы Ферменты NER узнают такой задержанный комплекс и процессируют его подобно комплексу XPA-XPC –h. HR 23 B
Повреждения ДНК могут вызвать остановку элонгирующей РНК-полимеразы Ферменты NER узнают такой задержанный комплекс и процессируют его подобно комплексу XPA-XPC –h. HR 23 B
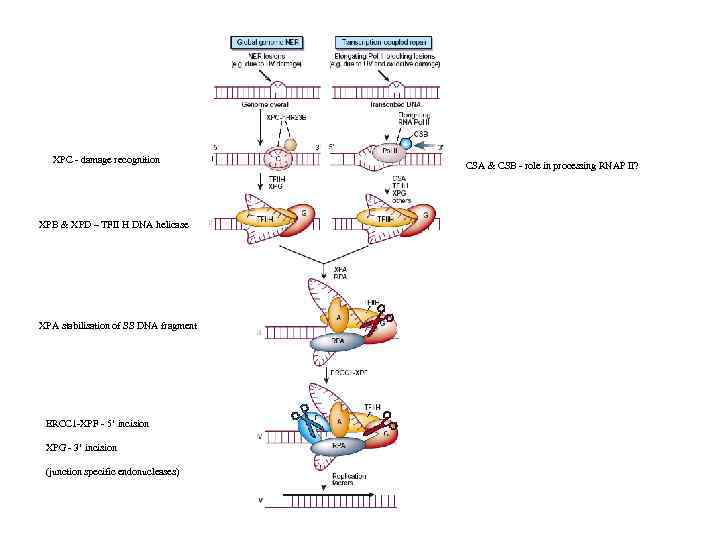 XPC - damage recognition XPB & XPD – TFII H DNA helicase XPA stabilisation of SS DNA fragment ERCC 1 -XPF - 5’ incision XPG - 3’ incision (junction specific endonucleases) CSA & CSB - role in processing RNAP II?
XPC - damage recognition XPB & XPD – TFII H DNA helicase XPA stabilisation of SS DNA fragment ERCC 1 -XPF - 5’ incision XPG - 3’ incision (junction specific endonucleases) CSA & CSB - role in processing RNAP II?


