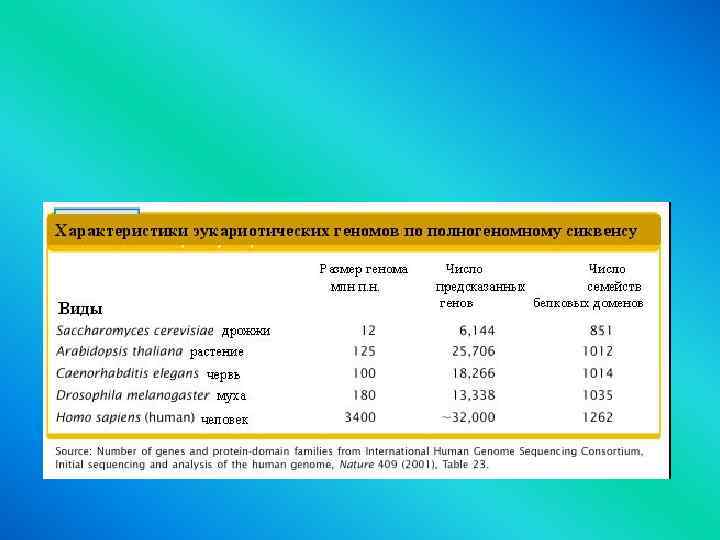
2 - Эу геном 2013 Вельков вв.ppt
- Количество слайдов: 112
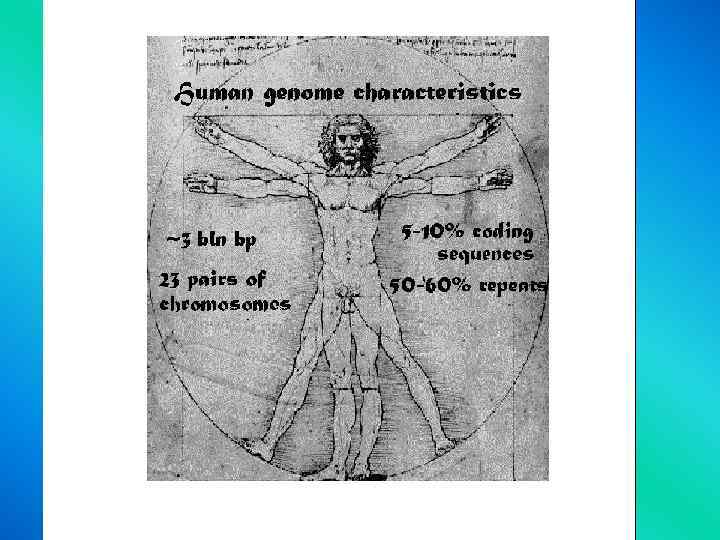
 Строение эукариотного генома Вельков ВВ 2013
Строение эукариотного генома Вельков ВВ 2013

 “The genetic information contained in each cell of the human body is approximately equivalent to a library of 4000 books. ” [500 pages per book & 300 words per page] -Carl Sagan -The Dragons of Eden, NY: Random House, 1977, p. 25
“The genetic information contained in each cell of the human body is approximately equivalent to a library of 4000 books. ” [500 pages per book & 300 words per page] -Carl Sagan -The Dragons of Eden, NY: Random House, 1977, p. 25
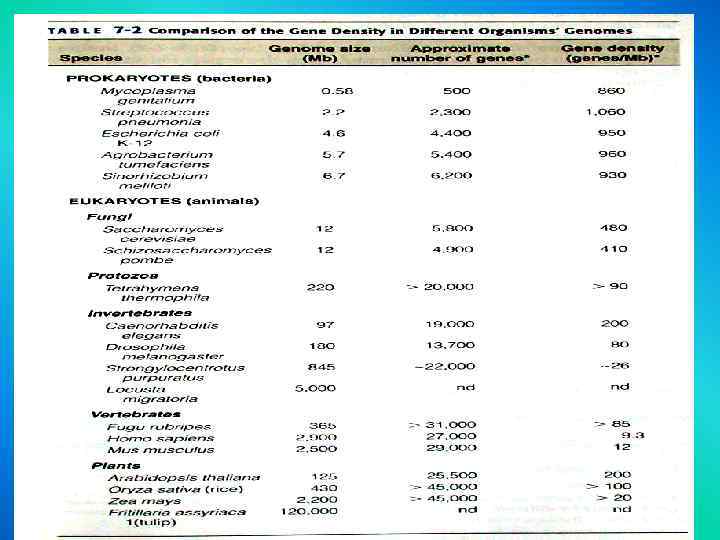
 Чем выше расположен организм на Древе Жизни и чем он сложнее, тем больше его геном и тем больше в нем генов …. ?
Чем выше расположен организм на Древе Жизни и чем он сложнее, тем больше его геном и тем больше в нем генов …. ?
 How about other life forms? ? Amoeba dubia: 670, 000, 000 “In the animal kingdom, the relationship between genome size and evolutionary status is not clear. One of the largest genomes belongs to a very small Amoeba proteus: 290, 000, 000 creature, Amoeba dubia. This protozoan genome has 670 billion units of DNA, or base pairs. The genome of a cousin, Amoeba proteus, has a mere 290 billion base pairs, making it 100 times larger than the human genome. ” Bufo bufo: 6, 900, 000 Man: 3, 000, 000 Sizing up genomes: Amoeba is King, Edward Winstead
How about other life forms? ? Amoeba dubia: 670, 000, 000 “In the animal kingdom, the relationship between genome size and evolutionary status is not clear. One of the largest genomes belongs to a very small Amoeba proteus: 290, 000, 000 creature, Amoeba dubia. This protozoan genome has 670 billion units of DNA, or base pairs. The genome of a cousin, Amoeba proteus, has a mere 290 billion base pairs, making it 100 times larger than the human genome. ” Bufo bufo: 6, 900, 000 Man: 3, 000, 000 Sizing up genomes: Amoeba is King, Edward Winstead
 Полная последовательность генома актинии Nematostella, представителя книдарий (Cnidaria), к ним относятся коралловые полипы, гидры и медузы. Выбор этого объекта определялся его «стратегическим» положением у самого основания эволюционного древа животных
Полная последовательность генома актинии Nematostella, представителя книдарий (Cnidaria), к ним относятся коралловые полипы, гидры и медузы. Выбор этого объекта определялся его «стратегическим» положением у самого основания эволюционного древа животных
 Губки – самые примитивные животные нет настоящих тканей, нервной системы и кишечника. Губки противопоставляются всем прочим, т. н. , «настоящим многоклеточным животным» (Eumetazoa). Среди Eumetazoa книдарии – самые примитивные
Губки – самые примитивные животные нет настоящих тканей, нервной системы и кишечника. Губки противопоставляются всем прочим, т. н. , «настоящим многоклеточным животным» (Eumetazoa). Среди Eumetazoa книдарии – самые примитивные
 Геном актинии 15 пар хромосом, около 450 млн пн оснований в 100 раз больше, чем у кишечной палочки, и в 6 раз меньше, чем у человека примерно 18 000 белок-кодирующих генов, что вполне сопоставимо с другими животными. Мобильные генетические элементы составляют 25% генома (вдвое меньше, чем у млекопитающих).
Геном актинии 15 пар хромосом, около 450 млн пн оснований в 100 раз больше, чем у кишечной палочки, и в 6 раз меньше, чем у человека примерно 18 000 белок-кодирующих генов, что вполне сопоставимо с другими животными. Мобильные генетические элементы составляют 25% генома (вдвое меньше, чем у млекопитающих).
 Человек унаследовал не менее 2/3 своих генов от общего с актинией предка; сама актиния — примерно столько же. Муха и круглый червь унаследовали от общего предка с актинией лишь 50% и 40% генов соответственно
Человек унаследовал не менее 2/3 своих генов от общего с актинией предка; сама актиния — примерно столько же. Муха и круглый червь унаследовали от общего предка с актинией лишь 50% и 40% генов соответственно
 Важнейшие генетические новации многоклеточных произошли на самых ранних этапах эволюции Последний общий предок актинии, человека и мухи жил около 700 млн лет назад иобладал весьма сложным геномом. Базовая генетическая «программа» развитияпервых животных, оказалась настолько удачной и гибкой, что последующая эволюция обеспечивалась в основном изменениями ее «настроек» , а не «архитектуры» .
Важнейшие генетические новации многоклеточных произошли на самых ранних этапах эволюции Последний общий предок актинии, человека и мухи жил около 700 млн лет назад иобладал весьма сложным геномом. Базовая генетическая «программа» развитияпервых животных, оказалась настолько удачной и гибкой, что последующая эволюция обеспечивалась в основном изменениями ее «настроек» , а не «архитектуры» .
 Геном человека более похож на геном актинии, чем геномы мухи и червя. Сходны не только набор генов, но и порядок их расположения в хромосомах. Около 80% генов общего предка Eumetazoa были унаследованы животными от одноклеточных предков (хоанофлагеллят, или воротничковых жгутиконосцев). геном на удивление мало изменился при становлении животного царства. Среди оставшихся 20% генов, аналогов которых нет у одноклеточных, много ключевых регуляторов развития. 25% этих новых генов содержат (домены), встречающиеся у одноклеточных, но в других комбинациях. Новые гены формируются из старых путем перекомбинирования фрагментов.
Геном человека более похож на геном актинии, чем геномы мухи и червя. Сходны не только набор генов, но и порядок их расположения в хромосомах. Около 80% генов общего предка Eumetazoa были унаследованы животными от одноклеточных предков (хоанофлагеллят, или воротничковых жгутиконосцев). геном на удивление мало изменился при становлении животного царства. Среди оставшихся 20% генов, аналогов которых нет у одноклеточных, много ключевых регуляторов развития. 25% этих новых генов содержат (домены), встречающиеся у одноклеточных, но в других комбинациях. Новые гены формируются из старых путем перекомбинирования фрагментов.
 Базовая генетическая программа первых животных, оказалась столь удачной и гибкой, что дальнейшая эволюция, связанная с усложнением организма, уже почти не требовала радикальных изменений самой программы. Достаточно было лишь небольших изменений в регуляторных участках ДНК
Базовая генетическая программа первых животных, оказалась столь удачной и гибкой, что дальнейшая эволюция, связанная с усложнением организма, уже почти не требовала радикальных изменений самой программы. Достаточно было лишь небольших изменений в регуляторных участках ДНК
 C-value paradox: Complexity does not correlate with genome size. 3. 4 x 109 bp Homo sapiens 6. 7 x 1011 bp Amoeba dubia
C-value paradox: Complexity does not correlate with genome size. 3. 4 x 109 bp Homo sapiens 6. 7 x 1011 bp Amoeba dubia
 The C-value paradox: What accounts for the often massive, counter-intuitive and seemingly arbitrary differences in genome size observed among eukaryotic organisms? The fruit fly Drosophila melanogaster 180 Mb The mountain grasshopper Podisma pedestris 18, 000 Mb The difference in genome size of a factor of 100 is difficult to explain in view of the apparently similar levels of evolutionary, developmental and behavioral complexity of these organisms.
The C-value paradox: What accounts for the often massive, counter-intuitive and seemingly arbitrary differences in genome size observed among eukaryotic organisms? The fruit fly Drosophila melanogaster 180 Mb The mountain grasshopper Podisma pedestris 18, 000 Mb The difference in genome size of a factor of 100 is difficult to explain in view of the apparently similar levels of evolutionary, developmental and behavioral complexity of these organisms.
 N-value paradox: Complexity does not correlate with gene number. ~25, 000 genes ~26, 000 genes ~50, 000 genes
N-value paradox: Complexity does not correlate with gene number. ~25, 000 genes ~26, 000 genes ~50, 000 genes
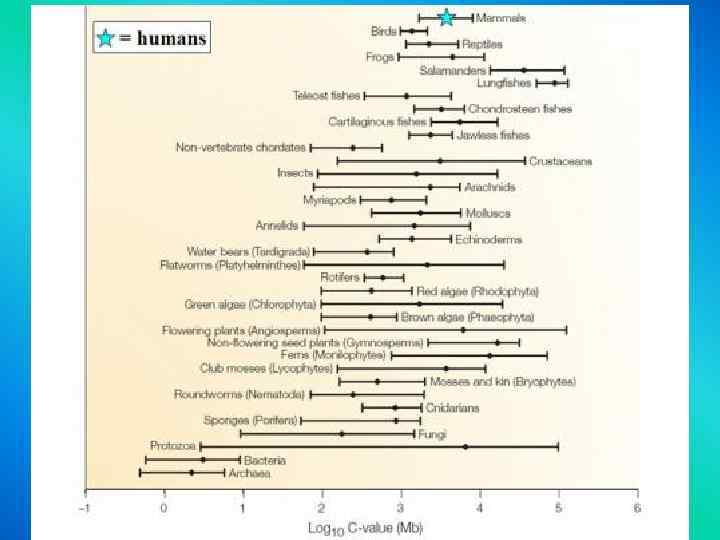
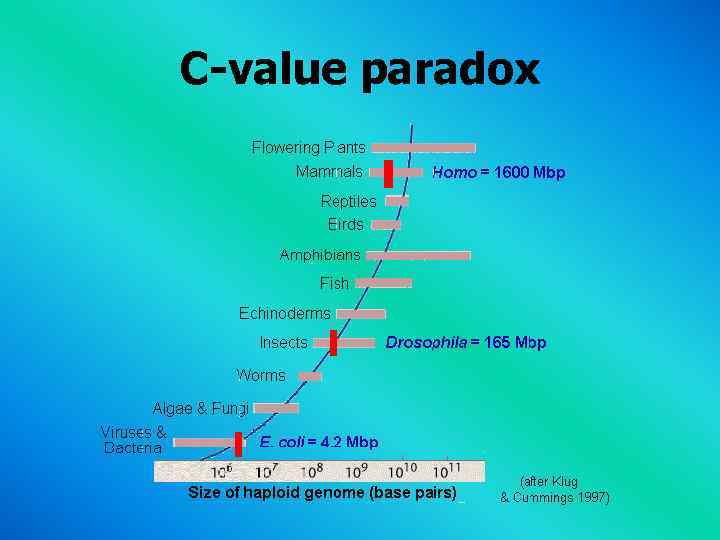 C-value paradox
C-value paradox

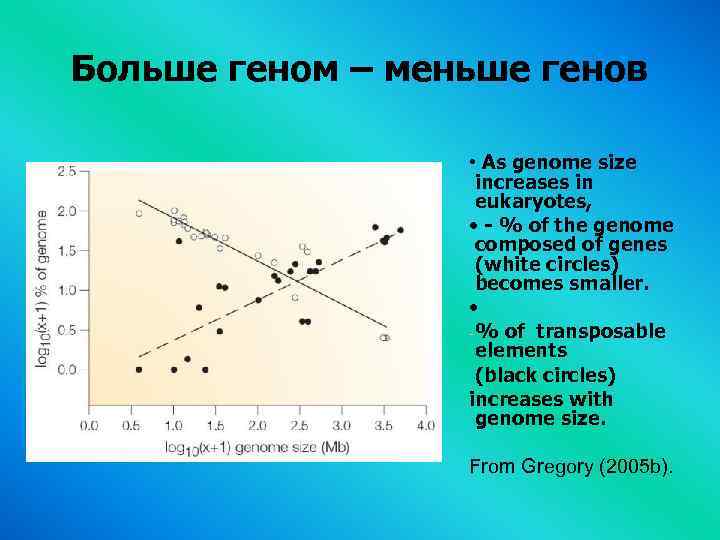 Больше геном – меньше генов • As genome size increases in eukaryotes, • - % of the genome composed of genes (white circles) becomes smaller. • - % of transposable elements (black circles) increases with genome size. From Gregory (2005 b).
Больше геном – меньше генов • As genome size increases in eukaryotes, • - % of the genome composed of genes (white circles) becomes smaller. • - % of transposable elements (black circles) increases with genome size. From Gregory (2005 b).
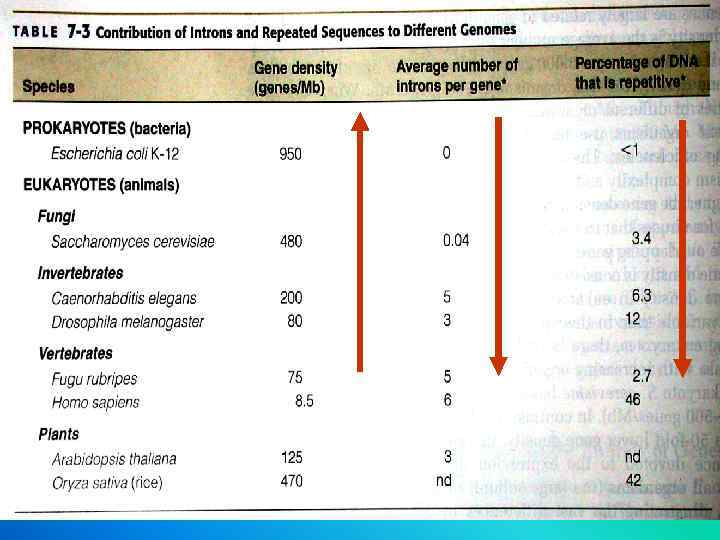
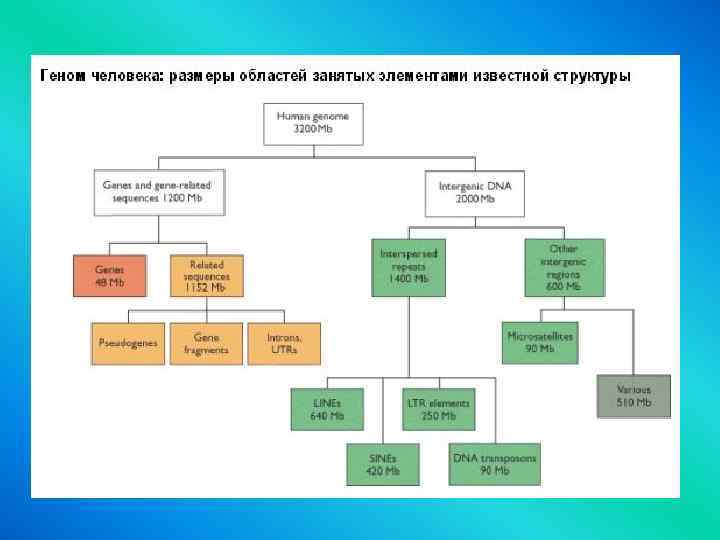
 Мусорная ДНК (? )
Мусорная ДНК (? )
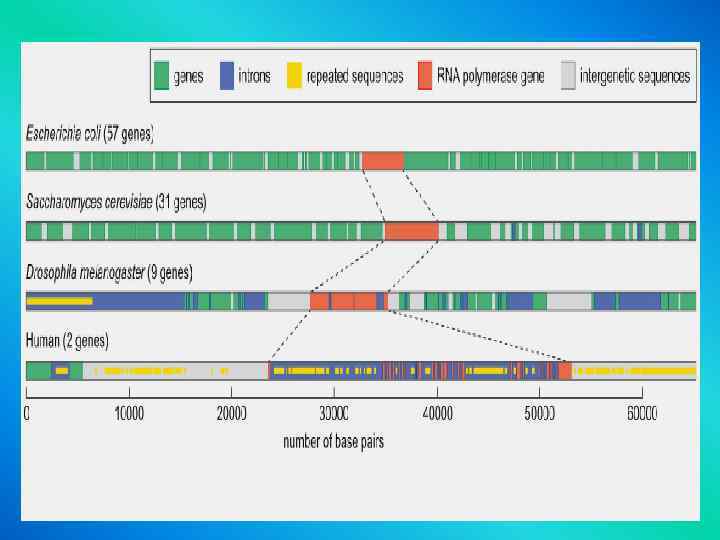
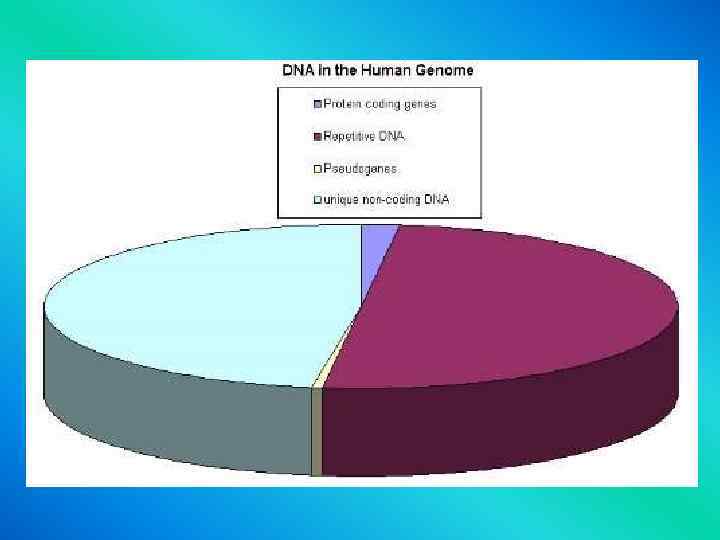
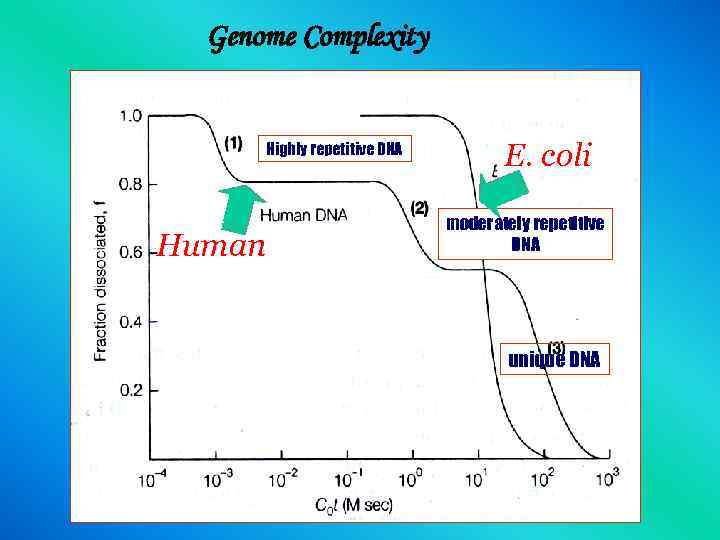 Genome Complexity Highly repetitive DNA Human E. coli moderately repetitive DNA unique DNA
Genome Complexity Highly repetitive DNA Human E. coli moderately repetitive DNA unique DNA
 Unique sequence DNA • The slowest to reassociate • Corresponds to coding regions of genes occurring in one or a few copies/haploid genome • All the DNA in E. coli genome has a unique sequence.
Unique sequence DNA • The slowest to reassociate • Corresponds to coding regions of genes occurring in one or a few copies/haploid genome • All the DNA in E. coli genome has a unique sequence.
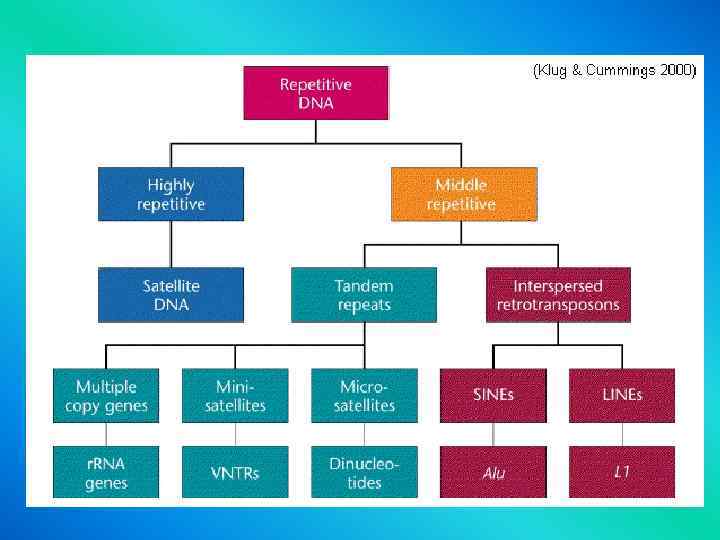
 Non-coding sequence DNA sequence that does not code for protein or RNA, including 1. Introns (unique sequence) in genes 2. DNA consisting of multiple repeats, can be tandemly repeated sequences (e. g. satellite DNA) 3. or interspersed repeats (e. g. Alu element) etc.
Non-coding sequence DNA sequence that does not code for protein or RNA, including 1. Introns (unique sequence) in genes 2. DNA consisting of multiple repeats, can be tandemly repeated sequences (e. g. satellite DNA) 3. or interspersed repeats (e. g. Alu element) etc.
 Repetitive DNA Tandem gene clusters: (1) moderately repetitive DNA consists of a number of types of repeated sequence. (2) genes whose products are required in unusually large quantities, e. g. there are 10 -10000 copies of r. DNA encoding 45 S precursor and X 100 copies of histone genes.
Repetitive DNA Tandem gene clusters: (1) moderately repetitive DNA consists of a number of types of repeated sequence. (2) genes whose products are required in unusually large quantities, e. g. there are 10 -10000 copies of r. DNA encoding 45 S precursor and X 100 copies of histone genes.
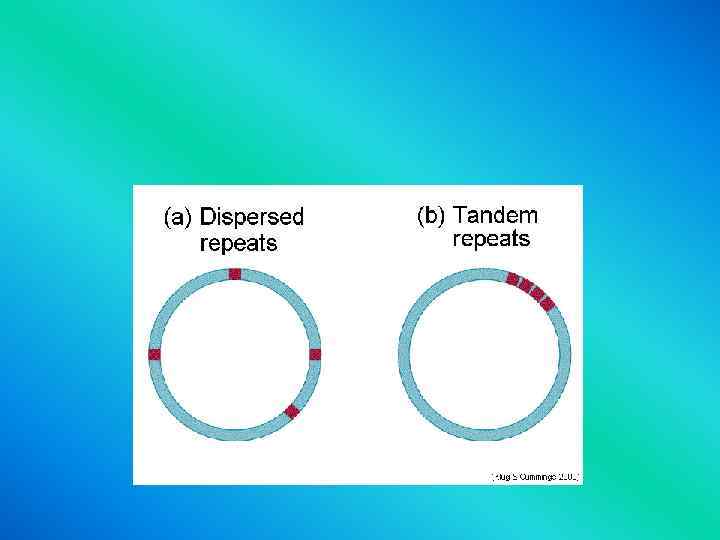
 Dispersed repetitive DNA 1. Moderately repetitive (x 100 - x 1000 copies) 2. Scattered throughout the genome Human Alu elements: 300 bp, 300 000 – 500 000 copies of 80 -90% identity Human L 1 element Alu + L 1= ~ 10% of human genome. Functions of these repetitive DNA are largely unknown
Dispersed repetitive DNA 1. Moderately repetitive (x 100 - x 1000 copies) 2. Scattered throughout the genome Human Alu elements: 300 bp, 300 000 – 500 000 copies of 80 -90% identity Human L 1 element Alu + L 1= ~ 10% of human genome. Functions of these repetitive DNA are largely unknown
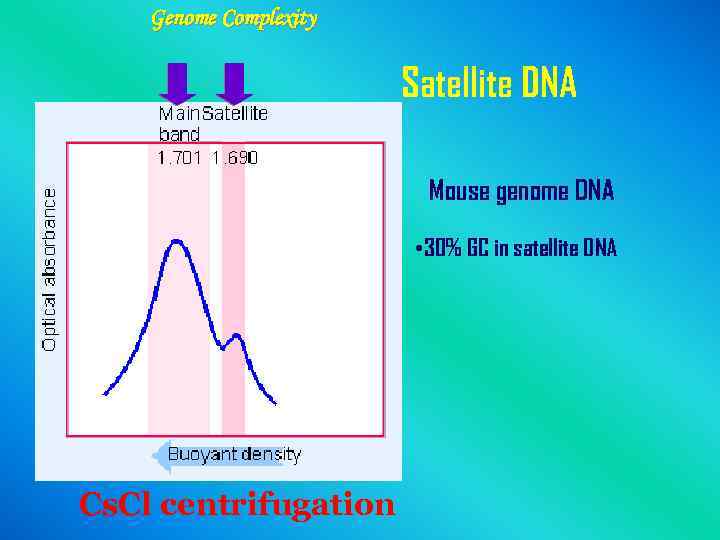 Genome Complexity Satellite DNA Mouse genome DNA • 30% GC in satellite DNA Cs. Cl centrifugation
Genome Complexity Satellite DNA Mouse genome DNA • 30% GC in satellite DNA Cs. Cl centrifugation
 Satellite DNA - Highly repetitive DNA (>10 x 6). - very short (2 to 20 -30 bp, mini- or micro-), in tandem arrays - concentrated near the centromeres and forms a large part of heterochromatin. - as separate band in buoyant density gradient - no function found, except a possible role in kinetochore binding - Minisatellite repeats are the basis of the DNA fingerprinting techniques
Satellite DNA - Highly repetitive DNA (>10 x 6). - very short (2 to 20 -30 bp, mini- or micro-), in tandem arrays - concentrated near the centromeres and forms a large part of heterochromatin. - as separate band in buoyant density gradient - no function found, except a possible role in kinetochore binding - Minisatellite repeats are the basis of the DNA fingerprinting techniques
 5’ – ATAAACTATAAACT – 3’ 3’ – TATTTGATATTTGA – 5’ n ACAAACT, 1. 1 x 107 bp, 25% genome ATAAACT, 3. 6 x 106 bp, 8% genome ACAAATT, 3. 6 x 106 bp, 8% genome AATATAG, cryptic Satellites comprise more than 40% of the genome Drosophila satellite DNA repeat (several million copies)
5’ – ATAAACTATAAACT – 3’ 3’ – TATTTGATATTTGA – 5’ n ACAAACT, 1. 1 x 107 bp, 25% genome ATAAACT, 3. 6 x 106 bp, 8% genome ACAAATT, 3. 6 x 106 bp, 8% genome AATATAG, cryptic Satellites comprise more than 40% of the genome Drosophila satellite DNA repeat (several million copies)
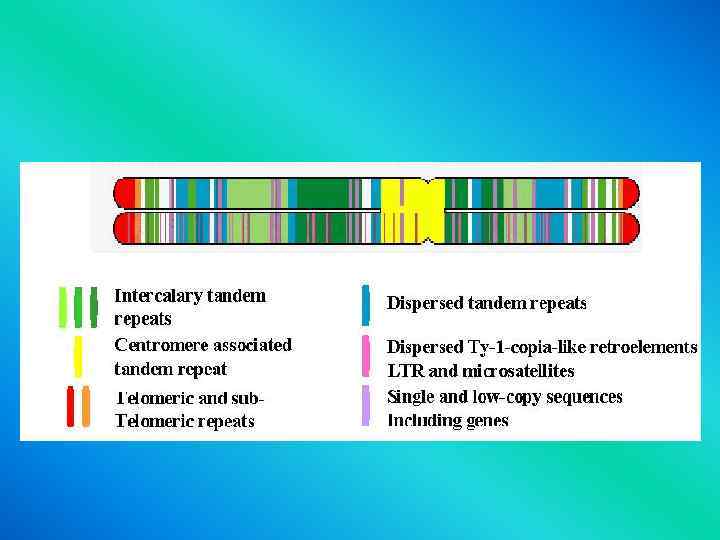

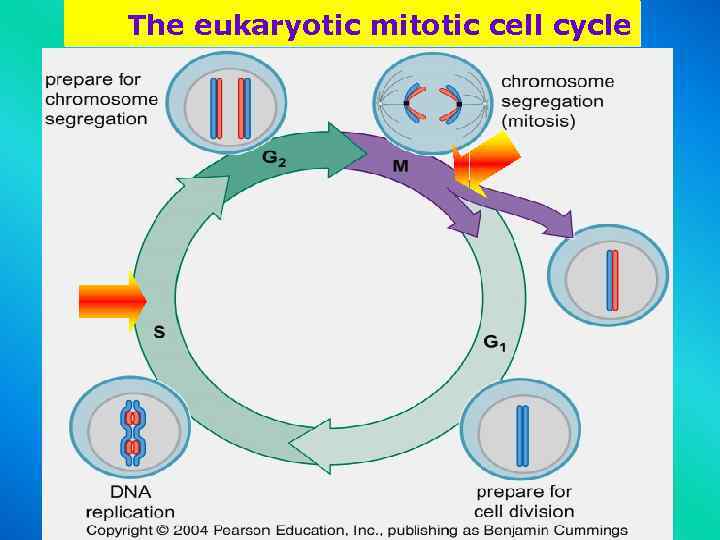 The eukaryotic mitotic cell cycle
The eukaryotic mitotic cell cycle
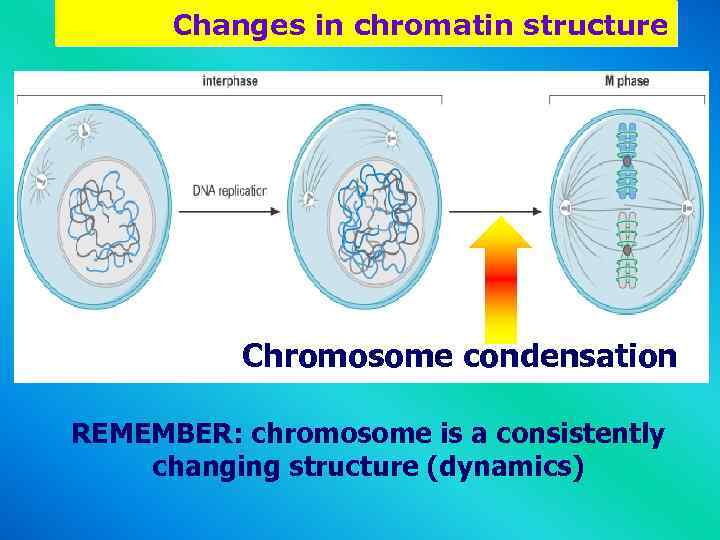 Changes in chromatin structure Chromosome condensation REMEMBER: chromosome is a consistently changing structure (dynamics)
Changes in chromatin structure Chromosome condensation REMEMBER: chromosome is a consistently changing structure (dynamics)
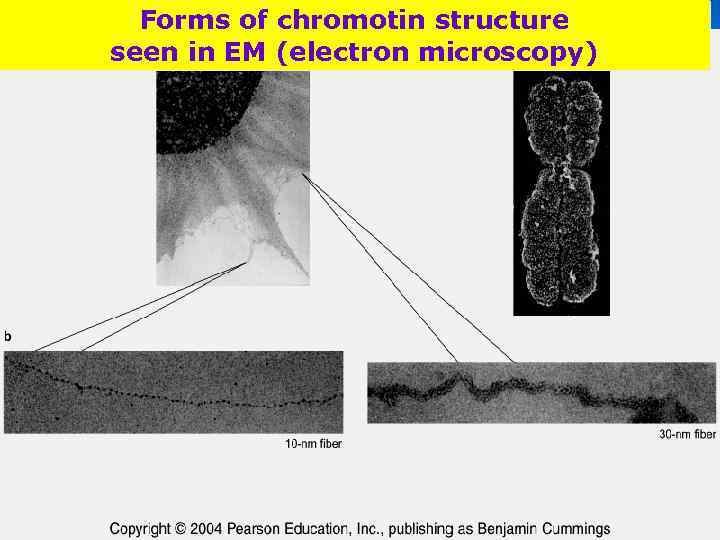 Forms of chromotin structure seen in EM (electron microscopy)
Forms of chromotin structure seen in EM (electron microscopy)
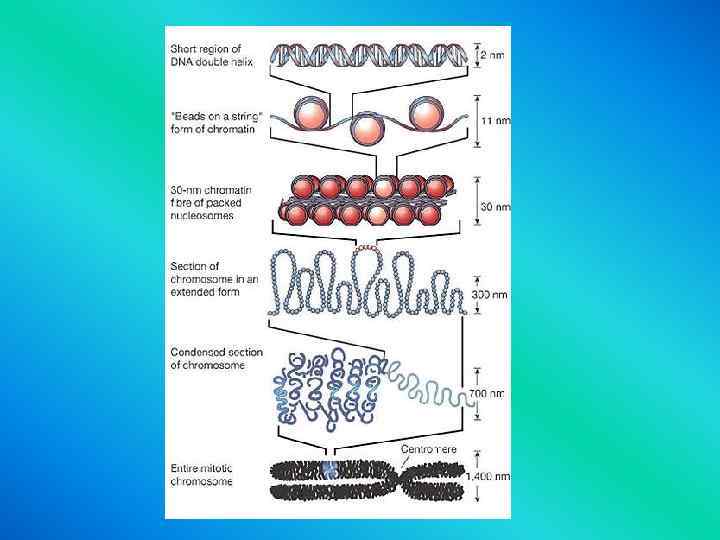
 Строение хромосомы
Строение хромосомы

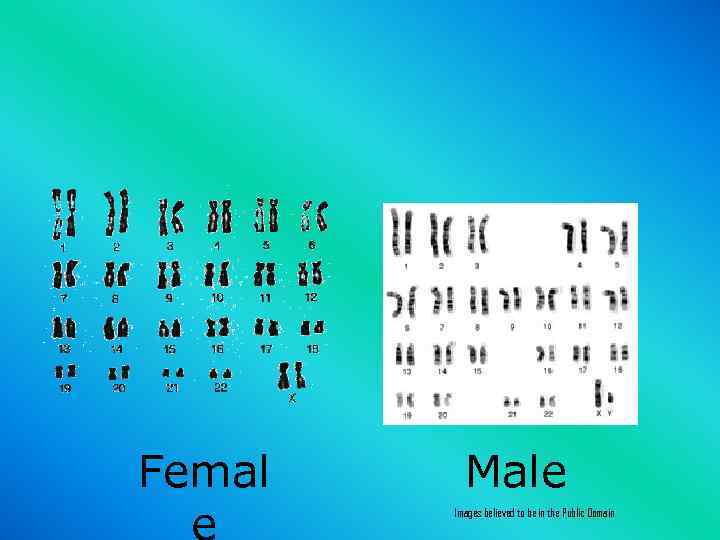 Femal Male Images believed to be in the Public Domain
Femal Male Images believed to be in the Public Domain
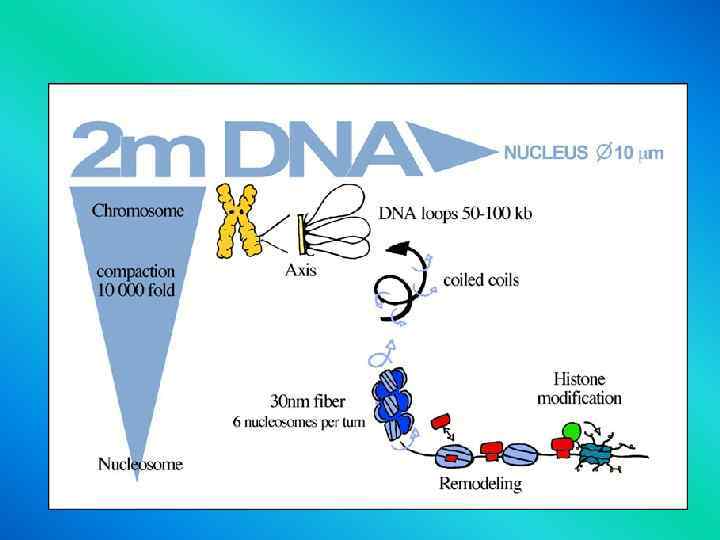
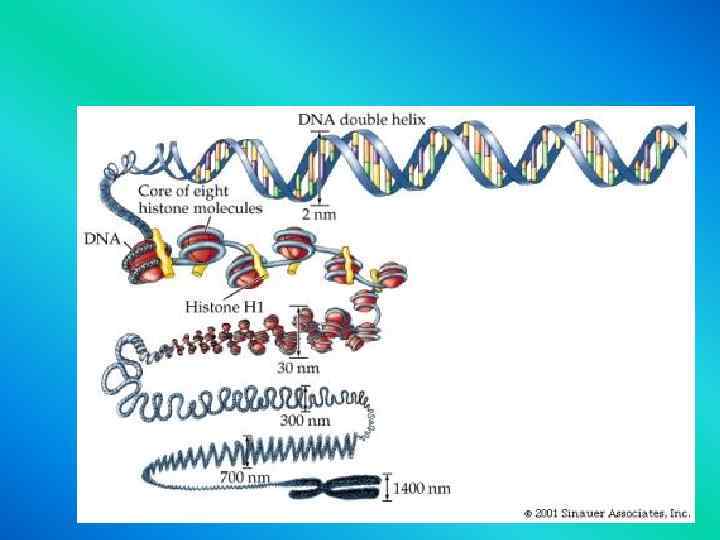
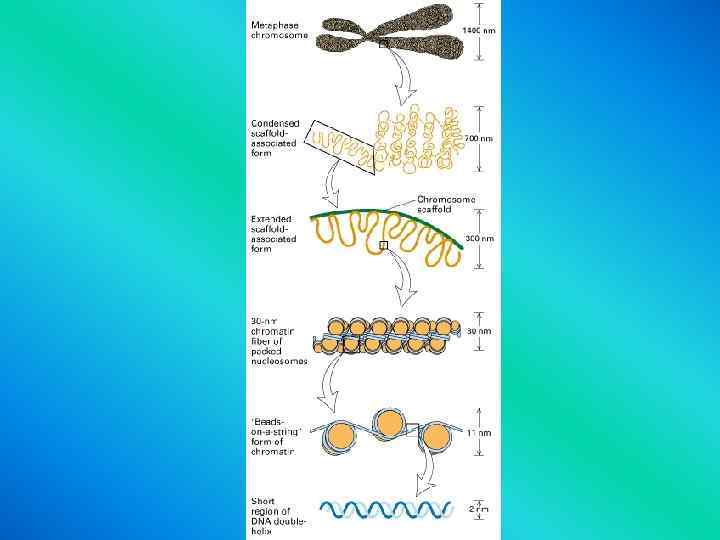
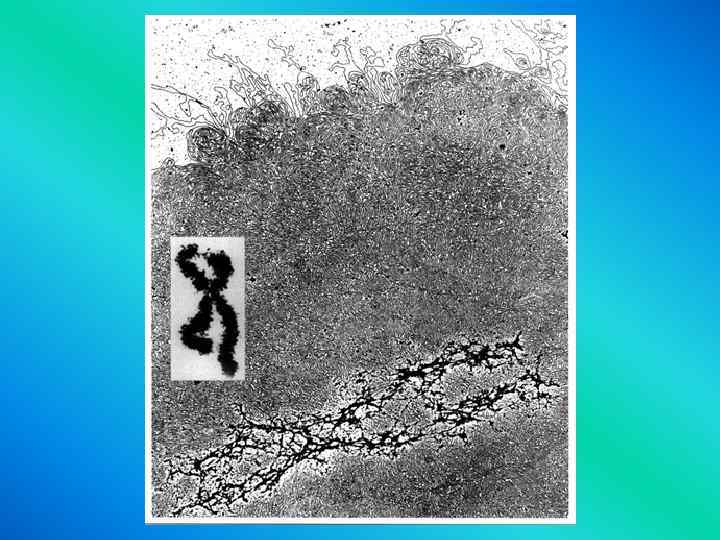
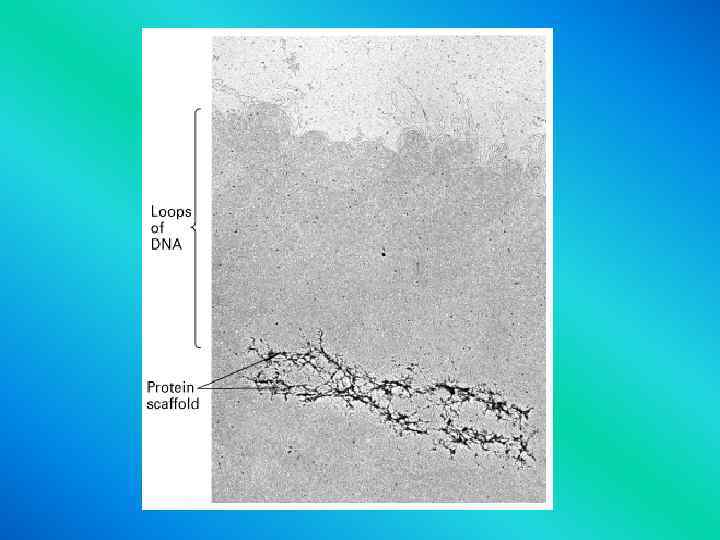
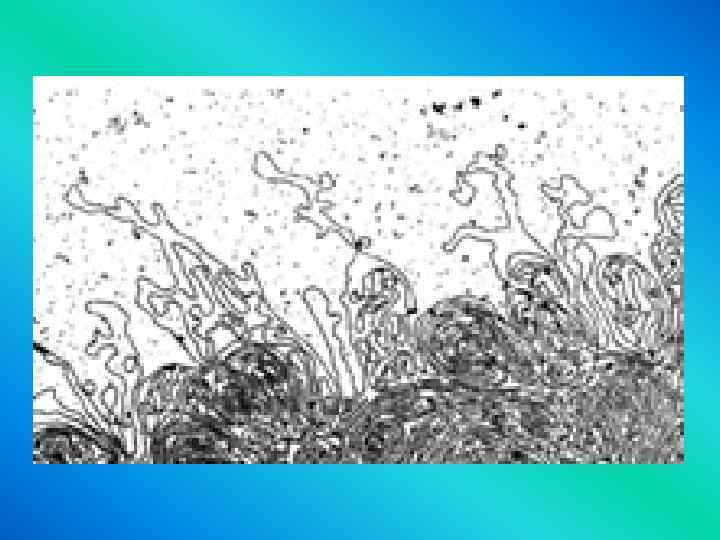
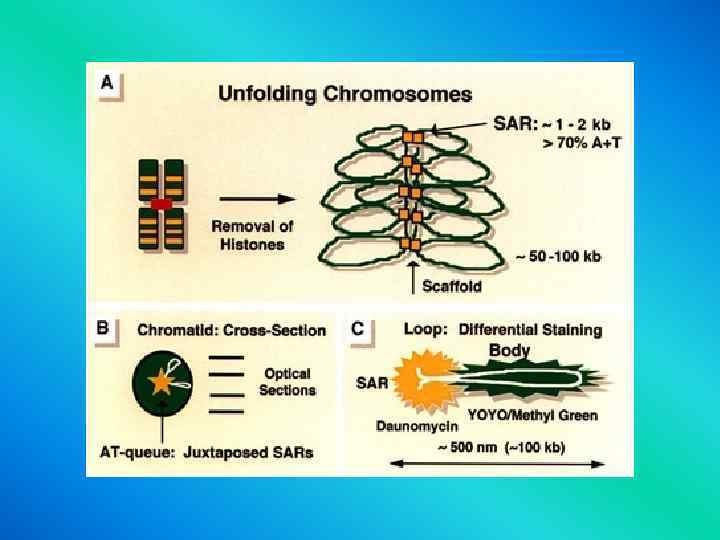
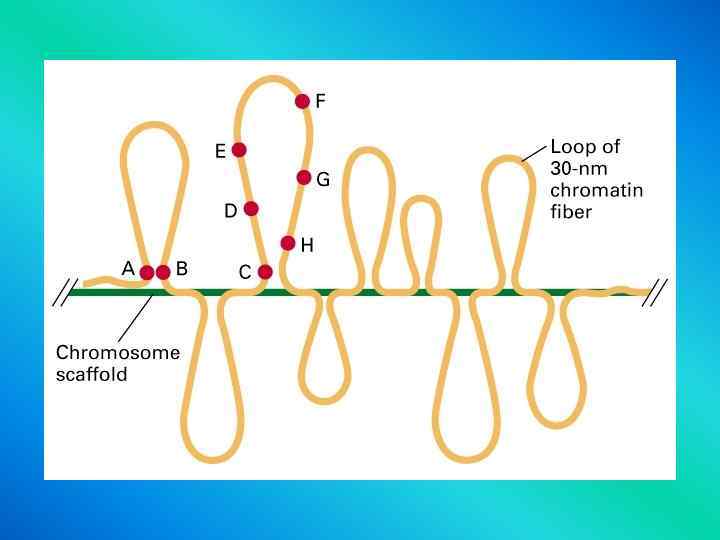
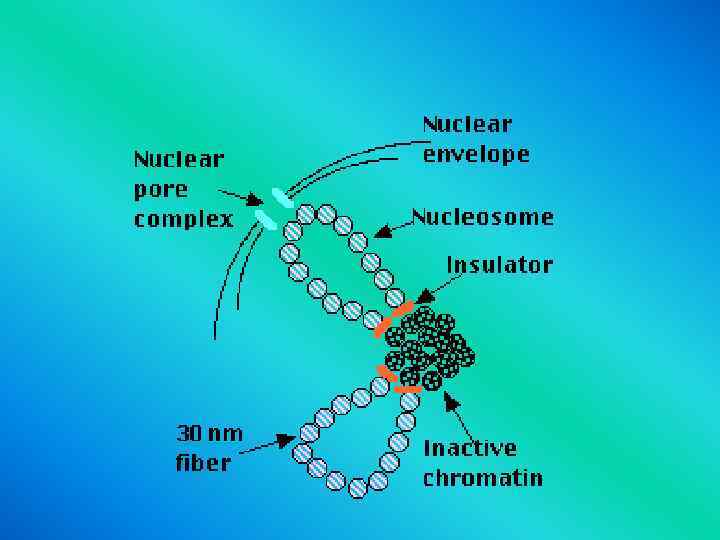
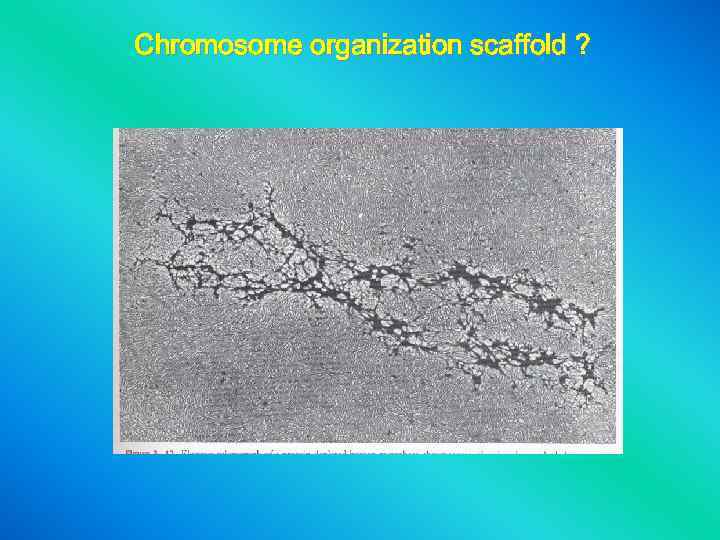 Chromosome organization scaffold ?
Chromosome organization scaffold ?
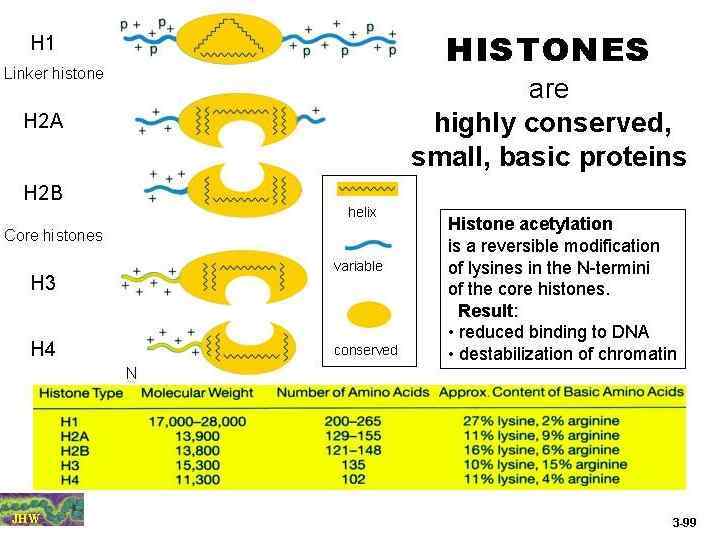
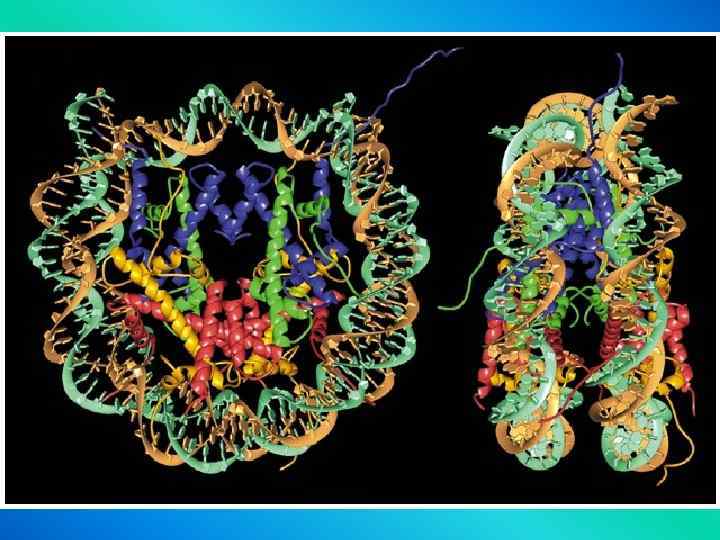
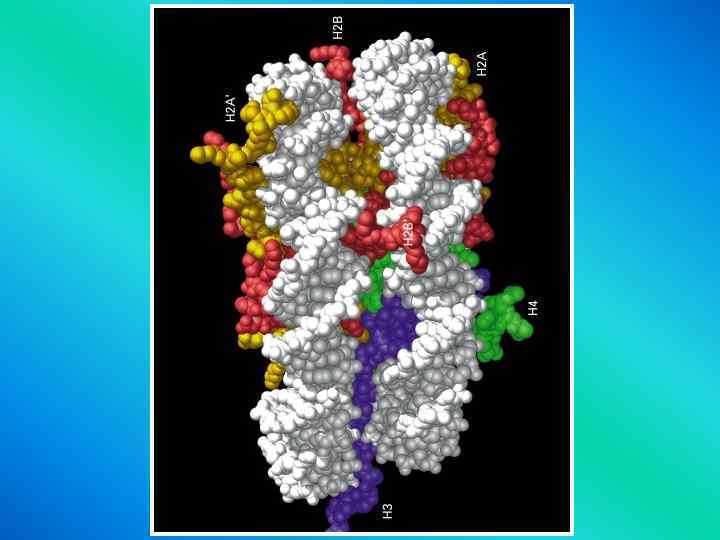
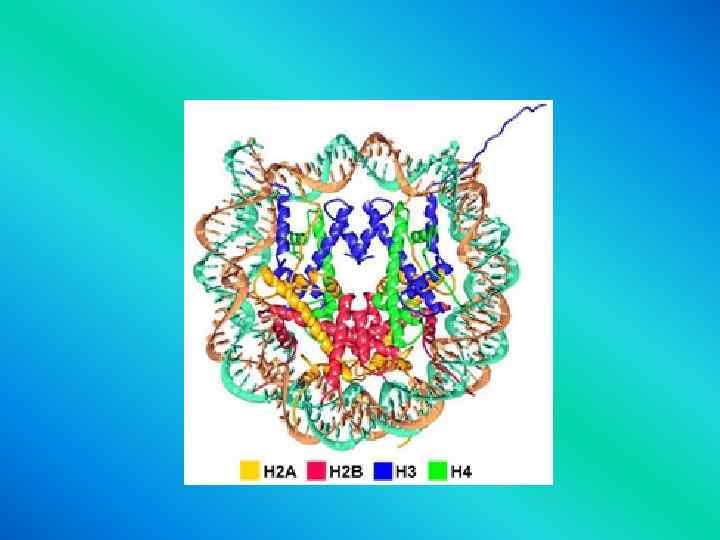

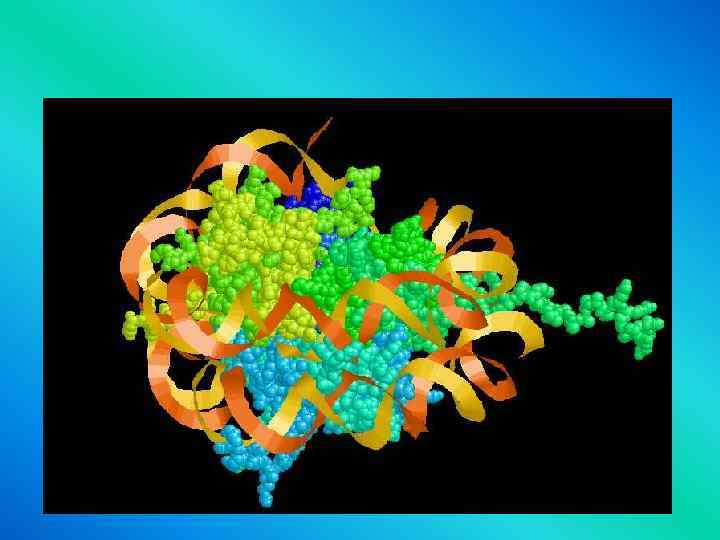
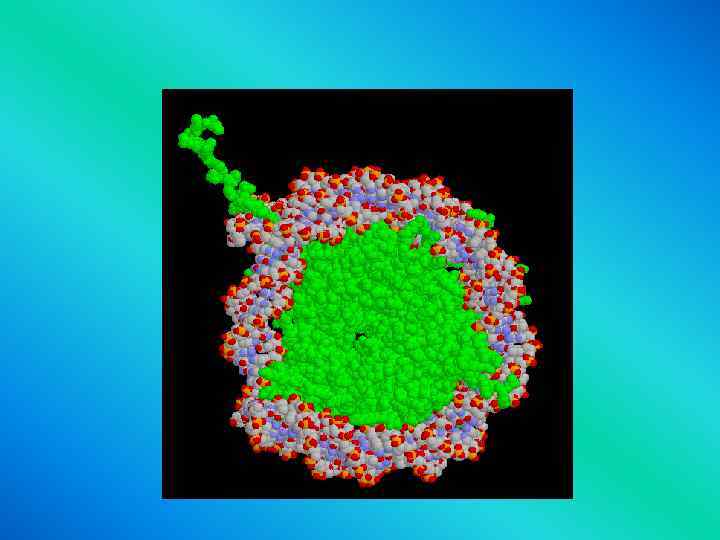
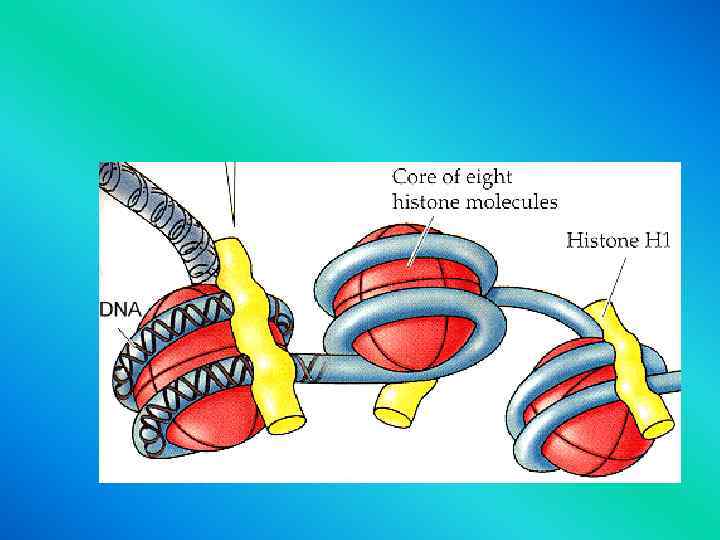
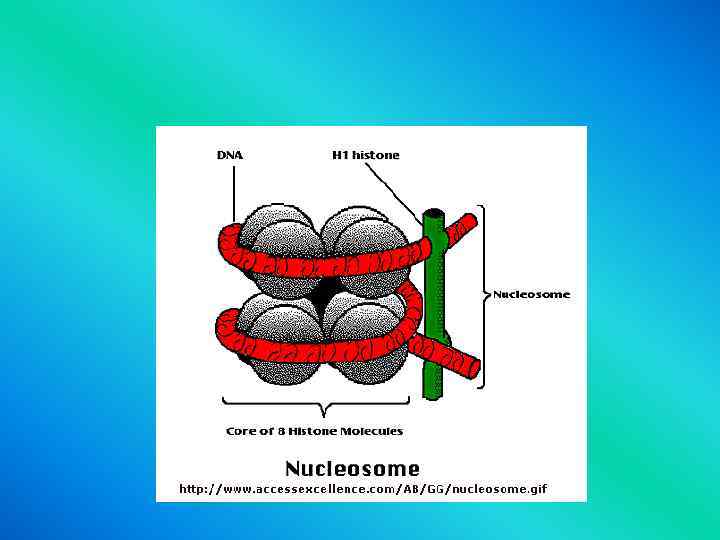
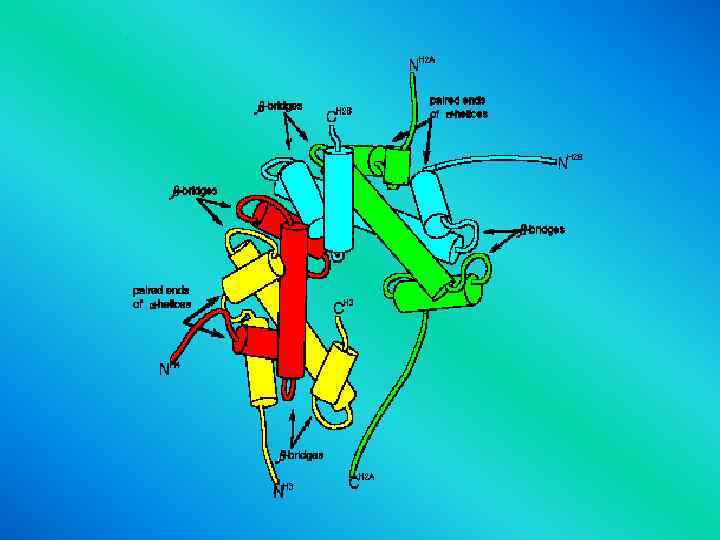
 • Histones are shown • in the following colors: • H 2 A - green H 2 B - blue H 3 - yellow H 4 - red H 5 - purple
• Histones are shown • in the following colors: • H 2 A - green H 2 B - blue H 3 - yellow H 4 - red H 5 - purple
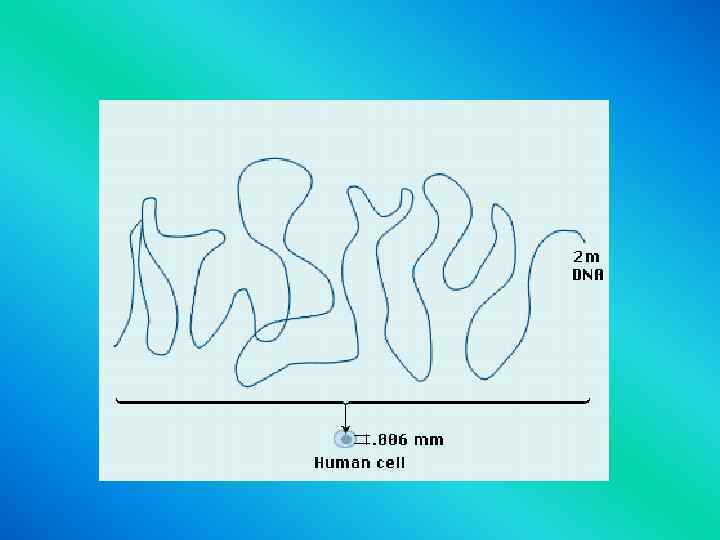
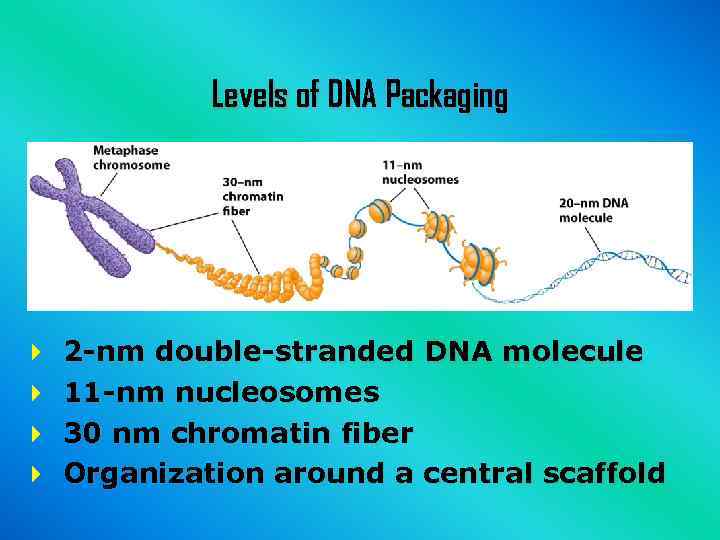 Levels of DNA Packaging 2 -nm double-stranded DNA molecule 11 -nm nucleosomes 30 nm chromatin fiber Organization around a central scaffold
Levels of DNA Packaging 2 -nm double-stranded DNA molecule 11 -nm nucleosomes 30 nm chromatin fiber Organization around a central scaffold
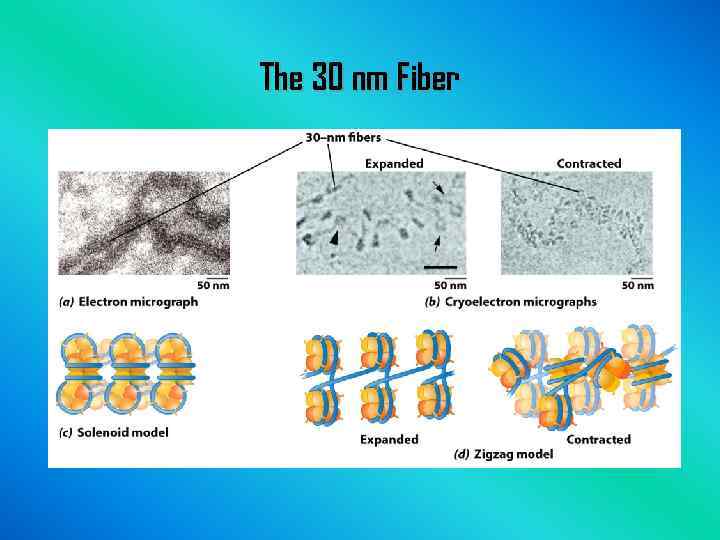 The 30 nm Fiber
The 30 nm Fiber
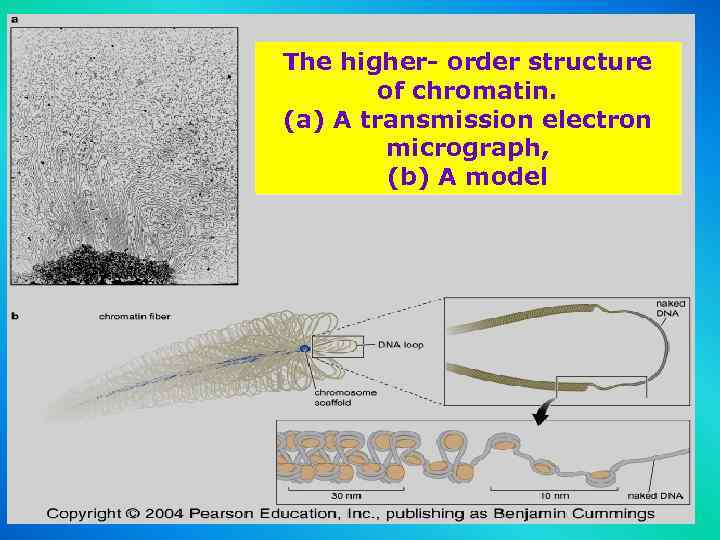 The higher- order structure of chromatin. (a) A transmission electron micrograph, (b) A model
The higher- order structure of chromatin. (a) A transmission electron micrograph, (b) A model
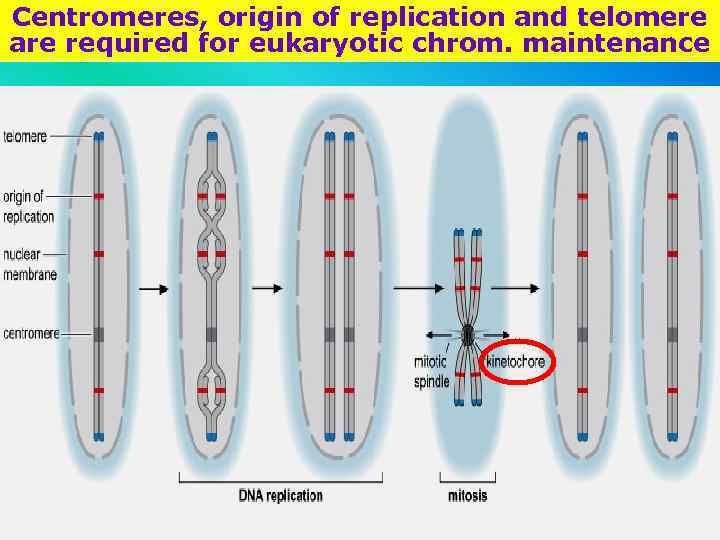 Centromeres, origin of replication and telomere are required for eukaryotic chrom. maintenance
Centromeres, origin of replication and telomere are required for eukaryotic chrom. maintenance
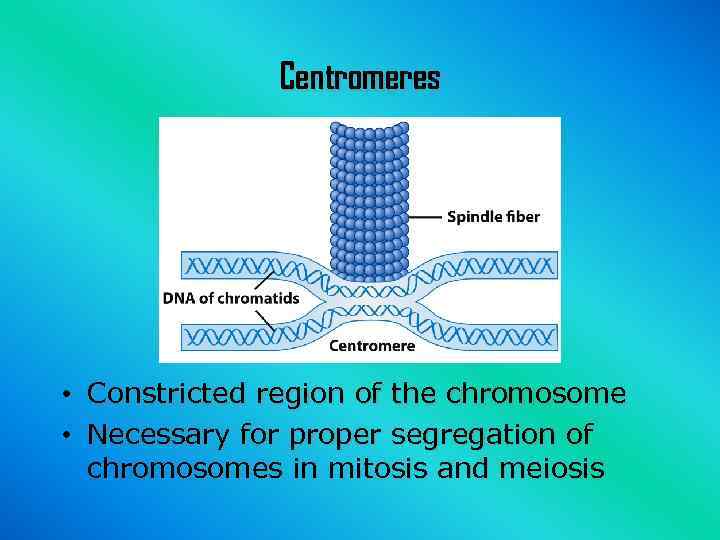 Centromeres • • Constricted region of the chromosome Necessary for proper segregation of chromosomes in mitosis and meiosis
Centromeres • • Constricted region of the chromosome Necessary for proper segregation of chromosomes in mitosis and meiosis
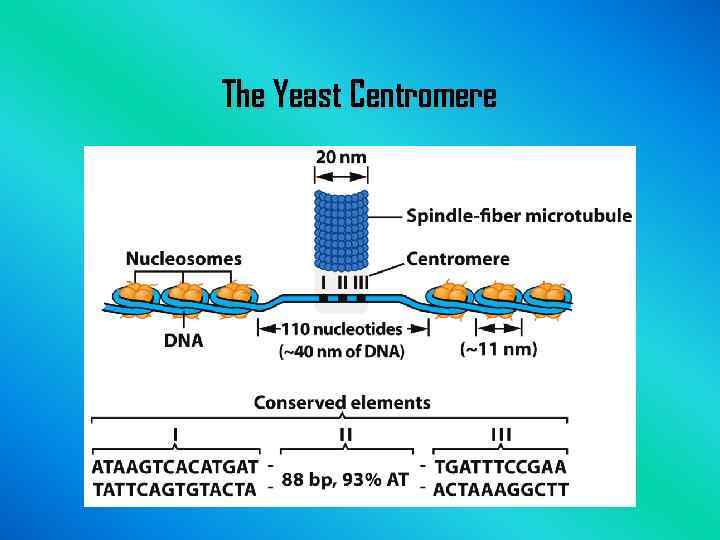 The Yeast Centromere
The Yeast Centromere
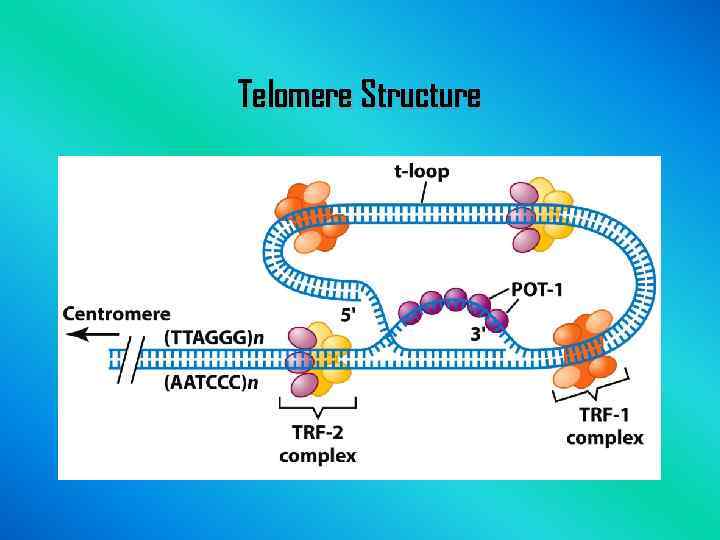 Telomere Structure
Telomere Structure
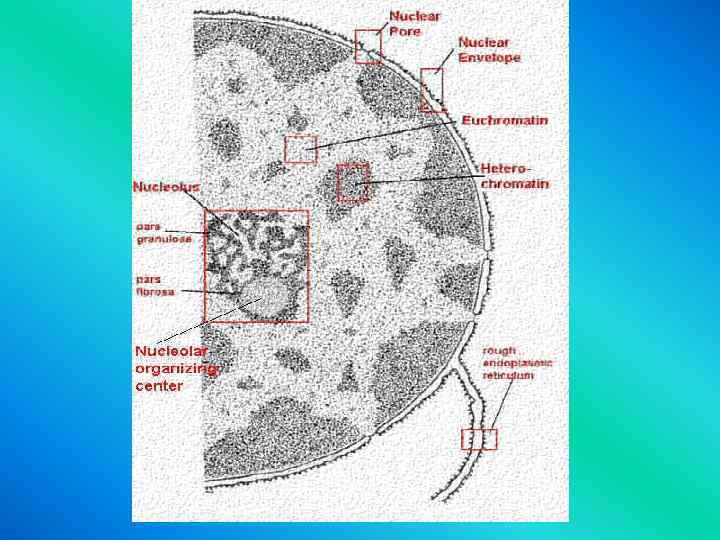
 Метилирование ДНК
Метилирование ДНК
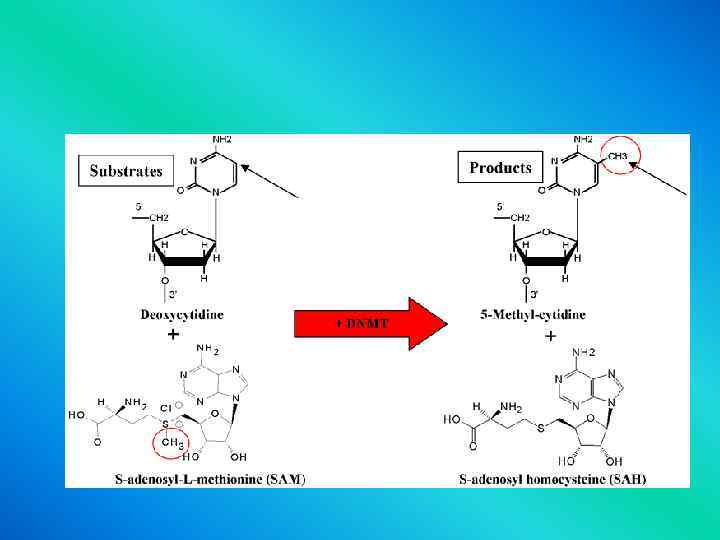
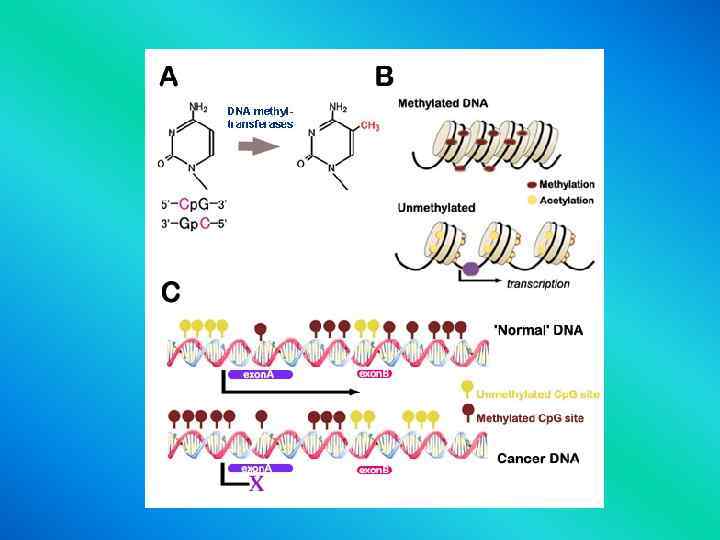
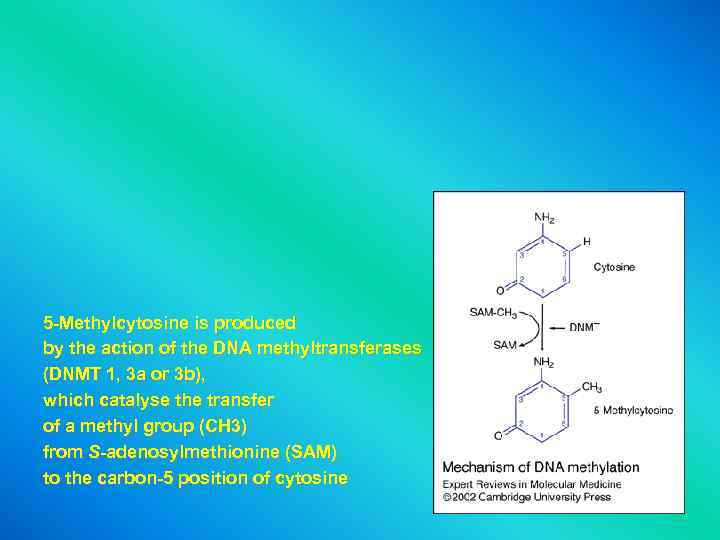 5 -Methylcytosine is produced by the action of the DNA methyltransferases (DNMT 1, 3 a or 3 b), which catalyse the transfer of a methyl group (CH 3) from S-adenosylmethionine (SAM) to the carbon-5 position of cytosine
5 -Methylcytosine is produced by the action of the DNA methyltransferases (DNMT 1, 3 a or 3 b), which catalyse the transfer of a methyl group (CH 3) from S-adenosylmethionine (SAM) to the carbon-5 position of cytosine
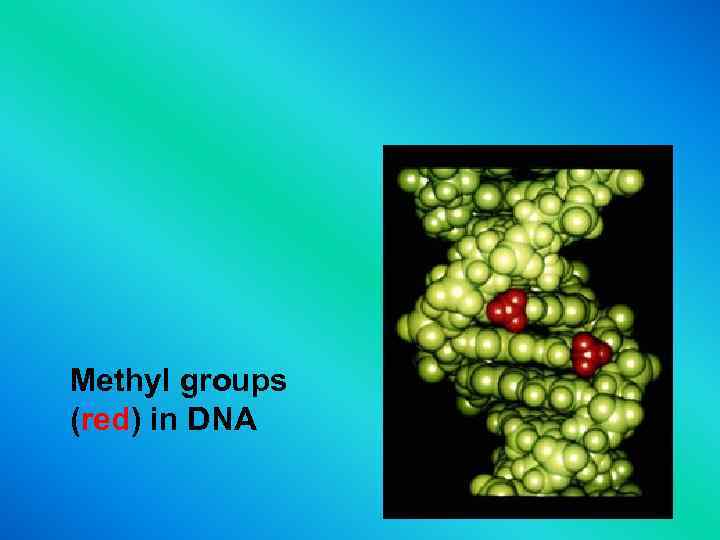 Methyl groups (red) in DNA red
Methyl groups (red) in DNA red
 Functions of DNA methylation in mammals Transcriptional gene silencing Chromatin compaction Genome stability Suppression of homologous recombination between repeats Genome defense X chromosome inactivation (females)
Functions of DNA methylation in mammals Transcriptional gene silencing Chromatin compaction Genome stability Suppression of homologous recombination between repeats Genome defense X chromosome inactivation (females)
 • Генетика предполагает, • а эпигенетика располагает“. • П. и Д. Медавары • В ДНК прокариот • модифицированы N 6 -метиладенин и 5 -метилцитозин • В ДНК высших эукариот - в основном 5 -метилцитозин • При метилировании • нуклеотидная последовательность ДНК не меняется, • метилирование – эпигенетическое событие. • • • Метилирование - стабильно наследуемая модификация, Которая обратима под воздействием деметилирующих агентов или ферментов и тем самым принципиально отличается от мутаций в ДНК.
• Генетика предполагает, • а эпигенетика располагает“. • П. и Д. Медавары • В ДНК прокариот • модифицированы N 6 -метиладенин и 5 -метилцитозин • В ДНК высших эукариот - в основном 5 -метилцитозин • При метилировании • нуклеотидная последовательность ДНК не меняется, • метилирование – эпигенетическое событие. • • • Метилирование - стабильно наследуемая модификация, Которая обратима под воздействием деметилирующих агентов или ферментов и тем самым принципиально отличается от мутаций в ДНК.
 • Метилирование – • 1) широкомасштабное подавление транскрпиции – компактизация хроматина, • 2) локальное (точечное) – препятствие взаимодействию с факторами транскрипции • Более 50% генов содержат Cp. G-островки: • Cp. G-островки есть, по-видимому, у всех housekeeping genes генов, • и у примерно 40% генов, выполняющих специализированные функции • Значительная часть тканеспецифических генов не имеет в своих Cp. G- островков
• Метилирование – • 1) широкомасштабное подавление транскрпиции – компактизация хроматина, • 2) локальное (точечное) – препятствие взаимодействию с факторами транскрипции • Более 50% генов содержат Cp. G-островки: • Cp. G-островки есть, по-видимому, у всех housekeeping genes генов, • и у примерно 40% генов, выполняющих специализированные функции • Значительная часть тканеспецифических генов не имеет в своих Cp. G- островков
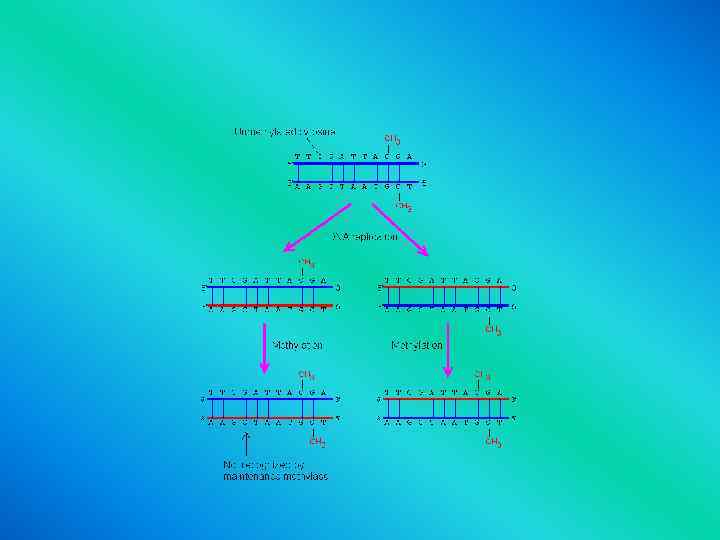
 • Волны метилирования • Во время эмбриогенеза закономерно сменяют друга "волны" метилирования- деметилирования • В итоге устанавливается строго определенный рисунок метилирования конкретных генов и генома в целом. • Он стабильно сохраняется в популяциях соматических клеток. • На стадии 1 -2 -клеточных делений идет тотальное деметилирование генома, устраняющее профиль, сформированный • в исходных половых клетках. • Оно продолжается до стадии • имплантации бластоциста
• Волны метилирования • Во время эмбриогенеза закономерно сменяют друга "волны" метилирования- деметилирования • В итоге устанавливается строго определенный рисунок метилирования конкретных генов и генома в целом. • Он стабильно сохраняется в популяциях соматических клеток. • На стадии 1 -2 -клеточных делений идет тотальное деметилирование генома, устраняющее профиль, сформированный • в исходных половых клетках. • Оно продолжается до стадии • имплантации бластоциста
 • «Активное деметилирование « генома , • остатки 5 -метилцитозина • удаляются из ДНК независимо от репликации • благодаря активности ДНК-деметилаз. • «Пассивное деметилирование» • подавление сопряженного с репликацией "поддерживающего" метилирования • (maitenance methylation) • ДНК-деметилаза узнает метилированные Cp. G • и трансформирует 5 -Ме. С в цитозин, не нарушая целостности ДНК
• «Активное деметилирование « генома , • остатки 5 -метилцитозина • удаляются из ДНК независимо от репликации • благодаря активности ДНК-деметилаз. • «Пассивное деметилирование» • подавление сопряженного с репликацией "поддерживающего" метилирования • (maitenance methylation) • ДНК-деметилаза узнает метилированные Cp. G • и трансформирует 5 -Ме. С в цитозин, не нарушая целостности ДНК
 • Глобальное метилирование генома • Начинается после имплантации бластоциста (метилирование de поvо, • профиль которого в целом сохраняется в соматических клетках взрослой особи) • • Метилируется геном • на стадии постимплантационного бластоциста в два этапа: • - собственно метилирование de novo, затрагивающее локальные участки и • - распространение (spreading) "пятна" метилирования на близлежащие области
• Глобальное метилирование генома • Начинается после имплантации бластоциста (метилирование de поvо, • профиль которого в целом сохраняется в соматических клетках взрослой особи) • • Метилируется геном • на стадии постимплантационного бластоциста в два этапа: • - собственно метилирование de novo, затрагивающее локальные участки и • - распространение (spreading) "пятна" метилирования на близлежащие области
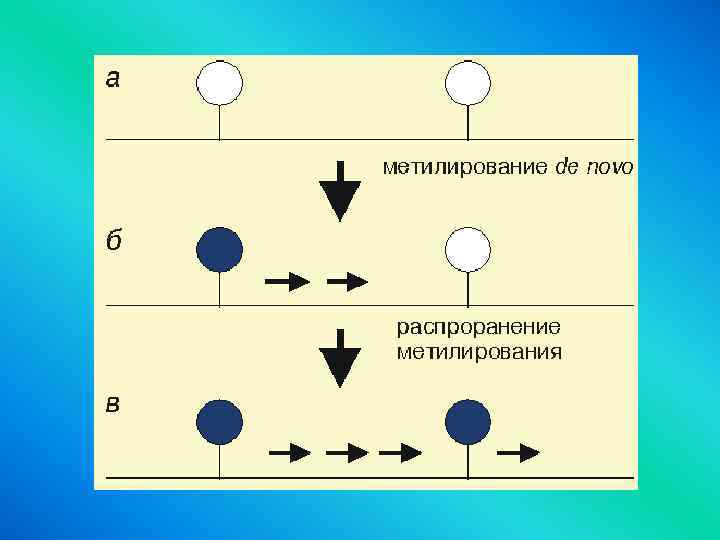
 • Две системы метилирования • Метилирование de novo, • вносящее элементы изменчивости • в профиль метилирования • Поддерживающее метилирование • функция – консервация • уже сформированного • профиля метилирования
• Две системы метилирования • Метилирование de novo, • вносящее элементы изменчивости • в профиль метилирования • Поддерживающее метилирование • функция – консервация • уже сформированного • профиля метилирования
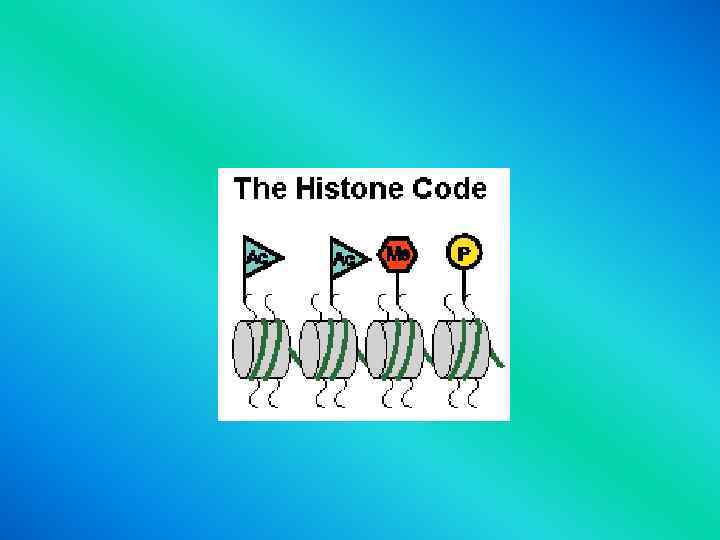
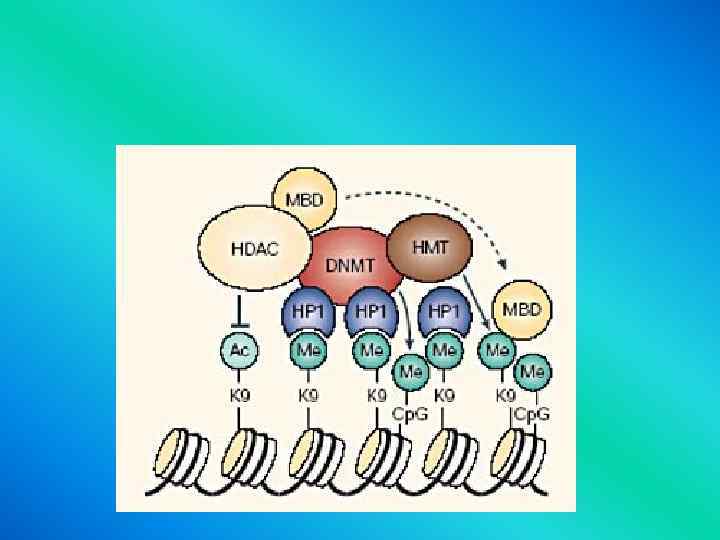
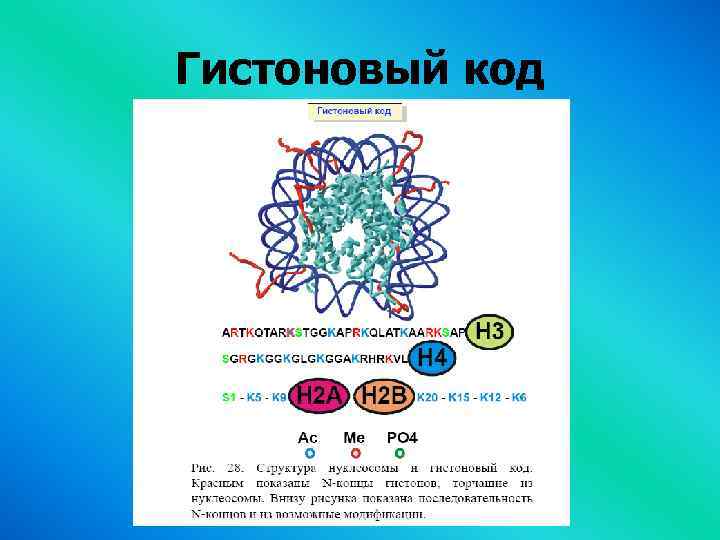 Гистоновый код
Гистоновый код

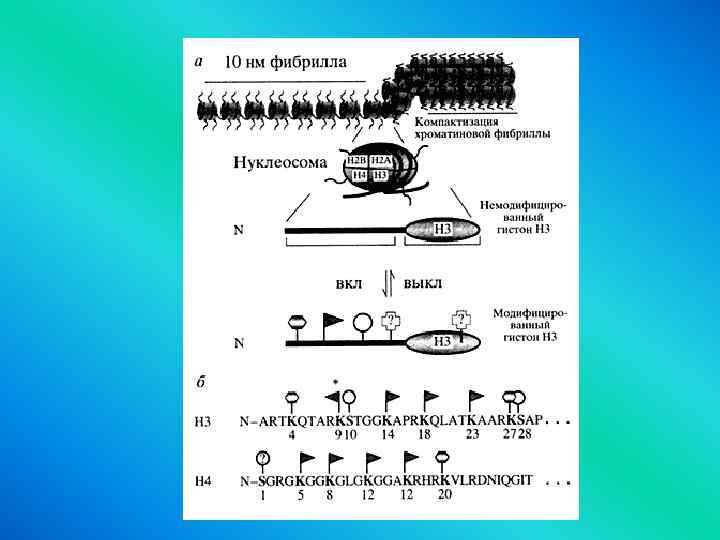
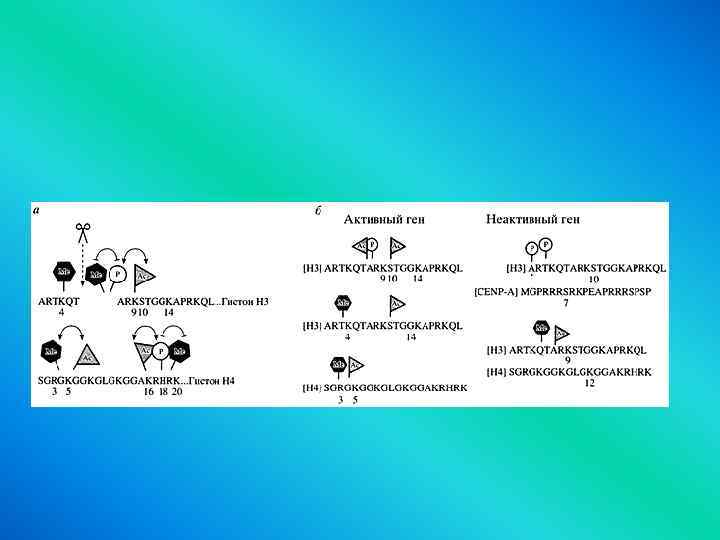
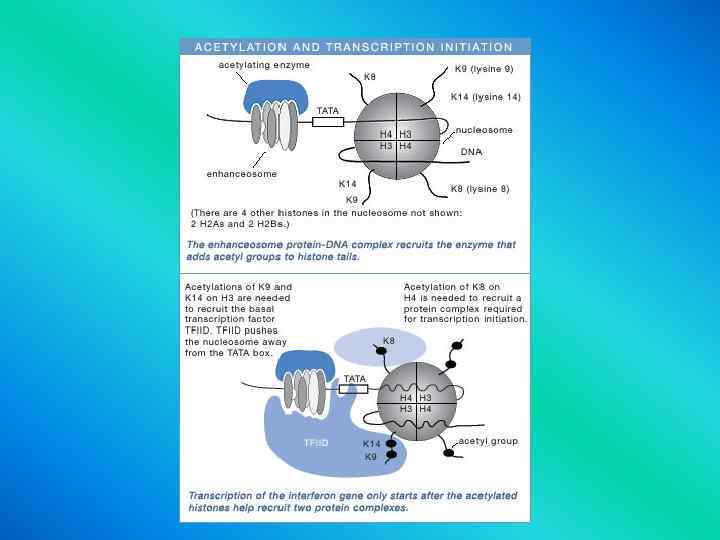
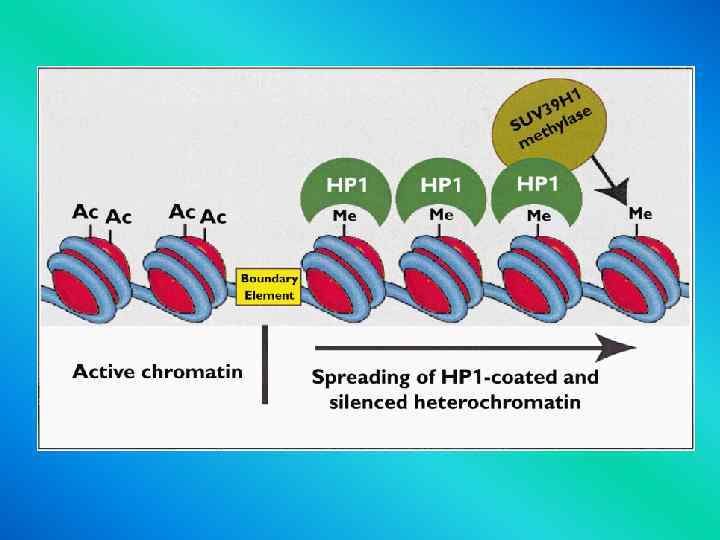
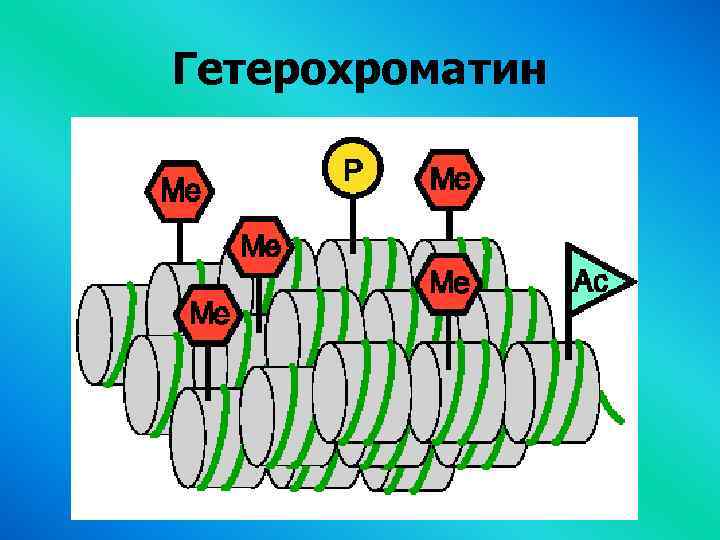 Гетерохроматин
Гетерохроматин
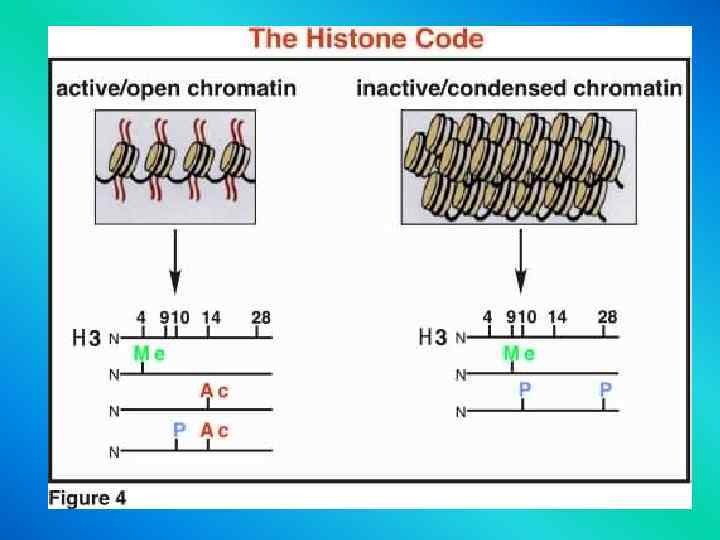
 Methylation of histone or of DNA usually turns a gene off. Acetylation of histone usually turns a gene on. Phosphorylation -- we're not sure what that does
Methylation of histone or of DNA usually turns a gene off. Acetylation of histone usually turns a gene on. Phosphorylation -- we're not sure what that does
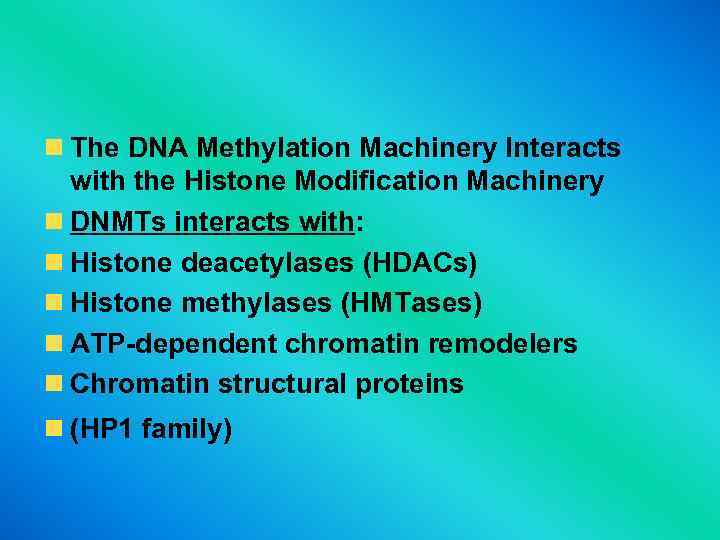 The DNA Methylation Machinery Interacts with the Histone Modification Machinery DNMTs interacts with: Histone deacetylases (HDACs) Histone methylases (HMTases) ATP-dependent chromatin remodelers Chromatin structural proteins (HP 1 family)
The DNA Methylation Machinery Interacts with the Histone Modification Machinery DNMTs interacts with: Histone deacetylases (HDACs) Histone methylases (HMTases) ATP-dependent chromatin remodelers Chromatin structural proteins (HP 1 family)
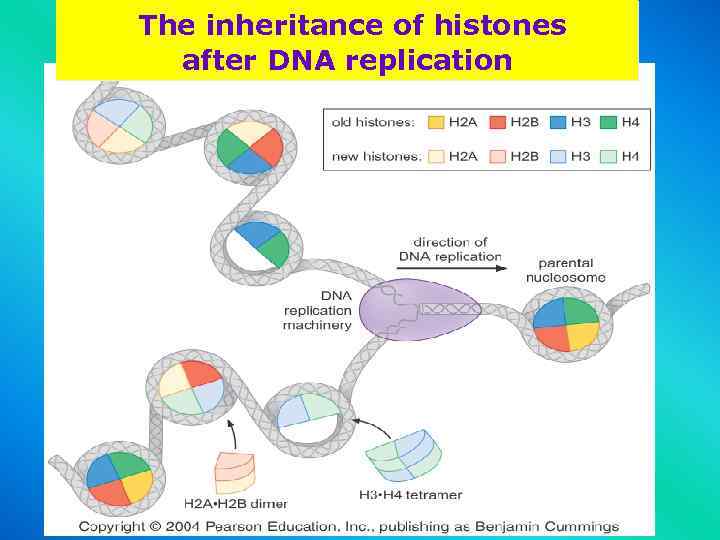 The inheritance of histones after DNA replication
The inheritance of histones after DNA replication
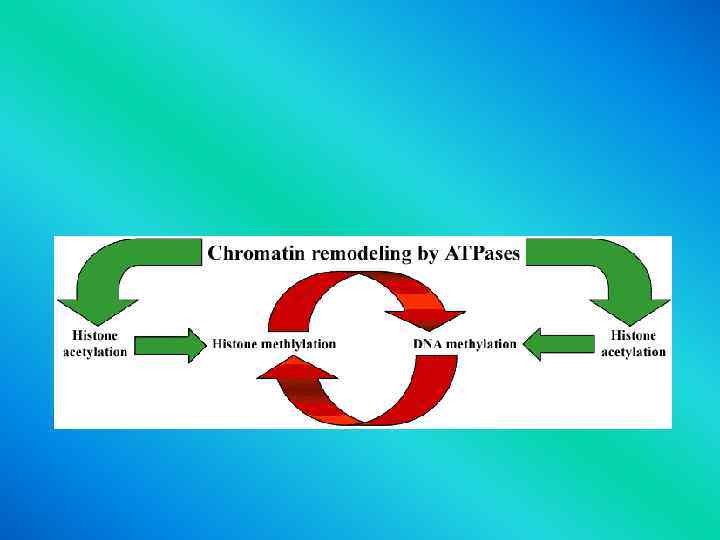
 Хроматин Cooperative and self-reinforcing organization of the chromatin & DNA-modifying machinery responsible for gene silencing in normal & malignant cells
Хроматин Cooperative and self-reinforcing organization of the chromatin & DNA-modifying machinery responsible for gene silencing in normal & malignant cells
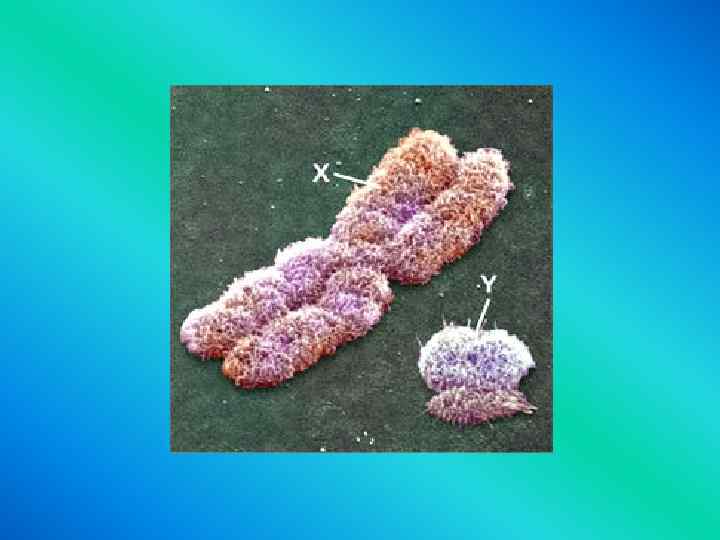

 Y chromosome Adam Mitochondrial Eve Lived approximately 50, 000 years ago Lived 166, 000 -249, 000 years ago Thomson, R. et al. (2000) Proc Natl Acad Sci U S A 97, 7360 -5 Cann, R. L. et al. (1987) Nature 325, 31 -6 Underhill, P. A. et al. (2000) Nat Genet 26, 358 -61 Vigilant, L. et al. (1991) Science 253, 1503 -7 Albrecht Dürer, The Fall of Man, 1504 Adam and Eve never met
Y chromosome Adam Mitochondrial Eve Lived approximately 50, 000 years ago Lived 166, 000 -249, 000 years ago Thomson, R. et al. (2000) Proc Natl Acad Sci U S A 97, 7360 -5 Cann, R. L. et al. (1987) Nature 325, 31 -6 Underhill, P. A. et al. (2000) Nat Genet 26, 358 -61 Vigilant, L. et al. (1991) Science 253, 1503 -7 Albrecht Dürer, The Fall of Man, 1504 Adam and Eve never met



