T cell epitope prediction 2014.pptx
- Количество слайдов: 34

State Research Center of Virology and Biotechnology Vector Предсказание Т-клеточных эпитопов с помощью методов машинного обучения Антонец Денис Викторович Барнаул, 2014

Ø CD 8+ T-лимфоциты играют важную роль в защите организма от вирусов и онкологических заболеваний Ø Цитотоксические CD 8+ T-лимфоциты узнают короткие пептиды (8 -11 а. к. о. ) процессированных антигенов, презентируемые молекулами MHC I класса
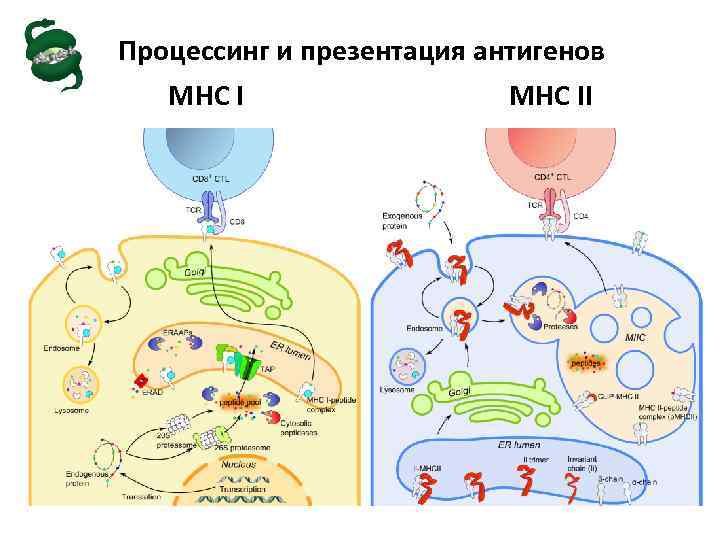
Процессинг и презентация антигенов MHC II

Ø CD 8+ T-лимфоциты играют важную роль в защите организма от вирусов и онкологических заболеваний Ø Цитотоксические CD 8+ T-лимфоциты узнают короткие пептиды (8 -11 а. к. о. ) процессированных антигенов, презентируемые молекулами MHC I класса Ø T клеточные эпитопы – пептиды, способные связывать молекулы MHC и активировать Т-клеточный ответ Ø T клеточные эпитопы используются для разработки новых полиэпитопных профилактических и иммунотерапевтических вакцин Ø Точное предсказание Т-клеточных эпитопов in silico в значительной степени сокращает материальные и временные затраты по сравнению с традиционными экспериментальными подходами
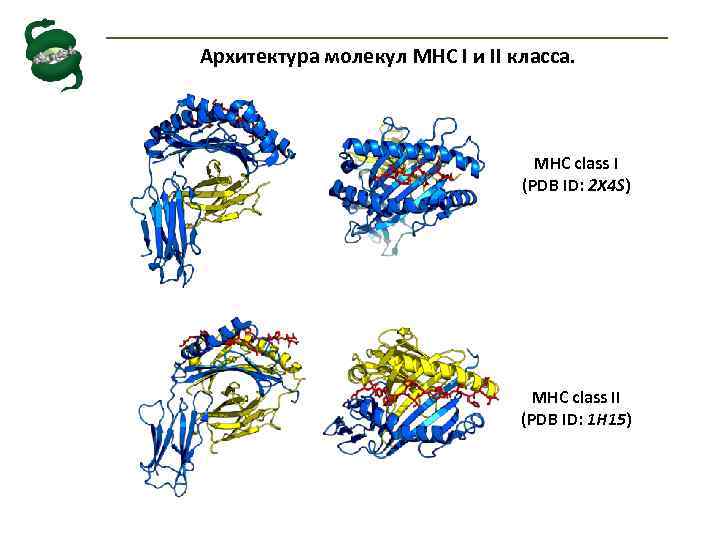
Архитектура молекул MHC I и II класса. MHC class I (PDB ID: 2 X 4 S) MHC class II (PDB ID: 1 H 15)
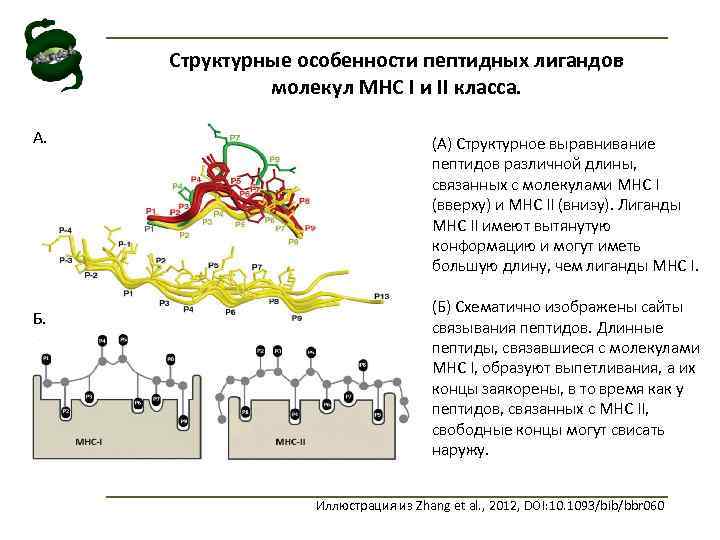
Структурные особенности пептидных лигандов молекул MHC I и II класса. A. Б. (A) Структурное выравнивание пептидов различной длины, связанных с молекулами MHC I (вверху) и MHC II (внизу). Лиганды MHC II имеют вытянутую конформацию и могут иметь большую длину, чем лиганды MHC I. (Б) Схематично изображены сайты связывания пептидов. Длинные пептиды, связавшиеся с молекулами MHC I, образуют выпетливания, а их концы заякорены, в то время как у пептидов, связанных с MHC II, свободные концы могут свисать наружу. Иллюстрация из Zhang et al. , 2012, DOI: 10. 1093/bib/bbr 060

Из всех возможных олигопептидов (число всех теоретически возможных нонапептидов составляет 209 – 5, 12× 1011) лишь около 3 % способны с достаточно высокой аффинностью связываться с молекулами MHC, и еще меньшее количество пептидов способно индуцировать Т-клеточный иммунный ответ (Assarsson et al. , 2007). Иммуногенность олигопептида в значительной степени определяется его способностью связываться с молекулой MHC (Lundegaard et al. 2010), следовательно, разработка моделей для точного предсказания пептидов, связывающихся с молекулами MHC, является необходимым условием надежного предсказания Т-клеточных эпитопов.

Подходы к предсказанию Т-клеточных эпитопов 1. Олигопептиды, способные связываться с молекулами MHC, имеют структуру амфипатической спирали (Berkower et al. , 1986). Уже в 1988 г. была показана его несостоятельность (Adorini et al. , 1988 b, 1988 a; Oldstone et al. , 1988; Rothbard, Taylor, 1988). 2. Поиск «якорных мотивов» – аминокислотных мотивов, характерных для пептидов, способных связываться с данной молекулой MHC (Falk et al. , 1991). SYFPEITHI (Rammensee et al. , 1999).

Подходы к предсказанию Т-клеточных эпитопов 3. Построение матриц с весовыми коэффициентами для различных аминокислот, занимающих определенные позиции в олигопептиде (Reche et al. , 2004, Parker et al. , 1994; Singh, Raghava, 2001). 4. Применение искусственных нейронных сетей и метода опорных векторов как для классификации пептидов (Dönnes, Elofsson, 2002), так и для построения функциональной зависимости меры аффинности от аминокислотной последовательности пептида (Nielsen et al. , 2004 b; Wan et al. , 2006). 5. Использование для предсказаний моделей пространственных структур комплексов пептидов с молекулами MHC.
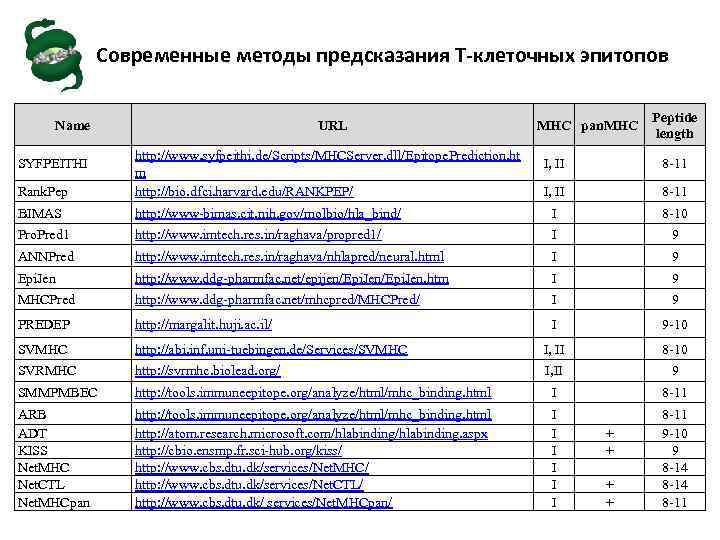
Современные методы предсказания Т-клеточных эпитопов Name URL MHC pan. MHC Peptide length Rank. Pep http: //www. syfpeithi. de/Scripts/MHCServer. dll/Epitope. Prediction. ht m http: //bio. dfci. harvard. edu/RANKPEP/ BIMAS http: //www-bimas. cit. nih. gov/molbio/hla_bind/ I 8 -10 Pro. Pred 1 http: //www. imtech. res. in/raghava/propred 1/ I 9 ANNPred http: //www. imtech. res. in/raghava/nhlapred/neural. html I 9 Epi. Jen http: //www. ddg-pharmfac. net/epijen/Epi. Jen. htm I 9 MHCPred http: //www. ddg-pharmfac. net/mhcpred/MHCPred/ I 9 PREDEP http: //margalit. huji. ac. il/ I 9 -10 SVMHC http: //abi. inf. uni-tuebingen. de/Services/SVMHC I, II 8 -10 SVRMHC http: //svrmhc. biolead. org/ I, II 9 SMMPMBEC http: //tools. immuneepitope. org/analyze/html/mhc_binding. html I 8 -11 ARB ADT KISS Net. MHC Net. CTL Net. MHCpan http: //tools. immuneepitope. org/analyze/html/mhc_binding. html http: //atom. research. microsoft. com/hlabinding. aspx http: //cbio. ensmp. fr. sci-hub. org/kiss/ http: //www. cbs. dtu. dk/services/Net. MHC/ http: //www. cbs. dtu. dk/services/Net. CTL/ http: //www. cbs. dtu. dk/ services/Net. MHCpan/ I I I 8 -11 9 -10 9 8 -14 8 -11 SYFPEITHI I, II 8 -11 + +

MHC binding affinity: where p. IC 50 is negative decimal logarithm of half maximal inhibitory concentration (in n. M) of the peptide, Pi is the certain amino acid residue at position i within the peptide and αi is coefficient, const – is the intercept Peptides with p. IC 50 values > 6. 3 were considered as MHC-binders

Перед построением моделей необходимо параметризовать пептиды A C D E F G H I K L M N P Q R S T V W Y 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 SYFPEITHI S Y F P E I T H I 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 1 0 0 0 Простейший способ параметризации – факторизация аминокислотных остатков All scales used here can be found at http: //tepredict. sourceforge. net/Used. Scales
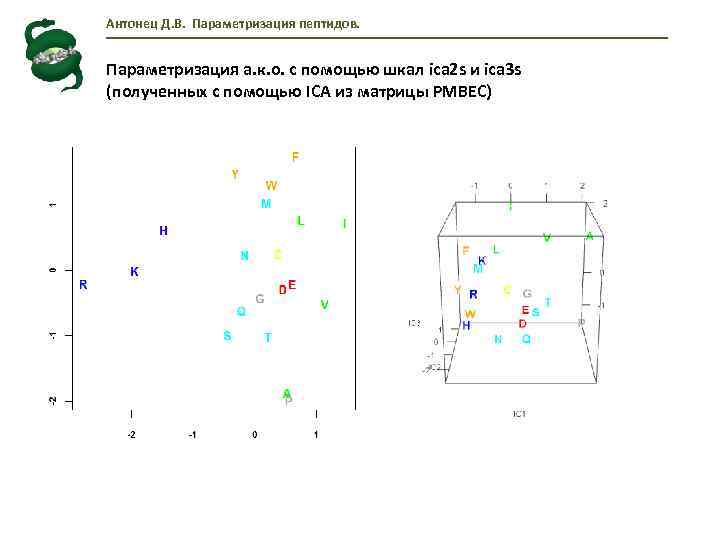
Антонец Д. В. Параметризация пептидов. Параметризация а. к. о. с помощью шкал ica 2 s и ica 3 s (полученных с помощью ICA из матрицы PMBEC)
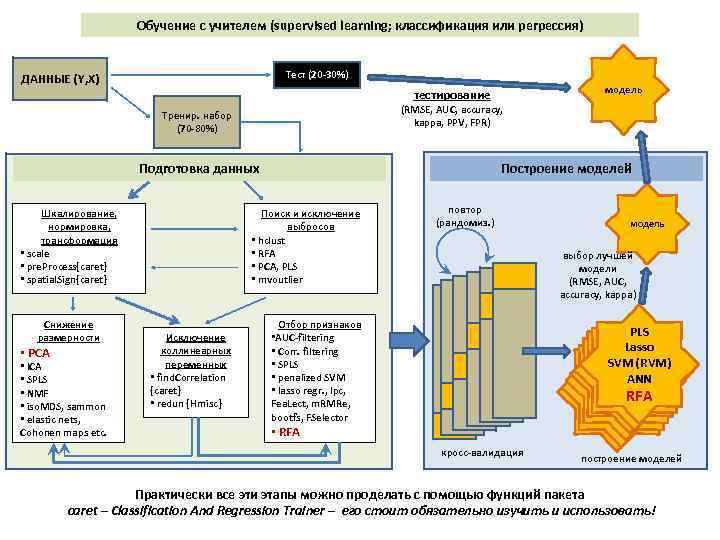
Обучение с учителем (supervised learning; классификация или регрессия) Тест (20 -30%) ДАННЫЕ (Y, X) (RMSE, AUC, accuracy, kappa, PPV, FPR) Тренир. набор (70 -80%) Подготовка данных Шкалирование, нормировка, трансформация • scale • pre. Process{caret} • spatial. Sign{caret} Снижение размерности • PCA • ICA • SPLS • NMF • iso. MDS, sammon • elastic nets, Cohonen maps etc. модель тестирование Построение моделей Поиск и исключение выбросов • hclust • RFA • PCA, PLS • mvoutlier Исключение коллинеарных переменных • find. Correlation {caret} • redun {Hmisc} повтор (рандомиз. ) модель выбор лучшей модели (RMSE, AUC, accuracy, kappa) Отбор признаков • AUC-filtering • Corr. filtering • SPLS • penalized SVM • lasso regr. , lpc, Fea. Lect, m. RMRe, bootfs, FSelector PLS PLS Lasso PLS (RVM) SVM Lasso SVM(RVM) Lasso SVM ANN Lasso(RVM) SVM ANN (RVM) SVM(RVM) ANN SVM ANN RFA RFA • RFA кросс-валидация построение моделей Практически все эти этапы можно проделать с помощью функций пакета caret – Classification And Regression Trainer – его стоит обязательно изучить и использовать!

Качество предсказаний оценивалось с помощью: 1. Специфичность TN/(TN + FP), (1 - FP/(TN+FP)) 2. Чувствительность TP/(TP + FN), (1 - FN/(TP+FN)); 3. AUC (Area Under the Curve) – area under Reciever Oparational Curve (Specificity vs. Sensitivity plot) 4. MSEP (Mean Squared Error of Prediction) – среднеквадратичная ошибка между предсказанными и измеренными значениями p. IC 50 5. Коэффициент корреляции Пирсона между предсказанными и измеренными значениями p. IC 50
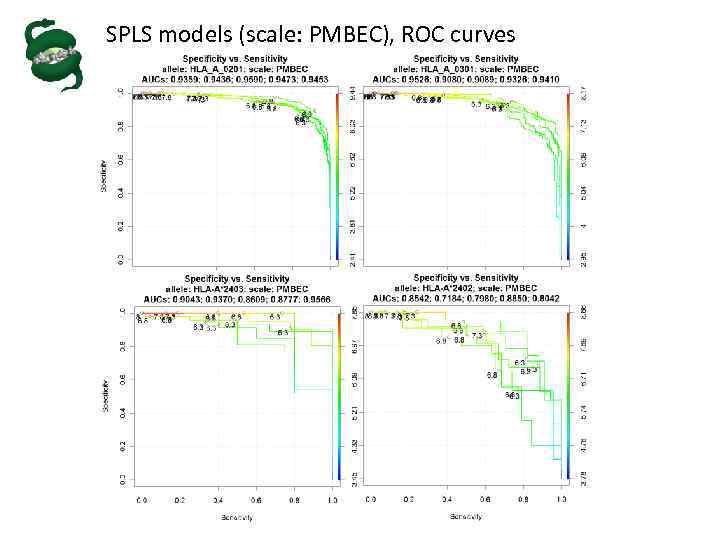
SPLS models (scale: PMBEC), ROC curves
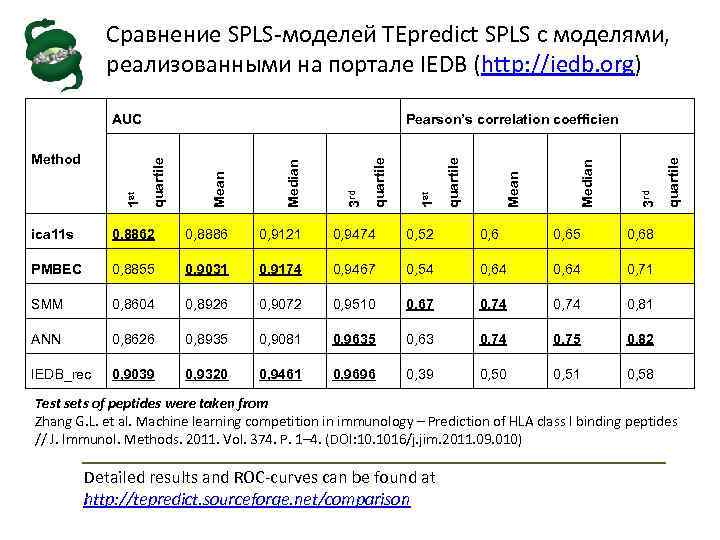
Сравнение SPLS-моделей TEpredict SPLS с моделями, реализованными на портале IEDB (http: //iedb. org) 0, 52 0, 65 0, 68 PMBEC 0, 8855 0, 9031 0, 9174 0, 9467 0, 54 0, 64 0, 71 SMM 0, 8604 0, 8926 0, 9072 0, 9510 0, 67 0, 74 0, 81 ANN 0, 8626 0, 8935 0, 9081 0, 9635 0, 63 0, 74 0, 75 0, 82 IEDB_rec 0, 9039 0, 9320 0, 9461 0, 9696 0, 39 0, 50 0, 51 quartile 0, 9474 0, 58 3 rd 0, 9121 Mean 0, 8886 1 st Median quartile 0, 8862 3 rd Median ica 11 s 1 st Mean Method Pearson’s correlation coefficien quartile AUC Test sets of peptides were taken from Zhang G. L. et al. Machine learning competition in immunology – Prediction of HLA class I binding peptides // J. Immunol. Methods. 2011. Vol. 374. P. 1– 4. (DOI: 10. 1016/j. jim. 2011. 09. 010) Detailed results and ROC-curves can be found at http: //tepredict. sourceforge. net/comparison

Разработка пан-MHC специфичных моделей для предсказания аффинности связывания олигопептидов с молекулами МНС Ø В базе данных IMGT/HLA содержится уже 11000 последовательностей различных аллельных вариантов молекул HLA Ø На сегодняшний день известно 7778 аллельных вариантов молекул HLA-A, HLA-B, и HLA-C Ø IEDB – наиболее полный источник результатов экспериментальных измерений аффинности связывания олигопептидов с молекулами MHC. Лишь для 50 аллельных вариантов молекул HLA I класса! Ø Аллельные варианты HLA могут быть сгруппированы на основе сходства их последовательностей и/или их специфичности по отношению к пептидам

Разработка пан-MHC специфичных моделей Lundegaard et al. , Immunome Res. 2010, 6 Suppl. 2: S 3. doi: 10. 1186/1745 -7580 -6 -S 2 -S 3.
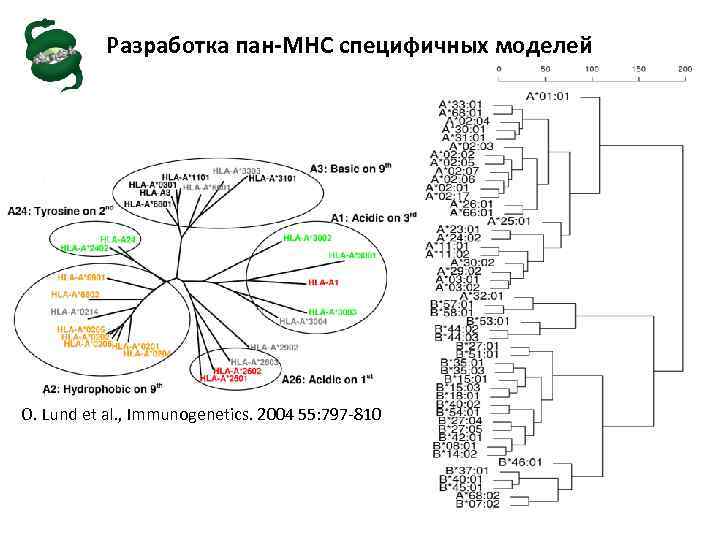
Разработка пан-MHC специфичных моделей O. Lund et al. , Immunogenetics. 2004 55: 797 -810

Developing pan-MHC specific models for predicting peptide-MHC binding affinity ei , {xi, yi} where ei – experimentally measured p. IC 50 value, x i – peptide, y i – HLA allele where ω is the {x, y} pair The joint kernel K can be decomposed as the product of Kx and Ky :

Developing pan-MHC specific models for predicting peptide-MHC binding affinity GSK has the following advantages: Ø it can take into account mutual similarity of amino acid residues Ø it can be used to compare peptides of different lengths GS kernel function was developed by Gigure et al. Giguère S, Marchand M, Laviolette F, Drouin A, Corbeil J. BMC Bioinformatics. 2013, 14: 82

Developing pan-MHC specific models for predicting peptide-MHC binding affinity Ø MHC-peptide binding data was selected from IEDB. 18094 peptide, HLA combinations were chosen for the training set, and 7696 – for testing. 50 unique HLA alleles 7310 unique peptides (with 68 8 aa peptides, 4397 – 9 aa, 2802 – 10 aa and 43 – 11 aa) Ø The model was built using RVM (relevance vector machine) technique realized in R package kernlab final model contained 3238 relevance vectors
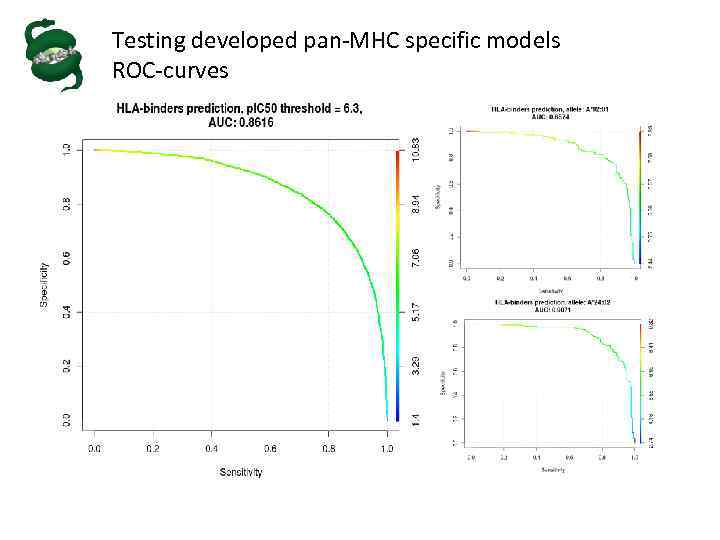
Testing developed pan-MHC specific models ROC-curves

Testing developed pan-MHC specific models Median Pearson’s corr. coeff. value (by HL A allele) was 0. 7324, the mean was 0. 7248 Median AUC values (by HL A allele) was 0. 8622, mean AUC was 0. 8490 Poor results were obtained for the following HLA alleles (Pearson’s coefficient < 0. 5): HLA-A*02: 17, -A*03: 02, -A*66: 01, -B*14: 02, B*35: 03, -B*42: 01 Thus, new RVM-based model demonstrated reasonable performance, which is comparable to our previous allele-specific models. Advantages: 1. It can be applied for peptides of any length 2. Predictions can be done for any HLA allele with known amino acid sequence.
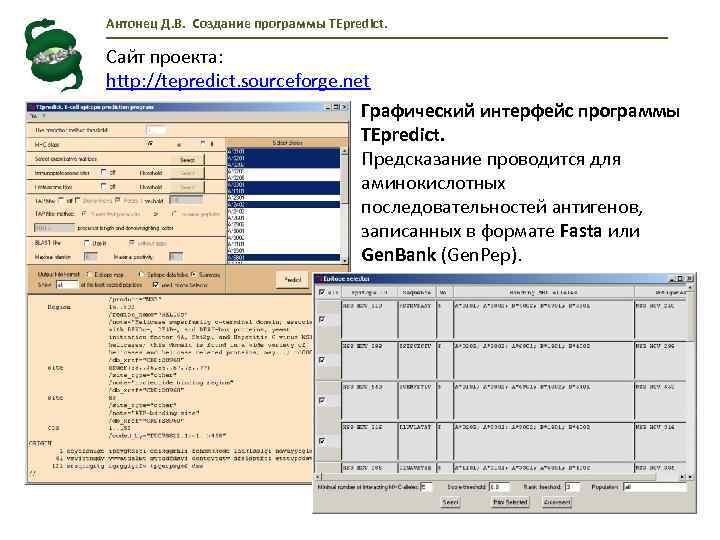
Антонец Д. В. Создание программы TEpredict. Сайт проекта: http: //tepredict. sourceforge. net Графический интерфейс программы TEpredict. Предсказание проводится для аминокислотных последовательностей антигенов, записанных в формате Fasta или Gen. Bank (Gen. Pep).
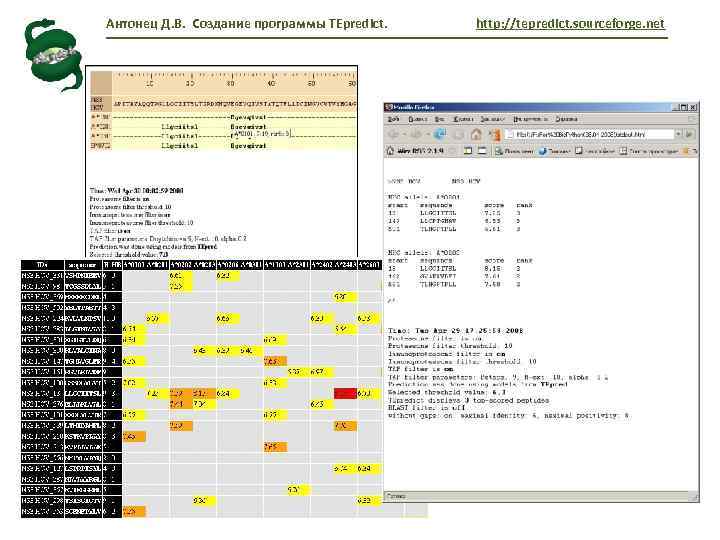
Антонец Д. В. Создание программы TEpredict. http: //tepredict. sourceforge. net

Антонец Д. В. Создание программы TEpredict. Ø Ø Ø http: //tepredict. sourceforge. net TEpredict имеет ряд дополнительных возможностей: предсказание протеасомного и/или иммунопротеасомного процессинга антигенов; предсказание аффинности связывания олигопептидов с TAP; выбор минимального количества олигопептидов, покрывающих заданный репертуар аллельных вариантов молекул HLA I с необходимым уровнем избыточности; исключение пептидов, имеющих значительное локальное сходство с белками человека (с помощью BLAST); автоматизация анализа, поскольку помимо графического интерфейса программа имеет интерфейс командной строки, и может быть использована как библиотека языка Python; помимо оригинальных моделей в программе реализованы методы Pro. Pred 1, n. HLAPred и Pro. Pred.
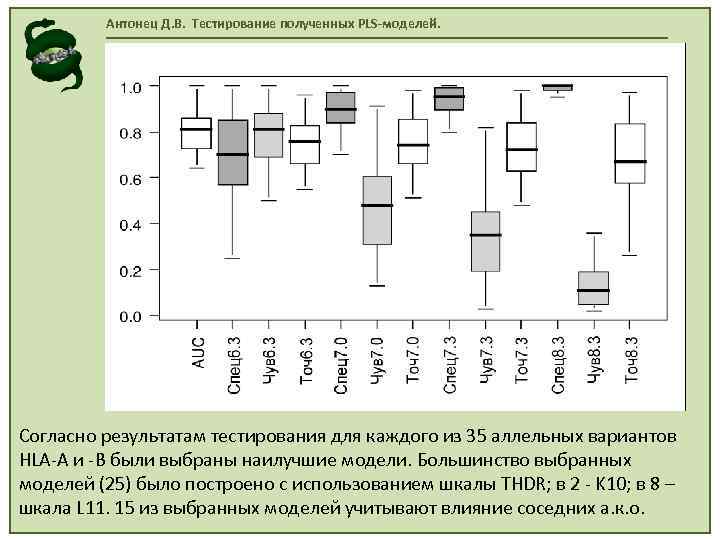
Антонец Д. В. Тестирование полученных PLS-моделей. Согласно результатам тестирования для каждого из 35 аллельных вариантов HLA-A и -B были выбраны наилучшие модели. Большинство выбранных моделей (25) было построено с использованием шкалы THDR; в 2 - K 10; в 8 – шкала L 11. 15 из выбранных моделей учитывают влияние соседних а. к. о.
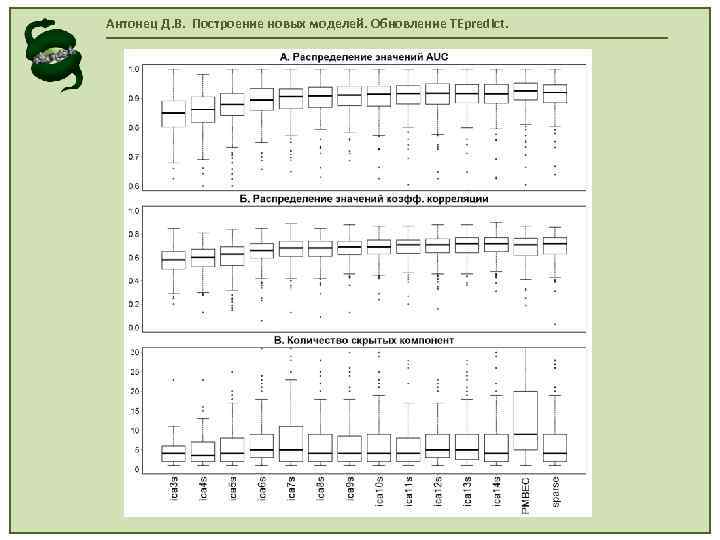
Антонец Д. В. Построение новых моделей. Обновление TEpredict.
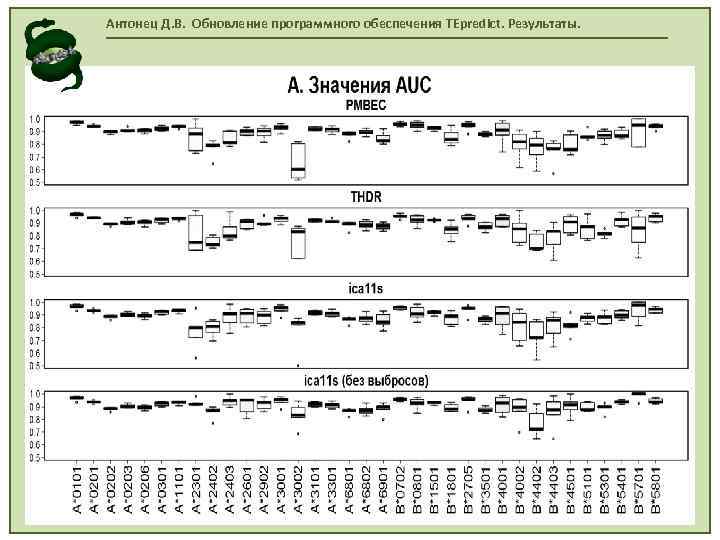
Антонец Д. В. Обновление программного обеспечения TEpredict. Результаты.
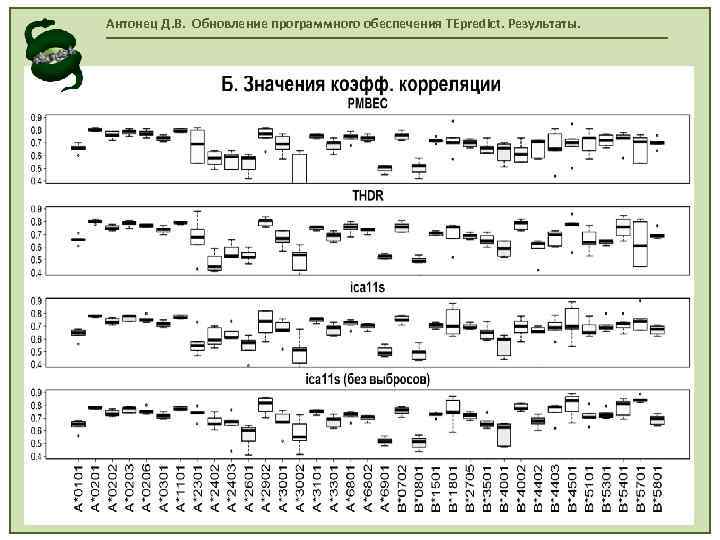
Антонец Д. В. Обновление программного обеспечения TEpredict. Результаты.
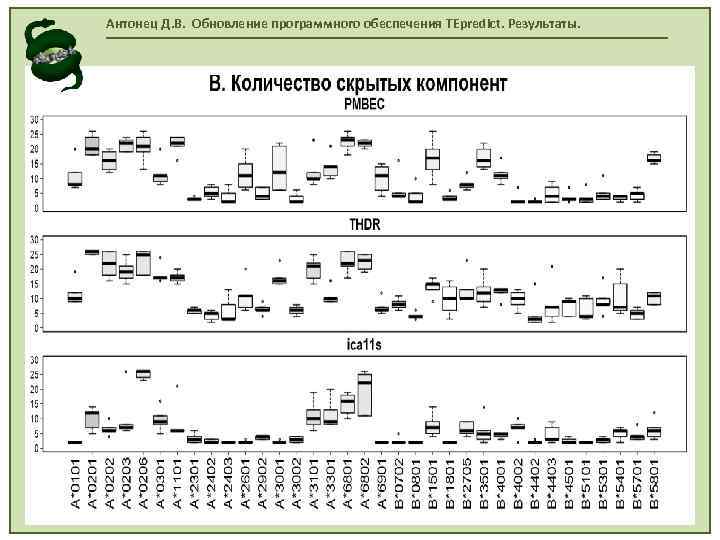
Антонец Д. В. Обновление программного обеспечения TEpredict. Результаты.
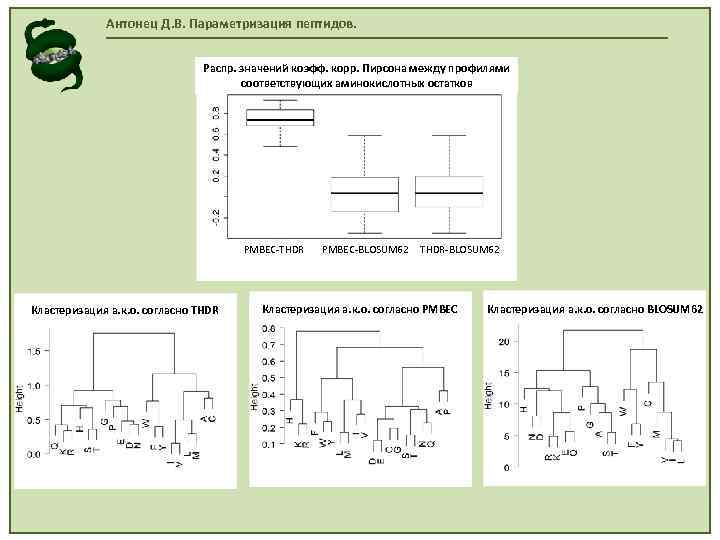
Антонец Д. В. Параметризация пептидов. Распр. значений коэфф. корр. Пирсона между профилями соответствующих аминокислотных остатков PMBEC-THDR Кластеризация а. к. о. согласно THDR PMBEC-BLOSUM 62 THDR-BLOSUM 62 Кластеризация а. к. о. согласно PMBEC Кластеризация а. к. о. согласно BLOSUM 62
T cell epitope prediction 2014.pptx