D--Users-CHELOMINA-Desktop-МАГИСТРЫ-2016-альтернативный сплайсинг.ppt
- Количество слайдов: 189

Сплайсинг • Общие сведения: 1. Большинство генов эукариот «разорваны» , включая белоккодирующие гены, гены т. РНК и р. РНК. Исключение составляют гены гистонов и немного других. 2. У прокариот мало интронов; большинство найдено у вирусов и архей. 3. Интроны были «открыты» через присутствие R петель, однонитиевых областей, наблюдаемых, когда ДНК-м. РНК гибриды (гетеродуплексы) были визуализированы под электронным микроскопом. Наблюдение R-петель хорошо совпадало с с более ранними наблюдениями, что пре-м. РНК (гетерогенная ядерная РНК) была длиннее предсказанной, основанной на длине м. РНК в цитоплазме иили аминокислотной последовательности. 4. В общем, объем интронных последовательностей значительно выше экзонных последовательностей. Инсулин человека: 3 экзона, 2 интрона ~ 12 длины гена; овальбумин цыпленка: 8 экзонов, 7 интронов ~ 34 гена; дигидрофолат-редуктаза: 56 экзонов, 55 интронов ~ 95% гена. 5. Некоторые интроны могут быть довольно большими. Например, один интрон гена Ubx дрозофилы составляет примерно 70 000 п. н. 6. Существует одно общее свойство для всех белок-кодирующих генов, 1 это сайты сплайсинга.

Сплайсинг Существует три разных типа вырезания интронов: для т. РНК, р. РНК и м. РНК. 1. Сплайсинг т. РНК является двустадийным, вовлекающим сплайсирующие эндонуклеазы, которые разрезают концы интронов, и сплайсирующие лигазы, которые соединяют две части т. РНК друг с другом. 2. Сплайсинг р. РНК осуществляется через самосплайсирующее или «автокаталитическое» разрезание. Это автокаталитическое разрезание не вовлекает ни источники внешней энергии, ни энзимы, это просто серия переноса фосфоэфирных связей. 2

3. Сплайсинг м. РНК (иранскрипты или белок-кодрующие гены) выполняется комплексом РНК-белковых структур, называемых сплайсосомами. - 5 молекул малых РНК U 1, U 2, U 4, U 5 и U 6 с размером 100 -215 нуклеотидов. - U 3 найдена только в ядрышках, следовательно, м. б. вовлечена в формирование рибосом. - малые РНК не существуют в виде свободных молекул, они связаны с белками, образуя sn. RNPs (малые ядерные рибонуклеопротеины). Начальным этапом является разрезание в 5’ сайте (↓GUинтрон), затем образуется связь между G и консервативным А на 3’ сайте с образованием структуры лассо. Следующий этап – разрезание в 3’ сайте и объединение двух экзонов через 5’ - 3’ диэфирную связь. Сплайсированная процессированная м. РНК затем поступает в цитоплазму для трансляции. 3

Интроны • Факты. • 1. Интроны у эукариот имеются давно. Сравнение гомологичных генов среди широкого круга таксонов показало сходство как их числа, так и локализации в геноме. Например, гены α- и β- глобинов млекопитающих, птиц и амфибий. Интроны этих генов высоко консервативны в числе и положении (не не последовательности нуклеотидов). Это указывает, что эта структурная организация генов существует более 500 миллионов лет. • 2. Отличия последовательностей меду интронами (даже у одного вида) высокое, указывающее, что интроны испытываю небольшое давление. • 3. Имеется множество примеров наличия инсерций или подвижных элементов в интронах, предполагающих, что возможные функции интронов видимо не связаны со специфическими последовательностями ДНК. 4

Интроны • Наиболее интересную спекуляцию относительно возможной функции интронов представляют интроны в exon shuffling. • 1. Большинство белков имеют несколько доменов. Эти домены включают субстратную регуляцию, ко-факторную регуляцию, каталитические области, аллостерические функции и т. п. Ранее обсуждаемые примеры включают ДНК-полимеразы. • 2. Много белков (например, энзимы) разделяют один или два домена, а через «exon shuffling» новые «гены» для похожих, но разных белков могут вовлекаться через рекомбинацию различных доменов (экзонов). Должно быть эволюционное преимущество, чтобы «новые» гены стали отдельными транскрипционными единицами. • 3. Имеется множество примеров наличия инсерций или подвижных элементов в интронах, предполагающих, что возможные функции интронов видимо не связаны со специфическими последовательностями ДНК. 5

Сплайсинг • В 1978 г. Филипп Шарп (Массачусетский технологический институт) открыл явление сплайсинга РНК (от англ. to splace - сшивать без узлов). • Экзоны - кодирующие участки генов. • Интроны - некодирующие участки генов. 6
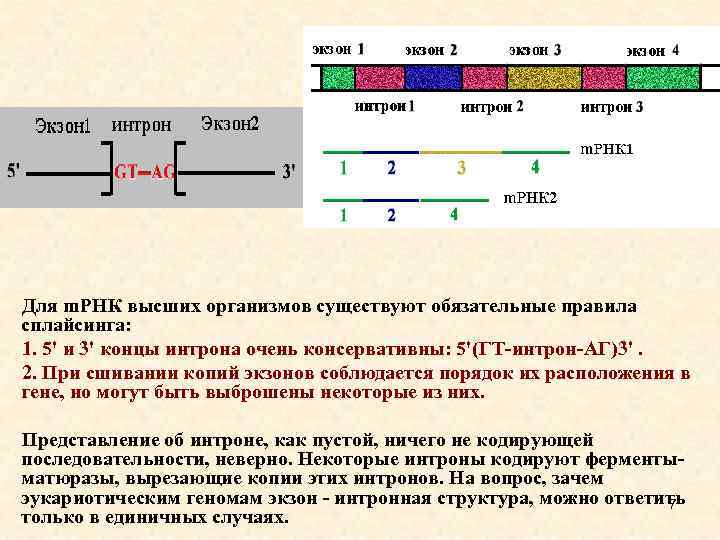
Для m. РНК высших организмов существуют обязательные правила сплайсинга: 1. 5' и 3' концы интрона очень консервативны: 5'(ГT-интрон-AГ)3'. 2. При сшивании копий экзонов соблюдается порядок их расположения в гене, но могут быть выброшены некоторые из них. Представление об интроне, как пустой, ничего не кодирующей последовательности, неверно. Некоторые интроны кодируют ферментыматюразы, вырезающие копии этих интронов. На вопрос, зачем эукариотическим геномам экзон - интронная структура, можно ответить 7 только в единичных случаях.

На долю интронов приходится в 5 -7 раз больше нуклеотидных пар, чем на долю экзонов. Количество экзонов в гене больше, чем интронов. Сплайсинг - вырезание копий интронов из про-m. РНК и сшивание копий экзонов с образованием m. РНК. Копии интронов гидролизуются до нуклеотидов. Сплайсинг показан для большинства m. РНК и некоторых t. РНК. У простейших найден автосплайсинг r. РНК. Сплайсинг показан даже для архей. Не существует единого механизма сплайсинга. Описано по крайней мере 5 разных механизмов. В ряде случаев сплайсинг осуществляют ферменты-матюразы. В некоторых случаях в процессе сплайсинга участвуют s. РНК. В случае автосплайсинга процесс происходит благодаря третичной структуре про-РНК. 8

• А. В начале происходит нуклеофильная атака донорного экзона, затем происходит разрезание, кусочек GU заворачивается и присоединяется к А. 9 Затем разрезается вторая часть, первый экзон соединяется со вторым, и образуется интрон.

Двухэтапный механизм автосплайсинга 1, 2 - экзоны 10

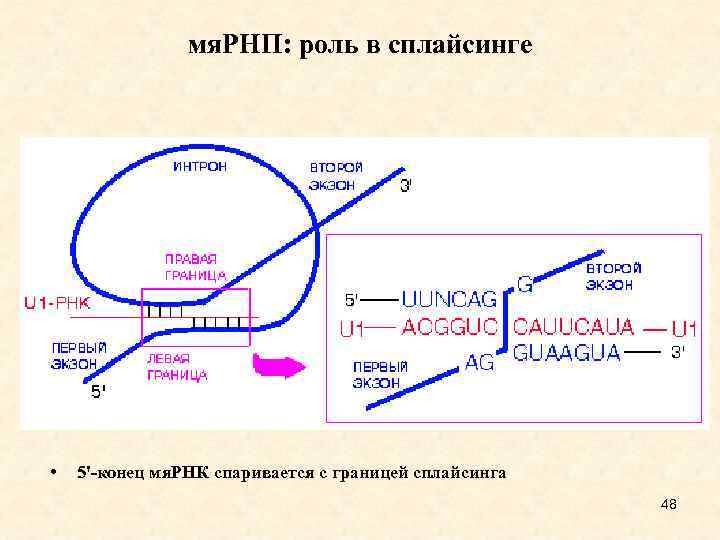
5'-конец мя. РНК спаривается с границей сплайсинга 11

• Вторичные структуры типичных интронов типа I (Tetrahymena thermophila), II и III групп (Euglena gracilis). Стрелками обозначены 3 и 5 сайты сплайсинга, а также места интеграции интронов, пунктирными линиями - сайты интронов, сближенные в пространстве, EBS и IBS - соответственно сайты связывания экзонов и интронов. 12
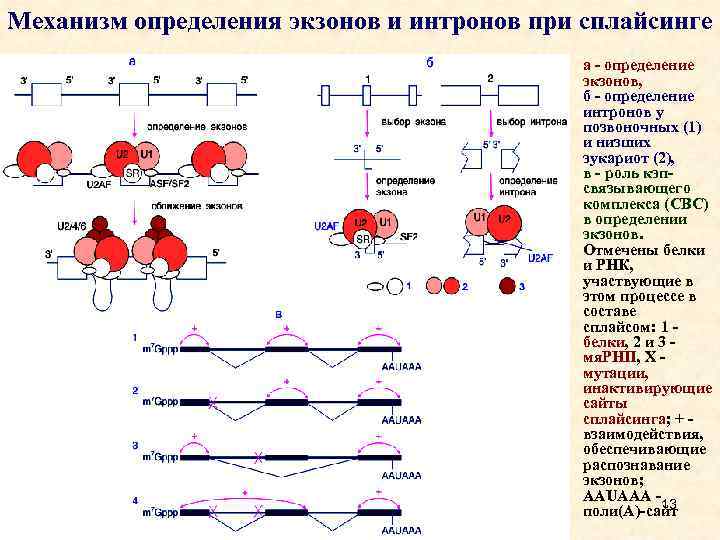
Механизм определения экзонов и интронов при сплайсинге а - определение экзонов, б - определение интронов у позвоночных (1) и низших эукариот (2), в - роль кэпсвязывающего комплекса (CBC) в определении экзонов. Отмечены белки и РНК, участвующие в этом процессе в составе сплайсом: 1 - белки, 2 и 3 - мя. РНП, X - мутации, инактивирующие сайты сплайсинга; + - взаимодействия, обеспечивающие распознавание экзонов; AAUAAA - 13 поли(А)-сайт

Альтернативный сплайсинг m. РНК кальцитонинового гена у млекопитающих (крыса) 14

• Во всех клетках есть кальцитониновый ген, но в клетках щитовидной железы он экспрессируется в виде гормона кальцитонина, а в клетках гипофиза - нейропептида CGRP (пептида, имеющего отношение к гену кальцитонина). Ген один, а белки получаются разные в результате сплайсинга m. РНК и процессинга полипептидов. В клетках других тканей этот ген не экспрессируется. • Сплайсинг осуществляется белковыми комплексами - сплайсосомами, в которых помимо ферментов, вырезающих и сшивающих участки проm. РНК, имеются белки, придающие про-m. РНК нужную конформацию, и несколько s. PНК. Сплайсосома непосредственно связана с ферментами, 15 занимающимися полиаденилированием.

Альтернативный сплайсинг в определении пола у дрозофилы 16
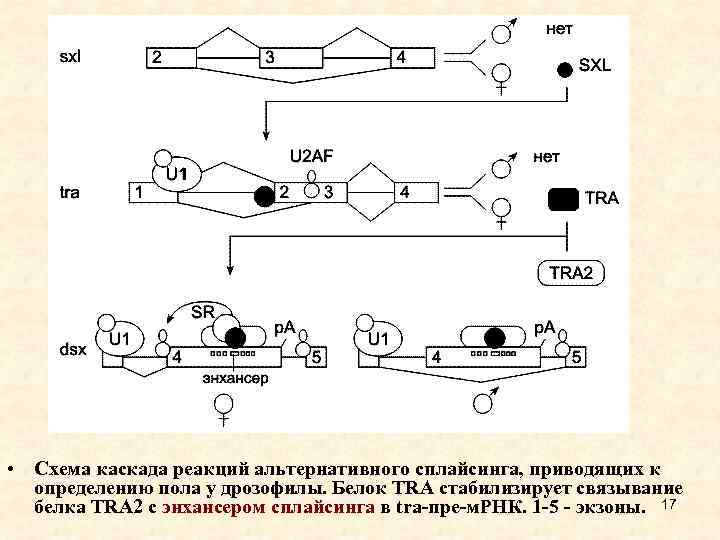
• Схема каскада реакций альтернативного сплайсинга, приводящих к определению пола у дрозофилы. Белок TRA стабилизирует связывание белка TRA 2 с энхансером сплайсинга в tra-пре-м. РНК. 1 -5 - экзоны. 17

Соматическая дифференциация пола у дрозофилы 18

Выбор сплайсосомой альтернативных сайтов сплайсинга 19

Механизм действия белков SR при обеспечении выбора сплайсомой альтернативных сайтов сплайсинга U 1 -мя. РНП. Эффективность вырезания интронов: а - 40% проксимальных, 40% дистальных; 20 б - 80% проксимальных, 20% дистальных
21

Взаимно исключаемые экзоны 22

23

Малые РНК • s. РНК обнаружены в количестве 103 -105 копий на клетку. Поскольку в большинстве случаев эти РНК обогащены урацилом, они называются U 1, U 2. . . Их размер от 100 до 300 нукл. Все они кодируются в ядре, но работают как в ядре (small nuclear - SN), так и в цитоплазме (small cytoplasmic - SC). • SNURPS - РНП (рибонуклеопротеидные комплексы) в ядре. sn. РНК входят в состав РНП, участвующих в полиаденилированиии и сплайсинге. • SCURPS - РНП в цитоплазме. Входят в состав информосом. • Малая РНК U 4 присутствует в комплексах, участвующих в полиаденилировании. Если получить антитела к белкам, связывающимся с U 4, то не происходит полиаденилирования и сплайсинга. • При красной волчанке (аутоимунном заболевании) вырабатываются антитела к белкам комплекса с U 4. • Гистоновая m. РНК не полиаденилируется потому, что s. РНКU 7, которая комплементарна 3'-концу гистоновой m. РНК, защищает ее от полиаденилирования. 24 • Малые РНК U 1, U 2, U 4, U 5, U 6 входят в состав сплайсосомы.

Вторичная структура малых РНК 25

• Если посмотреть, какую долю гена составляют экзоны, то самый большой известный транскрипт (у гена миодистрофина) имеет длину около 2, 5 миллионов нуклеотидов. У него в зрелую часть РНК переходит 14 тыс нуклеотидов (0, 6%), а остальные 99, 4% от первичного транскрипта выкидывается (интроны). • С ростом размеров гена в хромосоме его белок-кодирующая часть увеличивается незначительно, а количество интронов в гене растет. С ростом числа интронов растет число сайтов сплайсинга и вероятность их повреждения. Поэтому для генов с большим числом интронов потеря функции при мутации может быть связана не с белок- кодирующей частью ДНК, а с регуляторными элементами сплайсинга. • Секвенирование генома человека показало, что некоторые экзоны многократно повторены в геноме. Это могут быть повторы экзонов в составе одного гена, или присутствие одного и того же экзона в составе нескольких разных генов. Получается, что экзоны, основные элементы структуры РНК, то есть белок-кодирующие элементы, в процессе эволюции могут каким-то образом размножаться в геноме и «перетасовываться» между разными генами. Такое явление получило название exon shuffling – перетасовка экзонов. Ниже показаны разные белки, в которых содержатся одинаковые экзоны. Таким образом, оказывается, что эволюция – это нередко именно 26 блоковые изменения генома, а не точечные изменения.

1 27 2

3 Сейчас общеизвестно, что не менее 50% генов человека альтернативно сплайсируется. Альтернативный сплайсинг бывает разных типов (галочками показано, как вырезаются экзоны). 28 4

EST (expresstion sequence tag – "ярлыки экспрессируемых последовательностей "). EST – это короткие, прочитанные однократно (то есть весьма неточно), фрагменты цитоплазматической (сплайсированной, содержащей только экзоны) РНК. Если у нас есть геном, то мы можем эти EST картировать на геном и, тем самым, найти, где находятся интроны и экзоны. 29

Человек и мышь биологически очень похожи. Белки похожи – уровень сходства аминокислотных последовательностей 80%, также похожа значительная часть некодирующих областей генома. Практически у всех генов одинаково устроена экзон-интронная структура, для 99% генов 30 экзонная структура одинакова. Только 1% генов уникален у каждого генома, остальные гены имеют гомологи в другом геноме.

1 2 31

1 2 32

1 Возникает вопрос: Если геномы одинаковы, то может быть, и белки одинаковы? Непонятно, чем же человек отличается от мыши. Одинаково ли устроен альтернативный сплайсинг у мыши и человека? 2 Наивный подход для ответа на этот вопрос такой: возьмем весь набор альтернативного сплайсинга мыши и человека и сравним его. Этот подход неправильный, так как при исследовании альтернативного сплайсинга мы здесь имеем дело с EST. Если у человека EST просеквенировано несколько миллионов штук, то у мыши сделано всего несколько тысяч, поэтому там, где мы можем увидеть альтернативный сплайсинг у человека, можем ничего не увидеть у мыши, так как базы данных еще не совсем полные. Поэтому такое сравнение даст нам неправильный ответ. Правильный подход в данной ситуации заключается в следующем: мы на основе имеющихся данных на человеческой ДНК строим м. РНК, соответствующую белку, и затем этот белок проецируем на мышиный геном. Если оказывается, что для этого белка (или его части) нет кодирующих последовательностей в мышиной ДНК, то это значит, что тот экзон, который есть у человека, отсутствует в геноме у мыши. 2: У 52% человеческих генов есть такие экзоны, которых нет у мыши. И половина мышиных генов имеет такие изоформы, которых нет у 33 человека.

• Видно, что примерно треть генов человека имеет изоформы, которые в геноме мыши не кодируются, отсутствуют, и также в геноме человека отсутствуют мышиные экзоны. • Представленные данные показывают, что альтернативный сплайсинг – явление весьма распространенное, и что мышь сильно отличается от человека по альтернативному сплайсингу. • Можно сделать и эволюционное предположение. По-видимому, альтернативный сплайсинг допускает большую свободу для создания новых белков, или изменения функций 34 существующих белков, и в этом и состоит его связь с эволюцией.

• Пример: сверху изображены ДНК и ее изоформы у человека, а снизу – то же у мыши для белка р53, который участвует в регуляции клеточных процессов (раковое перерождение, апоптоз). Видно, что у мыши есть изоформа, которая теряет экзон, порождая стоп в другом месте. 35

• Интроны характерны для всех типов эукариотической РНК, но также найдены в рибосомальной РНК (р. РНК) и транспортной РНК (т. РНК) прокариот. Число и длина интронов очень различны в разных видах и среди разных генов одного организма. Гены млекопитающих, в том числе человека, и цветковых растений часто содержат многочисленные интроны, которые могут быть длиннее экзонов. Кодирующие последовательности, которые в конечном итоге и дают м. РНК, показаны черным, а интроны – белым. 36

• Сплайсинг: факторы эволюционные • Есть четыре предположения, отвечающие на вопросы - почему сплайсинг сильно распостранен у эукариот и как эволюция поддерживает наличие интронов. • Первое: посредством интронов обеспечивается рекомбинация внутри последовательностей, что ускоряет процесс образования новых генов. C этой точки зрения предки современных генов кодировали маленькие, простые полипептиды, а рекомбинация в интронах приводит к дупликации и пересортировке простых генов, приводящей к образованию современных сложных белков. Это допущение согласуется с наблюдениями о том, что экзоны часто коррелируют с функциональными доменами белков и гомологичные экзоны могут быть найдены в различных генах ( тасование экзонов ). 37

• Второе: интроны могут содержать регуляторные последовательности, такие как энхансеры в генах иммуноглобулинов. В некоторых случаях внутри интрона одного гена может присутствовать целый другой ген. Однако, если для компактных геномов такое обьяснение вполне приемлимо, маловероятно, что эукариоты с их огромными геномами поддерживают присутствие интронов лишь с целью кодирования. • Третье: дифференциальный сплайсинг поддерживает образование различных, но связанных белков и обеспечивает дополнительный механизм регуляции экспрессии генов. Возможно, что дифференциальный сплайсинг играет центральную роль в таких сложных процессах, как развитие. • Четвертое: механизм биогенезиса м. РНК должен эволюционировать вместе со сплайсингом. Для нескольких пре-м. РНК, таких как ранние гены SV 40, бета-глобин человека и др. сплайсинг необходим для образования стабильной цитоплазматичес- кой м. РНК; замена геномной последовательности последовательностью к. ДНК приводит к сильному уменьшению стабильной цитоплазматической РНК. В дрожжах же аналогичное не наблюдается. 38

• Интроны превалируют у высших эукариот над прокариотами • Согласно одной гипотезе интронами были насыщены предки прокариотических генов, но они их потеряли вследствие необходимости оптимизации их геномов с целью усиления скорости репликации их ДНК в быстро растущих организмах. Этот процесс "оптимизации" был очень эффективным: обнаружен всего лишь один прокариотический структурный ген с интронами (thymidylase synthase gene of phage T 4). В дрожжах только 5 -10% структурных генов содержат интроны. Несколько случаев делеции интронов зарегистрировано для эукариот. • • Интроны: размеры Эффективность сплайсинга очень сильно изменяется при значительном уменьшении длины интрона, и еще более сильно - при уменьшении расстояния между брэнч сайтом и 5' сайтом сплайсинга. При последовательном уменьшении этого расстояния процесс сплайсинга прекращается, однако он начинается снова при активировании нового ложного сайта сплайсинга слева от истинного. Эти эксперименты показывают, что минимальный размер функционального интрона равен приблизительно 80 нуклеотидам. Существование минимального размера интронов, вероятно отражает тот факт, что для сплайсинга необходимо взаимодействие РНК с множественными факторами сплайсинга. Однако существует природный вирусный интрон длины меньшей минимальной. 39

• Интроны: удерживание при сплайсинге • Простейшей формой альтернативного сплайсинга является включение сплайсируемого интрона в состав зрелой РНК. Ошибка в вырезании интрона приводит к вставке дополнительного пептидного сегмента, если не нарушается открытая рамка считывания. Досрочная терминация считывания или нарушение рамки приводит к функционально другому продукту или же к прекращению экспрессии гена. Примеры такого сплайсинга обнаруживаются для генов фибронектина и элемента Р транспозазы дрозофилы. 40

• Интроны РНК группы I • Многие митохондриальные м. РНК и р. РНК грибов содержат интроны. Большинство из них было названо интронами группы I на основании консервативных элементов, каждый длиной около 10 н-ов. Четыре консервативных последовательности были названы A, B, box 9 L , box 2. Другая пара последовательностей - 9 R и 9 R' не является высоко консервативной, но всегда комплементарна другу. Эти консервативные последовательности всегда находятся в одинаковом порядке в интроне, 5'-9 R'-A-B-9 L-9 R-2 -3'. Большинство интронов группы I содержат дополнительно внутреннюю управляющую последовательность. 41

• Интроны РНК группы I • внутренняя управляющая последовательность 42

• Интроны РНК группы II • Небольшая группа митохондриальных IVS имеет консервативные последовательности и вторичную структуру, отличную от интронов группы I. Они названы интронами группы II. Один из них, последний интрон дрожжевой митохондриальной м. РНК охидазы цитохрома, подвергается аутосплайсингу in vitro. Также как и сплайсинг интронов группы I, реакция требует присутствия ионов Mg 2+. Отличие состоит в отсутствии необходимости присутствия гуанозина или любого другого нуклеотида в качестве субстрата. • Механизм сплайсинга предшественников группы II имеет общие черты с механизмом сплайсинга пре-м. РНК группы I. Оба процесса являются двухэтапными реакциями трансэстерификации, при которых на первом этапе происходит разрыв 5' сайта сплайсинга, а на втором разрыв 3' сайта сплайсинга и сшивание экзонов. Второй этап совершенно одинаков у обеих групп, а первый различается. В интронах группы I нуклеофильным является 3' гидроксильная группа гуанозина связанного с РНК, в интронах группы II - 2' гидроксильная группа нуклеотида в цепочке РНК. Благодаря этой группе формируется структура лариата. 43

• Пре-м. РНК ядерные • Сплайсинг ядерных пре-м. РНК, также как и сплайсинг интронов группы II включает в себя формирование лариата. Хотя ядерные прем. РНК не подвергаются аутосплайсингу in vitro известно, что структура интронов играет ту же роль при регуляции реакции сплайсинга. Интроны содержат три консервативные последовательности: 5' сайт сплайсинга , 3' сайт сплайсинга и брэнч сайт. Мутации влияющие на сплайсинг подтверждают это, но сдругой стороны делетирование протяженных участков вне этих трех консервативных последовательностей не приводят к изменениям в процессе сплайсинга. Это сильно отличается от ситуации с аутосплайсингом , где значительная часть структуры РНК является консервативной и требуется для поддержания активной реакции. Неудивительно поэтому, что ядерные РНК требуют дополнительных факторов сплайсинга (транс-действующих элементов) к консервативным участкам. 44

• Экзоны: включение и исключение • Некоторые экзоны могут включаться и исключаться независимо от других экзонов. Обычно при этом рамка считывания остается прежней, хотя ее сдвиг или досрочная терминация могут быть результатом такого исключения. При большом числе экзонов в гене может генерироваться высокое разнообразие продуктов, как например у генов NCAM и тропонина-Т. Наблюдаемая гипервариабельность в N-концевом районе тропонина-Т основана на наличии 5 кассетных экзонов на 5' конце гена, что находится в согласии с теорией, допускающей образование до 64 разных Тn-T изоформ. 45

• Экзоны взаимно исключаемые при сплайсинге • Внутренние экзоны иногда являются взаимно исключаемыми. Один из пары всегда сплайсируется в м. РНК, но никогда не оба вместе. • Наилучшим примером является пара, найденная в генах альфа и бета тропомиозина. Экзоны 2 и 3 альфатропомиозина мыши сплайсируются строго взаимноисключающе и производят м. РНК содержащую экзоны 1, 2 и 4 в одних тканях и 1, 3 и 4 в других. показали, что природа такого сплайсинга обеспечивается близостью брэнч сайта экзона 3 к 5' сайту сплайсинга экзона 2. 46

• мя. РНК • • • Ядро эукариотических клеток содержит большое число видов различных малых РНК. Их размер варьирует от 100 до 300 нуклеотидов, а число молекул, приходящихся на одну клетку, состовляет примерно от 100000 до 1000000. Некоторые малые РНК синтезируются с помощью РНК-полимеразы III, другие- РНК-полимеразы II; среди таких транскриптов встречаются и некоторые имеющие кэп, как и молекулы м. РНК. Такие молекулы называют малыми ядерными РНК (sn. RNA). Природные малые РНК существуют в виде рибонуклеопротеиновых частиц ( мя. РНП или в обиходе snurps от английского sn. RNP). мя. РНП получают имена от мя. РНК , которую они содержат. Существуют шесть основных видов мя. РНП (U 1 -U 6) и несколько дополнительных (U 7 -U 10). РНКовая компонента мя. РНП состовляет 60 -120 nt. Все мя. РНК за исключением U 6 содержат 5' концевую структуру, содержащую уникальный нуклеозид 2, 2, 7 триметилгуанозин. мя. РНК присутствуют в клетке в виде рибонуклеопротеиновых частиц. Лернер и Стейтз предложили основное средство для описания структуры и функций мя. РНП продемонстрировав, что некоторые пациенты с аутоимунными заболеваниями несут антитела к мя. РНП; антигенными детерминантами являются мя. РНП-специфические белки. Рибонуклеопротеины содержат также в себе белковую компоненту, состоящую из ряда полипептидов, называемых Sm белками. Их обозначеня и мол. вес - 68. 000 ( 68 K ), 33. 000 ( A ), 32. 000 ( A' ), 29. 000 (B'), 28. 000 (B), 22. 000 (C), 16. 000 (D), 13. 000 (E), 12. 000 (F), 11. 000 (G). Белки B, B', D , E , F и G присутствуют в большинстве мя. РНП. U 1 содержит уникальные детерминанты A, C, и 68 К полипептид ( Fisher D. E. , 1983 ) полипептид А' и совершенно другой, отличный от В белок В‘. 47
мя. РНП: роль в сплайсинге • 5'-конец мя. РНК спаривается с границей сплайсинга 48

49

• Транскрипт первичный • Первичный РНК-транскрипт, или про-м. РНК, синтезированный на транскрипционной единице , в большинстве случаев длиннее, чем последовательность нуклеотидов, соответствующая конечному продукту (полипептиду, т. РНК, р. РНК). У эукариот первичный транскрипт (молекулярная масса от 106 до 1. 5. 107) может быть в 10 раз длиннее, чем м. РНК, поступающая для трансляции. Первичный РНК-транскрипт претерпевает изменения в совокупности называемые процессингом. При процессинге к нему сначала присоединяются колпачок и poly(A) , а затем в результате многократного сплайсинга он укорачивается, и одновременно происходит внутреннее метилирование с образованием 6 -метиладенозина. • Про-м. РНК и м. РНК всегда соединены ионными связями с белками и образуют рибонуклеопротеиновые частицы. 50

• Abstract Introns are integral elements of eukaryotic genomes that perform various important functions and actively participate in gene evolution. We review six distinct roles of spliceosomal introns: • (1) sources of non-coding RNA; • (2) carriers of transcription regulatory elements; • (3) actors in alternative and trans-splicing; • (4) enhancers of meiotic crossing over within coding sequences; • (5) substrates for exon shuffling; and • (6) signals for m. RNA export from the nucleus and nonsense-mediated decay. • We consider transposable capacities of introns and the current state of the long-lasting debate on the early-or-late origin of introns. Cumulative data on known types of contemporary exon shuffling and the estimation of the size of the underlying exon universe are also discussed. We argue that the processes central to introns-early (exon shuffling) and introns-late (intron insertion) theories are entirely compatible. Each has provided insight: the latter through elucidating the transposon capabilities of introns, and the former through understanding the importance of introns in genomic recombination leading to gene rearrangements and evolution. 51

Diseases caused by cis-acting mutations that disrupt use of alternative splice sites. Four diseases are caused by mutations that disrupt use of alternative splice sites. The splicing patterns favored by the mutations are indicated by the blue arrows. Red indicates alternatively spliced regions. (A) Familial isolated growth hormone deficiency type II (IGHD II) is caused by mutations in the growth hormone gene (GH-1). 5 ss indicates 5 splice site. (B) Frasier syndrome is caused by mutations in the WT-1 gene. (C) Frontotemporal dementia and Parkinsonism linked to Chromosome 17 (FTDP-17) are caused by mutations in the MAPT gene. Dotted lines indicate proposed RNA secondary structure. All mutations except 280 K (green) enhance exon 10 inclusion. The 280 K mutation enhances exon skipping. (D) Atypical cysticfibrosis is caused by polymorphisms of the CFTRgene. Pre-m. RNA splicing and human disease Nuno Andreґ Faustino and Thomas A. Cooper

Model of pathogenesis in myotonic dystrophy. CUGand CCUG-repeat RNA form imperfect hairpins that are bound by the double-stranded RNA-binding protein muscleblind (orange). Nuclear accumulation of repeatcontaining RNA elevates expression of CUG-BP (lavender) by an unknown mechanism. The consequences of muscleblind sequestration remain to be determined. Increased CUG-BP activity alters regulated splicing of three CUG-BP targets, ultimately resulting in the characteristic symptoms of DM.

The Regulation of Telomerase by Alternative Splicing of TERT Jiří Nehyba, Radmila Hrdličková and Henry R. Bose, Jr. University of Texas at Austin USA Key differences in the sequences of quail and chicken intron 10. Four large regions of DNA present in chicken TERT gene are not found in the corresponding quail intron (736, 23, 36, and 319 nt). B. A stop codon (indicated by asterisk), absent from the chicken cassette exon 7 A, is present in the corresponding quail sequence. C. Comparison of splice donor A site in chicken exon 2 (sd. A) with its quail sequence homolog, and splice donor A 2 (sd. A 2) site in the same exon with quail q. A 2 splice donor site (sdq. A 2). In contrast to the chicken isoform A, chicken isoform A 2 and quail isoform q. A 2 contain stop codons in exon 3 sequences (asterisks).

Example of exon loss coupled with AS: Lactadherin belongs to a family of mosaic glycoproteins widely expressed in a number of tissues and body fluids. The figure shows SMART(21) protein representations with included intron positions (vertical lines) mapped onto a Clusta. IW alignment of the orthologous genes in human and mouse (mfgm_human and mfgm_mouse). An AS variant in mouse (q 9 wts 3_mouse) is shown to skip the exon which is missing in human. This event is proven to be an exon loss in human rather than a gain in mouse because this exon is found in other organisms. Black boxes represent gaps in the alignment. Alternative splicing and evolution Stephanie Boue, * Ivica Letunic, and Peer Bork

REST-mediated transcriptome reprogramming in neuroendocrine transdifferentiation. The REST transcriptional complex represses transcription of neuronal genes through binding to the regulatory RE 1 sites located within promoter or enhancer regions of genes. This complex undergoes transcriptional and post-transcriptional changes during neuroendocrine transdifferentiation. REST transcripts are transcriptionally attenuated and alternatively spliced and the REST cofactor BHC 80 is alternative spliced. The resulting REST and BHC 80 isoforms lack important functional domains necessary for the repression (REST 4 isoform and the NEPC-specific BHC 80 isoform). Loss of REST-mediated repression leads to the emergence of theneuronal transcriptional program, including NEPC-specific splicing. The role of m. RNA splicing in prostate cancer Anna V Lapuk, Stanislav V Volik, Yuzhuo Wang, Colin C

Splicing regulation of the androgen receptor (AR) and phosphoinositide 3 -kinase (PI 3 K) signaling pathways. Members of the pathway whose transcripts are alternatively spliced in cancer are indicated with green decagons. Splice factors Sam 68, SRSF 1, and SRs as well as their regulators (SRPKs) that are altered in cancer transcriptionally or posttranscriptionally are marked with green ovals. Dark green indicates that altered function of a splice isoform or a splice factor has been observed in prostate cancer whereas light green–in other cancers. SRs: splice factors of the serine/arginine-rich protein family; SRPKs: SR protein kinases; p: phosphorylation; AS: alternative splicing. The signaling pathways of AR and PI 3 K have been adapted from Barlow and Shen. 100 *Pathway components for which new blocking agents are under investigation for advanced prostate cancer.


Interchromosomal растения Distant Neighboring Exon skipping Intron retention Mutually exclusive exons Alternative donor site Alternative acceptor site

Schematic representation of the sequence elements and proteins at 5' and 3' exon‑intron boundaries in an RNA transcript. The diagram illustrates the appropriate relative distributions of the molecules and core splicing signals with its consensus sequence in regulation of the alternative splicing. The enhancer elements [(exonic splicing enhancers (ESEs) and intronic splicing enhancers (ISEs)] are recognized by activator proteins (the SR protein family), and the silencer elements [exonic splicing silencers (ESSs) and intronic splicing silencers (ISSs)] are bound by repressor proteins [the heterogeneous nuclear ribonucleoproteins (hn. RNP) protein family]. These two protein families are engaged to promote or inhibit spliceosome assembly at weak splice sites, respectively.


Alternative splicing of human (and mouse) genes

• 24 -31% human vs. 17 -25% mouse elementary alternatives are not conserved • 55% human vs 36% mouse genes have at least one non-conserved variant • denser coverage of human genes by ESTs: – pick up rare (tissue- and stage-specific) => younger variants – pick up aberrant (non-functional) variants • 17 -24% m. RNA-derived elementary alternatives are nonconserved (compared to 25 -32% EST-derived ones)

• inclusion level is a good predictor of conservation – 98% constitutive exons are conserved – 98% major form exons are conserved – 28% minor form exons are conserved • inclusion level of conserved exons in human and mouse is highly correlated • Minor non-conserved form exons are errors? No: – – minor form exons are supported by multiple ESTs 28% of minor form exons are upregulated in one specific tissue 70% of tissue-specific exons are not conserved splicing signals of conserved and non-conserved exons are similar

Alternative splicing avoids disrupting domains (and nondomain units) Control: fix the domain structure; randomly place alternative regions

Alternative splicing Form

Abstract • Alternative splicing of m. RNAs by analysing the exon linkage relationship by alignment of ESTs to the genome sequence • Little effort has been made to investigate the relationship between cancers and alternative splicing • Alternative splicing assembler(ASA) • Of 4322 genes screened, 3498(81%) were observed with at least one alternative splicing variants. • Using Fisher’s test, alternative splicing variants

Introduction • About 45% of the human EST sequence are derived from cancer cells. • Alternative splicing is an important mechanism in higher eukaryotes for producing proteomics complexity • Approximately 30 -60% genes are alternative splicing as estimated by genomically aligned ESTs • Human could conceivably produce hundreds of thousands of different proteins by the estimated 35, 000 genes • Alternative splicing of pre-m. RNA is a versatile mechanism for regulating gene function at the posttranscription level

Alternative splicing of the mouse immunoglobulin μ heavy chain gene S-signal peptide C - constant region V- variable region green – membrane anchor Red- untranslated reg. yellow – end of coding reg. for secreted form

Alternative splicing allows us to hear multiple frequencies From Graveley B. R. (2001) Alternative splicing: increasing diversity in the proteomic world. Trends in Genetics 17: 100 -107.

Types of alternative splicing Every conceivable pattern of alternative splicing is found in Nature. Exons can have multiple 5'- or 3'-splice sites that are alternatively used (Fig. Ia, b). Cassette exons are fully contained exons that are alternatively used. Single cassette exons can reside between two constitutive exons such that the alternative exon is either included or skipped (Fig. Ic). Alternatively, multiple cassette exons can reside between two constitutive exons such that the splicing machinery must choose between them ( Fig. Id). Finally, introns can be retained in the m. RNA and become translated ( Fig. Ie). The constitutive exons are depicted as open boxes and alternative exons are shaded. The lines above and below the boxes show possible alternative splicing events. From Graveley B. R. (2001) Alternative splicing: increasing diversity in the proteomic world. Trends in Genetics 17: 100 -107.

Negative and positive control of alternative RNA splicing. (A) Negative control, in which a repressor protein binds to the primary RNA transcript in tissue 2, thereby preventing the splicing machinery from removing an intron sequence. (B) Positive control, in which the splicing machinery is unable to remove a particular intron sequence without assistance from an activator protein.

Cascade of regulated splicing that controls expression of sex-lethal (sxl), transformer (tra), and doublesex (dsx) genes in Drosophila embryos. For clarity, only the exons (light red boxes) and introns (blue lines) where regulated splicing occurs are shown. Splicing is indicated by dashed lines above (female) and below (male) the pre-m. RNAs. Dark red bands contain in-frame stop codons, which prevent synthesis of functional protein. Only female embryos produce functional Sxl protein, which represses splicing between exons 2 and 3 in sxl pre-m. RNA and between exons 1 and 2 in tra pre-m. RNA. In contrast, binding of the Tra-Tra 2 complex to dsx pre-m. RNA activates splicing between exons 3 and 4. As a result of this cascade of regulated splicing, distinct Dsx proteins are produced in female and male embryos. These repress transcription of genes required for sexual differentiation of the opposite sex. See text for further discussion. [Adapted from M. J. Moore et al. , 1993, in R. Gesteland J. Atkins, eds. , The RNA World, Cold Spring Harbor Press, pp. 303 -357. ]

Some cases of alternative polyadenylation are driven by alternative splicing

Transmembrane and secreted forms of immunoglobulins are derived from the same heavy-chain sequence by alternative polyadenylation. The polyadenylation factor Cst. F-64 regulates alternative processing of Ig. M heavy chain pre-m. RNA during B cell differentiation. Takagaki Y, Seipelt RL, Peterson ML, Manley JL. Cell 1996 Nov 29; 87(5): 941 -52
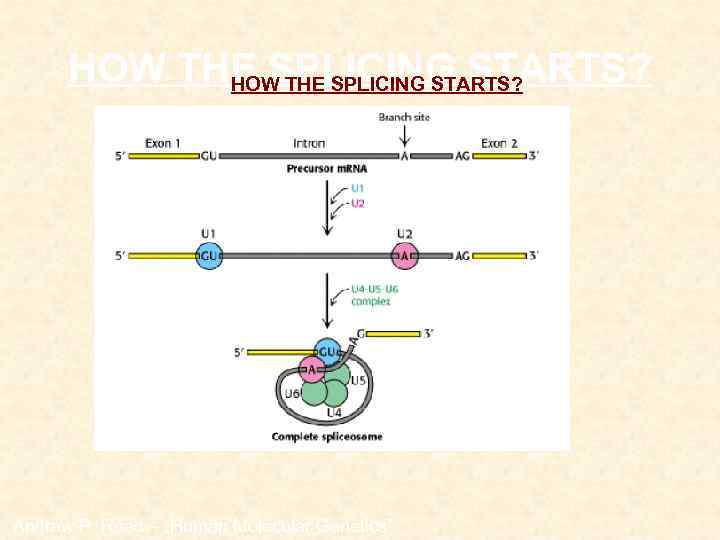
HOW THE SPLICING STARTS? Andrew P. Read – „Human Molecular Genetics”
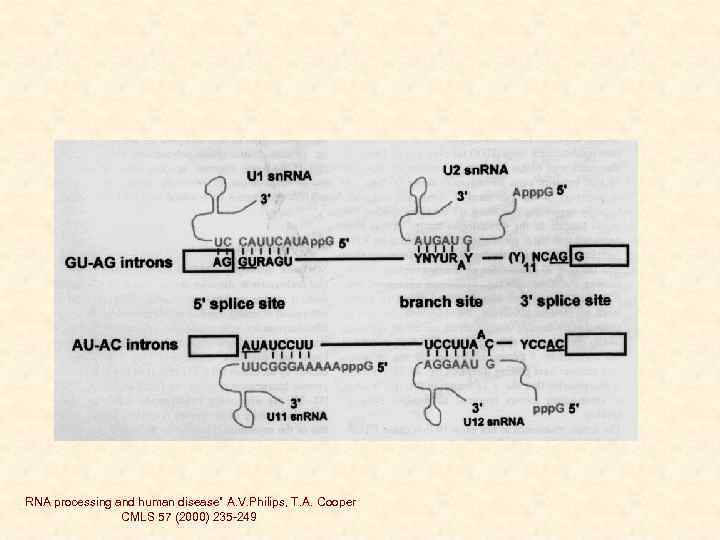
„RNA processing and human disease” A. V. Philips, T. A. Cooper CMLS 57 (2000) 235 -249

sn. RNP


antisense

Human genome 2. 91 billion base pairs 24, 500 genes 1. 5% exons (127 nucleotides) 24% introns (~3, 000 nucleotides) 75% intergenic (no genes) Average size of a gene is 27, 894 bases Contains an average of 8. 8 exons Titin contains 234 exons.

Gene number C. elegans ~19, 000 Drosophila melanogaster ~13, 600 Homo Sapiens ~24, 000 Mouse ~24, 000 Arabidopsis thaliana ~25, 500 Rice (Oryza sativa) ~50, 000

Compared to worm and fly, human has shorter exons and longer introns on the extremes of the distribution 150 nt – drosophila 125 nt – Ciona intes. 119 nt - human

5000 genes 234 introns Multi-introns genes 7. 8 introns per gene

Alternative splicing פוליפרימידין טראק 40% 18% 8% ? 3% isoform

81 איזופורמים 1

Alternative splicing regulate pro or anti apoptotic pathways. pro-apoptotic anti-apoptotic

150 белков 5 типов мя. РНК (U 1, U 2, U 3, U 4, U 5)

Пример альтернативного сплайсинга

Интроны • Последовательности РНК вырезаемые в процессе превращения первичного транскрипта в м. РНК • В основном характерны для более развитых организмов – Очень мало у бактерий – У эукариот особенно часто встречаются у высокоразвитых организмов, особенно в составе одного и того же гена • Гены без интронов • Например кодирующие гистоны • Также не процессируются

Интроны • Изначально описаны у Аденовируса и у гена куриного овальбумина – Ген и м. РНК были денатурированы и гибридизованы между собой – Электронная микроскопия обнаружила образование петель где присутствовали интроны

Интроны • 4 класса – ядерная и не-ядерная р. РНК, т. РНК и м. РНК – не-ядерная м. РНК не у животных – Ядерная м. РНК – т. РНК сплайсинг, требующий эндонуклеазу и АТФ

Первая группа • Катализируется самой м. РНК • Трансэстерификация не требует затраты энергии и протекает спонтанно • Интрон впоследствии разрушается • Требует участие Гуанозина, который остается на интроне • Гуанозин разрушает и вклинивается в 5’ конец интрона

Вторая группа • Подобно Группе 1 но с участием внутреннего Аденина • Образуется характерная структура Лассо • Также само-катализ

Сплайсинг ядерной м. РНК • С участием маленьких рибонуклеопротеиновых частиц - small nuclear ribonucleoprotein complexes (sn. RNP’s) – Содержащих sn. РНК – Необходимы для образования сплайсеосомы

Механизм сплайсинга • http: //vcell. ndsu. ed u/animations/mrna splicing/movie. htm

Сплайсинг ядерной м. РНК • Границы интрон-экзон распознаются малыми ядерными РНК • Консенсусные последовательности в интронах гибридизуются с sn. RNA’s • Белки и другие мя(sn)РНК собираются в сплайсеосому на транскрипте • Неспаренный А на 3’ конце интрона атакует 5’ границу интрон-экзон – Образуется лариатная структура • Нуклеотидное замещение • Образрование сплайсеосомы требует АТФ
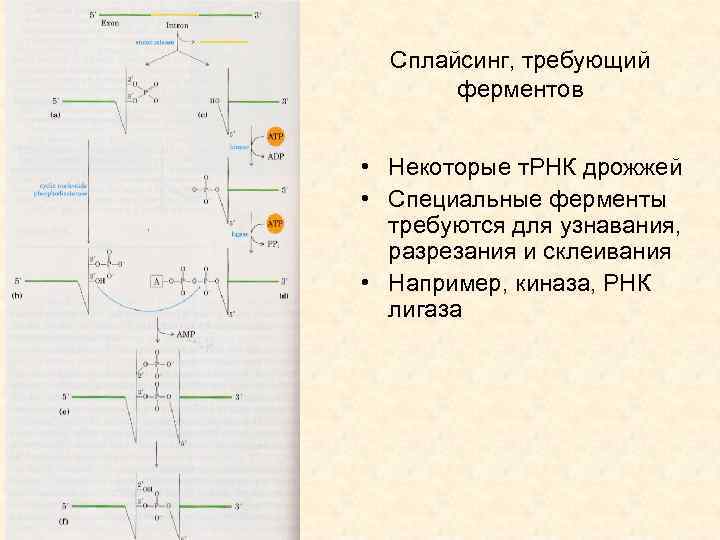
Сплайсинг, требующий ферментов • Некоторые т. РНК дрожжей • Специальные ферменты требуются для узнавания, разрезания и склеивания • Например, киназа, РНК лигаза

Альтернативный сплайсинг Пропускание экзонов • • • Целый экзон может быть пропущен Тяжелая цепь миозина в развитии плодовой мушки Исключение одной из границ 3’
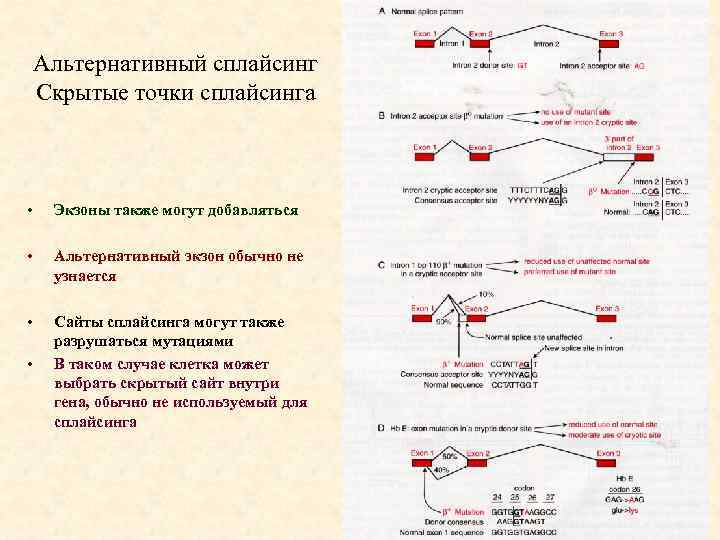
Альтернативный сплайсинг Скрытые точки сплайсинга • Экзоны также могут добавляться • Альтернативный экзон обычно не узнается • Сайты сплайсинга могут также разрушаться мутациями В таком случае клетка может выбрать скрытый сайт внутри гена, обычно не используемый для сплайсинга •

Примеры альтернативного сплайсинга • Не соответствует догме: 1 ген – 1 белок • http: //evolution-development. blogspot. com/ • http: //science. jrank. org/pages/2950/Gene. Splicing-Alternative-splicing. html

Частота случаев альтернативного сплайсинга в ортологичных генах уразных животных и человека по отношению к минимальному разнообразию (к частоте таких случаев в почках лягушки). По оси абсцисс отложено время расхождения эволюционных линий позвоночных, представленных в данном исследовании. Во всех тканях (они обозначены разными цветами) у приматов примерно в два раза выше частота альтернативного сплайсинга. Изображение из обсуждаемой статьи в Science

Процент одинаковых случаев альтернативного сплайсинга у человека по сравнению с другими девятью видами животных. Для каждой ткани (см. легенду во врезке) подсчет велся отдельно. Изображение из дополнительных материалов к обсуждаемой статье в Science

Общие экзоны, которые участвуют в альтернативном сплайсинге, у человека, мыши, курицы и лягушки. Видно, что у всех этих четырех животных вместе есть 41 общий экзон. Изображение из дополнительных материалов к обсуждаемой статье в Science

Вариации в строении цветков у генно-модифицированных

АЛЬТЕРНАТИВНЫЙ СПЛАЙСИНГ м. РНК Fas В КЛЕТКАХ ОПУХОЛЕВОГО ОЧАГА ПРИ РАКЕ ТОЛСТОГО КИШЕЧНИКА 2010 г. Н. А. Сахарнов 1, Д. В. Новиков 1, А. В. Алясова 2 , В. В. Новиков 1 1 Нижегородский госуниверситет им. Н. И. Лобачевского 2 Нижегородская государственная медицинская академия mbre@mail. ru Поступила в редакцию 07. 04. 2010 В клетках опухолевых очагов больных раком толстого кишечника выявлены 7 альтернативных форм м. РНК молекулы Fas и 15 различных спектров м. РНК. На III стадии рака толстого кишечника в образцах опухолей не обнаруживается м. РНК Fas с делецией 5 экзона, а на IV стадии отсутствует м. РНК Fas с делецией 4 экзона.

Существует несколько механизмов альтернативного сплайсинга: 1. Пропуск экзона или экзонной кассеты: в этом случае экзон может вырезаться из первичного транскрипта или сохраняться в нём. Это наиболее часто используемый механизм у млекопитающих. 2. Взаимоисключающие экзоны: из двух экзонов в конечном транскрипте сохраняется только один. 3. Использование альтернативного донорного сайта: есть несколько альтернативных 5'-участков сплайсинга (донорных сайтов), что изменяет 3'границу вышележащего (upstream) экзона. 4. Использование альтернативного акцепторного сайта: используются разные 3'-участки сплайсинга (акцепторные сайты), что меняет 5'-границы нижележащего (downstream) экзона. 5. Удержание интрона: интрон сохраняется в последовательности транскрипта. Если интрон находится в кодирующей последовательности, то он может кодировать стоп-кодон или же сдвигать рамку считывания. А это может привести к потере функциональности белка, поэтому данный механизм альтернативного сплайсинга используется крайне редко у млекопитающих.

Механизм сплайсинга 1. Сплайсинг осуществляется сплайсосомой — массивной структурой, в состав которой входит 5 малых ядерных нуклеопротеидных частиц (sn. RNP) — U 1, U 2, U 4, U 5 и U 6 (U 3 не участвует в сплайсинге) — и большого количества вспомогательных белков. Вместе они способны точно распознать сайты сплайсинга и катализировать 2 шага реакции сплайсинга. 2. Сборка сплайсосомы начинается с распознавания U 1 5’-сайта сплайсинга. После этого фактор сплайсинга 1 (SF 1) связывается в точке разветвления (branch point, обычно это аденозин). Такой комплекс носит название E’. Затем большая субъединица (65 k. Da) гетеродимерного вспомогательного фактора U 2 (U 2 AF) образует комплекс с полипиримидиновым участком (polypyrimidine tract), а малая субъединица (35 k. Da) U 2 AF связывается с 3’-терминальным AG на стыке интрона и экзона. Весь этот образовавшийся комплекс является АТФ-независимым и уже называется комплексом E. Позже, после замены SF 1 на U 2 в точке разветвления, комплекс Е становится АТФзависимым комплексом А. 3. Дальнейшее образование тройного U 4/U 6 -U 5 комплекса приводит к формированию комплекса В, который после экстенсивных конформационных изменений и перестроек превращается в каталитически активный комплекс С. U 6 занимает позицию U 1, а U 1 и U 4 уходят. Оставшийся комплекс претерпевает 2 реакции переэтерификации. В ходе первой реакции, 5’-конец интрона отщепляется от экзона, что перед ним, и присоединяется к сайту разветвления А через 2’, 5’-фосфодиэфирную связь. Во второй реакции переэтерификации, 3’-конец интрона отщепляется от экзона, что расположен далее по последовательности, и два экзона объединяются. Образовавшийся интрон высвобождается в форме лассо и позднее деградирует.

Регуляция Существуют различные способы регуляции альтернативного сплайсинга: • РНК-белковые взаимодействия, • РНК-РНК взаимодействия (при взаимоисключающих экзонах), • взаимодействие пре-м. РНК с некодирующими РНК, в том числе малыми ядрышковыми РНК и др. Несмотря на столь потенциальное разнообразие регуляторных механизмов, РНК-белковые взаимодействия считаются основными способами регуляции сплайсинга

Регуляторные элементы Участки последовательности РНК и регуляторы белков определяют, какой экзон удалить, а какой оставить. Существует 4 категории регуляторов: 1. энхансеры сплайсинга экзонов (exonic splicing enhancers, ESEs) 2. сайленсеры сплайсинга экзонов (exonic splicing silencers, ESSs) 3. энхансеры сплайсинга интронов (intronic splicing enhancers, ISEs) 4. сайленсеры сплайсинга интронов (intronic splicing silencers, ISSs)

Регуляторы, помогающие распознать сайт сплайсинга • Белки SR (серин(S)/аргинин(R)-богатые белки) играют важную роль в распознавании сайта сплайсинга. Например, когда они взаимодействуют с энхансерами экзонов (ESE), они помогают связываться U 1 с 5’-сайтом сплайсинга, а также U 2 AF и U 2 с 3’-сайтом сплайсинга. Эти взаимодействия (SR c энхансерами) происходят благодаря их RS доменам (содержат Arg-Ser повторы). Белки SR могут работать вместе с другими положительными регуляторами. • SR белок SRp 38 (также известный как TASR, NSSR, FUSIP 1 и SRrp 40) в дефосфорилированном действует как репрессор сплайсинга. Однако, недавние исследования показали, что в фосфорилированном состоянии он также функционирует в качестве активатора, зависящего от сплайсируемой последовательности

Ингибирование распознавания сайта сплайсинга • Ингибировать распознавание сайта сплайсинга можно разными способами. Первый, когда сайленсеры сплайсинга располагаются близко к сайту сплайсинга или к энхансерам сплайсинга, и, соответственно, может происходить стерическое ингибирование, при котором блокируется доступ к sn. RNP или положительным регуляторным факторам. Например, полипиримидин-связывающий белок (PTB; также известный как PTB 1 и hn. RNP I), связывается с полипиримидиновым участком и, тем самым, блокирует связывание U 2 AF с экзонами. Тканеспецифичные факторы сплайсинга FOX 1 и FOX 2, связываясь с интронной последовательностью, могут ингибировать формирование комплекса E', предотвращая связывание SF 1 с точкой разветвления. • Ингибиторы сплайсинга могут стерически мешать связыванию активаторов с энхансерами. Семейство белков Hu/ELAV ингибирует связывание U 1, конкурируя с белком TIA 1, который взаимодействует с AU-богатой областью, расположенной далее по последовательности в 5’-сайте сплайсинга экзона 23 а нейрофиброматозного типа 1. FOX 1 и FOX 2 также ингибируют формирование комплекса Е, связываясь с экзонной последовательностью, близко расположенной от ESE, где связываются TRA 2 и SRp 55. Из-за этого U 2 AF не может закрепиться

Регуляция сплайсинга в зависимости от позиции экзона • Действие цис-регуляторных элементов иногда зависит от позиции регулируемого экзона. Несколько белков, например такие как NOVA 1, NOVA 2, FOX 1, FOX 2, hn. RNP l-подобный, hn. RNP F и hn. RNP H, могут действовать либо как репрессоры, либо как активаторы в зависимости от места сайта связывания. • Позиция сплайсинга может определять действие факторов сплайсинга. Энхансеры могут располагаться таким образом, что, когда факторы сплайсинга связываются с ними, другой экзон лучше расположен для действия сплайсосомы. Или наоборот, элементы сайленсинга соревнуются с компонентами сплайсосомы или как-то изменяют структуру м. РНК, препятствуя узнаванию сайта сплайсинга.

Методы обнаружения альтернативного сплайсинга • Основным методом определения мест альтернативного сплайсинга в данный момент является секвенирование библиотек к. ДНК, полученных на матрице м. РНК. Данный метод называется секвенированием РНК. • Также возможно применение ДНК-микрочипов.

Альтернативный сплайсинг и заболевания Изменения в альтернативном сплайсинге могут вызывать разные заболевания: нейродегенеративные, сердечно-сосудистые, дыхательные заболевания, а также рак и болезнь Альцгеймера. В тканях, где происходит множество процессов (например головной мозг), требуется большое количество белков для выполнения различных функций. Любой дефект в альтернативном сплайсинге может резко изменить состав белков в клетке, вследствие чего и возникнут многие неврологические заболевания. Эти дефекты могут быть разделены на две основные группы: первичные и вторичные дефекты сплайсинга.

Первичные дефекты сплайсинга При первичных дефектах сплайсинга происходит мутация в последовательности, которая важна для правильного прохождения альтернативного сплайсинга. Следовательно, сплайсинг не происходит должным образом. Многие мутации, приводящие к заболеваниям, вызваны нарушениями сплайсинга пре-м. РНК. Примерами являются синдром Луи-Бара и нейрофиброматоз. Половина пациентов, страдающих этими заболеваниями, несет мутации, которые влияют на прохождение сплайсинга пре-м. РНК. Другой пример первичных нарушений сплайсинга был обнаружен у пациентов с лобновисочной деменцией (аутосомно-доминантное расстройство, связано с 17 хромосомой). Дефекты в сплайсинге приводят к изменению транскриптов тау-белков, ассоциированных с микротрубочками, - MAPT. Эти белки накапливаются в аксонах или растущих нейронах. Изменения в сплайсинге MAPT могут также привести к другим заболеваниям, таким как болезнь Альцгеймера, мышечная дистрофия, дисфункция глии и дегенерация спинного мозга. Мышечная дистрофия Дюшенна (МДД) — это результат мутаций в гене дистрофина. Мутации вызывают нарушения в сплайсинге и продукцию неправильных м. РНК. Разные мутации в пре-м. РНК дистрофина вызывают промежуточный вариант МДД, мышечную дистрофию Беккера (МДБ).

Вторичные дефекты сплайсинга Под вторичными дефектами сплайсинга подразумеваются мутации в регуляторных факторах, которые очень важны для процесса сплайсинга. Действие различных активаторов и/или репрессоров может определять пути прохождения альтернативного сплайсинга. В зависимости от того, какой регулятор действует, пре-м. РНК меняется. В некоторых случаях, это может привести к серьёзным нарушениям. Примером таких нарушений является Синдром Прадера-Вилли, генетическая болезнь, характеризующаяся интеллектуальными и поведенческими отклонениями. Фермент ацетилхолинэстераза (ACh. E) собирается из серии белков. В ходе альтернативного сплайсинга может получаться три изоформы белка, которые вызывают такие нейродегенеративные заболевания, как болезнь Альцгеймера и болезнь Паркинсона. Разбалансировка регуляторов сплайсинга может приводить к избытку ACh. E-R, изоформе ACh. E, найденной у пациентов, страдающих болезнью Альцгеймера.


RNA Splicing

Number of introns per gene in various eukaryotic species

THE CHEMISTRY OF RNA SPLICING Sequences within the RNA determine where splicing occurs

The intron is removed in a form called lariat and the flanking exons are joined

The structure of the three-way junction formed during the splicing reaction

Exons from different RNA molecules can be fused by Trans-splicing

Outline of the mechanisms used for three types of RNA splicing. (A) Three types of spliceosomes. The major spliceosome (left), the AT–AC spliceosome (middle), and the trans-spliceosome (right) are each shown at two stages of assembly. The U 5 sn. RNP is the only component that is common to all three spliceosomes. Introns removed by the AT–AC spliceosome have a different set of consensus nucleotide sequences from those removed by the major spliceosome. In humans, it is estimated that 0. 1% of introns are removed by the AT–AC spliceosome. In trans-splicing, the SL sn. RNP is consumed in the reaction because a portion of the SL sn. RNA becomes the first exon of the mature m. RNA.

THE SPLICEOSOME MACHINERY RNA splicing is carried out by a large complex called the spliceosome sn. RNP (small nuclear ribonuclear proteins)

Some RNA-RNA hybrids formed during the splicing reaction

Assembly, rearrangements, and catalysis within the spliceosome Steps of spliceosome-mediated splicing reaction

Several of the rearrangements that take place in the spliceosome during pre-m. RNA splicing. Shown here are the details for the yeast Saccharomyces cerevisiae, in which the nucleotide sequences involved are slightly different from those in human cells. (A) The exchange of U 1 sn. RNP for U 6 sn. RNP occurs before the first phosphoryl-transfer reaction (see Figure 6– 29). This exchange allows the 5' splice site to be read by two different sn. RNPs, thereby increasing the accuracy of 5' splice site selection by the spliceosome. (B) The branch-point site is first recognized by BBP and subsequently by U 2 sn. RNP; as in (A), this “check and recheck” strategy provides increased accuracy of site selection. The binding of U 2 to the branch-point forces the appropriate adenine (in red) to be unpaired and thereby activates it for the attack on the 5' splice site (see Figure 6– 29). This, in combination with recognition by BBP, is the way in which the spliceosome accurately chooses the adenine that is ultimately to form the branch point.

The spliceosome uses ATP hydrolysis to produce a complex series of RNA-RNA rearrangements . (C) After the first phosphoryl-transfer reaction (left) has occurred, U 5 sn. RNP undergoes a rearrangement that brings the two exons into close proximity for the second phosphoryl-transfer reaction (right). The sn. RNAs both position the reactants and provide (either all or in part) the catalytic site for the two reactions. The U 5 sn. RNP is present in the spliceosome before this rearrangement occurs; for clarity it has been omitted from the left panel. As discussed in the text, all of the RNA–RNA rearrangements shown in this figure (as well as others that occur in the spliceosome but are not shown) require the participation of additional proteins and ATP hydrolysis.

Self-splicing introns reveal that RNA can catalyze RNA splicing

Group I introns release a linear intron rather than a lariat

Proposed folding of the RNA catalytic regions for splicing of group II intron and pre-m. RNA

The Nobel Prize in Chemistry 1989 "for their discovery of catalytic properties of RNA" Sidney Altman Thomas R. Cech 1/2 of the prize Canada and USA Yale University New Haven, CT, USA University of Colorado Boulder, CO, USA b. 1939 b. 1947

How does the spliceosome find the splice sites reliably Errors produced by mistakes in splice-site selection

SR proteins recruit spliceosome components to the 5’ end and 3’ splice sites

A small group of introns are spliced by an alternative spliceosome composed of a different set of sn. RNps

ALTERNATIVE SPLICING Single gene can produce multiple products by alternative splicing

Five ways to splice an RNA

Alternative splicing of SV 40 T-antigen

Several mechanisms exist to ensure mutually exclusive splicing Steric hindrance

Combination of major and minor splice sites

Mutually exclusive splicing on a grand scale The multiple exons of the Drosophila Dscam gene

Splicing of Dscam Exon 6 uses a novel strategy

Alternative splicing is regulated by activators and repressors

Inhibition of splicing by hn. RNPI

Regulation of alternative splicing determines the sex of flies Early transcriptional regulation of Sx. I protein

A cascade of alternative splicing events determines the sex of a fly

EXON SHUFFLING Exons are shuffled by recombination to produce genes encoding new proteins

Genes made up parts of other genes


Evolutionary change in proteins. Domain shuffling.


there are two ways the splicing of one gene can change: mutations in cis mutations in trans DNA

1. The phenotype is determined by the proteome & transcriptome. 2. Selection acts on the phenotype, and is blind to the genotype. Therefore: two species/individuals that have different forms of a protein will be selected differently - even if the genes DNA sequence is identical. DN A DNA m. RNA 10% Species #1 (cell type 1) m. RNA 90% Species #2 (cell type 1)

Implications of alternative splicing in the evolution of a protein: ’Trying out’ a new domain splice site weak splice site present in intron weak splice site strengthened by a mutation both splice sites now used initial protein two proteins forms produced, one with a new domain derived from intron sequence

Modrek 2003

Alt. Exons that encode entire domains are selected for. Kriventseva 2003

Alternative splice sites are more likely to fall between domains than constitutive exons. Kriventseva 2003

New exons are shorter than average Fyodor 2003

Microarray Method of Splice Variant Detection

Each spot is the signal from one probe The colour is transformed into number by a scanner Strengths: • Can be scaled up (to an entire genome…) • Any splice variant type • Amenable to high throughput mathmatics & stats. Limitations: • Not very accurate • Complex, expensive

• alternative splicing affects the way that genomes evolve and the way that we think about genome complexity • How many proteins are produced in eukaryotic genomes? • How many genes do you need to make a complex multicellular organism? • How can the production of many splice variants contribute to the exploration of ’genomic sequence space’? • How stable are splice sites in evolution?

• Proteins evolve by many processes over long periods of time • Most genes in complex eukaryotes are alternatively spliced • It is not known how quickly alternative splicing evolves • We will compare orthologous transcripts in two species of nematodes to examine this ‘rate of evolution’ Using microarrays and RT-PCR Our arrays work effectively with synthetic RNAs, but are not very sensitive • RT-PCR is sensitive, but cant be scaled up

How are the splice sites identified? outcomes of 5’ ss mutants x 1. activates cryptic 5’ ss, but only if there is one within 100 -300 bp of original 5’ ss x 2. skip the exon altogether and ignore perfectly good 3’ and 5’ ss of the upstream intron

How are the splice sites identified? beta-globin mutants that create a new 3’ ss within an intron: x also create a new exon? ? ?

In multicellular organisms, exons are recognized as units prior to assembly of the spliceosome across the long introns. This “exon definition” step involves interactions between the splice sites across the exon and special sequences in the exon called Exonic Splicing Enhancers (ESE). The sequences in exons are selected to not just code for particular peptide sequences, but also for binding of regulatory proteins to ESE’s.

How are the splice sites identified? Boundaries between introns & exons are recognized through its interaction with multiple proteins either across exon or intron Intron definition: Uses intron as the unit of recognition mechanism. Complex forms through stabilized Exon 1 protein interactions across the intron RS 70 K SR SR SR U 2 AF 35 U 2 AF RS SF 2 RS Exon 2 A SF 1 U 1 sn. RNP Intron Definition SR Exon Definition: Complex can easily form stabilized protein interactions across the exon. Excises out the flanking introns SR U 2 AF 35 U 2 AF RS RS 70 K SR A RS SF 2 Exon SF 1 (Cote, Univ. of Ottawa) Exon Definition U 1 sn. RNP Stable interaction confirms accuracy of splice site choice

Why are exons preferentially recognized? Differential size distributions of exons (~50 to 300 nt) vs. introns (<100 -100, 000 nt) • SR protein - preferentially binds to exon sequences - mark the 5’ & 3’ splicing sites in conjunction w/ U 1 & U 2 during transcription • hn. RNP - heterogenous nuclear ribonucleoproteins (twice the diameter of nucleosome) - consists at least eight different proteins - compacts introns, thereby masking cryptic splicing sites - preferentially binds to introns, but also bind to exons, although less frequently

Cross-exon bridging interactions involve SR domains of U 2 AF, U 170 K And 1 or more SR-family proteins • ~12 in mammals (and # AS isoforms!) • Tissue-specific differences in concentration • RRMs vary in degree of sequence preferences Outstanding question: What triggers the switch from Exon- to Intron-Defined interactions?

Vertebrate external exons

Splicing is co-transcriptional and all introns assayed are spliced within 5 -10 minutes of transcription of the downstream exon and 3’ splice site, regardless of intron size (1 kb or 240 kb)

Defining an exon involves the specific stabilization or destabilization of splice site recognition Stabilization: exon inclusion Destabilization: exon skipping

Regulation of alternative splicing involves the specific stabilization or destabilization of splice site recognition Stabilization: exon inclusion Destabilization: exon skipping

How would you identify cis-regulatory sequences responsible for alternative splicing ? Mutational analysis finds an element necessary for exon inclusion Transfection Reporter Plasmid Alternatively spliced Not alternatively spliced Examine RNA Splicing of Transfected Splicing Reporters to identify cis-regulatory regions

Four classes of splicing regulatory elements: Exonic Splicing Enhancers, Exonic Splicing Silencers (ESS), Intronic Splicing Enhancers (ISE), and Intronic Splicing Silencers (ISS). + 1 3 - 1 2 1 - 2 + ESE 3 2 ESS 3 ISE 3 ISS

How would an Intronic Splicing Silencer work

SR proteins generally bind ESE, ESS, ISE, and ISSs

The SR Proteins are a family of proteins with a common domain structure of 1 or 2 RNP RNA binding domains (also called RRMs) and a C-terminal domain rich in SR dipeptides. These proteins are involved in many aspects of splicing, but most significantly they bind to Exonic Splicing Enhancers (ESEs) and stimulate spliceosome assembly at the adjacent sights. It is thought that most exons carry ESE’s and require SR proteins for exon recognition.

SR Proteins bind to specific RNA elements using their RNA binding domains similar to those in the Sex-Lethal protein.

Characterization of an ESE and SR protein in flies ·Sex differentiation in flies controlled by AS Cascade ·Dsx: weak 3’SS next to female-specific exon ·Tra/Tra 2 (females) promotes recruitment of U 2 AF ·Sequence-specific RRM -> binds 13 -nte. Repeats ·RS domain interacts w U 2 AF RS domain Proof of concept: Convert ESE to MS 2 binding site -> activated by MS 2: RS

hn. RNP function at ISSs hn. RNP contain RRMs but not SR domain Can block sterically, tighter binding affinity than U 2 AF

SR Proteins bind to CTD of pol. II: promote co-transcriptional splicing?

CTD of RNA pol II plays important role in pre-m. RNA splicing (Kornblihtt et al, 2004)

Does splice site strength affect alternative splicing?

A connection between chromatin and splicing include exon. IIIc by repress exon. IIIb include exon. IIIb, repress exon IIIc, via Epithelial splicing regulatory protein



Splicing regulation SR proteins create ’bridges’ to stabilize the spliceosome hn. RNP splicing enhancer splicing silencer In trypanosomes: • U 2 F 65 and 35 exist and do not interact. • U 2 F 65 interacts with SF 1. • Interacting SR proteins were identified. • hn. RNP proteins exist.
D--Users-CHELOMINA-Desktop-МАГИСТРЫ-2016-альтернативный сплайсинг.ppt