fbe6ea83f64949cef876cf3098fbdfbf.ppt
- Количество слайдов: 61

Скрытая наследуемость (Missing Heritability Problem) Рубанович А. В. Институт общей генетики им. Н. И. Вавилова РАН rubanovich@vigg. ru

6 ноября 2008 Brendan Maher вводит термин «missing heritability»

В чем проблема и откуда она взялась? GWAS (2002) - Genome. Wide Association Studies Ведь мы просмотрели все (или почти все? ) В начале века мы научились генотипировать одновременно сильно изменчивые сайты в генах по всем наиболее изменчивым генам (GWAS или широкогеномное сканирование) Оказалось: Мы отличаемся друг от друга примерно каждым тысячным нуклеотидом (1 -2 SNP на 1000 п. н. ), 99. 9% нуклеотидов но эффекты этих различий слишком малы, чтобы объяснить у нас одинаковы! изменчивость и наследуемость большинства признаков 1, 1 vs. 15 ! Что значит «слишком малы» : Где остальная наследуемость? Если (не дай Бог!) у Ваших родителей диабет I типа, то для Вас вероятность заболеть диабетом в 15 раз выше, чем для всех прочих. Т. е. признак наследуется, и должны быть генетические различия между здоровыми и диабетиками. GWAS находит эти различия - аллельные варианты для ~ 20 -40 SNP , p < 10 -8, но эффект мизерный! но при этом среди носителей «генотипов предрасположенности» доля больных диабетом лишь в 1, 1 раз больше

Still missing! Публикации 2010 -2011 (Nature, PNAS, Plo. S)

Красивая аналогия с астрофизикой: «dark matter» - 83% вселенной Замкнутая картина вселенной невозможна без постулата о существовании скрытой от нас «темной материи» «Missing mass» - Fritz Zwicky (1933) «Серебряная» Пуля Темная материя вокруг галактик Гало из «темной материи» , вокруг нашей Milky Way (компьютерная реконструкция)

Припомним некоторые понятия, чтобы говорить на одном языке Виды генетической изменчивости: q Структурная изменчивость: делеции, инсерции, дупликации (CNV), тандемные повторы (STR, VNTR) q Изменчивость отдельных нуклетидов: SNP – их более 10 млн. …GATCAGGCGATTACACGCAATG… …GATCATGCGATTACCCGCAATG… …GATCAGGCGATTACACGCAATG… …GATCAGGCGATTACCCGCAATG… …GATCATGCGATTACACGCAATG… …GATCAGGCGATTACCCGCAATG… SNP 1 G>T SNP 2 C>A Для 2 SNP возможны 4 гаплотипа GC GA TC TA Если SNP 1 и SNP 2 не сцеплены, то PGC = PG PC D = PGC – PG PC , D` = D/Dmax Неравновесие по сцеплению: D` = 1, если не все возможные гаплотипы встречаются в популяции (например, только GC, GA, TC) При D` ~ 1 в данных по SNP 1 содержится информация о SNP 2 (и наоборот)
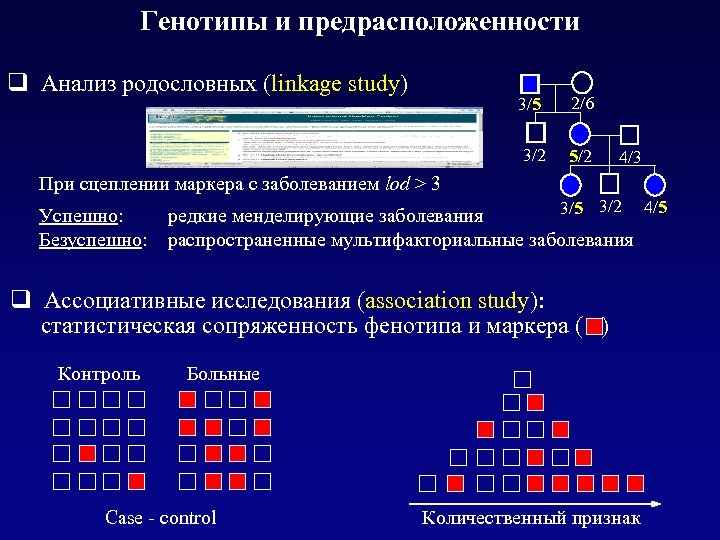
Генотипы и предрасположенности q Анализ родословных (linkage study) 3/5 2/6 3/2 5/2 4/3 При сцеплении маркера с заболеванием lod > 3 3/5 3/2 4/5 Успешно: редкие менделирующие заболевания Безуспешно: распространенные мультифакториальные заболевания q Ассоциативные исследования (association study): статистическая сопряженность фенотипа и маркера ( ) Контроль Больные Case - control Количественный признак
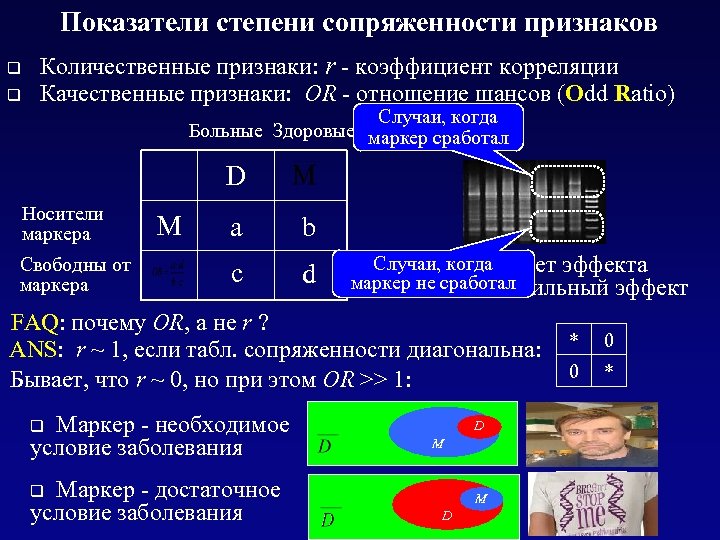
Показатели степени сопряженности признаков q Количественные признаки: r - коэффициент корреляции q Качественные признаки: OR - отношение шансов (Odd Ratio) Случаи, когда Больные Здоровые маркер сработал D Носители маркера Свободны от маркера M a b c d Случаи, когда OR = 1 - нет эффекта маркер не сработал OR >> 1 - сильный эффект FAQ: почему OR, а не r ? ANS: r ~ 1, если табл. сопряженности диагональна: * 0 Бывает, что r ~ 0, но при этом OR >> 1: Маркер - необходимое условие заболевания q Маркер - достаточное условие заболевания M M D * * * 0 D q 0 * *

Как характеризуют наследуемость? q В широком смысле: Доля изменчивости (вариансы, дисперсии) признака, Если Вы думаете, что r = 0. 7 – это «хорошая» корреляция, вспомните, что при обусловленная генетической изменчивостью этом изменчивость Y лишь на 50% обусловлена изменчивостью X (0. 72 = 0. 49) Это определение восходит к регрессионному анализу: Y ~ X Пока это все только слова – для вычисления корреляций нужны числа. Коэффициент Cor(Y, X)2 = Каким числом характеризовать генотип? детерминации = Доля изменчивости Y, обусловленная X Наследуемость = (Корреляция «генотип - фенотип» )2 Простейшая модель –аддитивность действия генов. Например, в случае, когда Р - предрасположенность к заболеванию Общее число «рисковых» аллелей G = во всех генах предрасположенности = GA q В узком смысле: = Cor(P, GA)2 = Доля «аддитивно объясненной» изменчивости фенотипа Всегда
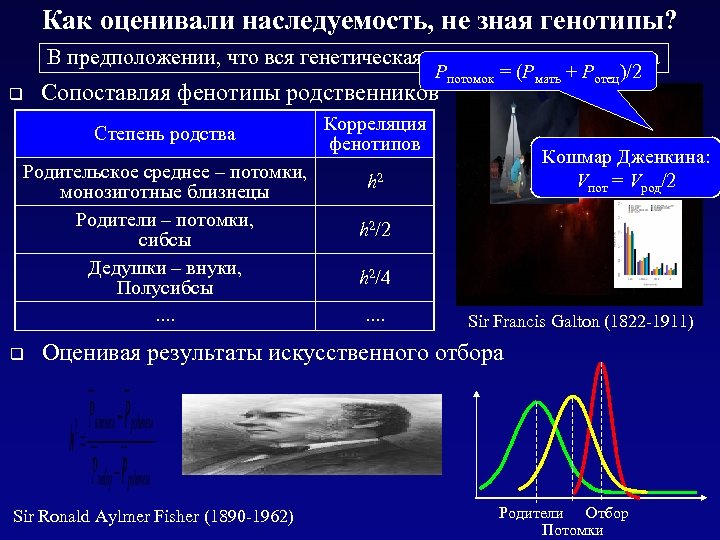
Как оценивали наследуемость, не зная генотипы? В предположении, что вся генетическая изменчивость аддитивна Рпотомок = (Рмать + Ротец)/2 q Сопоставляя фенотипы родственников Степень родства Корреляция фенотипов Родительское среднее – потомки, монозиготные близнецы Родители – потомки, сибсы Дедушки – внуки, Полусибсы h 2/2 …. Кошмар Дженкина: Vпот = Vрод/2 h 2/4 Sir Francis Galton (1822 -1911) q Оценивая результаты искусственного отбора Sir Ronald Aylmer Fisher (1890 -1962) Родители Отбор Потомки

Как оценивали наследуемость, не зная генотипы? В предположении, что вся генетическая изменчивость аддитивна q Сопоставляя фенотипы родственников Степень родства Корреляция фенотипов Родительское среднее – потомки, монозиготные близнецы Родители – потомки, сибсы Дедушки – внуки, Полусибсы h 2/2 …. Самое удивительное, что это работает! …. h 2 r. MZ = 2 r. DZ Для 86 признаков r. MZ отличается в среднем от h 2/4 Hill et. al, Plo. S Gen. , 2008 2 r. DZ не более, чем на 0, 5% q Оценивая результаты искусственного отбора r. MZ - 2 r. DZ
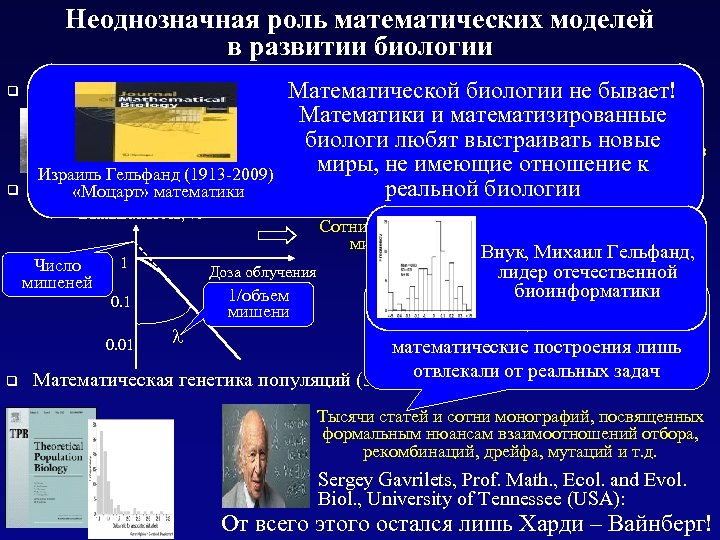
Неоднозначная роль математических моделей в развитии биологии Математической биологии не бывает! Математики и математизированные Тысячи работ с «оценками» наследуемости и числа биологи любят выстраивать новые генов по результатам селекционных экспериментов миры, не имеющие отношение к Израиль Гельфанд (1913 -2009) q «Оценки» числа мишеней и числа ударов в радиобиологии (40 -70 г. г. ) реальной биологии «Моцарт» математики q «Оценки» числа генов, ответственных за количественный признак (50 -60 г. г. ) Выживаемость, % Сотни работ с «оценками» числа и объемов мишеней по кривым выживаемости Внук, Михаил Гельфанд, Доза облучения лидер отечественной биоинформатики 1/объем Современные биоинформатики 0. 1 мишени полагают, что все эти 0. 01 математические построения лишь отвлекали от реальных задач q Математическая генетика популяций (30 -80 г. г) Число мишеней 1 Тысячи статей и сотни монографий, посвященных формальным нюансам взаимоотношений отбора, рекомбинаций, дрейфа, мутаций и т. д. Sergey Gavrilets, Prof. Math. , Ecol. and Evol. Biol. , University of Tennessee (USA): От всего этого остался лишь Харди – Вайнберг!
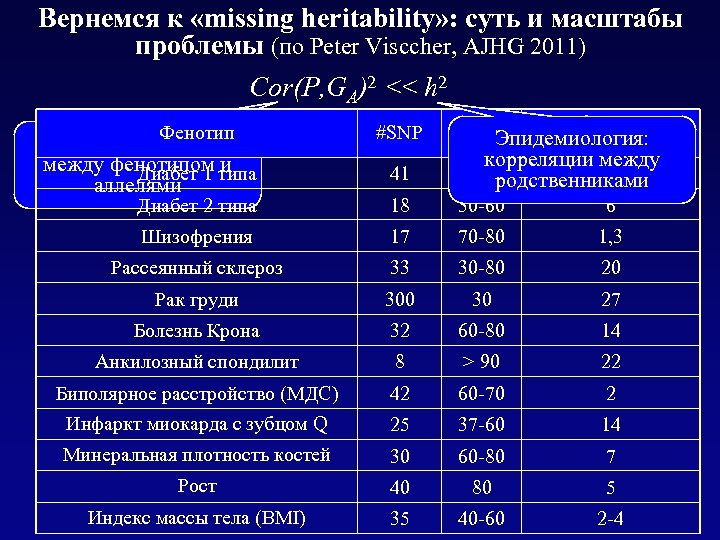
Вернемся к «missing heritability» : суть и масштабы проблемы (по Рeter Visccher, AJHG 2011) Cor(P, GA)2 << h 2 Фенотип GWAS: корреляции между фенотипом и Диабет 1 типа аллелями #SNP 41 % объясненной h 2, % Эпидемиология: наследуемости корреляции между 90 родственниками 30 Диабет 2 типа 18 30 -60 6 Шизофрения 17 70 -80 1, 3 Рассеянный склероз 33 30 -80 20 Рак груди 300 30 27 Болезнь Крона 32 60 -80 14 Анкилозный спондилит 8 > 90 22 Биполярное расстройство (МДС) Инфаркт миокарда с зубцом Q 42 60 -70 2 25 37 -60 14 Минеральная плотность костей 30 60 -80 7 Рост 40 80 5 Индекс массы тела (BMI) 35 40 -60 2 -4

Вернемся к «missing heritability» : суть и масштабы Генетика – не самая молодая наука. Почему только сейчас?

Генотипы и предрасположенности Хроники по Walter Bodmer (Nat. Genet. , 2008) Группы крови (1910 -1960) ~20 loci Иммуногенетика ~20 loci (1965 -…) HLA (Хр 6) Форез белков (1960 -1980) ~100 loci RFLP (1975 -…) >105 loci Ford (1945) – полиморфизм АВ 0 AB 0 и рак желудка (Aird, 1953) OR = 1. 3 4 с и болезнь Ходжкина (Amiel, 1967) OR = 2. 8 Анкилозный спондилит, псориаз, … OR = 100! Механизм до сих пор Осознание тотального характера неясен! генетической изменчивости Маркеры при анализе Убедительных и регулярных родословных, судебная взаимосвязей с фенотипами медицина не обнаружено ATCATCATC… VNTR, STR (1985 -…) >105 loci Гипервариабельны: эволюционные древа, идентификация личности GATCAGGCGA GATCCTGCGA GATCAGGCGA SNP (1998 -…) >108 loci PCR-генотипирование кандидидатных локусов, GWAS
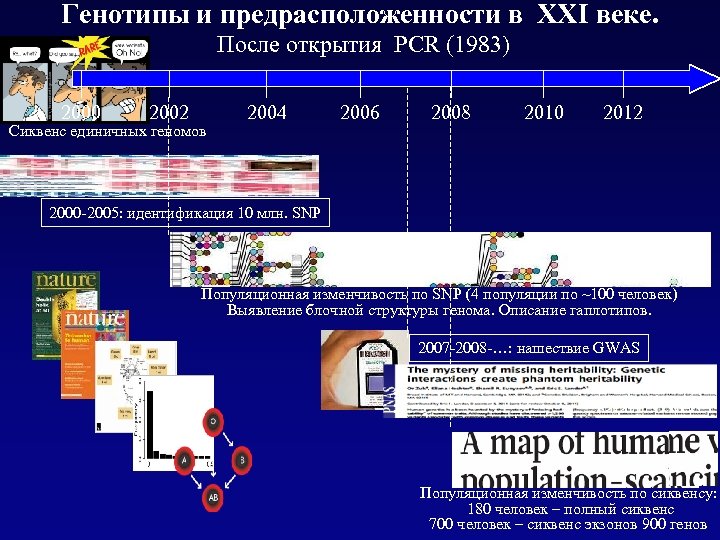
Генотипы и предрасположенности в XXI веке. После открытия PCR (1983) 2000 2002 Сиквенс единичных геномов 2004 2006 2008 2010 2012 2000 -2005: идентификация 10 млн. SNP Популяционная изменчивость по SNP (4 популяции по ~100 человек) Выявление блочной структуры генома. Описание гаплотипов. 2007 -2008 -…: нашествие GWAS Каталог GWAS Популяционная изменчивость по сиквенсу: 180 человек – полный сиквенс 700 человек – сиквенс экзонов 900 генов

Единый каталог гаплотипов по SNP www. hapmap. org Цель проекта: характеризация изменчивости нуклеотидов, которая встречается во всех популяциях человека - т. н. «common SNP» . Для них, как правило, Minor Allele Frequency (MAF) > 0. 1 Представлены только SNP! Между соседними буквами сотни База первичных данных доступна всем и выглядит так: консервативных нуклеотидов Популяция YRI, CEU, CHB либо JPT + 7 популяций (2007) # Гаплотипы по всем хромсомам GCCCAAATGCC…GCTTAGCACA ~ 60 -100 4. 7 млн. SNP (2010) «Горячая точка» Основная неожиданность: гаплотипы всех хромосом имеют блочную структуру, рекомбинации (их ~330 000) т. е. распадаются на независимые блоки, состоящие из скоррелированных SNP
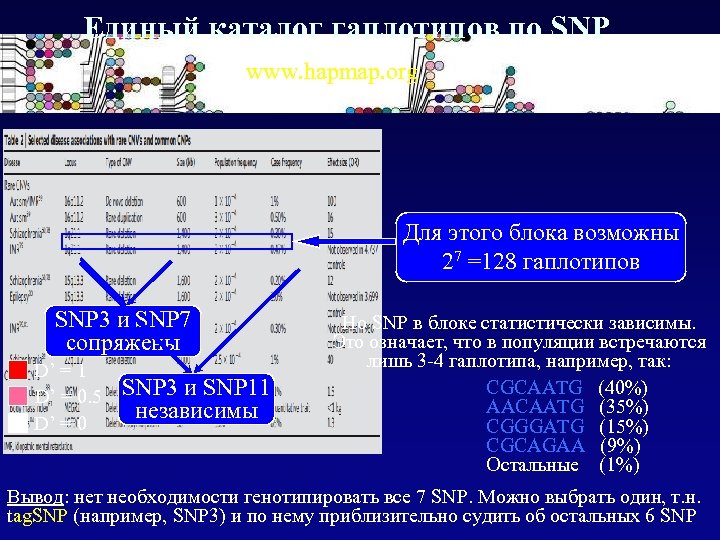
Единый каталог гаплотипов по SNP www. hapmap. org Для этого блока возможны 27 =128 гаплотипов SNP 3 и SNP 7 сопряжены D’ = 1 D’ = 0. 5 D’ = 0 SNP 3 и SNP 11 независимы Но SNP в блоке статистически зависимы. Это означает, что в популяции встречаются лишь 3 -4 гаплотипа, например, так: CGCAATG (40%) AACAATG (35%) CGGGATG (15%) CGCAGAA (9%) Остальные (1%) Вывод: нет необходимости генотипировать все 7 SNP. Можно выбрать один, т. н. tag. SNP (например, SNP 3) и по нему приблизительно судить об остальных 6 SNP
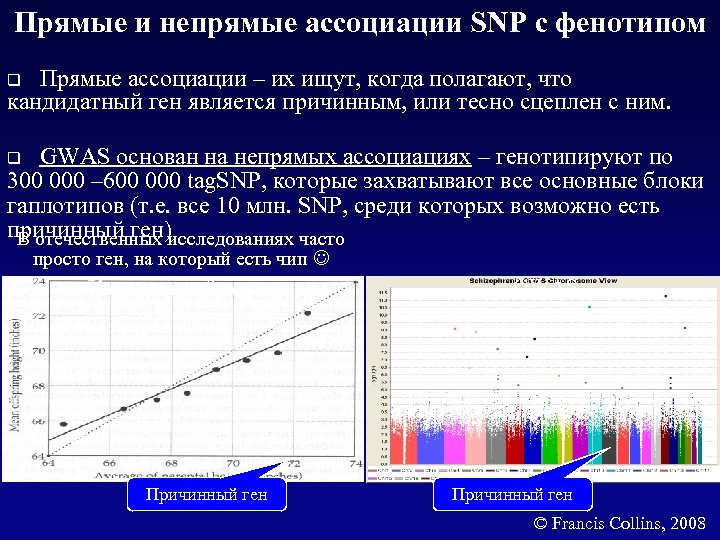
Прямые и непрямые ассоциации SNP с фенотипом q Прямые ассоциации – их ищут, когда полагают, что кандидатный ген является причинным, или тесно сцеплен с ним. q GWAS основан на непрямых ассоциациях – генотипируют по 300 000 – 600 000 tag. SNP, которые захватывают все основные блоки гаплотипов (т. е. все 10 млн. SNP, среди которых возможно есть причинный ген) В отечественных исследованиях часто просто ген, на который есть чип Кандидатный ген Причинный ген GWAS Причинный ген © Francis Collins, 2008
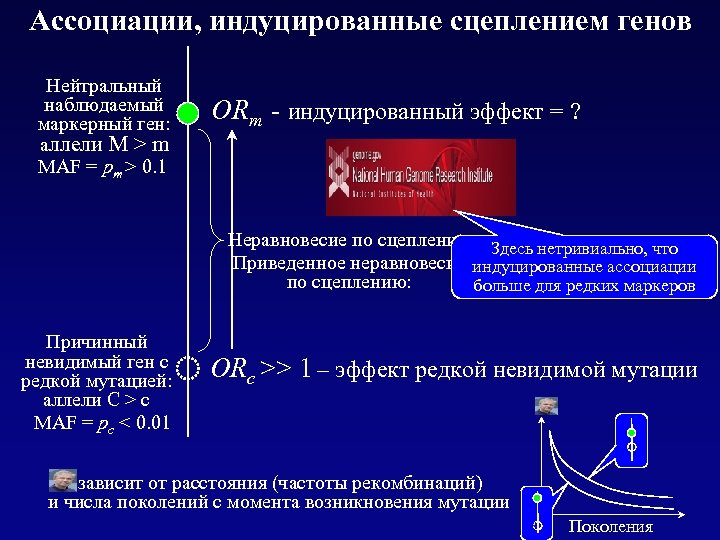
Ассоциации, индуцированные сцеплением генов Нейтральный наблюдаемый маркерный ген: аллели M > m MAF = pm > 0. 1 ORm - индуцированный эффект = ? Неравновесие по сцеплению: Здесь нетривиально, что D = pc, m – pc pm Приведенное неравновесие индуцированные ассоциации по сцеплению: больше для редких маркеров Причинный невидимый ген с редкой мутацией: аллели С > c MAF = pc < 0. 01 ORc >> 1 – эффект редкой невидимой мутации зависит от расстояния (частоты рекомбинаций) и числа поколений с момента возникновения мутации Поколения

Ассоциации, индуцированные сцеплением генов Нейтральный наблюдаемый маркерный ген: аллели: M > m MAF = pm > 0. 1 ORm - индуцированный эффект = ? Неравновесие по сцеплению: Приведенное неравновесие по сцеплению: Причинный невидимый ген с редкой мутацией: аллели С > c MAF = pc < 0. 01 D = pc, m – pc pm ORc >> 1 – эффект невидимый редкой мутации Скорей всего, почти всегда из 4 бывают лишь 3 гаплотипа: М C m C М c m c Тогда и

Как выглядят результаты GWAS? ATGGATTCTGGTATGTTCTAGCGCTTGCACCATCCCATTTAACTGTAAGAAGAAT TGCACGGTCCCAATTGCTCGAGAGATTTCTCTTTTACCTTTTTTTACTATTTTTCAC TCTCCCATAACCTCCTATATTGACTGATCTGTAATAACCACGATATTATTGGAAT AAATAGGGGCTTGAATTTGGAAAAAAACTGAAATATTTTCGTGATAAG TGATAGTGATATTCTTCTTTTATTTGCTACTGTTACTAAGTCTCATGTACTAACAT CGATTGCTTCATTCTTTTTGTTGCTATATGTTTAGAGGTTGCTGCTTTGGTT ATTGATAACGGTTCTGGTATGTGTAAAGCCGGTTTTGCCGGTGACGACGCTCCTC GTGCTGTCTTCCCATCTATCGTCGGTAGACACCAAGGTATCATGGTCGGT ATGGGTCAAAAAGACTCCTACGTTGGTGATGAACTCAATCCAAGAGAGGTATCT TGACTTTACGTTACCCAATTGAACACGGTATTGTCACCAACTGGGACGATATGGA Последовательности длиной 500 000 – 1 000 AAAGATCTGGCATCATACCTTCTACAACGAATTGAGAGTTGCCCCAGAAGAACA нуклеотидов для огромных выборок: CCCTGTTCTTTTGACTGAAGCTCCAATGAACCCTAAATCAAACAGAGAAAAGAT GACTCAAATTATGTTTGAAACTTTCAACGTTCCAGCCTTCTACGTTTCCAAG несколько тысяч больных и столько же здоровых! CCGTTTTGTCCTTGTACTCTTCCGGTAGAACTACTGGTATTGTTTTGGATTCCGGT GATGGTGTTACTCACGTCGTTCCAATTTACGCTGGTTTCTCTCTACCTCACGCCAT TTTGAGAATCGATTTGGCCGGTAGAGATTTGACTACTTGATGAAGATCTTG AGTGAACGTGGTTACTCTTTCTCCACCACTGCTGAAAGAGAAATTGTCCGTGACA При меньших выборках эффекты будут заведомо TCAAGGAAAAACTATGTTACGTCGCCTTGGACTTCGAACAAGAAATGCAAACCG CTGCTCAATCTTCTTCAATTGAAAAATCCTACGAACTTCCAGATGGTCAAGTCAT статистически незначимы (105 -106 сравнений ) CACTATTGGTAACGAAAGATTCAGAGCCCCAGAAGCTTTGTTCCATCCTTCTGTT TTGGGTTTGGAATCTGCCGGTATTGACCAAACTACTTACAACTCCATCATGAAGT GTGATGTCCGTAAGGAATTATACGGTAACATCGTTATGTCCGGTGGTAC CACCATGTTCCCAGGTATTGCCGAAAGAATGCAAAAGGAAATCACCGCTTTGGC TCCATCTTCCATGAAGGTCAAGATCATTGCTCCTCCAGAAAGTACTCCGTC

Как выглядят результаты GWAS: Manhattan Plot – достоверность вдоль хромосом Логарифм P-value 8 SNP, ассоциированных с шизофренией p = 10 -7 p = 10 -5 При GWAS необходим уровень значимости эффектов SNP ~ 10 -8 - 10 -7. По Бонферрони: 0. 05/5 105 = 10 -7 22 хромосомы

почечная карцинома GWAS of renal cell carcinoma identifies two susceptibility loci on 2 p 21 and 11 q 13. 3 M. Purdue, …(97 авторов)…, P. Brennan. Nature Genetics, 2011, 43, 1 Пики частоты рекомбинации Только для 2 SNP значимость ассоциаций подтвердилась в 2 независимых исследованиях (OR = 1. 18, p = 10 -7, MAF = 0. 9)
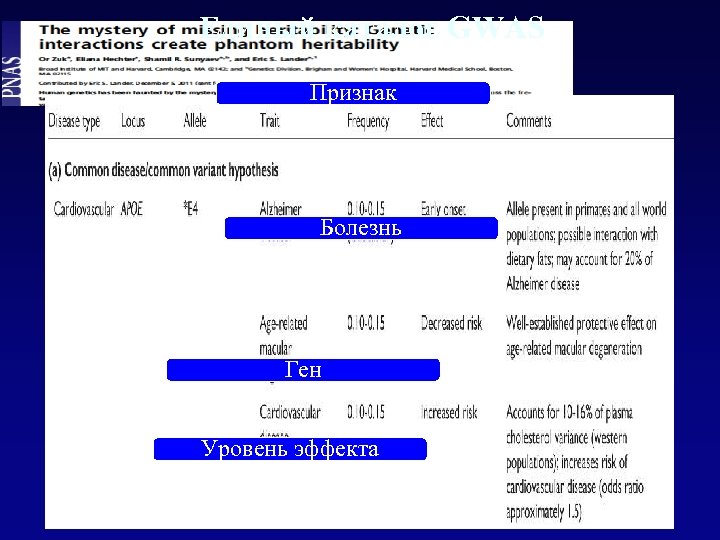
Единый каталог GWAS Признак Болезнь Ген Уровень эффекта

Сюрпризы GWAS Результаты GWAS устойчиво демонстрируют: L Плохую воспроизводимость В различных GWAS находят непересекающиеся группы ассоциированных SNP L Эффекты отдельных SNP крайне малы: При этом в чипах стараются tag. SNP Предрасположенность к заболеваниям - OR = 1. 1 – 1. 3 особенно плотно расположить Количественные признаки – менее 1% объясненной вариансы именно в кодирующих экзонах L Ассоциированные SNP в основном расположены в некодирующих районах (40%), либо в интронах (40%) и… L не сцеплены с несинонимичными заменами, которые могли бы повлиять на функционирование генного продукта L Ассоциированные SNP находят совсем не в тех генах или участках хромосом, в которых их рассчитывают найти
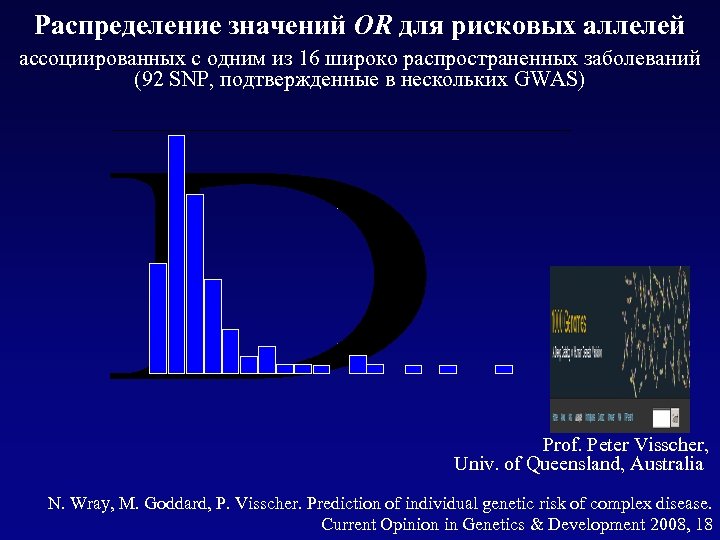
Распределение значений OR для рисковых аллелей ассоциированных с одним из 16 широко распространенных заболеваний (92 SNP, подтвержденные в нескольких GWAS) Prof. Peter Visscher, Univ. of Queensland, Australia N. Wray, M. Goddard, P. Visscher. Prediction of individual genetic risk of complex disease. Current Opinion in Genetics & Development 2008, 18

Сюрпризы GWAS Результаты GWAS устойчиво демонстрируют: L Плохую воспроизводимость В различных GWAS находят непересекающиеся группы ассоциированных SNP L Эффекты отдельных SNP крайне малы: Предрасположенность к заболеваниям - OR = 1. 1 – 1. 3 Количественные признаки – менее 1% объясненной вариансы L Ассоциированные SNP часто расположены в некодирующих районах, либо … L не сцеплены с несинонимичными заменами, которые могли бы повлиять на функционирование генного продукта L Ассоциированные SNP находят совсем не в тех генах или участках хромосом, в которых их рассчитывают найти

The Genome in the Form of GWAS as a “Teacher” of Unexpected Lessons and Connections Неожиданное участие генов Teri Manolio, Director of office for Population Genomics, NHGRI Неожиданные сочетания заболеваний T. Manolio. N Engl J Med 2010; 363

Очевидные недостатки GWAS L Требует огромных выборок (из-за множественности сравнений критический уровень значимости < 10 -7) L Статистические артефакты: сомнения всегда остаются! L GWAS основан на чипах, а не на секвенировании! Он видит только уже известные общие варианты, на которые можно изготовить чипы. GWAS не видит: - неизвестные ранее SNP - редкие мутации (MAF < 0. 01) - тандемные повторы (STR, VNTR) - структурную изменчивость (CNV) L При этом GWAS не регистрирует: - эффекты генов вблизи горячих точек рекомбинации - слабые эффекты (из-за требования pvalue < 10 -7) L Основной вариант GWAS - это «case - control» . Отсюда: - плохая воспроизводимость для различных популяций - крайняя чувствительность к стратификации выборок (этнос, пол, возраст, среда и т. д. )

Возможные объяснения парадокса «missing heritability» (по Peter Visccher, 2011) В порядке роста правдоподобия и не исключая друга q Оценки наследования по родословным ошибочны q Неаддитивность действия генов (эпистаз) q Взаимодействия генотип – среда (Gx. E) q Эпигенетические эффекты q Низкая мощность обнаружения малых эффектов «общих полиморфизмов» q Гетерогенность заболеваний – много различных заболеваний сходны по фенотипу q Общие варианты в проблемных участках генома, напр. , в горячих точках рекомбинации q Мутации, не относящиеся к SNP (напр. , CNV, STR) q Редкие мутации с большим эффектом
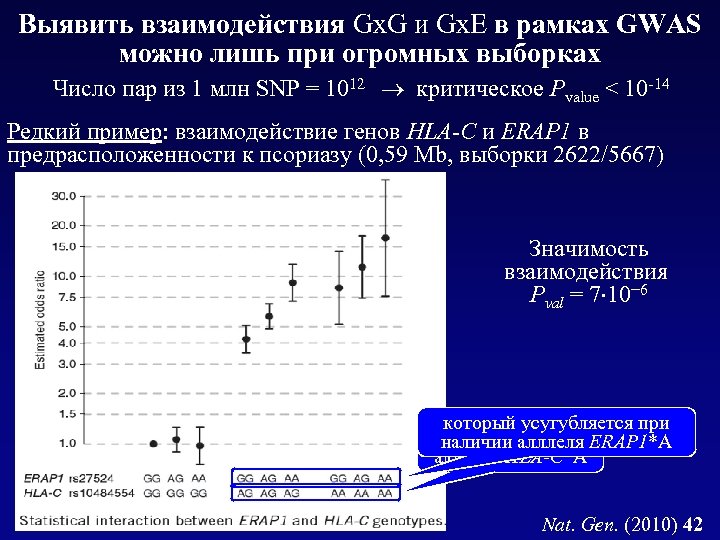
Выявить взаимодействия Gx. G и Gx. E в рамках GWAS можно лишь при огромных выборках Число пар из 1 млн SNP = 1012 критическое Pvalue < 10 -14 Редкий пример: взаимодействие генов HLA-C и ERAP 1 в предрасположенности к псориазу (0, 59 Mb, выборки 2622/5667) Значимость взаимодействия Pval = 7 10− 6 который усугубляется при Сильный эффект наличии алллеля ERAP 1*A алллеля HLA-C*A Nat. Gen. (2010) 42

Взаимодействия Gx. G могут быть причиной более высоких оценок наследуемости по родословным, чем по GWAS Тесно сцепленные несинонимичные SNP, MAF = 0. 5 SNP 1 A > G SNP 2 C > T Предположим, что для гаплотипов AC, GT – признак (в равной степени) AT, GC – признак Тогда эффект аллеля А в среднем = AC + AT = 0, и все «однолокусые» эффекты = 0 Наследуемость по родословным и по GWAS q Анализ родословных: 1 локус – 4 аллеля h 2 = Cor (родители, потомки) = 0, 71 0, 5 0, 25 Признак q GWAS: 2 биаллельных нейтральных локуса h 2 = 0

Возможные объяснения парадокса «missing heritability» (по Peter Visccher, 2011) В порядке роста правдоподобия и не исключая друга q Оценки наследования по родословным ошибочны q Неаддитивность действия генов (эпистаз) q Взаимодействия генотип – среда (Gx. E) q Эпигенетические эффекты q Низкая мощность обнаружения малых эффектов «общих полиморфизмов» q Гетерогенность заболеваний – много различных заболеваний сходны по фенотипу q Общие варианты в проблемных участках генома, напр. , в горячих точках рекомбинации q Мутации, не относящиеся к SNP (напр. , CNV, STR) q Редкие мутации с большим эффектом

Уровень метилирования Часто является эффективным биомаркером, D. Fradin et al. PLo. S ONE, 2012, 2 но его наследуемость сильно зависит от популяции и эффектов Gx. E Fraser et al. Population-specificity of human DNA methylation. Genome Biology, 2012, 13

Возможные объяснения парадокса «missing heritability» (по Peter Visccher, 2011) В порядке роста правдоподобия и не исключая друга q Оценки наследования по родословным ошибочны q Неаддитивность действия генов (эпистаз) q Взаимодействия генотип – среда (Gx. E) q Эпигенетические эффекты q Низкая мощность обнаружения малых эффектов «общих полиморфизмов» q Гетерогенность заболеваний – много различных заболеваний сходны по фенотипу q Общие варианты в проблемных участках генома, напр. , в горячих точках рекомбинации q Мутации, не относящиеся к SNP (напр. , CNV, STR) q Редкие мутации с большим эффектом

Гетерогенность заболеваний: слепота Complex genetic diseases: controversy over the Croesus code A. Wright, N. Hastie. Genome Biology. 2001, 2(8)

Гетерогенность эффектов (разнонаправленная плейотропия) Высокая частота во всех популяциях человека и приматов A. Wright, N. Hastie. Genome Biology. 2001, 2(8)

Возможные объяснения парадокса «missing heritability» (по Peter Visccher, 2011) В порядке роста правдоподобия и не исключая друга q Оценки наследования по родословным ошибочны q Неаддитивность действия генов (эпистаз) q Взаимодействия генотип – среда (Gx. E) q Эпигенетические эффекты q Низкая мощность обнаружения малых эффектов «общих полиморфизмов» q Гетерогенность заболеваний – много различных заболеваний Наиболее вероятное сходны по фенотипу объяснение парадокса «missing heritability» q Общие варианты в проблемных участках генома, напр. , в горячих точках рекомбинации q Мутации, не относящиеся к SNP (напр. , CNV, STR) q Редкие мутации с большим эффектом

Крупные CNV часто ассоциированы с психогенными заболеваниями Редкие CNV OR = 15, но частота в популяции ~ 0. 0002 Доля объясненной заболеваемости ~ 0. 28% T. Manolio et al. Finding the missing heritability of complex diseases «Общие» CNV Частота в популяции ~ 0. 55 Genome differed from the reference OR = 1, 3. Доля объясненной заболеваемости ~ 15% in only 0. 1% of SNPs, but in 1. 2% of CNVs. Nature, 2009, 461

Возможные объяснения парадокса «missing heritability» (по Peter Visccher, 2011) В порядке роста правдоподобия и не исключая друга q Оценки наследования по родословным ошибочны q Неаддитивность действия генов (эпистаз) q Взаимодействия генотип – среда (Gx. E) q Эпигенетические эффекты q Низкая мощность обнаружения малых эффектов «общих полиморфизмов» q Гетерогенность заболеваний – много различных заболеваний сходны по фенотипу q Общие варианты в проблемных участках генома, напр. , в горячих точках рекомбинации q Мутации, не относящиеся к SNP (напр. , CNV, STR) q Редкие мутации с большим эффектом

Проблема «Missing Heritability» возродила дискуссию «Rare vs. Common» q Гипотеза «common disease - rare variants» . Предрасположенность к широко распространенным заболеваниям определяется редкими аллелями (мутациями, MAF < 0. 01) с высокой пенетратностью (Pritchard, 2001; Prixhard & Cox, 2002) q Гипотеза «common disease - common variants» . Предрасположенность к широко распространенным заболеваниям определяется аллелями высоко полиморфных генов (Reich & Lander, 2001) GWAS фактически целиком основан на этой гипотезе 10 лет, 500 000$, 700 GWAS для 300 заболеваний, и вечные дебаты о природе фенотипической изменчивости

Проблема «Missing Heritability» возродила дискуссию «Rare vs. Common» Менделисты (1900 -1940) Биометрики У. Бейтсон К. Пирсон Г. де Фриз R. Fisher Классическая (1960 -1980) гипотеза Балансовая гипотеза A 1 B 1 C 1 D 1 E 1 G 1 F 1 A 2 B 2 C 2 D 2 E 2 G 2 F 2 + + + + m + + Г. Дж. Меллер Ф. Г. Добржанский Rare Jonathan Pritchard, Prof. of Howard Hughes Medical Institute (2000 -…) Common Eric Lander, Director of the Broad Inst. of Massachusetts, Harvard Univ.

Проблема «Missing Heritability» возродила дискуссию «Rare vs. Common» Хорхе Луис Борхес (1899 - 1986) «Быть может, всемирная история – это история различных интонаций при произнесении нескольких метафор»

Аргументы в пользу гипотезы «common disease - rare variants» (по Greg Gibson) Rare and common variants: twenty arguments Nature Rev. Genetics, Feb. 2012, 13 q Вредные мутации в "генах предрасположенности" находятся под действием отбора и не могут стать «common variants» q Ассоциации заболеваний с «общими» вариантами почти никогда не удается объяснить с функциональных позиций q Многие семейные формы заболеваний обусловлены редкими аллелями с высокой пенетрантностью q Редкие CNV часто ассоциированы с психогенными заболеваниями q Массовое секвенирование экзомов показывает, что несинонимичные замены имеют низкую MAF, а «общие» варианты преобладают в межгенных областях
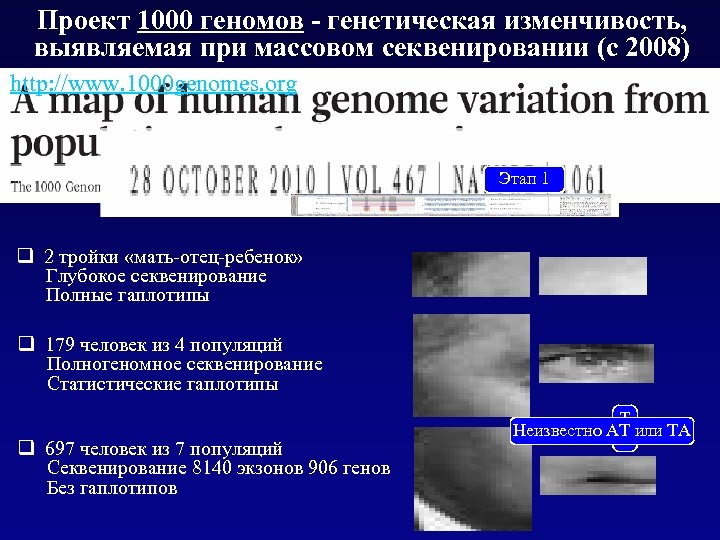
Проект 1000 геномов - генетическая изменчивость, выявляемая при массовом секвенировании (с 2008) http: //www. 1000 genomes. org Этап 1 q 2 тройки «мать-отец-ребенок» Глубокое секвенирование Полные гаплотипы q 179 человек из 4 популяций Полногеномное секвенирование Статистические гаплотипы q 697 человек из 7 популяций Секвенирование 8140 экзонов 906 генов Без гаплотипов Т Неизвестно АТ или ТА А

Проект 1000 геномов - генетическая изменчивость, выявляемая при массовом секвенировании Обнаружено # Из них новых, % SNP 15 млн. 55 Короткие Indel 1 млн. Крупные CNV 20 000 57 61 (делеции) 89 (дупликации) q В среднем каждый человек является носителем 250 -300 аллелей типа «loss-of-function variants» и … q носителем 50 -100 аллелей, ранее зарегистрированных как варианты, вовлеченные в наследственные заболевания (NHGRI) q de novo по 2 тройкам : в половых клетках зарегистрировано 1028 нуклеотидных замен (10 -8 на нуклеотид за поколение) Nature, 2010, 467

Проект 1000 геномов - генетическая изменчивость, выявляемая при массовом секвенировании Доля SNP Среди уникальных SNP преобладают стоп-кодоны Среди «общих» вариантов преобладают SNP в межгенных областях Частота аллеля (MAF) Уникальные SNP Nature, 2010, 467

Проект 1000 геномов - генетическая изменчивость, выявляемая при массовом секвенировании Несинонимичные SNP: Condel score Среди уникальных SNP преобладают ущербные Ущербные Доля SNP Мягкие Consensus Deleteriousness score of missense SNPs Частота аллеля (MAF) Уникальные SNP Среди «общих» SNP преобладают мягкие замены Nature, 2010, 467
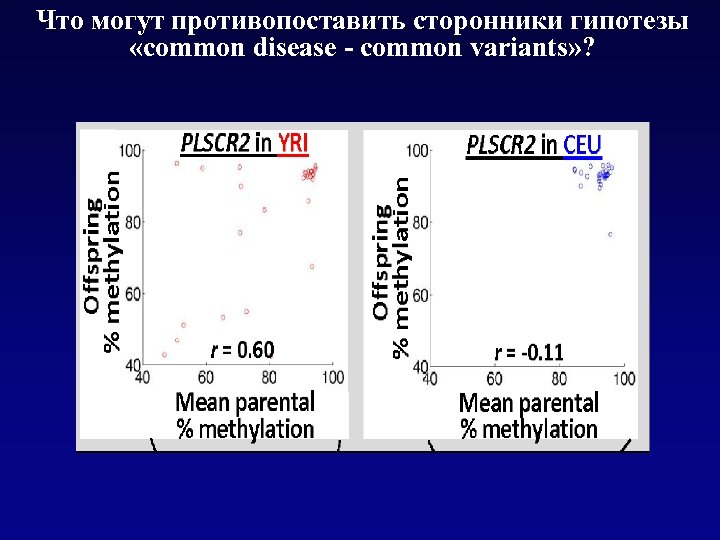
Что могут противопоставить сторонники гипотезы «common disease - common variants» ?

Что могут противопоставить сторонники гипотезы «common disease - common variants» ? q Для ряда заболеваний ассоциации с «общими» аллельными вариантами реально существуют и уверенно воспроизводятся

Для ряда заболеваний ассоциации с «общими» аллельными вариантами реально существуют Заболевание «Общий» полиморфизм Язва 12 -перстной кишки AB 0*В Тромбоз вен F 5*Leiden Болезнь Альцгеймера APOE*Е 4 ВИЧ CCR 5*Δ 32 Колоректальный рак Диабет I типа APC*3920 A PTPN 22 Диабет II типа PPARG, KCJN 11 © Gibson & Muse, A Primer of Genome Science, 2002

Что могут противопоставить сторонники гипотезы «common disease - common variants» ? q Для ряда заболеваний ассоциации с «общими» аллельными вариантами реально существуют и уверенно воспроизводятся q Для модельных объектов (мухи, мыши) проблемы с «missing heritability» не возникают q Распределение частот рисковых аллелей не соответствует гипотезе «common disease - rare variants»

Распределение частот рисковых аллелей не соответствует гипотезе «common disease - rare variants» Распределение частот рисковых аллелей для наиболее значимых результатов GWAS (genome. gov ) Теоретическое распределение частот рисковых аллелей для ассоциаций, индуцированных сцеплением с редкими вариантами 417 SNP – 17 заболеваний Мода ~ 0. 4! N. Wray, S. Purcell, P. Visscher. Synthetic Associations Created by Rare Variants Do Not Explain Most GWAS Results. PLo. S Biology, 2011, 9, 1

Что могут противопоставить сторонники гипотезы «common disease - common variants» ? q Для ряда заболеваний ассоциации с «общими» аллельными вариантами реально существуют и уверенно воспроизводятся q Для модельных объектов (мухи, мыши) проблемы с «missing heritability» не возникают Количественные признаки, которые «в 2 шагах от заболевания» q Распределение частот рисковых аллелей не соответствует (напр. , давление при диабете) гипотезе «common disease - rare variants» q Изменчивость «эндофентипов» удается удовлетворительно объяснить «общими» полиморфизмами q Редкие варианты не объясняют «эпидемиологические сдвиги» , напр. , рост диабета и заболеваний сердца в Индии и Китае за последние 10 лет q В недавних работах показано, что GWAS (common SNP) в состоянии объяснить 45% изменчивости роста человека ( «главный модельный признак» со времен Гальтона)
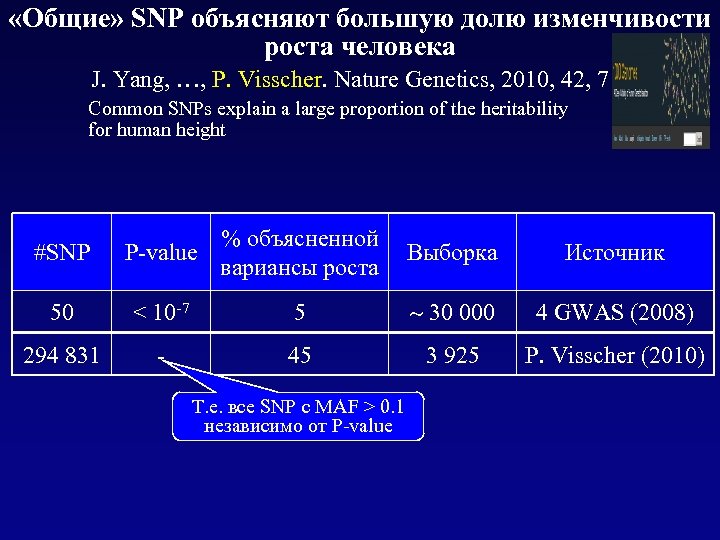
«Общие» SNP объясняют большую долю изменчивости роста человека J. Yang, …, P. Visscher. Nature Genetics, 2010, 42, 7 Common SNPs explain a large proportion of the heritability for human height #SNP % объясненной P-value Выборка вариансы роста Источник 50 < 10 -7 5 30 000 4 GWAS (2008) 294 831 - 45 3 925 P. Visscher (2010) Т. е. все SNP c MAF > 0. 1 независимо от P-value
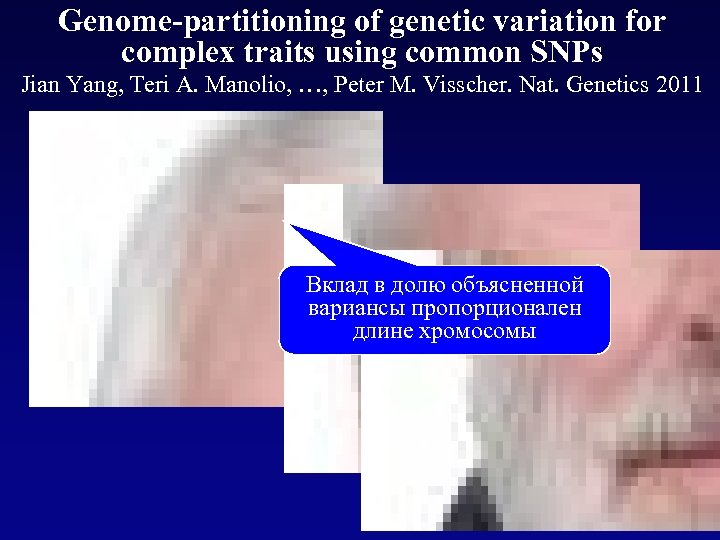
Genome-partitioning of genetic variation for complex traits using common SNPs Jian Yang, Teri A. Manolio, …, Peter M. Visscher. Nat. Genetics 2011 Вклад в долю объясненной вариансы пропорционален длине хромосомы
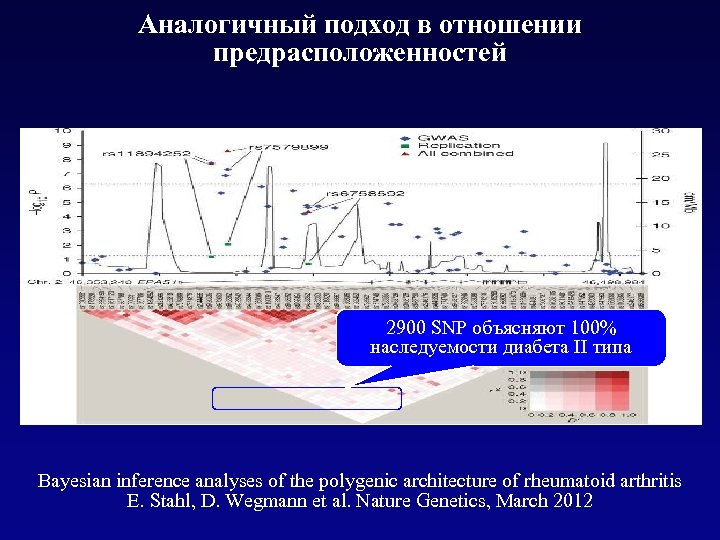
Аналогичный подход в отношении предрасположенностей 2900 SNP объясняют 100% наследуемости диабета II типа Bayesian inference analyses of the polygenic architecture of rheumatoid arthritis E. Stahl, D. Wegmann et al. Nature Genetics, March 2012

Как преодолеть кризис, связанный с «missing heritability» (из частных бесед): Евгений Рогаев, Univ. of Massachusetts Medical School, ИОГен РАН Идея GWAS порочна по своей сути. Только «глубокое секвенирование» позволит разобраться с генетикой предрасположенности. Юрий Дуброва, Dep. of Genetics Univ. of Leicester На практике биоинформатика обходится дороже чипов. И остается при этом самым слабым местом ассоциативных исследований. Никита Хромов-Борисов, СПб. ГМУ Прежде чем заниматься геномикой, следовало бы поначалу внедрить в клиническую практику регистрацию родословных. Это и дешевле и эффективнее.

Джон Крейг Вентер: ветеран войн - генетик - бизнесмен - организатор науки Der Spiegel Interview with Craig Venter http: //www. spiegel. de/international/world/0, 1518, 709174 -2, 00. html q SPIEGEL: “Why is it taking so long for the results of genome research to be applied in medicine? ” q VENTER: “Because we have, in truth, learned nothing from the genome other than probabilities. How does a 1 or 3 percent increased risk for something translate into the clinic? It is useless information. ” 07/29/2010

Всем спасибо, выводов не будет – «still missing» ! Но напоследок несколько советов: q Никогда не упускайте возможность полистать Nature q Почаще заглядывайте в «Hap. Map» , «NHGRI» и « 1000 Genomes» . Даже если Вы непосредственно не занимаетесь генотипированием. Все новости – там! Каталог GWAS q Попробуйте определить свой психотип: сделайте свой выбор между «rare» и «common» . Даже не пытайтесь занять промежуточную позицию! Слайды доступны всем!
fbe6ea83f64949cef876cf3098fbdfbf.ppt