4766f9b4f5885922d80715bc1976b3e0.ppt
- Количество слайдов: 52

ШТРИХ-КОДИРОВАНИЕ ВИДОВ НА ОСНОВЕ ДНК: ФИЛОГЕНЕТИКА, ТАКСОНОМИЯ, ПРОБЛЕМЫ И ОГРАНИЧЕНИЯ Картавцев Ю. Ф. Институт биологии моря имени А. В. Жирмунского Дальневосточного отделения Российской академии наук, Владивосток 690041; e-mail: kartavtsev_yu 48@hotmail. com

Первый наставник: академик РАН, проф. Ю. П. Алтухов 2002 г. (директор Института общей генетики им. Н. И. Вавилова)

МЕЖДУНАРОДНЫЙ ПРОЕКТ ШТРИХКОДИРОВАНИЯ ВИДОВ ЖИВЫХ ОРГАНИЗМОВ (1) • «Штрих-код жизни» (Barcoding of Life – http: //barcoding. si. edu/ ) – это международный проект, организованный в 2004 году и призванный объединить усилия генетиков, зоологов, ботаников, специалистов по информатике и электронике для того, чтобы каждому виду обитающих на Земле животных и растений дать специфическую метку. По этой метке любой организм, даже поврежденный, сохранившийся в виде какого-то фрагмента или находящийся на ранней стадии развития (не похожей на взрослое его состояние), мог бы быть точно определен. • Идея такого «штрих-кода» принадлежит канадскому биологу Полю Эберу. Он предложил в 2003 году в качестве видоспецифичной метки использовать последовательность нуклеотидов в ДНК гена, который есть у всех животных и растений. Это ген цитохромоксидазы 1 (субъединица 1 – Со-1, Cox 1 или COI; соответствующий белок принимает участие в процессах дыхания). Данный ген состоит примерно из 650 нуклеотидов.

МЕЖДУНАРОДНЫЙ ПРОЕКТ ШТРИХКОДИРОВАНИЯ ВИДОВ ЖИВЫХ ОРГАНИЗМОВ (2) • Идея штрих-кодирования видов широко поддержана в мире. Образовался международный Консорциум проекта “Штрих-код жизни”, в который к 2005 г. вступило 69 организаций из 31 страны (университеты, естественно-научные музеи, зоопарки, ботанические сады, агентства, занимающиеся природоохранной деятельностью, и т. д. ), а в 2006 г. их численность составила 133. Конечная цель проекта - идентифицировать все известные и пока неизвестные виды и дать возможность легко определять принадлежность того или иного организма к конкретному виду. Сейчас биологами описано около 1 млн. 700 видов животных и растений (не считая микробов). Предполагается, что всего существует не менее 10 млн. видов, то есть большинство их еще не выявлено. • Сформированы два подпроекта: Fish-BOL, по которому будут даны «штрих-коды» 29 000 видов морских и пресноводных рыб и «Птицы Северной Америки» (10 000 видов должны быть кодированы к 2010 году). К 2006 г. кодированы - 2453 видов рыб. В текущем году пройдет уже вторая международная конференция по описанной проблематике в рамках глобальной инициативы (Тайбэй, сентябрь 2007) и состоялось множество региональных международных симпозиумов.

МЕЖДУНАРОДНЫЙ ПРОЕКТ ШТРИХКОДИРОВАНИЯ ВИДОВ ЖИВЫХ ОРГАНИЗМОВ (3) Сопредседатели: P. Hebert и B. Ward •
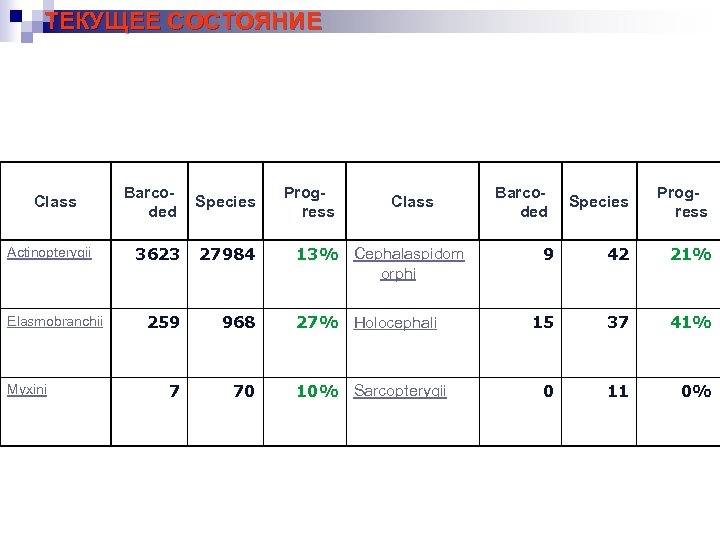
ТЕКУЩЕЕ СОСТОЯНИЕ Class Actinopterygii Elasmobranchii Myxini Barcoded Species 3623 27984 259 968 7 70 Progress Class 13% Cephalaspidom orphi 27% Holocephali 10% Sarcopterygii Barcoded Species Progress 9 42 21% 15 37 41% 0 11 0%
Регистрация и утилиты по штрих-кодированию (Bol. D; www. boldsystems. org) (1)

Регистрация и утилиты по штрих-кодированию (Bol. D; www. boldsystems. org) (2)
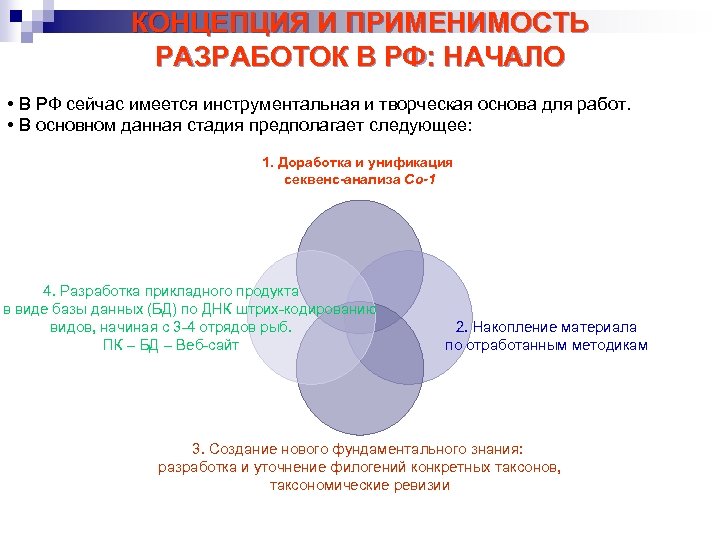
КОНЦЕПЦИЯ И ПРИМЕНИМОСТЬ РАЗРАБОТОК В РФ: НАЧАЛО • В РФ сейчас имеется инструментальная и творческая основа для работ. • В основном данная стадия предполагает следующее: 1. Доработка и унификация секвенс-анализа Co-1 4. Разработка прикладного продукта в виде базы данных (БД) по ДНК штрих-кодированию видов, начиная с 3 -4 отрядов рыб. ПК – БД – Веб-сайт 2. Накопление материала по отработанным методикам 3. Создание нового фундаментального знания: разработка и уточнение филогений конкретных таксонов, таксономические ревизии

КОНЦЕПЦИЯ ШТРИХ-КОДИРОВАНИЯ ВИДОВ РЫБ РФ • Создание базы данных будет сопровождаться разработкой специальной программы по переустройству музейных коллекций, начиная с коллекции музея Института биологии моря ДВО РАН (ИБМ), на современной основе. • Эта работа предполагает: 1) сопровождение всех типовых (паратиповых) образцов цветными цифровыми фотографиями, 2) ваучерным представлением типовых экземпляров рыб (подготовка технической документации и ее компьютерное обеспечение в соответствии с мировыми стандартами; Leviton et al. 1985; http: //www. asih. org/codons. pdf), 3) ведением специальных крио-коллекций спиртовых образцов тканей и 4) инкорпорированием данных в мировые базы данных, начиная с Fish-BOL (http: //www. fishbol ), Gen. Bank, NCBI (http: //www. ncbi. nlm. nih. gov/ ), Bol. D (www. boldsystems. org ) и Fish. Base (http: //fishbase. com/search. php ).
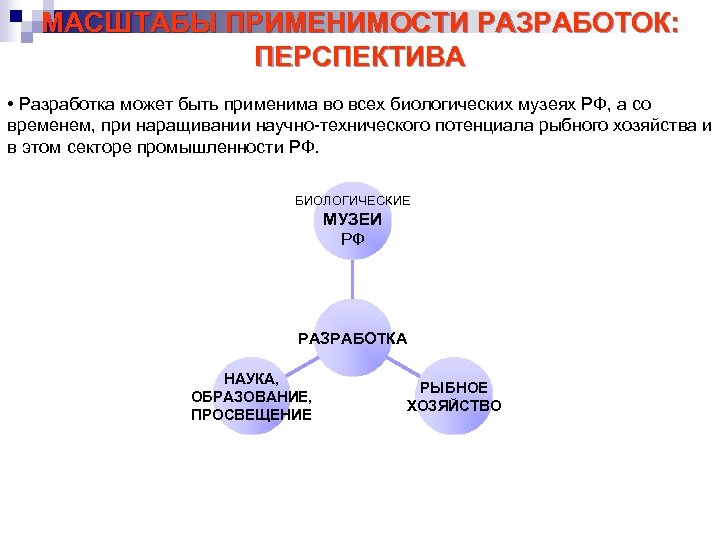
МАСШТАБЫ ПРИМЕНИМОСТИ РАЗРАБОТОК: ПЕРСПЕКТИВА • Разработка может быть применима во всех биологических музеях РФ, а со временем, при наращивании научно-технического потенциала рыбного хозяйства и в этом секторе промышленности РФ. БИОЛОГИЧЕСКИЕ МУЗЕИ РФ РАЗРАБОТКА НАУКА, ОБРАЗОВАНИЕ, ПРОСВЕЩЕНИЕ РЫБНОЕ ХОЗЯЙСТВО

МАСШТАБЫ ПРИМЕНИМОСТИ РАЗРАБОТОК: ПЕРСПЕКТИВА Потенциальный рынок использования данной услуги • В РФ функционирует 1071 ВУЗ (данные по 2004 г. ), во многих из которых имеются биологические или экологические специальности и, соответственно, потенциальная потребность в данной услуге. Кроме того, биологические музеи и экспозиции имеются в профильных институтах РАН, Госкомрыболовства и Министерства природных ресурсов. Биологические экспозиции и музеи имеют также большинство природных заповедников и заказников РФ, а также многие общеобразовательные школы. Число заинтересованных организаций в РФ может достигать много более 2000. Количество пользователей через систему Интернета в перспективе может измеряться десятками тысяч посещений соответствующих сайтов при их создании и развитии. Сложности • Прежде всего они, видятся в материальном обеспечении: для покупки необходимого экспериментального оборудования, химреактивов, компьютеров и программного обеспечения. • Творческие проблемы: Hebert et al. , 2004; Ward et al. , 2005; Картавцев, Ли, 2006; Ratnasingham, Hebert, 2007. Критика подхода: Moritz, Cicero, 2004; Funk, Omland, 2004.

ЧИСЛО ВИДОВ РЫБ РФ • В недавнем каталоге бесчерепных и рыб пресных и солоноватых вод России перечислены 557 видов и других таксонов (Богутская, Насека, 2004). Истинно пресноводных – 367 видов (Froesy, Pauly, 2005; World Wide Web Fishbase). • Приблизительное число морских видов – 568 (Froesy, Pauly, 2005; Fishbase, 2005). Т. е. , всего число видов рыб в России составляет 1125. Однако по новой сводке Соколовского и соавторов (2007) только число морских рыб Японского моря составляет 366 видов, а всего в РФ и сопредельных водах имеется 1153 видов рыбообразных и рыб из 170 семейств, 39 отрядов и 5 классов (Евсеенко, 2005). Таким образом, сегодняшняя оценка 1200 – 1400 видов, и это число после реализации программы Fish-BOL, видимо, сильно увеличиться. • Главное однако не это, а совершенно новый уровень описания имеющегося разнообразия, с точным документированием и сопровождением всех образцов цветными фото, ДНК штрих-кодом, а также бесплатным доступом к мировым базам данных.

РАЙОНЫ РАБОТ: НАЧАЛО n n Зона 1: Баренцево море, Белое море, Балтийское море, и Черное море. Главные участники: Институт общей генетики (ИОГен), Институт биологии гена (ИБГ), Зоологический институт (ЗИН), Всероссийский институт рыбного хозяйства и океанографии (ВНИРО), Полярный институт рыбного хозяйства и океанографии (ПИНРО), Московский государственный университет (МГУ) и др. Зона 2: Японское море, Охотское море, Берингово море. Главные участники: Институт биологии моря (ИБМ), Биолого-почвенный институт (БПИ), Институт биологических проблем севера (ИБПС), Дальневосточный государственный университет (ДВГУ), Тихоокеанский институт рыбного хозяйства и океанографии (ТИНРО-центр) и др.
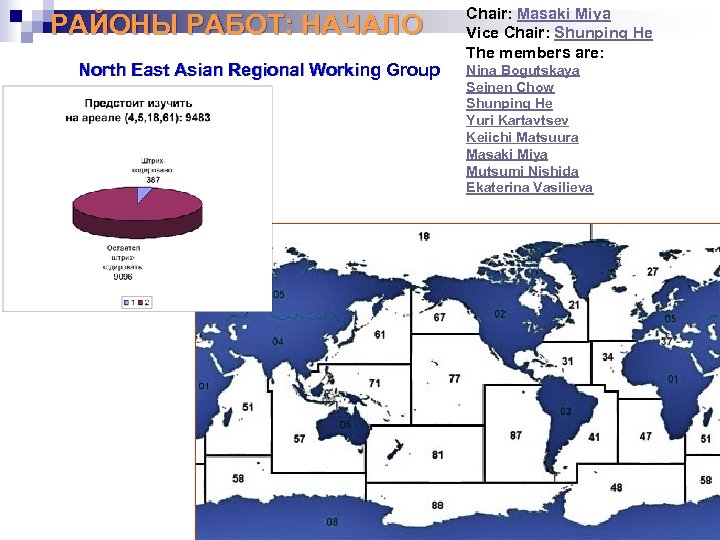
РАЙОНЫ РАБОТ: НАЧАЛО North East Asian Regional Working Group Chair: Masaki Miya Vice Chair: Shunping He The members are: Nina Bogutskaya Seinen Chow Shunping He Yuri Kartavtsev Keiichi Matsuura Masaki Miya Mutsumi Nishida Ekaterina Vasilieva

ИСТОЧНИКИ ФИНАНСИРОВАНИЯ Внутренние • Исследовательские гранты (РФФИ, ДВО и др. отделения РАН). • Программа РАН (с 2007 г. ? ). • Программа ДВО РАН (с 2007 г. ? ). • Программа Минобрнауки (? ). • Лоты Роснауки (? ). Внешние • Fish-BOL (поиск источников для исследовательских коллективов – FAO, World Bank etc. ).
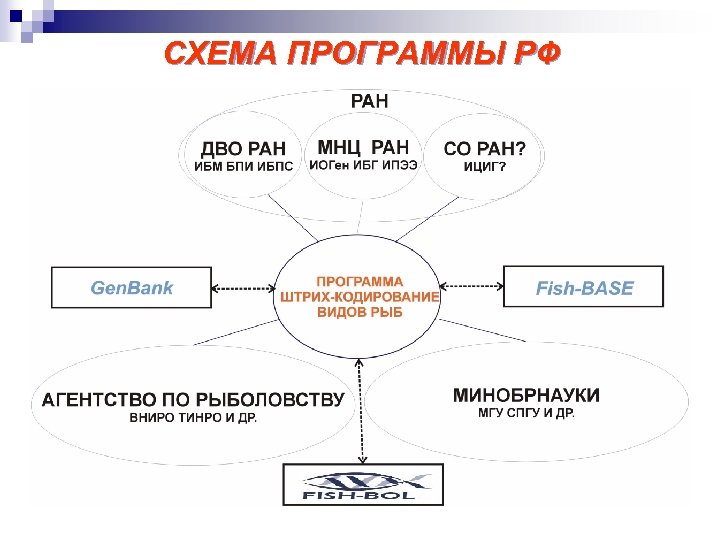
СХЕМА ПРОГРАММЫ РФ

ФИЛОГЕНЕТИКА: ПРОБЛЕМЫ, ОГРАНИЧЕНИЯ
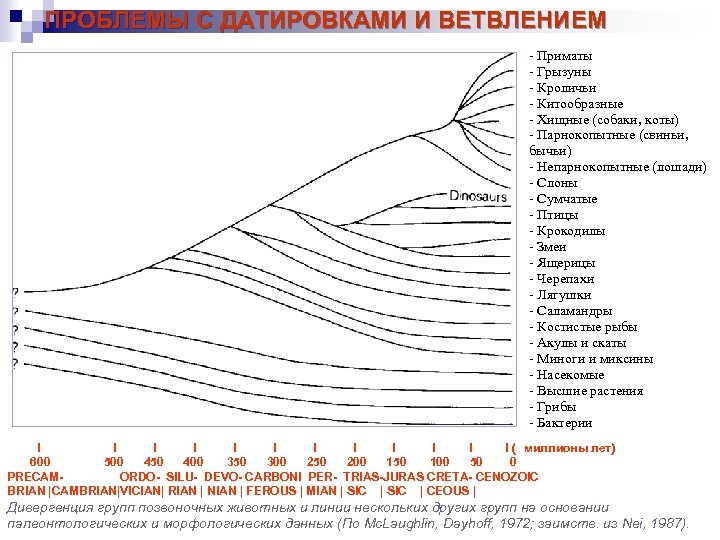
ПРОБЛЕМЫ С ДАТИРОВКАМИ И ВЕТВЛЕНИЕМ - Приматы - Грызуны - Кроличьи - Китообразные - Хищные (собаки, коты) - Парнокопытные (свиньи, бычьи) - Непарнокопытные (лошади) - Слоны - Сумчатые - Птицы - Крокодилы - Змеи - Ящерицы - Черепахи - Лягушки - Саламандры - Костистые рыбы - Акулы и скаты - Миноги и миксины - Насекомые - Высшие растения - Грибы - Бактерии l I I I l ( миллионы лет) 600 500 450 400 350 300 250 200 150 100 50 0 PRECAM- ORDO- SILU- DEVO- CARBONI PER- TRIAS-JURAS CRETA- CENOZOIC BRIAN |CAMBRIAN|VICIAN| RIAN | NIAN | FEROUS | MIAN | SIC | CEOUS | Дивергенция групп позвоночных животных и линии нескольких других групп на основании палеонтологических и морфологических данных (По Mc. Laughlin, Dayhoff, 1972; заимств. из Nei, 1987).

ВВЕДЕНИЕ n ДНК митохондрий (мт. ДНК) – это кольцевая молекула, длиной около 16 -18000 пн (пар нуклеотидов). Как показывают литературные данные, мт. ДНК всех рыб имеет сходную организацию (Lee et al. , 2001; Kim et al. , 2004; Kim et al. , 2005; Nagase et al. , 2005; Nohara et al. , 2005) и мало, чем отличается и у других позвоночных животных, включая человека (Anderson et al. , 1981; Bibb et al. , 1981; Wallace, 1992; Kogelnik et al. , 2005). Полный состав митоходриального генома (митогенома) включает: контрольный регион (CR или D петля), где сосредоточены сайт начала репликации и промоторы, большая (16 S) и малая (12 S) субъединицы р. РНК, 22 т. РНК и 13 полипептидных генов. n Филогенетические исследования обычно используют последовательности единичных генов, в том числе и генов ядерной ДНК, хотя в последние годы все чаше используют для этих целей и полный митогеном. Наиболее популярны в филогенетике последовательности генов цитохрома b (Cyt-b) и цитохром оксидазы 1 (Cо-1), которые используются для сравнения таксонов на уровне вид – семейство (Johns, Avise, 1998; Hebert et al. , 2004; Картавцев, Ли, 2006). Много последовательностей, несущих филогенетический сигнал, получено для разных групп также по гену 16 S р. РНК. n Последовательности отдельных генов могут давать различный филогенетический сигнал из-за различных темпов замен и конкретной эволюционной и/или демографической судьбы таксона (дифференцированная сортировка филетических линий). Это относится и к разным участкам одного и того же гена. Кроме того, при сопоставлении многочисленных таксонов, особенно высокого ранга, возникают проблемы с эффектами гомоплазии и недостаточной информационной емкостью наборов последовательностей для филогенетических целей (Hilish et al. , 1996, Miya et al. , 2001). Тем не менее, для идентификации видов, за редкими исключениями, достаточно сравнения даже относительно коротких последовательностей, например гена Со-1, 654 пн.

Применимость различных молекул в филогенетике и таксономии Вид Род Семейство Отряд Класс Спейсеры [ITS-1, 2] мт-ДНК я. ДНК, р. ДНК Наиболее значимые статистически результаты Достаточно значимые статистически результаты Тип

Некоторые из объектов A B Рис. 1. Внешний вид палтусовидной камбалы, Hypoglossus elasodon (A) и темной камбалы, Pseudopleuronectes obcurus (В).
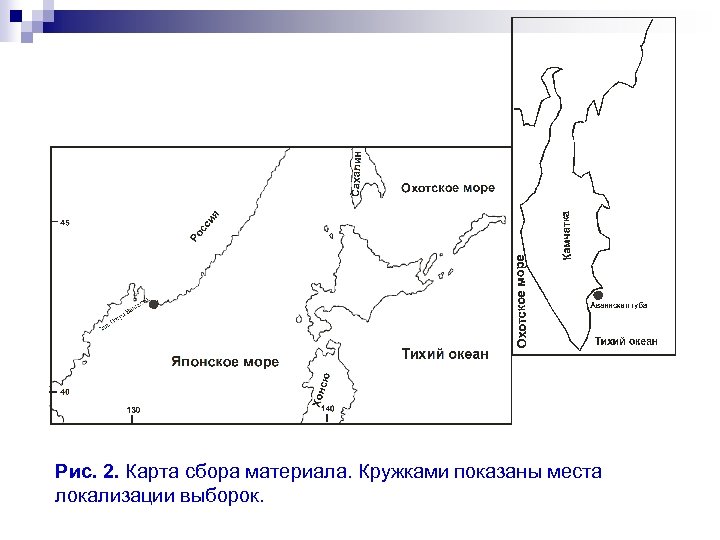
Рис. 2. Карта сбора материала. Кружками показаны места локализации выборок.
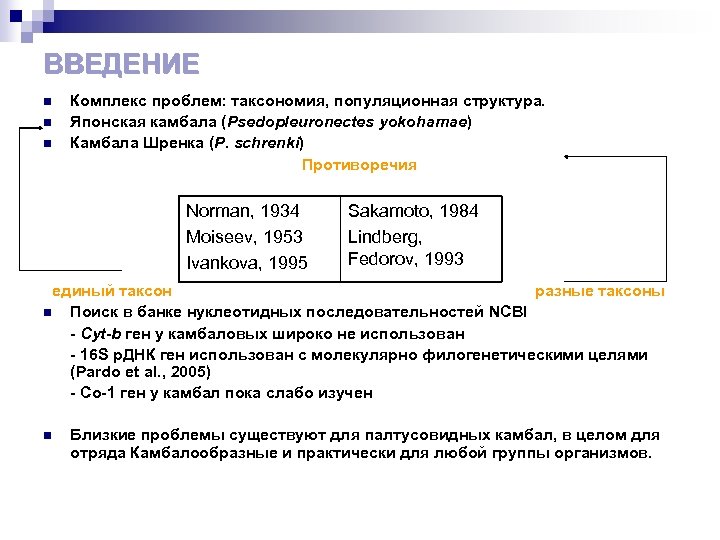
ВВЕДЕНИЕ n n n Комплекс проблем: таксономия, популяционная структура. Японская камбала (Psedopleuronectes yokohamae) Камбала Шренка (P. schrenki) Противоречия Norman, 1934 Moiseev, 1953 Ivankova, 1995 Sakamoto, 1984 Lindberg, Fedorov, 1993 единый таксон разные таксоны n Поиск в банке нуклеотидных последовательностей NCBI - Cyt-b ген у камбаловых широко не использован - 16 S р. ДНК ген использован с молекулярно филогенетическими целями (Pardo et al. , 2005) - Co-1 ген у камбал пока слабо изучен n Близкие проблемы существуют для палтусовидных камбал, в целом для отряда Камбалообразные и практически для любой группы организмов.

Основные вопросы n n Оценить нуклеотидное разнообразие у 6 видов камбал и выяснить согласуемость этих данных с результатами предыдущих исследований для Pseudopleuronectes yokohamae – P. schrenki и Hippoglossoides elassodon – H. robustus. Подтвердить или опровергнуть монофилетичность семейства Pleuronectidae.
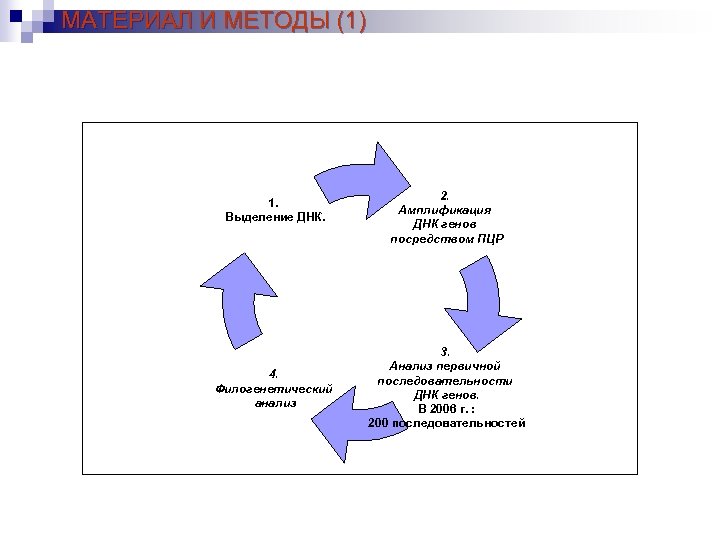
МАТЕРИАЛ И МЕТОДЫ (1) 1. Выделение ДНК. 4. Филогенетический анализ 2. Амплификация ДНК генов посредством ПЦР 3. Анализ первичной последовательности ДНК генов. В 2006 г. : 200 последовательностей

МАТЕРИАЛ И МЕТОДЫ (2) n n Амплификация мт. ДНК посредством полимеразной цепной реакции (ПЦР) Первоначально из скелетных мышц выделили тотальную ДНК с использованием обычной хлороформфенольной методики и осаждением ДНК спиртом. Для получения секвенс данных по гену Cyt-b для 6 видов камбаловых рыб сначала с помощью длинной ПЦР и праймеров L-12321 -Leu (5’-GGT CTT AGG AAC CAA AAA CTC TTG GTG CAA-3’) и S-LA-16 S-H (5’-TGC ACC ATT AGG ATG TCC TGA TCC AAC ATC-3’) получили продукты размером примерно 8 тпн. Далее использовали набор специфических праймеров, подобранных для Cyt-b камбаловых рыб: cytb-F, 5'-ATG GCC AAC CTC CGT AAA TCC CAC CCC CTT C-3' и cytb-R, 5'-CTG GGG CTC TGG ACG CTG AGC TAG TGC-3'. n Реакционная смесь (50 μl) для ПЦР включала: 5 μl 10 x ПЦР буфера (Ta. Ka. Ra, Japan), 5 μl L-праймера, 5 μl H-праймера, 8 μl смеси d. NTP, 1 μl затравки, 0. 5 μl Taq полимеразы (5 units/ μl ) и 30. 5 μl дистиллированной воды. ПЦР реакции проводили в ходе 35 -40 циклов: при режимах 98 o. C 25”, 55 -60 o. C - 30 -90” и 72 o. C - 60 -120” на цикл. Продукты ПЦР подвергали электрофорезу в 1. 0% агарозном геле. Окрашивание, осуществляли бромидом этидия и просматривали в УФ-свете. Было получено несколько ПЦР-продуктов и последовательно были клонированы на векторе p. CR 2. 1 (Invitrogen). Секвенирование реализовано с использованием коммерческих праймеров T 7 или M 13. n Анализ первичной последовательности ДНК n Меченные фрагменты ДНК анализировали на секвенаторе модели 373 S (Applied Biosystems Inc. ). n Последовательности видов камбал по гену Cyt-b легко идентифицировали по аналогии с другими известными последовательностями. n Близкие процедуры использованы при амплификации и севенировании гена Со-1 (детали – в докладе Шариной). n Филогенетический анализ n Последовательности ДНК были выровнены с помощью программы Clustal. W (Thompsn et al. , 1994), интегрированной в пакет MEGA-3 (Kumar et al. , 1993). Анализировали кодирующие (все позиции) и некодирующие участки последовательностей. Оптимальную модель для нуклеотидных замен для анализируемого набора последовательностей подбирали с использованием специального программного средства Modeltest 3. 7 (Posada, Grandall, 1998). Реконструкция древ основывалась на нескольких подходах, включая методы максимальной парсимонии (MP), максимального правдоподобия (ML) и байесовский (BA), с бутстреп-поддержкой во всех случаях, где это необходимо (Nei, Kumar, 2000; Felsenstein, 2004). Наилучшими для всего набора данных камбаловых рыб (20 последовательностей) оказались две модели замен: TVN+I+G и Tr. N+I+G. Вторая модель, Tr. N+I+G, признана наилучшей согласно информационного критерия Акаки (Akaike), тогда как вторая, TVN+I+G, оценена как наилучшая согласно теста максимального правдоподобия n

РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ: ДАННЫЕ ПО КАМБАЛООБРАЗНЫМ

Фрагмент выровненных последовательностей нуклеотидов гена Cyt-b 17 видов камбалообразных и других рыб
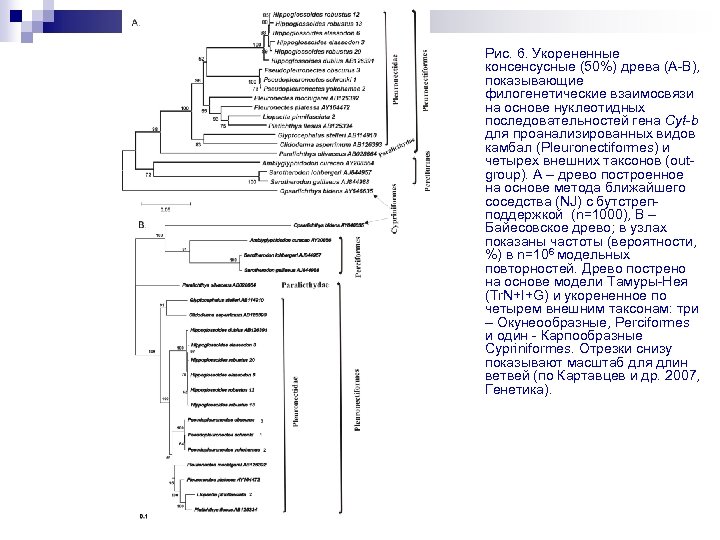
Рис. 6. Укорененные консенсусные (50%) древа (A-B), показывающие филогенетические взаимосвязи на основе нуклеотидных последовательностей гена Cyt-b для проанализированных видов камбал (Pleuronectiformes) и четырех внешних таксонов (outgroup). A – древо построенное на основе метода ближайшего соседства (NJ) с бутстрепподдержкой (n=1000), B – Байесовское древо; в узлах показаны частоты (вероятности, %) в n=106 модельных повторностей. Древо пострено на основе модели Тамуры-Нея (Tr. N+I+G) и укорененное по четырем внешним таксонам: три – Окунеообразные, Perciformes и один - Карпообразные Cypriniformes. Отрезки снизу показывают масштаб для длин ветвей (по Картавцев и др. 2007, Генетика).

Рис. 8. Укорененное Байесовское консенсусное (50%) древо, показывающее филогенетические взаимосвязи на основе нуклеотидных последовательностей гена Co-1 для 13 проанализированных видов камбал (Pleuronectiformes) и двух внешних таксонов (outgroup). В узлах показаны частоты (вероятности) в n=106 модельных повторностей. Древо построено на основе модели Тамуры-Нея (Tr. N+I+G) и укорененное по двум внешним таксонам: Окунеообразные, Perciformes. Отрезки снизу показывают масштаб для длин ветвей (наши не опубликованные данные).

Рис. 8. Укорененное консенсусное (50%) древо, показывающее филогенетические взаимосвязи на основе нуклеотидных последовательностей гена Cyt-b для 34 проанализированных видов камбал (Pleuronectiformes) и трех внешних таксонов (outgroup). Байесовское древо; в узлах показаны частоты (вероятности, %) в n=106 модельных повторностей. Древо пострено на основе модели Тамуры-Нея (Tr. N+I+G) и укорененное по трем внешним таксонам: Окунеообразные, Perciformes. Отрезки снизу показывают масштаб для длин ветвей (по Kartavtsev et al. , 2007, Marine Biol. ).
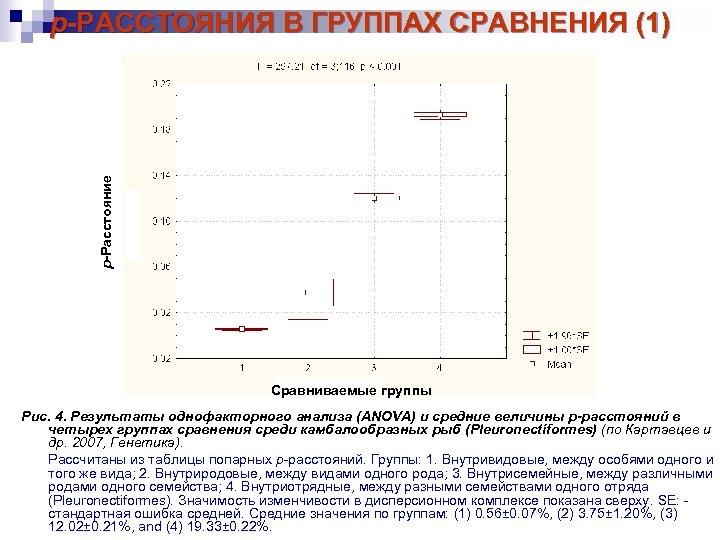
р-Расстояние p-РАССТОЯНИЯ В ГРУППАХ СРАВНЕНИЯ (1) Сравниваемые группы Рис. 4. Результаты однофакторного анализа (ANOVA) и средние величины p-расстояний в четырех группах сравнения среди камбалообразных рыб (Pleuronectiformes) (по Картавцев и др. 2007, Генетика). Рассчитаны из таблицы попарных p-расстояний. Группы: 1. Внутривидовые, между особями одного и того же вида; 2. Внутриродовые, между видами одного рода; 3. Внутрисемейные, между различными родами одного семейства; 4. Внутриотрядные, между разными семействами одного отряда (Pleuronectiformes). Значимость изменчивости в дисперсионном комплексе показана сверху. SE: стандартная ошибка средней. Средние значения по группам: (1) 0. 56± 0. 07%, (2) 3. 75± 1. 20%, (3) 12. 02± 0. 21%, and (4) 19. 33± 0. 22%.
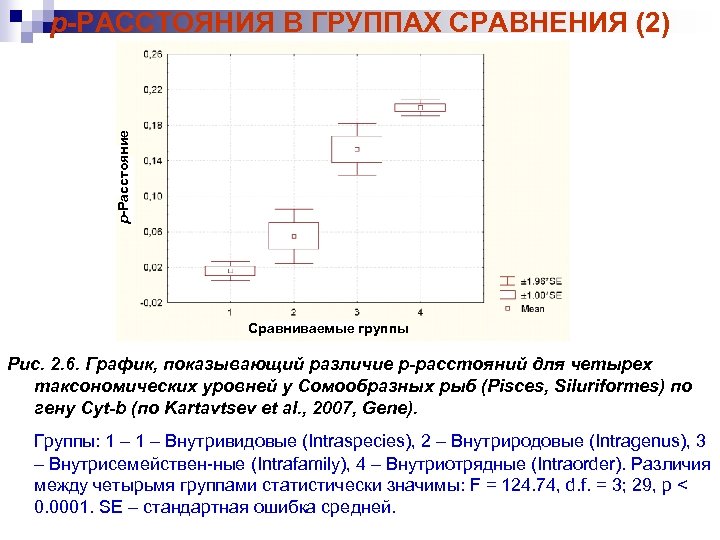
р-Расстояние p-РАССТОЯНИЯ В ГРУППАХ СРАВНЕНИЯ (2) Сравниваемые группы Рис. 2. 6. График, показывающий различие p-расстояний для четырех таксономических уровней у Сомообразных рыб (Pisces, Siluriformes) по гену Cyt-b (по Kartavtsev et al. , 2007, Gene). Группы: 1 – Внутривидовые (Intraspecies), 2 – Внутриродовые (Intragenus), 3 – Внутрисемействен-ные (Intrafamily), 4 – Внутриотрядные (Intraorder). Различия между четырьмя группами статистически значимы: F = 124. 74, d. f. = 3; 29, p < 0. 0001. SE – стандартная ошибка средней.
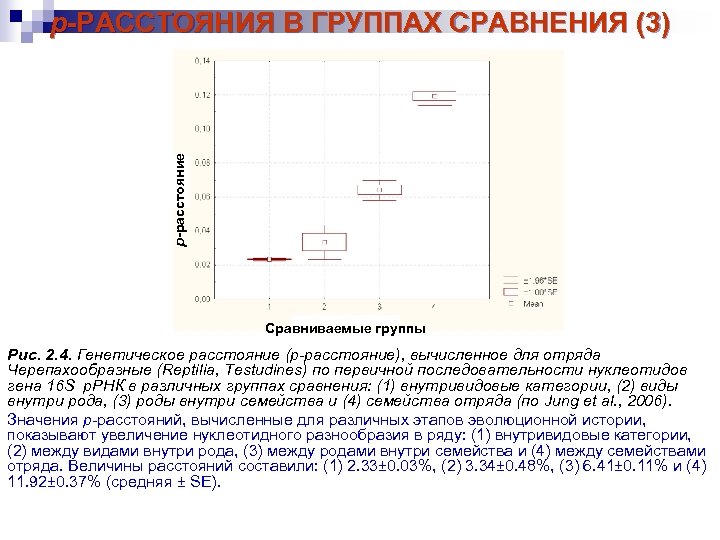
р-расстояние p-РАССТОЯНИЯ В ГРУППАХ СРАВНЕНИЯ (3) Сравниваемые группы Рис. 2. 4. Генетическое расстояние (p-расстояние), вычисленное для отряда Черепахообразные (Reptilia, Testudines) по первичной последовательности нуклеотидов гена 16 S р. РНК в различных группах сравнения: (1) внутривидовые категории, (2) виды внутри рода, (3) роды внутри семейства и (4) семейства отряда (по Jung et al. , 2006). Значения р-расстояний, вычисленные для различных этапов эволюционной истории, показывают увеличение нуклеотидного разнообразия в ряду: (1) внутривидовые категории, (2) между видами внутри рода, (3) между родами внутри семейства и (4) между семействами отряда. Величины расстояний составили: (1) 2. 33± 0. 03%, (2) 3. 34± 0. 48%, (3) 6. 41± 0. 11% и (4) 11. 92± 0. 37% (средняя ± SE).
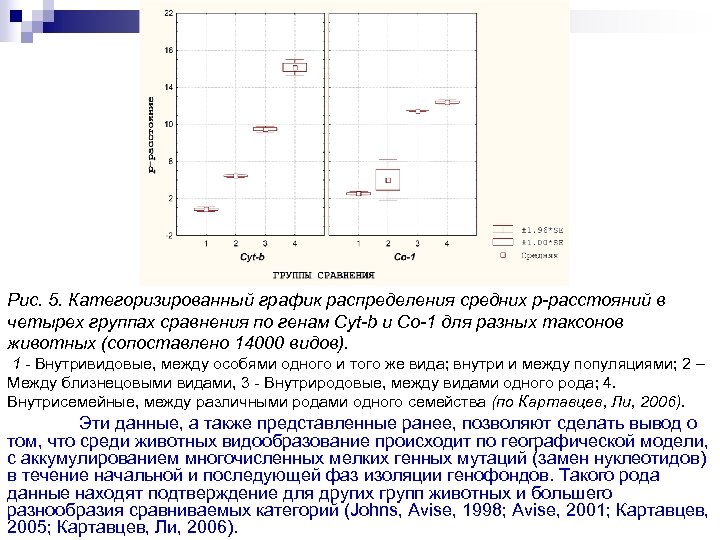
Рис. 5. Категоризированный график распределения средних р-расстояний в четырех группах сравнения по генам Cyt-b и Co-1 для разных таксонов животных (сопоставлено 14000 видов). 1 - Внутривидовые, между особями одного и того же вида; внутри и между популяциями; 2 – Между близнецовыми видами, 3 - Внутриродовые, между видами одного рода; 4. Внутрисемейные, между различными родами одного семейства (по Картавцев, Ли, 2006). Эти данные, а также представленные ранее, позволяют сделать вывод о том, что среди животных видообразование происходит по географической модели, с аккумулированием многочисленных мелких генных мутаций (замен нуклеотидов) в течение начальной и последующей фаз изоляции генофондов. Такого рода данные находят подтверждение для других групп животных и большего разнообразия сравниваемых категорий (Johns, Avise, 1998; Avise, 2001; Картавцев, 2005; Картавцев, Ли, 2006).
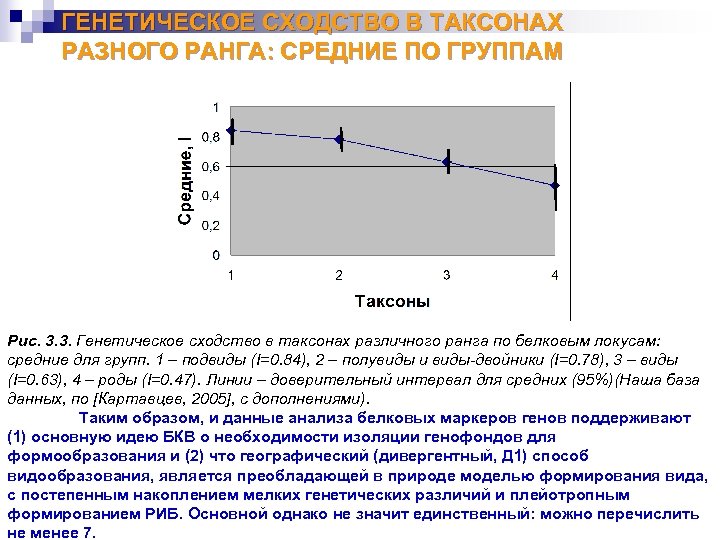
ГЕНЕТИЧЕСКОЕ СХОДСТВО В ТАКСОНАХ РАЗНОГО РАНГА: СРЕДНИЕ ПО ГРУППАМ Рис. 3. 3. Генетическое сходство в таксонах различного ранга по белковым локусам: средние для групп. 1 – подвиды (I=0. 84), 2 – полувиды и виды-двойники (I=0. 78), 3 – виды (I=0. 63), 4 – роды (I=0. 47). Линии – доверительный интервал для средних (95%)(Наша база данных, по [Картавцев, 2005], с дополнениями). Таким образом, и данные анализа белковых маркеров генов поддерживают (1) основную идею БКВ о необходимости изоляции генофондов для формообразования и (2) что географический (дивергентный, Д 1) способ видообразования, является преобладающей в природе моделью формирования вида, с постепенным накоплением мелких генетических различий и плейотропным формированием РИБ. Основной однако не значит единственный: можно перечислить не менее 7.
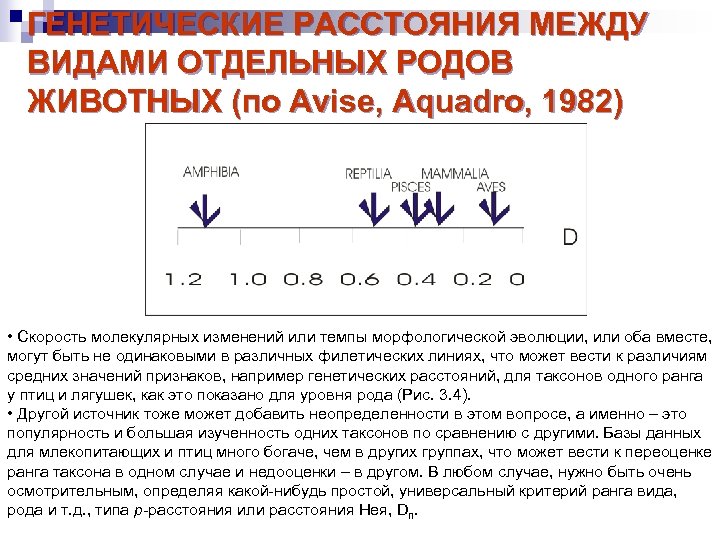
ГЕНЕТИЧЕСКИЕ РАССТОЯНИЯ МЕЖДУ ВИДАМИ ОТДЕЛЬНЫХ РОДОВ ЖИВОТНЫХ (по Avise, Aquadro, 1982) • Скорость молекулярных изменений или темпы морфологической эволюции, или оба вместе, могут быть не одинаковыми в различных филетических линиях, что может вести к различиям средних значений признаков, например генетических расстояний, для таксонов одного ранга у птиц и лягушек, как это показано для уровня рода (Рис. 3. 4). • Другой источник тоже может добавить неопределенности в этом вопросе, а именно – это популярность и большая изученность одних таксонов по сравнению с другими. Базы данных для млекопитающих и птиц много богаче, чем в других группах, что может вести к переоценке ранга таксона в одном случае и недооценки – в другом. В любом случае, нужно быть очень осмотрительным, определяя какой-нибудь простой, универсальный критерий ранга вида, рода и т. д. , типа р-расстояния или расстояния Нея, Dn.

СПОСОБЫ ВИДООБРАЗОВАНИЯ (СВ): ПОПУЛЯЦИОННО-ГЕНЕТИЧЕСКИЙ ВЗГЛЯД • ОТСУТСТВИЕ КОЛИЧЕСТВЕННОЙ ТЕОРИИ ВИДООБРАЗОВАНИЯ (КТВ) В СТЭ нет теории видообразования; нет модели которая обладала бы прогностической способностью. В нашем случае способностью предсказать видообразование, или по меньшей мере свойством отличать различные способы видообразования на базе некоторых количественных параметров или их эмпирических оценок. Попытки, которые сделаны в этом направлении (Avise, Wollenberg, 1997, Templeton, 1998) не удовлетворяют описанным выше критериям. В связи с этим была разработана схема и сформулирован алгоритмический подход (Картавцев и др. , 2002, Картавцев, 2005, Картавцев, Ли, 2006), позволяющие различать способы (модели) видообразования на основе ряда важнейших популяционногенетических параметров, и их оценок, имеющихся в литературе. Этот подход может заложить рамки будущей теории - генетической количественной теории видообразования (КТВ). • ВОЗМОЖНЫЕ ОСНОВЫ ДЛЯ КТВ В качестве основы для формулирования эволюционно-генетической концепции были использованы содержательные описания, сделанные Темплетоном (Templeton, 1981). В результате создана классификационная схема для 7 известных способов видообразования (Рис. 4. 1). Три элемента этой схемы для типов D 1 -D 3, дивергентного видообразования приведем для наглядности (Рис. 4. 2). Этот путь ведет к достаточно простой схеме, которая позволяет: (1) организовать дальнейшее исследование видообразования в различных группах организмов на основе выверенного генетического подхода и (2) вывести аналитические выражения (уравнения) для каждого способа видообразования (Рис. 4. 2).
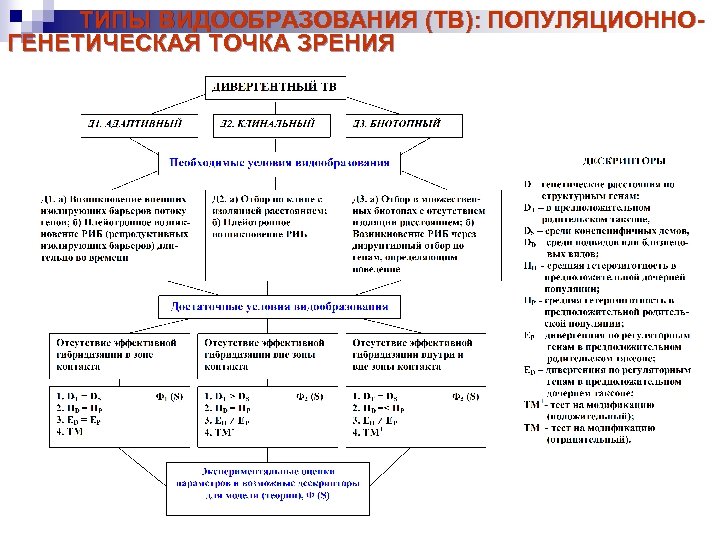
ТИПЫ ВИДООБРАЗОВАНИЯ (ТВ): ПОПУЛЯЦИОННОГЕНЕТИЧЕСКАЯ ТОЧКА ЗРЕНИЯ

ПУБЛИКАЦИИ АВТОРА ПО БЛИЗКОЙ ПРОБЛЕМАТИКЕ • Картавцев Ю. Ф. Молекулярная эволюция и популяционная генетика. Владивосток: Изд-во Дальневост. Гос. Унив. , 2005. 234 с. • Картавцев Ю. Ф. , Ли Д. С. Анализ нуклеотидного разнообразия по генам цитохрома b и цитохромоксидазы 1 на популяционном, видовом и родовом уровнях // Генетика, 2006. T. 42. № 4. 437 -461. • Jung S. -O. , Lee Y. -M, Kartavtsev Y. P. , Park I. -S. , Kim D. -S. , Lee J-S. The complete mitochondrial genome of the Korean soft-shelled turtle Pelodiscus sinensis // DNA Sequence, 2006. V. 17(6). P. 471 -483. • Sasaki T. , Kartavtsev Y. P. , Uematsu T. , Sviridov V. V. , Hanzawa N. Phylogenetic independence of Far Eastern Leuciscinae (Pisces: Cyprinidae) inferred from mitochondrial DNA analysis // Gene and Genetic Systems, 2007 (Accepted). • Kartavtsev Y. P. , Lee Y. -M, Jung S-O, Byeon H-K, Son Y-, Lee J-S. Complete mitochondrial genome in the bullhead torrent catfish, Liobagrus obesus (Siluriformes, Amblycipididae) and phylogenetic considerations // Gene, 2007 (Accepted). • Kartavtsev Y. P. , Park T. -J. , Vinnikov K. A. , Ivankov V. N. , Sharina S. N. , Lee J. -S. Cytochrome b (Cyt-b) gene sequences analysis in six flatfish species (Pisces, Pleuronectidae) with phylogenetic and taxonomic insights // J. Marine Biol. , 2007 (Accepted).

СПАСИБО ЗА ВНИМАНИЕ!
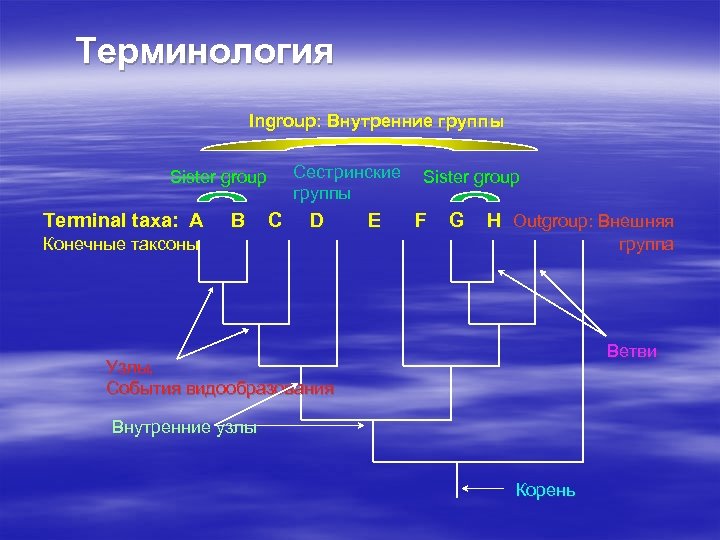
Терминология Ingroup: Внутренние группы Sister group Сестринские группы Sister group Terminal taxa: A B C D E F G H Outgroup: Внешняя Конечные таксоны группа Ветви Узлы, События видообразования Внутренние узлы Корень
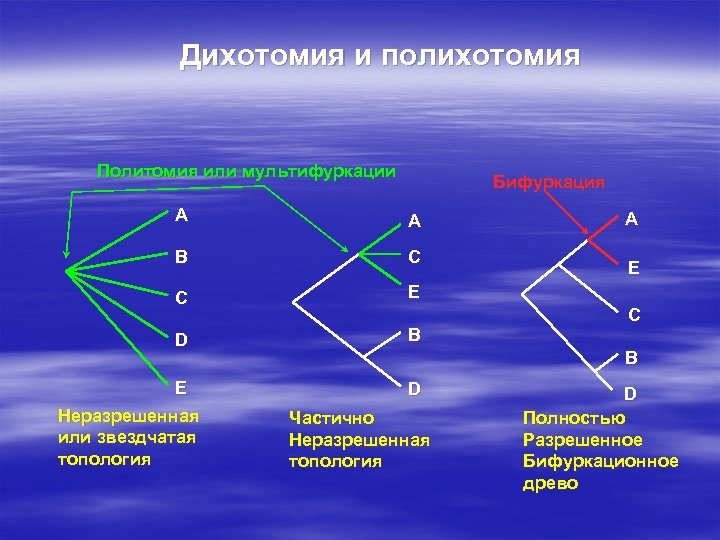
Дихотомия и полихотомия Политомия или мультифуркации Бифуркация A A B C C E D B E Неразрешенная или звездчатая топология A E C B D Частично Неразрешенная топология D Полностью Разрешенное Бифуркационное древо
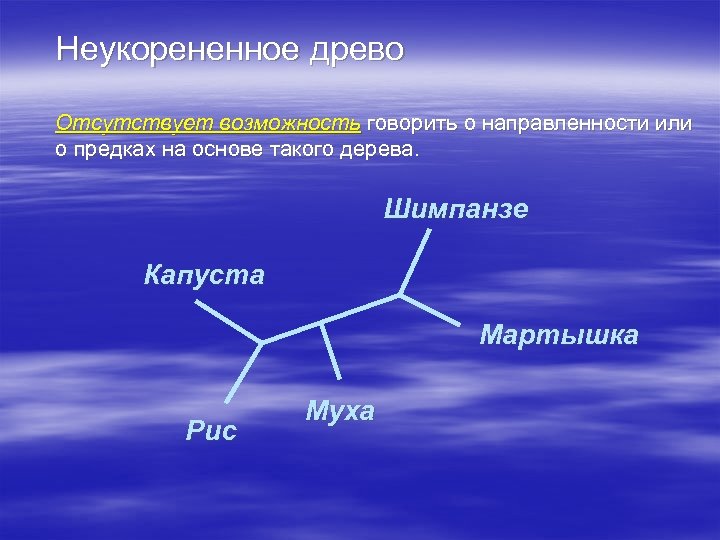
Неукорененное древо Отсутствует возможность говорить о направленности или о предках на основе такого дерева. Шимпанзе Капуста Мартышка Рис Муха
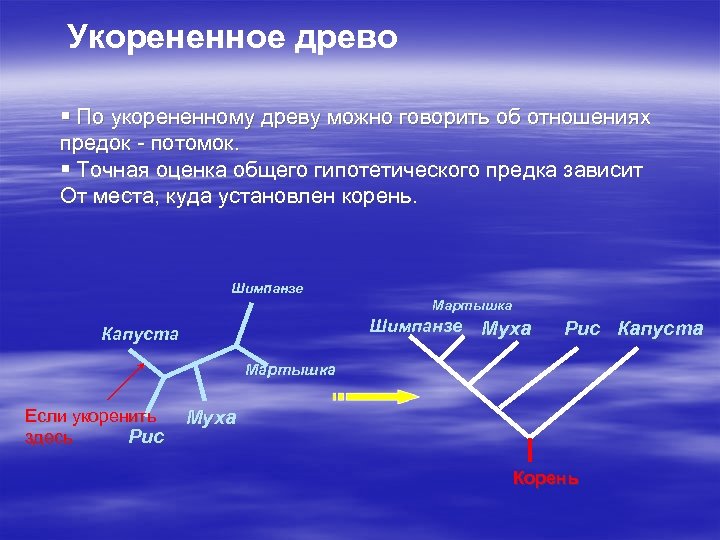
Укорененное древо § По укорененному древу можно говорить об отношениях предок - потомок. § Точная оценка общего гипотетического предка зависит От места, куда установлен корень. Шимпанзе Мартышка Шимпанзе Муха Капуста Рис Капуста Мартышка Если укоренить Рис здесь Муха Корень
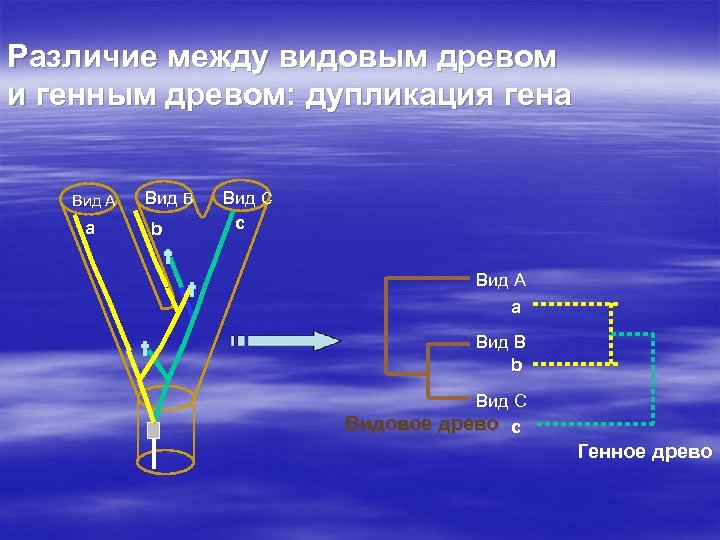
Различие между видовым древом и генным древом: дупликация гена Вид A a Вид B b Вид C c Вид A a Вид B b Вид C Видовое древо c Генное древо

Вскоре после видообразования сестринские таксоны с высокой вероятностью будут обнаруживать полифилетический статус генного древа После 4 N поколений сестринские таксоны окажутся с высокой вероятностью реципрокно монофилетичными Репродуктивная изоляция

СПИСОК ГЕНЕТИКОВ Москва 1. Захаров И. А. (ИОГен, чл-корр. РАН, зав. лабораторией, советник РАН) 2. Политов Д. В. (ИОГен, к. б. н. , зав. лабораторией) 3. Гордеева Н. В. (ИОГен, к. б. н. ) 4. Афанасьев К. И. (ИОГен, к. б. н. ) 5. Васильев В. П. (ИПЭЭ, д. б. н. ) 6. Рысков А. П. (ИБГ, чл-корр. РАН, зав. лабораторией) 6. Семенова С. А. (ИБГ, к. б. н. ) 7. Барминцев A. A. (ВНИРО, к. б. н. ) Владивосток 1. Картавцев Ю. Ф. (ИБМ, д. б. н. ) 2. Кухлевский А. Д. (ИБМ, к. б. н. ) 3. Шарина С. Н. (ИБМ, аспирант) 4. Шедько С. В. (БПИ, к. б. н. , зав. группой) 5. Чичвархин А. Ю. (БПИ, к. б. н. ) 6. Балакирев Е. С. (ДВГУ, д. б. н. , зав. лабораторией) 7. Винников К. А. (ДВГУ, аспирант) 8. Чичвархина O. В. (ИБМ, аспирант)

СПИСОК УЧАСТВУЮЩИХ ИХТИОЛОГОВ ДВ • ИБМ (4) Питрук Д. Л. (к. б. н. . , директор музея, зам. директора) Яковлев Ю. М. (к. б. н. ) Соколовский А. С. (к. б. н. ) Баланов А. А. (к. б. н. , зав. лабораторией) Долганов В. М. (д. б. н. ) • ДВГУ (4) Иванков В. Н. (д. б. н. ) Платошина Л. К. (к. б. н. ) Винников К. А. (аспирант) Рутенко О. А. (аспирант) • ИБПС (3) Черешнев И. А. (член-корр. РАН, директор) Шестаков А. В. (к. б. н. ) Грунин С. И. (аспирант) • ТИНРО-центр, Сах. НИРО, Камчат. НИРО и др. (7) Шунтов В. П. (д. б. н. , зав. лабораторией) Борисовец Е. Н. (к. б. н. , зав. лабораторией) Свиридов В. В. (к. б. н. ) Пушникова Г. М. (к. б. н. ) Карпенко В. В. (к. б. н. , зав. лабораторией) Золотухин С. Ф. (к. б. н. , зав. лабораторией) Рыбникова И. Г. (к. б. н. )

Подписка последовательностей в Gen. Bank (NCBI)

ВВЕДЕНИЕ n ДНК митохондрий (мт. ДНК) – это кольцевая молекула, длиной около 16 -18 тысяч пар нуклеотидов (тпн. ). Как показывают литературные данные, мт. ДНК всех рыб имеет сходную организацию (Lee et al. , 2001; Kim et al. , 2004; Kim et al. , 2005; Nagase et al. , 2005; Nohara et al. , 2005) и мало, чем отличается и у других позвоночных животных, включая человека (Anderson et al. , 1981; Bibb et al. , 1981; Wallace, 1992; Kogelnik et al. , 2005). Полный состав митоходриального генома (митогенома) включает: контрольный регион (CR или D петля), где сосредоточены сайт начала репликации и промоторы, большая (16 S) и малая (12 S) субъединицы р. РНК, 22 т. РНК и 13 полипептидных генов. n Филогенетические исследования обычно используют последовательности единичных генов, в том числе и генов ядерной ДНК, хотя в последние годы все чаше используют для этих целей и полный митогеном. Наиболее популярны в филогенетике последовательности генов цитохрома b (Cyt-b) и цитохром оксидазы 1 (Cо-1), которые используются для сравнения таксонов на уровне вид – семейство (Johns, Avise, 1998; Hebert et al. , 2004; Картавцев, Ли, 2006). Много последовательностей, несущих филогенетический сигнал, получено для разных групп также по гену 16 S р. РНК. Последовательности отдельных генов могут давать различный филогенетический сигнал из-за различных темпов замен и конкретной эволюционной и/или демографической судьбы таксона (дифференцированная сортировка филетических линий). Это относится и к разным участкам одного и того же гена. Кроме того, при сопоставлении многочисленных таксонов, особенно высокого ранга, возникают проблемы с эффектами гомоплазии и недостаточной информационной емкостью наборов последовательностей для филогенетических целей (Hilish et al. , 1996, Miya et al. , 2001). Тем не менее, для идентификации видов, за редкими исключениями, достаточно сравнение даже относительно коротких последовательностей, например гена цитохром оксидазы 1 (Со-1, 654 пн). n
4766f9b4f5885922d80715bc1976b3e0.ppt