Патрушв_Khimiki_3.ppt
- Количество слайдов: 70
 Репликация ДНК
Репликация ДНК

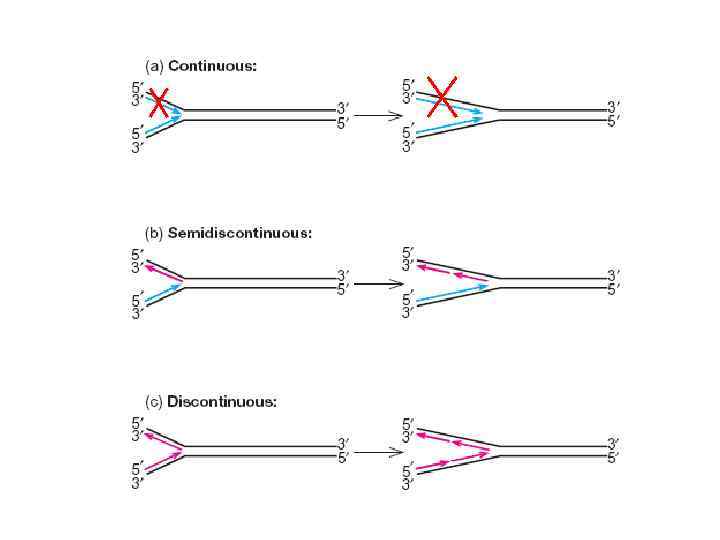
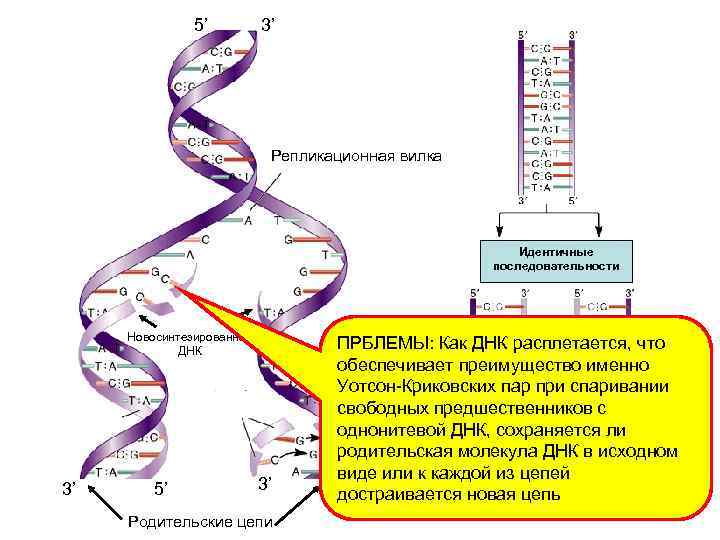 5’ 3’ Репликационная вилка Идентичные последовательности Новосинтезированная ДНК ПРБЛЕМЫ: Как ДНК расплетается, что обеспечивает преимущество именно Уотсон-Криковских пар при спаривании свободных предшественников с однонитевой ДНК, сохраняется ли родительская молекула ДНК в исходном виде или к каждой из цепей 3’ 5’ достраивается новая цепь Родительские цепи
5’ 3’ Репликационная вилка Идентичные последовательности Новосинтезированная ДНК ПРБЛЕМЫ: Как ДНК расплетается, что обеспечивает преимущество именно Уотсон-Криковских пар при спаривании свободных предшественников с однонитевой ДНК, сохраняется ли родительская молекула ДНК в исходном виде или к каждой из цепей 3’ 5’ достраивается новая цепь Родительские цепи
 В конце 50 х годов было сформулировано три модели репликации ДНК – Консервативная модель • Обе родительские цепи остаются вместе после репликации – Полуконсервативная модель • После репликации ДНК состоит из одной родительской и одной новосинтезированной цепи – Дисперсионная модель • После репликации каждая из цепей ДНК содержит фрагмерты родительской и новосинтезированной ДНК
В конце 50 х годов было сформулировано три модели репликации ДНК – Консервативная модель • Обе родительские цепи остаются вместе после репликации – Полуконсервативная модель • После репликации ДНК состоит из одной родительской и одной новосинтезированной цепи – Дисперсионная модель • После репликации каждая из цепей ДНК содержит фрагмерты родительской и новосинтезированной ДНК
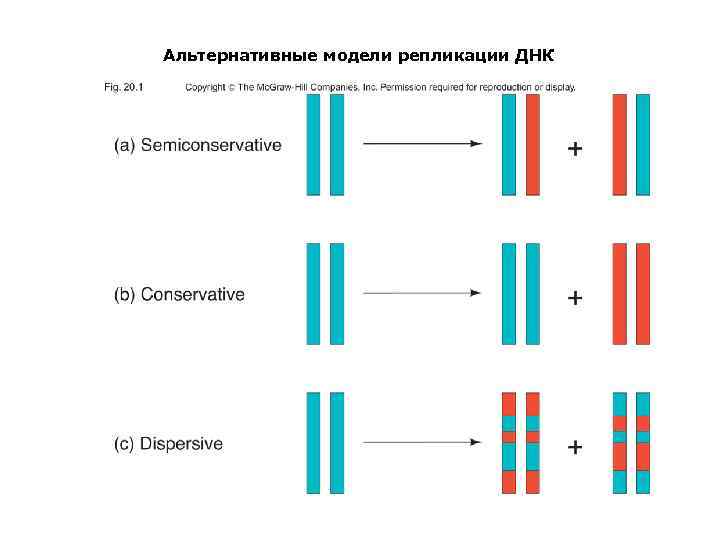 Альтернативные модели репликации ДНК
Альтернативные модели репликации ДНК
 • Опыт Мезельсона и Сталь 1958 – E. coli выращивают на среде, содержащей 15 N в течение длительного времени • Вся ДНК помечена тяжелым изотопом – Клетки переносят в среду, содержащую 14 N – После различных временных интервалов клетки отбирают и анализируют плавучую плотность ДНК посредством центрифугирования в градиенте плотности Cs. Cl
• Опыт Мезельсона и Сталь 1958 – E. coli выращивают на среде, содержащей 15 N в течение длительного времени • Вся ДНК помечена тяжелым изотопом – Клетки переносят в среду, содержащую 14 N – После различных временных интервалов клетки отбирают и анализируют плавучую плотность ДНК посредством центрифугирования в градиенте плотности Cs. Cl
 Центрифугирование в градиенте плотности Cs. Cl DNA 14 N 15 N 100000 g
Центрифугирование в градиенте плотности Cs. Cl DNA 14 N 15 N 100000 g
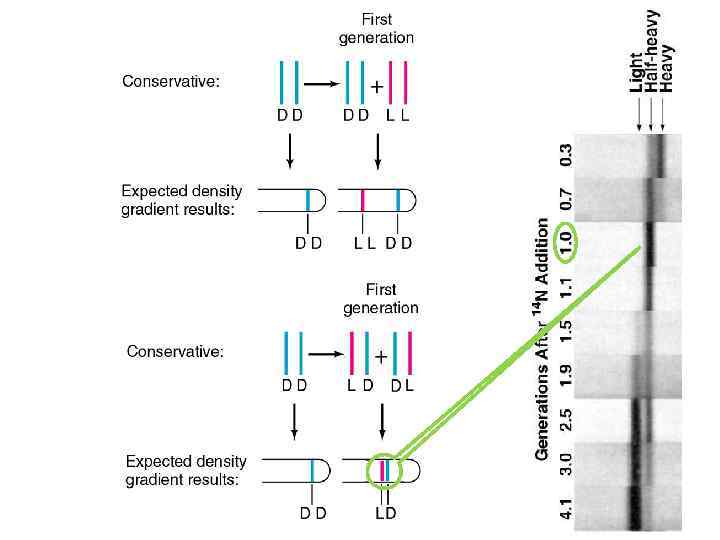

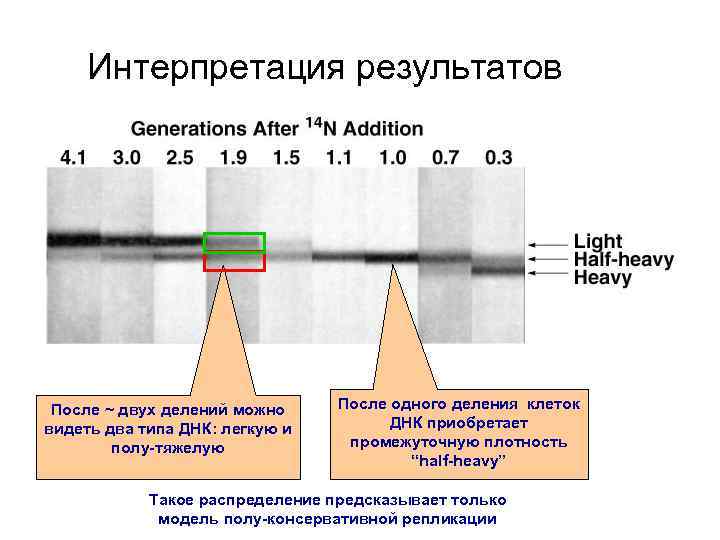 Интерпретация результатов После ~ двух делений можно После одного деления клеток видеть два типа ДНК: легкую и ДНК приобретает полу-тяжелую промежуточную плотность “half-heavy” Такое распределение предсказывает только модель полу-консервативной репликации
Интерпретация результатов После ~ двух делений можно После одного деления клеток видеть два типа ДНК: легкую и ДНК приобретает полу-тяжелую промежуточную плотность “half-heavy” Такое распределение предсказывает только модель полу-консервативной репликации
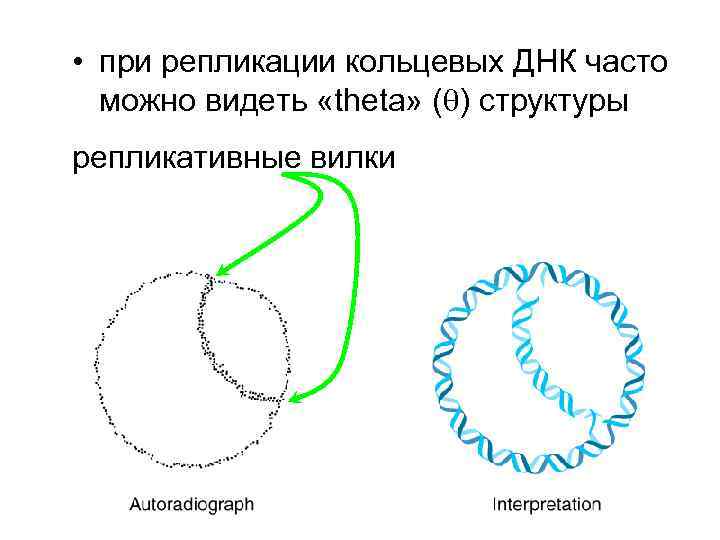 • при репликации кольцевых ДНК часто можно видеть «theta» ( ) структуры репликативные вилки
• при репликации кольцевых ДНК часто можно видеть «theta» ( ) структуры репликативные вилки
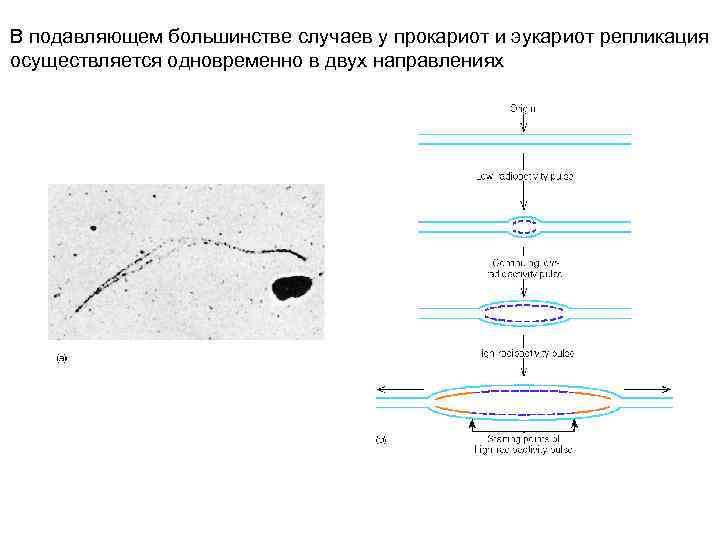 В подавляющем большинстве случаев у прокариот и эукариот репликация осуществляется одновременно в двух направлениях
В подавляющем большинстве случаев у прокариот и эукариот репликация осуществляется одновременно в двух направлениях
 1. Репликация ДНК осуществляется согласно полуконсервативной модели 2. Репликация ДНК происходит двунаправленно
1. Репликация ДНК осуществляется согласно полуконсервативной модели 2. Репликация ДНК происходит двунаправленно
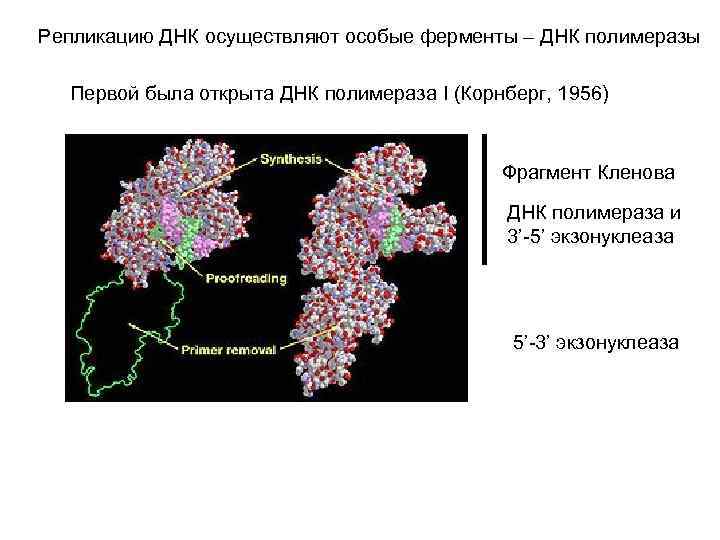 Репликацию ДНК осуществляют особые ферменты – ДНК полимеразы Первой была открыта ДНК полимераза I (Корнберг, 1956) Фрагмент Кленова ДНК полимераза и 3’-5’ экзонуклеаза 5’-3’ экзонуклеаза
Репликацию ДНК осуществляют особые ферменты – ДНК полимеразы Первой была открыта ДНК полимераза I (Корнберг, 1956) Фрагмент Кленова ДНК полимераза и 3’-5’ экзонуклеаза 5’-3’ экзонуклеаза
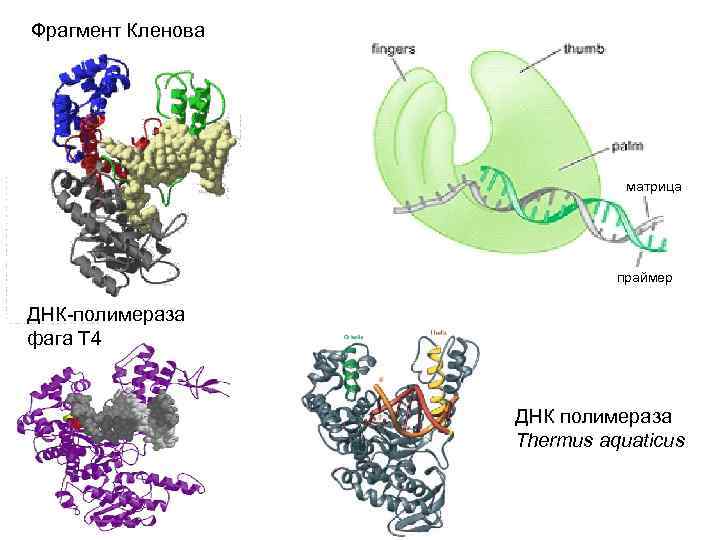 Фрагмент Кленова матрица праймер ДНК-полимераза фага Т 4 ДНК полимераза Thermus aquaticus
Фрагмент Кленова матрица праймер ДНК-полимераза фага Т 4 ДНК полимераза Thermus aquaticus
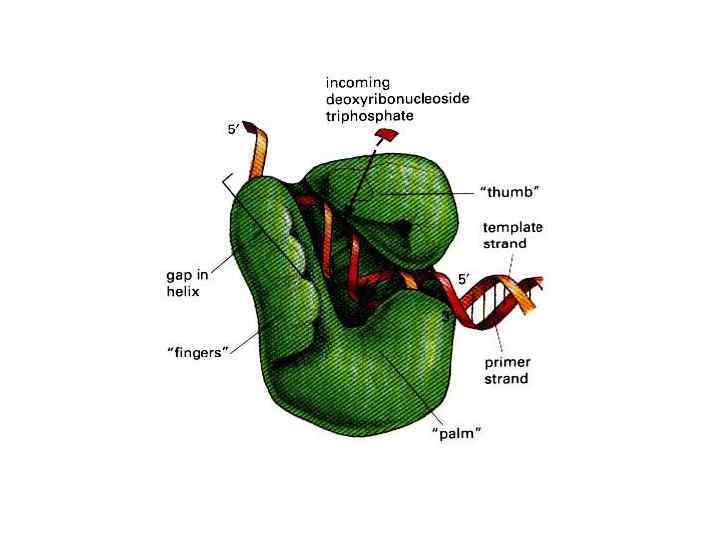
 DNA Polymerase-finger domain Fingers Thumb DNA Polymerase-palm domain Связывает d. NTP, и помещает один Содержит два активных центра, правильно спаренные d. NTP в каталитический – осуществляет полимеризацию, другой удаление ошибочно включенных центр нуклеотидов. Связывает матрицу и обеспечивает Полимеразный домен осуществляет экспонирование основания, с которым контроль правильности спаривания должен спариваться очередной d. NTP посредством контактов с основаниями в Palm области малой бороздки
DNA Polymerase-finger domain Fingers Thumb DNA Polymerase-palm domain Связывает d. NTP, и помещает один Содержит два активных центра, правильно спаренные d. NTP в каталитический – осуществляет полимеризацию, другой удаление ошибочно включенных центр нуклеотидов. Связывает матрицу и обеспечивает Полимеразный домен осуществляет экспонирование основания, с которым контроль правильности спаривания должен спариваться очередной d. NTP посредством контактов с основаниями в Palm области малой бороздки
 DNA Polymerase-thumb domain Прямо не участвует в катализе Взаимодействует с матрицей и синтезирующейся ДНК с тем, чтобы обеспечить правильную позиция праймера относительно активного центра, одновременно препятствуя «соскакиванию» полимеразы с ДНК
DNA Polymerase-thumb domain Прямо не участвует в катализе Взаимодействует с матрицей и синтезирующейся ДНК с тем, чтобы обеспечить правильную позиция праймера относительно активного центра, одновременно препятствуя «соскакиванию» полимеразы с ДНК
 ДНК полимераза использует один и тот же каталитический центр для включения в ДНК четырех разных нуклеотидов При этом важное значение имеет мониторинг правильного спаривания. Включение ошибочного нуклеотида резко снижает скорость катализа
ДНК полимераза использует один и тот же каталитический центр для включения в ДНК четырех разных нуклеотидов При этом важное значение имеет мониторинг правильного спаривания. Включение ошибочного нуклеотида резко снижает скорость катализа
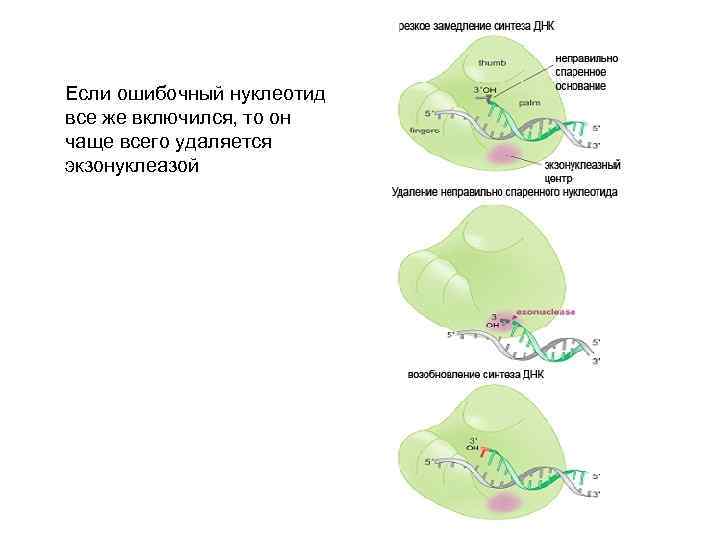 Если ошибочный нуклеотид все же включился, то он чаще всего удаляется экзонуклеазой
Если ошибочный нуклеотид все же включился, то он чаще всего удаляется экзонуклеазой
 С помощью генетических подходов было (утрата гена и ts мутации) было показано, что DNA pol I не является репликативным ферментом. У E. Coli есть еще две ДНК полимеразы (Pol II и Pol III). Репликацию осуществляет Pol III (показано посредством анализа ts мутаий). (холофермент) Mutant viable? Yes! No Function repair replication + DNA pol IV: mutagenesis + DNA pol V: error-prone repair
С помощью генетических подходов было (утрата гена и ts мутации) было показано, что DNA pol I не является репликативным ферментом. У E. Coli есть еще две ДНК полимеразы (Pol II и Pol III). Репликацию осуществляет Pol III (показано посредством анализа ts мутаий). (холофермент) Mutant viable? Yes! No Function repair replication + DNA pol IV: mutagenesis + DNA pol V: error-prone repair

 Важными характеристиками ДНК-полимераз являются точность работы (Fidelity) и процессивность. Точность работы обеспечивается наличием 3’ – 5’ экзонуклеазной активности, которая удаляет включенные по ошибке некомплементарные нуклеотиды. Pol I имеет полимеразный и 3’ – 5’ экзонуклеазный домены в составе одной полипептидной цепи. Pol III состоит из трех субъединиц, одна из которых ( ) обладает ДНК- полимеразной активностью, а другая – (ε) обладает 3’ – 5’ экзонуклеазной активностью. Функции третьей субъединицы (θ) не известны. -5 Pol III без экзонуклеазной субъединицы делает ошибки с частотой 10 При наличии экзонуклеазной активности вероятность ошибки составляет 10 -10
Важными характеристиками ДНК-полимераз являются точность работы (Fidelity) и процессивность. Точность работы обеспечивается наличием 3’ – 5’ экзонуклеазной активности, которая удаляет включенные по ошибке некомплементарные нуклеотиды. Pol I имеет полимеразный и 3’ – 5’ экзонуклеазный домены в составе одной полипептидной цепи. Pol III состоит из трех субъединиц, одна из которых ( ) обладает ДНК- полимеразной активностью, а другая – (ε) обладает 3’ – 5’ экзонуклеазной активностью. Функции третьей субъединицы (θ) не известны. -5 Pol III без экзонуклеазной субъединицы делает ошибки с частотой 10 При наличии экзонуклеазной активности вероятность ошибки составляет 10 -10
 Свойства ДНК полимераз ДНК полимеразы of E. coli_ pol III (core) Полимеризация: 5’ to 3’ yes yes Корректирующая экзонуклеаза: 3’ to 5’ yes yes Репарационная экзонуклеаза: 5’ to 3’ yes no no ДНК полимераза III является главным репликационным ферментом ДНК полимераза I участвует в заполнении брешей и удалении праймеров на отстающей цепи. Этот фермент также участвует в репарации повреждений ДНК
Свойства ДНК полимераз ДНК полимеразы of E. coli_ pol III (core) Полимеризация: 5’ to 3’ yes yes Корректирующая экзонуклеаза: 3’ to 5’ yes yes Репарационная экзонуклеаза: 5’ to 3’ yes no no ДНК полимераза III является главным репликационным ферментом ДНК полимераза I участвует в заполнении брешей и удалении праймеров на отстающей цепи. Этот фермент также участвует в репарации повреждений ДНК
 Schematic 3
Schematic 3

 Все три ДНК полимеразы E. Coli достраивают цепочку ДНК к 3’ концу предсуществующего праймера, используя в качестве матрицы однонитевую ДНК DNApol 5’ 3’ 5’ Все ДНК полимеразы нуждаются в праймере со свободной 3’ OH группой Все ДНК полимеразы работают только на однонитевой матрице Все ДНК полимеразы синтезируют ДНК в направлении 5’ - 3’ Процессивность собственно полимеразных субъединиц всех трех ДНК полимераз E. Coli является крайне низкой
Все три ДНК полимеразы E. Coli достраивают цепочку ДНК к 3’ концу предсуществующего праймера, используя в качестве матрицы однонитевую ДНК DNApol 5’ 3’ 5’ Все ДНК полимеразы нуждаются в праймере со свободной 3’ OH группой Все ДНК полимеразы работают только на однонитевой матрице Все ДНК полимеразы синтезируют ДНК в направлении 5’ - 3’ Процессивность собственно полимеразных субъединиц всех трех ДНК полимераз E. Coli является крайне низкой
 У прокариот фрагменты Оказаки имеют размер 1000 – 2000 н С помощью различных биохимических подходов было продемонстрировано, что на 5’ концах фрагментов Оказаки находятся короткие (10 - 12 н) последовательности РНК (праймеры) 32 альфа- P-ГТФ guanylyl transferase (RNA CAP) DNA RNA DNase I Благодаря наличию РНК на 5’ конце, фрагменты Оказаки не расщепляются 5’ экзонуклеазой фага лямбда
У прокариот фрагменты Оказаки имеют размер 1000 – 2000 н С помощью различных биохимических подходов было продемонстрировано, что на 5’ концах фрагментов Оказаки находятся короткие (10 - 12 н) последовательности РНК (праймеры) 32 альфа- P-ГТФ guanylyl transferase (RNA CAP) DNA RNA DNase I Благодаря наличию РНК на 5’ конце, фрагменты Оказаки не расщепляются 5’ экзонуклеазой фага лямбда
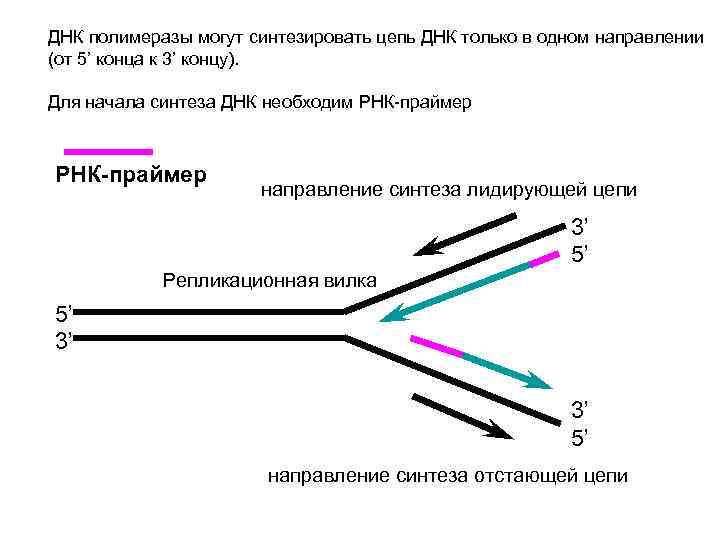 ДНК полимеразы могут синтезировать цепь ДНК только в одном направлении (от 5’ конца к 3’ концу). Для начала синтеза ДНК необходим РНК-праймер направление синтеза лидирующей цепи 3’ 5’ Репликационная вилка 5’ 3’ 5’ направление синтеза отстающей цепи
ДНК полимеразы могут синтезировать цепь ДНК только в одном направлении (от 5’ конца к 3’ концу). Для начала синтеза ДНК необходим РНК-праймер направление синтеза лидирующей цепи 3’ 5’ Репликационная вилка 5’ 3’ 5’ направление синтеза отстающей цепи
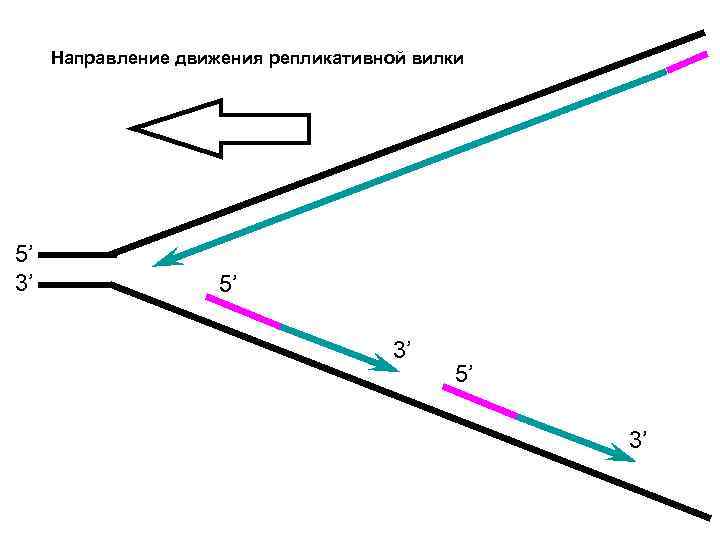 Направление движения репликативной вилки 5’ 3’ 5’ 3’ 5’ 3’
Направление движения репликативной вилки 5’ 3’ 5’ 3’ 5’ 3’
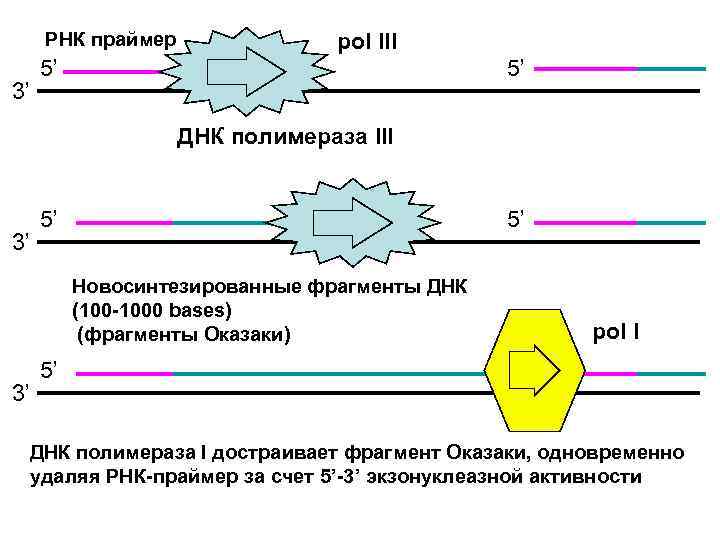 РНК праймер pol III 5’ 5’ 3’ ДНК полимераза III 5’ 3’ Новосинтезированные фрагменты ДНК (100 -1000 bases) (фрагменты Оказаки) pol I 5’ 3’ ДНК полимераза I достраивает фрагмент Оказаки, одновременно удаляя РНК-праймер за счет 5’-3’ экзонуклеазной активности
РНК праймер pol III 5’ 5’ 3’ ДНК полимераза III 5’ 3’ Новосинтезированные фрагменты ДНК (100 -1000 bases) (фрагменты Оказаки) pol I 5’ 3’ ДНК полимераза I достраивает фрагмент Оказаки, одновременно удаляя РНК-праймер за счет 5’-3’ экзонуклеазной активности
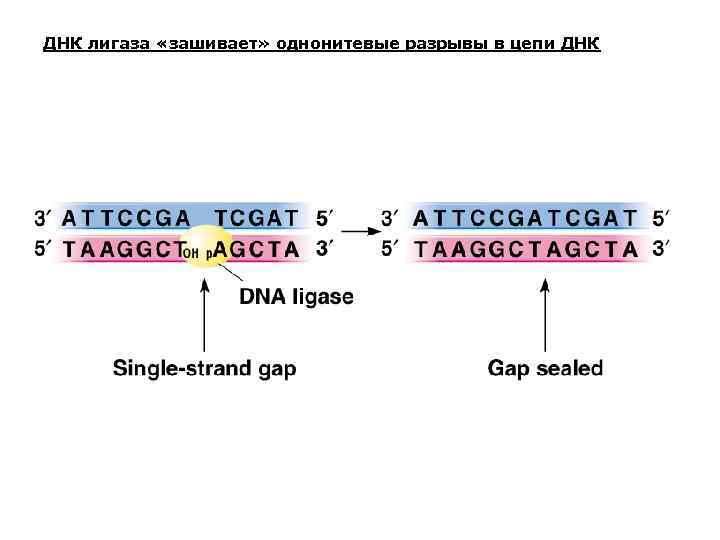 ДНК лигаза «зашивает» однонитевые разрывы в цепи ДНК
ДНК лигаза «зашивает» однонитевые разрывы в цепи ДНК
 Основные компоненты репликативной вилки E. Coli DNA гираза – генерирует негативные супервитки, которые компенсируют позитивные супервитки, накапливающиеся в результате pol III 3’ раскручивания ДНК 5’ 5’ 3’ B DNA лигаза G C белок, связывающийся с однонитевой ДНК праймаза (SSB) хеликаза, работающая в ori и в участках pol III начала синтеза фрагментов Оказаки Праймосома pol I
Основные компоненты репликативной вилки E. Coli DNA гираза – генерирует негативные супервитки, которые компенсируют позитивные супервитки, накапливающиеся в результате pol III 3’ раскручивания ДНК 5’ 5’ 3’ B DNA лигаза G C белок, связывающийся с однонитевой ДНК праймаза (SSB) хеликаза, работающая в ori и в участках pol III начала синтеза фрагментов Оказаки Праймосома pol I
 Раскручивание двойной спирали ДНК в ходе репликации приводит к возникновению положительных витков перед репликативной вилкой, которые убираются ДНК-топоизомеразами
Раскручивание двойной спирали ДНК в ходе репликации приводит к возникновению положительных витков перед репликативной вилкой, которые убираются ДНК-топоизомеразами
 Компоненты репликационной машины E. Coli dna. A Связывается с участком начала репликации Primasome dna. B хеликаза (расплетает ДНК в ori) dna. C связывается с dna. B dna. G праймаза (синтезирует РНК праймер) DNA gyrase вносит отрицательные супервитки перед репликативной вилкой Rep protein хеликаза (расплетает ДНК перед репликативной вилкой) SSB связывается с однонитевой ДНК DNA pol III главная репликативная полимераза DNA pol I удаляет РНК-праймеры и заполняет бреши DNA ligase «зашивает» однонитевые разрывы
Компоненты репликационной машины E. Coli dna. A Связывается с участком начала репликации Primasome dna. B хеликаза (расплетает ДНК в ori) dna. C связывается с dna. B dna. G праймаза (синтезирует РНК праймер) DNA gyrase вносит отрицательные супервитки перед репликативной вилкой Rep protein хеликаза (расплетает ДНК перед репликативной вилкой) SSB связывается с однонитевой ДНК DNA pol III главная репликативная полимераза DNA pol I удаляет РНК-праймеры и заполняет бреши DNA ligase «зашивает» однонитевые разрывы
 Обладающая полимеразной активностью субъединица ДНК полимеразы III характеризуется очень низкой процессивностью (10 нуклеотидов). Процессивность синтеза обеспечивается бета субъединицей, удерживающей полимеразу на ДНК. Для посадки бета-субъединицы на ДНК необходимы дополнительные белки
Обладающая полимеразной активностью субъединица ДНК полимеразы III характеризуется очень низкой процессивностью (10 нуклеотидов). Процессивность синтеза обеспечивается бета субъединицей, удерживающей полимеразу на ДНК. Для посадки бета-субъединицы на ДНК необходимы дополнительные белки
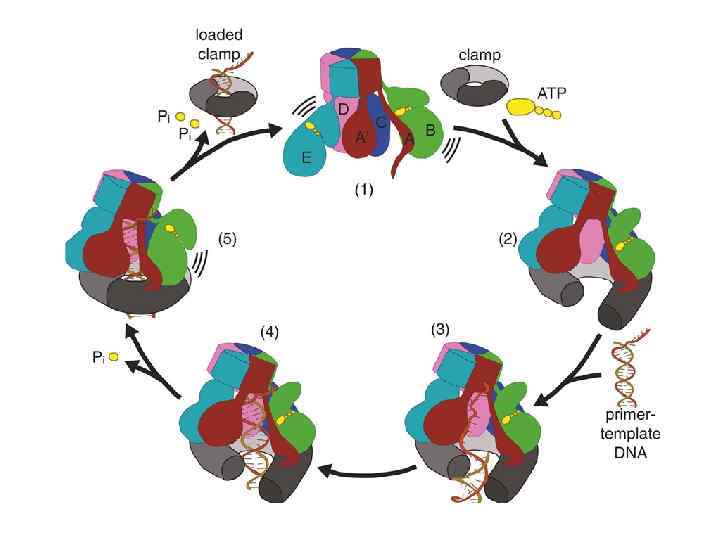
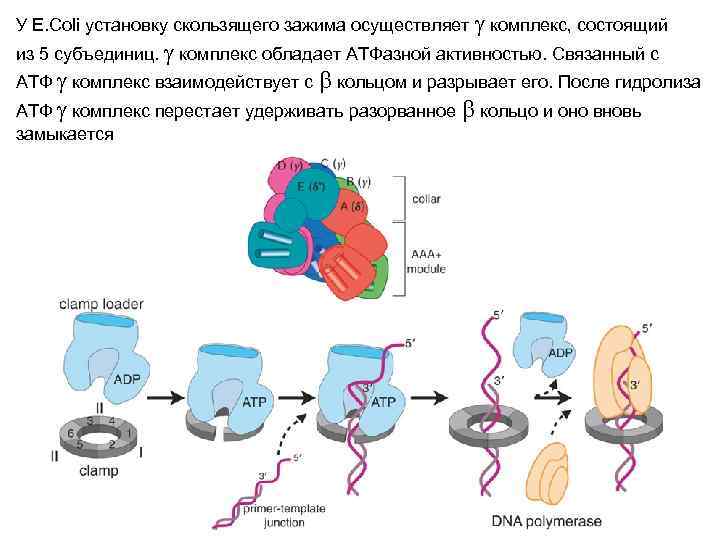 У E. Coli установку скользящего зажима осуществляет g комплекс, состоящий из 5 субъединиц. g комплекс обладает АТФазной активностью. Связанный с АТФ g комплекс взаимодействует с b кольцом и разрывает его. После гидролиза АТФ g комплекс перестает удерживать разорванное b кольцо и оно вновь замыкается
У E. Coli установку скользящего зажима осуществляет g комплекс, состоящий из 5 субъединиц. g комплекс обладает АТФазной активностью. Связанный с АТФ g комплекс взаимодействует с b кольцом и разрывает его. После гидролиза АТФ g комплекс перестает удерживать разорванное b кольцо и оно вновь замыкается
 Непосредственно осуществляющие репликацию ферменты организованы в единый комплекс, получивший название РЕПЛИСОМА В состав реплисомы входят: 2 DNA Pol III холофермента, состоящих из, α, ε и θ subunits. α субъединица обладает полимеразной активностью. ε обладает корректирующей экзонуклеазной активностью (3’ – 5’). θ субъединица стимулирует работу ε субъединицы. 2 β кольца (зажима), которые удерживают полимеразу на ДНК 2 t субъединицы, которые связывают 2 молекулы ДНК полимеразы 1 блок из 5 субъединиц (g комплекс) , которые осуществляют посадку b колец на праймеры для синтеза фрагментов Оказаки
Непосредственно осуществляющие репликацию ферменты организованы в единый комплекс, получивший название РЕПЛИСОМА В состав реплисомы входят: 2 DNA Pol III холофермента, состоящих из, α, ε и θ subunits. α субъединица обладает полимеразной активностью. ε обладает корректирующей экзонуклеазной активностью (3’ – 5’). θ субъединица стимулирует работу ε субъединицы. 2 β кольца (зажима), которые удерживают полимеразу на ДНК 2 t субъединицы, которые связывают 2 молекулы ДНК полимеразы 1 блок из 5 субъединиц (g комплекс) , которые осуществляют посадку b колец на праймеры для синтеза фрагментов Оказаки

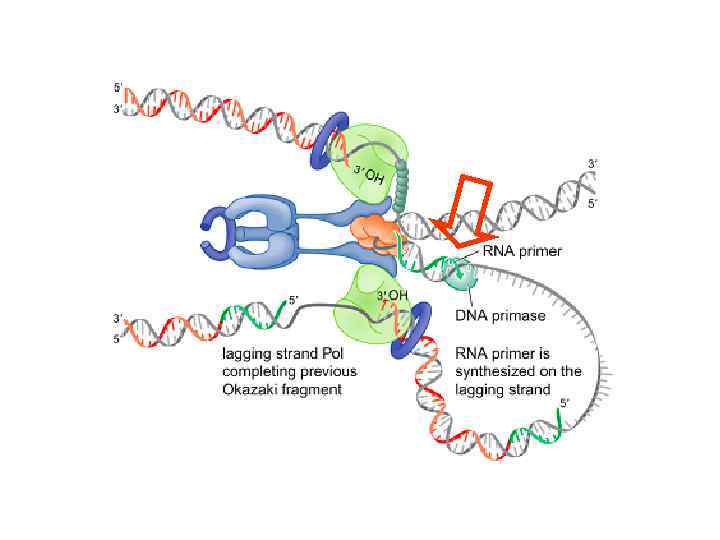
 ДНК-праймаза освобождается ДНК-полимераза III Освобождается из комплекса с ДНК и бета-кольцом, но остается связанной с репликативной вилкой
ДНК-праймаза освобождается ДНК-полимераза III Освобождается из комплекса с ДНК и бета-кольцом, но остается связанной с репликативной вилкой
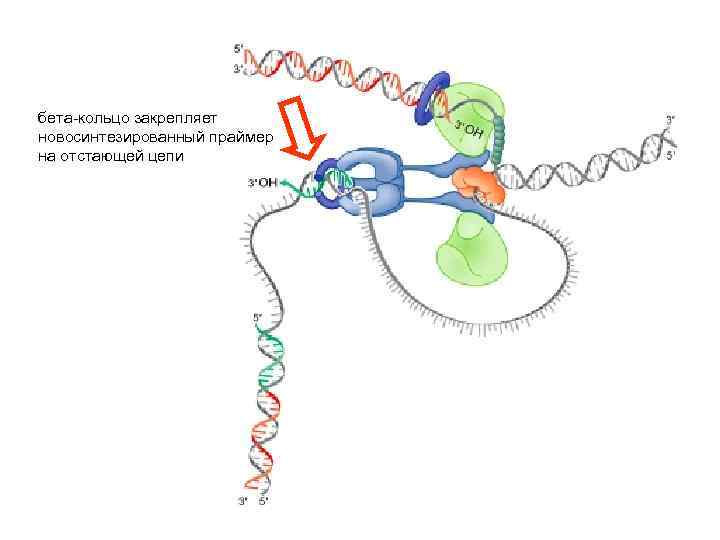 бета-кольцо закрепляет новосинтезированный праймер на отстающей цепи
бета-кольцо закрепляет новосинтезированный праймер на отстающей цепи
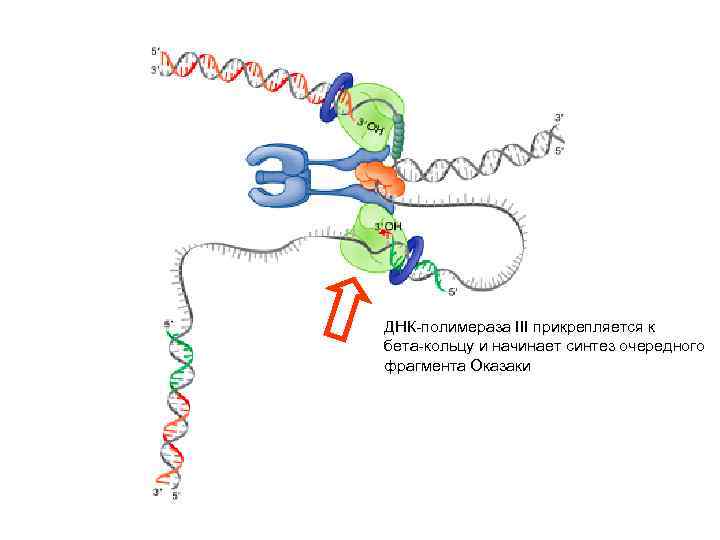 ДНК-полимераза III прикрепляется к бета-кольцу и начинает синтез очередного фрагмента Оказаки
ДНК-полимераза III прикрепляется к бета-кольцу и начинает синтез очередного фрагмента Оказаки
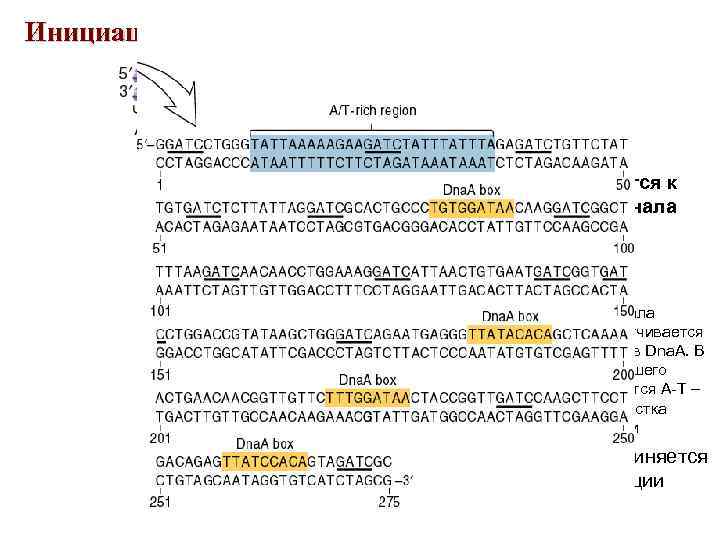 Инициация репликации ДНК на ori. C • Белки Dna. A присоединяются к Dna. A боксам в участке начала репликации – Часть участка начала репликации «накручивается на комплекс белков Dna. A. В результате возникшего напряжения плавится А-Т – богатый домен участка начала репликации белок (хеликаза) присоединяется к участку начала репликации
Инициация репликации ДНК на ori. C • Белки Dna. A присоединяются к Dna. A боксам в участке начала репликации – Часть участка начала репликации «накручивается на комплекс белков Dna. A. В результате возникшего напряжения плавится А-Т – богатый домен участка начала репликации белок (хеликаза) присоединяется к участку начала репликации
 Uses energy from ATP to unwind the duplex DNA Хеликаза начинает раскручивать ДНК в обоих направлениях, создавая возможность сборки двух репликативных комплексов SSB
Uses energy from ATP to unwind the duplex DNA Хеликаза начинает раскручивать ДНК в обоих направлениях, создавая возможность сборки двух репликативных комплексов SSB
 Репликация ДНК у эукариот Общие черты репликации ДНК у прокариот и эукариот 1. Полуконсервативный синтез ДНК 2. Двунаправленный синтез ДНК 3 Синтез в направлении 5’-3’. Соответственно, хотя бы одна цепь синтезируется в виде фрагментов Оказаки 4. Инициация синтеза ДНК посредством РНК-праймеров
Репликация ДНК у эукариот Общие черты репликации ДНК у прокариот и эукариот 1. Полуконсервативный синтез ДНК 2. Двунаправленный синтез ДНК 3 Синтез в направлении 5’-3’. Соответственно, хотя бы одна цепь синтезируется в виде фрагментов Оказаки 4. Инициация синтеза ДНК посредством РНК-праймеров

 Pol - PRIMASE DNA Pol (I): primase complex- bifunctional Heterotetrameric phosphoprotein 48 k. Da PRIMASE- initiates DNA synthesis (makes complementary RNA primer) 58 k. Da protein- tethers primase to 180 k. Da subunit 180 k. Da polymerase A subunit- extends RNA primer 70 k. Da subunit- no known catalytic function (may recruit pol : primase to the replication fork)
Pol - PRIMASE DNA Pol (I): primase complex- bifunctional Heterotetrameric phosphoprotein 48 k. Da PRIMASE- initiates DNA synthesis (makes complementary RNA primer) 58 k. Da protein- tethers primase to 180 k. Da subunit 180 k. Da polymerase A subunit- extends RNA primer 70 k. Da subunit- no known catalytic function (may recruit pol : primase to the replication fork)
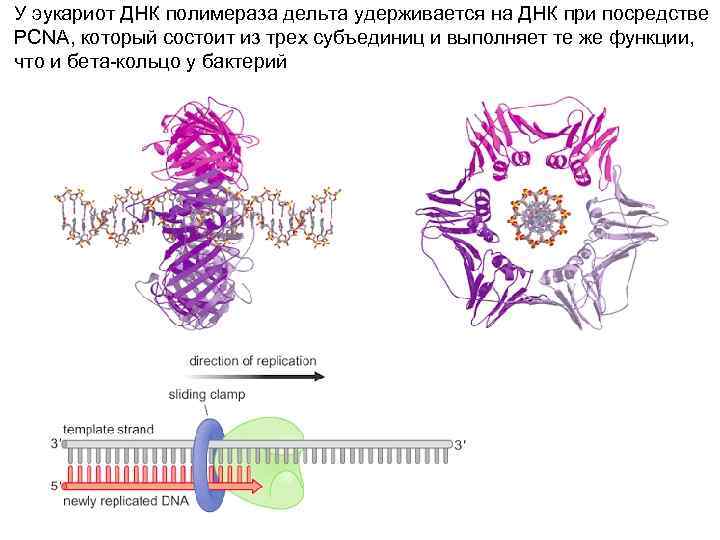 У эукариот ДНК полимераза дельта удерживается на ДНК при посредстве PCNA, который состоит из трех субъединиц и выполняет те же функции, что и бета-кольцо у бактерий
У эукариот ДНК полимераза дельта удерживается на ДНК при посредстве PCNA, который состоит из трех субъединиц и выполняет те же функции, что и бета-кольцо у бактерий
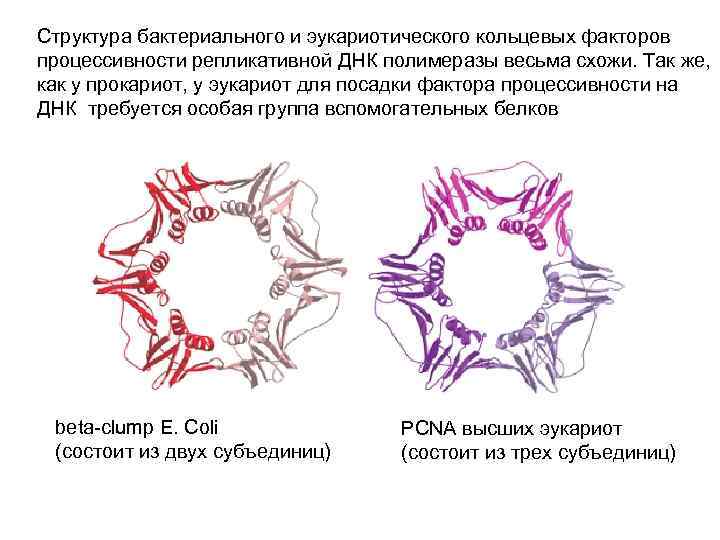 Структура бактериального и эукариотического кольцевых факторов процессивности репликативной ДНК полимеразы весьма схожи. Так же, как у прокариот, у эукариот для посадки фактора процессивности на ДНК требуется особая группа вспомогательных белков beta-clump E. Coli PCNA высших эукариот (состоит из двух субъединиц) (состоит из трех субъединиц)
Структура бактериального и эукариотического кольцевых факторов процессивности репликативной ДНК полимеразы весьма схожи. Так же, как у прокариот, у эукариот для посадки фактора процессивности на ДНК требуется особая группа вспомогательных белков beta-clump E. Coli PCNA высших эукариот (состоит из двух субъединиц) (состоит из трех субъединиц)
 Структура эукариотического комплекса, осуществляющего посадку скользящего зажима похожа на структуру бактериального g комплекса
Структура эукариотического комплекса, осуществляющего посадку скользящего зажима похожа на структуру бактериального g комплекса
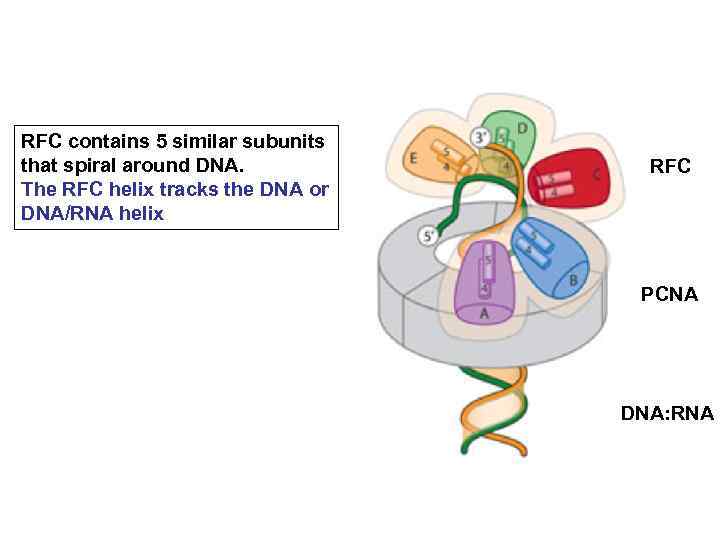 RFC contains 5 similar subunits that spiral around DNA. RFC The RFC helix tracks the DNA or DNA/RNA helix PCNA DNA: RNA
RFC contains 5 similar subunits that spiral around DNA. RFC The RFC helix tracks the DNA or DNA/RNA helix PCNA DNA: RNA
 RFC: PCNA crystal structure
RFC: PCNA crystal structure
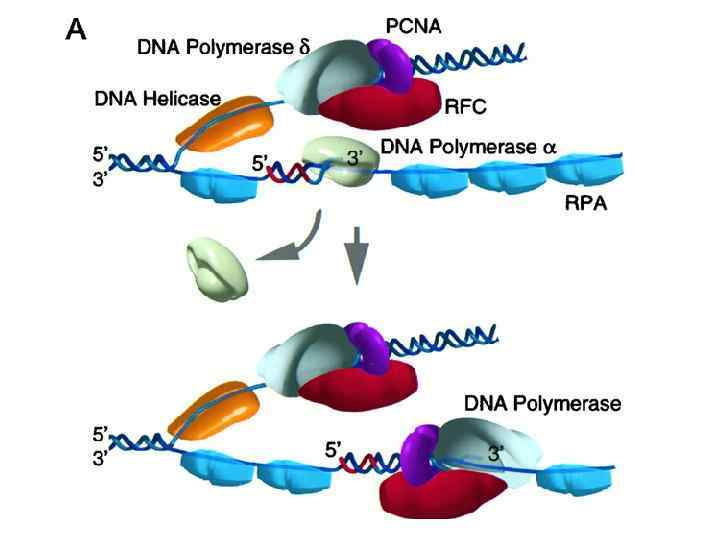
 Процессинг фрагментов Оказаки РНК ДНК, построенная Pol delta 7 -14 n Pol a (low fidelity) ~200 n ~ 20 n Это надо удалить и перезастроить
Процессинг фрагментов Оказаки РНК ДНК, построенная Pol delta 7 -14 n Pol a (low fidelity) ~200 n ~ 20 n Это надо удалить и перезастроить
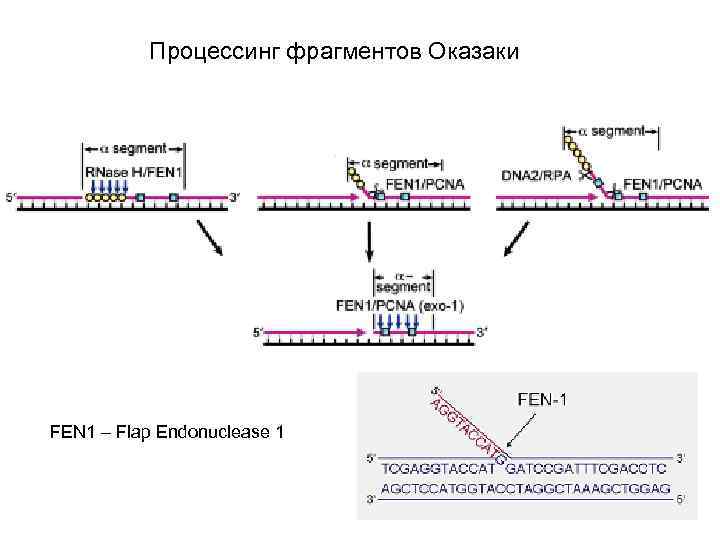 Процессинг фрагментов Оказаки FEN 1 – Flap Endonuclease 1
Процессинг фрагментов Оказаки FEN 1 – Flap Endonuclease 1
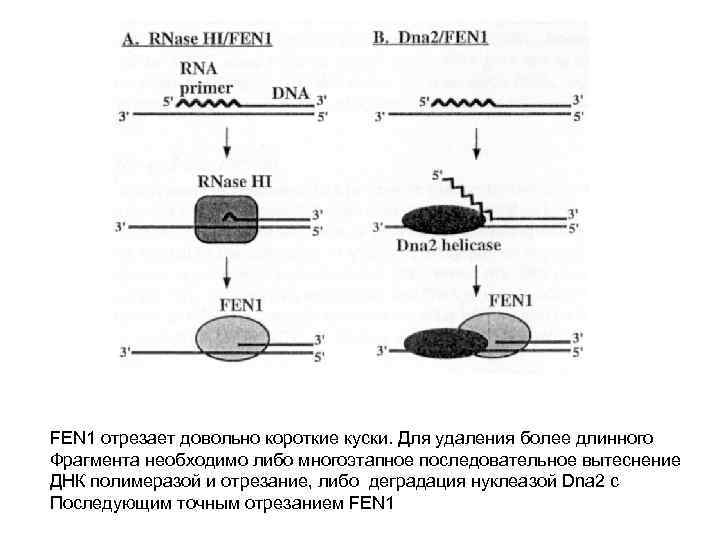 FEN 1 отрезает довольно короткие куски. Для удаления более длинного Фрагмента необходимо либо многоэтапное последовательное вытеснение ДНК полимеразой и отрезание, либо деградация нуклеазой Dna 2 с Последующим точным отрезанием FEN 1
FEN 1 отрезает довольно короткие куски. Для удаления более длинного Фрагмента необходимо либо многоэтапное последовательное вытеснение ДНК полимеразой и отрезание, либо деградация нуклеазой Dna 2 с Последующим точным отрезанием FEN 1
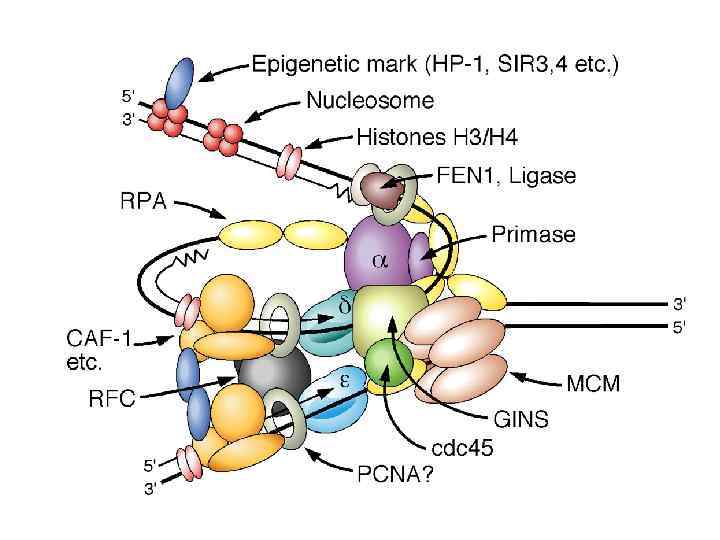
 Компоненты репликационной машины эукариот 1. DNA pol (I) + 2 primase subunits: синтезирует праймеры RNA-DNAi (3 -4 b) для синтеза ведущей и отстающей цепей. 2. DNA pol (III) : элонгация ведущей и отстающей цепей (возможно, элонгация ведущей цепи – pol e) 3. PCNA: (proliferating cell nuclear antigen) фактор процессивности pol d 4. Факторы репликации C (RF-C; посадка зажима, ATPase) and RF-A (связывание однонитевой ДНК). 5. Topoisomerases I & II: регулируют топологию ДНК.
Компоненты репликационной машины эукариот 1. DNA pol (I) + 2 primase subunits: синтезирует праймеры RNA-DNAi (3 -4 b) для синтеза ведущей и отстающей цепей. 2. DNA pol (III) : элонгация ведущей и отстающей цепей (возможно, элонгация ведущей цепи – pol e) 3. PCNA: (proliferating cell nuclear antigen) фактор процессивности pol d 4. Факторы репликации C (RF-C; посадка зажима, ATPase) and RF-A (связывание однонитевой ДНК). 5. Topoisomerases I & II: регулируют топологию ДНК.
 Вспомогательные белки репликативной машины эукариот PCNA 36 KD; нет видовой специфичности фактор процессивности pol d тример PCNA образует скользящий зажим, который удерживает pol d на ДНК RFA (Replication Factor A) субъединицы 70 KD, 34 KD, 11 KD; видовая специфичность белок, связывающий однонтиевую ДНК RFC (Replication Factor C) нет видовой специфичности активность АТФазы необходим для посадки скользящего зажима (PCNA)
Вспомогательные белки репликативной машины эукариот PCNA 36 KD; нет видовой специфичности фактор процессивности pol d тример PCNA образует скользящий зажим, который удерживает pol d на ДНК RFA (Replication Factor A) субъединицы 70 KD, 34 KD, 11 KD; видовая специфичность белок, связывающий однонтиевую ДНК RFC (Replication Factor C) нет видовой специфичности активность АТФазы необходим для посадки скользящего зажима (PCNA)
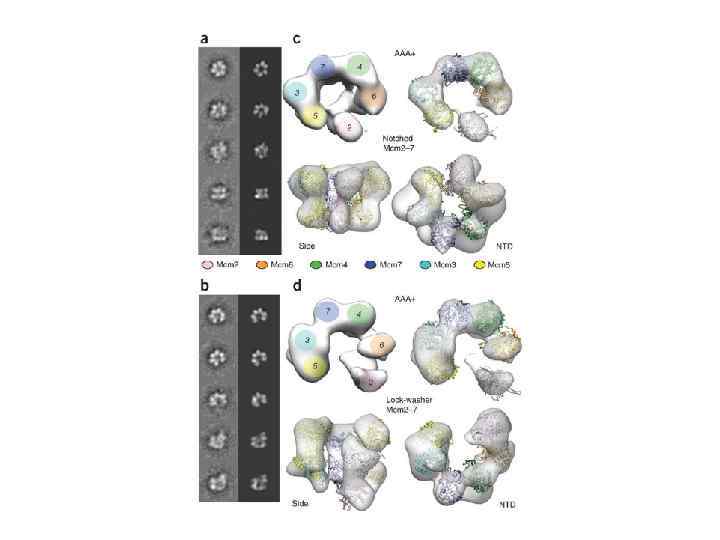
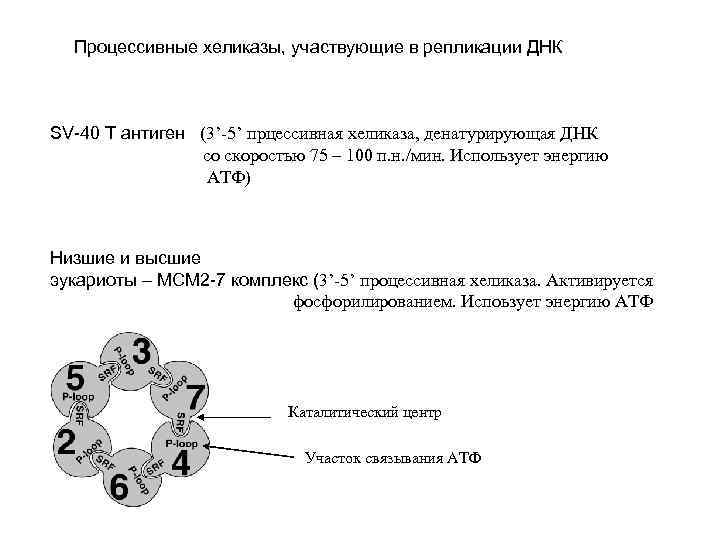 Процессивные хеликазы, участвующие в репликации ДНК SV-40 T антиген (3’-5’ прцессивная хеликаза, денатурирующая ДНК со скоростью 75 – 100 п. н. /мин. Использует энергию АТФ) Низшие и высшие эукариоты – MCM 2 -7 комплекс (3’-5’ процессивная хеликаза. Активируется фосфорилированием. Испоьзует энергию АТФ Каталитический центр Участок связывания АТФ
Процессивные хеликазы, участвующие в репликации ДНК SV-40 T антиген (3’-5’ прцессивная хеликаза, денатурирующая ДНК со скоростью 75 – 100 п. н. /мин. Использует энергию АТФ) Низшие и высшие эукариоты – MCM 2 -7 комплекс (3’-5’ процессивная хеликаза. Активируется фосфорилированием. Испоьзует энергию АТФ Каталитический центр Участок связывания АТФ

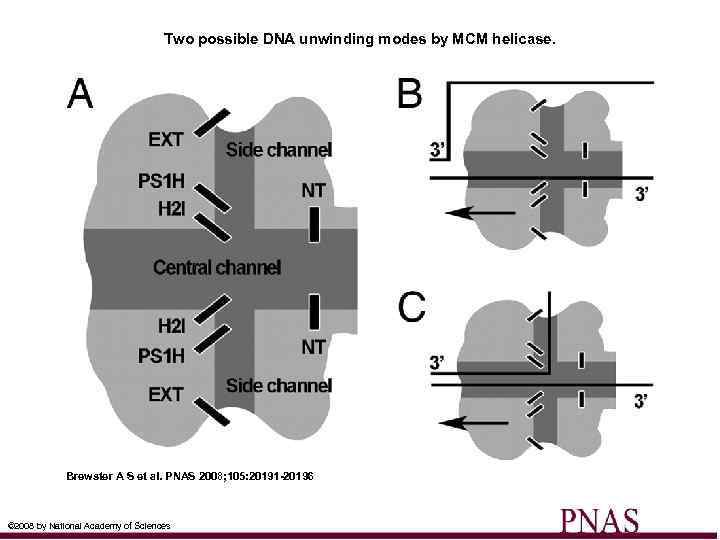 Two possible DNA unwinding modes by MCM helicase. Brewster A S et al. PNAS 2008; 105: 20191 -20196 © 2008 by National Academy of Sciences
Two possible DNA unwinding modes by MCM helicase. Brewster A S et al. PNAS 2008; 105: 20191 -20196 © 2008 by National Academy of Sciences
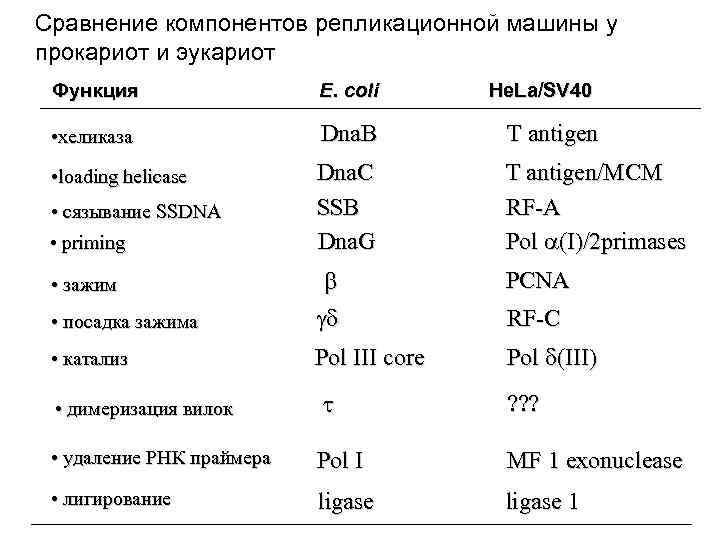 Сравнение компонентов репликационной машины у прокариот и эукариот Функция E. coli He. La/SV 40 • хеликаза Dna. B T antigen • loading helicase Dna. C T antigen/MCM • сязывание SSDNA SSB RF-A • priming Dna. G Pol (I)/2 primases • зажим b PCNA • посадка зажима gd RF-C • катализ Pol III core Pol d(III) • димеризация вилок t ? ? ? • удаление РНК праймера Pol I MF 1 exonuclease • лигирование ligase 1
Сравнение компонентов репликационной машины у прокариот и эукариот Функция E. coli He. La/SV 40 • хеликаза Dna. B T antigen • loading helicase Dna. C T antigen/MCM • сязывание SSDNA SSB RF-A • priming Dna. G Pol (I)/2 primases • зажим b PCNA • посадка зажима gd RF-C • катализ Pol III core Pol d(III) • димеризация вилок t ? ? ? • удаление РНК праймера Pol I MF 1 exonuclease • лигирование ligase 1

 Проблемы, которые решаются в репликационной вилке Проблема Решение ATP? нет однонитевой матрицы хеликаза + однонитевая матрица нестабильна SSB (RPA (euks)) - нет праймера праймаза (+) нет 3’-->5’ полимеразы репликационная вилка низкая процессивность DNA pol скользящий зажим - надо посадить зажим на ДНК установщих зажима ( /RFC) + отстающая цепь содержит Pol I 5’-->3’ exo, RNAse - фрагменты РНК разрывы в отстающей цепи DNA ligase + работа хеликазы генерирует ДНК топоизомеразы +/- супервитки
Проблемы, которые решаются в репликационной вилке Проблема Решение ATP? нет однонитевой матрицы хеликаза + однонитевая матрица нестабильна SSB (RPA (euks)) - нет праймера праймаза (+) нет 3’-->5’ полимеразы репликационная вилка низкая процессивность DNA pol скользящий зажим - надо посадить зажим на ДНК установщих зажима ( /RFC) + отстающая цепь содержит Pol I 5’-->3’ exo, RNAse - фрагменты РНК разрывы в отстающей цепи DNA ligase + работа хеликазы генерирует ДНК топоизомеразы +/- супервитки
 Геном человека кодирует по меньшей мере 15 ДНК полимераз, которые по структурной гомологии можно подразделить на 4 группы: A (Pols γ, ν and θ (Theta)) B (Pols α, δ, ε and ζ) X (Pols β, λ, μ, and terminal deoxyribonucleotidyltransferase) Y (Pols η (eta), ι, κ (σ), and Rev 1) γ репликация митохондриальной ДНК θ (Theta) репликация через апуриновые и апиримидиновые сайты – включение G/C ζ (zeta) репликация через различные повреждения λ, μ участвуют в процессинге концов ДНК в ходе NHEJ η репликация через тимидиновые димеры и G-G димеры, сшитые цис-платином κ (Trf 4/Pol σ ) необходима для соединения (когезии) хроматид
Геном человека кодирует по меньшей мере 15 ДНК полимераз, которые по структурной гомологии можно подразделить на 4 группы: A (Pols γ, ν and θ (Theta)) B (Pols α, δ, ε and ζ) X (Pols β, λ, μ, and terminal deoxyribonucleotidyltransferase) Y (Pols η (eta), ι, κ (σ), and Rev 1) γ репликация митохондриальной ДНК θ (Theta) репликация через апуриновые и апиримидиновые сайты – включение G/C ζ (zeta) репликация через различные повреждения λ, μ участвуют в процессинге концов ДНК в ходе NHEJ η репликация через тимидиновые димеры и G-G димеры, сшитые цис-платином κ (Trf 4/Pol σ ) необходима для соединения (когезии) хроматид


