dbdf869074eee9d34ae5b4d2c1f67639.ppt
- Количество слайдов: 61

Распознавание регуляторных сигналов Факультет биоинженерии и биоинформатики МГУ, второй курс. Май 2004 М. Гельфанд (лекции) Д. Равчеев (задания) А. Герасимова, Э. Пермина (занятия) В. Ю. Макеев (некоторые слайды)
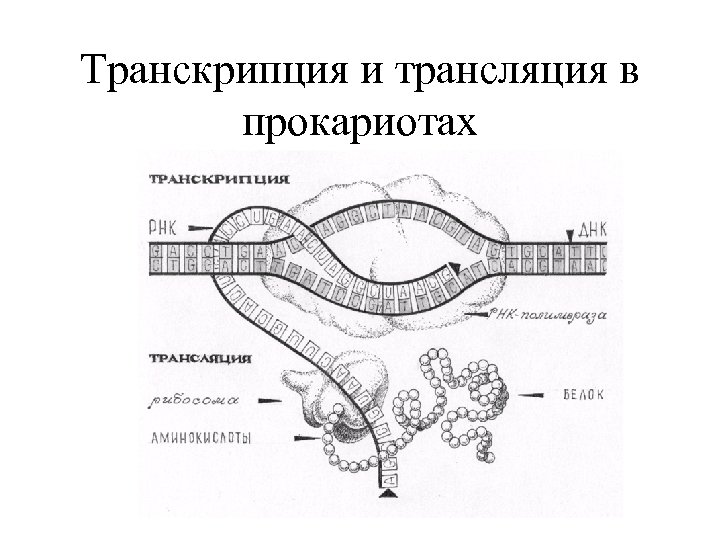
Транскрипция и трансляция в прокариотах
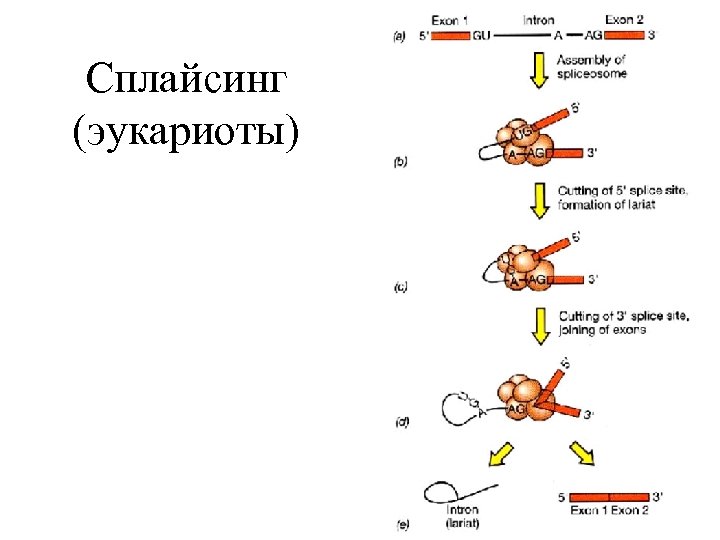
Сплайсинг (эукариоты)
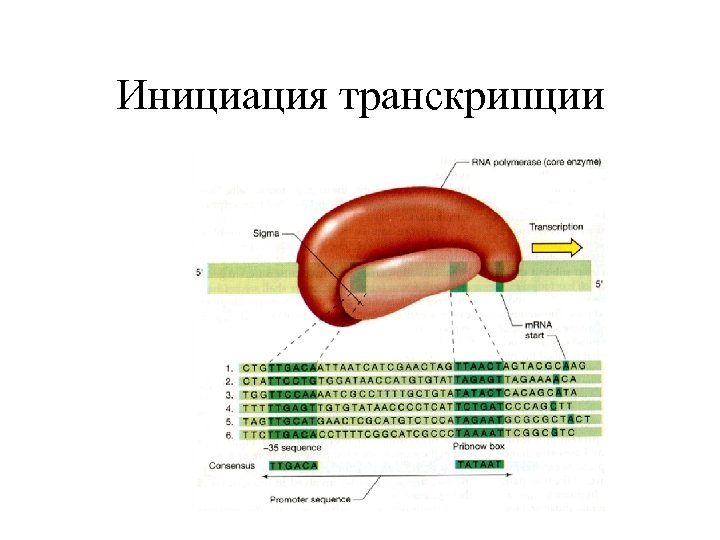
Инициация транскрипции
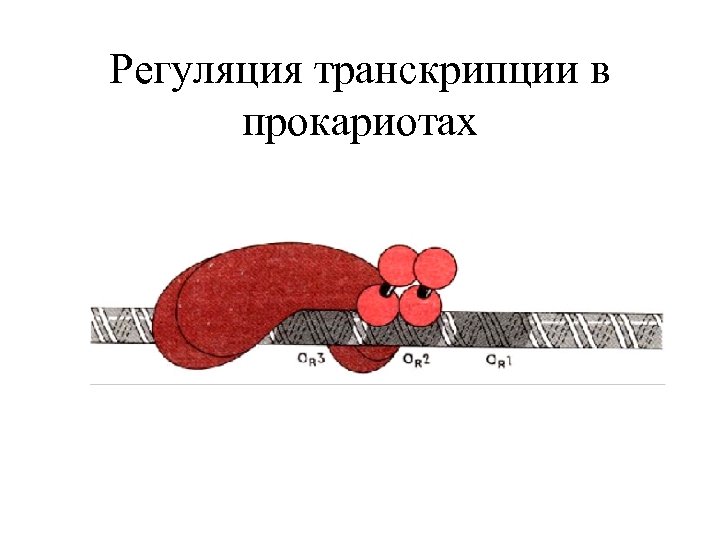
Регуляция транскрипции в прокариотах
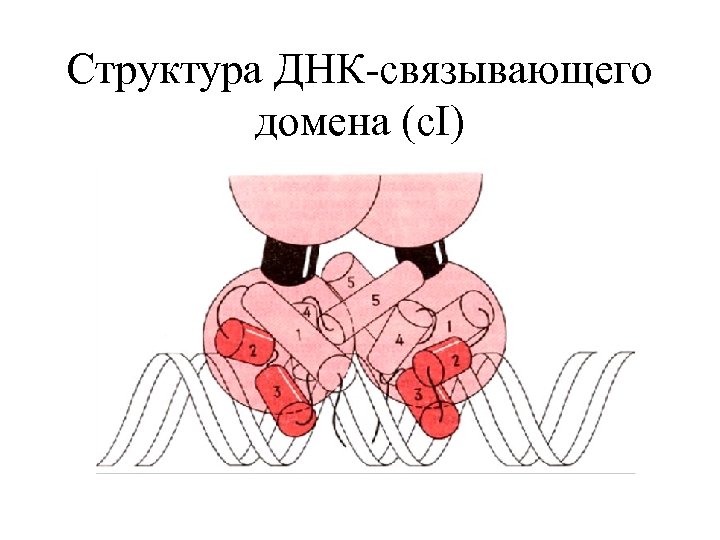
Структура ДНК-связывающего домена (c. I)
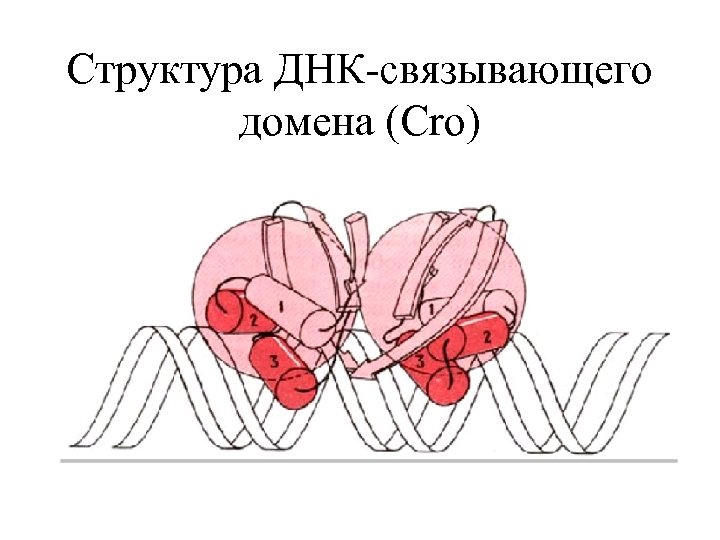
Структура ДНК-связывающего домена (Cro)
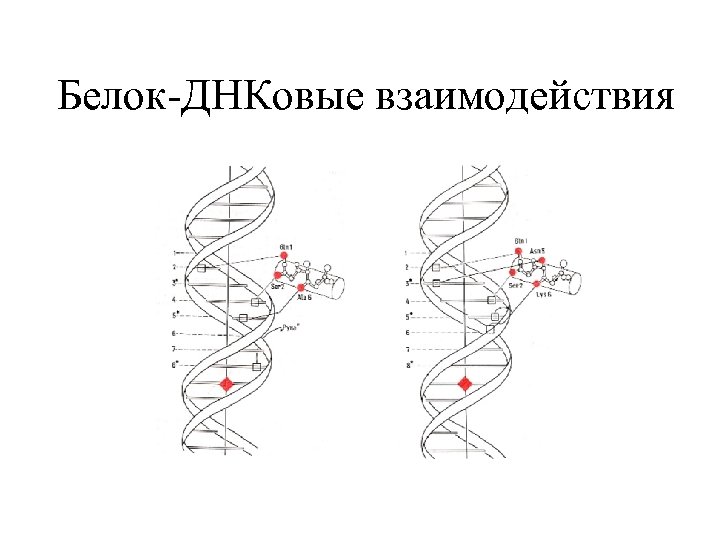
Белок-ДНКовые взаимодействия
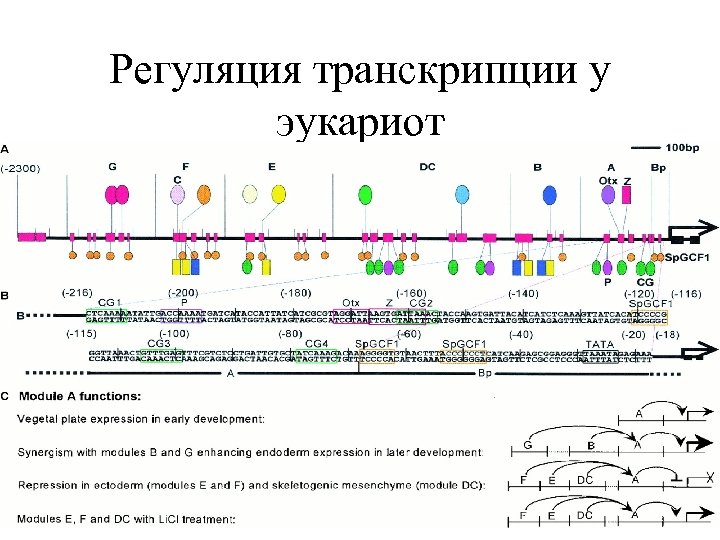
Регуляция транскрипции у эукариот
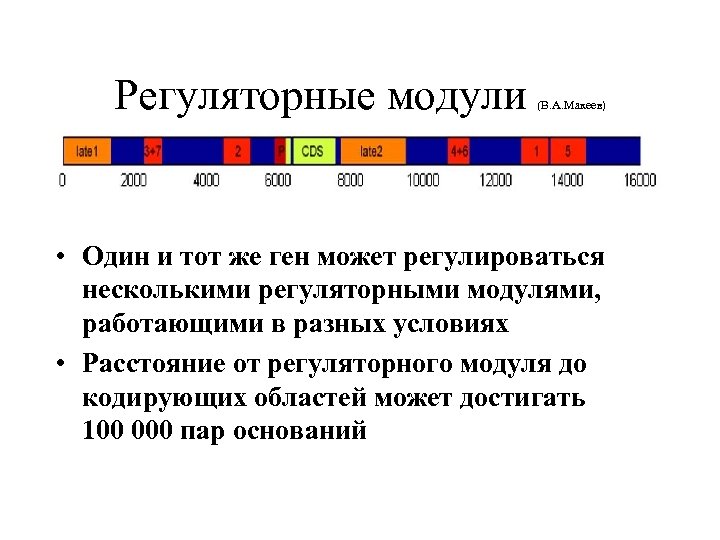
Регуляторные модули (В. А. Макеев) • Один и тот же ген может регулироваться несколькими регуляторными модулями, работающими в разных условиях • Расстояние от регуляторного модуля до кодирующих областей может достигать 100 000 пар оснований

Представление сигналов • Консенсус • Pattern ( «образец» - консенсус с вырoжденными позициями) • Позиционная весовая матрица (или профиль) positional weight matrix, PWM, profile • Логические правила • РНКовые сигналы – вторичная структура
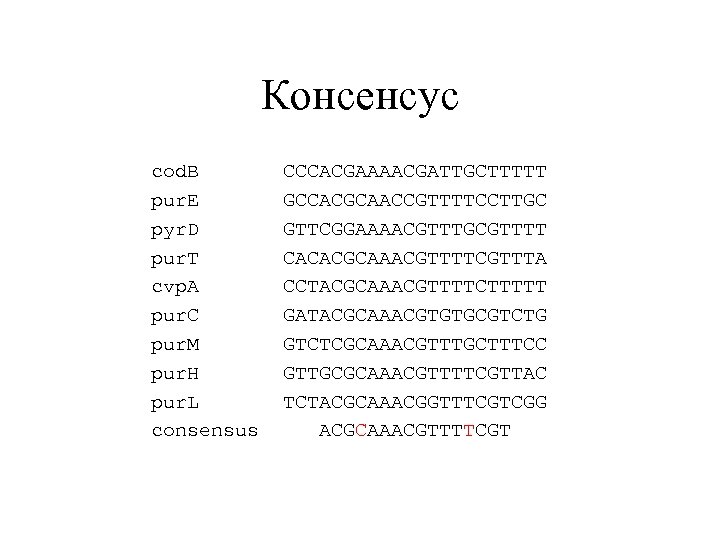
Консенсус cod. B CCCACGAAAACGATTGCTTTTT pur. E GCCACGCAACCGTTTTCCTTGC pyr. D GTTCGGAAAACGTTTGCGTTTT pur. T CACACGCAAACGTTTTCGTTTA cvp. A CCTACGCAAACGTTTTCTTTTT pur. C GATACGCAAACGTGTGCGTCTG pur. M GTCTCGCAAACGTTTGCTTTCC pur. H GTTGCGCAAACGTTTTCGTTAC pur. L TCTACGCAAACGGTTTCGTCGG consensus ACGCAAACGTTTTCGT

Образец cod. B CCCACGAAAACGATTGCTTTTT pur. E GCCACGCAACCGTTTTCCTTGC pyr. D GTTCGGAAAACGTTTGCGTTTT pur. T CACACGCAAACGTTTTCGTTTA cvp. A CCTACGCAAACGTTTTCTTTTT pur. C GATACGCAAACGTGTGCGTCTG pur. M GTCTCGCAAACGTTTGCTTTCC pur. H GTTGCGCAAACGTTTTCGTTAC pur. L TCTACGCAAACGGTTTCGTCGG consensus ACGCAAACGTTTTCGT pattern a. CGm. AAACGt. TTk. Ck. T
![Матрица частот Информационное содержание I = j b f(b, j)[log f(b, j) / p(b)] Матрица частот Информационное содержание I = j b f(b, j)[log f(b, j) / p(b)]](https://present5.com/presentation/dbdf869074eee9d34ae5b4d2c1f67639/image-14.jpg)
Матрица частот Информационное содержание I = j b f(b, j)[log f(b, j) / p(b)]
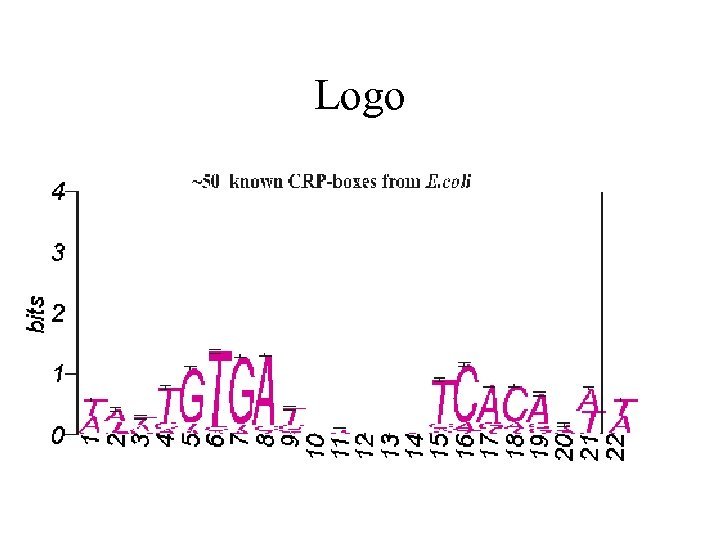
Logo

Позиционная весовая матрица (профиль)

• Вероятностная мотивировка: лог-правдоподобие (с точностью до линейного преобразования) • Ещё одна: z-score (при сообтветствующем основании логарифма) • Термодинамическая мотивировка: свободная энергия (в предположении независимости соседних позиций) • Псевдоотсчеты (pseudocounts)
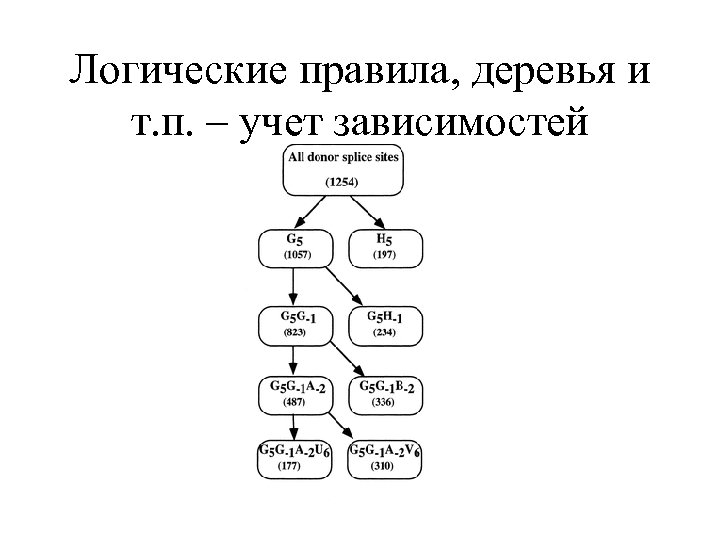
Логические правила, деревья и т. п. – учет зависимостей

Составление выборки • Начало: – – Gen. Bank специализированные банки данных литература (общоры) литература (оригинальные статьи) • Исправление ошибок • Проверка литературных данных • предсказанные сайты. • Удаление дубликатов

Перевыравнивание • Первоначальное выравнивание по биологическим признакам – промоторы: старт транскрипции – участки связывания рибосом: стартовый кодон – сайты сплайсинга: экзон-интронные границы • Выделение сигнала в скользящем окне • Перевыравание • и т. д. пока не сойдётся
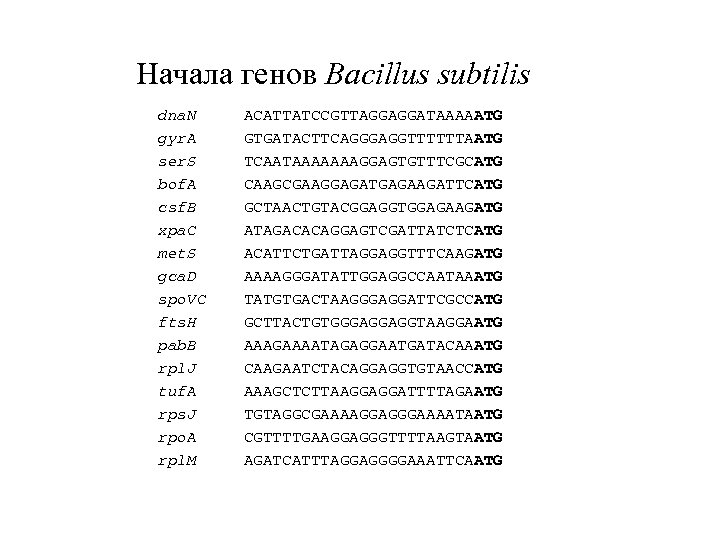
Начала генов Bacillus subtilis dna. N ACATTATCCGTTAGGAGGATAAAAATG gyr. A GTGATACTTCAGGGAGGTTTTTTAATG ser. S TCAATAAAAAAAGGAGTGTTTCGCATG bof. A CAAGCGAAGGAGATGAGAAGATTCATG csf. B GCTAACTGTACGGAGGTGGAGAAGATG xpa. C ATAGACACAGGAGTCGATTATCTCATG met. S ACATTCTGATTAGGAGGTTTCAAGATG gca. D AAAAGGGATATTGGAGGCCAATAAATG spo. VC TATGTGACTAAGGGAGGATTCGCCATG fts. H GCTTACTGTGGGAGGAGGTAAGGAATG pab. B AAAGAAAATAGAGGAATGATACAAATG rpl. J CAAGAATCTACAGGAGGTGTAACCATG tuf. A AAAGCTCTTAAGGAGGATTTTAGAATG rps. J TGTAGGCGAAAAGGAGGGAAAATAATG rpo. A CGTTTTGAAGGAGGGTTTTAAGTAATG rpl. M AGATCATTTAGGAGGGGAAATTCAATG

dna. N ACATTATCCGTTAGGAGGATAAAAATG gyr. A GTGATACTTCAGGGAGGTTTTTTAATG ser. S TCAATAAAAAAAGGAGTGTTTCGCATG bof. A CAAGCGAAGGAGATGAGAAGATTCATG csf. B GCTAACTGTACGGAGGTGGAGAAGATG xpa. C ATAGACACAGGAGTCGATTATCTCATG met. S ACATTCTGATTAGGAGGTTTCAAGATG gca. D AAAAGGGATATTGGAGGCCAATAAATG spo. VC TATGTGACTAAGGGAGGATTCGCCATG fts. H GCTTACTGTGGGAGGAGGTAAGGAATG pab. B AAAGAAAATAGAGGAATGATACAAATG rpl. J CAAGAATCTACAGGAGGTGTAACCATG tuf. A AAAGCTCTTAAGGAGGATTTTAGAATG rps. J TGTAGGCGAAAAGGAGGGAAAATAATG rpo. A CGTTTTGAAGGAGGGTTTTAAGTAATG rpl. M AGATCATTTAGGAGGGGAAATTCAATG cons. aaagtatataagggttaata. ATG num. 00100000110110000000111 760666658967228106888659666
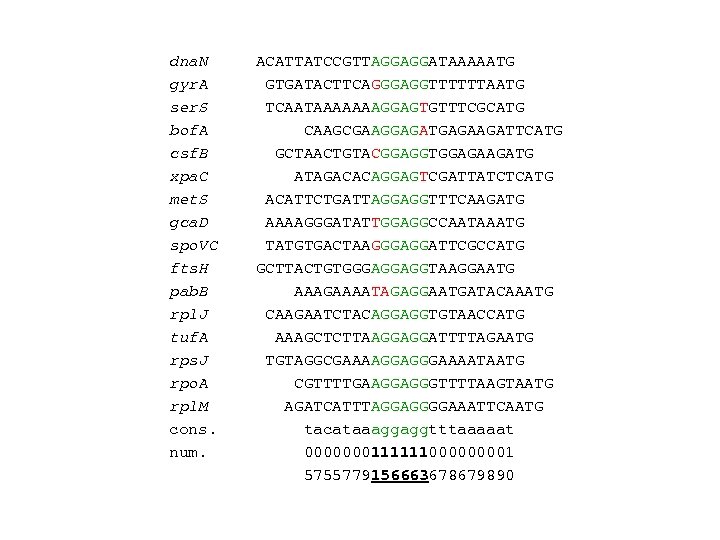
dna. N ACATTATCCGTTAGGAGGATAAAAATG gyr. A GTGATACTTCAGGGAGGTTTTTTAATG ser. S TCAATAAAAAAAGGAGTGTTTCGCATG bof. A CAAGCGAAGGAGATGAGAAGATTCATG csf. B GCTAACTGTACGGAGGTGGAGAAGATG xpa. C ATAGACACAGGAGTCGATTATCTCATG met. S ACATTCTGATTAGGAGGTTTCAAGATG gca. D AAAAGGGATATTGGAGGCCAATAAATG spo. VC TATGTGACTAAGGGAGGATTCGCCATG fts. H GCTTACTGTGGGAGGAGGTAAGGAATG pab. B AAAGAAAATAGAGGAATGATACAAATG rpl. J CAAGAATCTACAGGAGGTGTAACCATG tuf. A AAAGCTCTTAAGGAGGATTTTAGAATG rps. J TGTAGGCGAAAAGGAGGGAAAATAATG rpo. A CGTTTTGAAGGAGGGTTTTAAGTAATG rpl. M AGATCATTTAGGAGGGGAAATTCAATG cons. tacataaaggaggtttaaaaat num. 000000011111100001 5755779156663678679890
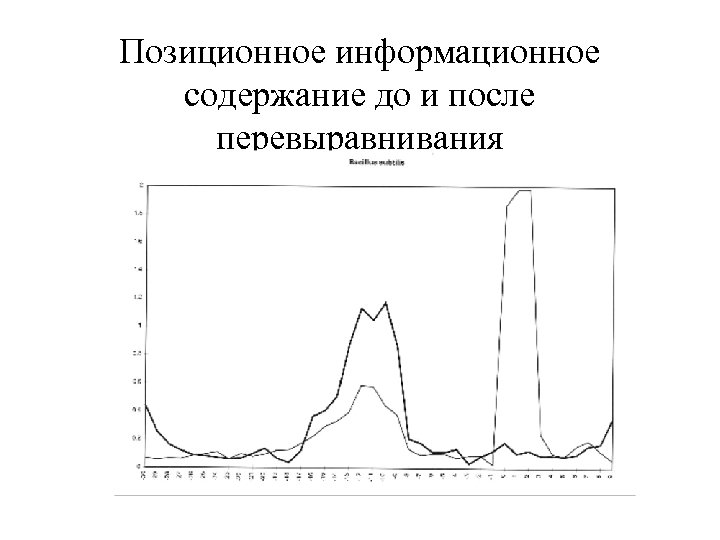
Позиционное информационное содержание до и после перевыравнивания
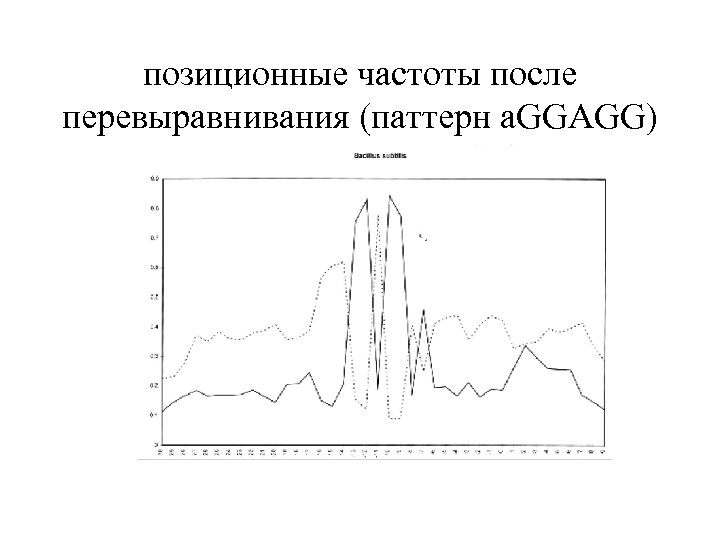
позиционные частоты после перевыравнивания (паттерн a. GGAGG)

Поиск сигнала с самого начала (ab initio) • “дискретные” подходы: считать слова и образцы • “непрерывные” подходы: оптимизация профиля

Как считать короткие слова • Рассмотрим все слова длины k (k-меры) • Для каждого k-мера вычислим количество последовательностей, которые его содержат – (не обязательно в точности) • Выберем самый частый k-мер

Проблема: Полный перебор возможен только для относительно коротких слов Предположение: если длинное слово встречается часто, его подслова тоже будут часто встречаться Решение: выбрать набор частых коротких слов и склеить в длинное

Как считать длинные слова • Рассмотрим некоторые k-меры • Для каждого k-мера вычислим количество последовательностей, которые его содержат – (не обязательно в точности) • Выберем самый частый k-мер

Проблема: «некоторые» k-меры - это какие? 1 я попытка: те, которые встречаются в выборке Но: сигнал (консенсусный k-мер) может и не встретиться.

2 я попытка: те, которые встречаются в выборке и похожие на них. Но: – опять же, сигнал может и не попасть в это множество; – а размер множества «похожих» слов растёт экспоненциально

Теоретико-графовый подход Каждый k-мер в каждой последовательности соответствует вершине. Два k-мера соединены ребром, если они похожи (например, отличаются не более, чем в h позиций, h<<k). Получается n-дольный граф (n – количество последовательностей). Сигнал соответствует клике (полному подграфу) – или по крайней мере плотному графу – с вершинами в каждой доле

Простой алгоритм • Удалить все вершины, которые не могут быть продолжены до полных графов – то есть, не имеют ребер во все доли • Из списка пар удалить все, которые … – то есть не образуют треугольники с третьими вершинами во всех долях • И т. д. . (не будет в такой форме работать для поиска плотных подграфов)

Оптимизация. Expectation - Maximization • Породим начальное множество профилей (например, каждый из имеющихся k-меров породит один профиль) • Для каждого профиля: – найти наилучшего представителя в каждой последовательности – обновить профиль • Повторять пока не сойдётся

Этот алгоритм сходится, но не может покинуть область локального максимума. Поэтому если начальное приближение было плохим, он сойдётся к ерунде. Решение: стохастическая оптимизация.

Имитация теплового отжига • Цель: максимизировать информационное содержание I I = j b f(b, j)[log f(b, j) / p(b)] • или любой другой функционал, измеряющий однородность множества сайтов

Алгоритм Обозначим: A – текущий сигнал (множество потенциальных сайтов), I(A) – его информационное содержание. B – сигнал, отличающийся от А выбором сайта в одной последовательности, I(B) – го информационное содержание. • если I(B) I(A), B принимается • если I(B) < I(A), B принимается с вероятностью P = exp [(I(B) – I(A)) / T] Температура T медленно снижается, первоначально она такова, что почти все изменения принимаются (Р близко к 1).

Gibbs sampler Опять, A – сигнал, I(A) – его информационное содержание. На каждом шаге в одной последовательности выбирается новый сайт с вероятностью P ~ exp [(I(Anew)] Для каждого потенциального сайта подсчитывается, сколько раз он был выбран. (Замечание: сигнал всё время меняется)
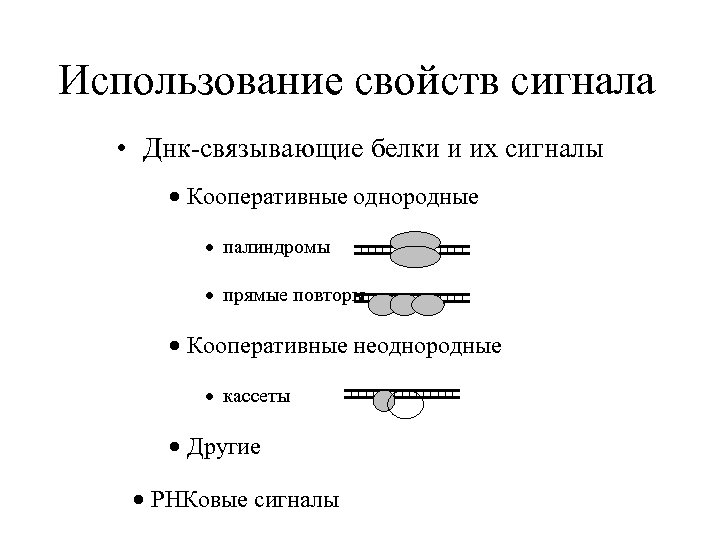
Использование свойств сигнала • Днк-связывающие белки и их сигналы · Кооперативные однородные · палиндромы · прямые повторы · Кооперативные неоднородные · кассеты · Другие · РНКовые сигналы

Распознавание: весовые матрицы (профили) Позиционные веса нуклеодтидов W(b, j)=ln(N(b, j)+0. 5) – 0. 25 iln(N(i, j)+0. 5) Вес потенциального сайта b 1…bk – это сумма соответствующих позиционных весов: S(b 1…bk ) = j=1, …, k. W(bj, j)
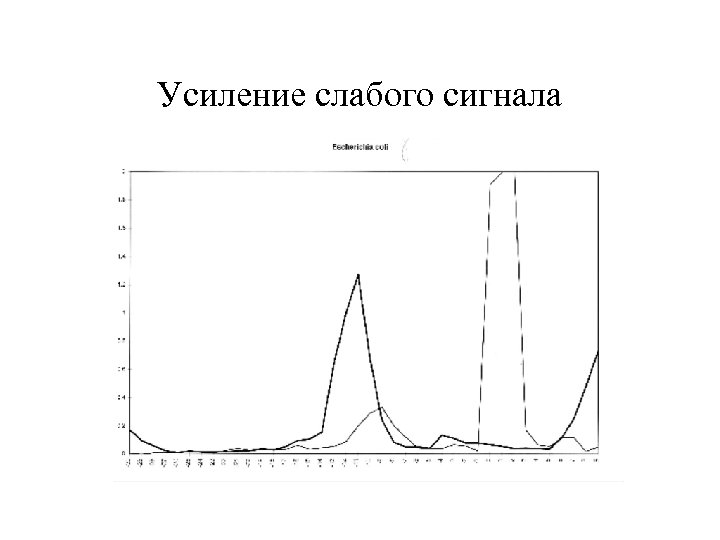
Усиление слабого сигнала
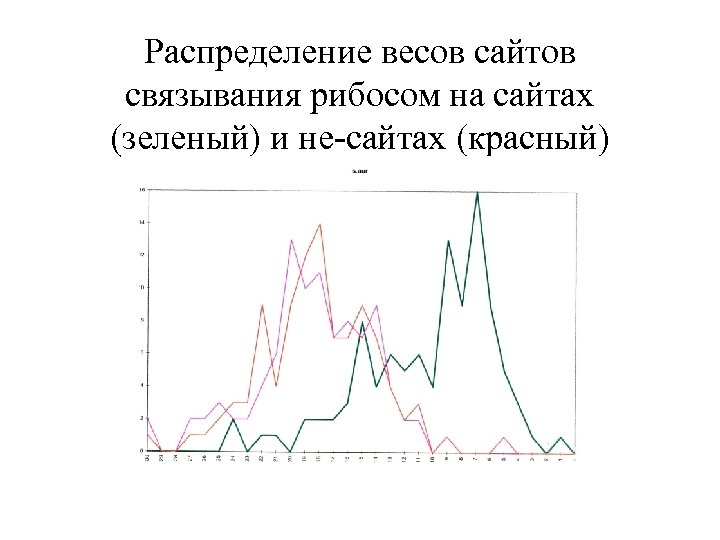
Распределение весов сайтов связывания рибосом на сайтах (зеленый) и не-сайтах (красный)

Нейронные сети: архитектура • 4 k входных нейронов (сенсоров), присутствие конкретного нуклеотида в конкретной позиции (да/нет) или 2 k нейронов (пурин/пиримидин, AT/GC) • один или более слоёв внутренних нейронов • один выходной нейрон (сайт/не-сайт)

• каждый нейрон связан соединениями с нейронами соседнего уровня • каждому соединению приписан вес Нейрон: • суммирует (с весами) входящие сигналы • сравнивает результат с порогом (или преобразует по заданному правилу) • если сумма выше порога, отправляет сигнал всем нейронам следующего уровня (или просто преобразованное значение)

Обучение: • Обработать сайты и не-сайты из обучающей выборки одни за одним, несколько полных итераций. • Для каждого объекта сделать предсказание. • Если оно неправильное, изменить веса. Сети отличаются архитектурой, способом обработки сигнала, расписанием обучения

Оценка качества алгоритмов Чувствительность: правильно предсказанные / все правильные Специфичность: правильно предсказанные / все предсказанные • Трудно составить тестирующую выборку: – неизвестные сайты – активация в определенных условиях – неспецифическое связывание

Промоторы E. coli • профиль, предсказывающий 1 сайт на 2000 нт, имеет чувствительность: – 25% на всех промоторах, – 60% на конститутивных (неактивируемых) промоторах
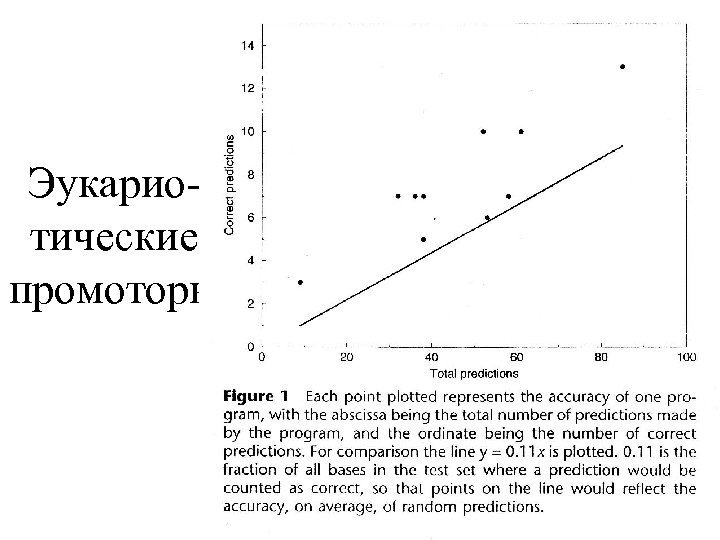
Эукариотические промоторы
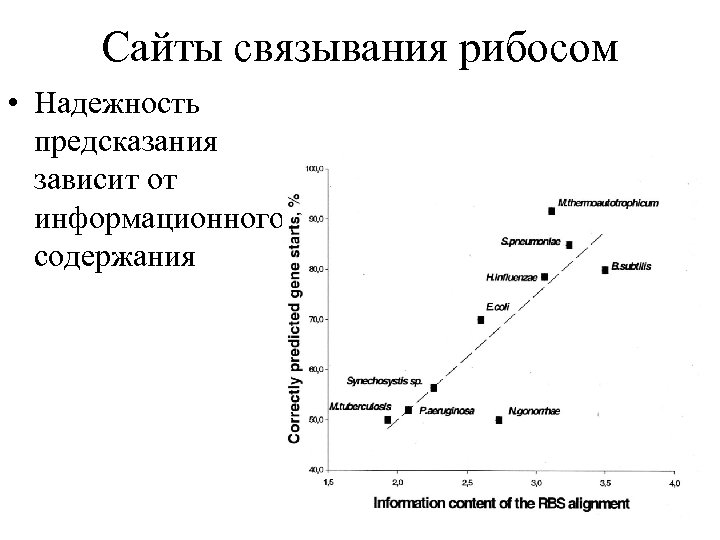
Сайты связывания рибосом • Надежность предсказания зависит от информационного содержания
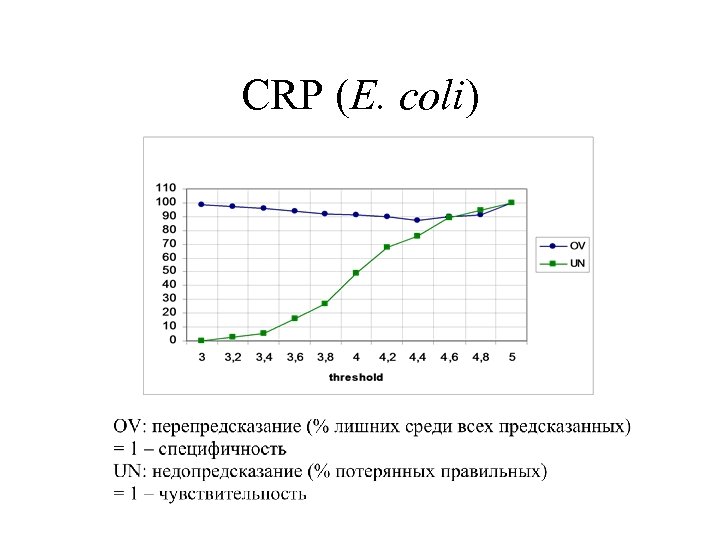
CRP (E. coli)

Запись Gen. Bank для гена E. coli

Что же делать? • филогенетическое картирование: правильные сайты консервативны

Консервативная область

Менее консервативная область

rbs. D в энтеробактериях: ответ Sty AGGGTTACACTGCGGC-CAGCGAAACGTTTCGCTAGTGGAGCAGAAAAATGAAGAAAGGC Sen AGGGTTACACTGCGGC-CAGCGAAACGTTTCGCTAGTGGAGCAGAAAAATGAAGAAAGGC Stm GGGGTTACACTGCGGC-CAGCGAAACGTTTCGCTAGTGGAGCAGAAAAATGAAGAAAGGC Eco AGGATTAAACTGTGGGTCAGCGAAACGTTTCGCTGATGGAGAA-AAAAATGAAAAAAGGC Ype TTTTCTAAACTCCTTGTTAGCGAAACGTTTCGCTCTTGGAGTA-GATCATGAAAAAAGGT ** ******** * * ***** Sty ACCGTACTCAACTCTGAAATCTCGTCGGTCATTTCCCGTCTGGGGCATACTGATACTCTG Sen ACCGTACTCAACTCTGAAATCTCGTCGGTCATTTCCCGTCTGGGGCATACTGATACTCTG Stm ACCGTACTCAACTCTGAAATCTCGTCGGTCATTTCCCGTCTGGGGCATACTGATACTCTG Eco ACCGTTCTTAATTCTGATATTTCATCGGTGATCTCCCGTCTGGGACATACCGATACGCTG Ype GTATTACTGAACGCTGATATTTCCGCGGTTATCTCCCGTCTGGGCCATACCGATCAGATT * ** ** ****** ***** *
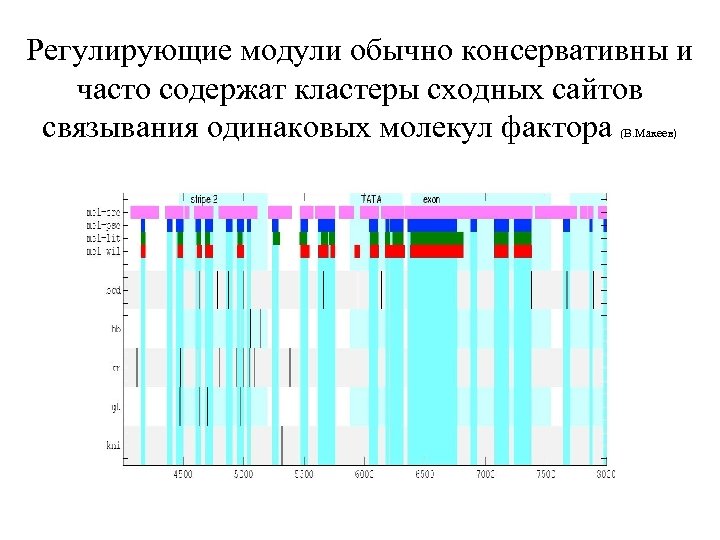
Регулирующие модули обычно консервативны и часто содержат кластеры сходных сайтов связывания одинаковых молекул фактора (В. Макеев)
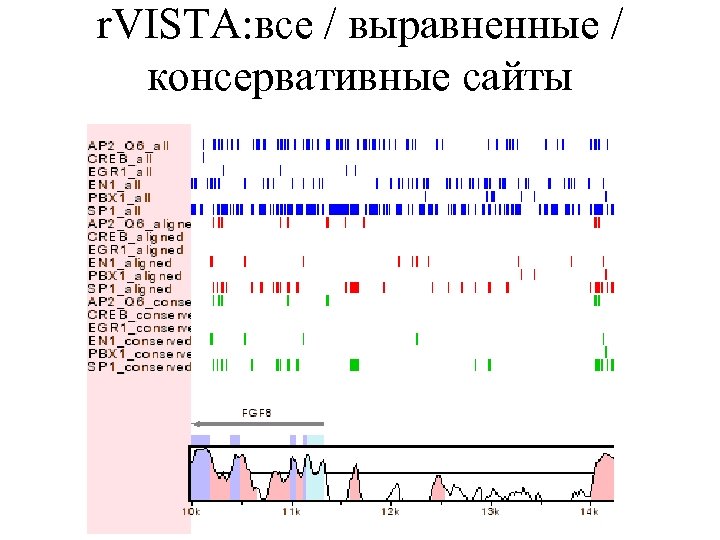
r. VISTA: все / выравненные / консервативные сайты

ура!
dbdf869074eee9d34ae5b4d2c1f67639.ppt