Базы-проверки мутаций.ppt
- Количество слайдов: 46
 Программы для проверки патогенности мутации: Poly. Phen-2, SIFT+Provean, Mutation taster. Прошлякова Татьяна Юрьевна
Программы для проверки патогенности мутации: Poly. Phen-2, SIFT+Provean, Mutation taster. Прошлякова Татьяна Юрьевна
 Poly. Phen-2
Poly. Phen-2
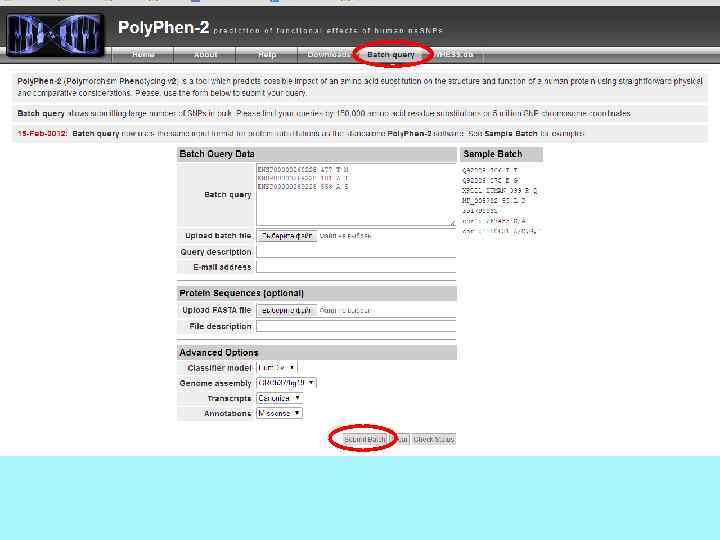
 Poly. Phen-2 (от слов Polymorphism «полиморфизм» и Phenotyping «фенотипирование» ) - программа, которая прогнозирует возможное влияние аминокислотной замены на структуру и функцию белка. Poly. Phen-2 определяет гомологов сходных последовательностей с помощью поиска BLAST в базе Uni. Ref 100. Выравнивание с запрашиваемой последовательностью производится не менее чем с 75 аминокислотами в ряду. Две пары наборов данных были использованы для тестирования и прогнозирования в Poly. Phen-2. • Первая пара Hum. Div, была составлена из всех повреждающих аллелей с известными воздействиями на функцию, включая заболевания, присутствующие в базе данных Uni. Prot. KB, вместе с различиями между белками человека и их близкородственных гомологов млекопитающих. • Вторая пара, Hum. Var, составлена из всех патогенных мутаций человека из базы Uni. Prot. KB, вместе с человеческими SNP (с частотой > 1%). Во вкладке «About» можно найти 4 статьи для более детального изучения.
Poly. Phen-2 (от слов Polymorphism «полиморфизм» и Phenotyping «фенотипирование» ) - программа, которая прогнозирует возможное влияние аминокислотной замены на структуру и функцию белка. Poly. Phen-2 определяет гомологов сходных последовательностей с помощью поиска BLAST в базе Uni. Ref 100. Выравнивание с запрашиваемой последовательностью производится не менее чем с 75 аминокислотами в ряду. Две пары наборов данных были использованы для тестирования и прогнозирования в Poly. Phen-2. • Первая пара Hum. Div, была составлена из всех повреждающих аллелей с известными воздействиями на функцию, включая заболевания, присутствующие в базе данных Uni. Prot. KB, вместе с различиями между белками человека и их близкородственных гомологов млекопитающих. • Вторая пара, Hum. Var, составлена из всех патогенных мутаций человека из базы Uni. Prot. KB, вместе с человеческими SNP (с частотой > 1%). Во вкладке «About» можно найти 4 статьи для более детального изучения.
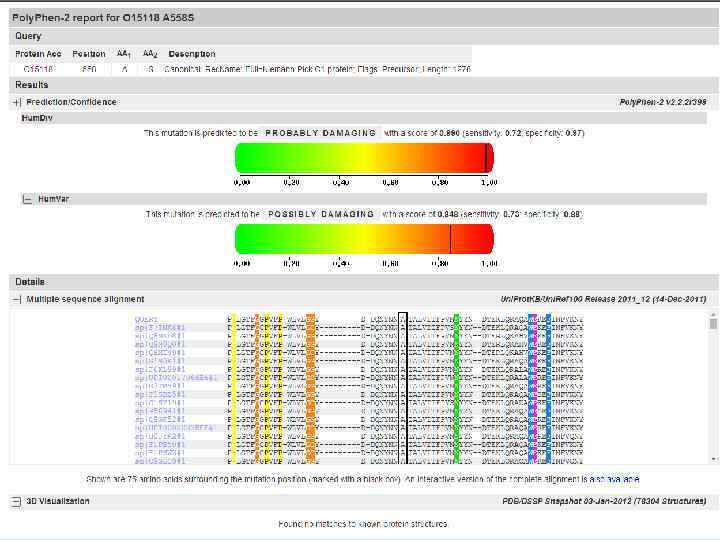
 Prediction - Прогноз Benign – доброкачественная замена Possibly damaging – возможно повреждающая (меньшая уверенность) Probably damaging – вероятно повреждающая (большая уверенность) Unknown – неизвестно *Score – счет Confidence – Уверенность Sensitivity – Чувствительность – это способность диагностического метода давать правильный результат, который определяется как доля истинно положительных результатов среди всех проведенных тестов; Specificity – Специфичность – это способность диагностического метода не давать при отсутствии заболевания ложноположительных результатов, который определяется как доля истинно отрицательных результатов
Prediction - Прогноз Benign – доброкачественная замена Possibly damaging – возможно повреждающая (меньшая уверенность) Probably damaging – вероятно повреждающая (большая уверенность) Unknown – неизвестно *Score – счет Confidence – Уверенность Sensitivity – Чувствительность – это способность диагностического метода давать правильный результат, который определяется как доля истинно положительных результатов среди всех проведенных тестов; Specificity – Специфичность – это способность диагностического метода не давать при отсутствии заболевания ложноположительных результатов, который определяется как доля истинно отрицательных результатов





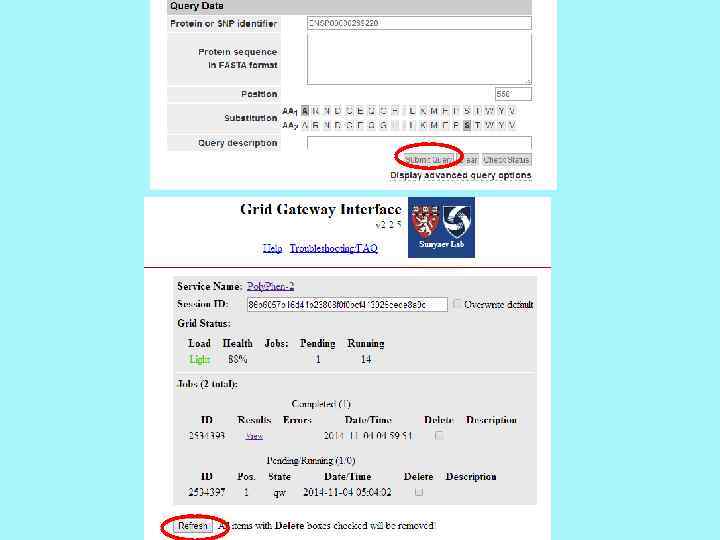
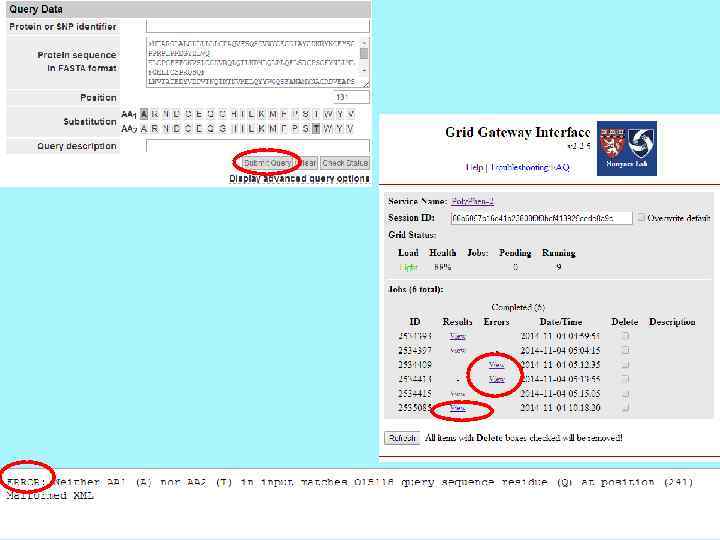
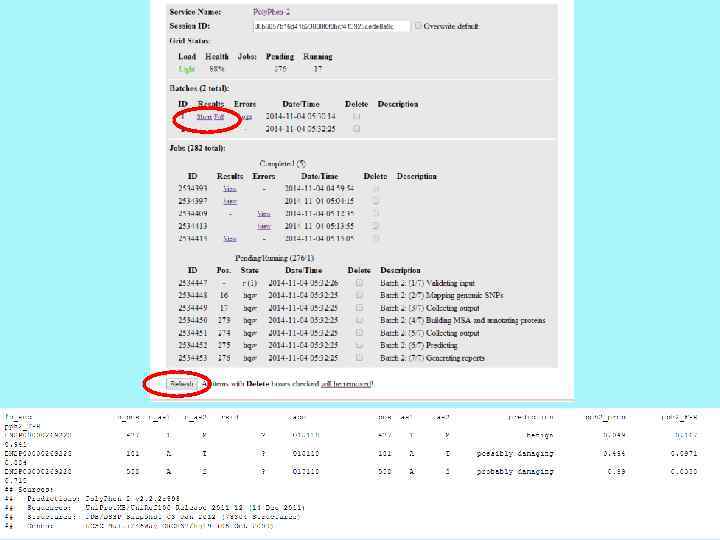
 Load показывает, насколько сильно сетка базы в настоящее время используется, Health отражает процент отлично функциональных узлов в сетке, Jobs – работа- перечислены общие количества всех Pending - ожидание и Running - запуск рабочих мест. В целом, эта информация позволяет получить оценку о том, как долго ваш запрос может занять временя до завершения. Job numeric ID - номер вашей загрузки. Сессии хранятся на сайте 3 месяца, можно раньше удалить самим. Pos. – position - Место в очереди. « 1» означает, что запросы в работу будут направлены немедленно, как только следующий свободный слот CPU будет доступен. State - Состояние работы, один из: qw - для очереди, в ожидании; t - для перевода в одном из узлов сетки для исполнения; или r - для запуска задач. Description – описание – можно писать самому, чтобы потом не путаться Errors- как проблемы сетки, так и неправильного ввода.
Load показывает, насколько сильно сетка базы в настоящее время используется, Health отражает процент отлично функциональных узлов в сетке, Jobs – работа- перечислены общие количества всех Pending - ожидание и Running - запуск рабочих мест. В целом, эта информация позволяет получить оценку о том, как долго ваш запрос может занять временя до завершения. Job numeric ID - номер вашей загрузки. Сессии хранятся на сайте 3 месяца, можно раньше удалить самим. Pos. – position - Место в очереди. « 1» означает, что запросы в работу будут направлены немедленно, как только следующий свободный слот CPU будет доступен. State - Состояние работы, один из: qw - для очереди, в ожидании; t - для перевода в одном из узлов сетки для исполнения; или r - для запуска задач. Description – описание – можно писать самому, чтобы потом не путаться Errors- как проблемы сетки, так и неправильного ввода.



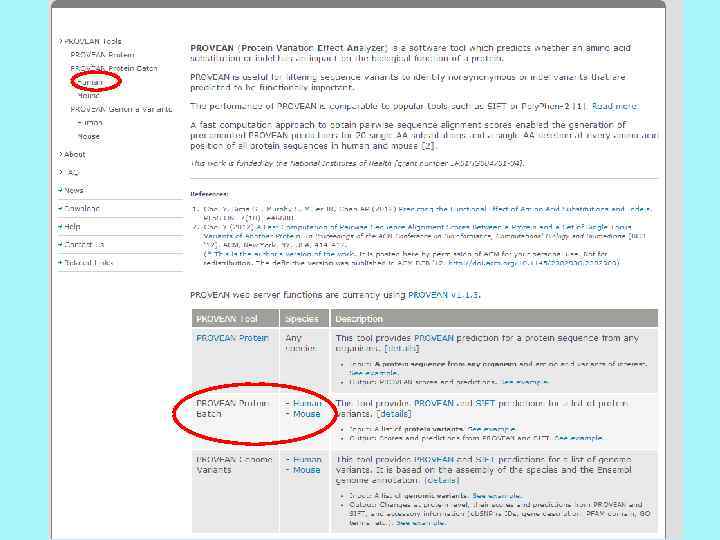
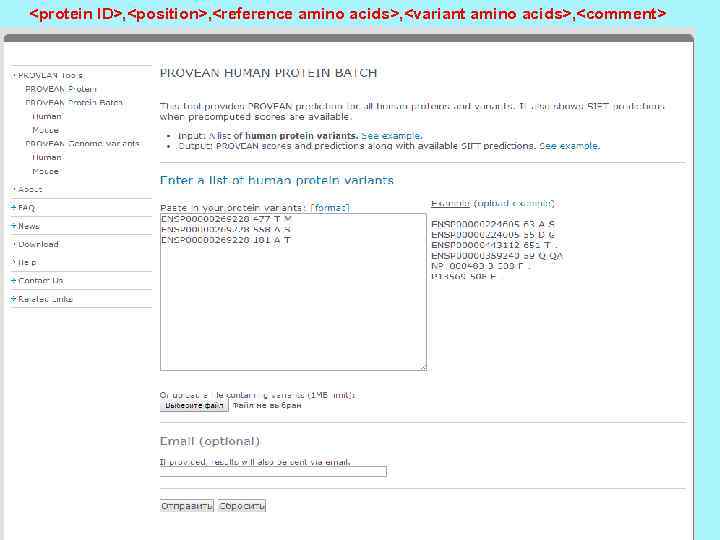
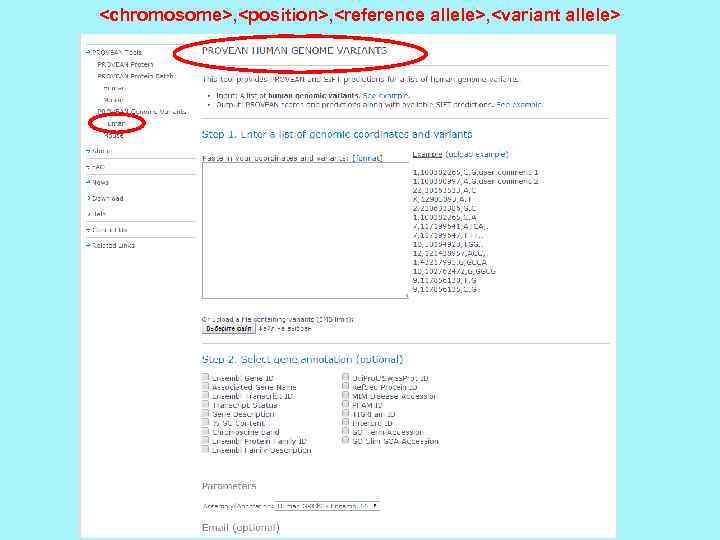
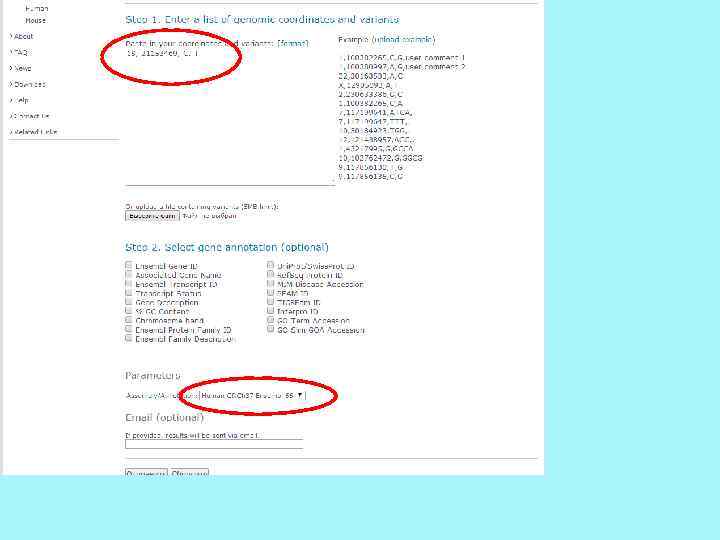
 SIFT+Provean
SIFT+Provean
 SIFT+Provean
SIFT+Provean
 SIFT score TOLERATED – толерантная, допустимая, переносимая, терпимая DAMAGING – повреждающая замена N/A – нет данных/не известно Позиции с вероятностью менее 0, 05 – DAMAGING Позиции больше или равны 0, 05 – TOLERATED
SIFT score TOLERATED – толерантная, допустимая, переносимая, терпимая DAMAGING – повреждающая замена N/A – нет данных/не известно Позиции с вероятностью менее 0, 05 – DAMAGING Позиции больше или равны 0, 05 – TOLERATED
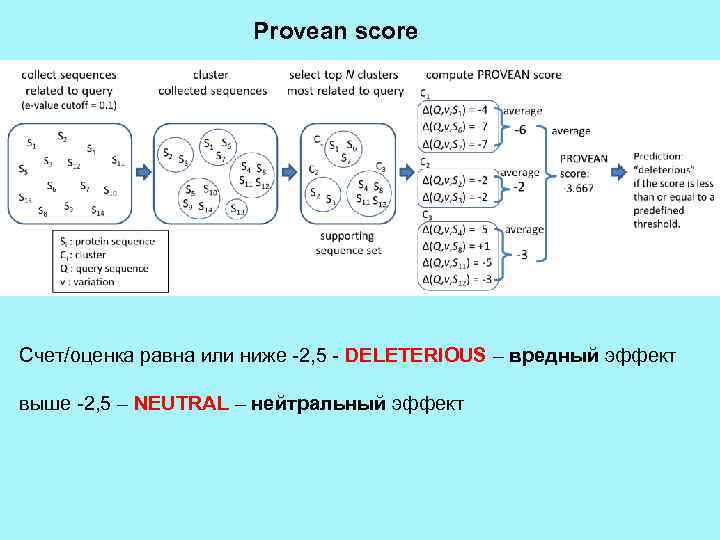 Provean score Счет/оценка равна или ниже -2, 5 - DELETERIOUS – вредный эффект выше -2, 5 – NEUTRAL – нейтральный эффект
Provean score Счет/оценка равна или ниже -2, 5 - DELETERIOUS – вредный эффект выше -2, 5 – NEUTRAL – нейтральный эффект
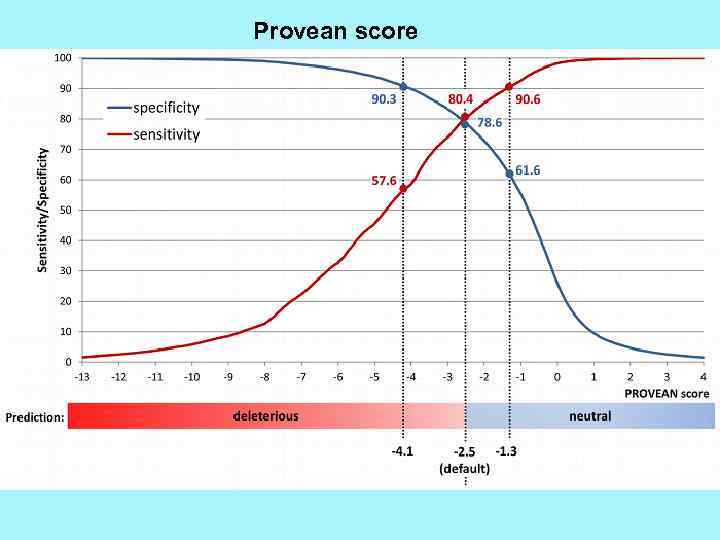 Provean score
Provean score
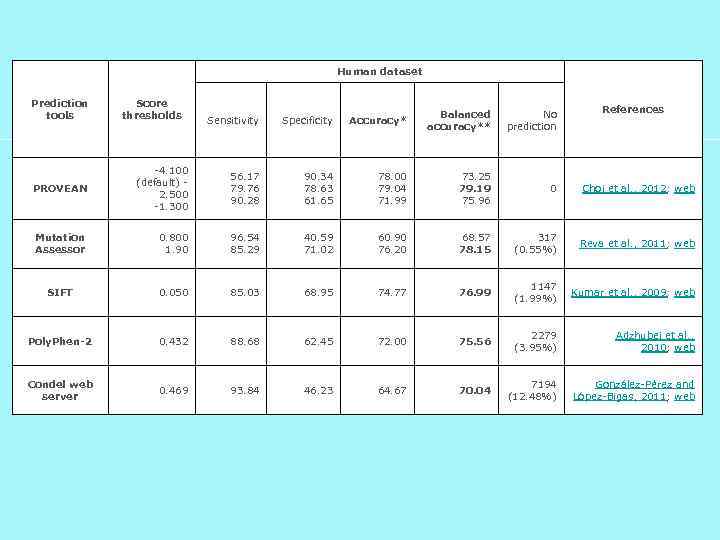 Human dataset Prediction tools Score thresholds No prediction References Sensitivity Specificity Accuracy* Balanced accuracy** PROVEAN -4. 100 (default) 2. 500 -1. 300 56. 17 79. 76 90. 28 90. 34 78. 63 61. 65 78. 00 79. 04 71. 99 73. 25 79. 19 75. 96 0 Choi et al. , 2012; web Mutation Assessor 0. 800 1. 90 96. 54 85. 29 40. 59 71. 02 60. 90 76. 20 68. 57 78. 15 317 (0. 55%) Reva et al. , 2011; web SIFT 0. 050 85. 03 68. 95 74. 77 76. 99 1147 (1. 99%) Kumar et al. , 2009; web Poly. Phen-2 0. 432 88. 68 62. 45 72. 00 75. 56 2279 (3. 95%) Adzhubei et al. , 2010; web Condel web server 0. 469 93. 84 46. 23 64. 67 70. 04 7194 (12. 48%) González-Pérez and López-Bigas, 2011; web
Human dataset Prediction tools Score thresholds No prediction References Sensitivity Specificity Accuracy* Balanced accuracy** PROVEAN -4. 100 (default) 2. 500 -1. 300 56. 17 79. 76 90. 28 90. 34 78. 63 61. 65 78. 00 79. 04 71. 99 73. 25 79. 19 75. 96 0 Choi et al. , 2012; web Mutation Assessor 0. 800 1. 90 96. 54 85. 29 40. 59 71. 02 60. 90 76. 20 68. 57 78. 15 317 (0. 55%) Reva et al. , 2011; web SIFT 0. 050 85. 03 68. 95 74. 77 76. 99 1147 (1. 99%) Kumar et al. , 2009; web Poly. Phen-2 0. 432 88. 68 62. 45 72. 00 75. 56 2279 (3. 95%) Adzhubei et al. , 2010; web Condel web server 0. 469 93. 84 46. 23 64. 67 70. 04 7194 (12. 48%) González-Pérez and López-Bigas, 2011; web
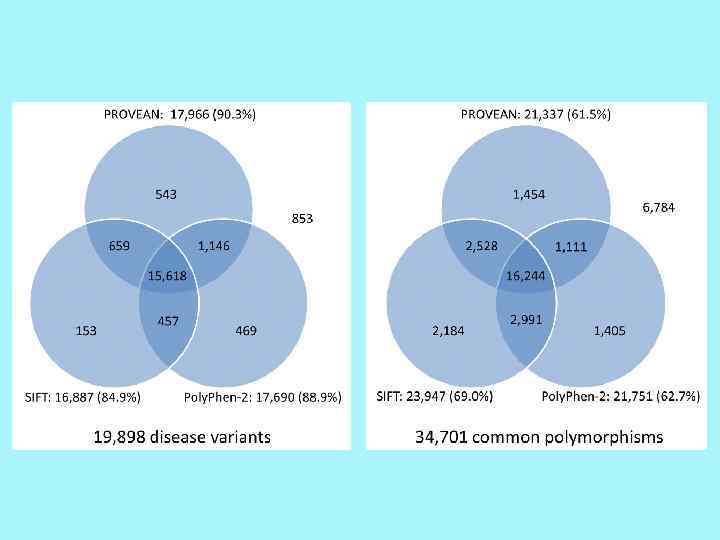
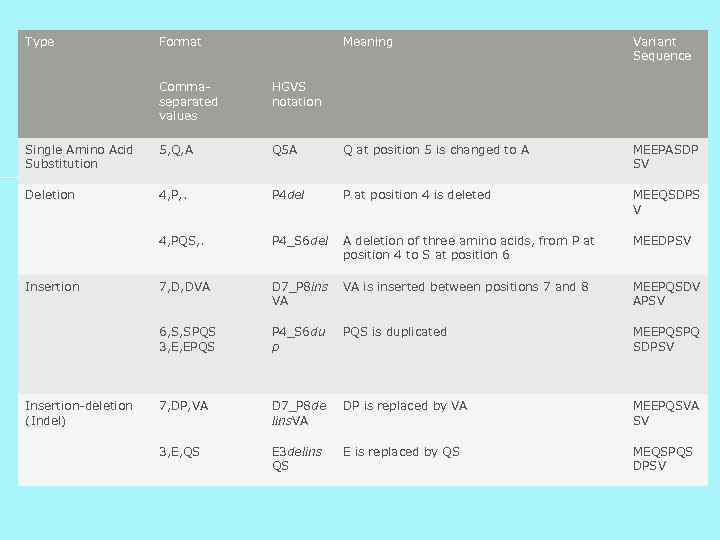 Type Format Meaning Variant Sequence Commaseparated values HGVS notation Single Amino Acid Substitution 5, Q, A Q 5 A Q at position 5 is changed to A MEEPASDP SV Deletion 4, P, . P 4 del P at position 4 is deleted MEEQSDPS V 4, PQS, . P 4_S 6 del A deletion of three amino acids, from P at position 4 to S at position 6 MEEDPSV 7, D, DVA D 7_P 8 ins VA VA is inserted between positions 7 and 8 MEEPQSDV APSV 6, S, SPQS 3, E, EPQS P 4_S 6 du p PQS is duplicated MEEPQSPQ SDPSV 7, DP, VA D 7_P 8 de lins. VA DP is replaced by VA MEEPQSVA SV 3, E, QS E 3 delins QS E is replaced by QS MEQSPQS DPSV Insertion-deletion (Indel)
Type Format Meaning Variant Sequence Commaseparated values HGVS notation Single Amino Acid Substitution 5, Q, A Q 5 A Q at position 5 is changed to A MEEPASDP SV Deletion 4, P, . P 4 del P at position 4 is deleted MEEQSDPS V 4, PQS, . P 4_S 6 del A deletion of three amino acids, from P at position 4 to S at position 6 MEEDPSV 7, D, DVA D 7_P 8 ins VA VA is inserted between positions 7 and 8 MEEPQSDV APSV 6, S, SPQS 3, E, EPQS P 4_S 6 du p PQS is duplicated MEEPQSPQ SDPSV 7, DP, VA D 7_P 8 de lins. VA DP is replaced by VA MEEPQSVA SV 3, E, QS E 3 delins QS E is replaced by QS MEQSPQS DPSV Insertion-deletion (Indel)
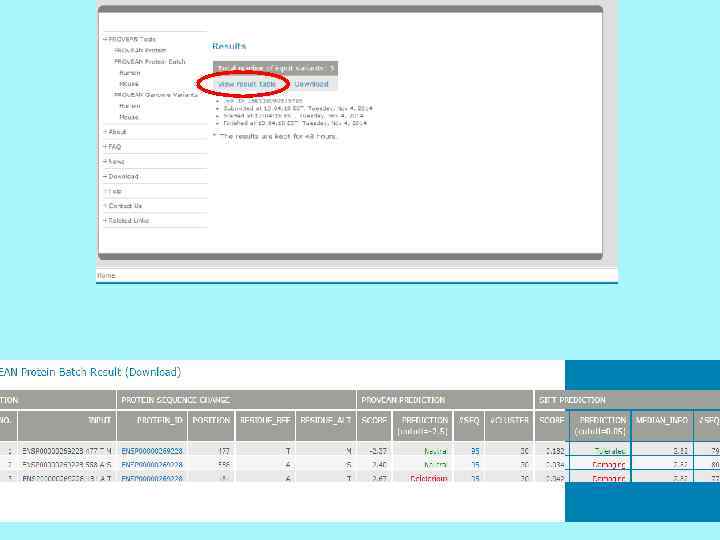


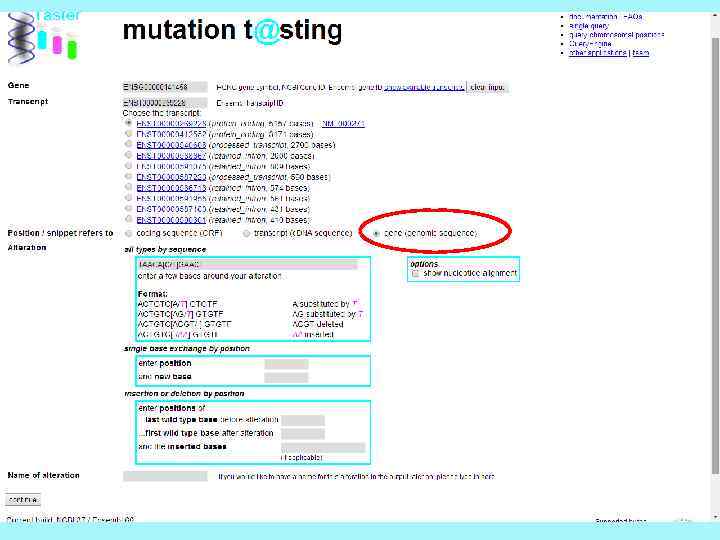
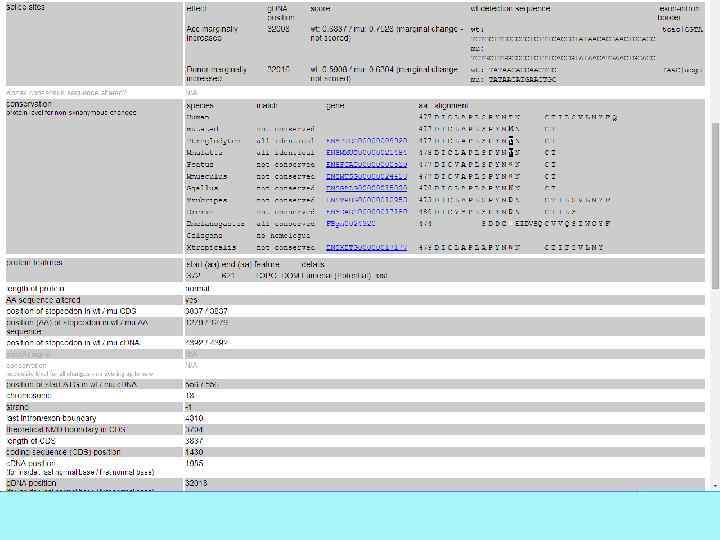
 Mutation taster
Mutation taster
 Prediction - предсказание Polymorphism – полиморфизм Disease causing – вызывает заболевание Принцип метода. Сравнение искомой замены с >390000 известных мутаций болезни из HGMD и >6800000 безвредных однонуклеотидных полиморфизмов и INDEL полиморфизмов > 1000 из проекта Геном. База данных содержит так называемые нормативные функции из базы данных Ensembl, такие как модификации гистонов и хроматинных областей или сайтов связывания транскрипционных факторов. Если изменение является «истинным» SNP (что подтверждается существованием каждого из трех генотипов АА, АВ, ВВ в Нар. Мар данных или наличием в литературе в гомозиготном состоянии в >4 случаях), то автоматически прогноз будет - полиморфизм.
Prediction - предсказание Polymorphism – полиморфизм Disease causing – вызывает заболевание Принцип метода. Сравнение искомой замены с >390000 известных мутаций болезни из HGMD и >6800000 безвредных однонуклеотидных полиморфизмов и INDEL полиморфизмов > 1000 из проекта Геном. База данных содержит так называемые нормативные функции из базы данных Ensembl, такие как модификации гистонов и хроматинных областей или сайтов связывания транскрипционных факторов. Если изменение является «истинным» SNP (что подтверждается существованием каждого из трех генотипов АА, АВ, ВВ в Нар. Мар данных или наличием в литературе в гомозиготном состоянии в >4 случаях), то автоматически прогноз будет - полиморфизм.




 Заключение Необходимо пользоваться всеми 4 базами для проверки патогенности замены. Отдельно хочется сказать, что наименее информативной программой является Mutation taster, однако его лучше использовать в самом начале, чтобы сама программа указала на номер нуклеотида и белка, а так же выдала информацию о положении в геноме. Затем лучше пользоваться Poly. Phen-2 поскольку ожидание результата в этой программе может доходить до целого дня. Программа SIFT+Provean выдает результаты быстро и пользоваться этой программой легче всего.
Заключение Необходимо пользоваться всеми 4 базами для проверки патогенности замены. Отдельно хочется сказать, что наименее информативной программой является Mutation taster, однако его лучше использовать в самом начале, чтобы сама программа указала на номер нуклеотида и белка, а так же выдала информацию о положении в геноме. Затем лучше пользоваться Poly. Phen-2 поскольку ожидание результата в этой программе может доходить до целого дня. Программа SIFT+Provean выдает результаты быстро и пользоваться этой программой легче всего.
 Спасибо за внимание!
Спасибо за внимание!


