lecture 5.ppt
- Количество слайдов: 85

Причины появления повреждений в ДНК • Ошибки репликации • Повреждения ДНК эндогенными агентами Гидролиз (депуринизация, дезаминирование) • Повреждения ДНК экзогенными агентами облучение повреждение химическими агентами (например, алкилирование) • Репликация «через повреждения» с использованием полимераз, отличающихся низкой точностью копирования
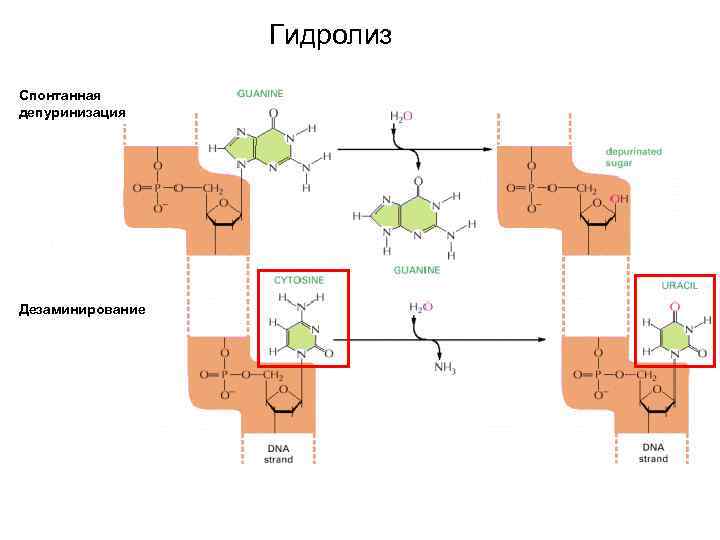
Гидролиз Спонтанная депуринизация Дезаминирование
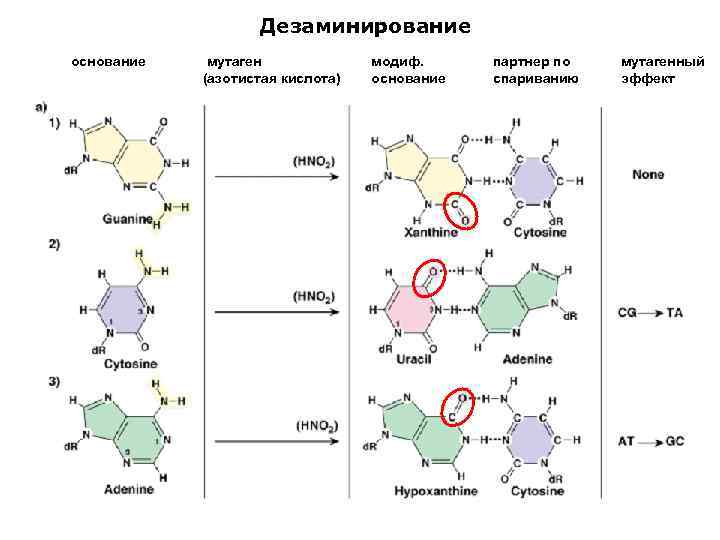
Дезаминирование основание мутаген (азотистая кислота) модиф. основание партнер по спариванию мутагенный эффект
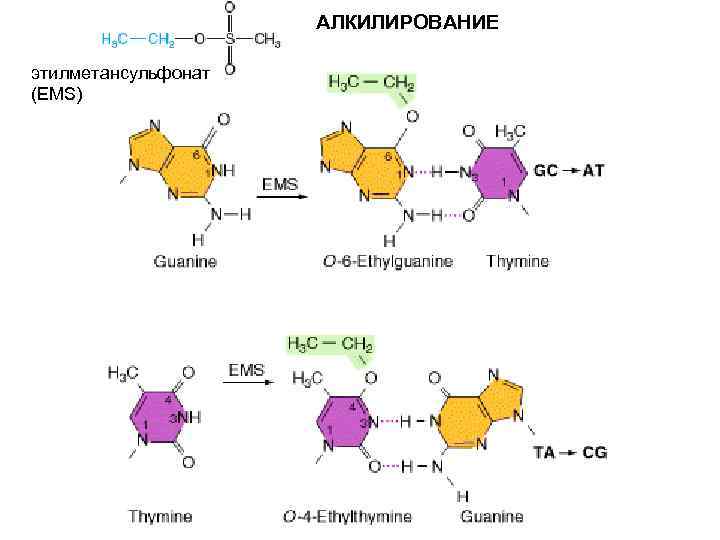
АЛКИЛИРОВАНИЕ этилметансульфонат (EMS)
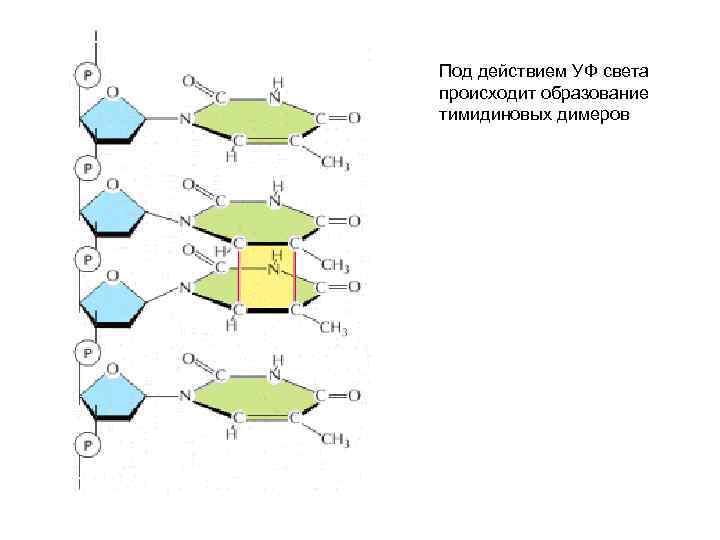
Под действием УФ света происходит образование тимидиновых димеров

Cтратегии коррекции повреждений • Ошибки репликации: • • исправление ошибок ДНК полимеразой (3’-5’ экзонуклеаза), репарация неспаренных оснований (mismatch repair) • Повреждение ДНК эндогенными и экзогенными агентами: • • Прямое удаление повреждений Эксцизия оснований (base excision repair) Эксцизия нуклеотидов (nucleotide excision repair) Рекомбинация и черезблоковый синтез особыми полимеразами (не удаляет ошибок но позволяет продолжить репликацию)
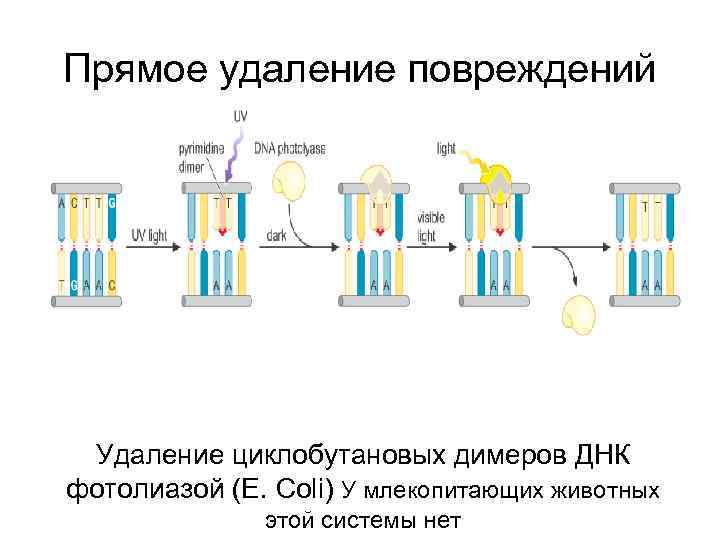
Прямое удаление повреждений Удаление циклобутановых димеров ДНК фотолиазой (E. Coli) У млекопитающих животных этой системы нет
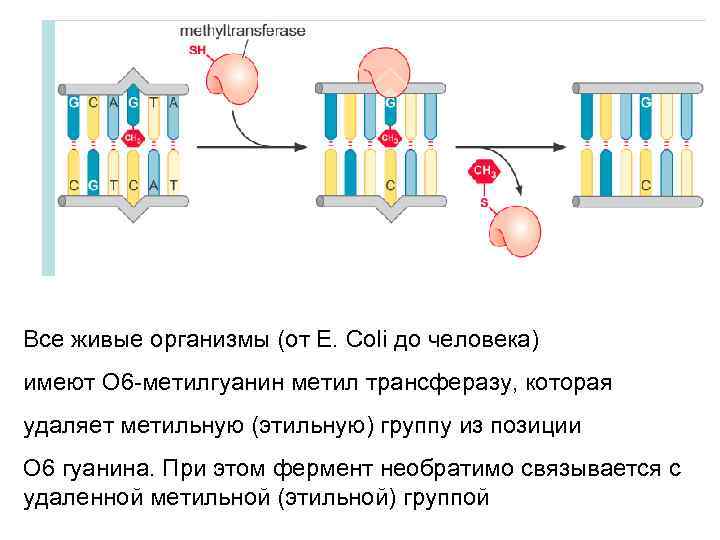
Все живые организмы (от E. Coli до человека) имеют О 6 -метилгуанин метил трансферазу, которая удаляет метильную (этильную) группу из позиции О 6 гуанина. При этом фермент необратимо связывается с удаленной метильной (этильной) группой

Другие повреждения удаляются с помощью более сложных механизмов Эксцизия оснований (base excision repair) удаляется один поврежденный нуклеотид Эксцизия нуклеотидов (nucleotide excision repair) удаляется достаточно протяженный олигонуклеотид (12 -13 н. у E. Coli и 24 -32 н. у эукариотических организмов)

Base excision repair (BER) эксцизия (вырезание) оснований прокариоты и эукариоты • Основной путь удаления модифицированных (в том числе окисленных) оснований и включенного по ошибке урацила • Различные ДНК-гликозилазы адресно узнают поврежденные основания и удаляют их, разрезая гликозидную связь. При этом возникает AP (апуриновый/апиримидиновый) сайт. • В клетке существует несколько АР эндонуклеаз, которые разрезают фосфодиэфирный остов ДНК рядом с AP сайтом • AP nucleotide удаляется экзонуклеазой/дезоксирибофосфодиэстеразой и «брешь» застраивается ДНК полимеразой
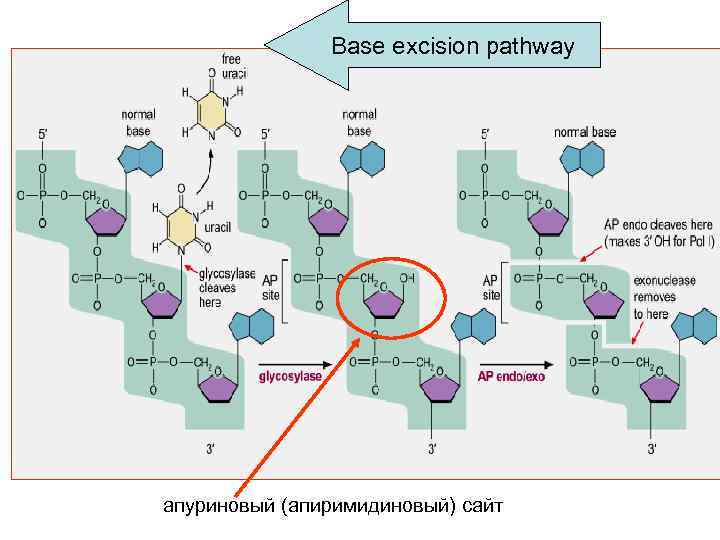
Base excision pathway апуриновый (апиримидиновый) сайт
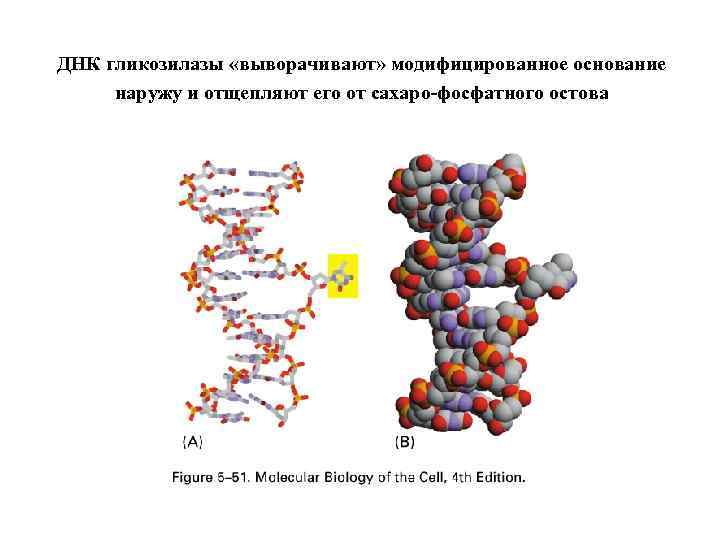
ДНК гликозилазы «выворачивают» модифицированное основание наружу и отщепляют его от сахаро-фосфатного остова
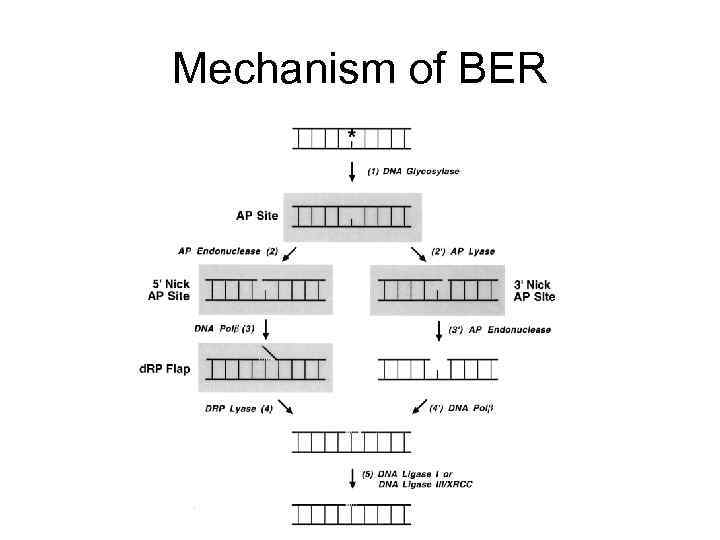
Mechanism of BER
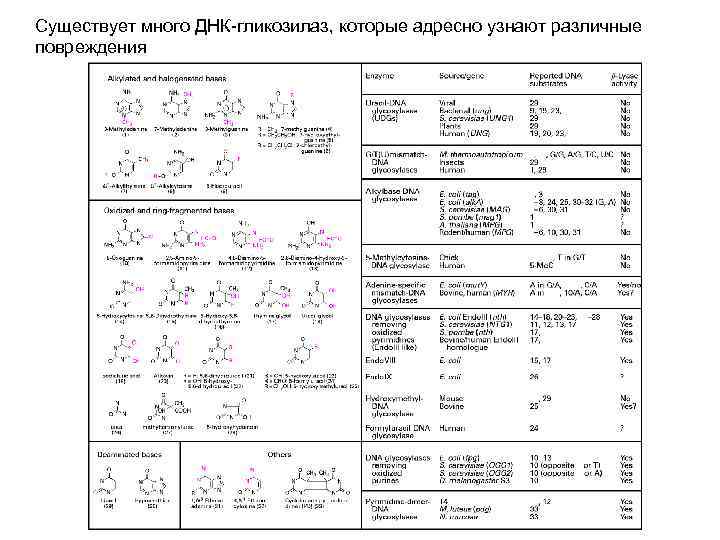
Существует много ДНК-гликозилаз, которые адресно узнают различные повреждения

ДНК-гликозизазы, присутствующие в клетках человека DNA glycosylase Specific for MBD 4 Uracil MPG Ethenoadenine, hypoxanthine, 3 methyladenine NTH 1 Cytosine glycol, dihydrouracil, formamidopyrimidine, thymine glycol OGG 1 Formamidopyrimidine, 8 -oxoguanine SMUG 1 Uracil TDG Ethenocytosine, uracil UNG Uracil, 5 -hydroxyuracil

Основные типы повреждений, которые удаляются посредством BER (большая часть не блокирует репликацию) • Окисленные основания, в том числе 8 -oкси-G, который спаривается с А, вызывая GC --> TA трансверсии • Дезоксиурацил • Различные продукты алкилирования оснований (например, 3 -me. A) • Спонтанно возникающие апуриновые сайты

Эксцизия нуклеотидов (nucleotide excision repair) прокариоты и эукриоты Этот механизм используется для коррекции «серьезных» повреждений, которые блокируют репликацию (у человека таковыми являются, в частности, тимидиновые димеры). 1. Особые белки узнают поврежденные участки ДНК и привлекают специальные нуклеазы, которые вносят разрывы на некотором расстоянии перед повреждением и на некотором расстоянии после него. 2. Фрагмент ДНК, содержащий повреждение, удаляется, и образовавшаяся брешь застраивается ДНК полимеразой


У E. Coli четыре белка участвуют в эксцизии нуклеотидов: Uvr. A, Uvr. B, Uvr. C, Uvr. D 1. Uvr. A и Uvr. B сканируют ДНК и выявляют поврежденные места 2. После выявления поврежденного участка Uvr. A освобождается из комплекса, а Uvr. B вызывает локальную денатурацию поврежденного участка и привлекает Uvr. C. 3. Комплекс Uvr. BС вносит однонитевые разрывы с 5’и 3’- конца от повреждения 4. DNA хеликаза Uvr. D обеспечивает удаление из дуплекса фрагмента ДНК, содержащего повреждение. 5. DNA Pol I застраивает брешь и лигаза «зашивает» однонитевой разрыв.
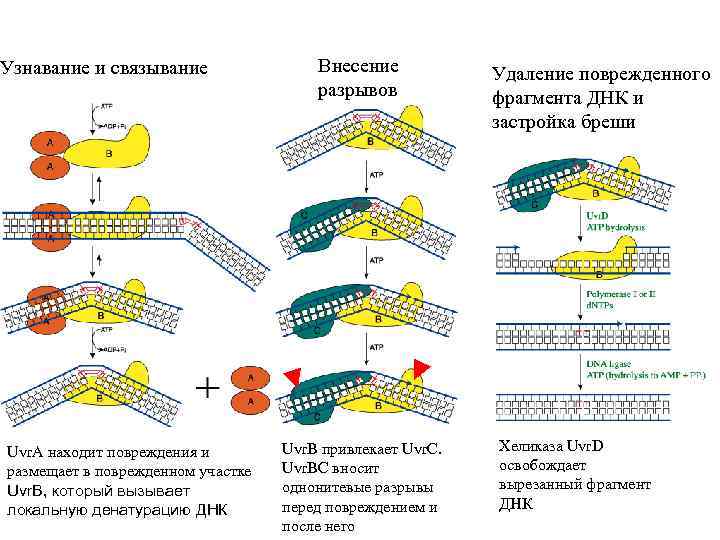
Узнавание и связывание Uvr. A находит повреждения и размещает в поврежденном участке Uvr. B, который вызывает локальную денатурацию ДНК Внесение разрывов Uvr. B привлекает Uvr. C. Uvr. BC вносит однонитевые разрывы перед повреждением и после него Удаление поврежденного фрагмента ДНК и застройка бреши Хеликаза Uvr. D освобождает вырезанный фрагмент ДНК
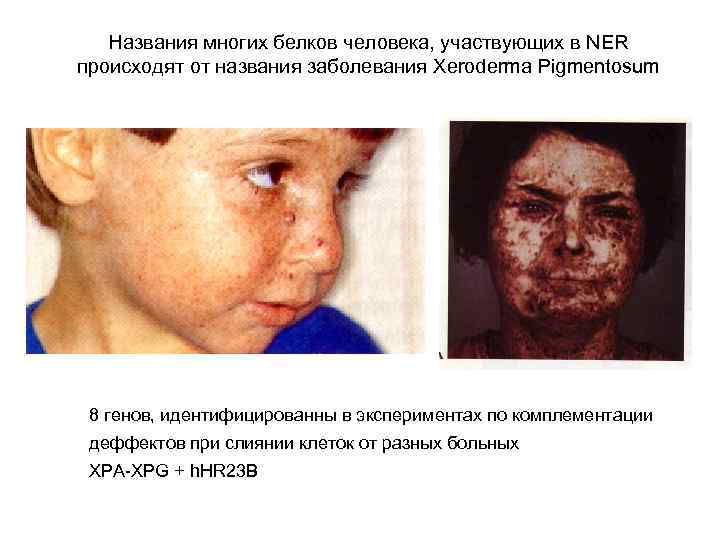
Названия многих белков человека, участвующих в NER происходят от названия заболевания Xeroderma Pigmentosum 8 генов, идентифицированны в экспериментах по комплементации деффектов при слиянии клеток от разных больных XPA-XPG + h. HR 23 B
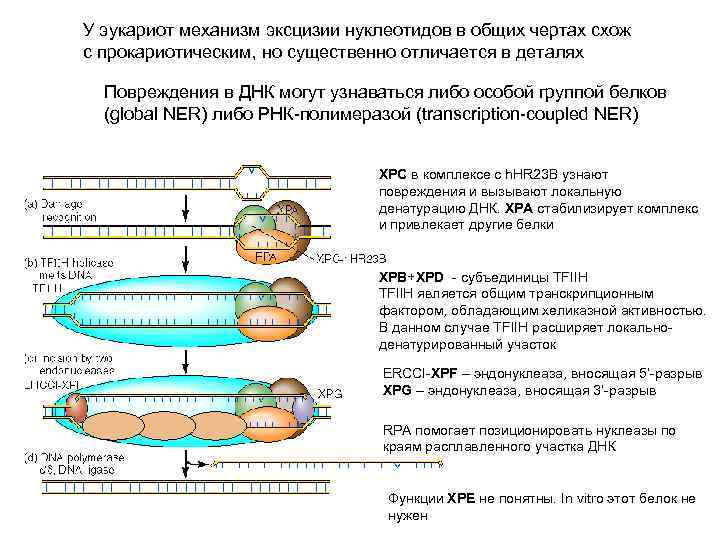
У эукариот механизм эксцизии нуклеотидов в общих чертах схож с прокариотическим, но существенно отличается в деталях Повреждения в ДНК могут узнаваться либо особой группой белков (global NER) либо РНК-полимеразой (transcription-coupled NER) XPC в комплексе с h. HR 23 B узнают повреждения и вызывают локальную денатурацию ДНК. XPA стабилизирует комплекс и привлекает другие белки XPB+XPD - субъединицы TFIIH является общим транскрипционным фактором, обладающим хеликазной активностью. В данном случае TFIIH расширяет локальноденатурированный участок ERCCI-XPF – эндонуклеаза, вносящая 5’-разрыв XPG – эндонуклеаза, вносящая 3’-разрыв RPA помогает позиционировать нуклеазы по краям расплавленного участка ДНК Функции XPE не понятны. In vitro этот белок не нужен
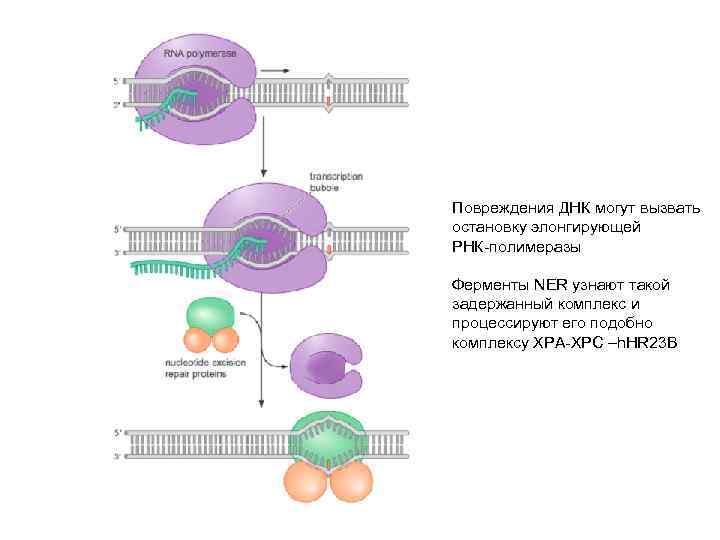
Повреждения ДНК могут вызвать остановку элонгирующей РНК-полимеразы Ферменты NER узнают такой задержанный комплекс и процессируют его подобно комплексу XPA-XPC –h. HR 23 B
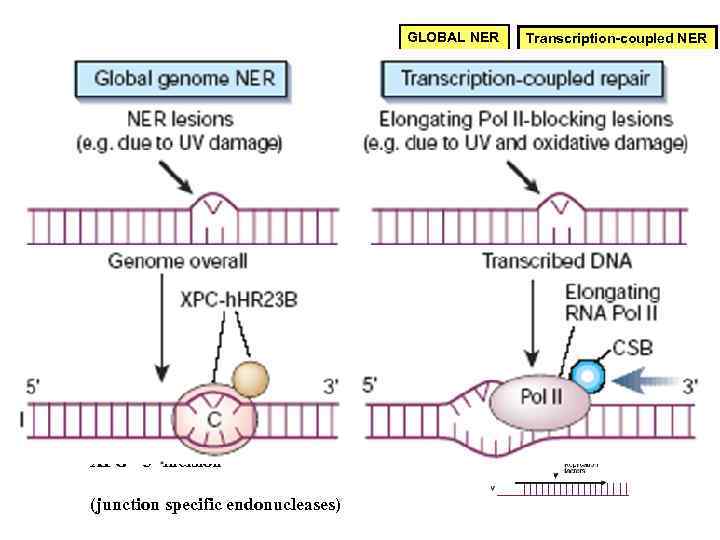
GLOBAL NER Transcription-coupled NER HPC-h. HR 23 B Pol II XPC - damage recognition TFIIH XPB & XPD – TFII H DNA helicase XPG XPA stabilisation of SS DNA fragment ERCC 1 -XPF - 5’ incision XPG - 3’ incision (junction specific endonucleases) XPG

Исправление ошибок репликации (Мismatch repair) • ДНК полимеразы (даже те, у которых есть корректирующая активность) все равно делают ошибки, которые надо исправлять • Система репарации ошибок репликации должна • 1. Быстро находить ошибки • 2. Различать родительскую и новосинтезированную • цепь с тем, чтобы в неспаренном участке • заменить ошибочно включенный нуклеотид
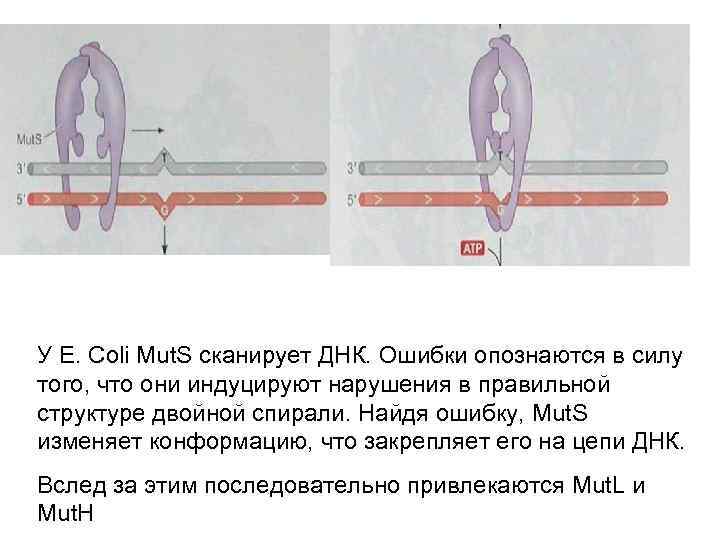
У E. Coli Mut. S сканирует ДНК. Ошибки опознаются в силу того, что они индуцируют нарушения в правильной структуре двойной спирали. Найдя ошибку, Mut. S изменяет конформацию, что закрепляет его на цепи ДНК. Вслед за этим последовательно привлекаются Mut. L и Mut. H
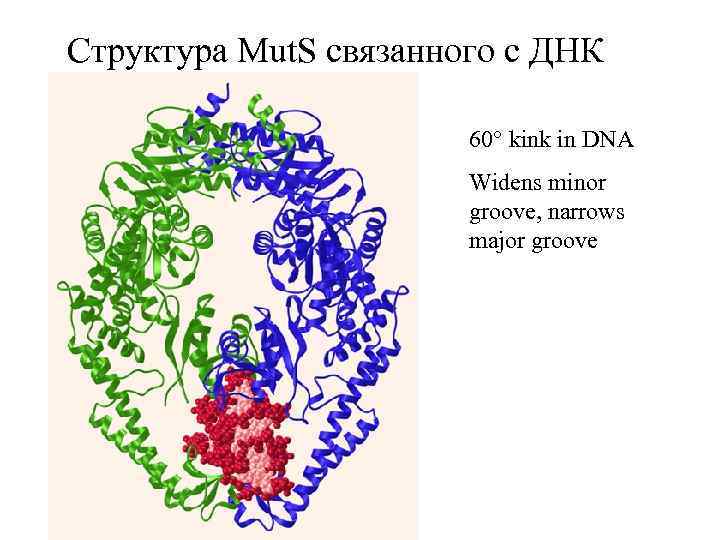
Структура Mut. S связанного с ДНК 60° kink in DNA Widens minor groove, narrows major groove

Что узнается ? • Mut. S димер (у дрожжей, Msh 2/Msh 3 или Msh 2/Msh 6 гетеродимер) • Эксперименты по связыванию с ДНК in vitro и репарации гетеродуплексов in vivo показали, что MMR узнает все комбинации неспаренных оснований, кроме C: C, а также короткие <4 п. н. делеции и инсерции ( «инделы» ) • неправильные пары G: T and A: C и инсерции/делеции в 1 п. особенно хорошо узнаются. Эти нарушения являются наиболее частыми ошибками ДНК-полимераз
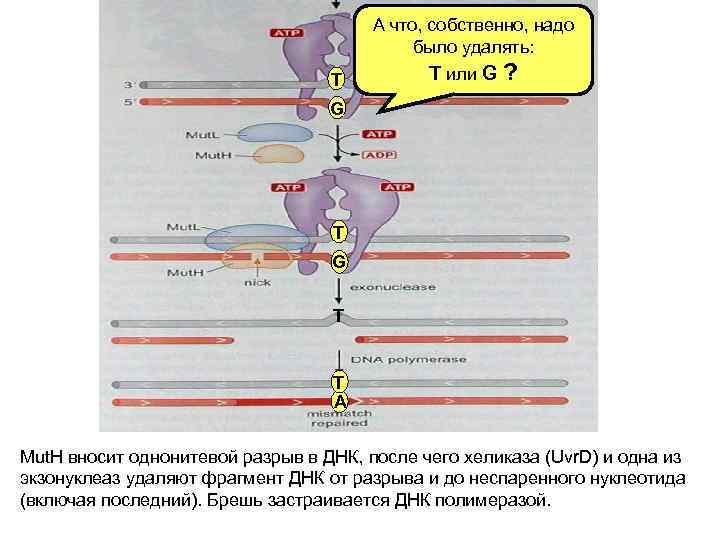
А что, собственно, надо было удалять: T T или G ? G T T A Mut. H вносит однонитевой разрыв в ДНК, после чего хеликаза (Uvr. D) и одна из экзонуклеаз удаляют фрагмент ДНК от разрыва и до неспаренного нуклеотида (включая последний). Брешь застраивается ДНК полимеразой.
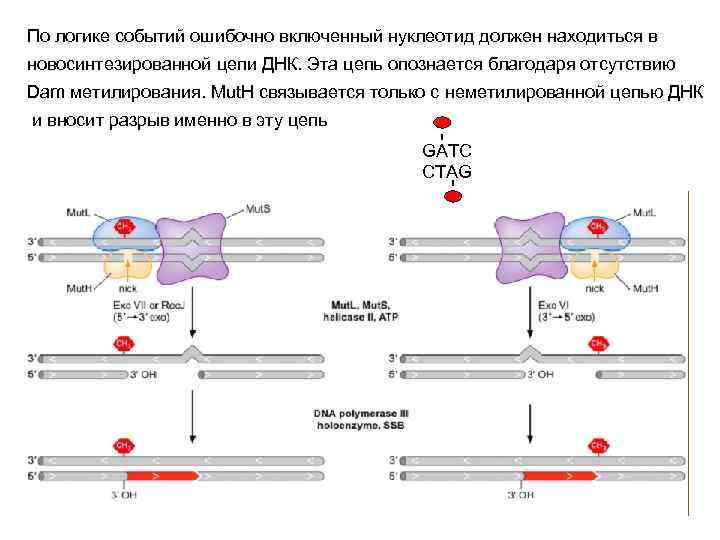
По логике событий ошибочно включенный нуклеотид должен находиться в новосинтезированной цепи ДНК. Эта цепь опознается благодаря отсутствию Dam метилирования. Mut. H связывается только с неметилированной цепью ДНК и вносит разрыв именно в эту цепь GATC CTAG

В эукариотических клетках также существует система коррекции ошибок репликации Обнаружены гомологи Mut. S и Mut. L; гомолога Mut. H не обнаружено гомологи Mut. S (MSH — Mut. S homolog) образуют два гетеродимерных комплекса -- MSH 2 -MSH 6 (Mut. Sα) узнает неспаренные нуклеотиды и короткие «инделы» -- MSH 2 -MSH 3 (Mut. Sβ) узнает длинные «инделы» Механизм распознавания новосинтезированной цепи не известен наличие разрывов?


А если не успели все починить а ДНК уже реплицируется?
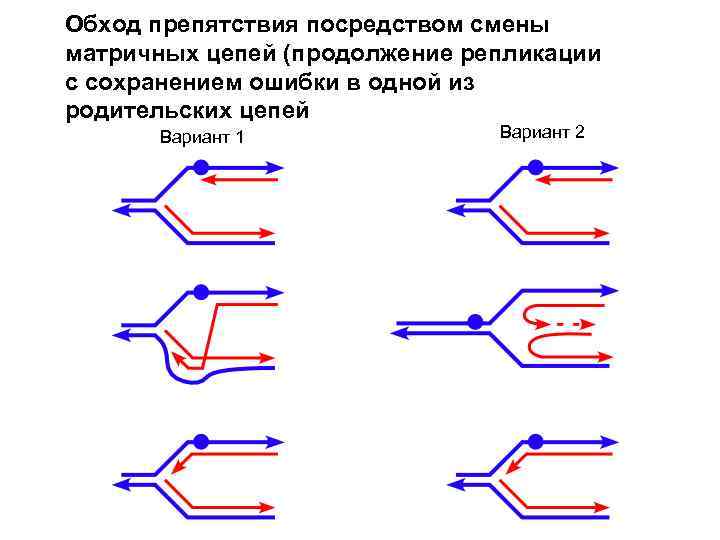
Обход препятствия посредством смены матричных цепей (продолжение репликации с сохранением ошибки в одной из родительских цепей Вариант 1 Вариант 2
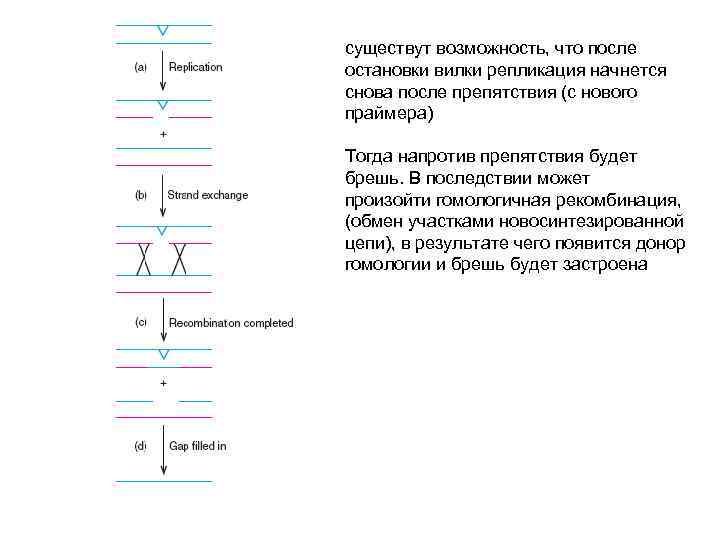
существут возможность, что после остановки вилки репликация начнется снова после препятствия (с нового праймера) Тогда напротив препятствия будет брешь. В последствии может произойти гомологичная рекомбинация, (обмен участками новосинтезированной цепи), в результате чего появится донор гомологии и брешь будет застроена

SOS репарация у Е. Сoli Прежде всего об остановке репликации надо сообщить Rec A Cвязывается с однонитевой ДНК и образует ДНК-белковые филаменты Однонитевые участки ДНК образуются при остановке репликативных вилок Lex A Мастер-регулятор транскрипции генов, кодирующих участвующие в репарации повреждений ДНК белки (31 ген или более) Димеры Lex A связываются с SOS боксами (20 п. н. консенсусы) в операторах генов репарации и ингибируют транскрипцию
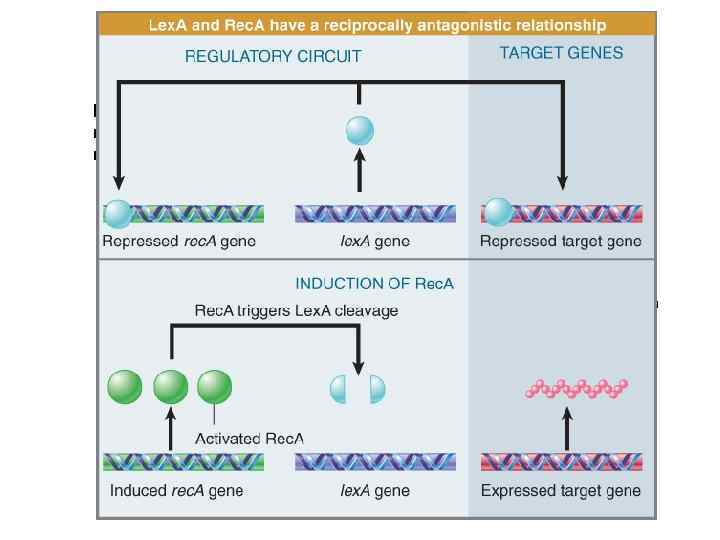
Филаменты Rec A стимулируют аутопротеолиз Lex A При снижении концентрации Lex A сначала активируются, гены, контролируемые операторами, в состав которых входят низкоафинные Lex A боксы lex. A, rec. A, uvr. B, and uvr. D При дальнейшем снижении уровня Lex A активируются гены, обеспецивающие осуществление репарации с ошибками (мутазы) Umu. DC оперон

Umu. D повергается автопротеолитическому расщеплению с образованием активного фрагмента Umu. D’ активирует черезблоковую полимеразу Umu. C комплекс (Umu. D’)2 -Umu. C теперь называют ДНК полимераза V эта полимераза осуществляет репликацию через AP сайты, тимидиновые димеры и ряд других повреждений
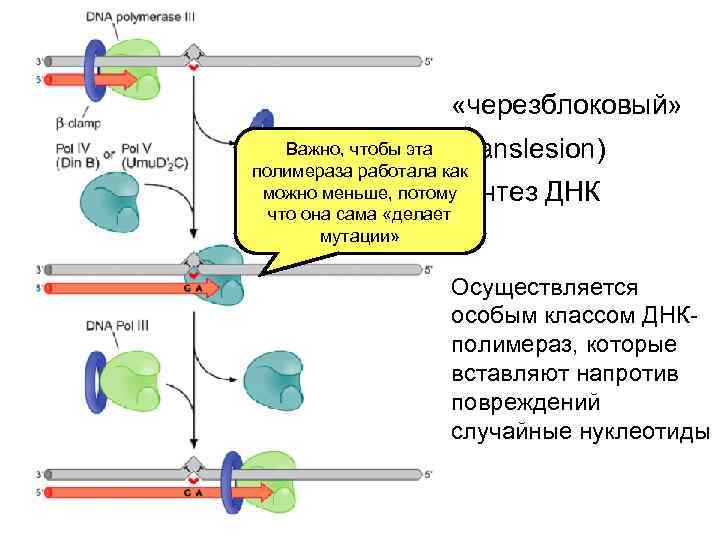
«черезблоковый» Важно, чтобы эта (translesion) полимераза работала как можно меньше, потому синтез ДНК что она сама «делает мутации» Осуществляется особым классом ДНКполимераз, которые вставляют напротив повреждений случайные нуклеотиды

«черезблоковый» синтез катализируемой «мутасомой» (DNA pol V)” Cowcatcher model G При наличии препятствия на пути ДНК поллимеразы III, эта полимераза диссоциирует от ДНК. Хеликаза продолжает работать, генерируя однотитевой участок, с которым связывается Rec. A Pol V связывается со свободным праймером. Для эффективного связывания необходим контакт с Rec A и с бета-зажимом Непосредственно в месте повреждения с ДНК связывается SSB Locomotive Pol V начинает синтез ДНК, одновременно вытесняя Rec A cowcatcher филамент. Одновременно происходит процессивная диссоциация Rec A филамента с противоположного конца, сопряденная с (скотосбраыватель) гидролизом ATФ. Pham P et al. PNAS 2001; 98: 8350 -8354 © 2001 by The National Academy of Sciences После удаления всего филамента Rec A Pol V диссоциирует от ДНК, освобождая место для Pol III. Таким образом время работы Pol V определяется временем существования Rec A филамента. Pol V успевает включить несколько нуклеотидов. Напротив тимидинового димера ТТ чаще всего включается GA
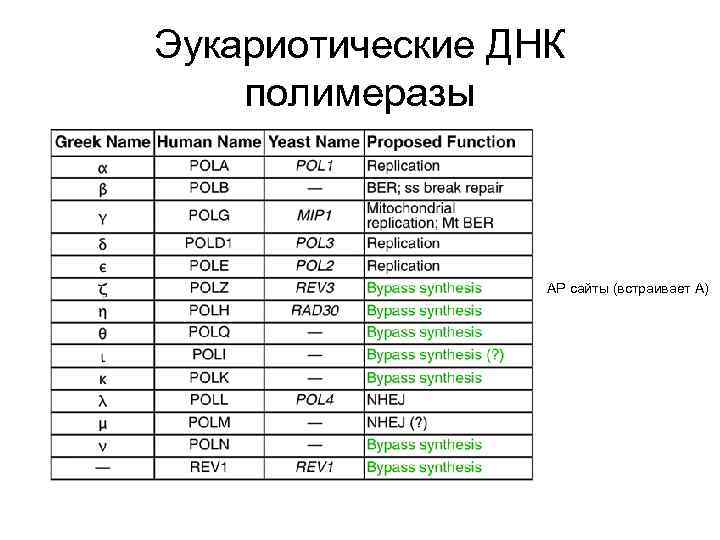
Эукариотические ДНК полимеразы АР сайты (встраивает А)

Репарация двунитевых разрывов Двунитевые разрывы в ДНК возникают: под действием ионизирующего излучения под действием некоторых химических агентов, в частности, ингибиторов ДНК топоизомеразы II Существует два основных пути репарации двунитевых разрывов: гомологичная рекомбинация негомологичное соединение концов ДНК
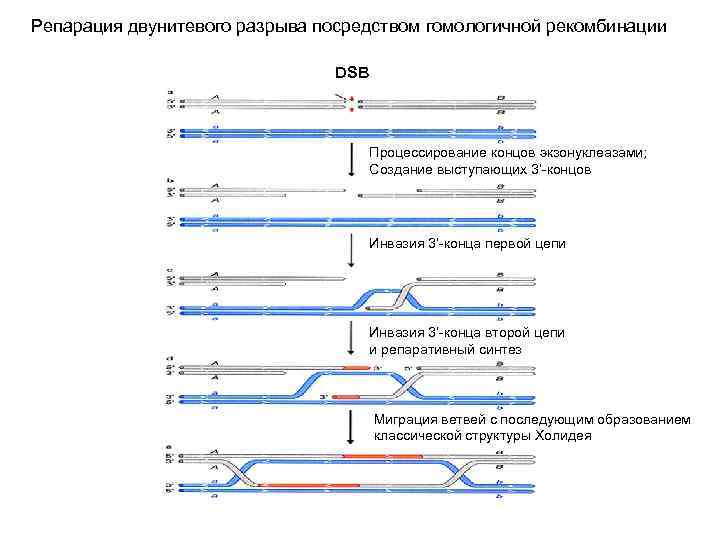
Репарация двунитевого разрыва посредством гомологичной рекомбинации DSB Процессирование концов экзонуклеазами; Создание выступающих 3’-концов Инвазия 3’-конца первой цепи Инвазия 3’-конца второй цепи и репаративный синтез Миграция ветвей с последующим образованием классической структуры Холидея

Для репарации двунитевых разрывов с использованием системы гомологичной рекомбинации необходимы: Донор гомологии (например гомологичная хромосома или сестринская хроматида) Белок, облегчающий инвазию цепи, и другие компоненты системы гомологичной рекомбинации
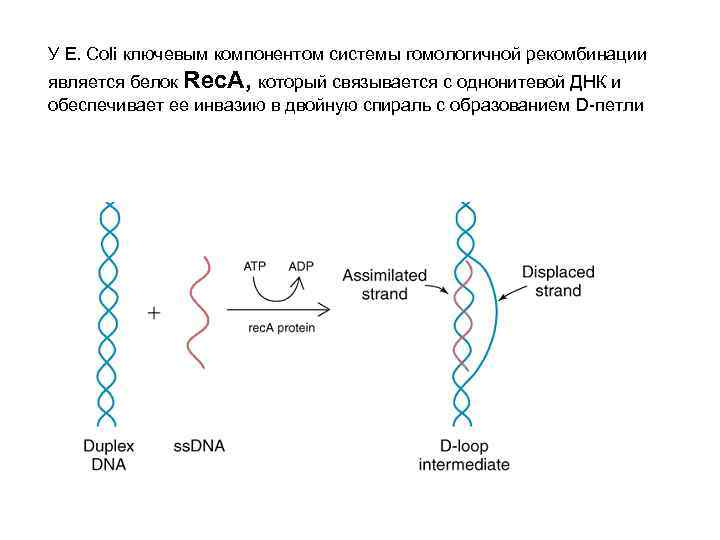
У E. Coli ключевым компонентом системы гомологичной рекомбинации является белок Rec. A, который связывается с однонитевой ДНК и обеспечивает ее инвазию в двойную спираль с образованием D-петли
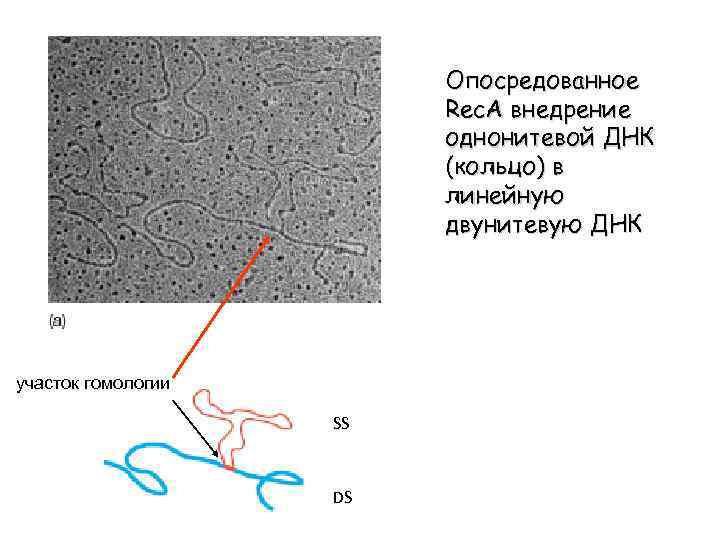
Опосредованное Rec. A внедрение однонитевой ДНК (кольцо) в линейную двунитевую ДНК участок гомологии SS 22. 13 DS Weaver, R. F. in Molecular Biology, 1999 Mc. Graw-Hill

Homologous Recombination Repairs ds Breaks В эукариотических клетках гомологом rec. A является Rad 51 Покрытая Rad 51 однонитевая ДНК внедряется в гомологичный участок сестринской хроматиды с образованием D петли 3’-конец внедрившейся цепи достраивается ДНК полимеразой и отжигается с 3’-концом комплементарной цепи исходного дуплекса Бреши застраиваются и однонитевые разрывы лигируются
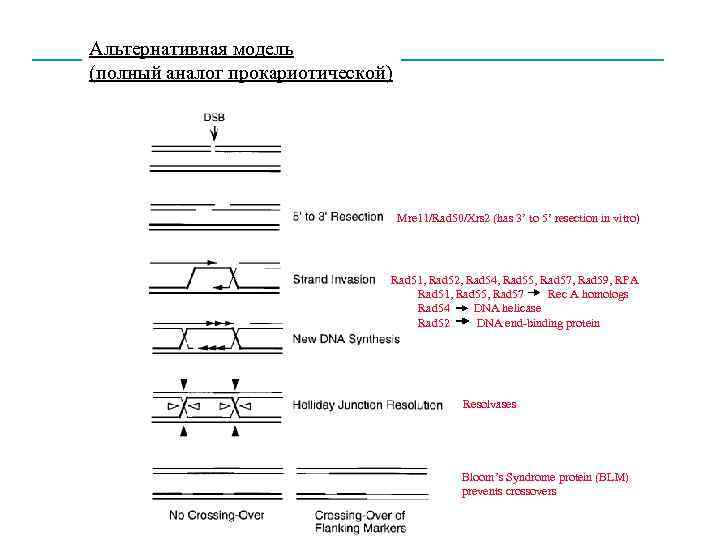
Альтернативная модель (полный аналог прокариотической) Mre 11/Rad 50/Xrs 2 (has 3’ to 5’ resection in vitro) Rad 51, Rad 52, Rad 54, Rad 55, Rad 57, Rad 59, RPA Rad 51, Rad 55, Rad 57 Rec A homologs Rad 54 DNA helicase Rad 52 DNA end-binding protein Resolvases Bloom’s Syndrome protein (BLM) prevents crossovers
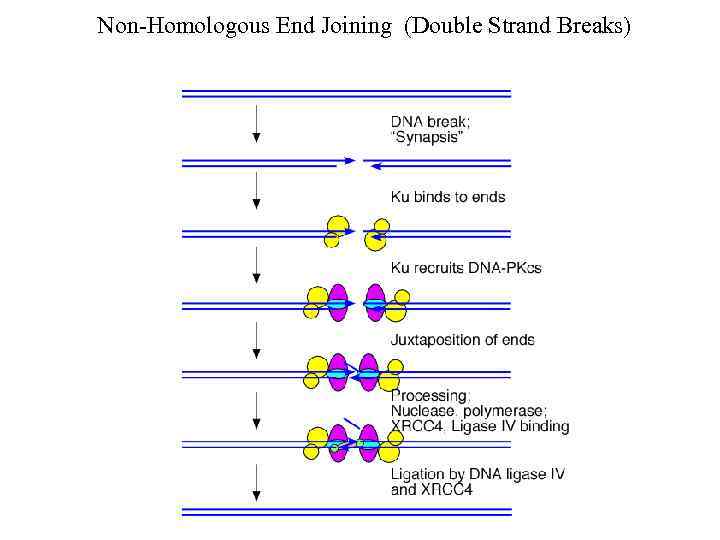
Non-Homologous End Joining (Double Strand Breaks)



РЕКОМБИНАЦИЯ ДНК Рекомбинация ДНК является одним из важнейших биологических процессов, повышающий уровень разнообразия геномов В рекомбинации ДНК участвуют многие механизмы, задействованные в репарации двунитевых разрывов по пути гомологической рекомбинации
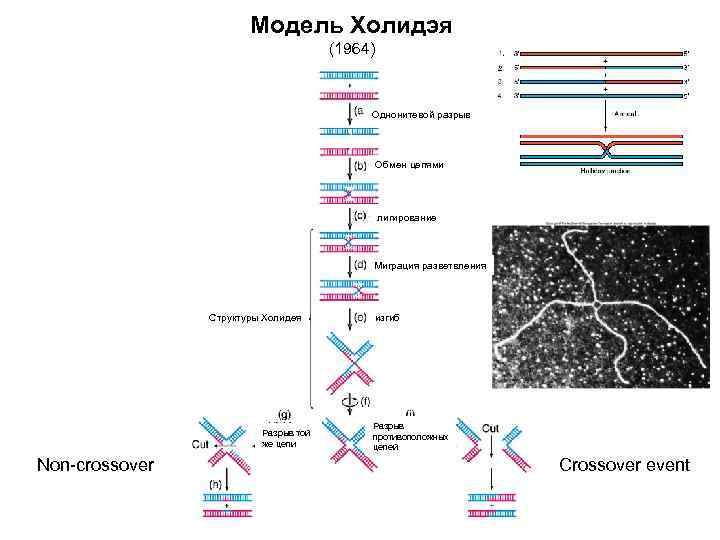
Модель Холидэя (1964) Однонитевой разрыв Обмен цепями лигирование Миграция разветвления Структуры Холидея Разрыв той же цепи Non-crossover изгиб Разрыв противоположных цепей Crossover event
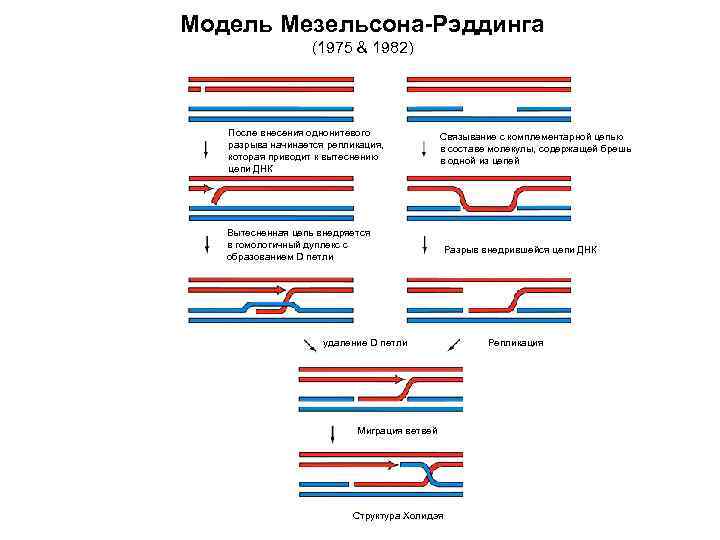
Модель Мезельсона-Рэддинга (1975 & 1982) После внесения однонитевого разрыва начинается репликация, которая приводит к вытеснению цепи ДНК Связывание с комплементарной цепью в составе молекулы, содержащей брешь в одной из цепей Вытесненная цепь внедряется в гомологичный дуплекс с образованием D петли удаление D петли Миграция ветвей Структура Холидэя Разрыв внедрившейся цепи ДНК Репликация

Рекомбинация в клетках E. coli Rec. BCD путь Rec. BCD комплекс вносит однонитевой разрыв рядом с 3’ концом сайта (5’-GCTGGTGG-3’) расплетает ДНК. сайты встречаются в среднем 1 раз на 5000 пн. Фрагмент однонитевой ДНК покрывается Rec. A белком, что обеспечивает его внедрение в гомологичный дуплекс В D петлю вносится разрыв (предположительно комплексом Rec. BCD) В результате происходит реципроктный обмен комплементарными цепями. После лигирования концов возникает типичная структура Холидэя Белки Ruv. A и Ruv. B (оба обладающие хеликазной и АТФазной активностями) обеспецивают миграцию ветвей. Ruv C внисит разрывы в структуру Холидэя, приводя к высвобождению двух молекул двунитевой ДНК
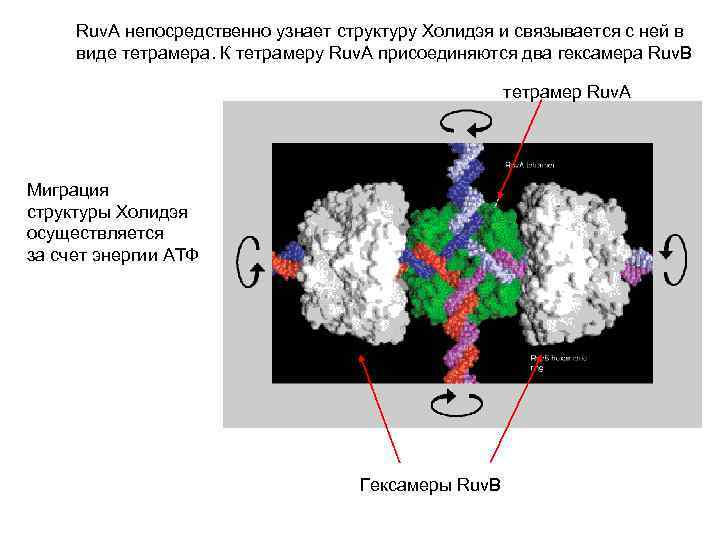
Ruv. A непосредственно узнает структуру Холидэя и связывается с ней в виде тетрамера. К тетрамеру Ruv. A присоединяются два гексамера Ruv. B тетрамер Ruv. A Миграция структуры Холидэя осуществляется за счет энергии АТФ Гексамеры Ruv. B


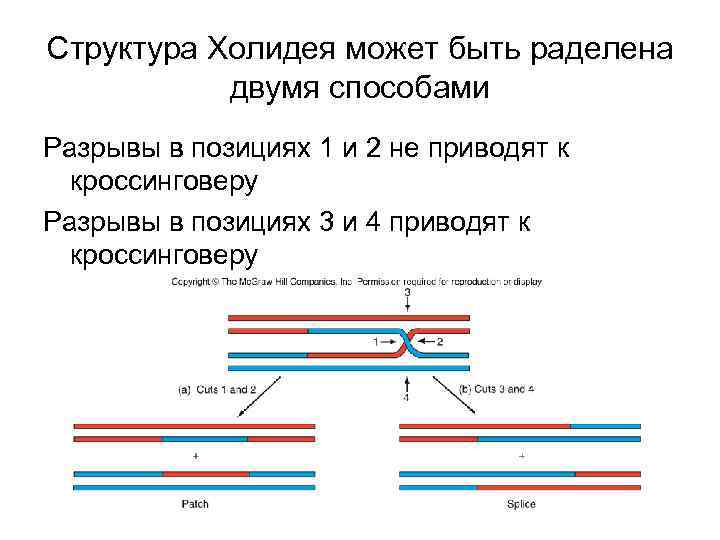
Структура Холидея может быть раделена двумя способами Разрывы в позициях 1 и 2 не приводят к кроссинговеру Разрывы в позициях 3 и 4 приводят к кроссинговеру
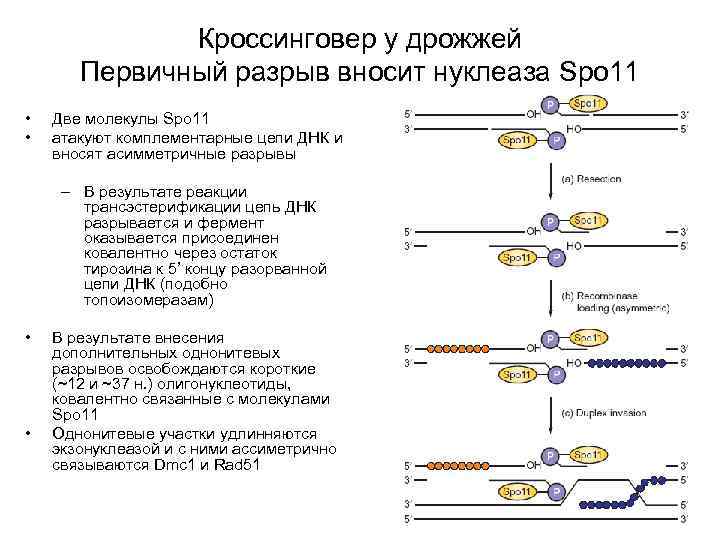
Кроссинговер у дрожжей Первичный разрыв вносит нуклеаза Spo 11 • • Две молекулы Spo 11 атакуют комплементарные цепи ДНК и вносят асимметричные разрывы – В результате реакции трансэстерификации цепь ДНК разрывается и фермент оказывается присоединен ковалентно через остаток тирозина к 5’ концу разорванной цепи ДНК (подобно топоизомеразам) • • В результате внесения дополнительных однонитевых разрывов освобождаются короткие (~12 и ~37 н. ) олигонуклеотиды, ковалентно связанные с молекулами Spo 11 Однонитевые участки удлинняются экзонуклеазой и с ними ассиметрично связываются Dmc 1 и Rad 51


Dmc 1 мейотический аналог Rad 51. Связывается с одним из однонитевых концов

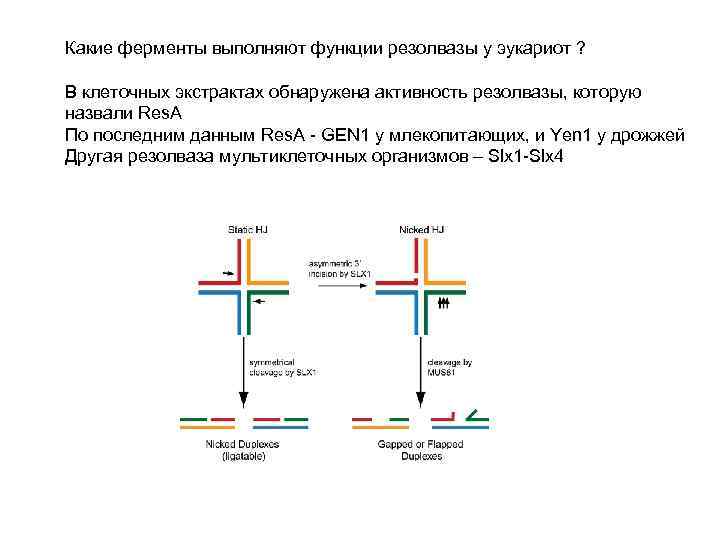
Какие ферменты выполняют функции резолвазы у эукариот ? В клеточных экстрактах обнаружена активность резолвазы, которую назвали Res. A По последним данным Res. A - GEN 1 у млекопитающих, и Yen 1 у дрожжей Другая резолваза мультиклеточных организмов – Slx 1 -Slx 4

ГЕННАЯ КОНВЕРСИЯ – «превращение» одного аллеля в другой
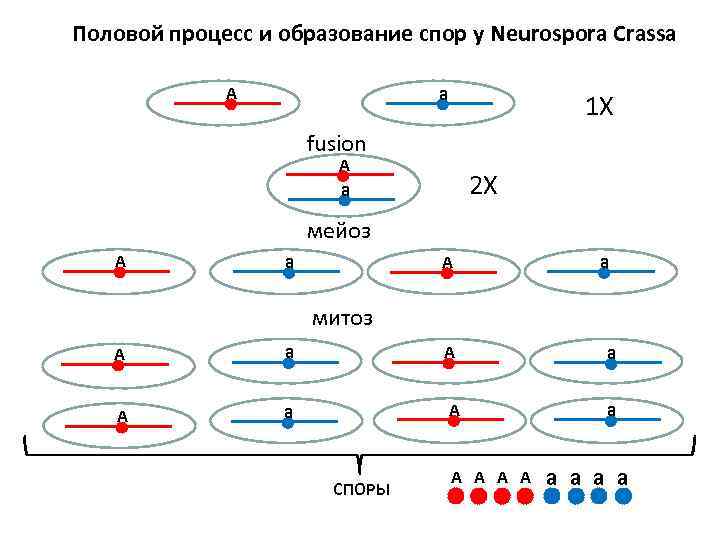
Половой процесс и образование спор у Neurospora Crassa а А 1 X fusion А 2 X а мейоз А а митоз А а А а СПОРЫ А А a a
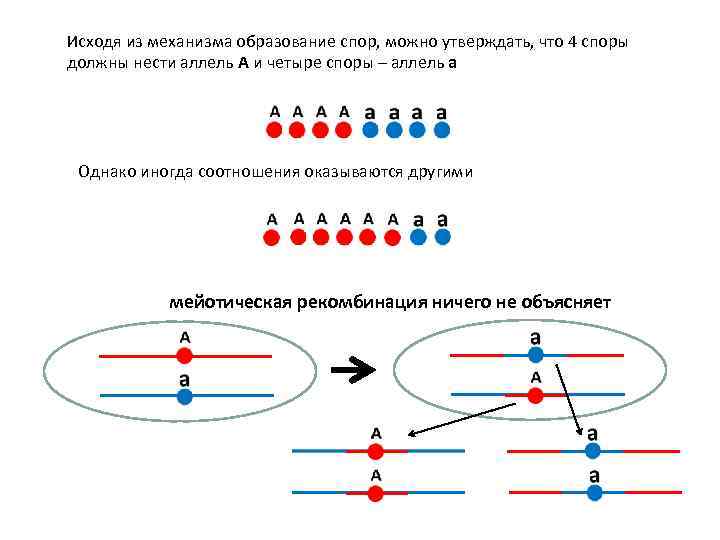
Исходя из механизма образование спор, можно утверждать, что 4 споры должны нести аллель A и четыре споры – аллель a Однако иногда соотношения оказываются другими мейотическая рекомбинация ничего не объясняет


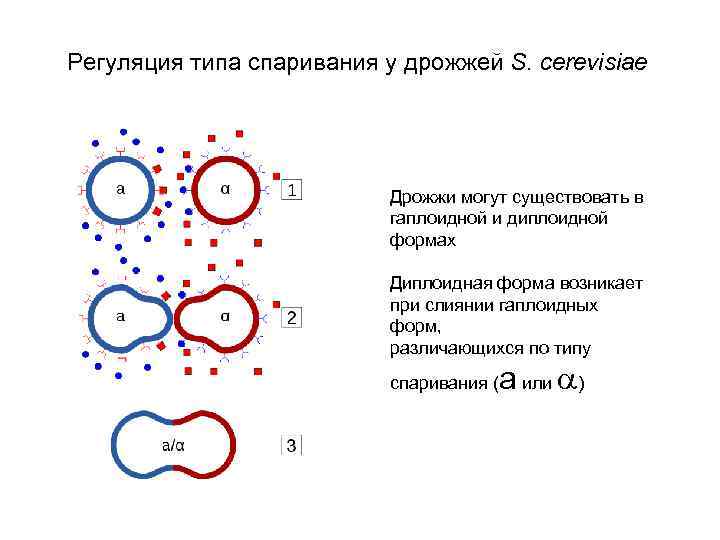
Регуляция типа спаривания у дрожжей S. cerevisiae Дрожжи могут существовать в гаплоидной и диплоидной формах Диплоидная форма возникает при слиянии гаплоидных форм, различающихся по типу спаривания ( a или a)
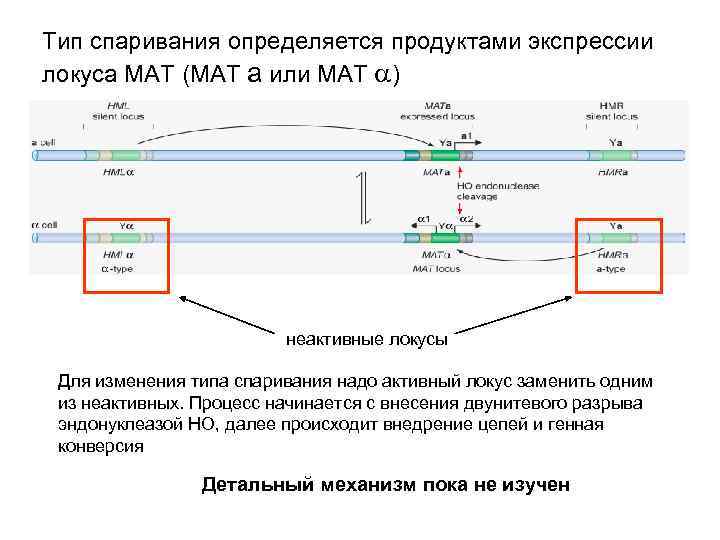
Тип спаривания определяется продуктами экспрессии локуса MAT (MAT a или MAT a) неактивные локусы Для изменения типа спаривания надо активный локус заменить одним из неактивных. Процесс начинается с внесения двунитевого разрыва эндонуклеазой HO, далее происходит внедрение цепей и генная конверсия Детальный механизм пока не изучен
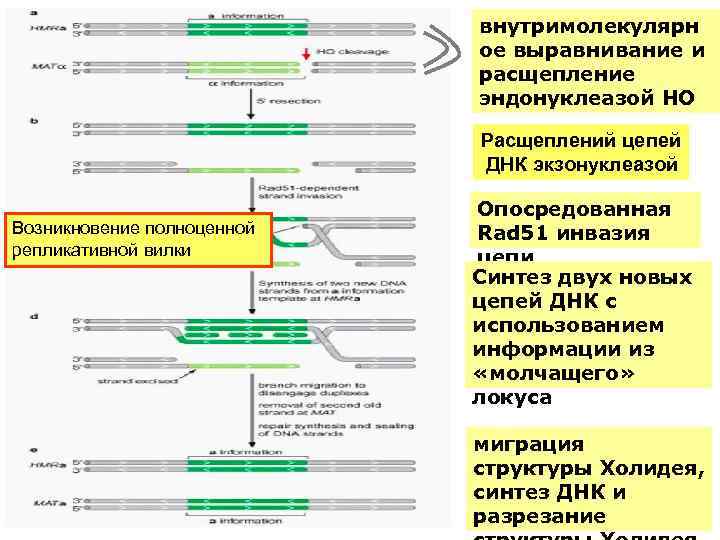
внутримолекулярн ое выравнивание и расщепление эндонуклеазой HO Расщеплений цепей ДНК экзонуклеазой Возникновение полноценной репликативной вилки Опосредованная Rad 51 инвазия цепи Синтез двух новых цепей ДНК c использованием информации из «молчащего» локуса миграция структуры Холидея, синтез ДНК и разрезание

Обмен участками может происходить и между сестринскими хроматидами
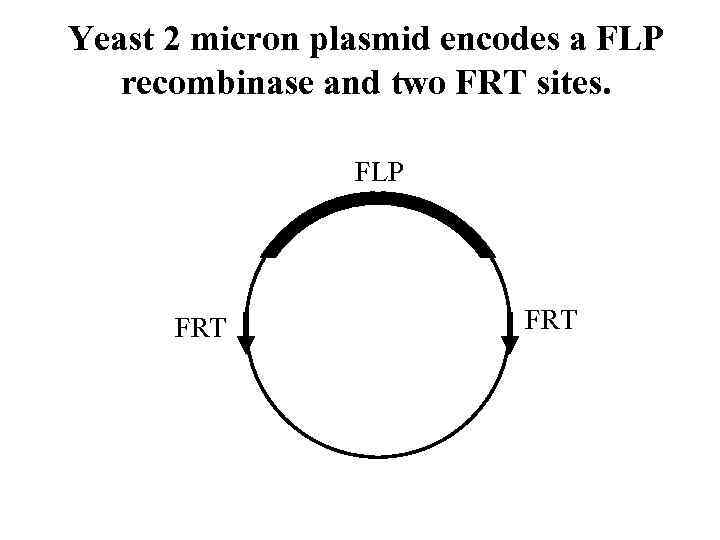
Yeast 2 micron plasmid encodes a FLP recombinase and two FRT sites. FLP FRT

FLP - Flippase FRT - Flippase Recognition Targets FRT – 34 bp последовательность ДНК с инвертированными 13 bp повторами на концах. FRT имеет направление благодаря асимметричному внутреннему участку 5'-GAAGTTCCTATTCtctagaaa. GTATAGGAACTTC-3'. 3’-CTTCAAGGATAAGagatcttt. CATATCCTTGAAG-5’ Флиппаза связывается с инвертированными повторами



в основном у прокариот сходство с топоизомеразами сохранение энергии при связывании белка с 3’ концом разорванной цепи ДНК Дальнейшие процессы идут без затраты энергии Альтернативная активация субъединиц связанных с «верхней» и «нижней» цепями ДНК исключает возможность появления двунитевых разрывов в молекуле ДНК
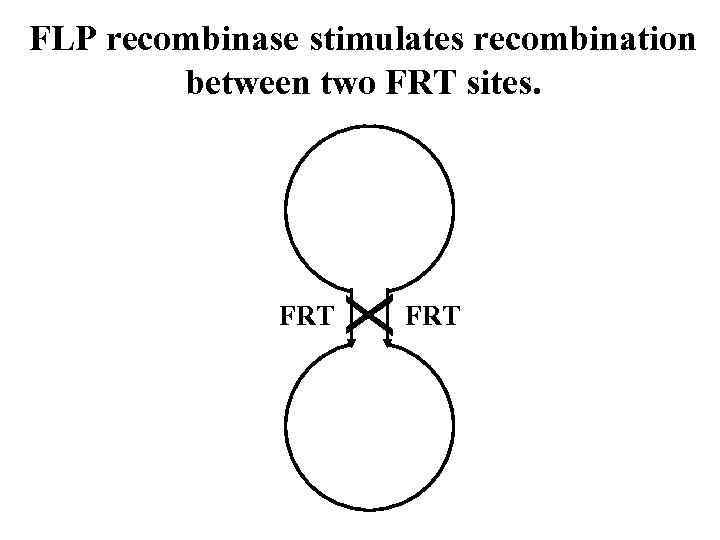
X FLP recombinase stimulates recombination between two FRT sites. FRT
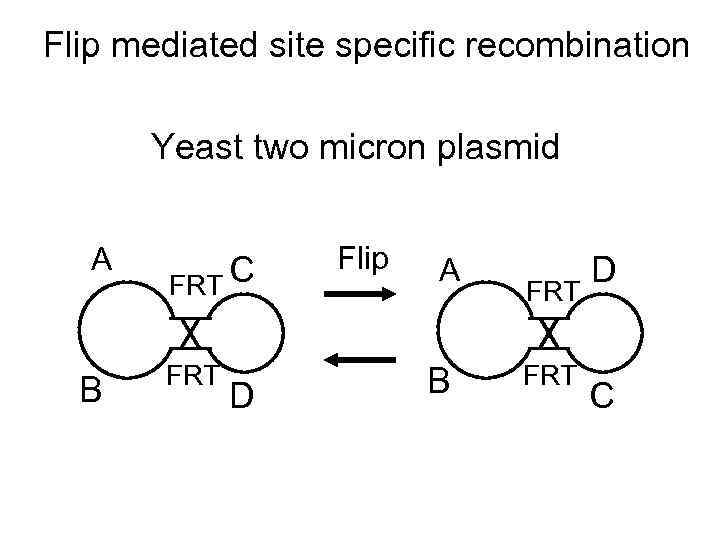
Flip mediated site specific recombination Yeast two micron plasmid A B FRT C FRT D Flip A B FRT D C
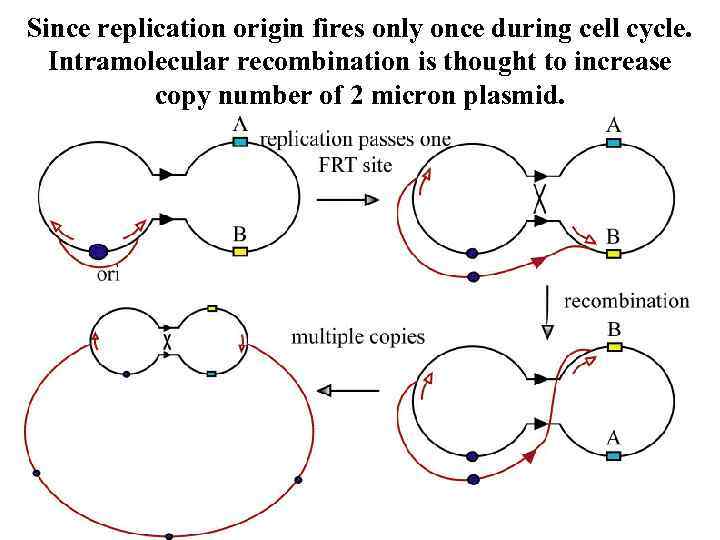
Since replication origin fires only once during cell cycle. Intramolecular recombination is thought to increase copy number of 2 micron plasmid.
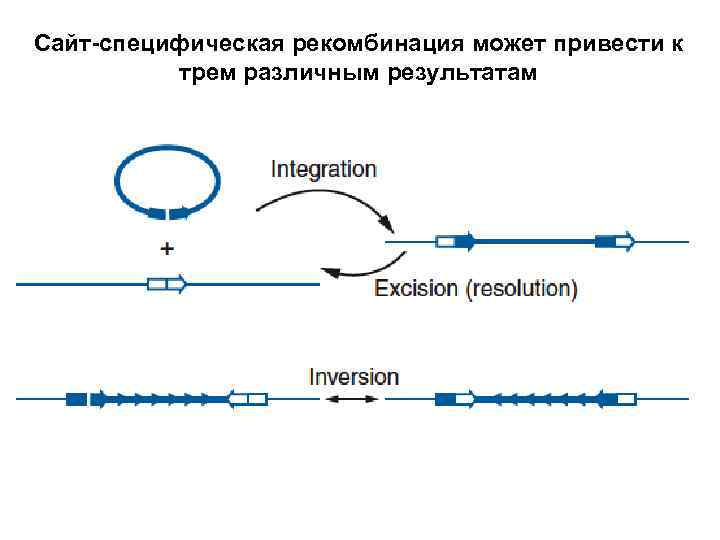
Сайт-специфическая рекомбинация может привести к трем различным результатам
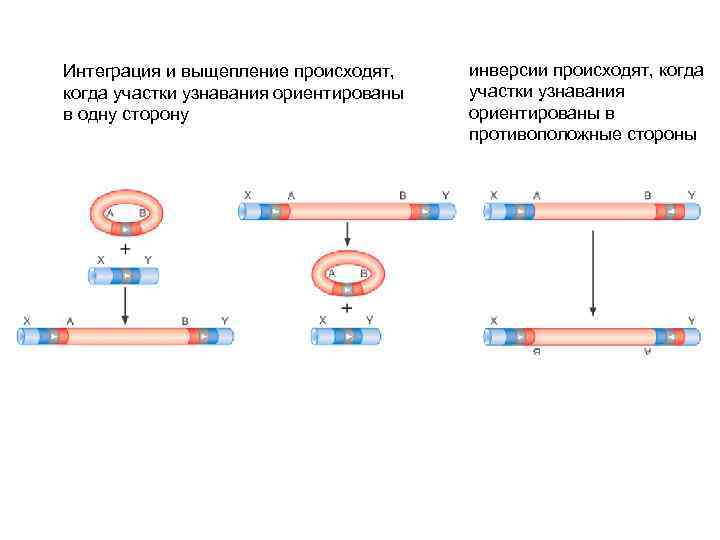
Интеграция и выщепление происходят, когда участки узнавания ориентированы в одну сторону инверсии происходят, когда участки узнавания ориентированы в противоположные стороны

В ходе работы рекомбиназ возникает временны промежуточный комплекс рекомбиназы с разорванной цепью ДНК, который запасает энергию, необходимую для последующего соединения цепей Существует два класса сайт-специфических рекомбиназ, различающихся по аминокислотному остатку активного центра, участвующему в образовании ковалентной связи с ДНК: тирозиновые и сериновые
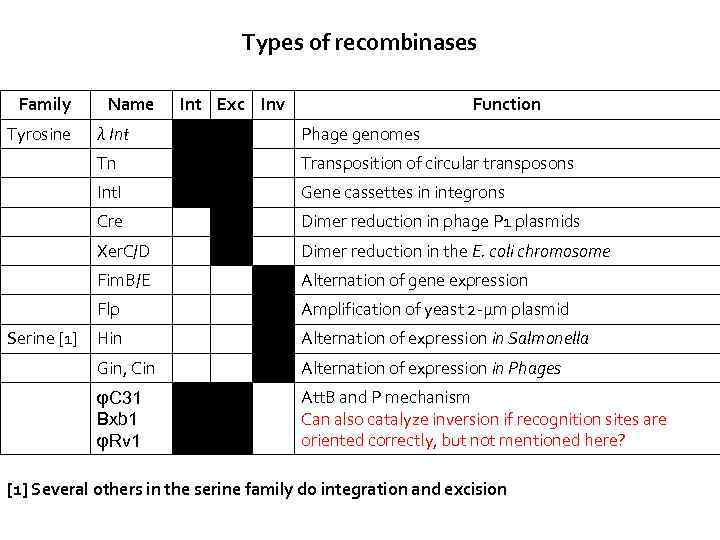
Types of recombinases Family Tyrosine Name Int Exc Inv Function Phage genomes Tn Transposition of circular transposons Int. I Gene cassettes in integrons Cre Dimer reduction in phage P 1 plasmids Xer. C/D Dimer reduction in the E. coli chromosome Fim. B/E Alternation of gene expression Flp Amplification of yeast 2 -μm plasmid Hin Alternation of expression in Salmonella Gin, Cin Alternation of expression in Phages φC 31 Bxb 1 φRv 1 Serine [1] λ Int Att. B and P mechanism Can also catalyze inversion if recognition sites are oriented correctly, but not mentioned here? [1] Several others in the serine family do integration and excision
lecture 5.ppt