2ee4565eaeb8e7e864d8e07fb16aa0ba.ppt
- Количество слайдов: 43

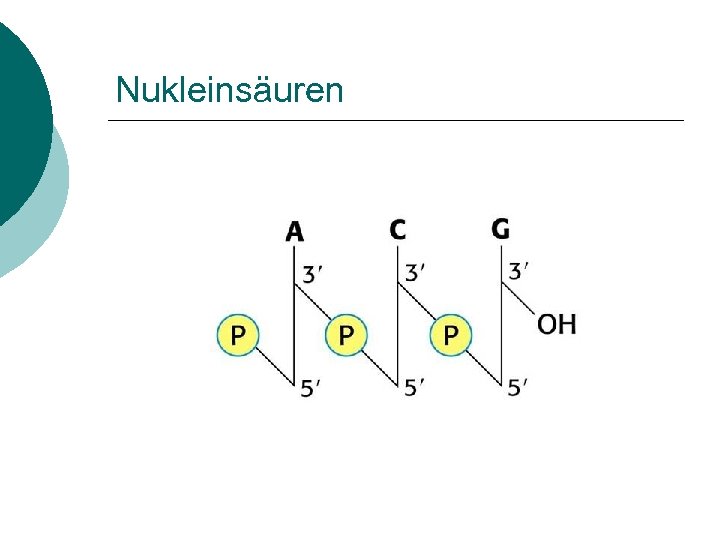
Nukleinsäuren
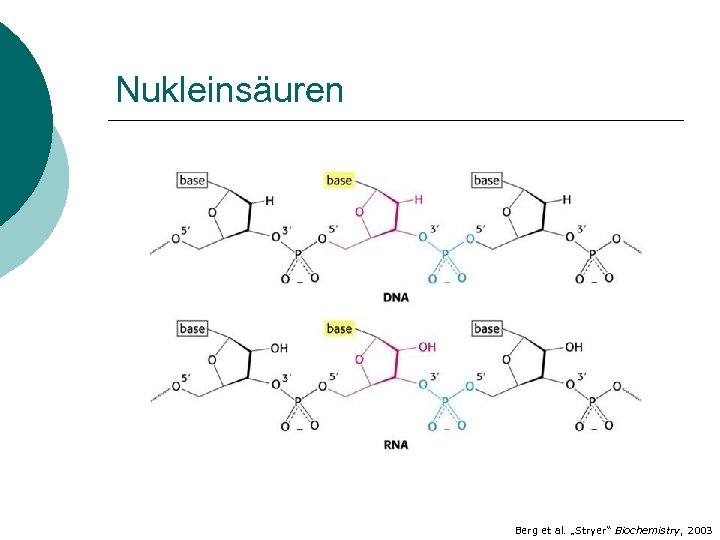
Nukleinsäuren Berg et al. „Stryer“ Biochemistry, 2003
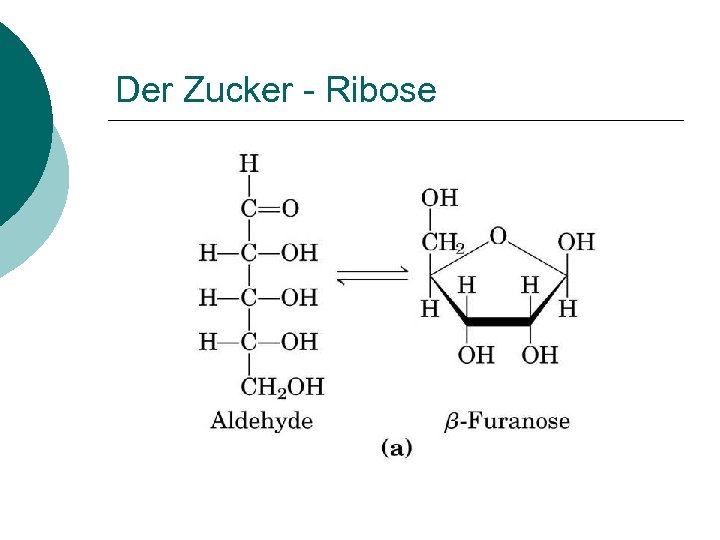
Der Zucker - Ribose
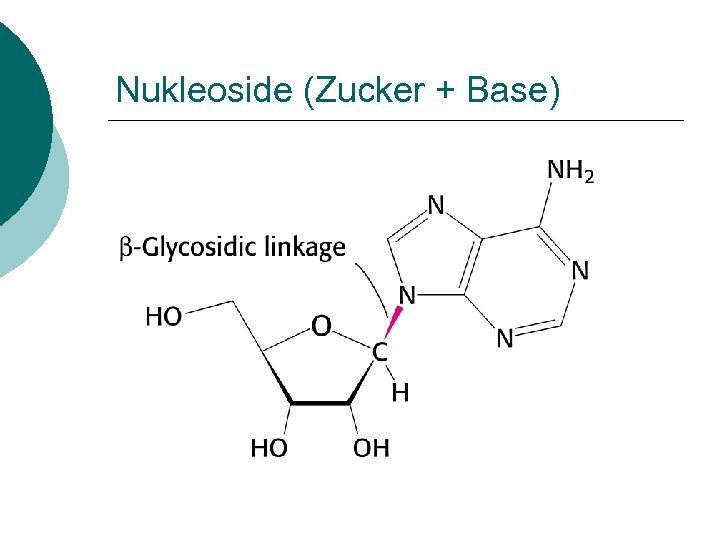
Nukleoside (Zucker + Base)
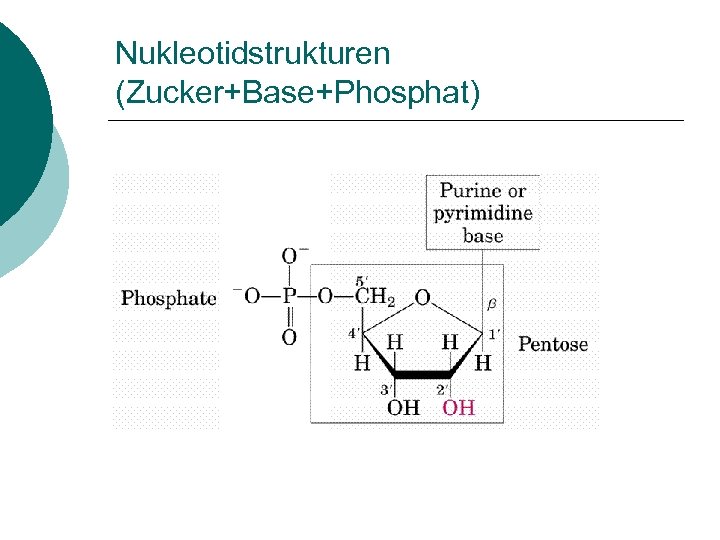
Nukleotidstrukturen (Zucker+Base+Phosphat)
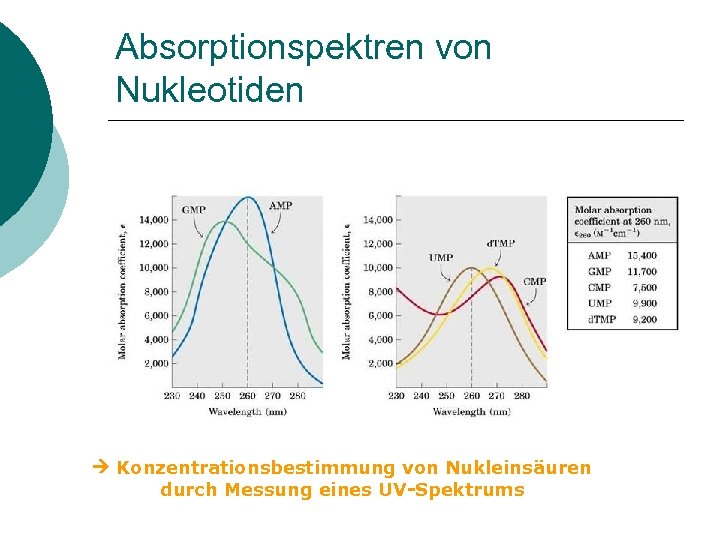
Absorptionspektren von Nukleotiden Konzentrationsbestimmung von Nukleinsäuren durch Messung eines UV-Spektrums
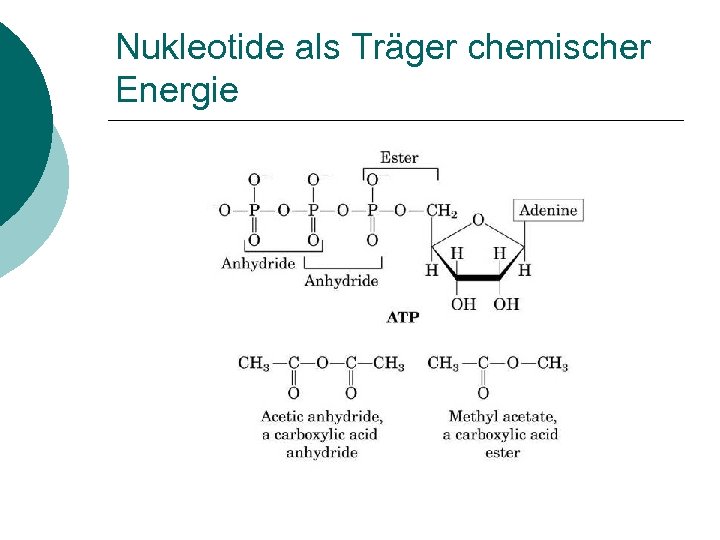
Nukleotide als Träger chemischer Energie
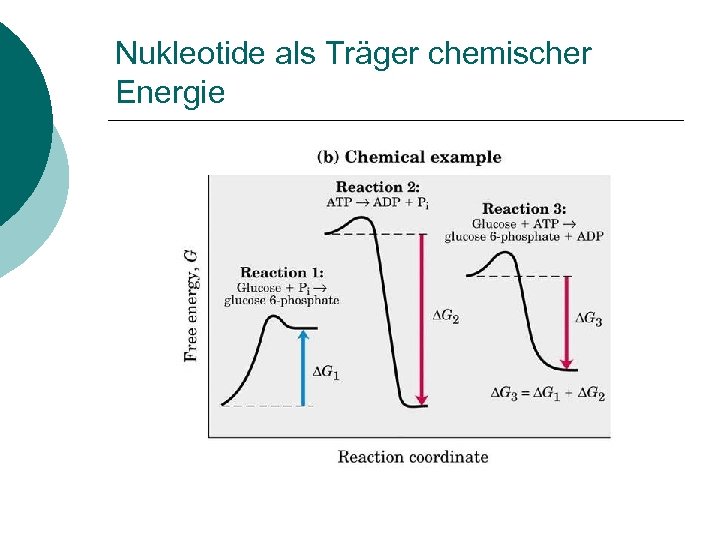
Nukleotide als Träger chemischer Energie
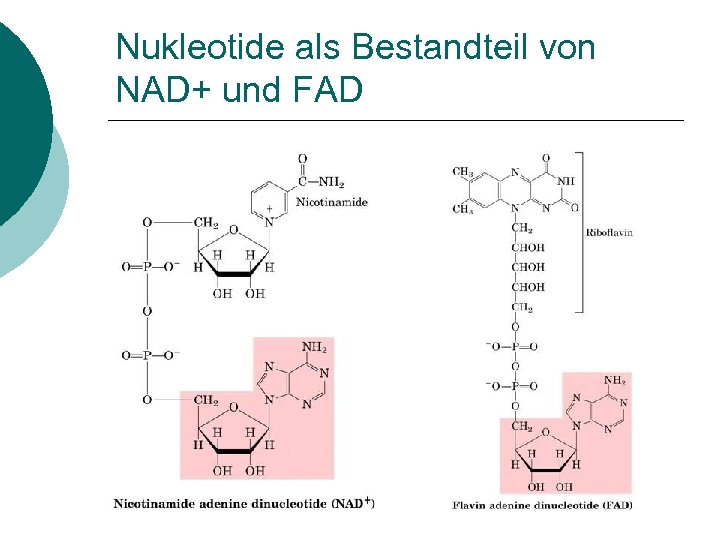
Nukleotide als Bestandteil von NAD+ und FAD
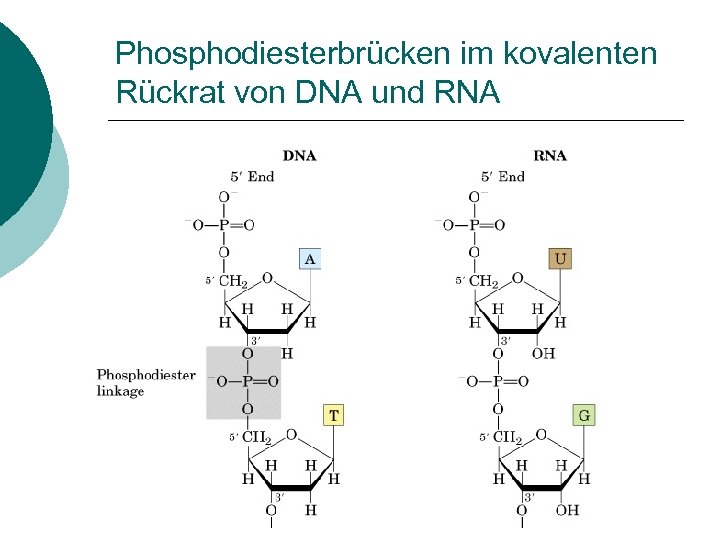
Phosphodiesterbrücken im kovalenten Rückrat von DNA und RNA
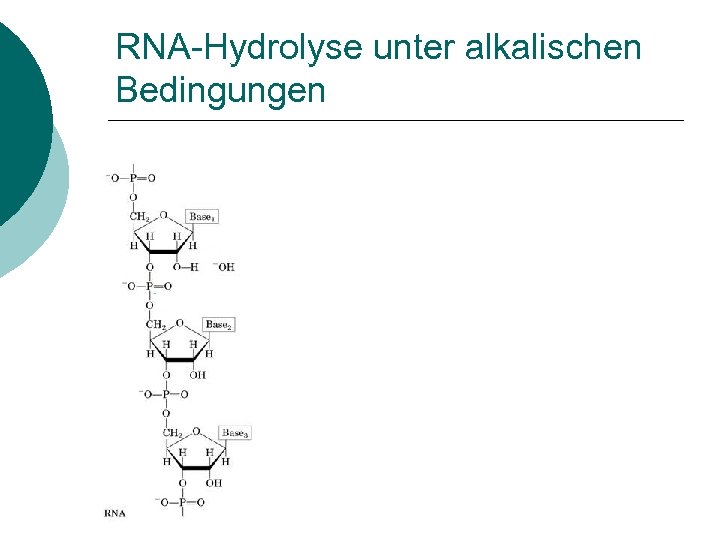
RNA-Hydrolyse unter alkalischen Bedingungen
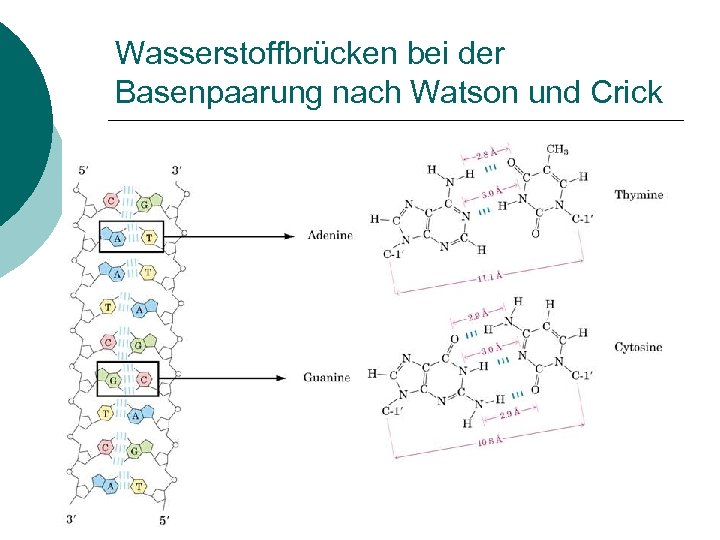
Wasserstoffbrücken bei der Basenpaarung nach Watson und Crick
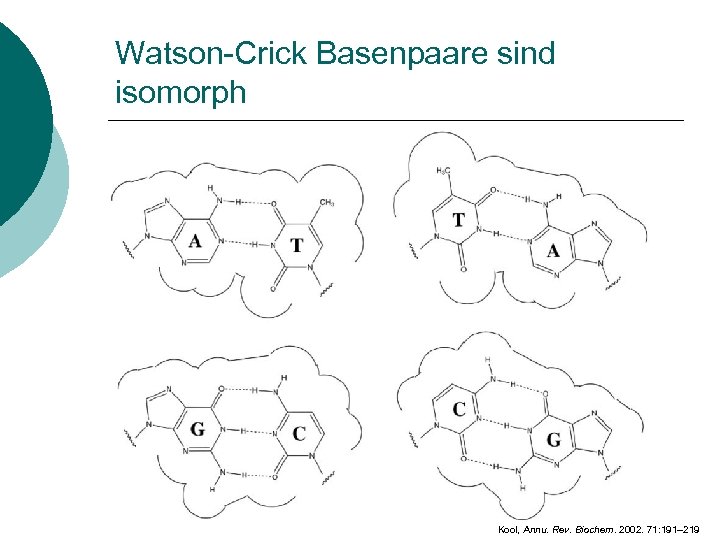
Watson-Crick Basenpaare sind isomorph Kool, Annu. Rev. Biochem. 2002. 71: 191– 219
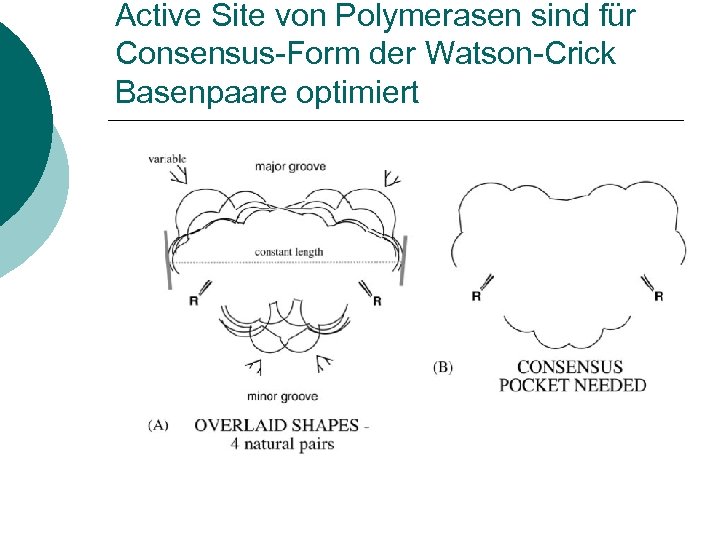
Active Site von Polymerasen sind für Consensus-Form der Watson-Crick Basenpaare optimiert
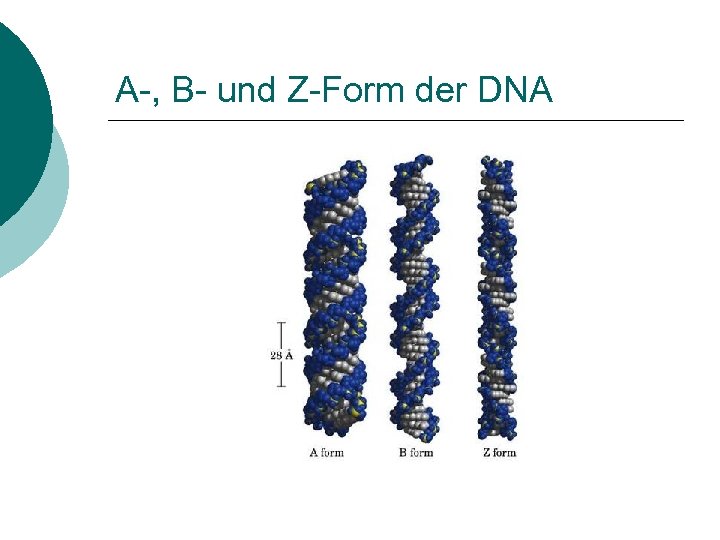
A-, B- und Z-Form der DNA
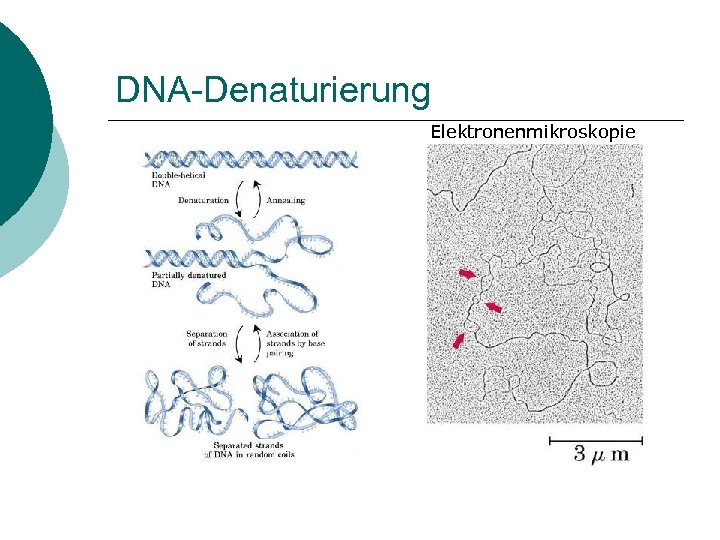
DNA-Denaturierung Elektronenmikroskopie
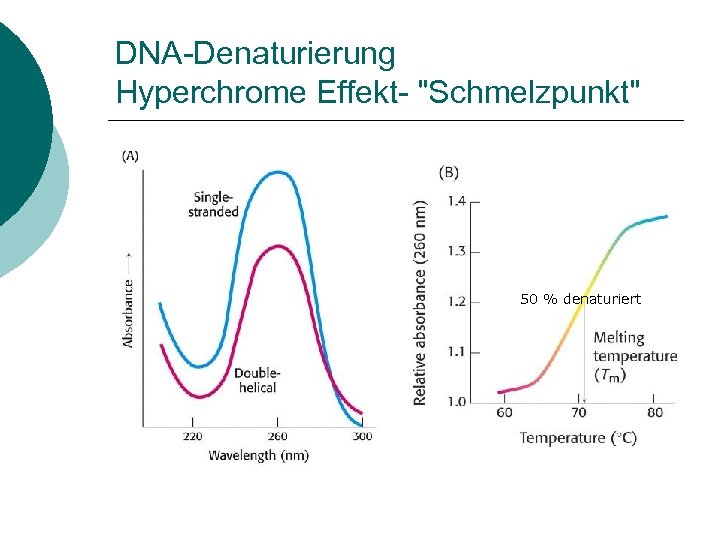
DNA-Denaturierung Hyperchrome Effekt- "Schmelzpunkt" 50 % denaturiert
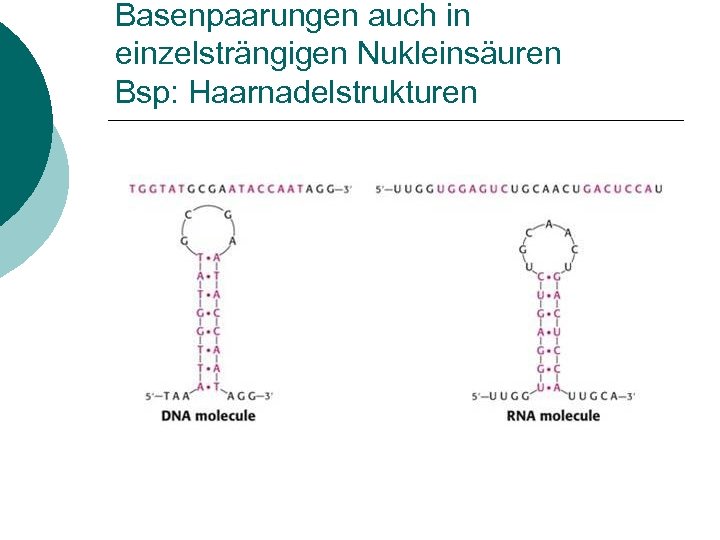
Basenpaarungen auch in einzelsträngigen Nukleinsäuren Bsp: Haarnadelstrukturen
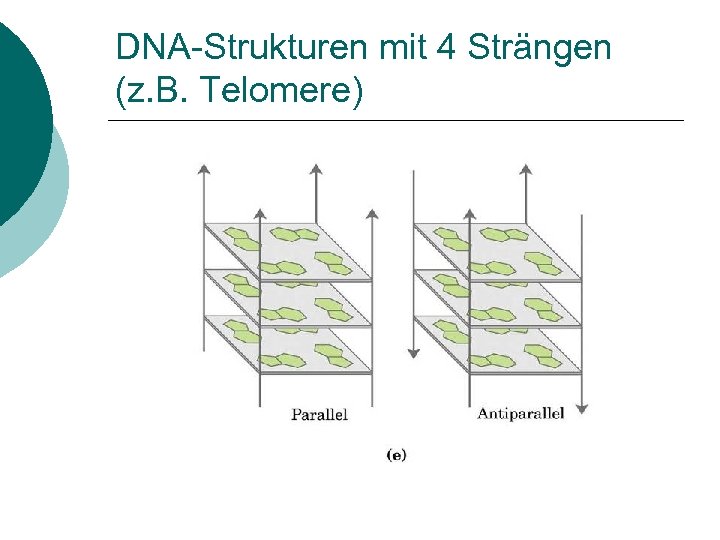
DNA-Strukturen mit 4 Strängen (z. B. Telomere)
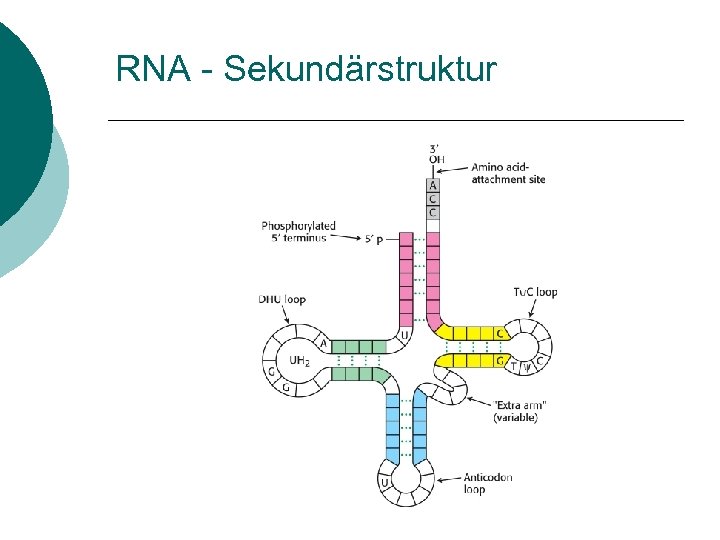
RNA - Sekundärstruktur
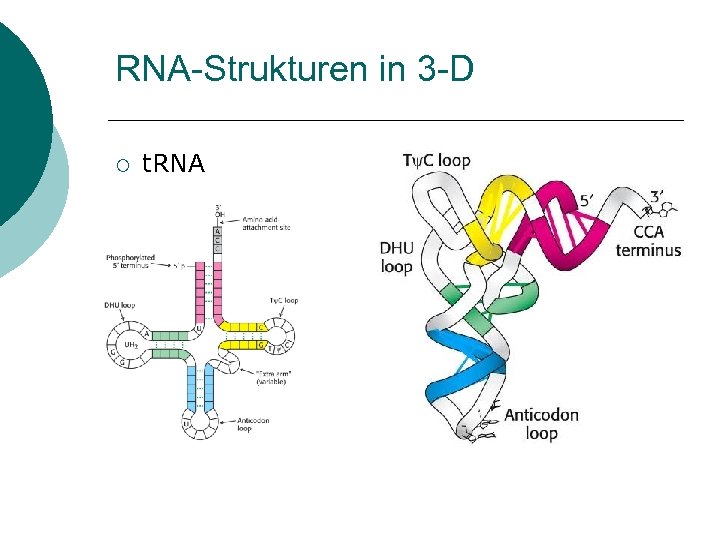
RNA-Strukturen in 3 -D ¡ t. RNA
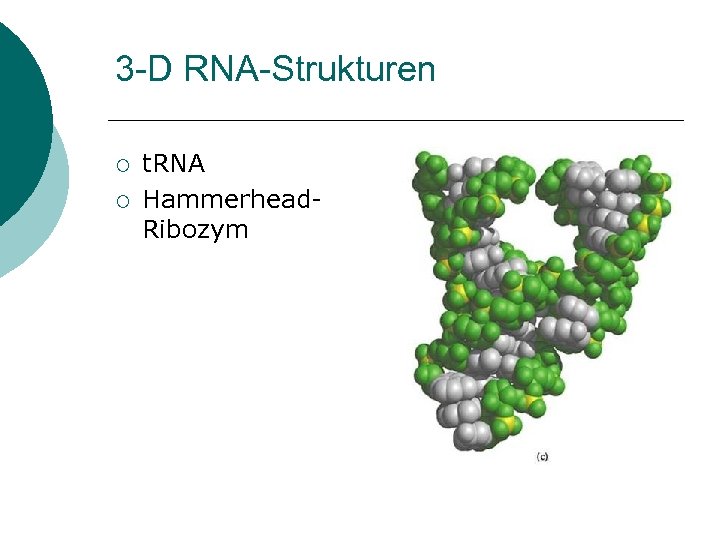
3 -D RNA-Strukturen ¡ ¡ t. RNA Hammerhead. Ribozym
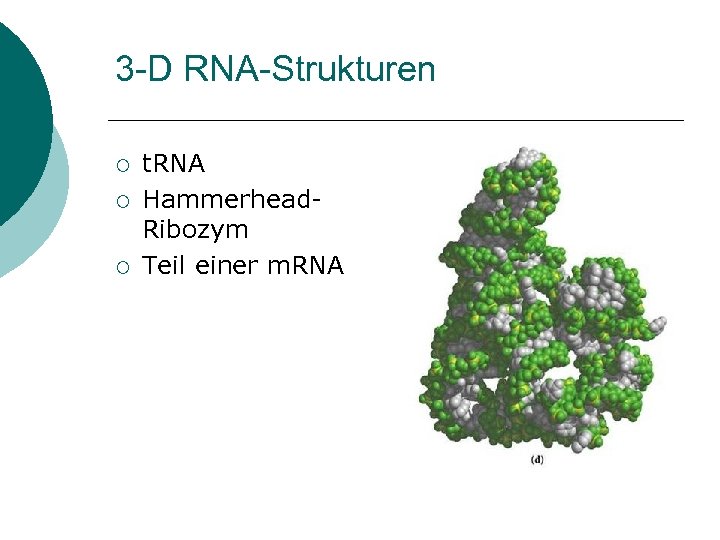
3 -D RNA-Strukturen ¡ ¡ ¡ t. RNA Hammerhead. Ribozym Teil einer m. RNA
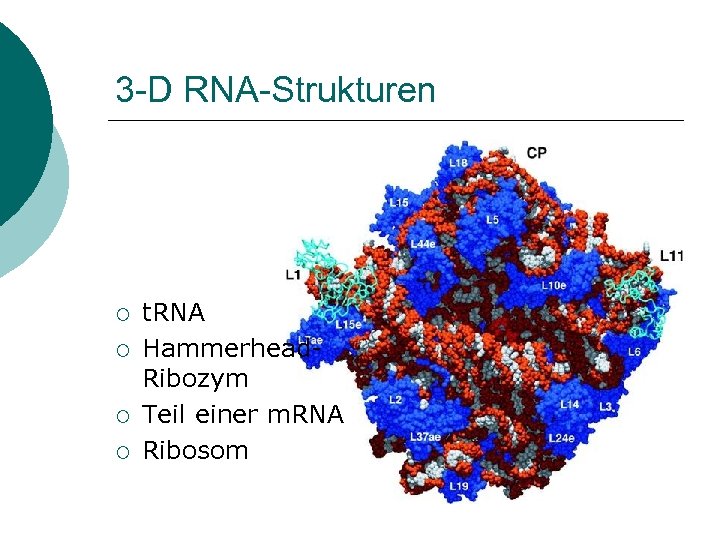
3 -D RNA-Strukturen ¡ ¡ t. RNA Hammerhead. Ribozym Teil einer m. RNA Ribosom
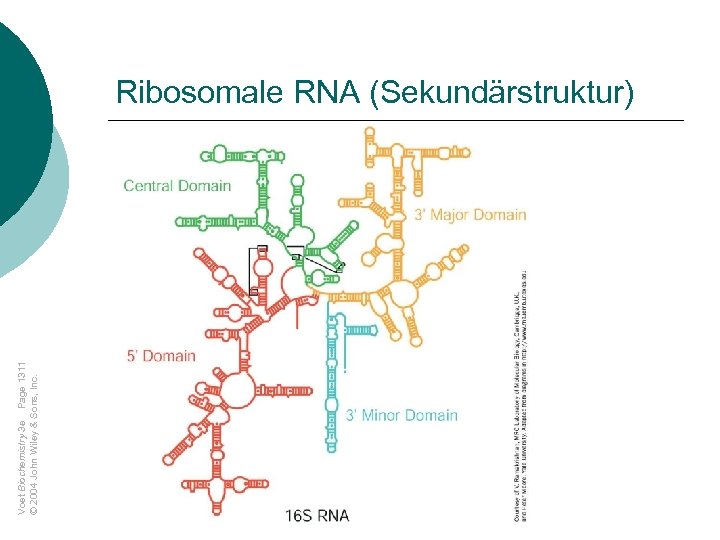
Voet Biochemistry 3 e Page 1311 © 2004 John Wiley & Sons, Inc. Ribosomale RNA (Sekundärstruktur)
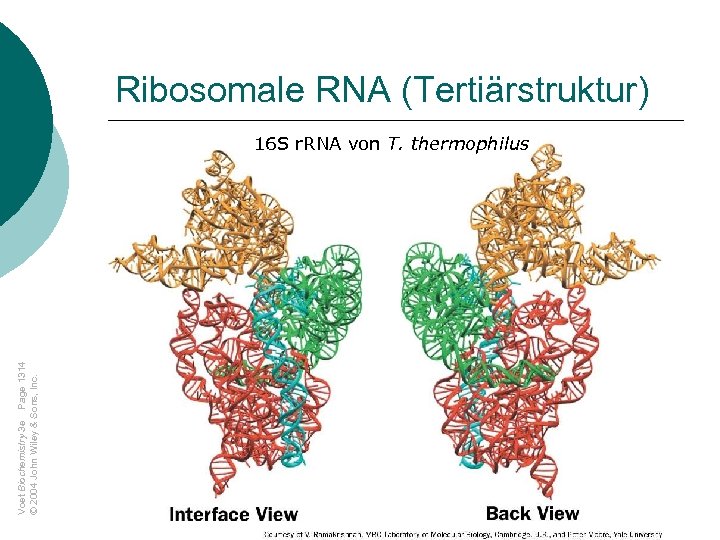
Ribosomale RNA (Tertiärstruktur) Voet Biochemistry 3 e Page 1314 © 2004 John Wiley & Sons, Inc. 16 S r. RNA von T. thermophilus
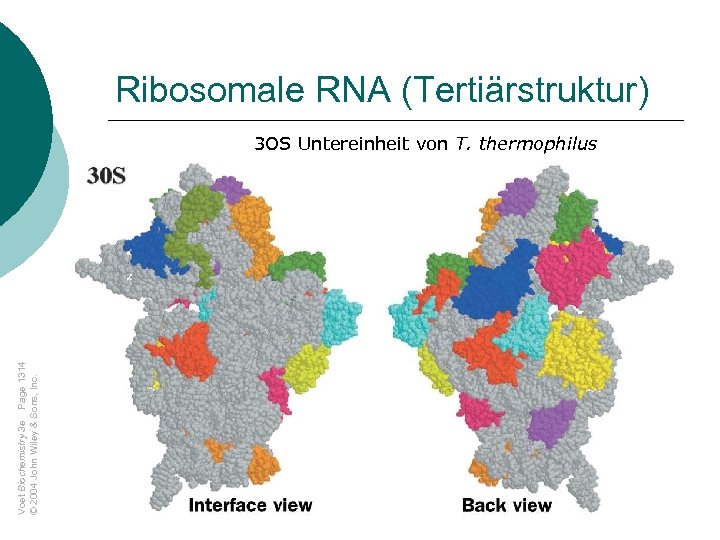
Ribosomale RNA (Tertiärstruktur) Voet Biochemistry 3 e Page 1314 © 2004 John Wiley & Sons, Inc. 3 OS Untereinheit von T. thermophilus
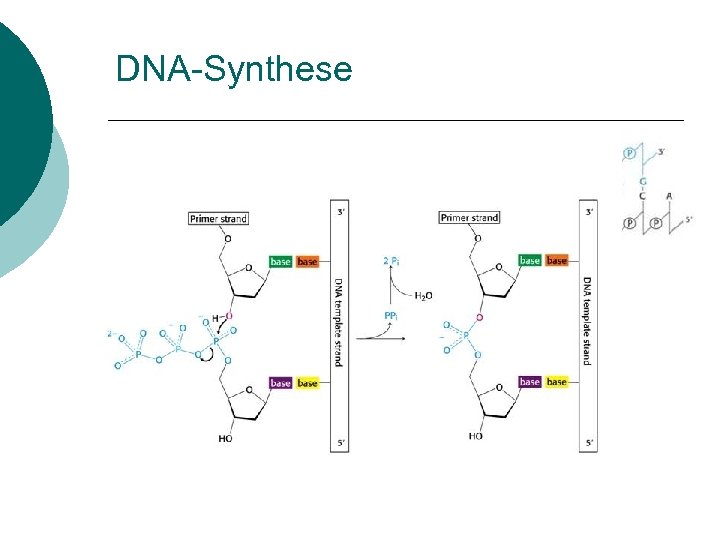
DNA-Synthese
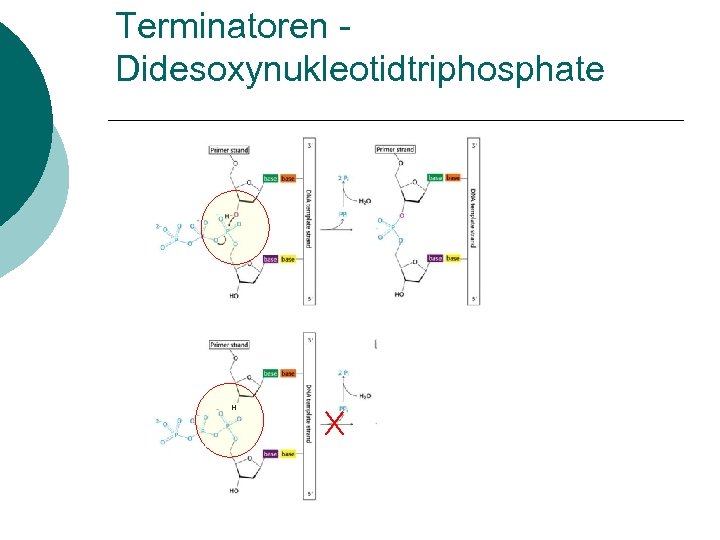
Terminatoren Didesoxynukleotidtriphosphate H
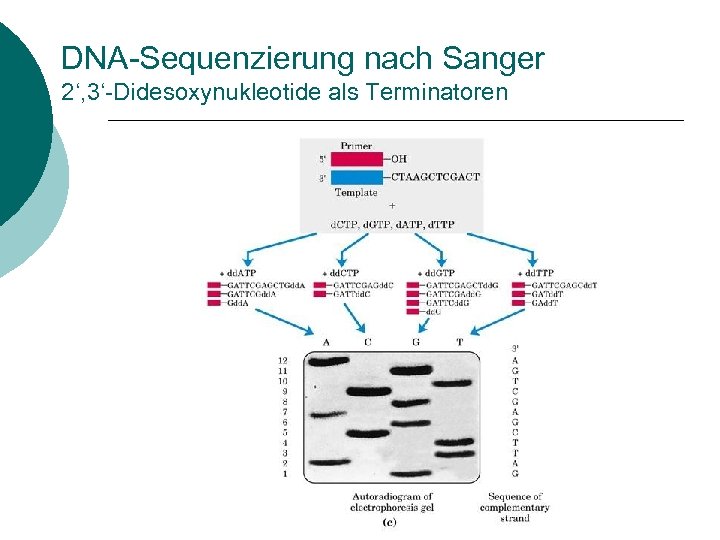
DNA-Sequenzierung nach Sanger 2‘, 3‘-Didesoxynukleotide als Terminatoren
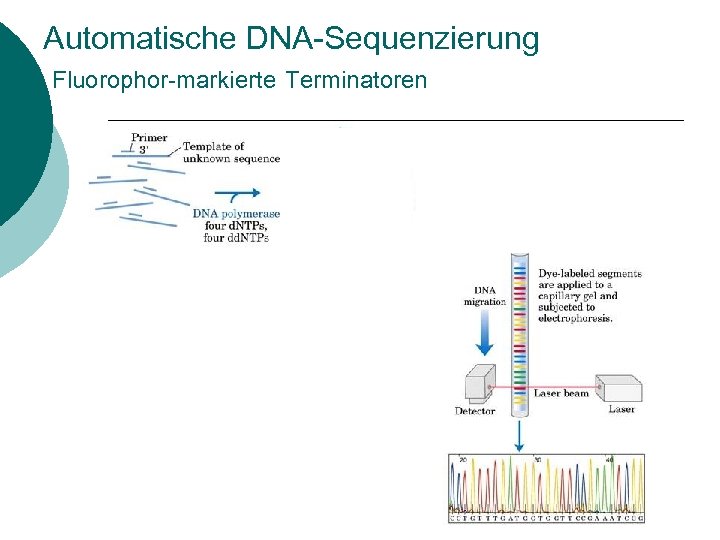
Automatische DNA-Sequenzierung Fluorophor-markierte Terminatoren
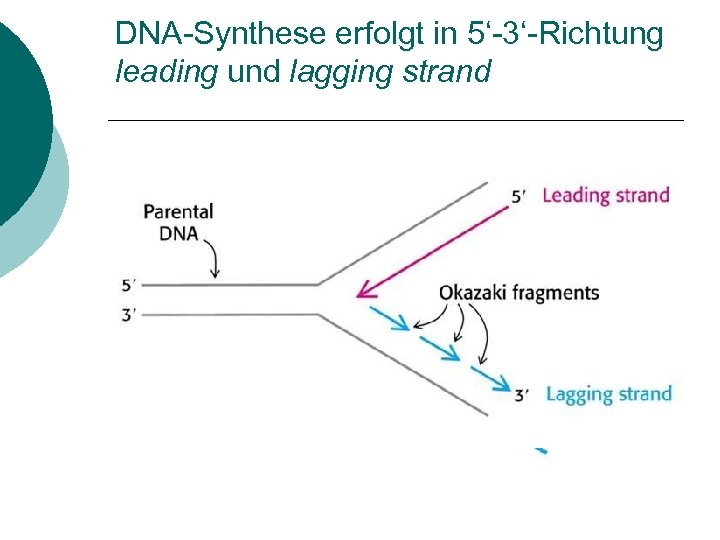
DNA-Synthese erfolgt in 5‘-3‘-Richtung leading und lagging strand
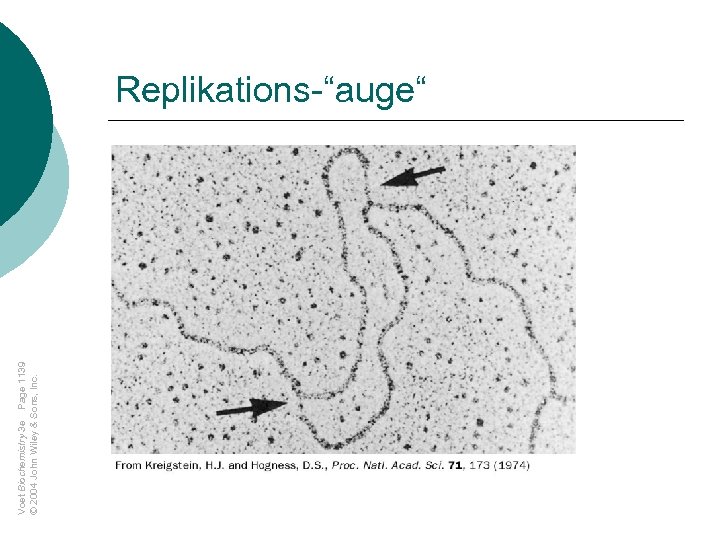
Voet Biochemistry 3 e Page 1139 © 2004 John Wiley & Sons, Inc. Replikations-“auge“
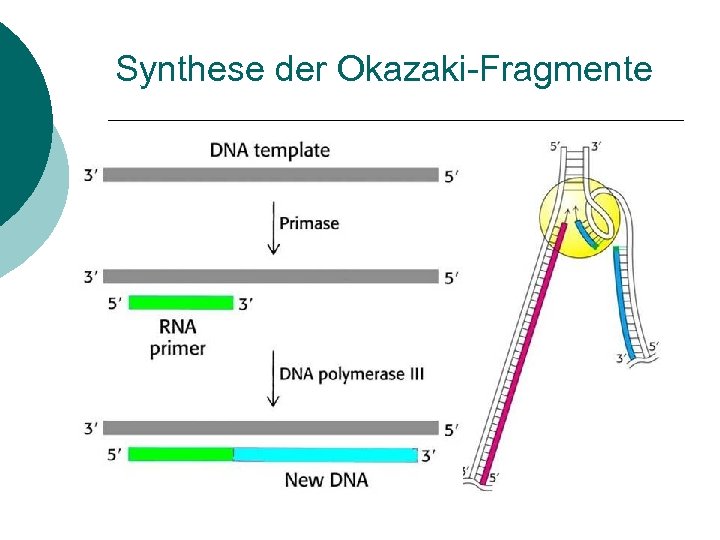
Synthese der Okazaki-Fragmente
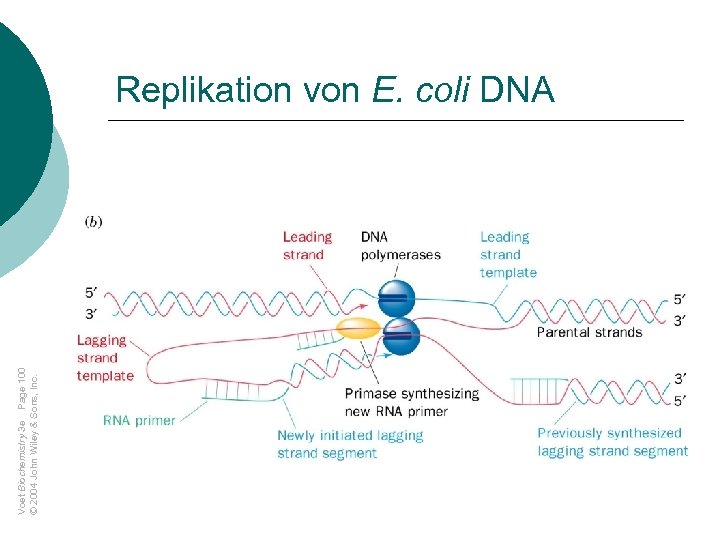
Voet Biochemistry 3 e Page 100 © 2004 John Wiley & Sons, Inc. Replikation von E. coli DNA
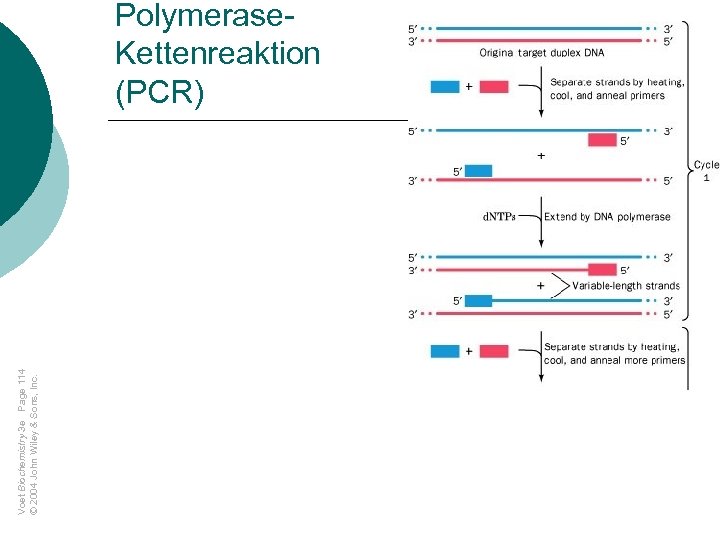
Voet Biochemistry 3 e Page 114 © 2004 John Wiley & Sons, Inc. Polymerase. Kettenreaktion (PCR)
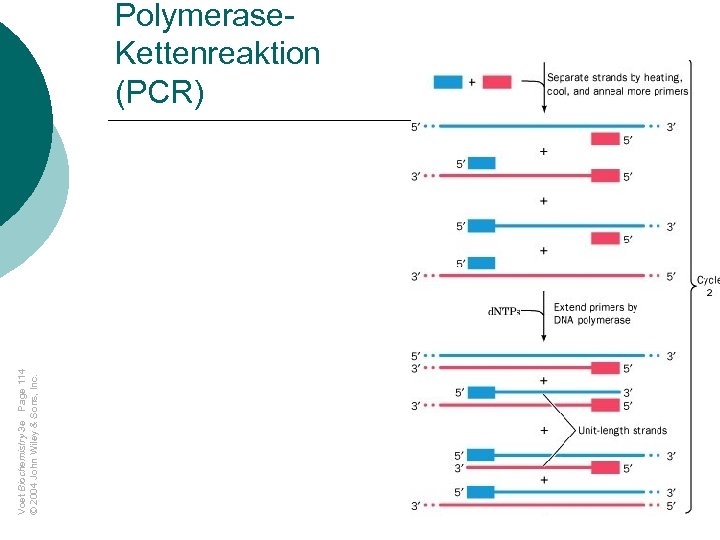
Voet Biochemistry 3 e Page 114 © 2004 John Wiley & Sons, Inc. Polymerase. Kettenreaktion (PCR)
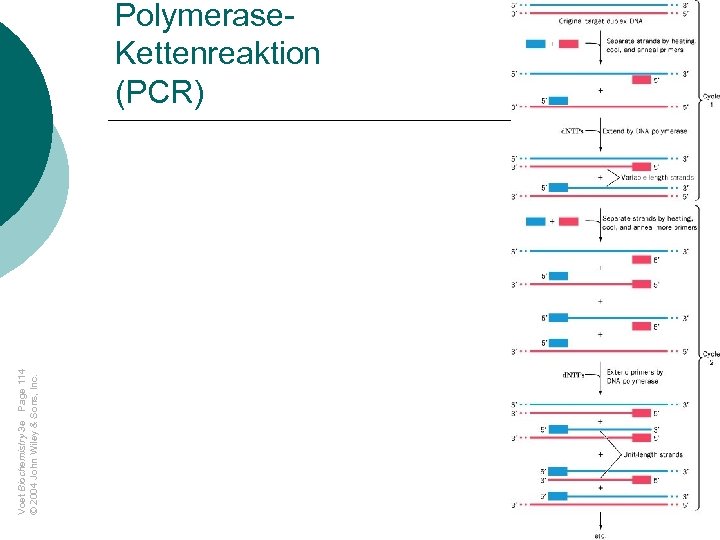
Voet Biochemistry 3 e Page 114 © 2004 John Wiley & Sons, Inc. Polymerase. Kettenreaktion (PCR)

DNA-Replikation versus PCR ¡ Template-DNA ¡ Primer = 3‘-Ende der DNA (leading strand) bzw. RNA -Primer (lagging strand) ¡ Primer = synthetische Oligodesoxynukleodtide ¡ d. NTPs (d. ATP, d. GTP, d. CTP, d. TTP) ¡ z. B. Taq-DNA-Polymerase ¡ thermische Denaturierung des DNA-Doppelstranges ¡ DNA-Polymerase ¡ ATP-abhängige. DNAHelikase entwindet Doppelstrang
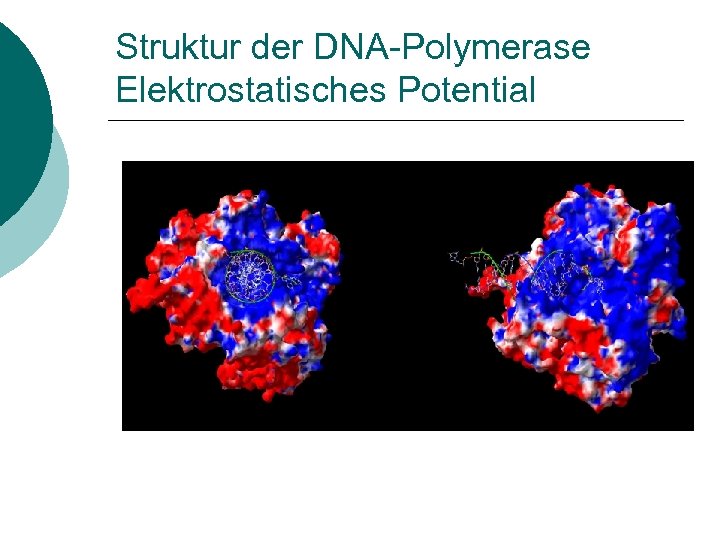
Struktur der DNA-Polymerase Elektrostatisches Potential

Nukleinsäuren und PCR Praktischer Teil ¡ ¡ Amplifikation des Gens eines DNAReparaturproteins mittels PCR Abhängigkeit der Produktmenge von l l ¡ Zyklenzahl der Anwesenheit einer Kompetitors (verkürzte Variante des Gens) Analyse der PCR-Produkte mittels Agarosegelelektrophorese

Nukleinsäuren und PCR Lernziele ¡ ¡ Nukleinsäure (Chemie und Struktur) DNA-Replikation Polymerase-Ketten-Reaktion (PCR) DNA-Sequenzierung
2ee4565eaeb8e7e864d8e07fb16aa0ba.ppt