1db88bf1778e8eb55d3589eef03ffb82.ppt
- Количество слайдов: 63

Негеномные данные М. Гельфанд «Сравнительная геномика» Би. Би 4 курс, Осень 2009

Массовые негеномные данные • • • (ко)-экспрессия Белок-ДНКовые взаимодействия Состав и концентрации белков Белок-белковые взаимодействия Одинарные и двойные мутанты (жизнеспособность, фенотипы) • Всякие разные эксперименты (text mining) • Много баз данных • Мало удобных средств анализа

Expression arrays (a. k. a. олигонуклеотидные чипы) • Олигонуклеотиды (зонды) нанесены на подложку • Туда же льют (радиоактивно или флуоресцентно – можно 2 метки) меченую РНК или к. ДНК (например – один раунд с затравкой поли. Т) – получаем (комплементарные цепи) 3’-концы генов • Меряем в каждой лунке интенсивность – она пропорциональна концентрации

«техническая» биоинформатика • Как подобрать зонды – Геномные соображения • Не участок малой сложности (прилипнет к чему угодно) • Не повтор (прилипнет ко мноим копиям) • Уникальность (семейства генов) – Физические соображения • • • Одинаковые температуры плавления Отсутствие комплементарности между зондами Отсутствие вторичной структуры в олигонуклеотиде Отсутствие вторичной структуры в мишени Как переделать интенсивности в концентрации (анализ изображений) Нормировка измерений в одном эксперименте / однородной серии экспериментов – – – Учет различий в условиях гибридизации и т. п. Учет неравномерности по полю Вычитание фона Нормировка интенсивностей флуоресцентных меток (регрессия) Логарифмы, потому что хотим • • нормального распределения ошибок приблизительно равномерного разброса наблюдений по шкале приблизительно колообразного распределения наблюдений вычитать, а не делить при анализе (статистические методы так лучше работают)
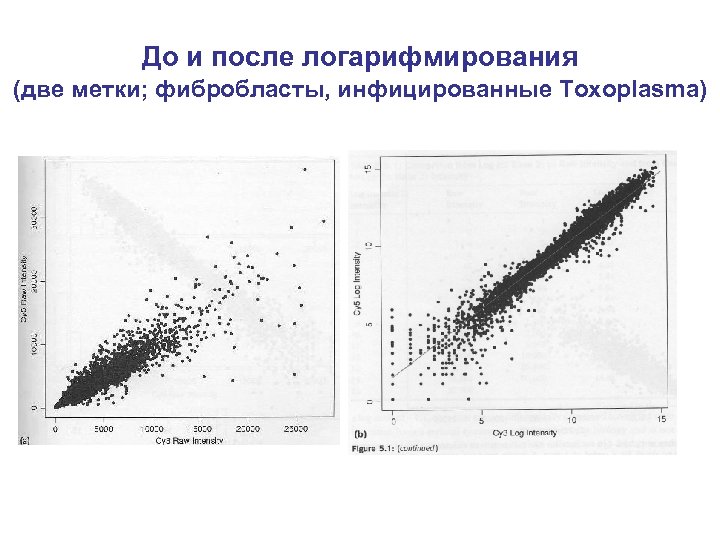
До и после логарифмирования (две метки; фибробласты, инфицированные Toxoplasma)
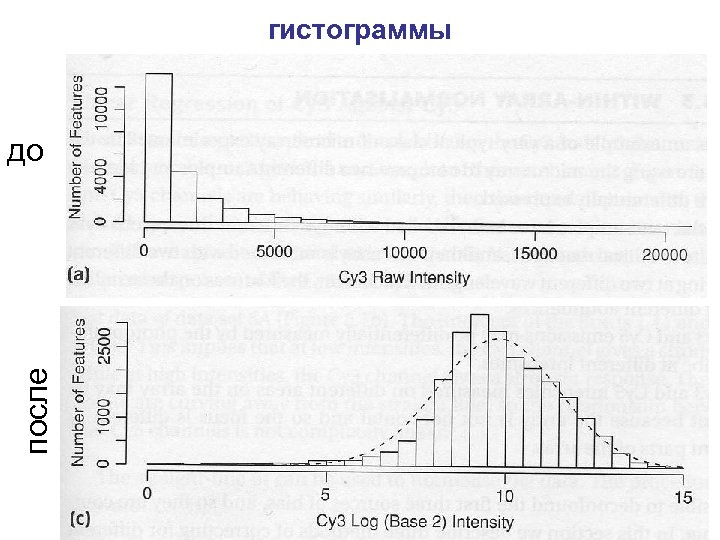
гистограммы после до

Виды чипов • к. ДНКовые – ПЦР-амплификация известных генов – (можно сколь угодно либерально – брать все сомнительные гены) – Надо учитывать альтернативный сплайсинг, полиаденилирование, промоторы (трудно) – В зависимости от способа приготовления меченой к. ДНК, можно предпочитать 3’-концы • Выстилающие (tiling) – Весь геном – по ~30 нуклеотидов со сдвигом ~10 нуклеотидов… – … за исключением повторов, микросателлитов и т. п.

Нормировка • Для применения статистических методов надо, чтобы распределения были сравнимы (одинаковые средние и среднеквадратичные отклонения) • для этого для каждого массива (первоначальных) данных вычитают среднее и делят на среднеквадратичное отклонение • у получившегося распределения среднее равно 0, среднеквадратичное отклонение равно 1

Типичные задачи • классификация измерений (например, диагностика) • выбор генов, дифференциально экспрессирующихся в двух классах (диагностика, до/после) • поиск групп ко-регулируемых генов

один ген, много измерений в двух независимых группах – одинаков ли уровень экспрессии? • стандартные методы сравнения двух распределений – параметрические (t-тест) – предполагается нормальность распределений, но: • выборки неоднородные, • распределения не только не нормальные, но могут быть бимодальные, • Outliers => ложно-положительные результаты (принимаем желаемое за действительное) – непараметрические (Уилкоксона-Манна-Уитни) • небольшая статистическая сила => ложно-отрицательные результаты (можно пропустить значимый эффект)

bootstrapping • берем те же данные, распределяем случайным образом по группам того же объема, и проводим такой же тест на сравнение распределений (например, вычисляем t-статистику) • но теперь не смотрим в таблицу tраспределения, а производим эту операцию очень много раз и получаем распределение статистики на имеющихся данных • и смотрим, какова же значимость

один ген, много пар измерений – есть ли разница? • вычитаем первое измерение из второго и проверяем «среднее=0? » • те же тесты, что и раньше

множественное тестирование • статистическая значимость 1% означает, что в случайных данных 1 из 100 измерений (генов) будет иметь такое значение • тысячи генов • консервативный подход – поправка Бонферрони (умножать уровень значимости на количество генов) – слишком консервативно – ничего не значимо

bootstrapping • альтернативный подход – посчитать, сколько генов пройдет данный порог на p-value в случайных данных того же объема • эта доля генов – false positives • выбрать уровень значимости с приемлемым уровнем false positives • поправка Бонферрони соответствует 0% false positives

классификация выборок (тканей, диагнозов и т. п. ): распознавание образов • k ближайших соседей • перцептрон и линейный дискриминантный анализ: измерения (скажем, пациенты) – точки в многомерном пространстве (координаты – уровни экспрессии генов); проводим плоскость, которая наилучшим образом разделяет выборки • нейронный сети (обобщение) • квадратичный анализ • support vector machines (другое обобщение)

понижение размерности и over-interpretation • можно обойтись меньшим числом измерений (гены-маркеры) • не следует трактовать различия в уровне экспрессии как непосредственные причины (скажем, болезни) • можно выбрать другой набор генов, который даст такое же качество диагноза

валидация • стандартные правила гигиены теории распознавания образов • обучающая выборка – подбор параметров • тестирующая выборка – подбор модели • экзамен – ОДИН РАЗ
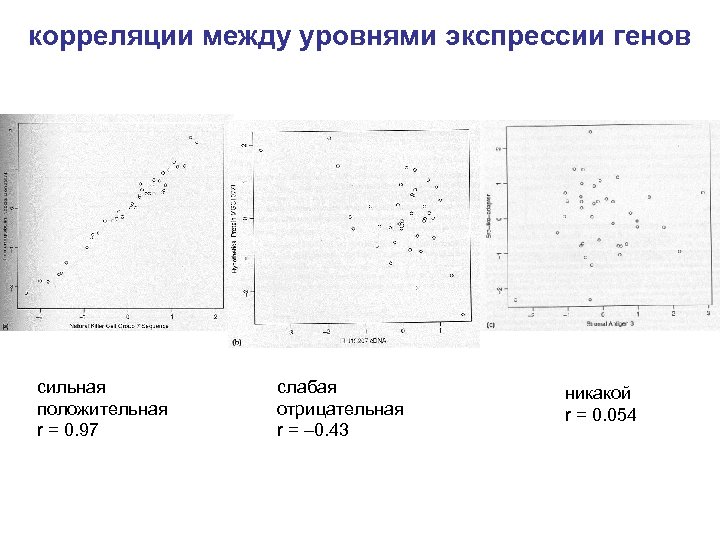
корреляции между уровнями экспрессии генов сильная положительная r = 0. 97 слабая отрицательная r = – 0. 43 никакой r = 0. 054

временные ряды • клеточный цикл (синхронизированные культуры) или последствия вмешательства • измерения уровней экспрессии через фиксированные отрезки времени • кластеризация полученных профилей экспрессии
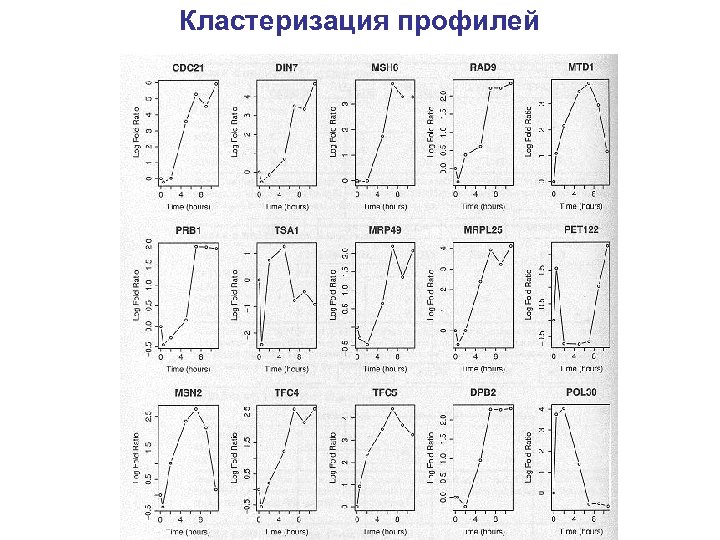
Кластеризация профилей
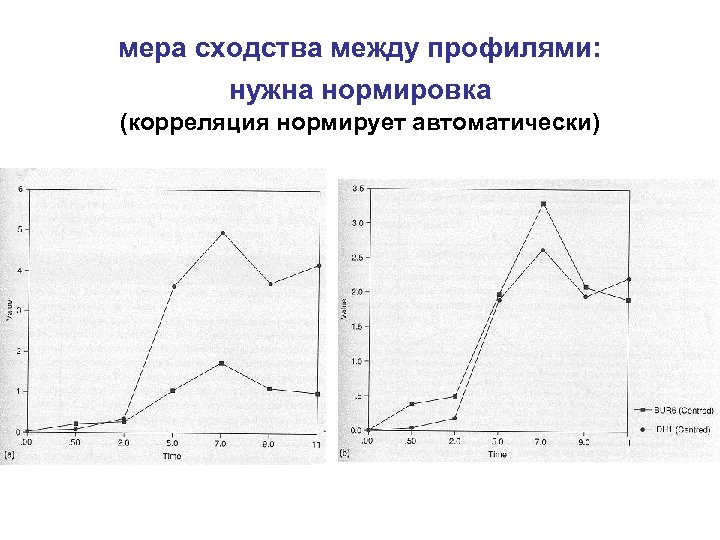
мера сходства между профилями: нужна нормировка (корреляция нормирует автоматически)
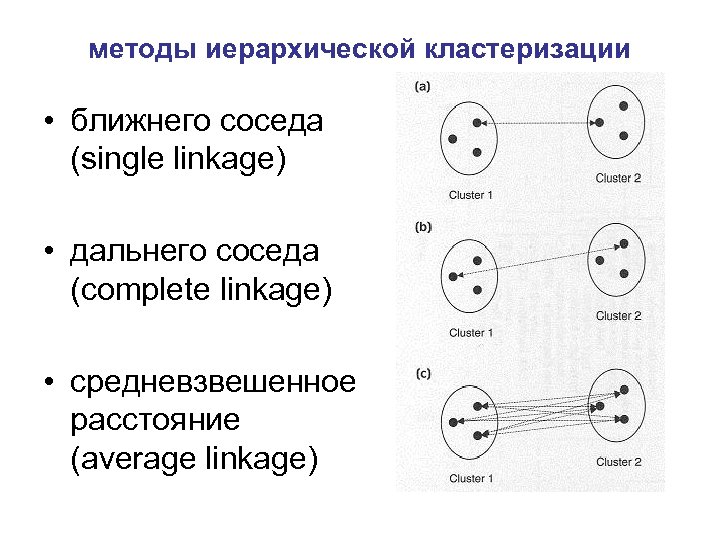
методы иерархической кластеризации • ближнего соседа (single linkage) • дальнего соседа (complete linkage) • средневзвешенное расстояние (average linkage)

мера близости – корреляция, метод кластеризации – среднеевзвешенное

кластеризация k-средних k-means clustering • • количество кластеров задается заранее создать случайные кластеры найти центроиды перераспределить точки – отнести точку к тому кластеру, к центроиду которого эта точка ближе • повторить пока не сойдется • не получается иерархической системы: система кластеров при k+1 не сводится к разделению одного из k кластеров на два

разнообразные условия – двойная кластеризация (biclustering) • если по столбцам – не времена, а различные условия (да еще и из разных экспериментов), то не обязательно корреляция должна наблюдаться на всем множестве условий • двойная кластеризация: выделение групп генов и групп условий, т. ч. экспрессия этих генов скоррелирована в этих условиях

базы данных результатов экспериментов по анализу экспрессии • Array. Express http: //www. ebi. ac. uk/microarray/Array. Express/arrayxpress. html • Stanford Microarray Database http: //genome-www 5. stanford. edu/Micro. Array/NDEV/index. shtml • GEO (Gene Expression Omnibus) http: //www. ncbi. nlm. nih. gov/geo/ • Стандартизованная форма данных об эксперименте (MIAME: Minimal Iinformation About a Microarray Experiment)
GEO
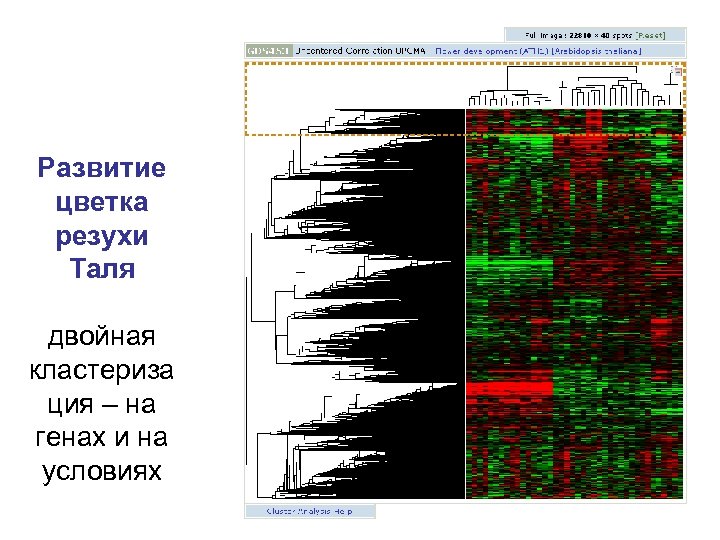
Развитие цветка резухи Таля двойная кластериза ция – на генах и на условиях
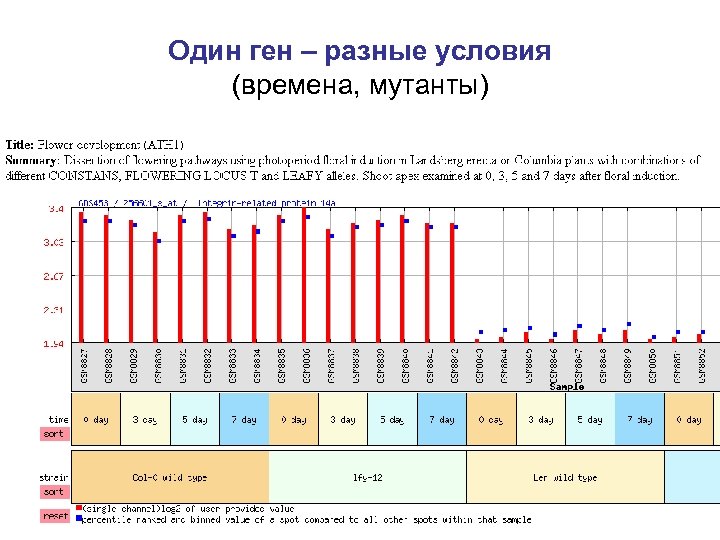
Один ген – разные условия (времена, мутанты)

программы • R (GNU) • Matlab • Expression Profiler (EBI) http: //www. ebi. ac. uk/microarray/Expression. Profiler/ep. html

Expression Profiler

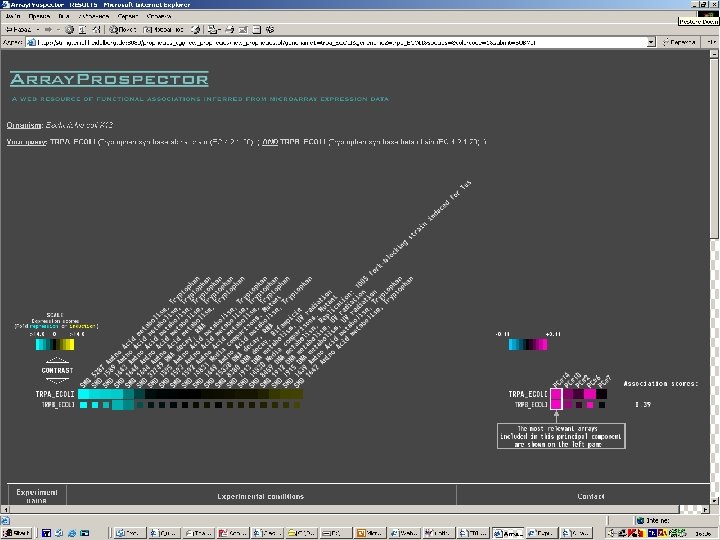
http: //string. embl-heidelberg. de: 8080/ prophecies_html/prophecies. html
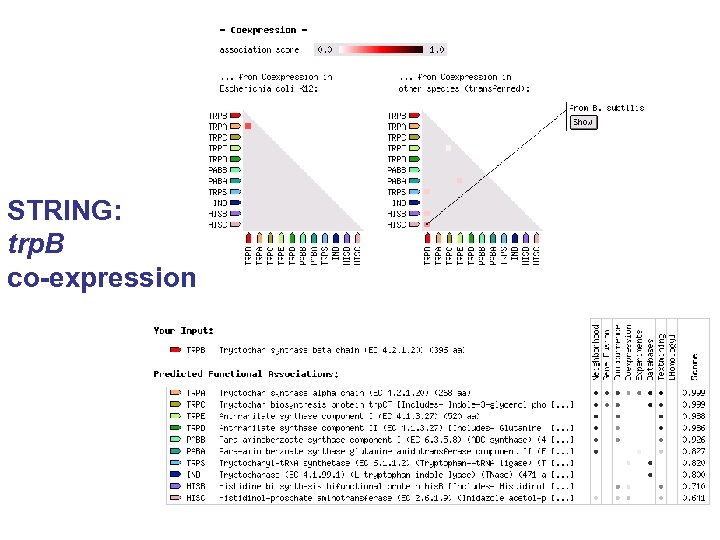
STRING: trp. B co-expression

протеомика – двумерный форез • двумерный форез – пятна (масса/заряд) • масс-спектрометрия – пептиды • в обоих случаях нужен геном. Для фореза – точные гены (старты! точнее даже белки без сигнальных пептидов)
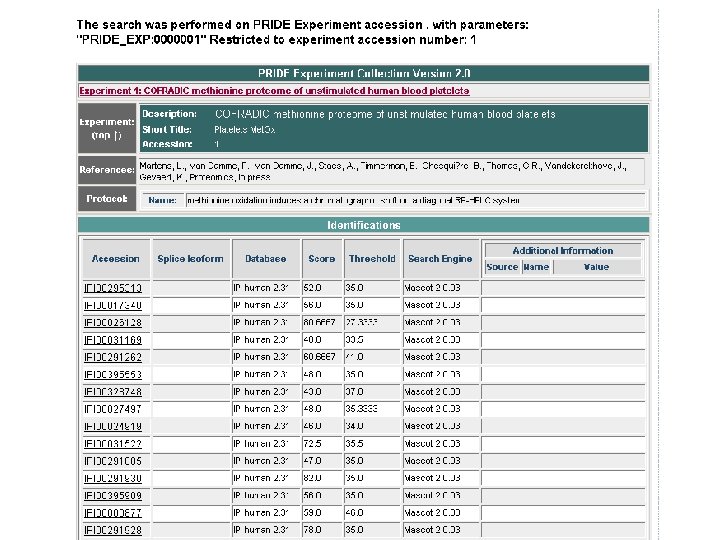
Pride

протеомика – белковые чипы • белки наносятся на подложку, потом можно измерять активность (например, связывание)

белок-белковые взаимодействия • комплексы – масс-спектрометрия • дрожжевые двугибридные системы – GAL 4 – два домена: димеризационный (связывает галактозу) и ДНК-связывающий – димер связывается с оператором – димеризационный и ДНК-связывающий домены могут быть в разных белках (связь через тестируемые белки)

Protein-protein interactions • MIPS, mammals: http: //mips. gsf. de/proj/ppi/ • MIPS, yeast: http: //mips. gsf. de/ proj/yeast/CYGD/interaction/ • MRC, links: http: //www. hgmp. mrc. ac. uk/ Genome. Web/prot-interaction. html • DIP, many model organisms: http: //dip. doe-mbi. ucla. edu/ dip/Main. cgi
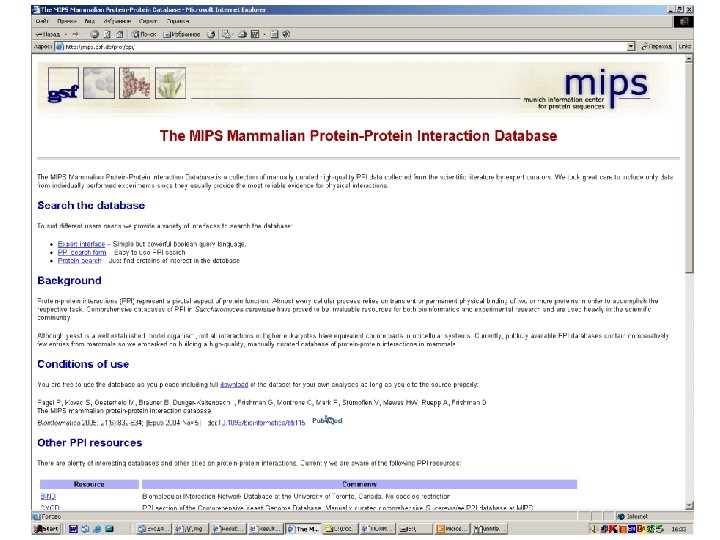
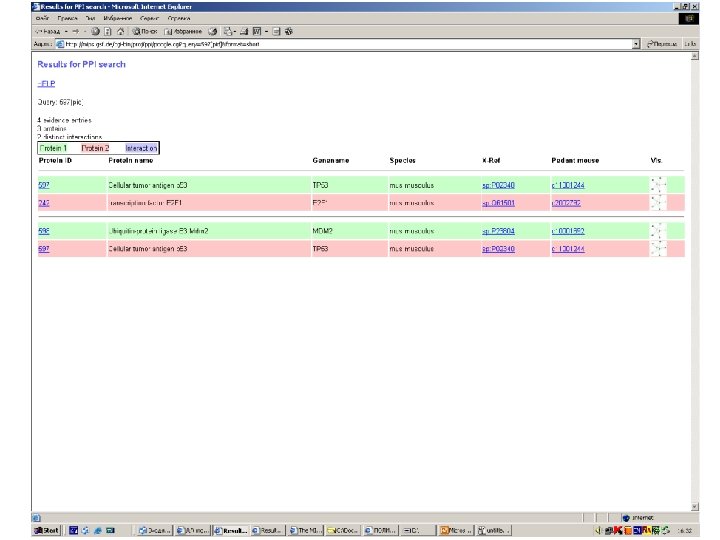
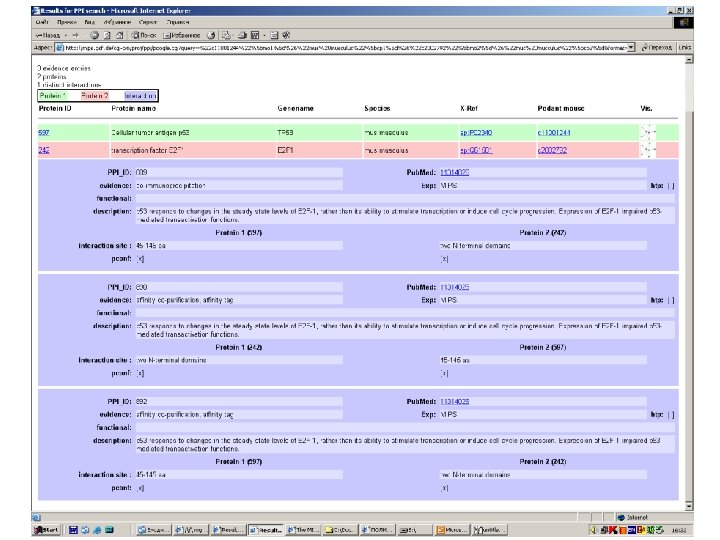
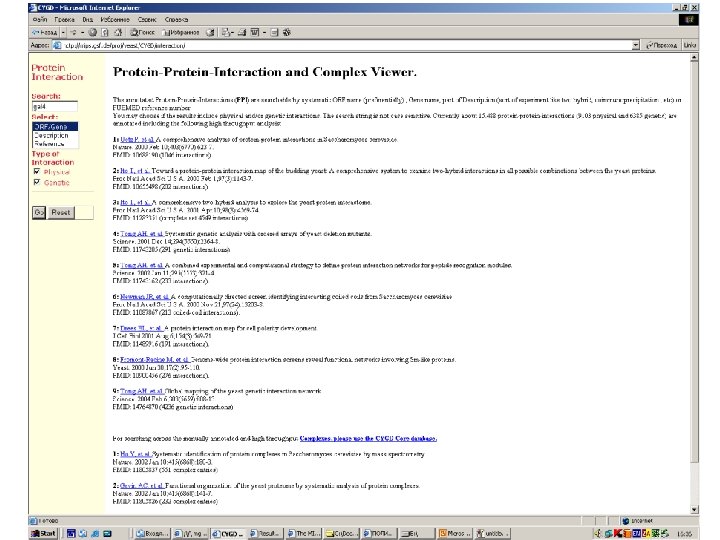
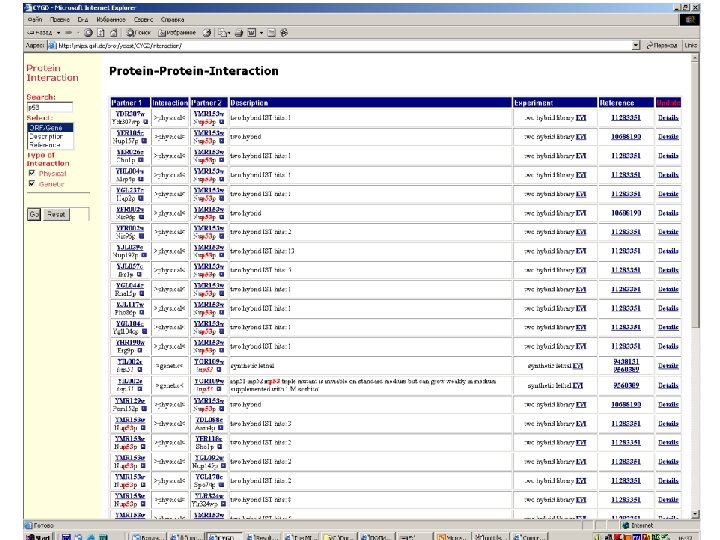
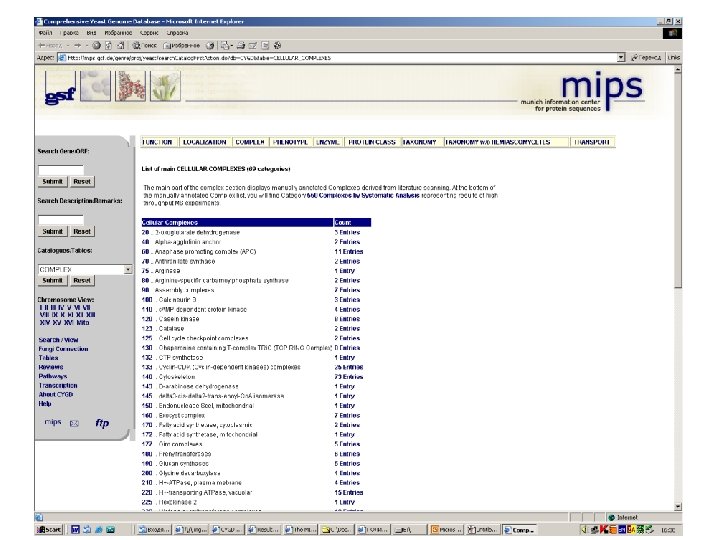

STRING: trp. B highthroughput experiments
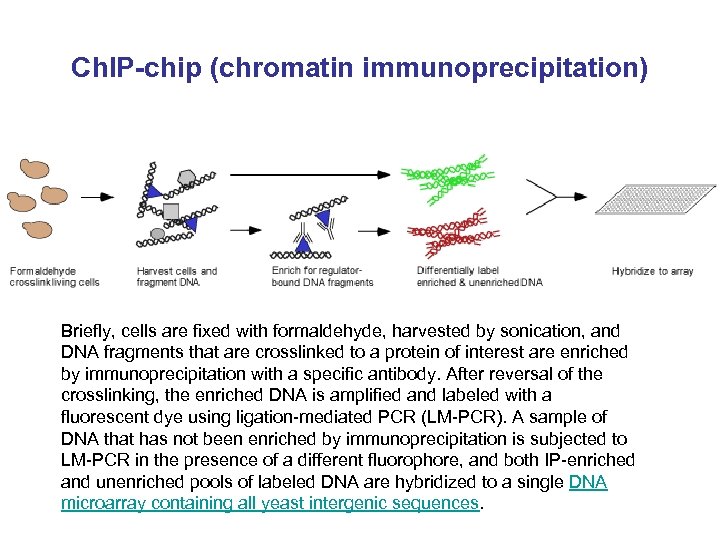
Ch. IP-chip (chromatin immunoprecipitation) Briefly, cells are fixed with formaldehyde, harvested by sonication, and DNA fragments that are crosslinked to a protein of interest are enriched by immunoprecipitation with a specific antibody. After reversal of the crosslinking, the enriched DNA is amplified and labeled with a fluorescent dye using ligation-mediated PCR (LM-PCR). A sample of DNA that has not been enriched by immunoprecipitation is subjected to LM-PCR in the presence of a different fluorophore, and both IP-enriched and unenriched pools of labeled DNA are hybridized to a single DNA microarray containing all yeast intergenic sequences.

фенотипы • essentiality – 18. 7% non-essential genes in yeast – 14. 4% non-essential genes in E. coli – … в «богатых» условиях • RNAi • synthetic lethals
MIPS

Как это используют • Так же, как любые слабые соображения
![Ингибитор РНКазы L [Huynen, Gabaldon] • COG 1245 – присутствует во всех эукариотах и Ингибитор РНКазы L [Huynen, Gabaldon] • COG 1245 – присутствует во всех эукариотах и](https://present5.com/presentation/1db88bf1778e8eb55d3589eef03ffb82/image-55.jpg)
Ингибитор РНКазы L [Huynen, Gabaldon] • COG 1245 – присутствует во всех эукариотах и археях • У человека RPLI 1 – ингибитор РНКазы L (интерферонзависимый путь деградации (вирусных) РНК) • Но РНКазы L у архей нет. Что же там делает этот белок?

• Филогенетический профиль (присутствие во всех археях и эукариотах, отсутствие во всех бактериях) => 55 COGов – Трансляция, биогенез рибосом, транскрипция, репликация, рекомбинация, репарация • В эукариотах – коэкспрессия с рибосомными белками процессинга р. РНК • В дрожжах – белок-белковое взаимодействие с HCR 1 (процессинг р. РНК) • Эксперимент (трипаносома): слабая экспрессия RLI 1 => уменьшение общего уровня синтеза белков • N-концевой домен содержит 4 цистеина – связывание с РНК? • Предсказание: RLI 1 – фактор процессинга р. РНК • Эксперимент: и впрямь – мутанты по RLI 1 имеют дефект процессинга пре-р. РНК – в дрожжах RLI 1 связан с незрелыми и зрелыми малыми субъединицами рибосом
![Систематический анализ генов дрожжей [Kemmeren et al. 2005] • Белок-белковые взаимодействия • Корреляция профилей Систематический анализ генов дрожжей [Kemmeren et al. 2005] • Белок-белковые взаимодействия • Корреляция профилей](https://present5.com/presentation/1db88bf1778e8eb55d3589eef03ffb82/image-57.jpg)
Систематический анализ генов дрожжей [Kemmeren et al. 2005] • Белок-белковые взаимодействия • Корреляция профилей экспрессии • Ко-локализация • Сходство фенотипов

KRE 33 “killer toxin resistant”, no GO annotation • 20 связанных генов • 13: часть комплекса U 3 sno. RNP (процессинг р. РНК) • 4: метаболизм м. РНК
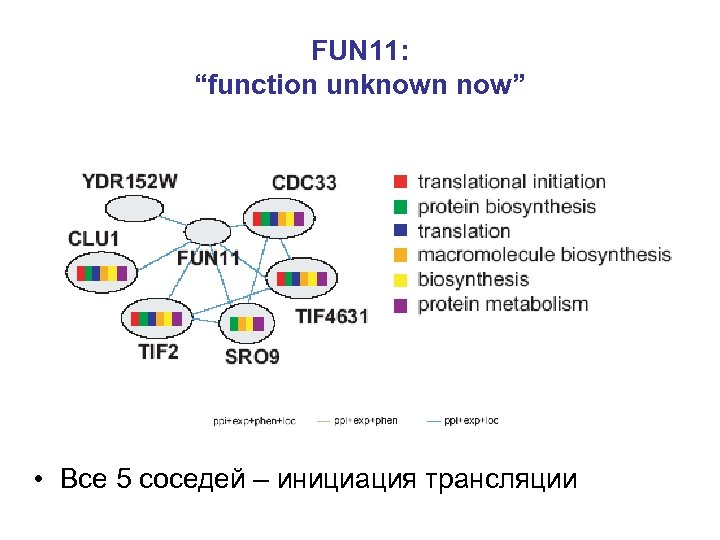
FUN 11: “function unknown now” • Все 5 соседей – инициация трансляции
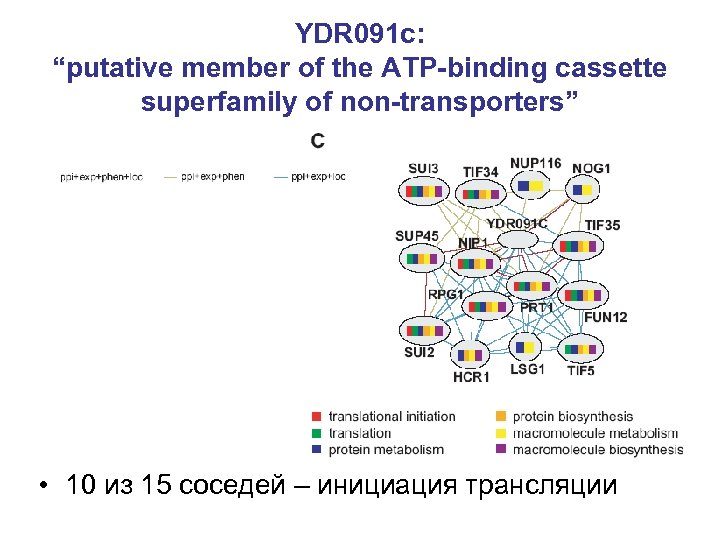
YDR 091 c: “putative member of the ATP-binding cassette superfamily of non-transporters” • 10 из 15 соседей – инициация трансляции

YGR 205 w – HSP 104 (ppi+exp+loc) • HSP 104: белок теплового шока; мутанты термочувствительны. Шаперон. • Коэкспрессия HSP 104 и YGR 205 w наблюдается при разных шоках • Предсказание: YGR 205 w тоже участвует в ответе на стресс • Эксперимент: мутанты по YGR 205 w имеют повышенную термоустойчивость • Та же система, противоположное действие – разные фенотипы
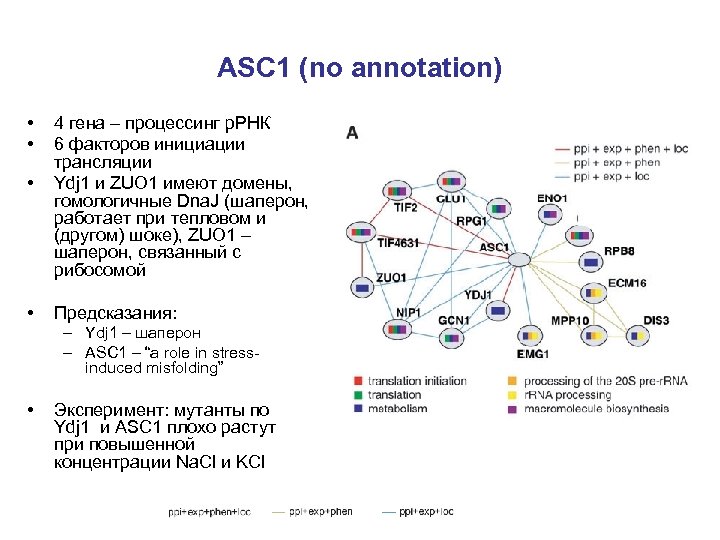
ASC 1 (no annotation) • • 4 гена – процессинг р. РНК 6 факторов инициации трансляции Ydj 1 и ZUO 1 имеют домены, гомологичные Dna. J (шаперон, работает при тепловом и (другом) шоке), ZUO 1 – шаперон, связанный с рибосомой Предсказания: – Ydj 1 – шаперон – ASC 1 – “a role in stressinduced misfolding” • Эксперимент: мутанты по Ydj 1 и ASC 1 плохо растут при повышенной концентрации Na. Cl и KCl
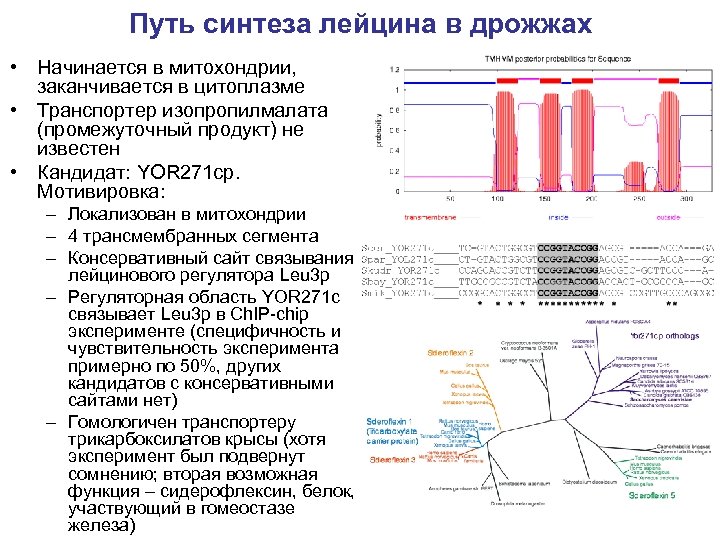
Путь синтеза лейцина в дрожжах • Начинается в митохондрии, заканчивается в цитоплазме • Транспортер изопропилмалата (промежуточный продукт) не известен • Кандидат: YOR 271 cp. Мотивировка: – Локализован в митохондрии – 4 трансмембранных сегмента – Консервативный сайт связывания лейцинового регулятора Leu 3 p – Регуляторная область YOR 271 c связывает Leu 3 p в Ch. IP-chip эксперименте (специфичность и чувствительность эксперимента примерно по 50%, других кандидатов с консервативными сайтами нет) – Гомологичен транспортеру трикарбоксилатов крысы (хотя эксперимент был подвернут сомнению; вторая возможная функция – сидерофлексин, белок, участвующий в гомеостазе железа)
1db88bf1778e8eb55d3589eef03ffb82.ppt