Лекция_2.pptx
- Количество слайдов: 37
 Методы изучения регуляторных районов генов Лекция II Меркулова Татьяна Ивановна Институт цитологии и генетики СО РАН
Методы изучения регуляторных районов генов Лекция II Меркулова Татьяна Ивановна Институт цитологии и генетики СО РАН
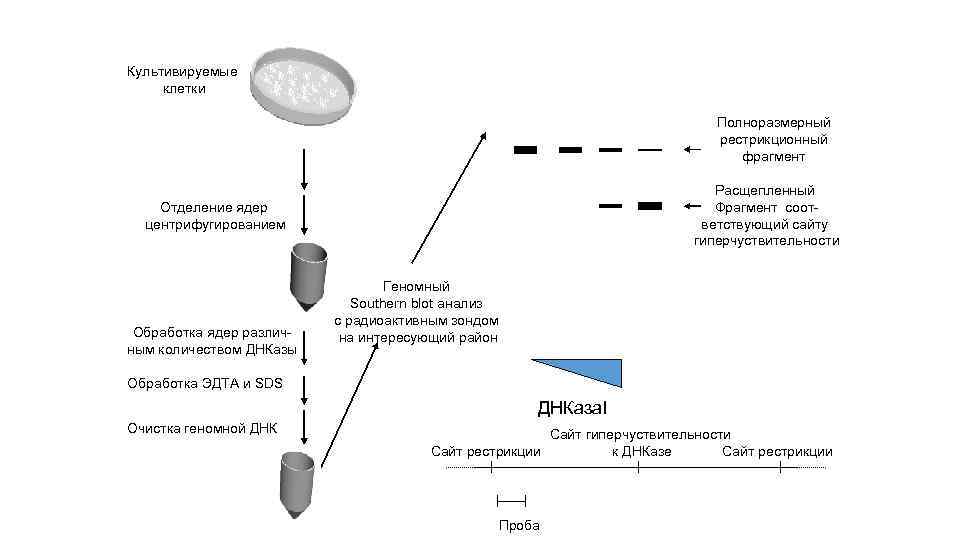 Культивируемые клетки Полноразмерный рестрикционный фрагмент Расщепленный Фрагмент соответствующий сайту гиперчуствительности Отделение ядер центрифугированием Обработка ядер различным количеством ДНКазы Геномный Southern blot анализ с радиоактивным зондом на интересующий район Обработка ЭДТА и SDS ДНКаза. I Очистка геномной ДНК Сайт гиперчуствительности к ДНКазе Сайт рестрикции Проба
Культивируемые клетки Полноразмерный рестрикционный фрагмент Расщепленный Фрагмент соответствующий сайту гиперчуствительности Отделение ядер центрифугированием Обработка ядер различным количеством ДНКазы Геномный Southern blot анализ с радиоактивным зондом на интересующий район Обработка ЭДТА и SDS ДНКаза. I Очистка геномной ДНК Сайт гиперчуствительности к ДНКазе Сайт рестрикции Проба
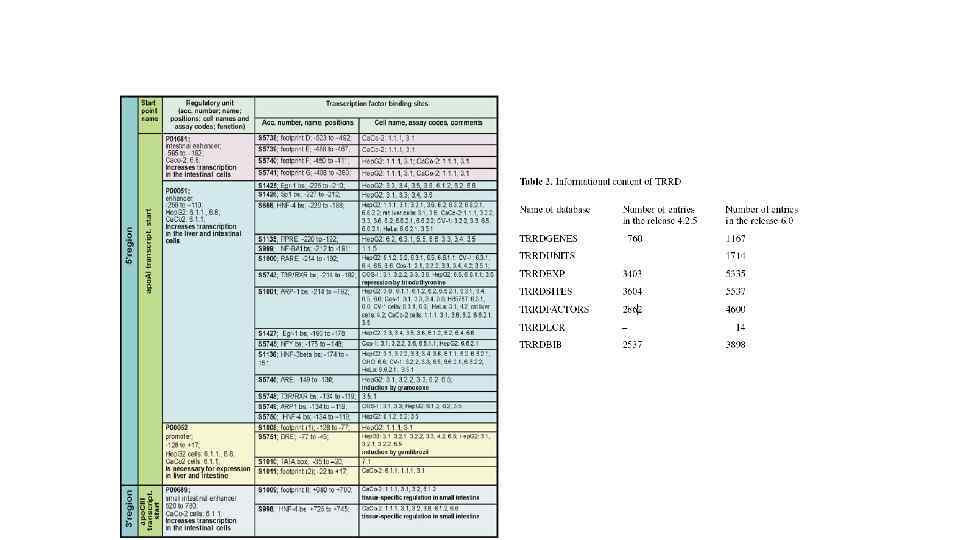
 Регуляторные районы гена тирозинаминотрансферазы (ТАТ) крысы Конститутивный печень-специфический энхансер ТАТ в районе – 11 т. п. н. от старта транскрипции. IIBP -10550 -10540 HNF 3 -10530 -10520 -10510 -10500 -10490 -10480 Печень-специфический глюкокортикоид-индуцибельный энхансер (GRU) ТАТ в районе -5, 5 т. п. н. от старта транскрипции C/EBP -5480 -5470 HNF 3 GRE -5460 -5450 C/EBP -5440 -5430 -5420 -5410 C/EBP -5400 -5390 -5380 C/EBP HNF 3 -5370 -5360 -5350 -5340 HNF 3 -5330 -5320 -5310 -5300 Печень-специфический c. AMP-индуцибельный энхансер ТАТ в районе – 3, 6 т. п. н. от старта транскрипции. IRE HNF 4 CRE/GME -3660 -3650 -3640 -3630 -3620 -3610 -3600 -3590 -3580 Печень-специфический глюкокортикоид-индуцибельный энхансер (GRU) ТАТ в районе – 2, 5 т. п. н. от старта транскрипции GRE -2610 Ets -2600 -2590 -2580 C/EBP -2570 -2560 -2550 -2540 -2530 -2520 GRE Ets -2510 -2500 HNF 3 C/EBP -2490 -2480 -2470 -2460 Sp 1 HNF 3 -2450 -2440 -2430 -2420 -2410 -2400 -2390 Регуляторные элементы промотора ТАТ. C/EBP HNF 1 -290 -280 -270 HNF 1 LIVER -260 -250 -240 -230 -220 -210 -200 -190 -180 -170 -160 NF-Y -150 -140 -130 -120 -110 -100 -90 -80 -70 -60
Регуляторные районы гена тирозинаминотрансферазы (ТАТ) крысы Конститутивный печень-специфический энхансер ТАТ в районе – 11 т. п. н. от старта транскрипции. IIBP -10550 -10540 HNF 3 -10530 -10520 -10510 -10500 -10490 -10480 Печень-специфический глюкокортикоид-индуцибельный энхансер (GRU) ТАТ в районе -5, 5 т. п. н. от старта транскрипции C/EBP -5480 -5470 HNF 3 GRE -5460 -5450 C/EBP -5440 -5430 -5420 -5410 C/EBP -5400 -5390 -5380 C/EBP HNF 3 -5370 -5360 -5350 -5340 HNF 3 -5330 -5320 -5310 -5300 Печень-специфический c. AMP-индуцибельный энхансер ТАТ в районе – 3, 6 т. п. н. от старта транскрипции. IRE HNF 4 CRE/GME -3660 -3650 -3640 -3630 -3620 -3610 -3600 -3590 -3580 Печень-специфический глюкокортикоид-индуцибельный энхансер (GRU) ТАТ в районе – 2, 5 т. п. н. от старта транскрипции GRE -2610 Ets -2600 -2590 -2580 C/EBP -2570 -2560 -2550 -2540 -2530 -2520 GRE Ets -2510 -2500 HNF 3 C/EBP -2490 -2480 -2470 -2460 Sp 1 HNF 3 -2450 -2440 -2430 -2420 -2410 -2400 -2390 Регуляторные элементы промотора ТАТ. C/EBP HNF 1 -290 -280 -270 HNF 1 LIVER -260 -250 -240 -230 -220 -210 -200 -190 -180 -170 -160 NF-Y -150 -140 -130 -120 -110 -100 -90 -80 -70 -60
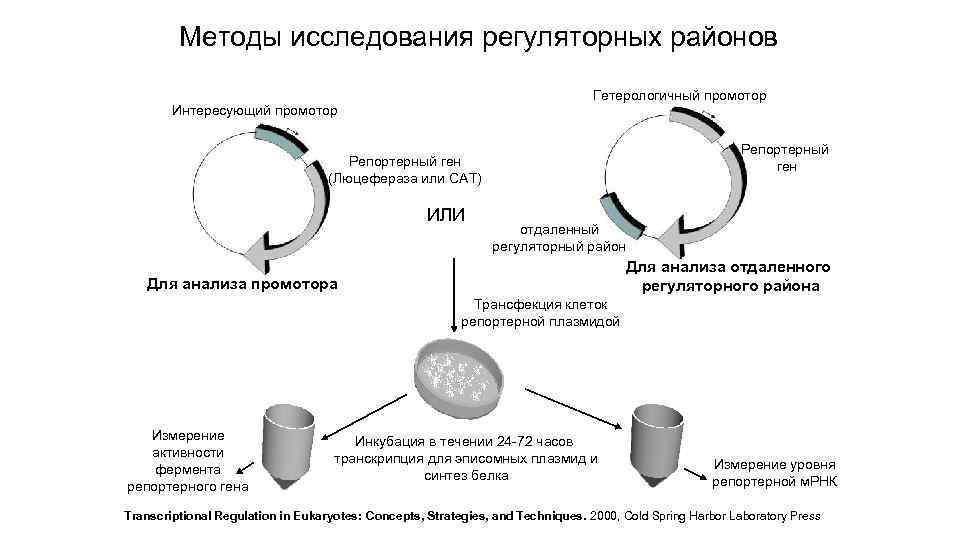 Методы исследования регуляторных районов Гетерологичный промотор Интересующий промотор Репортерный ген (Люцефераза или САТ) ИЛИ отдаленный регуляторный район Для анализа отдаленного регуляторного района Для анализа промотора Трансфекция клеток репортерной плазмидой Измерение активности фермента репортерного гена Инкубация в течении 24 -72 часов транскрипция для эписомных плазмид и синтез белка Измерение уровня репортерной м. РНК Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold Spring Harbor Laboratory Press
Методы исследования регуляторных районов Гетерологичный промотор Интересующий промотор Репортерный ген (Люцефераза или САТ) ИЛИ отдаленный регуляторный район Для анализа отдаленного регуляторного района Для анализа промотора Трансфекция клеток репортерной плазмидой Измерение активности фермента репортерного гена Инкубация в течении 24 -72 часов транскрипция для эписомных плазмид и синтез белка Измерение уровня репортерной м. РНК Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold Spring Harbor Laboratory Press
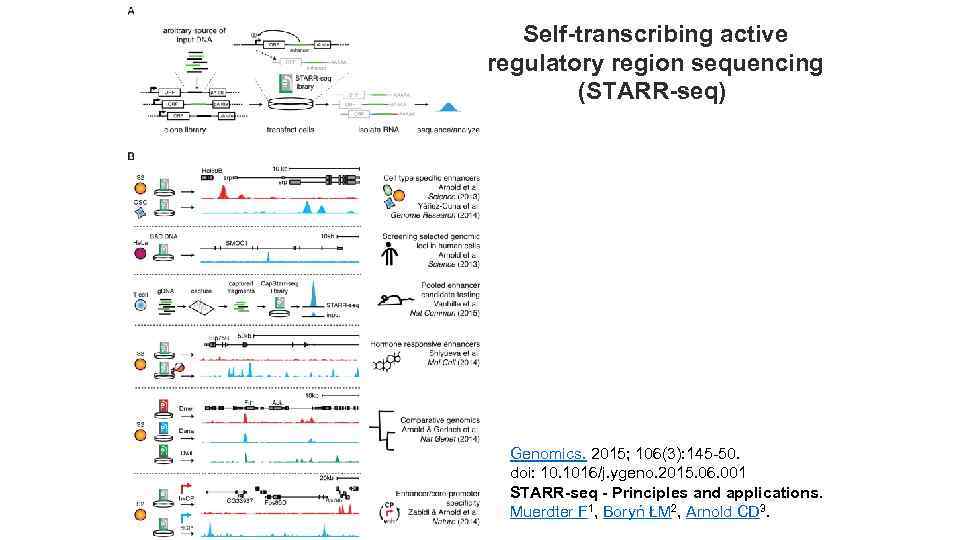 Self-transcribing active regulatory region sequencing (STARR-seq) Genomics. 2015; 106(3): 145 -50. doi: 10. 1016/j. ygeno. 2015. 06. 001 STARR-seq - Principles and applications. Muerdter F 1, Boryń ŁM 2, Arnold CD 3.
Self-transcribing active regulatory region sequencing (STARR-seq) Genomics. 2015; 106(3): 145 -50. doi: 10. 1016/j. ygeno. 2015. 06. 001 STARR-seq - Principles and applications. Muerdter F 1, Boryń ŁM 2, Arnold CD 3.
 Регуляторные районы гена тирозинаминотрансферазы (ТАТ) крысы Конститутивный печень-специфический энхансер ТАТ в районе – 11 т. п. н. от старта транскрипции. IIBP -10550 -10540 HNF 3 -10530 -10520 -10510 -10500 -10490 -10480 Печень-специфический глюкокортикоид-индуцибельный энхансер (GRU) ТАТ в районе -5, 5 т. п. н. от старта транскрипции C/EBP -5480 -5470 HNF 3 GRE -5460 -5450 C/EBP -5440 -5430 -5420 -5410 C/EBP -5400 -5390 -5380 C/EBP HNF 3 -5370 -5360 -5350 -5340 HNF 3 -5330 -5320 -5310 -5300 Печень-специфический c. AMP-индуцибельный энхансер ТАТ в районе – 3, 6 т. п. н. от старта транскрипции. IRE HNF 4 CRE/GME -3660 -3650 -3640 -3630 -3620 -3610 -3600 -3590 -3580 Печень-специфический глюкокортикоид-индуцибельный энхансер (GRU) ТАТ в районе – 2, 5 т. п. н. от старта транскрипции GRE -2610 Ets -2600 -2590 -2580 C/EBP -2570 -2560 -2550 -2540 -2530 -2520 GRE Ets -2510 -2500 HNF 3 C/EBP -2490 -2480 -2470 -2460 Sp 1 HNF 3 -2450 -2440 -2430 -2420 -2410 -2400 -2390 Регуляторные элементы промотора ТАТ. C/EBP HNF 1 -290 -280 -270 HNF 1 LIVER -260 -250 -240 -230 -220 -210 -200 -190 -180 -170 -160 NF-Y -150 -140 -130 -120 -110 -100 -90 -80 -70 -60
Регуляторные районы гена тирозинаминотрансферазы (ТАТ) крысы Конститутивный печень-специфический энхансер ТАТ в районе – 11 т. п. н. от старта транскрипции. IIBP -10550 -10540 HNF 3 -10530 -10520 -10510 -10500 -10490 -10480 Печень-специфический глюкокортикоид-индуцибельный энхансер (GRU) ТАТ в районе -5, 5 т. п. н. от старта транскрипции C/EBP -5480 -5470 HNF 3 GRE -5460 -5450 C/EBP -5440 -5430 -5420 -5410 C/EBP -5400 -5390 -5380 C/EBP HNF 3 -5370 -5360 -5350 -5340 HNF 3 -5330 -5320 -5310 -5300 Печень-специфический c. AMP-индуцибельный энхансер ТАТ в районе – 3, 6 т. п. н. от старта транскрипции. IRE HNF 4 CRE/GME -3660 -3650 -3640 -3630 -3620 -3610 -3600 -3590 -3580 Печень-специфический глюкокортикоид-индуцибельный энхансер (GRU) ТАТ в районе – 2, 5 т. п. н. от старта транскрипции GRE -2610 Ets -2600 -2590 -2580 C/EBP -2570 -2560 -2550 -2540 -2530 -2520 GRE Ets -2510 -2500 HNF 3 C/EBP -2490 -2480 -2470 -2460 Sp 1 HNF 3 -2450 -2440 -2430 -2420 -2410 -2400 -2390 Регуляторные элементы промотора ТАТ. C/EBP HNF 1 -290 -280 -270 HNF 1 LIVER -260 -250 -240 -230 -220 -210 -200 -190 -180 -170 -160 NF-Y -150 -140 -130 -120 -110 -100 -90 -80 -70 -60
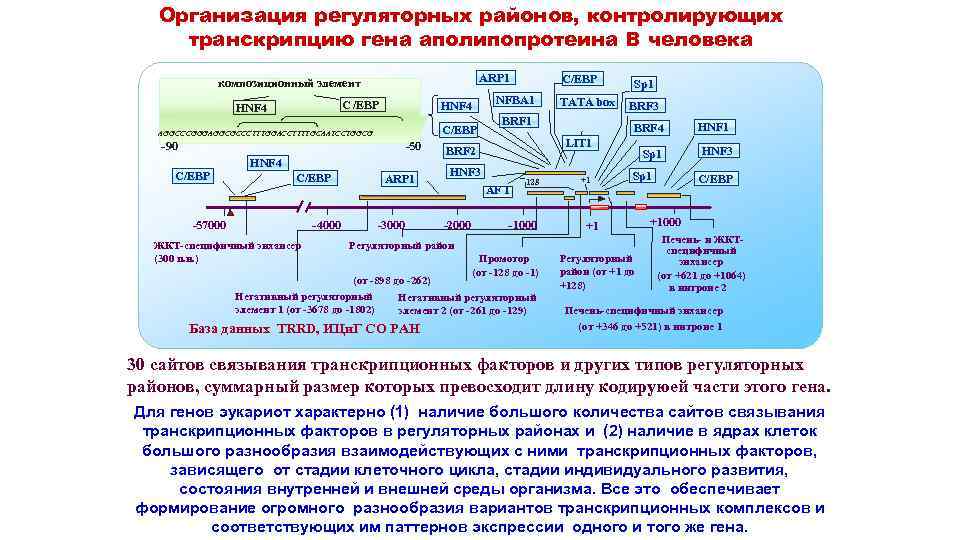 Организация регуляторных районов, контролирующих транскрипцию гена аполипопротеина В человека ARP 1 композиционный элемент С /EBP HNF 4 C/EBP AGGCCCGGGAGGCGCCCTTTGGACCTTTTGCAATCCTGGCG - 90 -50 C/EBP HNF 4 C/EBP -57000 -4000 ЖКТ-специфичный энхансер (300 п. н. ) ARP 1 -3000 C/EBP NFBA 1 BRF 3 BRF 4 LIT 1 BRF 2 HNF 3 AF 1 -2000 TATA box Sp 1 -- 128 -1000 +1 Sp 1 +1 Регуляторный район Промотор (от -128 до -1) (от -898 до -262) Негативный регуляторный элемент 1 (от -3678 до -1802) элемент 2 (от -261 до -129) База данных TRRD, ИЦи. Г СО РАН Регуляторный район (от +1 до +128) HNF 1 HNF 3 C/EBP +1000 Печень- и ЖКТспецифичный энхансер (от +621 до +1064) в интроне 2 Печень-специфичный энхансер (от +346 до +521) в интроне 1 30 сайтов связывания транскрипционных факторов и других типов регуляторных районов, суммарный размер которых превосходит длину кодируюей части этого гена. Для генов эукариот характерно (1) наличие большого количества сайтов связывания транскрипционных факторов в регуляторных районах и (2) наличие в ядрах клеток большого разнообразия взаимодействующих с ними транскрипционных факторов, зависящего от стадии клеточного цикла, стадии индивидуального развития, состояния внутренней и внешней среды организма. Все это обеспечивает формирование огромного разнообразия вариантов транскрипционных комплексов и соответствующих им паттернов экспрессии одного и того же гена.
Организация регуляторных районов, контролирующих транскрипцию гена аполипопротеина В человека ARP 1 композиционный элемент С /EBP HNF 4 C/EBP AGGCCCGGGAGGCGCCCTTTGGACCTTTTGCAATCCTGGCG - 90 -50 C/EBP HNF 4 C/EBP -57000 -4000 ЖКТ-специфичный энхансер (300 п. н. ) ARP 1 -3000 C/EBP NFBA 1 BRF 3 BRF 4 LIT 1 BRF 2 HNF 3 AF 1 -2000 TATA box Sp 1 -- 128 -1000 +1 Sp 1 +1 Регуляторный район Промотор (от -128 до -1) (от -898 до -262) Негативный регуляторный элемент 1 (от -3678 до -1802) элемент 2 (от -261 до -129) База данных TRRD, ИЦи. Г СО РАН Регуляторный район (от +1 до +128) HNF 1 HNF 3 C/EBP +1000 Печень- и ЖКТспецифичный энхансер (от +621 до +1064) в интроне 2 Печень-специфичный энхансер (от +346 до +521) в интроне 1 30 сайтов связывания транскрипционных факторов и других типов регуляторных районов, суммарный размер которых превосходит длину кодируюей части этого гена. Для генов эукариот характерно (1) наличие большого количества сайтов связывания транскрипционных факторов в регуляторных районах и (2) наличие в ядрах клеток большого разнообразия взаимодействующих с ними транскрипционных факторов, зависящего от стадии клеточного цикла, стадии индивидуального развития, состояния внутренней и внешней среды организма. Все это обеспечивает формирование огромного разнообразия вариантов транскрипционных комплексов и соответствующих им паттернов экспрессии одного и того же гена.
 Первые исследования регуляторных элементов Payvar F. , Wrange O. , et al. Purified glucocorticoid receptors bind selectively in vitro to a cloned DNA fragment whose transcription is regulated by glucocorticoids in vivo. Proc Natl Acad Sci U S A. 1981 Nov; 78(11): 6628 -32.
Первые исследования регуляторных элементов Payvar F. , Wrange O. , et al. Purified glucocorticoid receptors bind selectively in vitro to a cloned DNA fragment whose transcription is regulated by glucocorticoids in vivo. Proc Natl Acad Sci U S A. 1981 Nov; 78(11): 6628 -32.
![Первые исследования регуляторных элементов Selectivity of receptor binding to MTV 2 -EE [32 P]DNA: Первые исследования регуляторных элементов Selectivity of receptor binding to MTV 2 -EE [32 P]DNA:](https://present5.com/presentation/1/53400281_452834427.pdf-img/53400281_452834427.pdf-10.jpg) Первые исследования регуляторных элементов Selectivity of receptor binding to MTV 2 -EE [32 P]DNA: competition by excess unlabeled DNA. In one experiment (lanes A-G), 100 ng of end-labeled Eco. RI fiagments from p. MTV 2 were incubated with or without 500 ng of unlabeled closed circular p. BR 322 or p. MTV 2 DNA either in the presence or absence of glucocorticoid receptor for 60 min at 40 C; DNA bound to nitrocellulose filters was eluted analyzed on agarose gels. Lanes: A, DNA prior to filter binding; B, DNA bound in the absence of added protein; C, DNA bound in the presence of glucocorticoid receptor; D and E, DNA bound in the absence of glucocorticoid receptor but in the presence of a 5 -fold excess of unlabeled p. BR 322 orp. MTV 2 DNA, respectively; F and G, DNA bound in the presence of glucocorticoid receptor together with a 5 -fold excess of unlabeled p. BR 322 or p. MTV 2 DNA, respectively. In a second experiment (lanes H-L), 100 ng of the labeled p. MTV 2 fragments were first incubated in the presence or absence of glucocorticoid receptor for 30 min at 40 C; 500 ng of either RSF 2124 or p. MTV 2 DNA was then added and incubation was continued for an additional 30 min at 40 C. Lanes: H and I, DNA bound in the absence of glucocorticoid receptor but in the presence of added p. MTV 2 or RSF 2124 DNA, respectively; J, DNA bound in the presence of glucocorticoid receptor; K and L, DNA bound in the presence of glucocorticoid receptor and subsequently challenged with a 5 -fold excess of unlabeled p. MTV 2 or RSF 2124 DNA, respectively. Payvar F. , Wrange O. , et al. Purified glucocorticoid receptors bind selectively in vitro to a cloned DNA fragment whose transcription is regulated by glucocorticoids in vivo. Proc Natl Acad Sci U S A. 1981 Nov; 78(11): 6628 -32.
Первые исследования регуляторных элементов Selectivity of receptor binding to MTV 2 -EE [32 P]DNA: competition by excess unlabeled DNA. In one experiment (lanes A-G), 100 ng of end-labeled Eco. RI fiagments from p. MTV 2 were incubated with or without 500 ng of unlabeled closed circular p. BR 322 or p. MTV 2 DNA either in the presence or absence of glucocorticoid receptor for 60 min at 40 C; DNA bound to nitrocellulose filters was eluted analyzed on agarose gels. Lanes: A, DNA prior to filter binding; B, DNA bound in the absence of added protein; C, DNA bound in the presence of glucocorticoid receptor; D and E, DNA bound in the absence of glucocorticoid receptor but in the presence of a 5 -fold excess of unlabeled p. BR 322 orp. MTV 2 DNA, respectively; F and G, DNA bound in the presence of glucocorticoid receptor together with a 5 -fold excess of unlabeled p. BR 322 or p. MTV 2 DNA, respectively. In a second experiment (lanes H-L), 100 ng of the labeled p. MTV 2 fragments were first incubated in the presence or absence of glucocorticoid receptor for 30 min at 40 C; 500 ng of either RSF 2124 or p. MTV 2 DNA was then added and incubation was continued for an additional 30 min at 40 C. Lanes: H and I, DNA bound in the absence of glucocorticoid receptor but in the presence of added p. MTV 2 or RSF 2124 DNA, respectively; J, DNA bound in the presence of glucocorticoid receptor; K and L, DNA bound in the presence of glucocorticoid receptor and subsequently challenged with a 5 -fold excess of unlabeled p. MTV 2 or RSF 2124 DNA, respectively. Payvar F. , Wrange O. , et al. Purified glucocorticoid receptors bind selectively in vitro to a cloned DNA fragment whose transcription is regulated by glucocorticoids in vivo. Proc Natl Acad Sci U S A. 1981 Nov; 78(11): 6628 -32.
 Связывание глюкокортикоидного рецептора с фрагментами гена триптофаноксигеназы крысы А. Схема ДНК λТО 2. ▌ - экзоны гена TDO 2, - старт транскрипции, R, L - правое и левое плечи вектора (λch 4 A), ● - сайты рестрикции Eco. RI, I-V - фрагменты рестрикции λТО 2. - исследованный Данеш У. и сотр. участок гена; х - выявленные ими GREs Б. Радиоавтограф геля после электрофореза Eco. RI фрагментов λТО 2 в 0, 6% агарозе (1 - исходная ДНК, 2 - фрагменты ДНК, извлеченные из комплекса ДНК-рецептор-антитело на инактивированном S. Aureus). В. Радиоавтограф геля после электрофореза фрагментов II и III в 1% агарозе (1 - фрагменты, задерживающиеся на нитроцеллюлозных фильтрах в присутствии рецептора, 2 - то же в отсутствие рецептора, 3 - исходная ДНК). Merkulov V. M. , Merkulova T. I. “Nucleotide sequence of a fragment of the rat tryptophan oxygenase gene showing high affinity to glucocorticoid receptor in vitro”. // Biochim. Biophys. Acta 1992. V. 1132. P. 100 -102
Связывание глюкокортикоидного рецептора с фрагментами гена триптофаноксигеназы крысы А. Схема ДНК λТО 2. ▌ - экзоны гена TDO 2, - старт транскрипции, R, L - правое и левое плечи вектора (λch 4 A), ● - сайты рестрикции Eco. RI, I-V - фрагменты рестрикции λТО 2. - исследованный Данеш У. и сотр. участок гена; х - выявленные ими GREs Б. Радиоавтограф геля после электрофореза Eco. RI фрагментов λТО 2 в 0, 6% агарозе (1 - исходная ДНК, 2 - фрагменты ДНК, извлеченные из комплекса ДНК-рецептор-антитело на инактивированном S. Aureus). В. Радиоавтограф геля после электрофореза фрагментов II и III в 1% агарозе (1 - фрагменты, задерживающиеся на нитроцеллюлозных фильтрах в присутствии рецептора, 2 - то же в отсутствие рецептора, 3 - исходная ДНК). Merkulov V. M. , Merkulova T. I. “Nucleotide sequence of a fragment of the rat tryptophan oxygenase gene showing high affinity to glucocorticoid receptor in vitro”. // Biochim. Biophys. Acta 1992. V. 1132. P. 100 -102
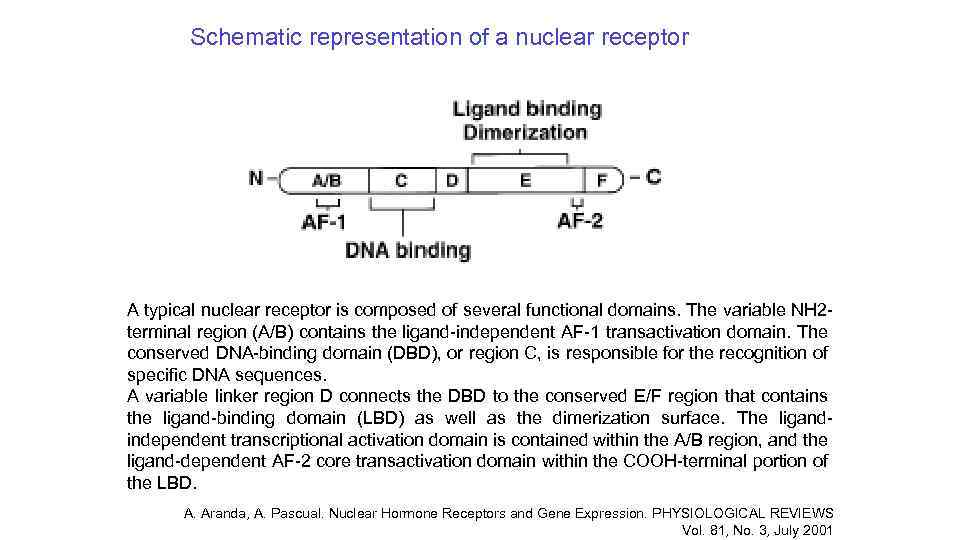 Schematic representation of a nuclear receptor A typical nuclear receptor is composed of several functional domains. The variable NH 2 terminal region (A/B) contains the ligand-independent AF-1 transactivation domain. The conserved DNA-binding domain (DBD), or region C, is responsible for the recognition of specific DNA sequences. A variable linker region D connects the DBD to the conserved E/F region that contains the ligand-binding domain (LBD) as well as the dimerization surface. The ligandindependent transcriptional activation domain is contained within the A/B region, and the ligand-dependent AF-2 core transactivation domain within the COOH-terminal portion of the LBD. A. Aranda, A. Pascual. Nuclear Hormone Receptors and Gene Expression. PHYSIOLOGICAL REVIEWS Vol. 81, No. 3, July 2001
Schematic representation of a nuclear receptor A typical nuclear receptor is composed of several functional domains. The variable NH 2 terminal region (A/B) contains the ligand-independent AF-1 transactivation domain. The conserved DNA-binding domain (DBD), or region C, is responsible for the recognition of specific DNA sequences. A variable linker region D connects the DBD to the conserved E/F region that contains the ligand-binding domain (LBD) as well as the dimerization surface. The ligandindependent transcriptional activation domain is contained within the A/B region, and the ligand-dependent AF-2 core transactivation domain within the COOH-terminal portion of the LBD. A. Aranda, A. Pascual. Nuclear Hormone Receptors and Gene Expression. PHYSIOLOGICAL REVIEWS Vol. 81, No. 3, July 2001
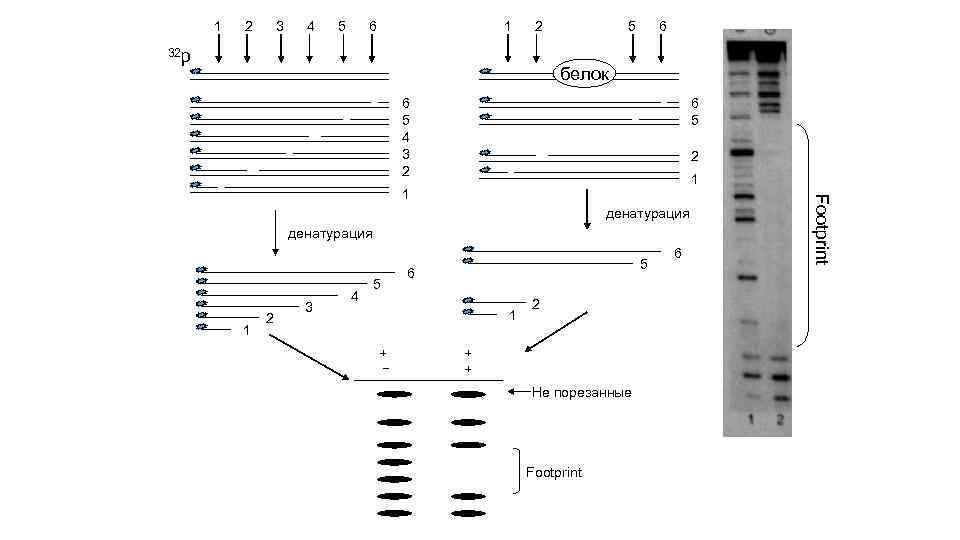 1 2 3 4 5 6 1 2 5 6 32 р белок 6 5 4 3 2 6 5 2 1 денатурация 1 2 3 4 5 5 6 1 + 2 + + Не порезанные Footprint 6 Footprint 1
1 2 3 4 5 6 1 2 5 6 32 р белок 6 5 4 3 2 6 5 2 1 денатурация 1 2 3 4 5 5 6 1 + 2 + + Не порезанные Footprint 6 Footprint 1
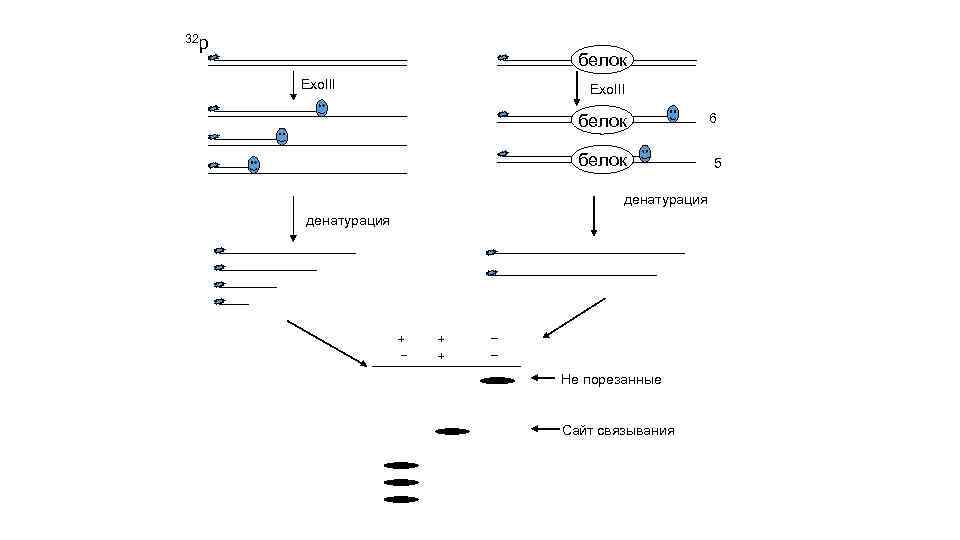 32 р белок Exo. III белок денатурация + + + Не порезанные Сайт связывания 6 5
32 р белок Exo. III белок денатурация + + + Не порезанные Сайт связывания 6 5
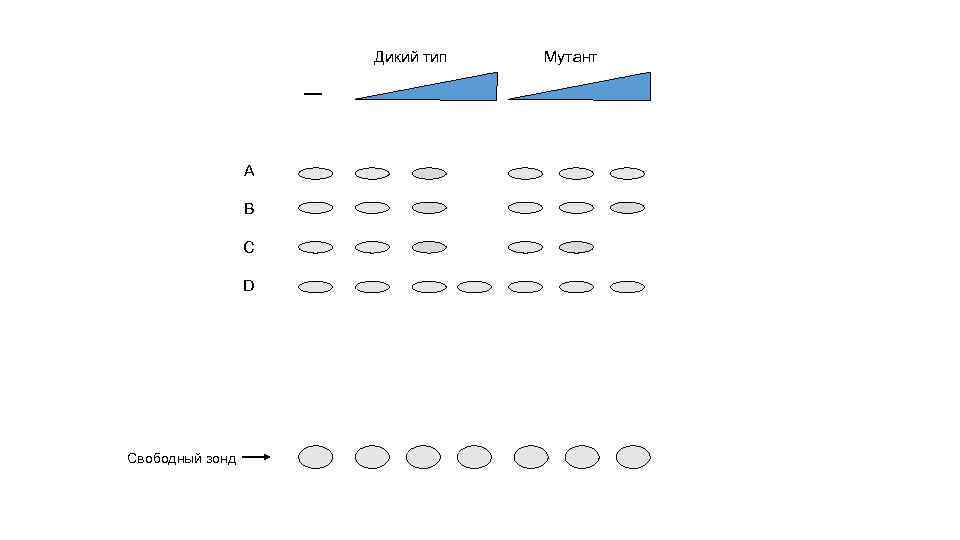 Дикий тип А В С D Свободный зонд Мутант
Дикий тип А В С D Свободный зонд Мутант
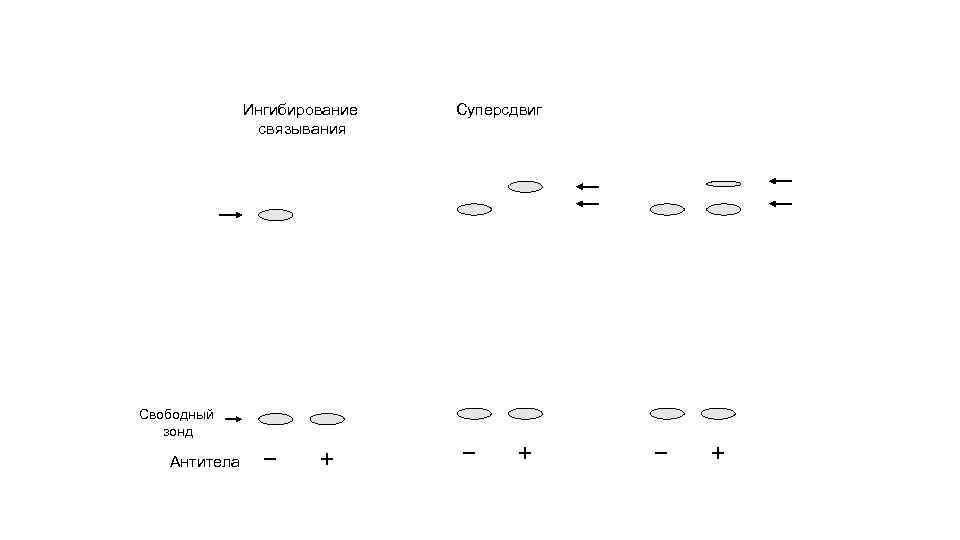 Ингибирование связывания Свободный зонд Антитела Суперсдвиг
Ингибирование связывания Свободный зонд Антитела Суперсдвиг
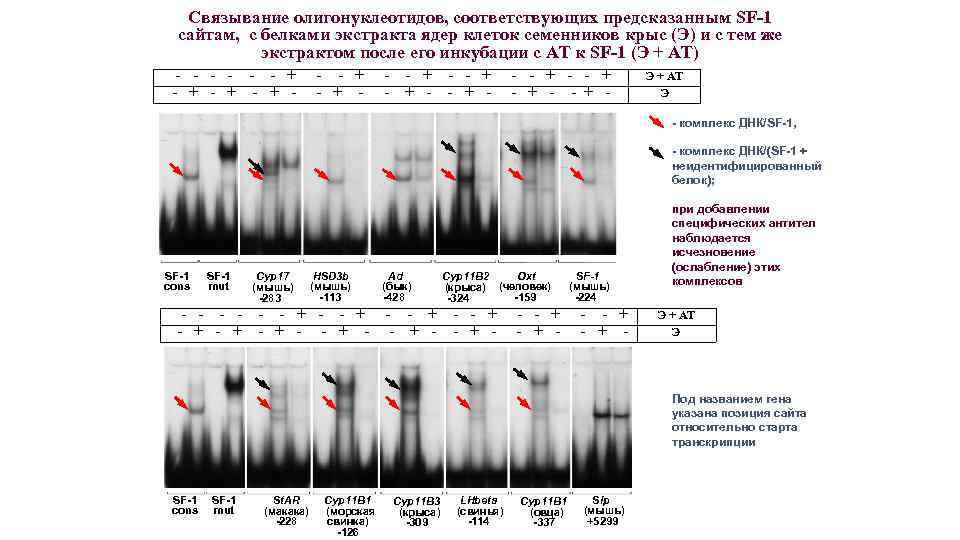 Связывание олигонуклеотидов, соответствующих предсказанным SF-1 сайтам, с белками экстракта ядер клеток семенников крыс (Э) и с тем же экстрактом после его инкубации c АТ к SF-1 (Э + АТ) - - - + - + - - - + - - + - Э + АТ Э - комплекс ДНК/SF-1, - комплекс ДНК/(SF-1 + неидентифицированный белок); SF-1 cons SF-1 mut Cyp 17 (мышь) -283 HSD 3 b (мышь) -113 - - - + - + - Ad (бык) -428 Oxt SF-1 Cyp 11 B 2 (крыса) (человек) (мышь) -159 -224 -324 - - + - + - при добавлении специфических антител наблюдается исчезновение (ослабление) этих комплексов Э + АТ Э Под названием гена указана позиция сайта относительно старта транскрипции SF-1 cons SF-1 mut St. AR (макака) -228 Cyp 11 B 1 (морская свинка) -126 LHbeta Cyp 11 B 3 (крыса) (свинья) -114 -309 Slp Cyp 11 B 1 (овца) (мышь) +5299 -337
Связывание олигонуклеотидов, соответствующих предсказанным SF-1 сайтам, с белками экстракта ядер клеток семенников крыс (Э) и с тем же экстрактом после его инкубации c АТ к SF-1 (Э + АТ) - - - + - + - - - + - - + - Э + АТ Э - комплекс ДНК/SF-1, - комплекс ДНК/(SF-1 + неидентифицированный белок); SF-1 cons SF-1 mut Cyp 17 (мышь) -283 HSD 3 b (мышь) -113 - - - + - + - Ad (бык) -428 Oxt SF-1 Cyp 11 B 2 (крыса) (человек) (мышь) -159 -224 -324 - - + - + - при добавлении специфических антител наблюдается исчезновение (ослабление) этих комплексов Э + АТ Э Под названием гена указана позиция сайта относительно старта транскрипции SF-1 cons SF-1 mut St. AR (макака) -228 Cyp 11 B 1 (морская свинка) -126 LHbeta Cyp 11 B 3 (крыса) (свинья) -114 -309 Slp Cyp 11 B 1 (овца) (мышь) +5299 -337
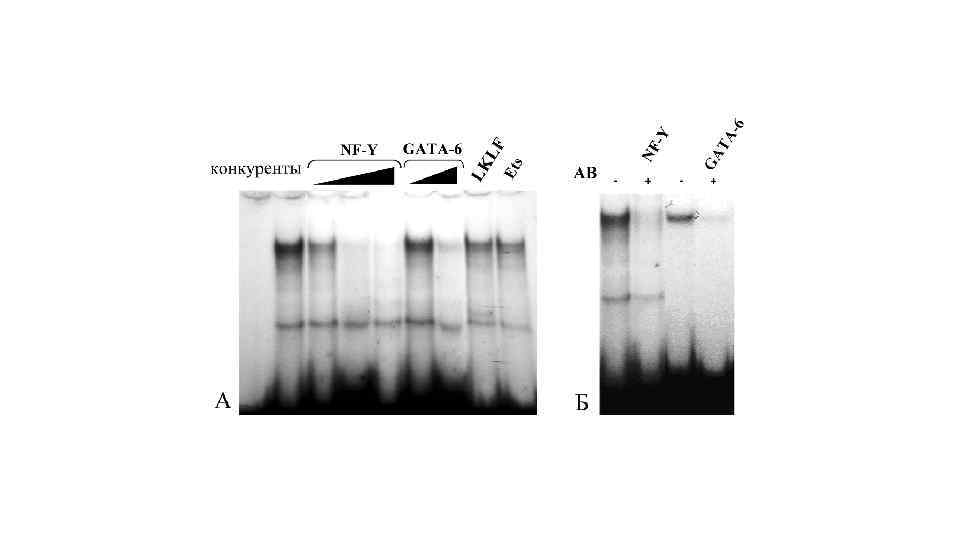
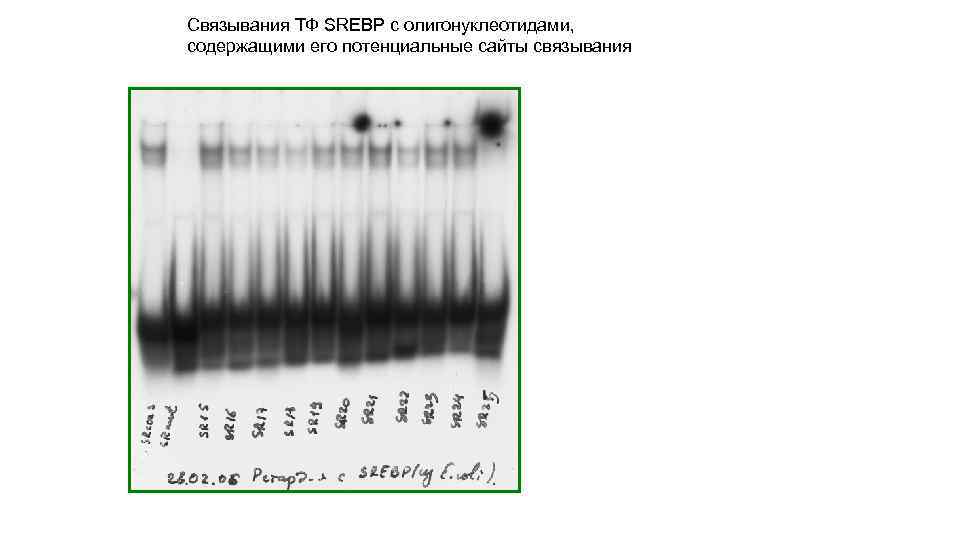 Связывания ТФ SREBP c олигонуклеотидами, содержащими его потенциальные сайты связывания
Связывания ТФ SREBP c олигонуклеотидами, содержащими его потенциальные сайты связывания
 Частотные матрицы и варианты консенсуса GRbs, полученные в результате анализа 25 (А) и 81 (B) экспериментально выявленных сайтов Позитивные GREs (n = 25) 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 A 5 15 10 55 10 55 40 15 20 15 0 5 5 0 5 C 25 0 20 10 75 25 25 15 35 0 0 0 25 100 10 G 45 65 20 0 0 10 15 25 25 5 95 5 5 0 0 T 25 20 50 35 15 10 20 45 20 80 5 90 65 0 85 Cons G G T A C A N N N T G T T C T (n = 81) [GR-TRRD] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 A 38 13 44 66 6 72 26 32 25 8 0 6 6 1 15 C 14 6 17 9 79 14 37 13 25 2 2 3 13 94 16 G 37 71 20 9 10 5 17 24 29 3 97 2 8 2 5 T 11 10 19 16 5 9 20 31 21 87 1 89 73 3 68 Cons G/A G А A C A N N N T G T T C T Частоты встречаемости нуклеотидов выражены в %
Частотные матрицы и варианты консенсуса GRbs, полученные в результате анализа 25 (А) и 81 (B) экспериментально выявленных сайтов Позитивные GREs (n = 25) 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 A 5 15 10 55 10 55 40 15 20 15 0 5 5 0 5 C 25 0 20 10 75 25 25 15 35 0 0 0 25 100 10 G 45 65 20 0 0 10 15 25 25 5 95 5 5 0 0 T 25 20 50 35 15 10 20 45 20 80 5 90 65 0 85 Cons G G T A C A N N N T G T T C T (n = 81) [GR-TRRD] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 A 38 13 44 66 6 72 26 32 25 8 0 6 6 1 15 C 14 6 17 9 79 14 37 13 25 2 2 3 13 94 16 G 37 71 20 9 10 5 17 24 29 3 97 2 8 2 5 T 11 10 19 16 5 9 20 31 21 87 1 89 73 3 68 Cons G/A G А A C A N N N T G T T C T Частоты встречаемости нуклеотидов выражены в %
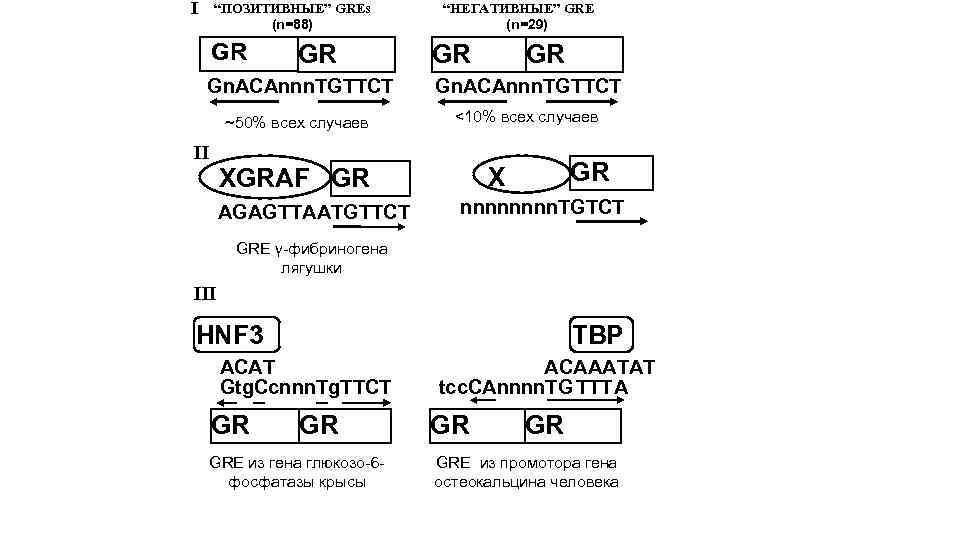 I “ПОЗИТИВНЫЕ” GREs (n=88) GR GR “НЕГАТИВНЫЕ” GRE (n=29) GR GR Gn. ACAnnn. TGTTCT ~50% всех случаев <10% всех случаев II AGAGTTAATGTTCT GR X XGRAF GR nnnn. TGTCT GRE γ-фибриногена лягушки III HNF 3 TBP ACAT Gtg. Ccnnn. Tg. TTCT GR GR GRE из гена глюкозо-6 фосфатазы крысы ACAAATAT tcc. CAnnnn. TG TTT A GR GR GRE из промотора гена остеокальцина человека
I “ПОЗИТИВНЫЕ” GREs (n=88) GR GR “НЕГАТИВНЫЕ” GRE (n=29) GR GR Gn. ACAnnn. TGTTCT ~50% всех случаев <10% всех случаев II AGAGTTAATGTTCT GR X XGRAF GR nnnn. TGTCT GRE γ-фибриногена лягушки III HNF 3 TBP ACAT Gtg. Ccnnn. Tg. TTCT GR GR GRE из гена глюкозо-6 фосфатазы крысы ACAAATAT tcc. CAnnnn. TG TTT A GR GR GRE из промотора гена остеокальцина человека
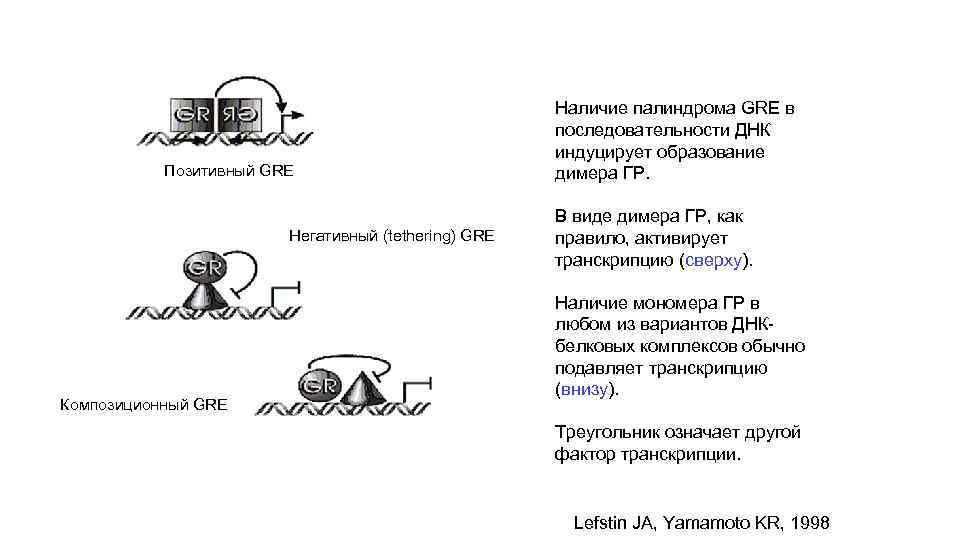 Позитивный GRE Негативный (tethering) GRE Композиционный GRE Наличие палиндрома GRE в последовательности ДНК индуцирует образование димера ГР. В виде димера ГР, как правило, активирует транскрипцию (сверху). Наличие мономера ГР в любом из вариантов ДНКбелковых комплексов обычно подавляет транскрипцию (внизу). Треугольник означает другой фактор транскрипции. Lefstin JA, Yamamoto KR, 1998
Позитивный GRE Негативный (tethering) GRE Композиционный GRE Наличие палиндрома GRE в последовательности ДНК индуцирует образование димера ГР. В виде димера ГР, как правило, активирует транскрипцию (сверху). Наличие мономера ГР в любом из вариантов ДНКбелковых комплексов обычно подавляет транскрипцию (внизу). Треугольник означает другой фактор транскрипции. Lefstin JA, Yamamoto KR, 1998
 Тонкий делеционный анализ промоторного района для выявления HRE (элемент, ответственный за реакцию на гипоксию) The indicated 5’ or 3’ deletion mutant of the ROR 4 reporter was transfected along with p. SVgal into Caco 2 cells. Eighteen hours after transfection the cells were cultured for 24 h under normoxia (open bar) or hypoxia (closed bar). Relative luciferase activity is represented, normalized to that in the transfected cells cultured under normoxia. Miki N, Ikuta M, Matsui T. Hypoxia-induced activation of the retinoic acid receptor-related orphan receptor a 4 gene by an interaction between hypoxia-inducible factor-1 and Sp 1. J Biol Chem. 2004 Apr 9; 279(15): 15025 -31.
Тонкий делеционный анализ промоторного района для выявления HRE (элемент, ответственный за реакцию на гипоксию) The indicated 5’ or 3’ deletion mutant of the ROR 4 reporter was transfected along with p. SVgal into Caco 2 cells. Eighteen hours after transfection the cells were cultured for 24 h under normoxia (open bar) or hypoxia (closed bar). Relative luciferase activity is represented, normalized to that in the transfected cells cultured under normoxia. Miki N, Ikuta M, Matsui T. Hypoxia-induced activation of the retinoic acid receptor-related orphan receptor a 4 gene by an interaction between hypoxia-inducible factor-1 and Sp 1. J Biol Chem. 2004 Apr 9; 279(15): 15025 -31.
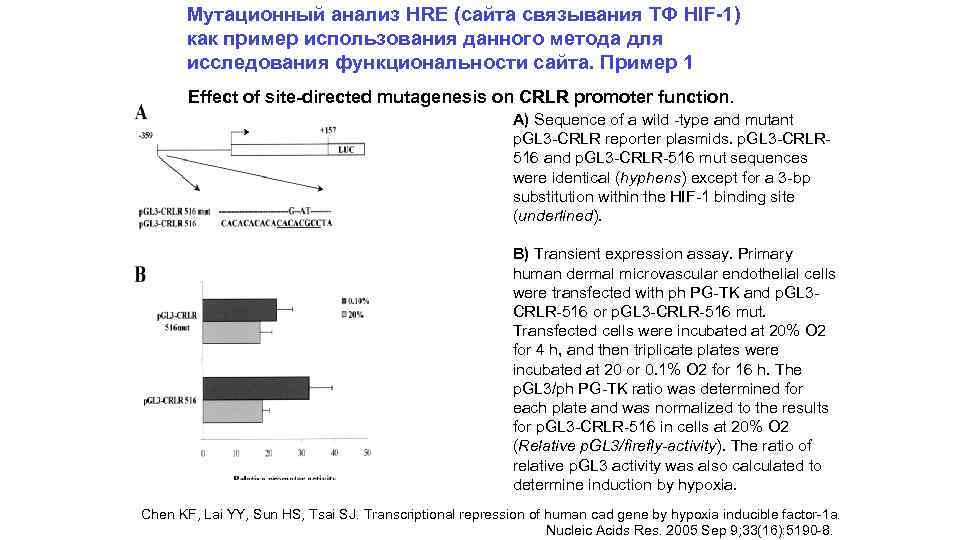 Мутационный анализ HRE (сайта связывания ТФ HIF-1) как пример использования данного метода для исследования функциональности сайта. Пример 1 Effect of site-directed mutagenesis on CRLR promoter function. A) Sequence of a wild -type and mutant p. GL 3 -CRLR reporter plasmids. p. GL 3 -CRLR 516 and p. GL 3 -CRLR-516 mut sequences were identical (hyphens) except for a 3 -bp substitution within the HIF-1 binding site (underlined). B) Transient expression assay. Primary human dermal microvascular endothelial cells were transfected with ph PG-TK and p. GL 3 CRLR-516 or p. GL 3 -CRLR-516 mut. Transfected cells were incubated at 20% O 2 for 4 h, and then triplicate plates were incubated at 20 or 0. 1% O 2 for 16 h. The p. GL 3/ph PG-TK ratio was determined for each plate and was normalized to the results for p. GL 3 -CRLR-516 in cells at 20% O 2 (Relative p. GL 3/firefly-activity). The ratio of relative p. GL 3 activity was also calculated to determine induction by hypoxia. Chen KF, Lai YY, Sun HS, Tsai SJ. Transcriptional repression of human cad gene by hypoxia inducible factor-1 a. Nucleic Acids Res. 2005 Sep 9; 33(16): 5190 -8.
Мутационный анализ HRE (сайта связывания ТФ HIF-1) как пример использования данного метода для исследования функциональности сайта. Пример 1 Effect of site-directed mutagenesis on CRLR promoter function. A) Sequence of a wild -type and mutant p. GL 3 -CRLR reporter plasmids. p. GL 3 -CRLR 516 and p. GL 3 -CRLR-516 mut sequences were identical (hyphens) except for a 3 -bp substitution within the HIF-1 binding site (underlined). B) Transient expression assay. Primary human dermal microvascular endothelial cells were transfected with ph PG-TK and p. GL 3 CRLR-516 or p. GL 3 -CRLR-516 mut. Transfected cells were incubated at 20% O 2 for 4 h, and then triplicate plates were incubated at 20 or 0. 1% O 2 for 16 h. The p. GL 3/ph PG-TK ratio was determined for each plate and was normalized to the results for p. GL 3 -CRLR-516 in cells at 20% O 2 (Relative p. GL 3/firefly-activity). The ratio of relative p. GL 3 activity was also calculated to determine induction by hypoxia. Chen KF, Lai YY, Sun HS, Tsai SJ. Transcriptional repression of human cad gene by hypoxia inducible factor-1 a. Nucleic Acids Res. 2005 Sep 9; 33(16): 5190 -8.
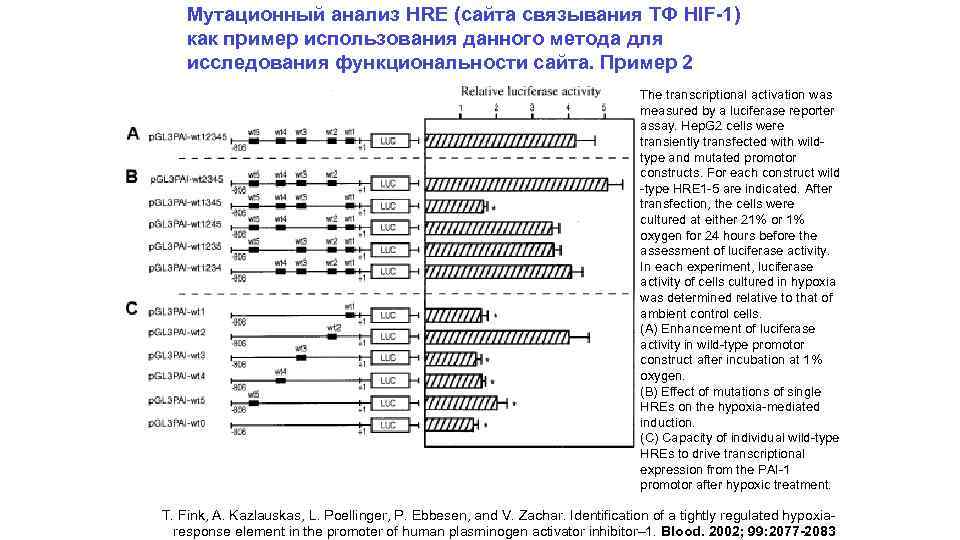 Мутационный анализ HRE (сайта связывания ТФ HIF-1) как пример использования данного метода для исследования функциональности сайта. Пример 2 The transcriptional activation was measured by a luciferase reporter assay. Hep. G 2 cells were transiently transfected with wildtype and mutated promotor constructs. For each construct wild -type HRE 1 -5 are indicated. After transfection, the cells were cultured at either 21% or 1% oxygen for 24 hours before the assessment of luciferase activity. In each experiment, luciferase activity of cells cultured in hypoxia was determined relative to that of ambient control cells. (A) Enhancement of luciferase activity in wild-type promotor construct after incubation at 1% oxygen. (B) Effect of mutations of single HREs on the hypoxia-mediated induction. (C) Capacity of individual wild-type HREs to drive transcriptional expression from the PAI-1 promotor after hypoxic treatment. T. Fink, A. Kazlauskas, L. Poellinger, P. Ebbesen, and V. Zachar. Identification of a tightly regulated hypoxiaresponse element in the promoter of human plasminogen activator inhibitor– 1. Blood. 2002; 99: 2077 -2083
Мутационный анализ HRE (сайта связывания ТФ HIF-1) как пример использования данного метода для исследования функциональности сайта. Пример 2 The transcriptional activation was measured by a luciferase reporter assay. Hep. G 2 cells were transiently transfected with wildtype and mutated promotor constructs. For each construct wild -type HRE 1 -5 are indicated. After transfection, the cells were cultured at either 21% or 1% oxygen for 24 hours before the assessment of luciferase activity. In each experiment, luciferase activity of cells cultured in hypoxia was determined relative to that of ambient control cells. (A) Enhancement of luciferase activity in wild-type promotor construct after incubation at 1% oxygen. (B) Effect of mutations of single HREs on the hypoxia-mediated induction. (C) Capacity of individual wild-type HREs to drive transcriptional expression from the PAI-1 promotor after hypoxic treatment. T. Fink, A. Kazlauskas, L. Poellinger, P. Ebbesen, and V. Zachar. Identification of a tightly regulated hypoxiaresponse element in the promoter of human plasminogen activator inhibitor– 1. Blood. 2002; 99: 2077 -2083
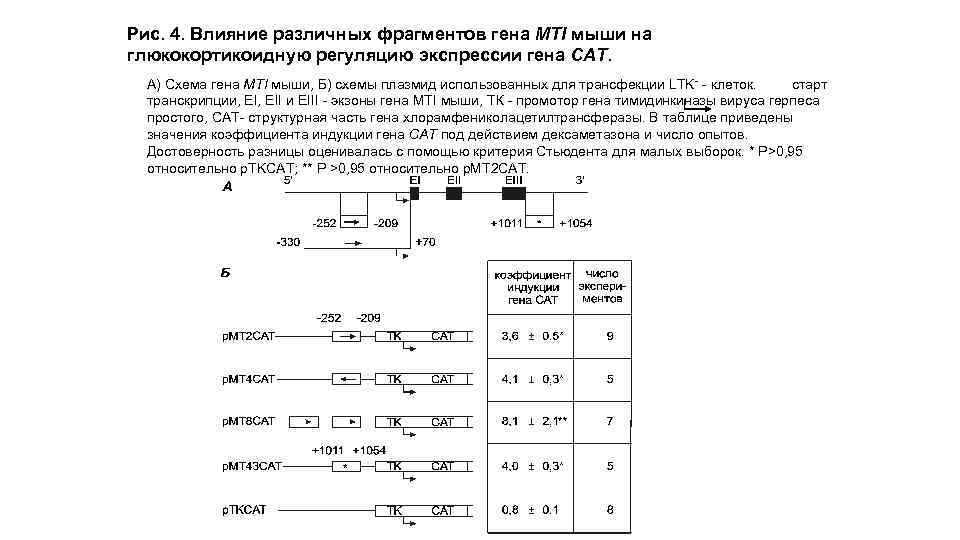 Рис. 4. Влияние различных фрагментов гена МТI мыши на глюкокортикоидную регуляцию экспрессии гена САТ. А) Схема гена МТI мыши, Б) схемы плазмид использованных для трансфекции LTK- - клеток. старт транскрипции, EII и EIII - экзоны гена МТI мыши, ТК - промотор гена тимидинкиназы вируса герпеса простого, САТ- структурная часть гена хлорамфениколацетилтрансферазы. В таблице приведены значения коэффициента индукции гена САТ под действием дексаметазона и число опытов. Достоверность разницы оценивалась с помощью критерия Стьюдента для малых выборок. * P>0, 95 относительно p. TKCAT; ** P >0, 95 относительно p. MT 2 CAT.
Рис. 4. Влияние различных фрагментов гена МТI мыши на глюкокортикоидную регуляцию экспрессии гена САТ. А) Схема гена МТI мыши, Б) схемы плазмид использованных для трансфекции LTK- - клеток. старт транскрипции, EII и EIII - экзоны гена МТI мыши, ТК - промотор гена тимидинкиназы вируса герпеса простого, САТ- структурная часть гена хлорамфениколацетилтрансферазы. В таблице приведены значения коэффициента индукции гена САТ под действием дексаметазона и число опытов. Достоверность разницы оценивалась с помощью критерия Стьюдента для малых выборок. * P>0, 95 относительно p. TKCAT; ** P >0, 95 относительно p. MT 2 CAT.
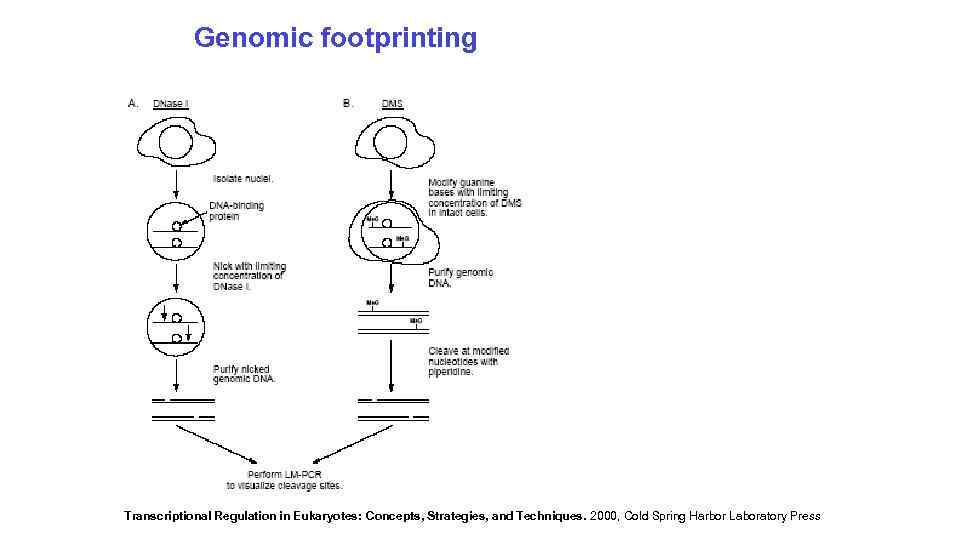 Genomic footprinting Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold Spring Harbor Laboratory Press
Genomic footprinting Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold Spring Harbor Laboratory Press
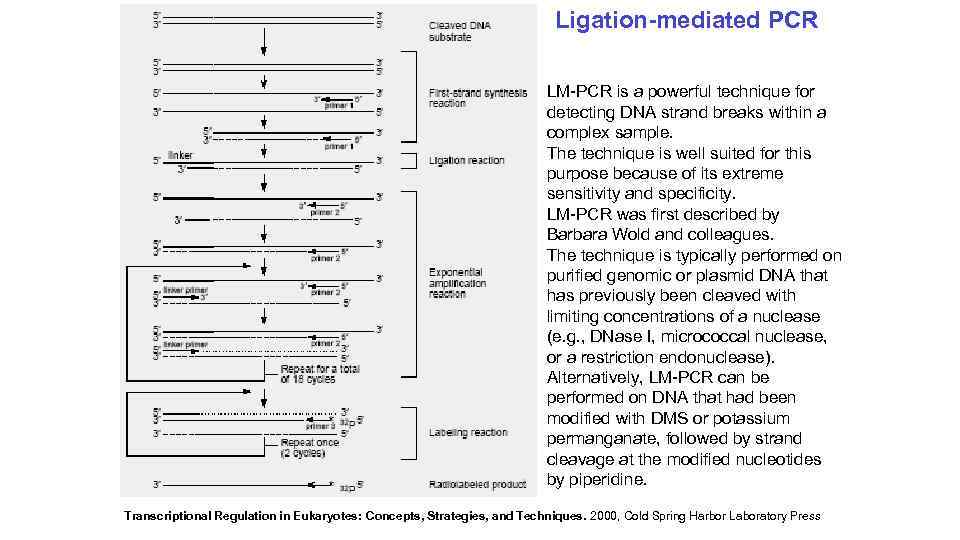 Ligation-mediated PCR LM-PCR is a powerful technique for detecting DNA strand breaks within a complex sample. The technique is well suited for this purpose because of its extreme sensitivity and specificity. LM-PCR was first described by Barbara Wold and colleagues. The technique is typically performed on purified genomic or plasmid DNA that has previously been cleaved with limiting concentrations of a nuclease (e. g. , DNase I, micrococcal nuclease, or a restriction endonuclease). Alternatively, LM-PCR can be performed on DNA that had been modified with DMS or potassium permanganate, followed by strand cleavage at the modified nucleotides by piperidine. Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold Spring Harbor Laboratory Press
Ligation-mediated PCR LM-PCR is a powerful technique for detecting DNA strand breaks within a complex sample. The technique is well suited for this purpose because of its extreme sensitivity and specificity. LM-PCR was first described by Barbara Wold and colleagues. The technique is typically performed on purified genomic or plasmid DNA that has previously been cleaved with limiting concentrations of a nuclease (e. g. , DNase I, micrococcal nuclease, or a restriction endonuclease). Alternatively, LM-PCR can be performed on DNA that had been modified with DMS or potassium permanganate, followed by strand cleavage at the modified nucleotides by piperidine. Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques. 2000, Cold Spring Harbor Laboratory Press
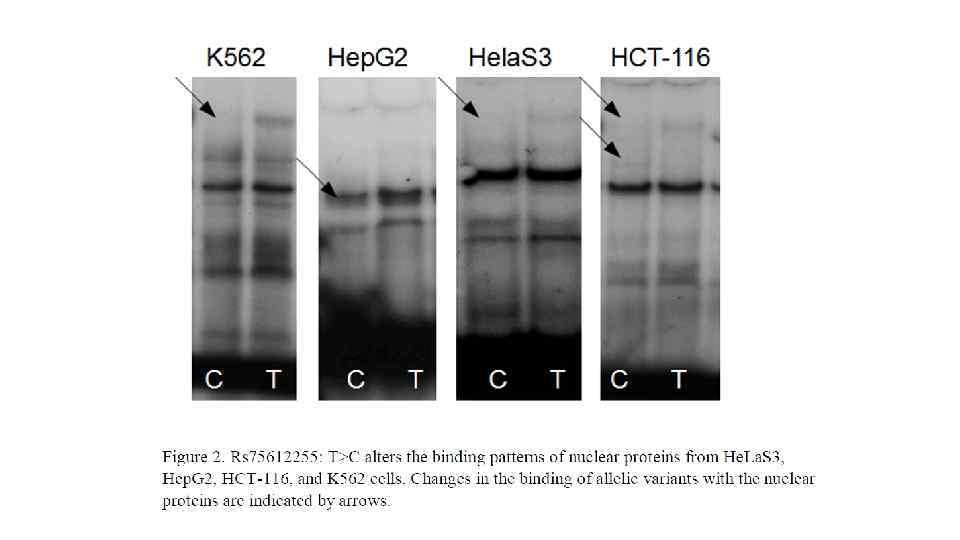
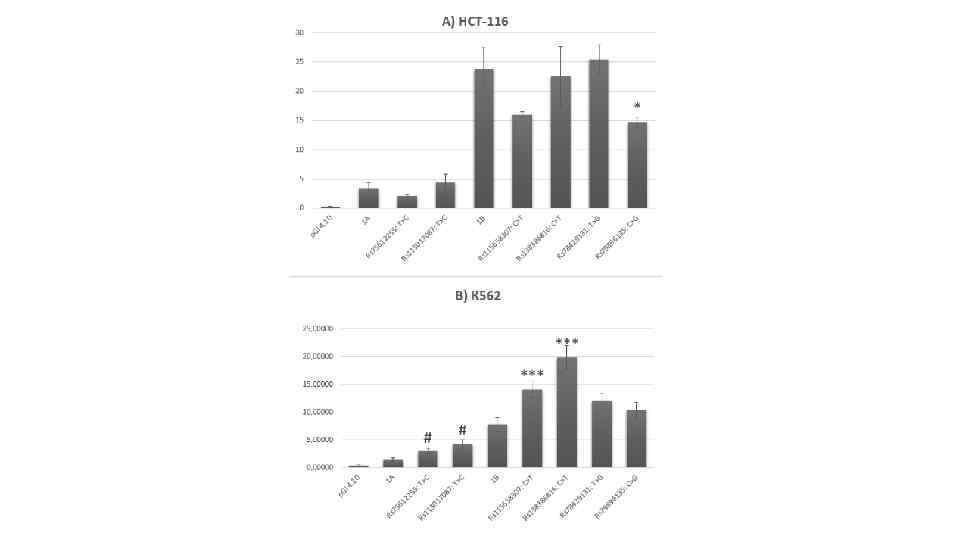
 In vivo genomic footprinting demonstrates involvement of CRE-II site in SMN 2 gene expression Intact nuclei isolated from brain and liver cells of Smn/ mice expressing eight copies of human SMN 2 gene were exposed to limited dimethylsulfate treatment, and genomic DNA was isolated. The DNA was then subjected to piperidine treatment followed by LMPCR amplification of the SMN 2 promoter. The LM-PCR products were separated on 6% sequencing gel and exposed to x-ray film. N, naked DNA, where DNA was treated with dimethyl sulfate and piperidine after isolation; C, DNA isolated from control cells treated in vivo with dimethyl sulfate. The stars and arrows indicate hypersensitive and protected G residues, respectively. A, lower strand spanning from 210 to 283 bp. B, upper strand spanning from 312 to 443 bp of the SMN 2 promoter S. Majumder, S. Varadharaj, K. Ghoshal et al. Identification of a Novel Cyclic AMP-response Element (CRE-II) and the Role of CREB-1 in the c. AMP-induced Expression of the Survival Motor Neuron (SMN) Gene //The Journal of Biological Chemistry / Vol. 279, No. 15, Issue of April 9, pp. 14803– 14811, 2004
In vivo genomic footprinting demonstrates involvement of CRE-II site in SMN 2 gene expression Intact nuclei isolated from brain and liver cells of Smn/ mice expressing eight copies of human SMN 2 gene were exposed to limited dimethylsulfate treatment, and genomic DNA was isolated. The DNA was then subjected to piperidine treatment followed by LMPCR amplification of the SMN 2 promoter. The LM-PCR products were separated on 6% sequencing gel and exposed to x-ray film. N, naked DNA, where DNA was treated with dimethyl sulfate and piperidine after isolation; C, DNA isolated from control cells treated in vivo with dimethyl sulfate. The stars and arrows indicate hypersensitive and protected G residues, respectively. A, lower strand spanning from 210 to 283 bp. B, upper strand spanning from 312 to 443 bp of the SMN 2 promoter S. Majumder, S. Varadharaj, K. Ghoshal et al. Identification of a Novel Cyclic AMP-response Element (CRE-II) and the Role of CREB-1 in the c. AMP-induced Expression of the Survival Motor Neuron (SMN) Gene //The Journal of Biological Chemistry / Vol. 279, No. 15, Issue of April 9, pp. 14803– 14811, 2004
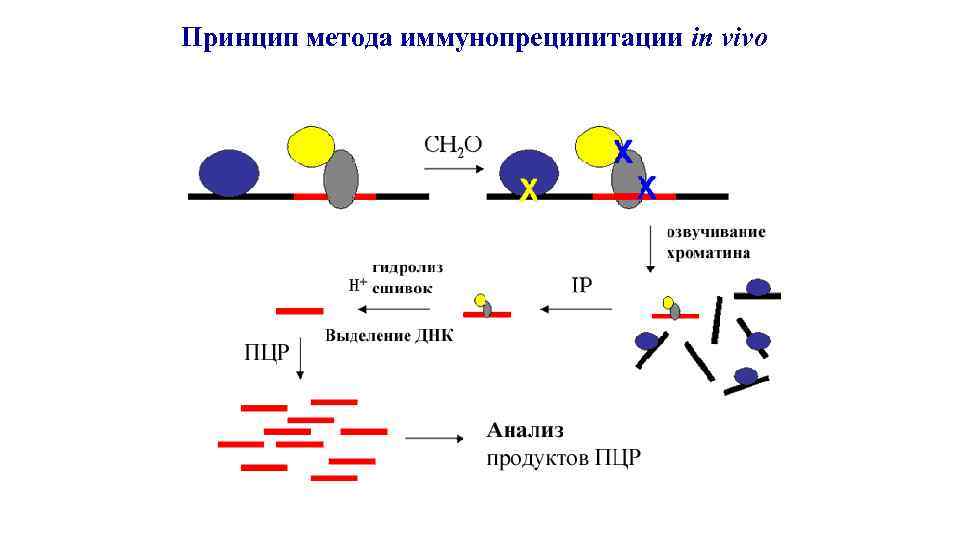 Принцип метода иммунопреципитации in vivo
Принцип метода иммунопреципитации in vivo
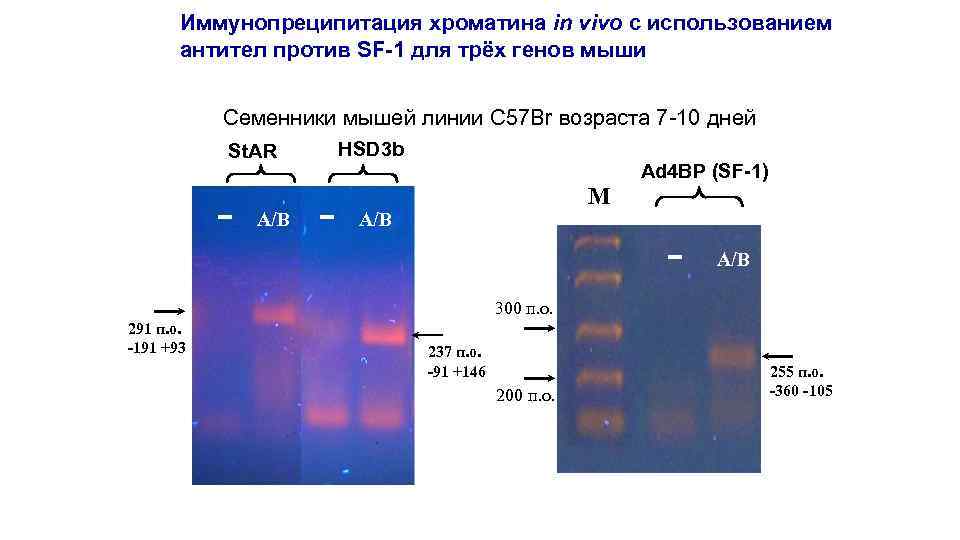 Иммунопреципитация хроматина in vivo с использованием антител против SF-1 для трёх генов мыши Семенники мышей линии C 57 Br возраста 7 -10 дней HSD 3 b St. AR - A/B - М A/B Ad 4 BP (SF-1) - A/B 300 п. о. 291 п. о. -191 +93 237 п. о. -91 +146 200 п. о. 255 п. о. -360 -105
Иммунопреципитация хроматина in vivo с использованием антител против SF-1 для трёх генов мыши Семенники мышей линии C 57 Br возраста 7 -10 дней HSD 3 b St. AR - A/B - М A/B Ad 4 BP (SF-1) - A/B 300 п. о. 291 п. о. -191 +93 237 п. о. -91 +146 200 п. о. 255 п. о. -360 -105
 TRANSFAC and its module TRANSCompel: transcriptional gene regulation in eukaryotes V. Matys*, O. V. Kel-Margoulis, E. Fricke, I. Liebich, S. Land, A. Barre-Dirrie, I. Reuter, D. Chekmenev, M. Krull, K. Hornischer, N. Voss, P. Stegmaier, B. Lewicki-Potapov, H. Saxel, A. E. Kel and E. Wingender Table TRANSFAC Rel. 7. 0 FACTOR 6133 Homo sapiens 1040 Mus musculus 765 D. melanogaster 233 A. thaliana 1751 S. Cerevisiae 368 SITE 7915 MATRIX 398 GENE (all entries) 2397 H. sapiens 608 M. musculus 417 D. melanogaster 145 A. thaliana 115 S. cerevisiae 195 GENE (entries with SITE links) 1504 CLASS 50 CELL 1307 TRANSCompel Rel. 7. 0 COMPEL (composite elements) 322
TRANSFAC and its module TRANSCompel: transcriptional gene regulation in eukaryotes V. Matys*, O. V. Kel-Margoulis, E. Fricke, I. Liebich, S. Land, A. Barre-Dirrie, I. Reuter, D. Chekmenev, M. Krull, K. Hornischer, N. Voss, P. Stegmaier, B. Lewicki-Potapov, H. Saxel, A. E. Kel and E. Wingender Table TRANSFAC Rel. 7. 0 FACTOR 6133 Homo sapiens 1040 Mus musculus 765 D. melanogaster 233 A. thaliana 1751 S. Cerevisiae 368 SITE 7915 MATRIX 398 GENE (all entries) 2397 H. sapiens 608 M. musculus 417 D. melanogaster 145 A. thaliana 115 S. cerevisiae 195 GENE (entries with SITE links) 1504 CLASS 50 CELL 1307 TRANSCompel Rel. 7. 0 COMPEL (composite elements) 322
 Transcription regulatory regions database (TRRD): its status in 2002 Kolchanov N. A. , Ignatieva E. V. , Ananko E. A. , Podkolodnaya O. A. , Stepanenko I. L. , Merkulova T. I. , Pozdnyakov M. A. , Podkolodny N. L. , Naumochkin A. N. , Romashchenko A. G.
Transcription regulatory regions database (TRRD): its status in 2002 Kolchanov N. A. , Ignatieva E. V. , Ananko E. A. , Podkolodnaya O. A. , Stepanenko I. L. , Merkulova T. I. , Pozdnyakov M. A. , Podkolodny N. L. , Naumochkin A. N. , Romashchenko A. G.