ения геномов. Секвенирование.ppt
- Количество слайдов: 77

Методы изучения геномов Секвенирование Д. б. н. , профессор Мельнов Сергей Борисович

Современные подходы к секвенированию ДНК, их достоинства и недостатки Секвенированием называют один из методов расшифровки нуклеотидной последовательности нуклеиновых кислот в молекуле ДНК. Автором этого метода по праву считают Ф. Сэнгера, который в начале семидесятых годов изобрел первый метод прямого ферментативного секвенирования ДНК.

Существует два основных метода секвенирования; метод Максама Гилберта (основан на химическом расщеплении ДНК по одному основанию) и метод Сэнгера (дидезокси метод). Метод Сэнгера более надежен и прост в исполнении, и на практике его используют чаше.

В Оксфордской лаборатории был разработан метод, который позволяет распознавать индивидуальные азотистые основания в нити ДНК. Благодаря этой технологии, по мнению ученых, появилась возможность создания простого и более дешевого метода секвенирования ДНК.

В основе этой методики лежит разрушение нитей ДНК на индивидуальные нуклеотиды, которые проходят через белок «нанопору» , закрепленную на двойном липидном слое. Помещение пористого белка в солевой раствор и приложение к нему напряжения приводит к протеканию тока через нанопоры, а это, в свою очередь, приводит к тому, что «адаптер» (трубчатая молекула на основе циклодекстрина, встроенная в белок) связывает каждое основание по мере его прохождения через поры. Во время связывания основания с порами понижается сила тока, при этом для каждого из четырех азотистых оснований наблюдается свойственный только ему уровень уменьшения этого показателя, что и позволяет их различить.

Секвенатор с пористым детектором позволяет считывать ДНК основание за основанием. Причем, отсутствует необходимость в дополнительных операциях, направленных на приготовление образца, обработку и хранение данных.

Команда исследователей из Бостонского Университета (Boston University) предложила новый метод ДНК секвенирования на кремниевом чипе. Как говорят специалисты, элегантное технологическое решение, которое возможно благодаря нанотехнологиям, сможет сделать секвенирование генома более быстрым и дешевым.

Профессор биомедицины, Амит Меллер (Amit Meller), в новой работе показал, что с помощью электромагнитных полей можно сделать быструю сортировку фрагментов ДНК. Приложив между слоев кремния и проводника напряжение, через 4 х нанометровую нанопору перемещаются отрезки ДНК. При этом нужно небольшое количество отрезков ДНК для детектора, что ранее ученые достигали с большим трудом.

Традиционно для секвенирования нужно предварительно изготовить миллионы копий молекул ДНК, а это очень много для анализа. В текущем исследовании можно обойтись гораздо меньшим количеством. Кроме успешного метода секвенирования, ученые смогут ближе понять электрофизику молекул ДНК и их поведения в наножидкостных системах. Как говорят исследователи, с помощью секвенатора на нанопоре возможно добиться уменьшения необходимых для секвенирования ДНК фрагментов до 10000– 100000.

Метод Сэнгера Обычному ныне ферментативному методу секвенирования ДНК с помощью дидезокситерминаторов предшествовал так называемый плюс/минус метод.

В его основе также лежало построение второй радиоактивно меченной (за счет присутствия в реакционной смеси одного меченого наряду с тремя немеченными д. НТФ) цепи ДНК по существующей матрице с помощью ДНК полимеразы I (Кленовского фрагмента). В качестве затравки использовался короткий синтетический олигонуклеотидный десятизвенный праймер. На втором этапе гетерогенные продукты удлинения данного праймера отделяли хроматографией на колонке от непрореагировавших ингредиентов и делились на восемь частей. Четыре части были использованы в минус системе, где в каждой реакции присутствовало по три разных д. НТФ, а четвертый отсутствовал.

Дальнейшее построение цепи новой порцией Кленовского фрагмента ДНК полимеразы I приводило к удлинению цепи ДНК в каждой реакции до тех пор, пока в матрице не оказывался нуклеотид, комплементарный отсутствующему. Вторые четыре части с добавлением только одного д. НТФ (в каждую – разный) выдерживались в плюс системе, где в качестве фермента использовалась 3'→ 5‘ экзонуклеазная активность ДНК полимеразы фага Т 4. Так, последовательное отщепление д. НМФ с 3‘ конца ДНК шло до тех пор, пока не встречался д. НМФ, д. НТФ которого присутствовал в реакционной смеси и тогда в действие вступала уже полимеразная активность Т 4 ДНК полимеразы и фрагмент ДНК приобретал свой окончательный размер.

По завершению всех этих реакций радиоактивно меченные продукты разделялись в полиакриламидном гель электрофорезе и экспонировались на рентгеновскую пленку, по которой можно было восстановить нуклеотидную последовательность секвенируемого участка ДНК.

В основе метода секвенирования ДНК, разработанного Сэнгером и соавт. , называемого также методом секвенирования путем терминации цепи, лежал принцип ферментативного построения комплементарной цепи ДНК по существующей одноцепочечной матрице при происходящем в разных местах цепи ДНК ингибировании ее дальнейшего роста.

Составляющими секвенирования являлись: • одноцепочечная матрица ДНК; • короткая затравочная молекула, комплементарная определенному участку матрицы; • ДНК полимераза; • 2‘ дезоксинуклеотид 5‘ трифосфаты (д. НТФ); • 2‘ 3‘ дидезоксинуклеотид 5‘ трифосфаты (дд. НТФ); • Реакционный буфер с ионами Mg 2+

Ключевым моментом секвенирования являлась терминация построения комплементарной цепи ДНК, происходящая при включении ДНК полимеразой модифицированных аналогов природных субстратов дидезоксинуклеотид трифосфатов, являющихся известными ингибиторами ДНК полимеразы. Отсутствие второго гидроксильного остатка в З' положении рибозного кольца у дидезоксипроизводных приводила к невозможности присоединения к ним следующего нуклеотида, и рост цепи таким обра зом прерывался.

Терминирование происходило одновременно как бы в двух режимах заданном и случайном. Заданность определялась тем, что в одной отдельной пробирке наряду со всеми четырьмя д. НТФ (один из которых был радиоактивно меченным) присутствовал только один какой нибудь конкретный дд. НТФ, случайность же происходила от того, что включение данного дд. НМФ в растущую цепь ДНК было произвольным, но, естественно, с учетом принципа комплементарности. То есть происходила некая конкуренция между д. НТФ и дд. НТФ при их выборе ДНК полимеразой. Поскольку в четырех реакционных пробирках (по типу оснований А, С, G и Т) присутствовало огромное количество молекул секвенируемой ДНК, во много раз превосходящее ту длину фрагмента, которую могла построить ДНК полимераза, то по теории вероятности каждое положение этого типа оснований во фрагменте ДНК оказывалось представленным соответствующим дд. НМФ.

Другим ключевым моментом являлся гетерогенный размер полученных фрагментов ДНК. Их гетерогенность определялась тем, что все З' концы фрагментов вновь синтезируемой цепи ДНК во всех четырех реакционных пробирках были терминированы соответствующими дд. НМФ и, таким образом, представляли собой полный набор всех возможных длин в пределах секвенируемого участка, построенного ДНК полимеразой. Что касается 5' конца всех этих фрагментов ДНК, принадлежащих новой цепи, то он должен быть для всех фрагментов строго одинаковым и принадлежать 5' концу исходной затравочной молекулы.

Продукты реакций терминирования подвергались денатурации и уже одноцепочечные меченые фрагменты, имевшие гомогенный 5' конец и гетерогенные З' концы, разделялись по длине высоковольтным электрофорезом высокого разрешения в полиакриламидном геле, позволяющим разделять фрагменты ДНК, отличающиеся всего на один нуклеотид, в четырех соседних дорожках, соответствующих специфическим терминирующим реакциям, например в последовательности Т, С, G, А. По завершению электрофореза гель экспонировался на рентгеновскую пленку и по прошествии некоторого времени (обычно от одних до двух суток) с проявленной пленки можно было "читать" последовательность нуклеотидов секвенируемого участка ДНК, начиная с нижней части геля и последовательно поднимаясь вверх по этим четырем дорожкам, соответствующим одному фрагменту ДНК.
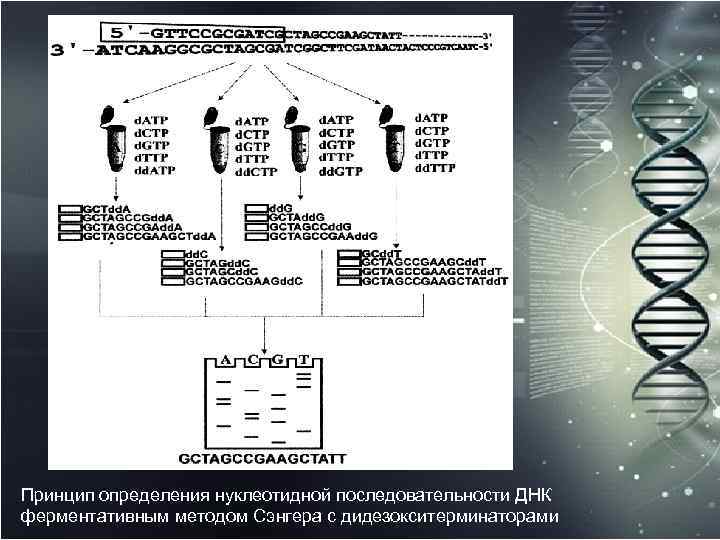
Принцип определения нуклеотидной последовательности ДНК ферментативным методом Сэнгера с дидезокситерминаторами

Автоматическое секвенирование В настоящее время под автоматическим секвенированием ДНК понимается, в первую очередь, электрофоретическое разделение флуоресцентно меченных продуктов терминирующих реакций с помощью специальных приборов автоматических секвенаторов ДНК. Другой аспект автоматизации процесса секвенирования ДНК заключается в наработке матриц для их последующего ферментативного секвенирования, а также проведении секвенирующих реакций (как в ферментативном методе, так и в методе химической деградации) с помощью лабораторных биороботов.

Выяснение начальной последовательности секвенируемого фрагмента ДНК а) Если о нем ничего неизвестно, то можно воспользоваться «устаревшим» методом Максама и Гилберта, который для первых десятков нуклеотидов достаточно быстро дает вполне надежные сведения.

б) В случае, секвенирования определенного гена, направляющего биосинтез вполне конкретного белка известной структуры, и если есть основание полагать, что этот ген (или хотя бы его начало) входит в состав обследуемого фрагмента ДНК, то можно, используя ЧИП методику, по 6— 7 первым аминокислотам данного белка определить последовательность в 18— 21 рибонуклеотидов в ведущей его синтез и. РНК, а значит и такой же длины начальную последовательность дезоксирибонуклеотидов соответствующего гена. Синтезированный праймер в этом случае просто воспроизведет расшифрованную часть и. РНК.

Присоединение к началу секвенируемого фрагмента изве стной последовательности ДНК для синтеза праймера по ней Эту операцию проделывают при помощи возможности встраивания фрагментов чужеродной ДНК, длиною в тысячи нуклеотидов, в плазмиды. Можно естественным образом связать начало (и конец) секвенируемого фрагмента ДНК с известной последовательностью нуклеотидов плазмиды.

Для того, чтобы точно узнать в какое место плазмиды включился фрагмент ДНК и как его потом «вырезать» обратно вместе с известным, ему предшествующим куском плазмиды — достаточным по своим размерам для посадки на него надежного праймера, необходимо подобрать две рестриктазы, сайты узнавания которых в пределах поликлонового сайта отстояли бы друг от друга достаточно далеко, на расстояние, большее, чем длина будущего праймера секвенирования.

Одна рестриктаза, сайт узнавания которой находился бы в «конце» поликлонового сайта (если определились с направлением будущего секвенирования), должна послужить для встраивания подлежащего секвенированию фрагмента ДНК. Вторая — для его последующего вырезания из плазмиды вместе с участком поликлонового сайта, разделявшим сайты узнавания обеих рестриктаз. Вся последовательность нуклеотидов на этом участке известна и потому синтезировать комплементарный к нему праймер не представит труда.

Последующее секвенирование таким образом удлиненного фрагмента ДНК либо начнется с его первого нуклеотида, либо с нескольких предшествующих нуклеотидов, относящихся к копированию присоединенного участка поликлонового сайта. Эти нуклеотиды легко узнать и отделить от последовательности самого исследуемого фрагмента ДНК. В последнее время для умножения количества секвенируемой ДНК клонированием и создания места посадки праймера стали широко использовать бактериофаг М 13.

В процессе секвенирования матричная ДНК должна все время оставаться строго однонитевой, без возможных «шпилек» — локальных двунитевых участков, образующихся в силу наличия в этой ДНК близко расположенных, пусть небольших, но комплементарных последовательностей. То же самое относится и к меченым отрезкам ДНК в процессе их разделения электрофорезом. В процессе комплементарного синтеза по методу Сэнгера (он повторяется многократно — в несколько циклов) такая опасность не возникает, так как в каждом цикле имеется стадия предварительной полной денатурации при температуре 96°С, а само копирование осуществляется при температуре 60°С. Но электрофорез приходится проводить в сильно денатурирующей среде: 80% ном растворе формамида, содержащим 5 т. М раствор ЭДТА.

Необходимо заботиться о том, чтобы при электрофорезе количество флюоресцентно меченого материала в каждой полосе (даже такой, в которой содержится наименьшее число отрезков ДНК) было достаточным для надежной регистрации. Вместе с тем, этого желательно добиться без чрезмерного расходования исходного материала ДНК, который обычно дефицитен. Поэтому реакцию ограниченного (включением дидезоксирибонуклеотида) матричного синтеза на том реальном количестве секвенируемой ДНК, которое направляется в эксперимент, повторяют многократно. Это делается точно так же, как в симметричной ПЦР реакции. Но с тем отличием, что используется только один, начальный праймер ( «асимметричная ПЦР реакция» ). Этап элонгации заканчивается по достижении конца матрицы (если он не был остановлен раньше). Комплементарный синтез от второго праймера в обратном направлении здесь не фигурирует.

Готовят 20 мкл инкубационной смеси, в которой содержится: одно или двунитевая нить ДНК секвенируемого фрагмента (в последнем случае будет копироваться только одна из нитей — та, для которой синтезирован праймер), избыточное количество 4 х нормальных дезоксирибонуклеозидтрифосфатов, ограниченное количество 4 х флюоресцентных дидезоксирибонуклеозидтрифосфатов, термостабильная Taq ДНК полимераза и в достаточном количестве синтетический праймер (все это в буфере с Mg. Cl 2). Инкубационная смесь прогревается предварительно в тонкостенной полипропиленовой пробирочке емкостью в 0, 2 или 0, 5 мл при 96°С для полной денатурации ДНК. Затем ее помещают во встроенный в прибор термоблок, где автоматически осуществляется 25 последовательных циклов, каждый из которых состоит из следующих, очень быстро сменяющих друга термических экспозиций: — 10 секунд при 96°С, — 5 секунд при 50°С, — 4 минуты при 60°С.

Экспозиция при 96°С освобождает однонитевые матричные молекулы секвенируемой ДНК от новосинтезированных укороченных копий своей последовательности вместе с праймерами и Taq ДНК полимеразой. При 50 °С новые молекулы праймера гибридизуются с начальными участками освободившихся матриц. Вслед за ними на эти матрицы садятся и молекулы Taq ДНК полимеразы. В течение 4 х минут они ведут комплементарный синтез ДНК вплоть до момента случайного включения флюоресцентно меченого дидезоксирибонуклеотида.

Количество копий при таком однонаправленном синтезе не увеличивается многократно, как при симметричной ПЦР реакции с двумя праймерами, а возрастает в лучшем случае всего в 25 раз — по числу циклов. Однако и этого оказывается достаточно для того, чтобы ограничить количество исходно вносимой ДНК 0, 2 или 0, 5 микрограммами (соответственно для однонитевой или двунитевой молекулы). В интересах реальной ориентировки, не мешает заметить, что для фрагмента двунитевой ДНК длиной в 1000 пар нуклеотидов в 0, 5 мкгр содержится более 10 копий этого фрагмента. В каждом из циклов ПЦР реакции все они снова выступают в качестве матриц для случайным образом укороченных копирований. Это обеспечивает в конце концов достаточное число одинаковых по длине отрезков флюоресцентно меченой ДНК в любой полосе электрофоретического разделения.

Во избежание флюоресцентного фона смесь этих отрезков следует очистить от неиспользованных меченых диде зоксинулеозидтрифосфатов. С этой целью новосинтезированные отрезки ДНК (вместе с исходными, немеченными матрицами) осаждают добавлением 1 мл 70% го этанола и центрифугированием в микропробирках на 1, 5 мл. Осадок растворяют в 4 мкл 80% ного формамида с ЭДТА, прогревают при 96°С 2 минуты и в количестве 2 мкл вносят на старт трека пластины ПААГ.

Пиросеквенирование Метод пиросеквенирования ДНК основан на многошаговой детекции неорганического пирофосфата, образующегося из д. НТФ (или дд. НТФ) в процессе присоединения ДНК полимеразой оответствующего с нуклеотидмонофосфата к 3' концу растущей цепи ДНК, иммобилизованной на твердом носителе

Чтобы понять принцип работы необходимо рассмотреть ряд ферментативных реакций: ДНК полимераза (d. NMP)n + d. NTP → (d. NMP)n+1 + РРi АТФ сульфурилаза PPi + APS → ATP + SO 2 4 Люцифераза ATP + люциферин + O 2 → AMP + PPi + оксилюциферин + CO 2 + hv где, (d. NMP)n ДНК, а n число нуклеотидных остатков, PPi неорганический пирофосфат, APS аденозин 5' фосфосульфат, hv квант света.

Как видно из приведенной серии последовательных реакций, образующийся в процессе присоединения очередного нуклеотида неорганический пирофосфат затем под действием АТФ сульфурилазы преобразует аденозинфосфосульфат в молекулу АТФ, которая, в свою очередь, необходима для осуществления ферментативной реакции окисления люциферина люциферазой с выделением квантов света.

Принцип высокопроизводительного пиросеквенирования ДНК Весь геном, все его молекулы ДНК, случайным образом фрагментируются на кусочки по 300 – 500 пар оснований. Затем комплементарные цепи фрагмента разделяются, к каждой цепи фрагментов пришивается одинаковый для всех олигонуклеотид «адаптер» , который позволяет отдельным цепям налипать на пластиковые бусинки. (Последовательность этого олигонуклеотида позволяет позднее в процессе секвенирования распознавать ДНК матрицу. )

При этом смесь разъединённых на комплементарные цепи фрагментов разбавляют таким образом, что каждая бусинка получает лишь по одной индивидуальной цепи. Каждая бусинка оказывается заключённой в капельку, окруженную маслом и содержащую смесь для осуществления полимеразной цепной реакции (ПЦР), которая и проходит отдельно в каждой капельке эмульсии (так называемая эмульсионная ПЦР, э. ПЦР). Это приводит к «клональной амплификации» цепей ДНК, т. е. к тому, что на поверхности бусинки удерживается уже не одна, а около 10 млн. копий ( «клонов» ) уникальной ДНК матрицы. Далее эмульсия разрушается, вновь двуцепочечные фрагменты ДНК (образовавшиеся в ходе ПЦР) разделяются, и бусинки, несущие одноцепочечные копии ДНК матрицы, помещаются в лунки «предметного стекла» — слайда особой конструкции. Каждая лунка такого слайда образует отдельный пиколитровый «реактор» , в котором и будет происходить реакция секвенирования.

Слайд представляет собой срез блока, полученного путём нескольких раундов вытягивания и сплавления оптических волокон. В результате каждой итерации, диаметр индивидуальных волокон уменьшается по мере того, как волокна формируют пучки шестигранной упаковки увеличивающегося поперечного диаметра. Каждое волокно имеет сердечник диаметром 44 мкм, окружённый 2– 3 мкм слоем плакировки (оболочки). Затем сердечники вытравливаются, и в результате получаются лунки ≈55 мкм глубиной, с расстоянием ≈50 мкм между центрами соседних лунок. Объём таких «реакторов» — 75 пиколитров; плотность размещения на поверхности слайда — 480 лунок на квадратный миллиметр. Каждый слайд несёт около 1, 6 миллионов лунок, в каждую из которых попадает одна бусинка с ДНК матрицей. Слайд помещается в проточную камеру таким образом, что над отверстиями лунок создаётся канал высотой 300 мкм, по которому в лунки поступают необходимые реактивы.

Доставляемые в проточную камеру реактивы текут в слое, перпендикулярном оси лунок. Такая конфигурация позволяет одновременно осуществлять реакции на бусинках, несущих ДНК матрицы, внутри отдельных лунок. Добавление и удаление реагентов и продуктов реакции происходит за счёт конвекционного и диффузионного переноса. Временные рамки диффузии между потоком и лунками составляют порядка 10 секунд и зависят от высоты проточной камеры и глубины лунок.

Глубина лунок тщательным образом рассчитана исходя из следующих соображений: • Лунки должны быть достаточно глубокими, чтобы бусинки, несущие ДНК матрицу, не выскакивали из них под действием конвекции; • Они должны быть достаточно глубокими, чтобы исключить диффузию продуктов реакции из лунок, где имело место включение нуклеотида, в лунки, где включения не произошло (см. ниже); • Лунки должны быть мелкими настолько, сколько требуется для осуществления быстрой диффузии нуклеотидов в лунку и быстрого вымывания оставшихся нуклеотидов и продуктов реакции в конце каждого цикла, что, в свою очередь, необходимо для обеспечения высокой продуктивности секвенирования и снижения расходов реактивов.

Помимо бусинок с ДНК матрицей, в каждую лунку «насыпают» ещё бусинок помельче — каждая с «сидящими» на её поверхности (иммобилизованными) ферментами, необходимыми для пирофосфатного секвенирования. Нуклеотиды (одного вида за раз) и другие реактивы, необходимые для реакции секвенирования, подаются последовательно в проточную камеру, куда помещается слайд. Каждый раз, когда определённый нуклеотид встраивается в растущую цепь ДНК в какой нибудь из лунок, в ней высвобождается молекула пирофосфата, которая, в свою очередь, является необходимым предшественником компонента другой ферментативной реакции. Её катализирует особый фермент, люцифераза светлячка Photinus pyralis. Но для её осуществления необходим аденозинтрифосфат (АТФ).

Новообразованный пирофосфат превращается в лунке в АТФ под действием ещё одного фермента — АТФ сульфурилазы. И тогда люцифераза окисляет люциферин до оксилюциферина, а эта реакция сопровождается хемилюминесценцией — по простому, маленькой вспышкой света. Дно слайда находится в оптическом контакте с оптико волоконным световодом, подключённым к прибору с зарядовой связью (CCD сенсор, charge coupled device). Это позволяет регистрировать излучаемые фотоны со дна каждой индивидуальной лунки, в которой произошло встраивание известного нуклеотида.
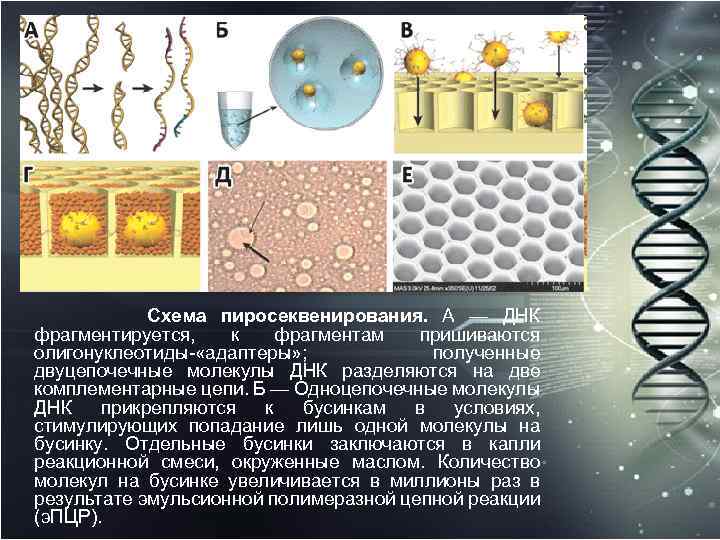
Схема пиросеквенирования. А — ДНК фрагментируется, к фрагментам пришиваются олигонуклеотиды «адаптеры» ; полученные двуцепочечные молекулы ДНК разделяются на две комплементарные цепи. Б — Одноцепочечные молекулы ДНК прикрепляются к бусинкам в условиях, стимулирующих попадание лишь одной молекулы на бусинку. Отдельные бусинки заключаются в капли реакционной смеси, окруженные маслом. Количество молекул на бусинке увеличивается в миллионы раз в результате эмульсионной полимеразной цепной реакции (э. ПЦР).
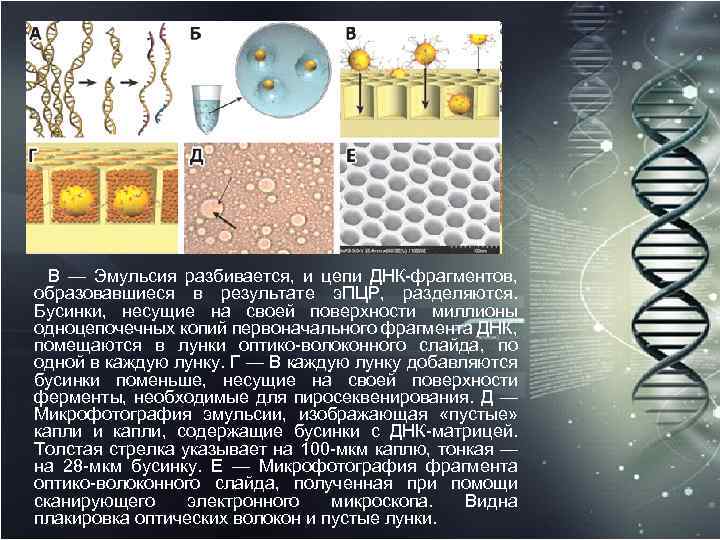
В — Эмульсия разбивается, и цепи ДНК фрагментов, образовавшиеся в результате э. ПЦР, разделяются. Бусинки, несущие на своей поверхности миллионы одноцепочечных копий первоначального фрагмента ДНК, помещаются в лунки оптико волоконного слайда, по одной в каждую лунку. Г — В каждую лунку добавляются бусинки поменьше, несущие на своей поверхности ферменты, необходимые для пиросеквенирования. Д — Микрофотография эмульсии, изображающая «пустые» капли и капли, содержащие бусинки с ДНК матрицей. Толстая стрелка указывает на 100 мкм каплю, тонкая — на 28 мкм бусинку. Е — Микрофотография фрагмента оптико волоконного слайда, полученная при помощи сканирующего электронного микроскопа. Видна плакировка оптических волокон и пустые лунки.

Связывая зарегистрированные от каждой лунки вспышки с типом нуклеотида, присутствующего в проточной камере в данный момент времени, компьютер последовательно отслеживает рост цепочек ДНК в сотнях тысяч лунок одновременно. Время, необходимое для протекания ферментативной реакции, производящей детектируемую «вспышку» , составляет порядка 0, 02– 1, 5 секунд. Таким образом, скорость реакции определяется скоростью массопереноса, что оставляет место для улучшений за счёт ускорения доставки реактивов. После поступления в проточную камеру каждого нуклеотида, она промывается раствором, содержащим фермент апиразу. Таким образом перед тем, как «запустить» в камеру следующий нуклеотид, из всех лунок удаляются любые нуклеотиды, остававшиеся там от предыдущего раунда.

Включение того или иного нуклеотида детектируется в результате высвобождения неорганического пирофосфата и последующего излучения света. Определить лунки, содержащие бусинки с ДНК матрицей, можно, прочитав «последовательность ключ» адаптерного олигонуклеотида, пришитого к началу каждой ДНК матрицы. Из регистрируемого сигнала вычитается уровень фона, затем сигнал нормализуется и корректируется. Интенсивность нормализованного сигнала для каждой конкретной лунки во время поступления в проточную камеру определённого нуклеотида пропорциональна числу встроенных нуклеотидов, если таковые имеются.

Линейность зависимости сохраняется для гомополимеров длиной как минимум в восемь нуклеотидов. При таком секвенировании синтезом очень небольшое число ДНК матриц на каждой бусинке теряет синхронизм, т. е. вырываются вперёд или начинают отставать от других матриц. Причиной этому прежде всего служат остающиеся в лунке нуклеотиды или неполное удлинение цепи. Исправление таких сдвигов необходимо, поскольку потеря синхронизма создаёт кумулятивный эффект, сильно снижающий качество прочтения при увеличении его длины. Исходя из подробной модели лежащих в основе этого эффекта физических процессов, сотрудники компании 454 разработали особый алгоритм, позволяющий оценивать и вносить поправки на «перелёт» и неполную достройку цепи, происходящие в отдельных лунках.

Перед тем, как составить и «записать» окончательную последовательность прочитанной ДНК, из всего массива данных для дальнейшей работы необходимо отобрать высококачественные прочтения и отбросить некачественные. Отбор основывается на наблюдении, что в прочтениях низкого качества велика доля сигналов, не позволяющих отличить циклы, в течение которых произошло включение нуклеотида, от циклов без включения. Такие двусмысленные сигналы — причина ошибок в записи последовательности тдельных рочтений. о п Чтобы увеличить число пригодных для использования прочтений, компанией 454 была разработана особая мера, позволяющая оценивать ab initio вероятность правильного определения нуклеотида в каждой конкретной позиции отдельных прочтений.

Высокая точность расшифровки последовательности достигается тем, что система осуществляет многочисленное прочтение одного и того же фрагмента, что позволяет построить единую обобщённую (так называемую консенсусную) последовательность. Отдельные прочтения одного и того же участка ДНК выравниваются относительно друга исходя из интенсивности сигналов в момент протекания через камеру того или иного нуклеотида, а не на основе последовательности этих прочтений. Затем соответствующие сигналы усредняют, и только тогда записывают полученную последовательность. Такой подход значительно улучшает качество расшифровки последовательности и предоставляет возможность оценки её качества.

Бисульфитное секвенирование Бисульфитное секвенирование (Bisulfite sequencing) — это применение бисульфитной обработки к ДНК для определения паттерна метилирования.

Бисульфитное секвенирование основано на том факте, что обработка ДНК бисульфитом превращает остатки цитозина в урацил, но не затрагивает остатки 5 метилцитозина. Дальнейший анализ сводится к различению однонуклеотидных олиморфизмов, п и к разделению в сиквенсах цитозинов и тиминов. Другие методы вместо обработки бисульфитом для различения метилирования аллелей предполагают специфическую по метилированию рестрикцию и иммунопреципитацию метилированной ДНК (Me. DIP).
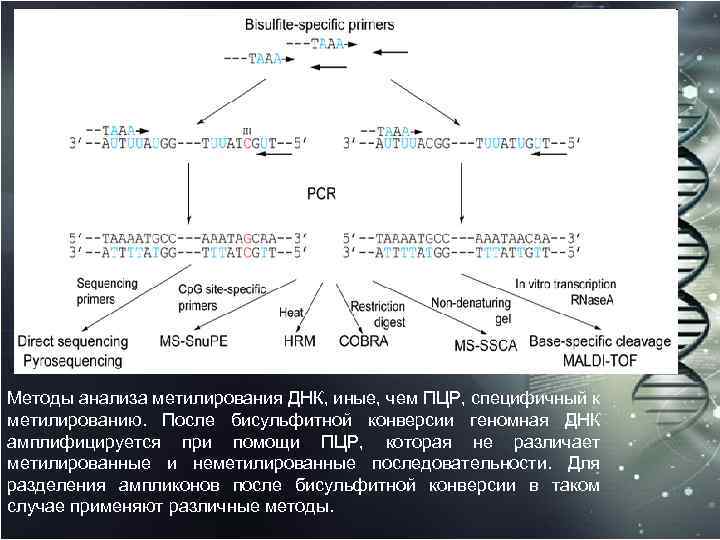
Методы анализа метилирования ДНК, иные, чем ПЦР, специфичный к метилированию. После бисульфитной конверсии геномная ДНК амплифицируется при помощи ПЦР, которая не различает метилированные и неметилированные последовательности. Для разделения ампликонов после бисульфитной конверсии в таком случае применяют различные методы.

Мультиплексное секвенирование ДНК Основано на одновременном использовании целого набора плазмидных векторов plex 00 20, было разработано первоначально для метода секвенирования ДНК путем химической деградации с целью повышения его производительности.

Главный принцип секвенирования ДНК с помощью мультиплексного подхода заключается в одновременном проведении терминирующих реакций со всеми секвенируемыми матрицами ДНК, клонированными изначально в различных специализированных векторах. Затем следует их разделение в секвенирующем полиакриламидном геле, перенос на мембранный фильтр и фиксация перенесенных фрагментов ДНК на этом фильтре. Таким образом, на фильтре оказываются фиксированными "невидимые" лестницы полос всех исследуемых фрагментов ДНК. Для их выявления необходимы этапы молекулярной гибридизации с мечеными олигонуклеотидными зондами, гомологичными каждый раз фрагменту только одного определенного вектора из пула использованных.

20 самостоятельных плазмидных векторов plex 00 20 несут по два различающихся участка для отжига праймеров каждый, что составляет 40 вариантов "тэгов". Аналогичными "тэгами" располагают и одиннадцать М 13 р1 ех и пять Plex векторов. В результате гибридизации с каким либо меченым олигонуклеотидным праймером после радиоавтографии на рентгеновской пленке становились видны полосы ДНК, принадлежащие только одной конкретной матрице. Для выявления следующего клона необходим этап полного удаления первого меченого зонда и повторная гибридизация с новым зондом. И так столько раз, сколько матриц в различающихся векторах было первоначально взято в исследование.

Необходимым условием осуществления мультиплексного секвенирования ДНК является перенос фрагментов ДНК из полиакриламидного секвенирующего геля на мембранный фильтр, поскольку проведение всех этих последовательных процедур гибридизации, окрашивания, отмывки возможно только с мембранным фильтром. Второе условие заключается в прочной сорбции фрагментов ДНК на данном фильтре, выдерживающей многократные инкубации в различных растворах. В противном случае мультиплексное секвенирование будет просто невозможно. Немаловажное значение имеет тип используемой метки.
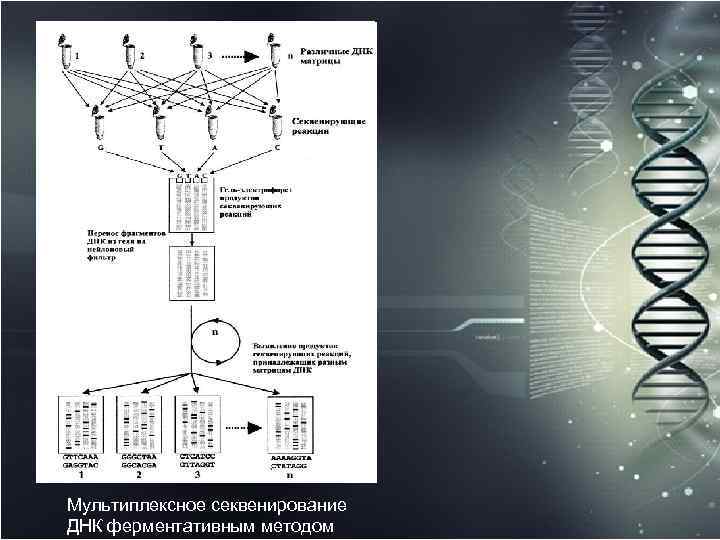
Мультиплексное секвенирование ДНК ферментативным методом

Обычно мультиплексное секвенирование проводится с немеченными фрагментами ДНК и уже потом выявление на фильтре секвенируемых полос ДНК зависит от специфической пробы, меченной тем или иным соединением (радиоактивностью, биотином и др. ), что делает неизбежным этап блот гибридизации. Однако было предложено интересное решение по мультиплексному секвенированию нескольких матриц ДНК, не требующее этапа гибридизации со специфическими пробами. Суть этого способа заключается в одновременном разделении в одном геле по разному меченных фрагментов ДНК, принадлежащих конкретным матрицам, и поэтому этот метод назвали мультиплексным мечением.

В качестве праймеров были использованы олигонуклеотиды, меченные по 5' концу разными гаптенами: биотином, дигоксигенином, 2, 4 динитрофенилом и флуоресцеином. В результате терминирующих реакций, секвенирующего электрофореза, переноса фрагментов ДНК из геля на мембранный фильтр, проводимых одновременно со всеми этими матрицами, появилась возможность сразу, без этапа гибридизации, последовательно выявлять на фильтре полосы ДНК, соответствующие определенным матрицам, что в значительной мере способствовало более быстрому получению данных. Одно из главных отличий этого подхода от обычного мультиплексного секвенирования заключается в том, что здесь электрофоретическому разделению подвергаются продукты терминирующих реакций, уже несущие определенные, различные для каждой матрицы метки.

Для выявления меченных ими фрагментов ДНК, перенесенных на мембранный фильтр, необходимо проведение соответствующих последовательных процедур. Для подобного секвенирования не требуется клонирования или переклонирования вставки в специализированном векторе, поэтому в качестве зондов (в данном случае секвенирующих праймеров) могут выступать стандартные универсальные праймеры, меченные по 5' концу различными гаптенами. Таким образом, становятся необходимы только этапы связывания конъюгата антител к соответствующему гаптену и щелочной фосфатазы (или комплекса стрептавидин/щелочная фосфатаза для биотина) и затем с помощью хемилюминесценции 1, 2 диоксетанового субстрата выявления полос ДНК на фильтре.
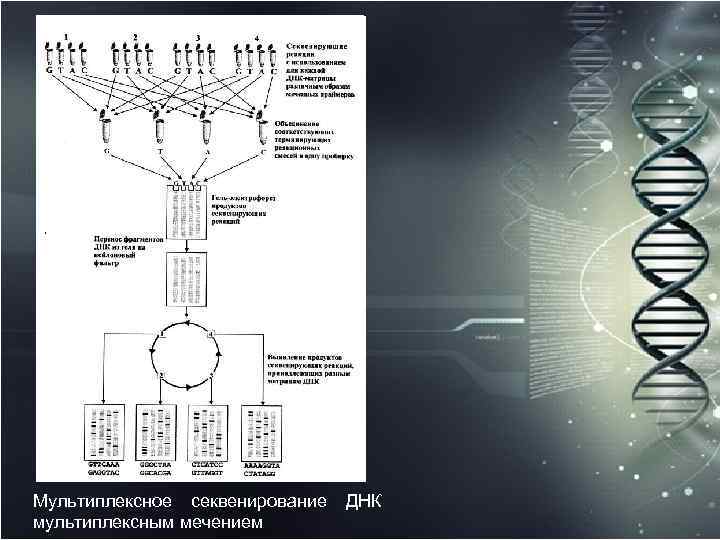
Мультиплексное секвенирование ДНК мультиплексным мечением

Наибольшую чувствительность имеет система с биотинилированным праймером. В 8 раз худшая чувствительность отмечалась при использовании флуоресцеинового гаптена и в 16 для дигоксигенина и 2, 4 динитрофенила. Поскольку при многократных этапах выявления полос ДНК и их последующего удаления с фильтра все же происходит незначительный смыв сорбированных фрагментов ДНК, то в цитируемой работе рекомендовалось последовательное выявление секвенируемой ДНК, несущей дигоксигениновый и/или динитрофенильный гаптены, затем флуоресцеиновыи гаптен, и в завершение биотинилированную метку.

Секвенирование с помощью нанопор Исследовательская группа Амита Меллера (Amit Meller) сообщает о революционной работе по детектированию молекул ДНК, проходящих через нанопоры в кремнии. Для продавливания длинных нитей ДНК через поры (ширина которых составляет около 4 нм) исследователи используют электрическое поле. Измерения позволяют фиксировать прохождение через нанопору единичной молекулы нуклеиновой кислоты.

Результаты исследования демонстрируют возможность определения и анализа меньших количеств ДНК, чем это было возможно до настоящего времени. Он добавляет, что секвенирование или профилирование генома с помощью нанопор позволит уменьшить количества ДНК, необходимые для анализа.

В группе Меллера было решено использовать электрические поля, локализованные около «исходящих отверстий» нанопор для воздействия на длинные отрицательно заряженные нити ДНК и протаскивания их через нанопоры, после прохождения которых молекула ДНК может быть изучена подробнее. Такой подход позволяет использовать меньшее количество копий ДНК для дальнейшего анализа.

В процессе исследования неожиданно было обнаружено, что нити ДНК с большей длиной проходят через поры с большей скоростью. Это обстоятельство позволяет говорить о том, что система из нанопор может быть оптимизирована для детектирования длинных нитей ДНК, состоящих из десятков тысяч и даже большего количества пар азотистых оснований. Это обстоятельство может привести к существенному ускорению процедуры анализа генома, позволяя непосредственно анализировать длинные цепи ДНК, вместо того чтобы нарезать их и восстанавливать их структуру по фрагментам коротких отрезков.

Существующие технологии амплификации ДНК ограничивают размеры анализируемой ДНК тысячей пар оснований. Поскольку новый метод позволяет избавиться от процедуры амплификации, он не только снижает стоимость, временные затраты и уровень ошибок, характерные для анализа ДНК прежними методами, но и позволяет изучать нити ДНК, гораздо более длинные, чем возможно вследствие ограничений, характерных для существующих методов. Для изменения параметров электрического поля около пор они применили градиенты концентрации солей, которые позволили ускорить время захвата молекул ДНК полем, таким образом, способствуя более быстрому их прохождению через нанопоры и уменьшению количества ДНК, необходимых для точных измерений.

Виды и марки секвенаторов Современные секвенаторы можно разделить по типу проводимого электрофореза на капиллярные и те, в которых разделение происходит в гелевых пластинах. Последние могут также различаться по количеству детектируемых красителей: один, два или четыре. Капиллярные секвенаторы выпускаются только в варианте, использующем детекцию четырех флуоресцентных красок.

Наиболее успешным секвенатором начала 90 х стал ABI 373 (Applied Biosystems). В этой модели используется одновременная детекция четырех красителей (TAMRA, FAM, ROX, JOE), возбуждаемых излучением аргонового лазера. Лазер генерирует непрерывное излучение в сине зеленой области спектра в диапазоне 488 514 нм. Разделение фрагментов проводится в геле толщиной 0. 4 мм. Возбуждение флуорофора и следующая за этим эмиссия флуоресценции происходят при прохождении меченным фрагментом ДНК зоны сканирования. Для уменьшения эффекта перекрывания спектров эмиссии, получаемый сигнал пропускается через колесо с четырьмя последовательно заменяемыми фильтрами. В результате, первичные данные представляют собой наборы из четырех чисел (в соответствии с сигналом, прошедшим через каждый из фильтров) для каждой точки сканирования (по ширине геля), собранные через равные интервалы времени в течение всего электрофореза.

Среди секвенаторов, детектирующих флуоресценцию одного красителя, можно выделить секвенатор Micro. Gene Blaster фирмы Visible Genetics. Micro. Gene Blaster позволяет одновременно проводить анализ четырех образцов, меченных Cy 5. 5, на 16 ти дорожках. Разделение происходит в одноразовых Micro. Cel кассетах, заполняемых фотополимеризуемым акриламидом. Высота кассет варьирует от 140 до 280 мм, что позволяет секвенировать от 300 (Micro. Cel 300 Cassette) до 700 (Micro. Cel 700 Cassette) нуклеотидов. В качестве источника излучения используется He. Ne лазер с длинной волны 632. 8 нм. Луч лазера направляется не через, а между стеклами, т. е. поперек геля. Это позволило отказаться от любых движущихся частей и, следовательно, значительно повысить надежность прибора и уменьшить его размеры.

Интересные секвенаторы, работающие на двух красителях в инфракрасной области спектра, выпускает фирма LI COR. Ее основная машина IR 2 DNA Sequencer. IR 2 несёт два твердофазных полупроводниковых лазера, один из которых генерирует излучение с длиной волны 680 нм, а другой 780 нм. В качестве красителей используются IRDye 700 и IRDye 800 или их производные. Детекция каждого из флуорофоров осуществляется своим фотодиодом. Причем, возбуждение/детекция красителей происходит не одновременно, а последовательно. Подобная двухканальная система позволяет: во первых, избежать перекрывания спектров эмиссии (они разнесены на 100 нм), и во вторых проводить одновременное двунаправленное секвенирование (сразу с двух праймеров, каждый из которых мечен своим красителем). Разделение фрагментов в IR 2 проводится в гелях с высотой от 18 до 48 см и шириной 25 см, способных вмещать от 32 до 96 дорожек. Толщина гелей может варьировать от 0. 2 мм до 0. 4 мм. За один прогон IR 2 DNA Sequencer способен определять более 1000 нуклеотидов и является по этому показателю рекордсменом.

Конкурентом IR 2 DNA Sequencer стал секвенатор ABI Prism 377, который пришел на смену ABI 373. Основное новшество этой машины заключается в замене оптической фильтрации флуоресценции, которая использовалась в ABI 373 для разделения спектров испускания красителей, на "виртуальную" фильтрацию. В процессе сканирования эмиссия флуоресценции отделяется от отраженного света лазера, рассеивается посредством дифракционной решетки (это позволяет разложить исходный световой пучок на спектральные составляющие) и, наконец, фиксируется CCD камерой. Т. е. в итоге кванты света преобразуются в пиксели во всем промежутке спектра от 514 до 680 нм. Какой именно набор пикселей преобразовать в пик на хроматограмме задается заранее через интерфейс программы Data Collection в зависимости от используемых флуорофоров и способа введения метки. Выпускается несколько модификаций ABI 377. Самый простой вариант обеспечивает автоматический обсчет 18 ти образцов, самый продвинутый 96 ти. Расстояние до точки сканирования (т. е. дистанция разгонки) может варьировать в зависимости от размеров используемых стекол и составлять 12, 36 или 48 см (при толщине геля 0. 2 или 0. 4 мм). Максимально возможный размер определяемых фрагментов 800 900 нуклеотидов.

Схожая технология записи и обработки данных используется в другом секвенаторе фирмы Applied Biosystems ABI Prism 310. Это однокапиллярный секвенатор, способный в автоматическом режиме последовательно просканировать 96 образцов. Полный цикл для каждого из них составляет приблизительно полтора часа и включает: электрокинетическую инжекцию образца в капилляр, электрофорез, возбуждение/детекцию эмиссии флуоресценции и смену полимерного материала в капилляре перед инжекцией следующего образца. За указанное время ABI 310 способен прочитать приблизительно 450 нуклеотидов.

Зачем нужно секвенирование Применений у расшифровки последовательности нуклеотидов и аминокислот очень много. Больше всего геномные технологии используются при работе с организмами, применяемыми в биотехнологиях, такими как бактерии и дрожжи. Во первых, чтобы понимать, какие белки есть у бактерий, и на основании этого прогнозировать их особенности. Например, если у бактерии нет фермента, расщепляющего какой то сахар, то и растить ее на питательной среде из этого сахара бессмысленно. Во вторых, последовательность ДНК очень важна в генной инженерии микроорганизмов. Там нужно точно знать, в каком месте отрезать какой то ген и в какое место его следует переставить.

Важно секвенирование и для человека. Во первых, в таких прикладных аспектах, как криминалистика. Впрочем, для этих целей полномасштабная расшифровка всего генома не нужна. Для этих целей вообще используется митохондриальная ДНК: не геномные молекулы в ядре клетки, а находящиеся в митохондриях. Они короче и быстрее мутируют, так что вариабельность у них выше, а анализировать их намного проще. Более сложно и интересно использование в медицине; существует огромное количество в разной мере наследственных заболеваний. Гены, которые отвечают за эти болезни, полезно знать, чтобы проводить диагностику предрасположенности. Есть болезни мультигенные, например сахарный диабет или рассеянный склероз. В таких случаях необходимо сочетание определенных вариантов нескольких генов, чтобы болезнь развилась. Например, сахарный диабет чаще возникает у людей со второй группой крови. И так как нет какого то единственного гена, ответственного за подобные заболевания, желательно анализировать уже полный геном.

Спасибо за внимание!
ения геномов. Секвенирование.ppt