моб эл-ты генома.ppt
- Количество слайдов: 99

Курс лекций «МОБИЛЬНЫЕ ЭЛЕМЕНТЫ ГЕНОМА» А. П. Галкин Основные определения: Мобильные элементы – автономно реплицирующиеся последовательности ДНК, которые перемещаются по геному в результате ферментативных реакций, но, в отличии от вирусов, не передаются между клетками или организмами. Транспозиция – ферментативный процесс перемещения мобильных элементов из одного сайта генома в другой.

«There are genetic elements capable of transposing to different chromosomal locations in maize plants» Mc. Clintock B. Am J Bot. 1945; 32: 671– 678; Mc. Clintock B. Year Book Carnegie Inst Washington. 1948; 47: 155– 169 Отзыв журнала PNAS на статью Mc. Clintock 1950 г. : “… the presented thesis, and evidence for it, could not be accepted by the majority of geneticists or by other biologists” Цитата из обзора Fedoroff N. Proc Natl Acad Sci U S A. 2000 Jun 20; 97(13): 7002 -7. Transposons and genome evolution in plants.
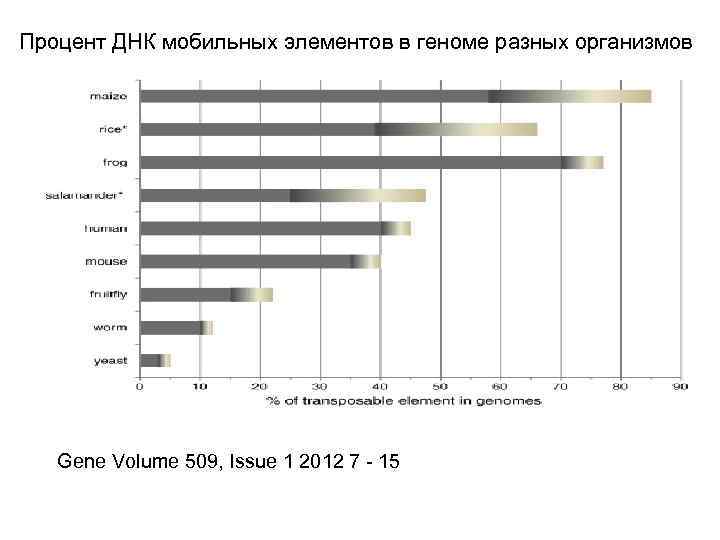
Процент ДНК мобильных элементов в геноме разных организмов Gene Volume 509, Issue 1 2012 7 - 15
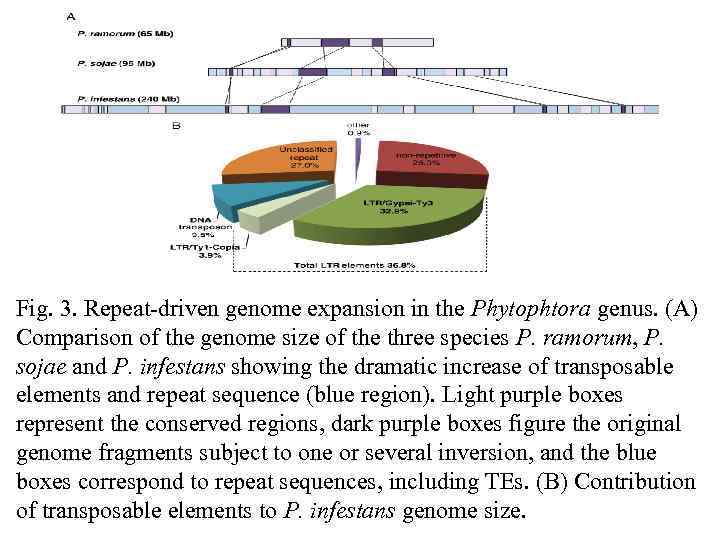
Fig. 3. Repeat-driven genome expansion in the Phytophtora genus. (A) Comparison of the genome size of the three species P. ramorum, P. sojae and P. infestans showing the dramatic increase of transposable elements and repeat sequence (blue region). Light purple boxes represent the conserved regions, dark purple boxes figure the original genome fragments subject to one or several inversion, and the blue boxes correspond to repeat sequences, including TEs. (B) Contribution of transposable elements to P. infestans genome size.
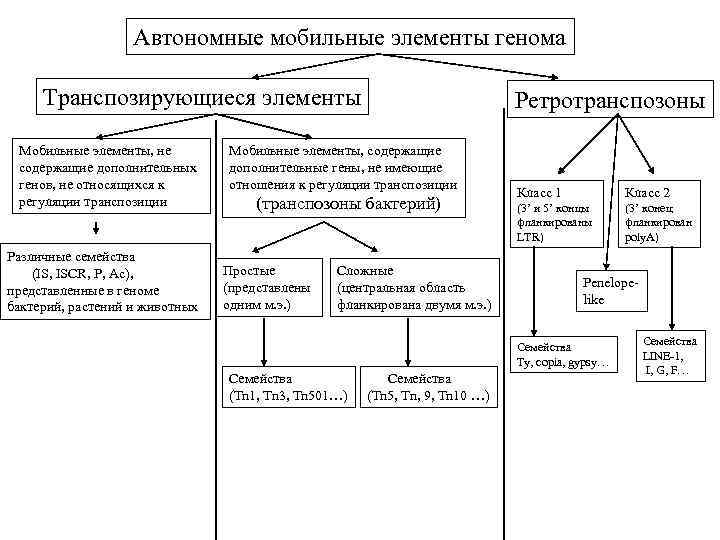
Автономные мобильные элементы генома Транспозирующиеся элементы Мобильные элементы, не содержащие дополнительных генов, не относящихся к регуляции транспозиции Различные семейства (IS, ISCR, P, Ac), представленные в геноме бактерий, растений и животных Ретротранспозоны Мобильные элементы, содержащие дополнительные гены, не имеющие отношения к регуляции транспозиции (транспозоны бактерий) Простые (представлены одним м. э. ) Сложные (центральная область фланкирована двумя м. э. ) Класс 1 Класс 2 (3’ и 5’ концы фланкированы LTR) (3’ конец фланкирован poly. A) Penelopelike Семейства Ty, copia, gypsy… Семейства (Tn 1, Tn 3, Tn 501…) Семейства (Tn 5, Tn, 9, Tn 10 …) Семейства LINE-1, I, G, F…

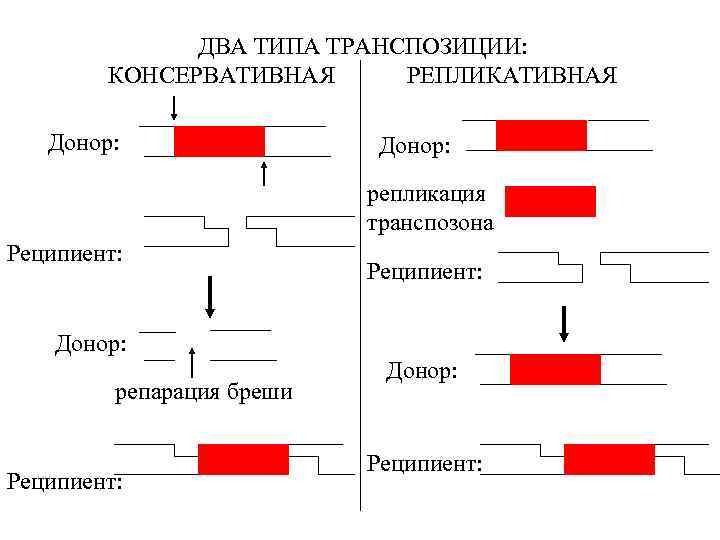
ДВА ТИПА ТРАНСПОЗИЦИИ: КОНСЕРВАТИВНАЯ РЕПЛИКАТИВНАЯ Донор: репликация транспозона Реципиент: Донор: репарация бреши Реципиент: Донор: Реципиент:

МОБИЛЬНЫЕ ЭЛЕМЕНТЫ БАКТЕРИЙ IS-элементы и IS-подобные элементы в составе сложных транспозонов IS

ISCR-элементы (Insertion Sequence Common Regions) ISCR elements are related to IS 91 family elements. ISCR elements display characteristic features of the IS 91 family group of elements that include IS 91, IS 1294 and IS 801. These features are a large transposase ORF indicated in yellow that shares sequence motifs, terminator (ter. IS) and origin of replication (ori. IS). This is especially highlighted by the high sequence identity at the ori. IS as depicted in the blue box where nucleotides present in the majority of sequences are highlighted. The large arrow indicates the direction of replicative transposition and cotransposed genes are found adjacent to the ter. IS end of the element. IS 91 is often associated with virulence factors such as elt. A/B toxin in pathogenic enterohemolytic and enterotoxigenic strains of Escherichia coli and ISCR elements are often found adjacent to antibiotic resistance genes such as bla. OXA-45. (FEMS Microbiology, V 35, Issue 5, pages 912– 935.
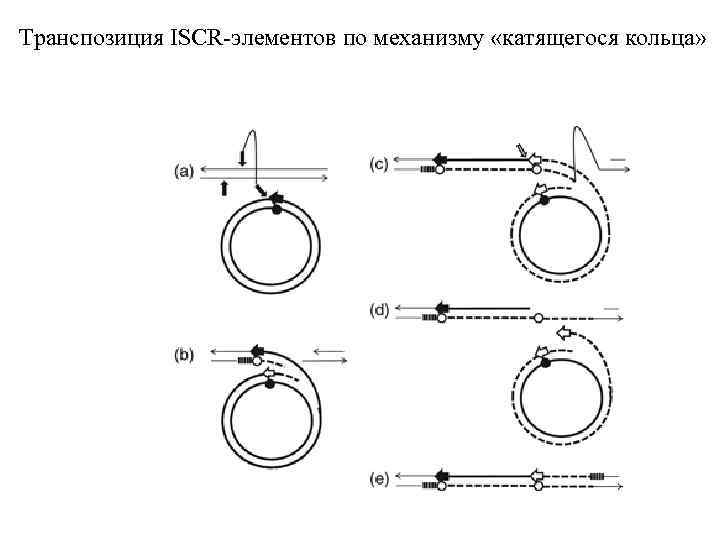
Транспозиция ISCR-элементов по механизму «катящегося кольца»
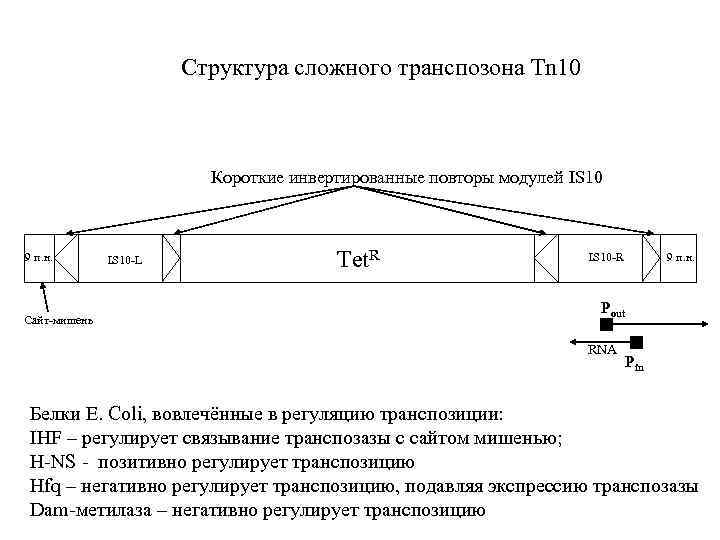
Структура сложного транспозона Tn 10 Короткие инвертированные повторы модулей IS 10 9 п. н. Сайт-мишень IS 10 -L Tet. R IS 10 -R 9 п. н. Pout RNA Pin Белки E. Coli, вовлечённые в регуляцию транспозиции: IHF – регулирует связывание транспозазы с сайтом мишенью; H-NS - позитивно регулирует транспозицию Hfq – негативно регулирует транспозицию, подавляя экспрессию транспозазы Dam-метилаза – негативно регулирует транспозицию
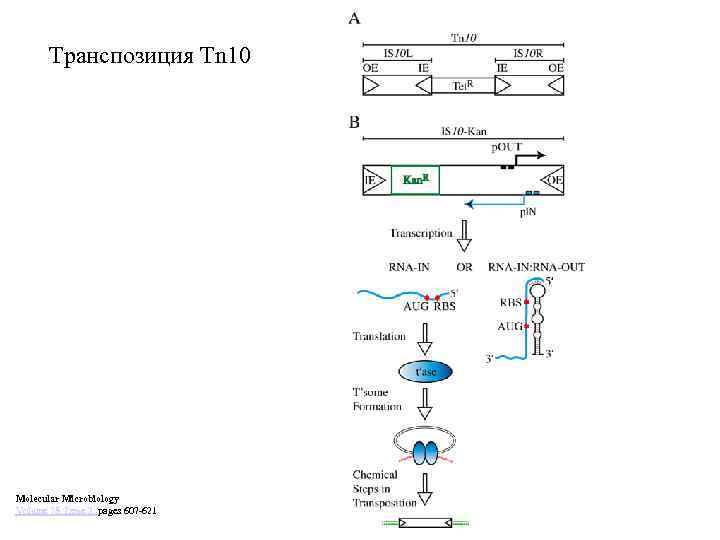
Транспозиция Tn 10 Molecular Microbiology Volume 78, Issue 3, pages 607 -621

Structure of Tn 10/IS 10 including a ‘marked’ IS 10 and summary of the major steps in Tn 10/IS 10 transposition. OE and IE are outside and inside ends, respectively, and include binding sites for the IS 10 encoded transposase protein. For IS 10 -Kan promoters p. IN and p. OUT (blue and black squares respectively) and the corresponding RNA transcripts, RNA-IN and RNA-OUT (blue and black lines with arrows), are shown. RNA-OUT is an anti-sense RNA to RNA-IN (transposase transcript) and pairing of these RNAs blocks ribosome binding to RNA-IN by sequestering the ribosome binding site (RBS). Translation of RNA-IN generates the transposase protein, which binds transposon ends to form a transpososome. Subsequent to transpososome assembly the chemical steps in transposition are initiated.
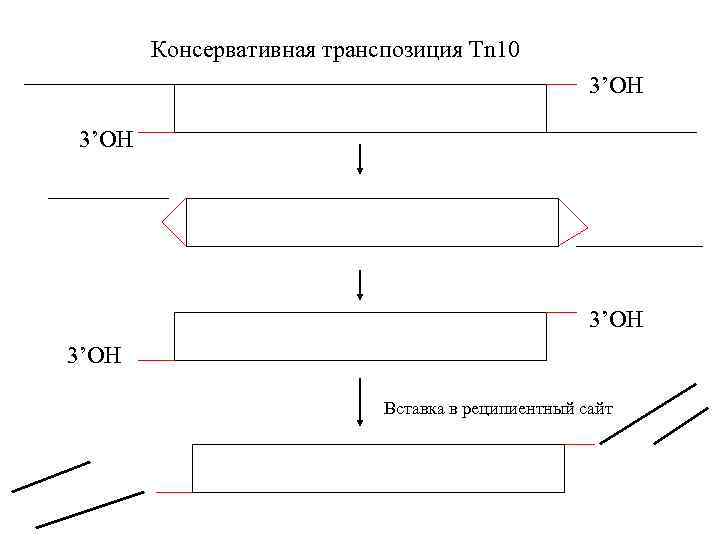
Консервативная транспозиция Tn 10 3’OH Вставка в реципиентный сайт
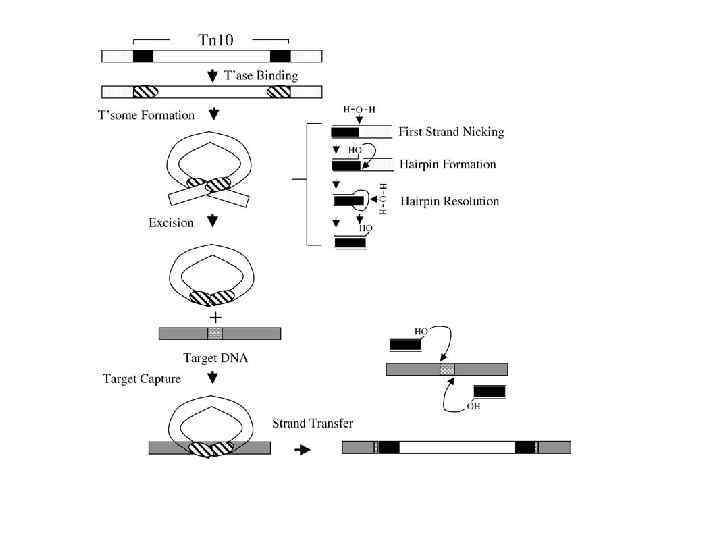

Mechanism of Tn 10 transposition. Tn 10 is shown imbedded within donor DNA. Transposase (striped oval) is shown bound to OE sequences (black rectangles). After mediating transpososome formation, transposase catalyzes the chemical steps in excision. Once the flanking donor DNA is removed from both transposon ends the transpososome binds a target DNA (gray rectangle) and catalyzes the chemical steps in the strand transfer reaction. Host repair functions then fill in the 9 bp gaps created by the strand transfer reaction, leading to the duplication of the original short target site (checkered rectangle).
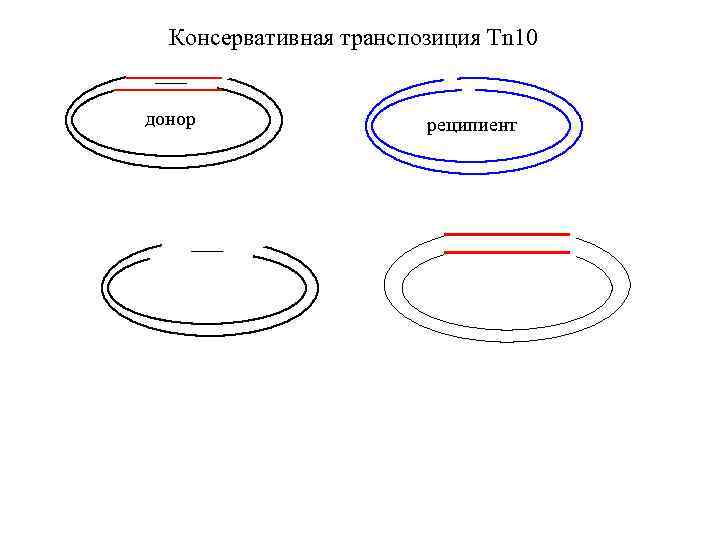
Консервативная транспозиция Tn 10 донор реципиент
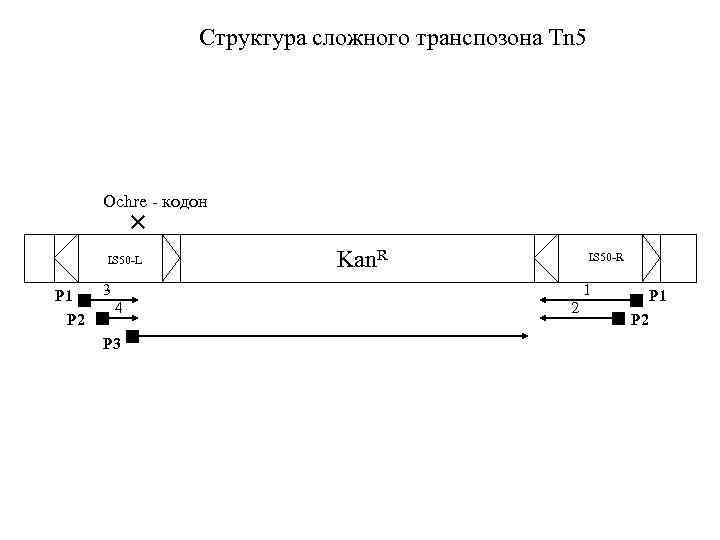
Структура сложного транспозона Tn 5 Ochre - кодон IS 50 -L P 1 P 2 Kan. R IS 50 -R 3 1 4 P 3 2 P 1 P 2
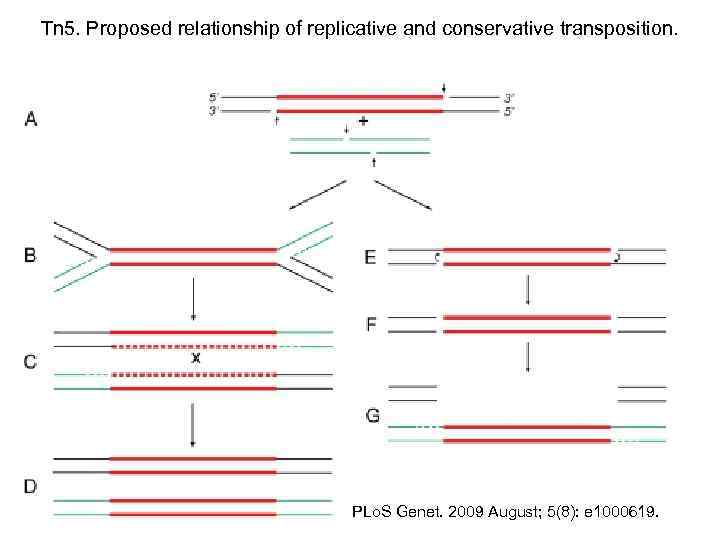
Tn 5. Proposed relationship of replicative and conservative transposition. PLo. S Genet. 2009 August; 5(8): e 1000619.

Both processes start by nicking (short vertical arrows) of the transposon ends to expose the 3′-OH termini (A). At some point (see below), the target DNA is also cleaved to provide short protruding 5′-PO 4 ends. In replicative transposition (left), strand-transfer takes place by joining the 3′ ends to 5′ ends of the target DNA in a concerted cleavage and joining reaction to form the “Shapiro intermediate” (B). As a result of replication of the intermediate, the donor and recipient replicons become fused to form a cointegrate (C) carrying one directly repeated copy of the transposon at each junction. Consequently, the cointegrate is an unstable structure that is resolved by rec. A-dependent generalized recombination (as in Tn 5; A. Ahmed, unpublished results) or tnp. R-specified site-specific recombination (as in Tn 3 [22]). The donor and recipient replicons are thereby separated, each harboring one copy of the transposon (D). If the target DNA is located within the donor replicon itself (intramolecular transposition), maturation of the Shapiro intermediate produces a replicative inversion (as shown in Figure 2) or an adjacent deletion (Figure 3). This process is highly efficient in transposons like Mu and Tn 3 [2], [9]. In conservative transposition (right), the 3′ ends engage in hairpin formation at both ends of the transposon (E) [7]. Following hairpin resolution (F), the free 3′ ends of the excised transposon are joined to 5′ ends from the target DNA (G), and the gaps are filled to complete the insertion process. The fate of the donor DNA containing a large gap (G) is not known: it could be degraded or undergo double-strand gap repair to regenerate the transposon sequence. This process is highly efficient in transposons like Tn 10 [4], [8]. In Tn 5, hairpin formation is not efficient (i. e. , is leaky), so that a small proportion of the initial 3′ nicks remains free to engage in strand-transfer. Hence, the transposon displays properties of both conservative and replicative transposition concomitantly [5], [6]. These reactions are carried out by the respective transposases, which, by oligomerization, bring the end sequences of the transposon together to form a synaptic complex. For clarity, however, the transposon is shown as a straight line. The donor DNA sequence is shown in black, transposon DNA sequence is in red, and the recipient DNA sequence is in green. Replication and gap repair are indicated by dashed lines. The crossover event that resolves the cointegrate (C) is indicated by “x. ”
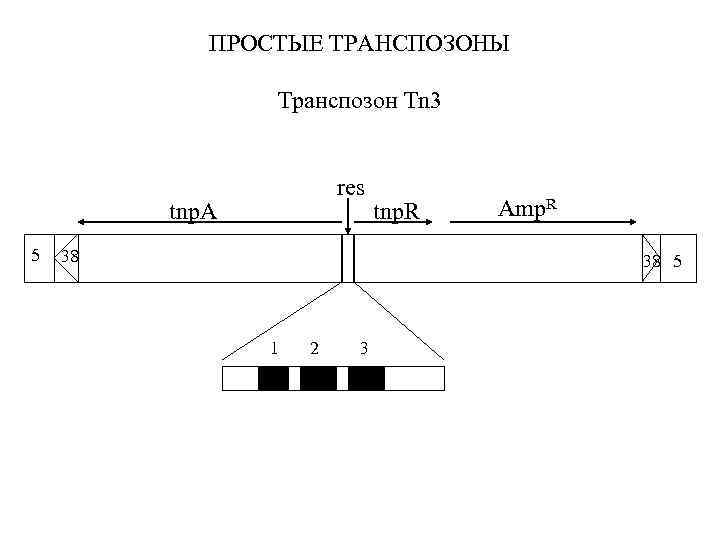
ПРОСТЫЕ ТРАНСПОЗОНЫ Транспозон Tn 3 tnp. A 5 res 38 tnp. R Amp. R 38 5 1 2 3
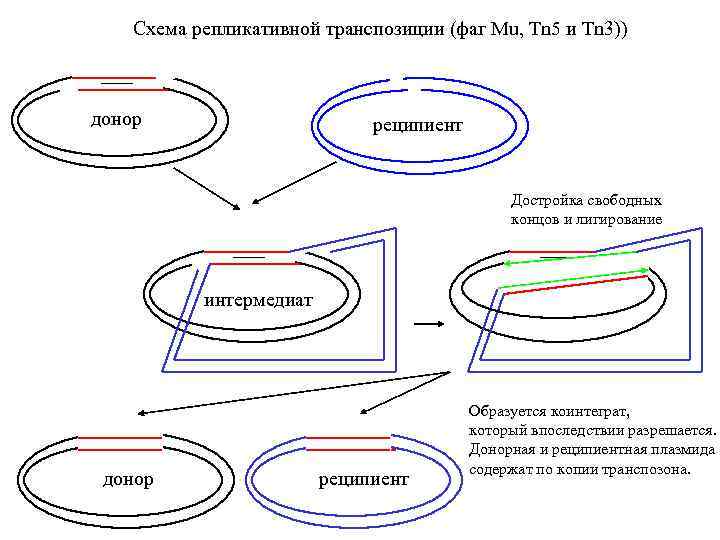
Схема репликативной транспозиции (фаг Mu, Tn 5 и Tn 3)) донор реципиент Достройка свободных концов и лигирование интермедиат донор реципиент Образуется коинтеграт, который впоследствии разрешается. Донорная и реципиентная плазмида содержат по копии транспозона.
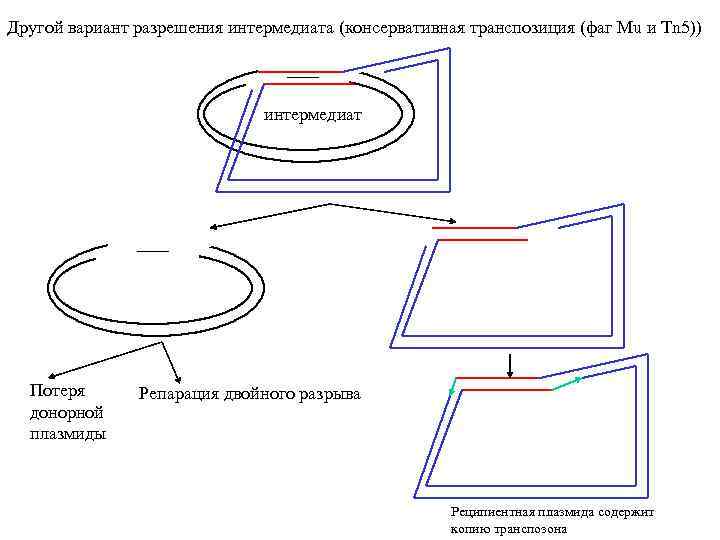
Другой вариант разрешения интермедиата (консервативная транспозиция (фаг Mu и Tn 5)) интермедиат Потеря донорной плазмиды Репарация двойного разрыва Реципиентная плазмида содержит копию транспозона

Механизм мультикопийного ингибирования инсерции бактериальных транспозонов и бактериофагов CRISPR – clustered regularly interspaced short palindromic repeats
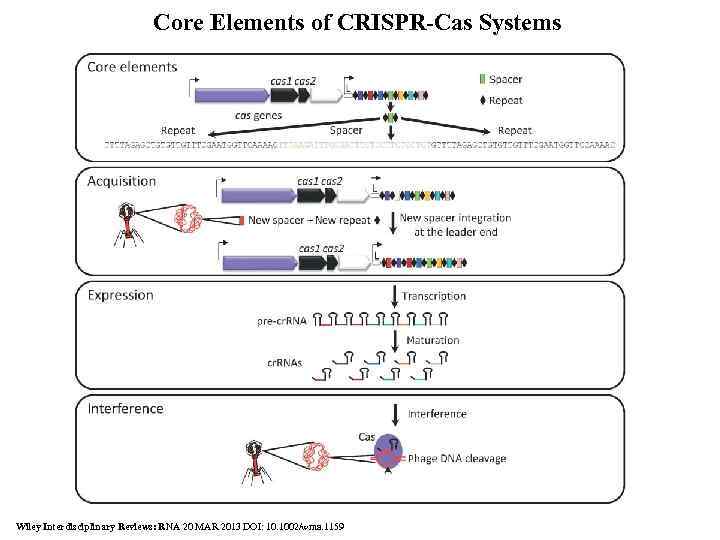
Core Elements of CRISPR-Cas Systems Wiley Interdisciplinary Reviews: RNA 20 MAR 2013 DOI: 10. 1002/wrna. 1159

CRISPR-Cas elements and mechanism of action. The various elements that constitute CRISPR-Cas systems are graphically depicted, including cas genes (with the universal cas 1 and cas 2), the leader (L), spacers (boxes), repeats (diamonds). Adaptive immunity is build through acquisition of new spacers at the leader end, which are derived from invasive DNA (here, phage DNA). In the expression stage, the repeatspacer array is then transcribed as a full-length pre-cr. RNA, which is processed by cleavage into mature cr. RNAs. The mature RNAs then form a ribonucleoprotein complex with Cas proteins to mediate interference by guiding endonucleases toward homologous nucleic acid sequences.
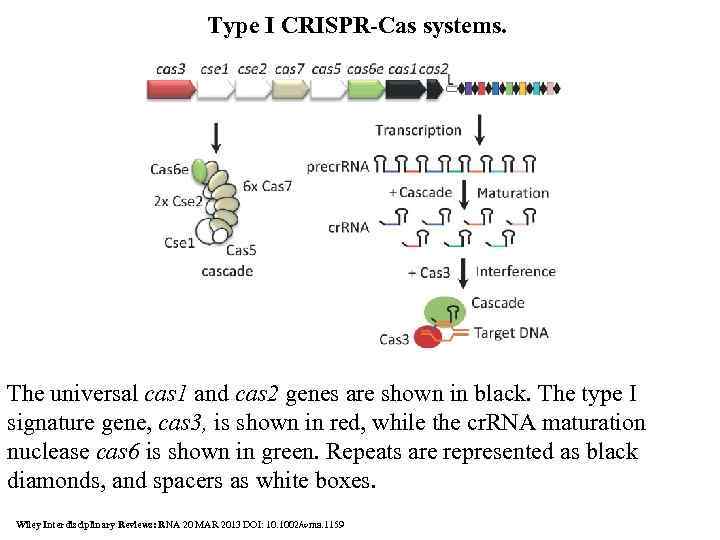
Type I CRISPR-Cas systems. The universal cas 1 and cas 2 genes are shown in black. The type I signature gene, cas 3, is shown in red, while the cr. RNA maturation nuclease cas 6 is shown in green. Repeats are represented as black diamonds, and spacers as white boxes. Wiley Interdisciplinary Reviews: RNA 20 MAR 2013 DOI: 10. 1002/wrna. 1159

The signature gene for type I CRISPR-Cas systems is cas 3, which encodes a nuclease involved in the cleavage of the target DNA. 22 A signature feature of type I systems (Figure 2) is the CRISPR-associated complex for antiviral defense (Cascade) which is responsible for the processing of cr. RNA and involved in recognition of target DNA. 9 Another key protein in the mechanism of action of type I systems is Cas 6, an endoribonuclease which cleaves the pre-cr. RNA within the CRISPR repeat sequence during the cr. RNA maturation process. Studies of model type I systems have documented the involvement of Cascade in CRISPR-mediated interference, whereby the Cascade: cr. RNA ribonucleoprotein complex targets homologous invasive DNA and triggers R-loop formation for subsequent cleavage by the signature protein Cas 3. 36– 44 An outline of CRISPR-mediated immunity in type I systems is provided in Figure 2. The molecular processes responsible for cr. RNA biogenesis and maturation are highly precise processing events. The CRISPR repeat-spacer array is first transcribed as a long transcript defined as the precursor CRISPR RNA (pre-cr. RNA). The pre-cr. RNA subsequently undergoes a single maturation step, where the Cas 6 endonuclease cleaves the transcript within the CRISPR repeat sequence, typically as part of the Cascade complex. Generally, cleavage occurs at the base of the stem-loop structure formed by the palindromic CRISPR repeat sequence, generating a cr. RNA consisting of a full spacer flanked by a short 5′-handle (∼ 8 nt) and a 3′-stem loop derived from the CRISPR repeat (∼ 17– 21 nt). The beauty of the system is illustrated by the seahorse-like structure of the Cascade complex, which includes a Cas 7 hexameric backbone, a Cse 1 tail and a Cas 6 e head.
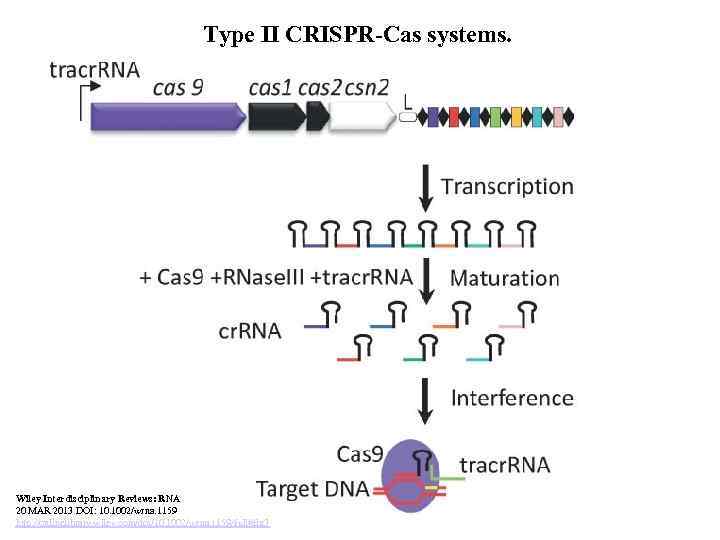
Type II CRISPR-Cas systems. Wiley Interdisciplinary Reviews: RNA 20 MAR 2013 DOI: 10. 1002/wrna. 1159 http: //onlinelibrary. wiley. com/doi/10. 1002/wrna. 1159/full#fig 3

Figure 3. Type II CRISPR-Cas systems. Details are provided for the prototypical Streptococcus thermophilus DGCC 7710 type II-A system. The universal cas 1 and cas 2 genes are shown in black. The type II signature gene, cas 9, is shown in purple. Repeats are represented as black diamonds, and spacers as white boxes. For type II systems, the signature gene is the idiosyncratic cas 9, 22 which encodes a large protein involved in both cr. RNA biogenesis and target DNA cleavage 54– 59 (Figure 3). Another peculiar molecular element unique to type II systems is the tracr. RNA, 49 which includes a sequence (∼ 25 nt) complementary to the CRISPR repeat, which specifically trans-activates pre-cr. RNA cleavage within the CRISPR repeat, and sets the stage for cr. RNA maturation and processing by the housekeeping endoribonuclease RNAse III, which acts as a dicing effector. 55 The three component system tracr. RNA-Cas 9 -RNAse III is responsible for the maturation of an approximately 40 nt cr. RNA which then forms a ribonucleoprotein complex to mediate interference through ds. DNA cleavage. 56 Tracr. RNA base pairs with each CRISPR repeat sequence within the pre-cr. RNA to form a double-stranded RNA template which is specifically cleaved by RNAse III in the presence of Cas 9. 55 Target ds. DNA cleavage is mediated by HNH and Ruv. C domains in Cas 9 (Figures 3 and 5), which cleave the complementary and noncomplementary strands, respectively, exactly 3 nt away from the 3′-end of the proto-spacer, which is 2 bp away from the PAM. 54, 57, 58 Interestingly, two type II-A systems in S. thermophilus cleave target phage and plasmid DNA at the same position, though they recognize different PAMS, namely NNAGAAW for CRISPR 1 and NGGNG for CRISPR 3. 54, 58, 59
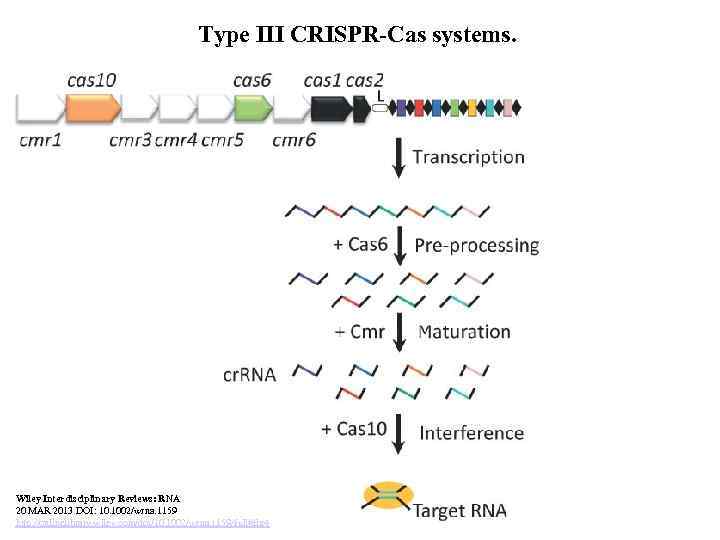
Type III CRISPR-Cas systems. Wiley Interdisciplinary Reviews: RNA 20 MAR 2013 DOI: 10. 1002/wrna. 1159 http: //onlinelibrary. wiley. com/doi/10. 1002/wrna. 1159/full#fig 4

Figure 4. Type III CRISPR-Cas systems. Details are provided for the prototypical Pyrococcus furiosus type III-B system. The universal cas 1 and cas 2 genes are shown in black. The type III signature gene, cas 10, is shown in orange, while the cr. RNA maturation nuclease cas 6 is shown in green. Repeats are represented as black diamonds, and spacers as white boxes. The signature gene for type III CRISPR-Cas systems is cas 10, 22 which encodes a nuclease implicated in target nucleic acid interference. In type III systems (Figure 4), once transcribed, the pre-cr. RNA subsequently undergoes two maturation steps: first, the Cas 6 endonuclease cleaves within the CRISPR repeat; second, protein complexes (analogous to the type I Cascade) further trim the cleaved pre-cr. RNA to generate a shorter mature cr. RNA. 16, 34, 60– 63 For type III systems, cr. RNA processing and maturation operate in a ruler mechanism manner, whereby Cas 6 cleaves within the 5′-end of the transcribed CRISPR repeat to generate a 8 -nt 5′-handle. 16, 60, 62 The secondary processing, which occurs through a ruler mechanism anchored at the primary cr. RNA processing site, is mediated by additional Cas genes. 62 The approximately 67 nt Cas 6 -generated cr. RNA is further trimmed at the 3′ to generate a 39– 45 nt mature cr. RNA which drives subsequent interference. In type III-B systems, the Cas RAMP module (CMR) effector complex specifically targets RNAs. 34, 61 Csm and Cmr Cas proteins are implicated in cr. RNA maturation in type III-A and type III-B CRISPR-Cas systems, respectively. Similar to type I systems, cr. RNA guides Cascade-like complexes toward the cognate nucleic acid for cleavage. Likewise, similar to type I systems, the endoribonuclease Cas 6 is involved in cleavage of pre-cr. RNA within the CRISPR repeat sequence.
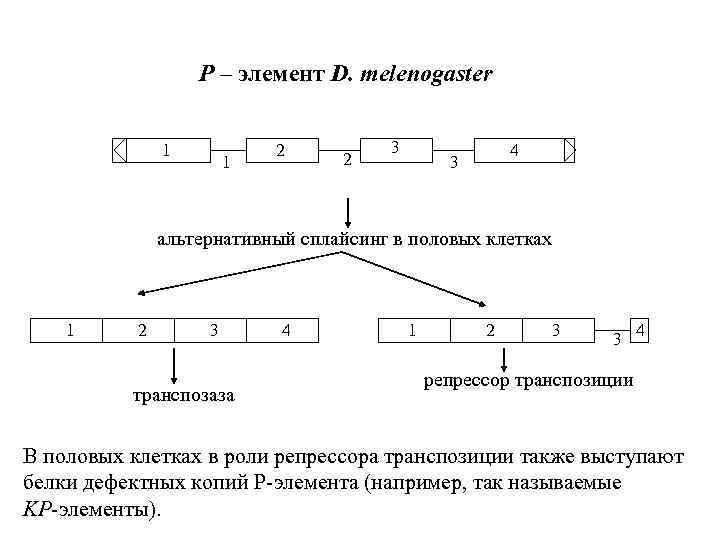
P – элемент D. melenogaster 1 1 2 2 3 4 3 альтернативный сплайсинг в половых клетках 1 2 3 транспозаза 4 1 2 3 3 4 репрессор транспозиции В половых клетках в роли репрессора транспозиции также выступают белки дефектных копий P-элемента (например, так называемые KP-элементы).

Факторы, регулирующие индукцию и репрессию транспозиции P-элементов в половых клетках -Наличие полноразмерных Р-элементов, кодирующих транспозазу и репрессор; -Наличие делетированных копий, в частности КР, влияет на репрессию транспозиции по мужскому и женскому типу; - Локализация полноразмерных копий в геноме - Регуляция цитотипа за счёт комплекса pi. RNA/Piwi
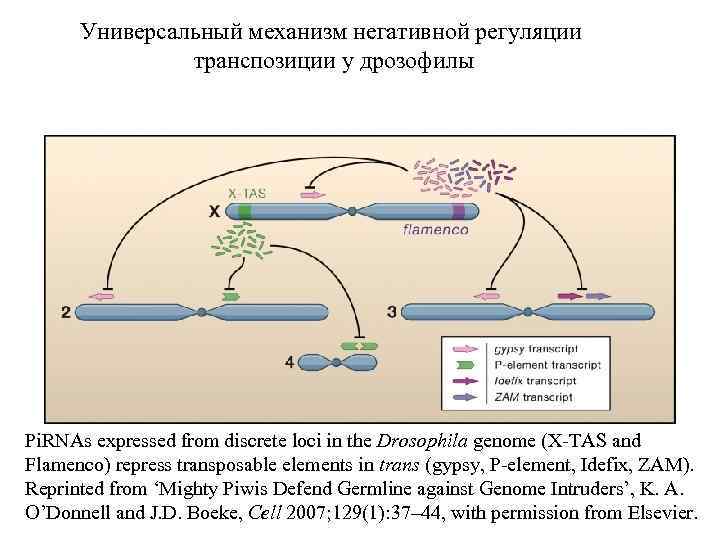
Универсальный механизм негативной регуляции транспозиции у дрозофилы Pi. RNAs expressed from discrete loci in the Drosophila genome (X-TAS and Flamenco) repress transposable elements in trans (gypsy, P-element, Idefix, ZAM). Reprinted from ‘Mighty Piwis Defend Germline against Genome Intruders’, K. A. O’Donnell and J. D. Boeke, Cell 2007; 129(1): 37– 44, with permission from Elsevier.
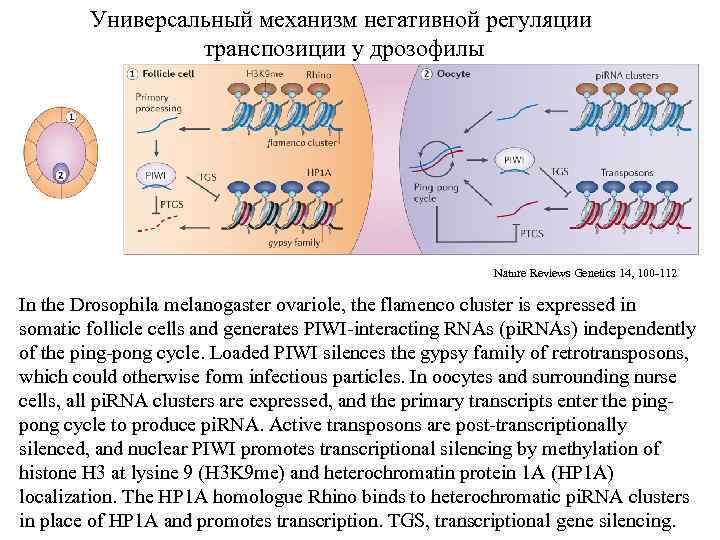
Универсальный механизм негативной регуляции транспозиции у дрозофилы Nature Reviews Genetics 14, 100 -112 In the Drosophila melanogaster ovariole, the flamenco cluster is expressed in somatic follicle cells and generates PIWI-interacting RNAs (pi. RNAs) independently of the ping-pong cycle. Loaded PIWI silences the gypsy family of retrotransposons, which could otherwise form infectious particles. In oocytes and surrounding nurse cells, all pi. RNA clusters are expressed, and the primary transcripts enter the pingpong cycle to produce pi. RNA. Active transposons are post-transcriptionally silenced, and nuclear PIWI promotes transcriptional silencing by methylation of histone H 3 at lysine 9 (H 3 K 9 me) and heterochromatin protein 1 A (HP 1 A) localization. The HP 1 A homologue Rhino binds to heterochromatic pi. RNA clusters in place of HP 1 A and promotes transcription. TGS, transcriptional gene silencing.

In the Drosophila melanogaster ovariole, the flamenco cluster is expressed in somatic follicle cells and generates PIWI-interacting RNAs (pi. RNAs) independently of the ping-pong cycle. Loaded PIWI silences the gypsy family of retrotransposons, which could otherwise form infectious particles. In oocytes and surrounding nurse cells, all pi. RNA clusters are expressed, and the primary transcripts enter the pingpong cycle to produce pi. RNA. Active transposons are post-transcriptionally silenced, and nuclear PIWI promotes transcriptional silencing by methylation of histone H 3 at lysine 9 (H 3 K 9 me) and heterochromatin protein 1 A (HP 1 A) localization. The HP 1 A homologue Rhino binds to heterochromatic pi. RNA clusters in place of HP 1 A and promotes transcription. TGS, transcriptional gene silencing.

Универсальный механизм негативной регуляции транспозиции у дрозофилы
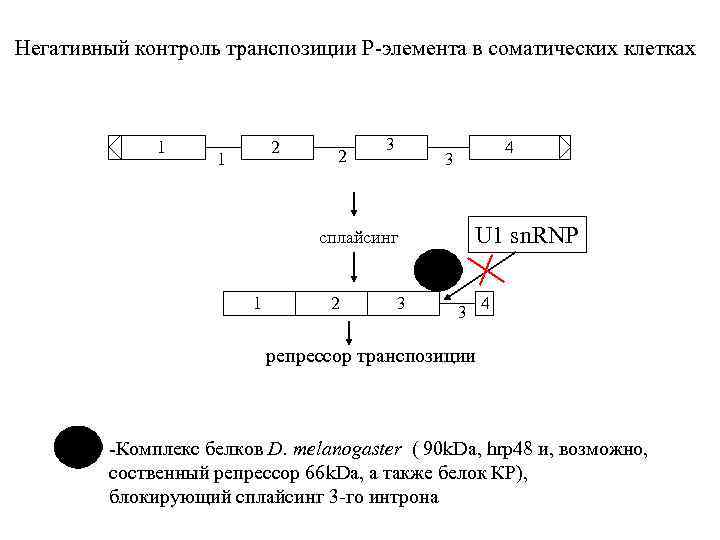
Негативный контроль транспозиции P-элемента в соматических клетках 1 2 3 сплайсинг 1 2 4 3 3 U 1 sn. RNP 3 4 репрессор транспозиции -Комплекс белков D. melanogaster ( 90 k. Da, hrp 48 и, возможно, соственный репрессор 66 k. Da, а также белок КР), блокирующий сплайсинг 3 -го интрона

Модель репарации двунитевого разрыва, опосредованного транспозицией P-элемента (SDSA) Произошло вырезание копии P-элемента в одной из сестринских хроматид.
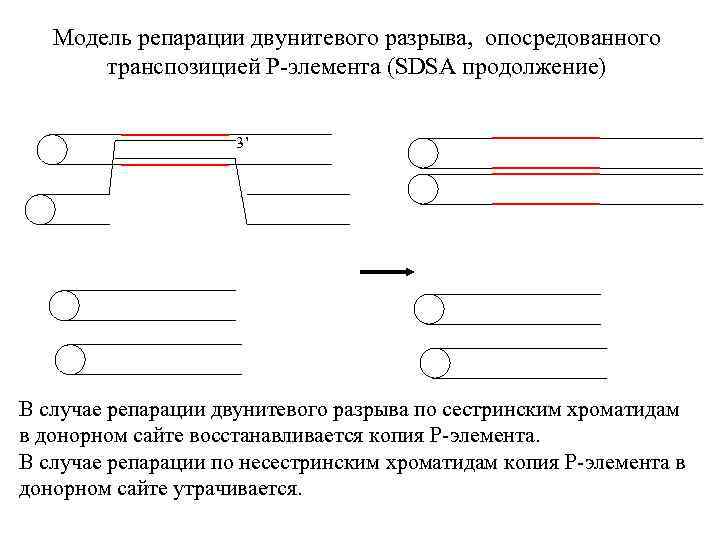
Модель репарации двунитевого разрыва, опосредованного транспозицией P-элемента (SDSA продолжение) 3’ В случае репарации двунитевого разрыва по сестринским хроматидам в донорном сайте восстанавливается копия P-элемента. В случае репарации по несестринским хроматидам копия P-элемента в донорном сайте утрачивается.
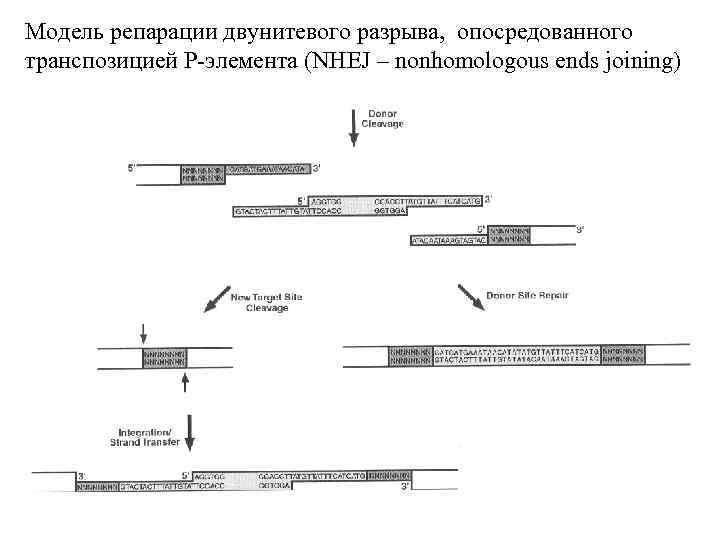
Модель репарации двунитевого разрыва, опосредованного транспозицией P-элемента (NHEJ – nonhomologous ends joining)

Синдром Р-М гибридного дисгенеза При скрещивании самцов, в геноме которых представлены P-элементы с самками у которых Р-элементы отсутствуют у гибридного потомства отмечается комплекс нарушений, который включает: высокие частоты мутаций, хромосомных абераций и рекомбинации у самок и самцов, а также нарушение расхождения по полу от 1: 1. Понятие Р и М цитотипа.
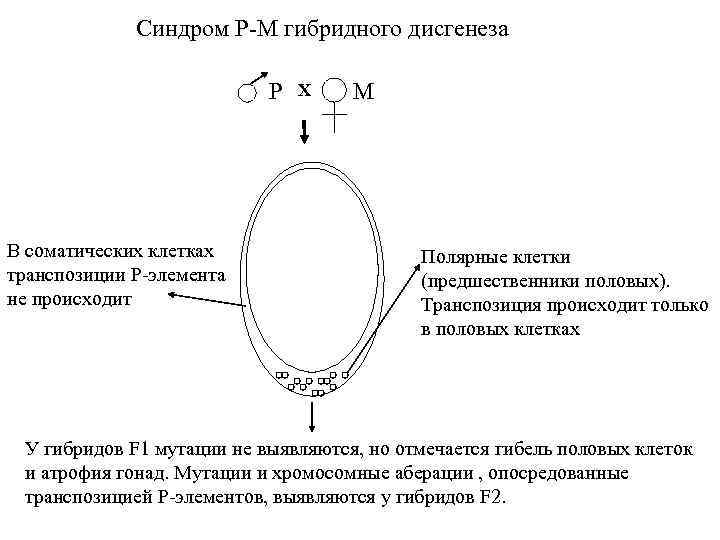
Синдром Р-М гибридного дисгенеза P В соматических клетках транспозиции P-элемента не происходит X M Полярные клетки (предшественники половых). Транспозиция происходит только в половых клетках У гибридов F 1 мутации не выявляются, но отмечается гибель половых клеток и атрофия гонад. Мутации и хромосомные аберации , опосредованные транспозицией P-элементов, выявляются у гибридов F 2.
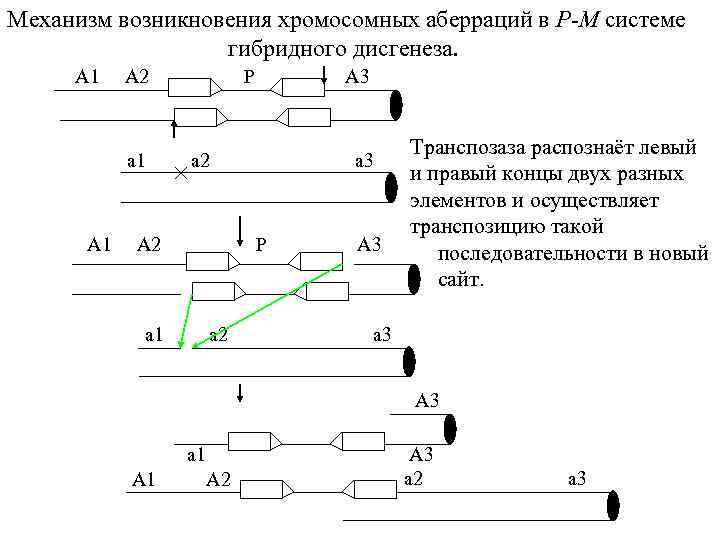
Механизм возникновения хромосомных аберраций в P-M системе гибридного дисгенеза. A 1 A 2 P A 3 Транспозаза распознаёт левый и правый концы двух разных элементов и осуществляет транспозицию такой A 1 A 2 P A 3 последовательности в новый сайт. a 1 a 2 a 3 A 3 a 1 A 1 A 2 A 3 a 2 a 3
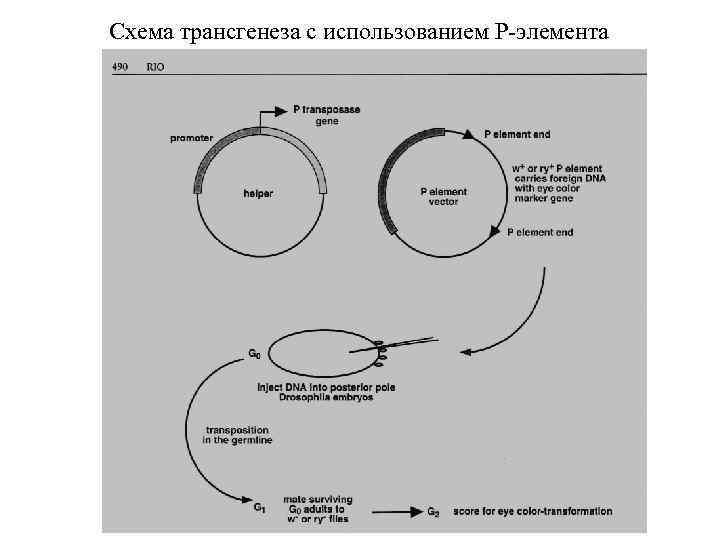
Схема трансгенеза с использованием Р-элемента
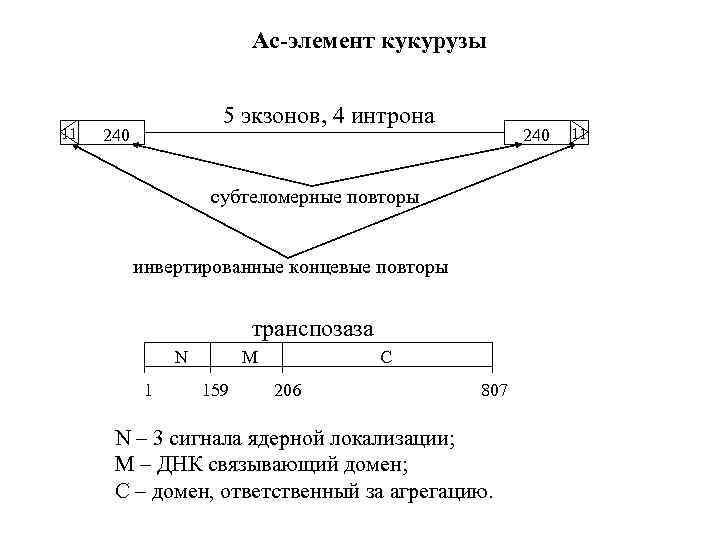
Ac-элемент кукурузы 11 240 5 экзонов, 4 интрона субтеломерные повторы инвертированные концевые повторы транспозаза N M C 1 159 206 807 N – 3 сигнала ядерной локализации; M – ДНК связывающий домен; С – домен, ответственный за агрегацию. 240 11
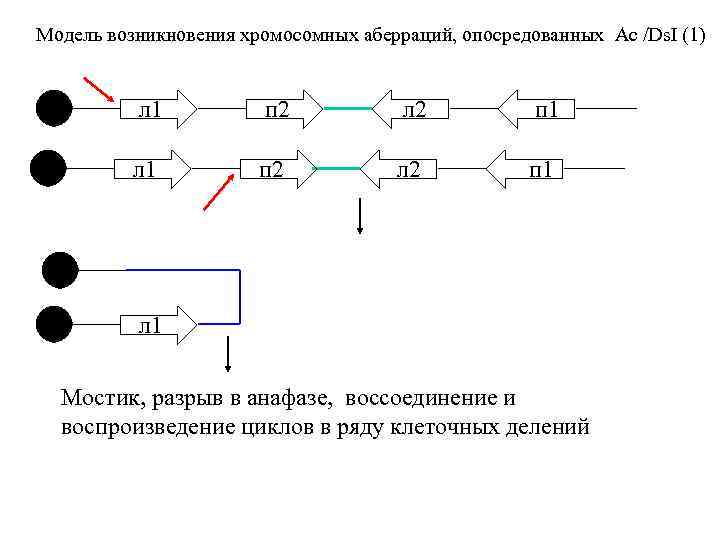
Модель возникновения хромосомных аберраций, опосредованных Ac /Ds. I (1) л 1 п 2 л 2 п 1 л 1 Мостик, разрыв в анафазе, воссоединение и воспроизведение циклов в ряду клеточных делений
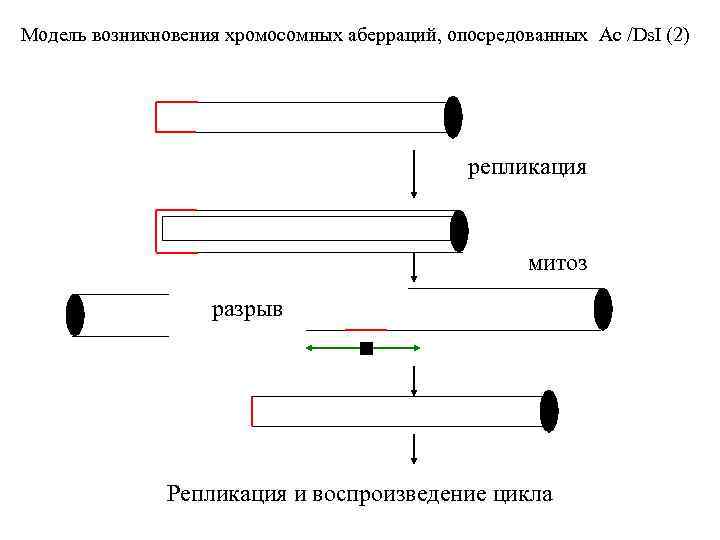
Модель возникновения хромосомных аберраций, опосредованных Ac /Ds. I (2) репликация митоз разрыв Репликация и воспроизведение цикла
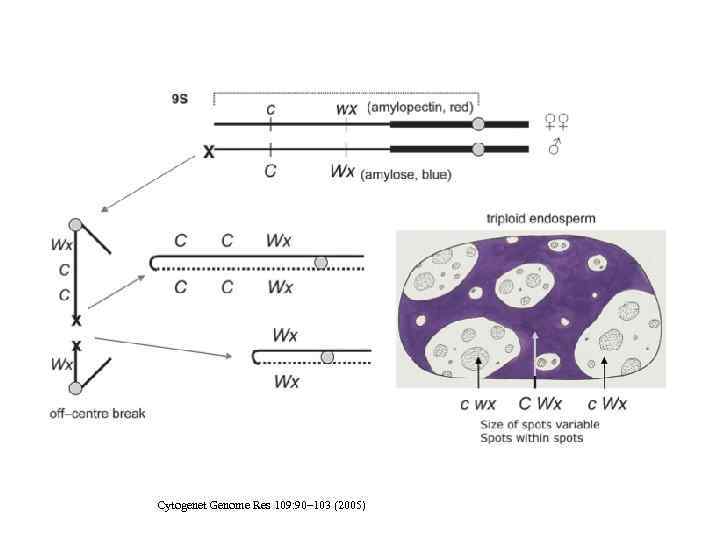
Cytogenet Genome Res 109: 90– 103 (2005)
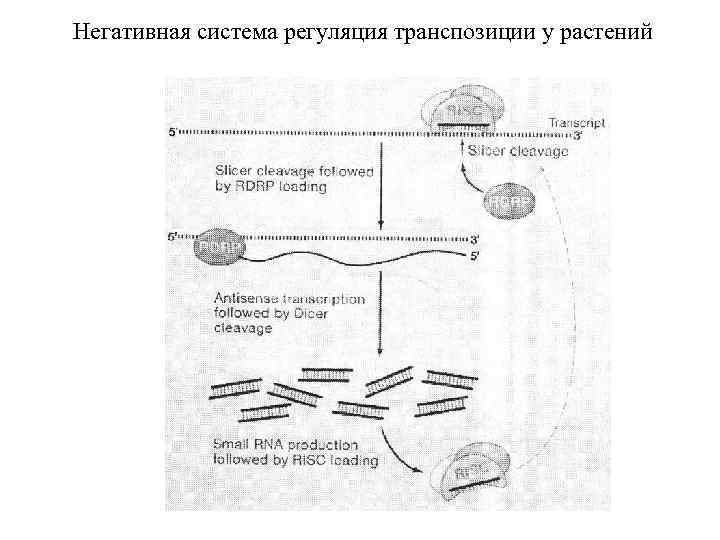
Негативная система регуляция транспозиции у растений


A model of double lock regulation in transposon activity by si. RNAs in Arabidopsis. Transposon-related RNAs transcribed by Pol IV subsequently produce si. RNAs with RDR and DCL proteins. An RNAi pathway degrades the transcription of transposons as indicated by the production of 21 nt si. RNA. Binding of 24 nt si. RNA to the AGO 4 -containing RNA induced silencing complex (RISC) targets RNA-directed DNA methylation (Rd. DM) to the chromatin. Rd. DM introduces de novo cytosine methylation and suppresses transcription of transposons. Previous observations indicated that si. RNA function to regulate transposition of mobile elements in Arabidopsis (Mirouze et al. 2009; Ito et al. 2011), yet it remains unclear if the si. RNAs target extrachromosomal DNA for degradation or inhibit integration into the host genome, or some combination therein.
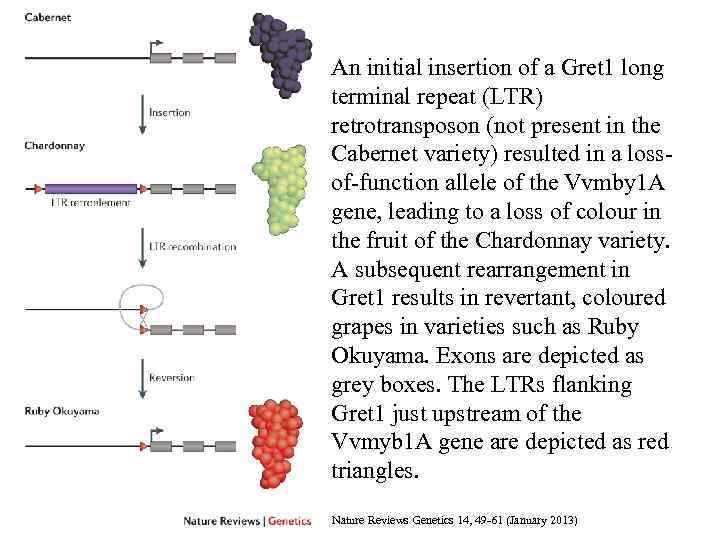
An initial insertion of a Gret 1 long terminal repeat (LTR) retrotransposon (not present in the Cabernet variety) resulted in a lossof-function allele of the Vvmby 1 A gene, leading to a loss of colour in the fruit of the Chardonnay variety. A subsequent rearrangement in Gret 1 results in revertant, coloured grapes in varieties such as Ruby Okuyama. Exons are depicted as grey boxes. The LTRs flanking Gret 1 just upstream of the Vvmyb 1 A gene are depicted as red triangles. Nature Reviews Genetics 14, 49 -61 (January 2013)
Nature Reviews Genetics 14, 49 -61 (January 2013)

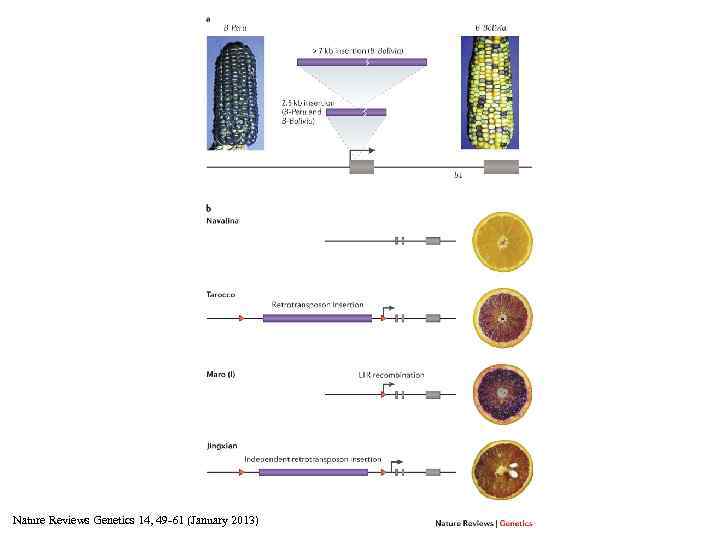
a | Effects of transposable element (TE) insertions on expression of the b 1 gene in maize. TE insertions into the first exon the b 1 gene in the B-Peru allele resulted in a shift in expression of this gene from vegetative tissues to the seed. A subsequent insertion in the B-Bolivia allele resulted in reduced and variegated expression of this gene. b | The role of TE insertions in the development of blood oranges. The insertion of Rider, a long terminal repeat (LTR) retrotransposon upstream of the Ruby gene resulted in cold-dependent expression of that gene in the fruit. The Navalina variant carries the functional native Ruby gene, which shows limited expression in the fruit flesh. Tarocco carries a Ruby allele that has an insertion of a retrotransposon immediately upstream of the Ruby coding sequence. The LTR of the retroelement provides a novel promoter that drives expression in the flesh of the fruit, resulting in the distinctive red colouration illustrated here. The LTR also confers responsiveness to cold temperatures. Recombination between the LTRs resulted in enhanced expression found in Maro (I). Jingxian is an independently derived blood orange variant that contains a distinct but related LTR retrotransposon that also confers both tissue specificity and cold responsiveness. The LTRs flanking the retrotransposons just upstream of the Ruby gene are depicted as red triangles. Exons are depicted as grey boxes. Images of corn in panel a are taken, with permission, from Ref. 51 © (1999) National Academy of Sciences USA. Images of blood oranges in panel b are taken, with permission, from Ref. 54 © (2012) American Society of Plant Biologists.
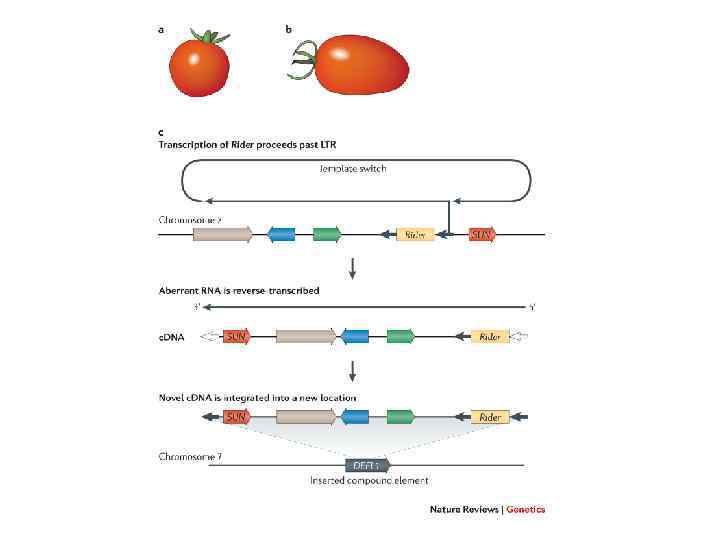

a | Most strains of cultivated tomatoes (Solanum lycopersicum) retain the round fruit shape characteristic of their wild ancestor Solanum pimpinellifolium. b | The oval fruits of some strains of cultivated tomatoes, such as Roma, result from retroposition of the IQ domain 12 (IQD 12) gene. c | This results in tissue-specific expression of IQD 12 owing to the presence of an ectopic promoter via the following three stages. First, transcription of long terminal repeat (LTR) retrotransposons normally initiates within one LTR and terminates at the end of the second LTR. However, in this case it appears that transcription continues through several other genes (green, blue and brown arrows). Template switching then results in continued transcription of the IQD 12, which is just upstream of the retrotransposon, after which transcription is terminated. The result is a long transcript that includes the retrotransposon as well as several other genes, including IQD 12. Second, this transcript is reverse-transcribed, resulting in a c. DNA. Third, this c. DNA is then integrated into a gene, DEFL 1, which is expressed in developing fruit. This leads to the expression of IQD 12 in the fruit because it is now under the control of the DEFL 1 promoter.

Ретротранспозоны класса I Обобщённая структура copia-подобных элементов дрозофилы (mdg 1, copia, 412, B 104, 297), IAP-элементов мыши и Ty дрожжей LTR gag pol LTR РНК Одна или несколько рамок считывания. В результате процессинга Образуется белок оболочки, RT-RH, протеаза и интеграза
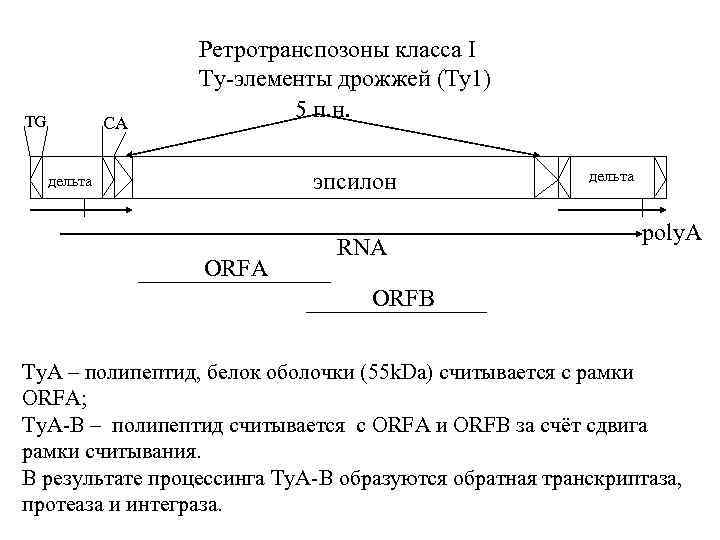
TG СА Ретротранспозоны класса I Ty-элементы дрожжей (Ty 1) 5 п. н. эпсилон дельта ORFA RNA дельта poly. A ORFB Ty. A – полипептид, белок оболочки (55 k. Da) считывается с рамки ORFA; Ty. A-B – полипептид считывается с ORFA и ORFB за счёт сдвига рамки считывания. В результате процессинга Ty. A-B образуются обратная транскриптаза, протеаза и интеграза.
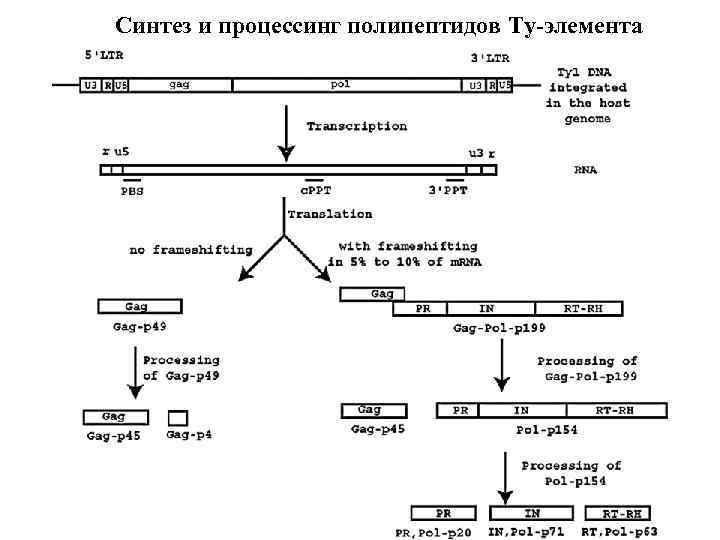
Синтез и процессинг полипептидов Ty-элемента

Механизм обратной транскрипции
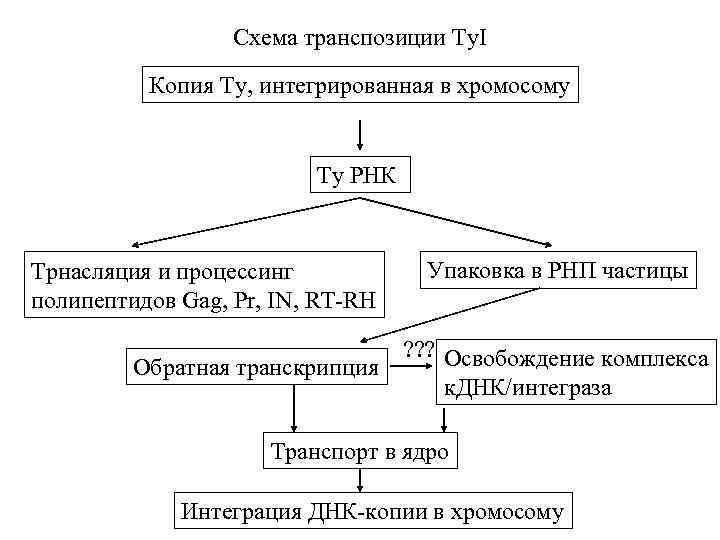
Схема транспозиции Ty. I Копия Ty, интегрированная в хромосому Ty РНК Трнасляция и процессинг полипептидов Gag, Pr, IN, RT-RH Обратная транскрипция Упаковка в РНП частицы ? ? ? Освобождение комплекса к. ДНК/интеграза Транспорт в ядро Интеграция ДНК-копии в хромосому
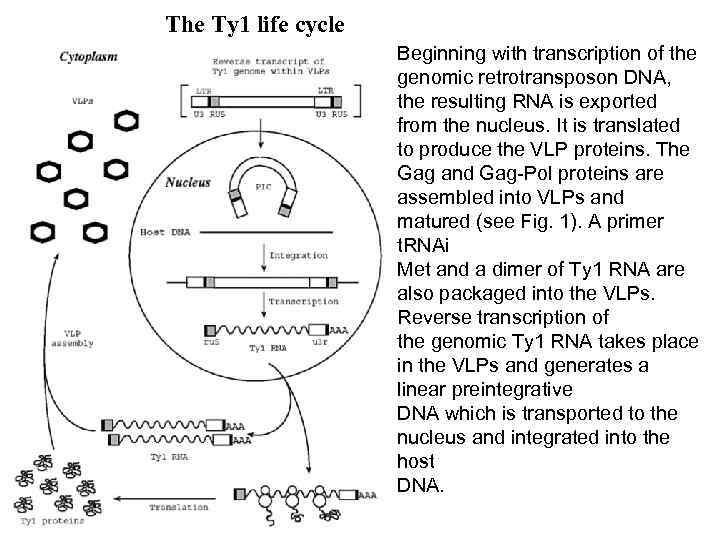
The Ty 1 life cycle Beginning with transcription of the genomic retrotransposon DNA, the resulting RNA is exported from the nucleus. It is translated to produce the VLP proteins. The Gag and Gag-Pol proteins are assembled into VLPs and matured (see Fig. 1). A primer t. RNAi Met and a dimer of Ty 1 RNA are also packaged into the VLPs. Reverse transcription of the genomic Ty 1 RNA takes place in the VLPs and generates a linear preintegrative DNA which is transported to the nucleus and integrated into the host DNA.

Интеграция ДНК-копии ретротранспозона класса I в хромосому

Ретротранспозоны класса I (продолжение) Ретровирус Gypsy LTR gag pol env LTR РНК Env - ген инфекционности, обеспечивающий способность ретровирусов проникать из клетки в клетку и из организма в организм. Дрозофилы, мутантные по локусу flamenco, в геноме которых представлен gypsy продуцируют gypsy-RNP частицы. Гомогенат из таких особей, содержащий gypsy-RNP частицы, добавленный в пищу особям, не содержащим gypsy-элементов, обладает инфекционными свойствами. Gypsy интегрирует в геном как соматических, так и половых клеток.

Ретротранспозоны класса II Ретротранспозон L 1 человека До 500 тыс копий на геном, примерно 100 из них интактны. 0. 1% наследственных заболеваний связано с транспозицией L 1 , 1/3 генома человека представляет собой псевдогены, реплицированные за счёт ретротранспозиции, опосредованной L 1. EN – эндонуклеаза; RT – обратная транскриптаза; C – цистеин богатый мотив, белок оболочки.
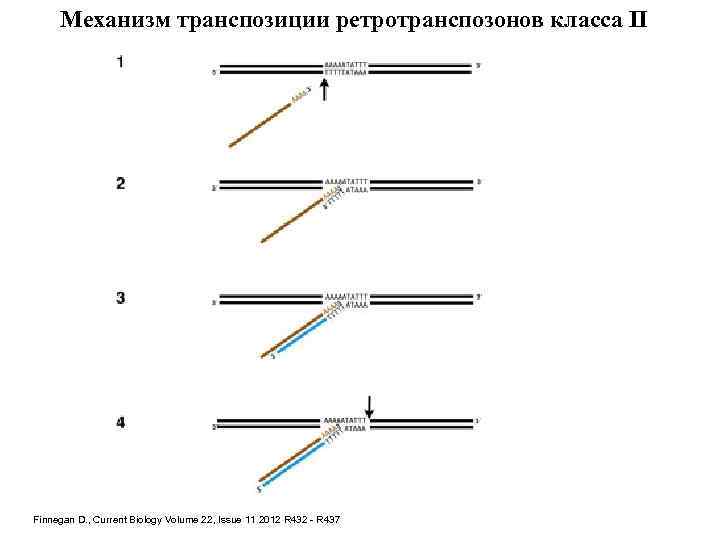
Механизм транспозиции ретротранспозонов класса II Finnegan D. , Current Biology Volume 22, Issue 11 2012 R 432 - R 437

Механизм транспозиции ретротранспозонов класса II (продолжение) Finnegan D. , Current Biology Volume 22, Issue 11 2012 R 432 - R 437

Non-LTR retrotransposons: the RNA transposition intermediate (brown) together with reverse transcriptase and nuclease (not shown) associates with chromosomal DNA (black) at an A/T rich sequence and the nuclease makes a single strand break adjacent to a run of thymines (1). This allows the poly(A) sequence at the 3′ end of the RNA to base pair with DNA at the break (2). Reverse transcriptase can then synthesise DNA (blue) using the 3′ OH at the break as primer (3). The nuclease makes a break in the opposite strand of chromosomal DNA a few nucleotides from the first (4). Template RNA is removed by RNAse H (5) allowing the new 3′ OH to prime synthesis of the second DNA strand (6) and host repair enzymes to complete integration (7).

МОБИЛЬНЫЕ ЭЛЕМЕНТЫ В ГЕНОМЕ ЧЕЛОВЕКА
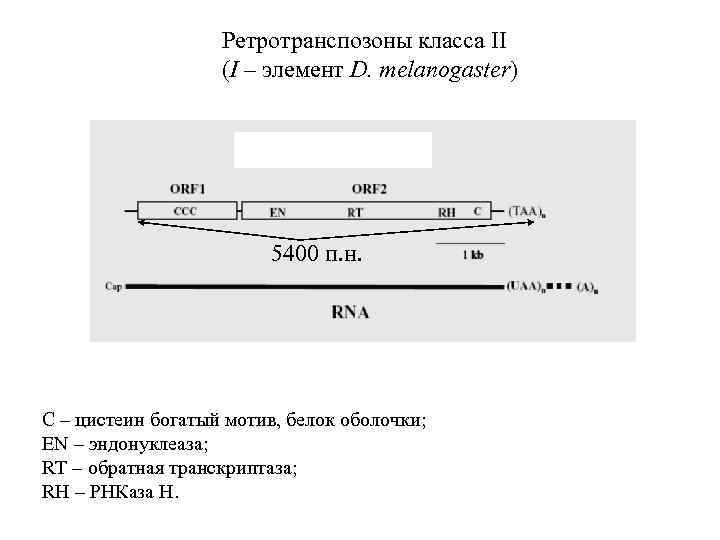
Ретротранспозоны класса II (I – элемент D. melanogaster) 5400 п. н. C – цистеин богатый мотив, белок оболочки; EN – эндонуклеаза; RT – обратная транскриптаза; RH – РНКаза Н.
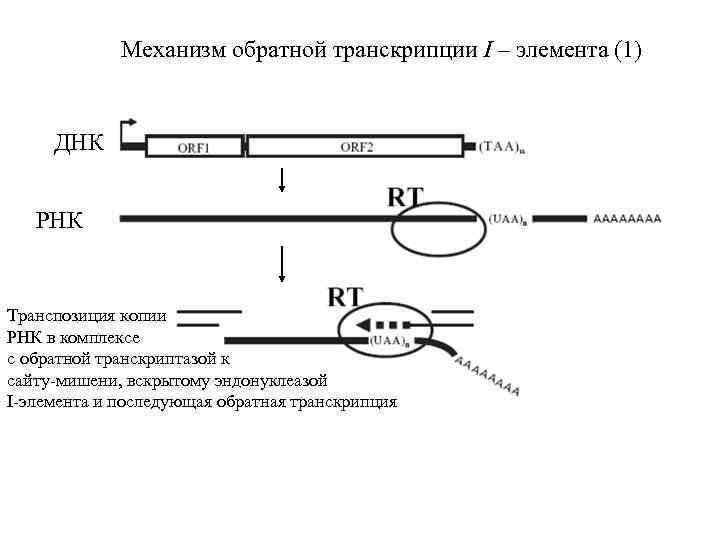
Механизм обратной транскрипции I – элемента (1) ДНК РНК Транспозиция копии РНК в комплексе с обратной транскриптазой к сайту-мишени, вскрытому эндонуклеазой I-элемента и последующая обратная транскрипция
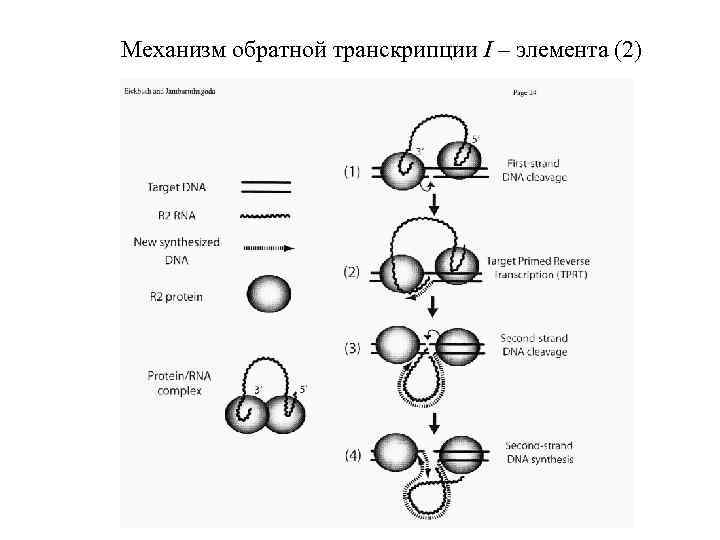
Механизм обратной транскрипции I – элемента (2)
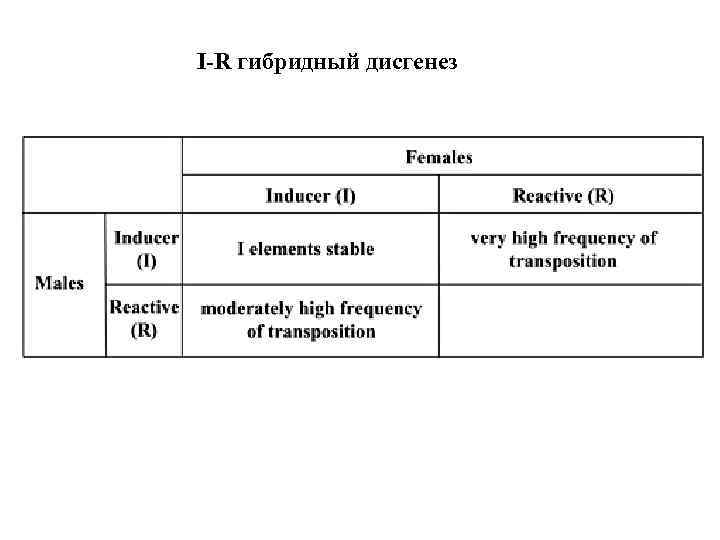
I-R гибридный дисгенез

Penelope-like retrotransposones Выявлены у насекомых, млекопитающих, растений и грибов RT сильно отличается от других ретротранспозонов и сходна c RT теломераз У некоторых организмов выявляются в субтеломерных районах LTR у некоторых представителей отсутствуют, ориентация варьирует. Uri – домен, имеющий гомологию с эндонуклеазой бактерий.
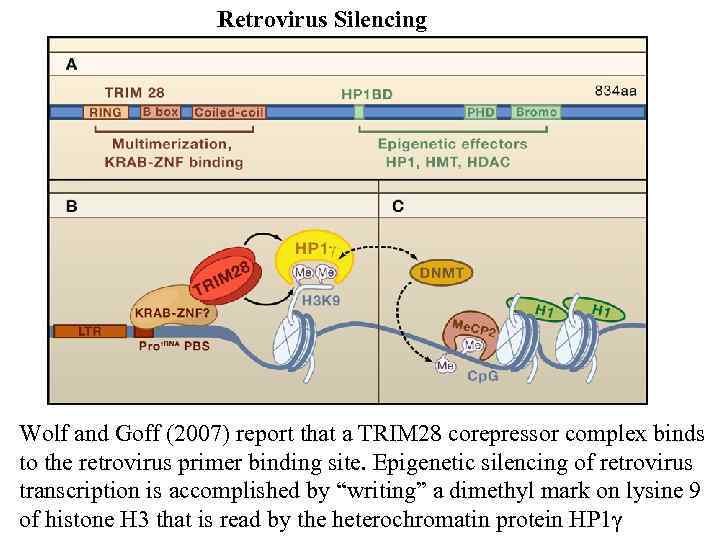
Retrovirus Silencing Wolf and Goff (2007) report that a TRIM 28 corepressor complex binds to the retrovirus primer binding site. Epigenetic silencing of retrovirus transcription is accomplished by “writing” a dimethyl mark on lysine 9 of histone H 3 that is read by the heterochromatin protein HP 1γ

Ретрогены Перемещаются путём обратной транскрипции, не содержат концевых повторов, на 3’-конце имеется поли-А последовательность или сигнал полиаденилирования, размер дупликации сайта-мишени варьирует. Широко представлены у млекопитающих, реже у других позвоночных и растений, а у беспозвоночных отсутствуют. Вероятно, перемещаются за счёт чужой обратной транскриптазы и РНП-частиц ретровирусов или ретротранспозонов. Подразделяются на : - процессированные псевдогены полипептидов (актин человека, альфа-тубулин крысы, альфа-глобин крысы, бета-тубулин человека и т. д. ); - процессированные псевдогены малых цитоплазматических РНК (т. РНК, Alu-SINE приматов, SINE 1 мыши). Сохраняют внутрение промоторы для РНК полимеразы III и транскрибируются; - процессированные псевдогены малых ядерных РНК (U 1, U 2, U 3). По сравнению с исходным геном укорочен 3’-конец, не имеют внутреннего промотора, синтезируются с помощью РНК полимеразы III.
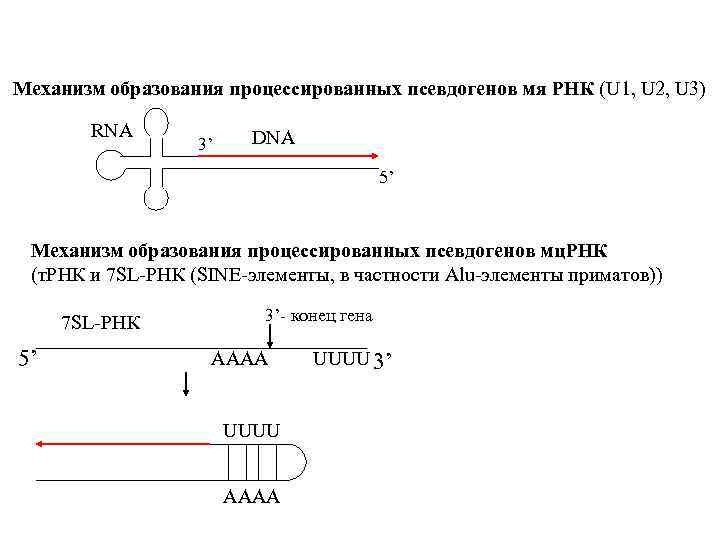
Механизм образования процессированных псевдогенов мя РНК (U 1, U 2, U 3) RNA 3’ DNA 5’ Механизм образования процессированных псевдогенов мц. РНК (т. РНК и 7 SL-РНК (SINE-элементы, в частности Alu-элементы приматов)) 7 SL-РНК 5’ 3’- конец гена AAAA UUUU 3’

Роль мобильных элементов в геноме 1. Инсерции МЭ и хромосомные аберрации, опосредованные МЭ, чаще всего нейтральны или вредны. Некоторые инсерции ретротранспозонов усиливают экспрессию прилежащих генов и иногда адаптивны, а в других случаях приводят к развитию патологий, в частности, вызывают кансерогенез. 2. Перестройки, опосредованные МЭ обеспечивают пластичность генома. 3. Ретротранспозоны класса II участвуют в репарации двунитевых разрывов. 4. МЭ составляют существенную часть геномов многих организмов и играют важную роль в структурно-функциональной организации генома (стабильность хр-м, инактивация хроматина , инактивация Х-хромосомы, регуляторные последовательности). 5. Транспозиция псевдогенов приводит к возникновению новых генов. 6. Возможная роль МЭ в негативной регуляции активности вирусов. 7. Доместикация МЭ – возникновение систем, использующих транспозицию для регуляции жизненно-важных процессов. 8. Теломераза - неавтономный ретротранспозон, возникновение которого способствовало появлению линейных хромосом и многоклеточных организмов

L 1 – эелемент активирует экспрессию apo(a)
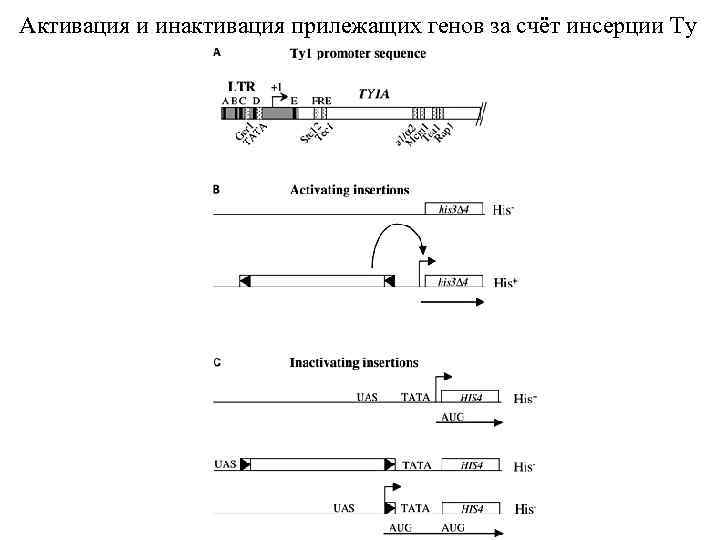
Активация и инактивация прилежащих генов за счёт инсерции Ty
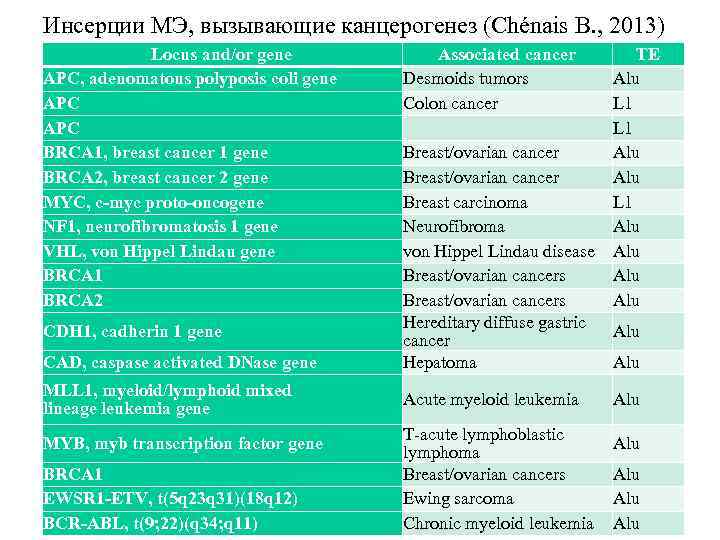
Инсерции МЭ, вызывающие канцерогенез (Chénais B. , 2013) Locus and/or gene APC, adenomatous polyposis coli gene APC BRCA 1, breast cancer 1 gene BRCA 2, breast cancer 2 gene MYC, c-myc proto-oncogene NF 1, neurofibromatosis 1 gene VHL, von Hippel Lindau gene BRCA 1 BRCA 2 CAD, caspase activated DNase gene TE Alu L 1 Breast/ovarian cancer Alu Breast carcinoma L 1 Neurofibroma Alu von Hippel Lindau disease Alu Breast/ovarian cancers Alu Hereditary diffuse gastric Alu cancer Hepatoma Alu MLL 1, myeloid/lymphoid mixed lineage leukemia gene Acute myeloid leukemia Alu MYB, myb transcription factor gene T-acute lymphoblastic lymphoma Breast/ovarian cancers Ewing sarcoma Chronic myeloid leukemia Alu CDH 1, cadherin 1 gene BRCA 1 EWSR 1 -ETV, t(5 q 23 q 31)(18 q 12) BCR-ABL, t(9; 22)(q 34; q 11) Associated cancer Desmoids tumors Colon cancer Alu Alu
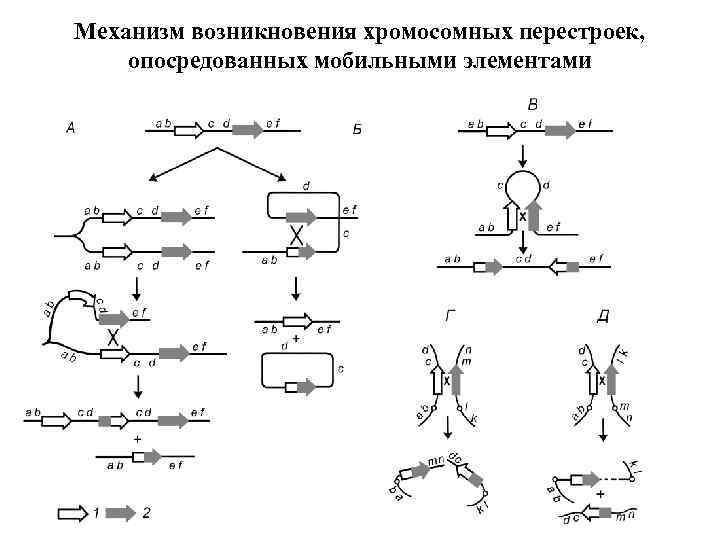
Механизм возникновения хромосомных перестроек, опосредованных мобильными элементами

А – делеции и дупликации; Б – делеции без дупликаций; В – инверсии; Г – транслкации; Д – образование мостов и фрагментов в анафазе I: I – исходная копия мигрирующего элемента, 2 – копия мигрирующего элемента, переместившаяся в новое место.

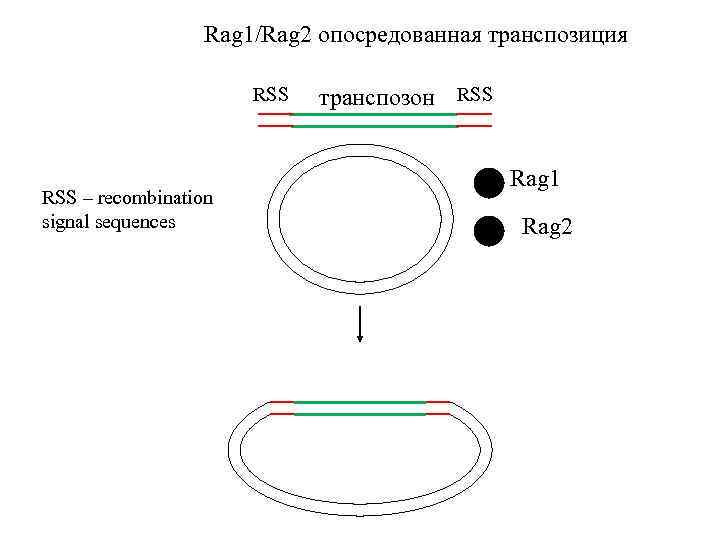
Rag 1/Rag 2 опосредованная транспозиция RSS – recombination signal sequences транспозон RSS Rag 1 Rag 2

Транспозиция кассет в локусе типа спаривания у дрожжей

Транспозиция кассет в локусе типа спаривания у дрожжей (продолжение) MATa
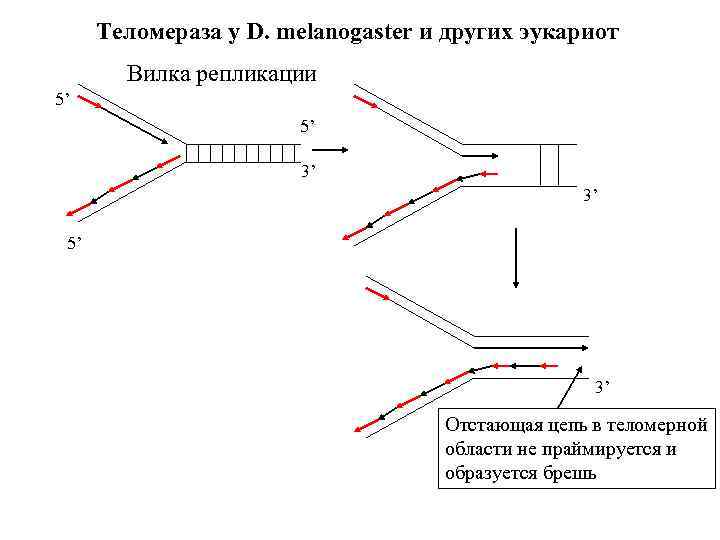
Теломераза у D. melanogaster и других эукариот Вилка репликации 5’ 5’ 3’ 3’ 5’ 3’ Отстающая цепь в теломерной области не праймируется и образуется брешь
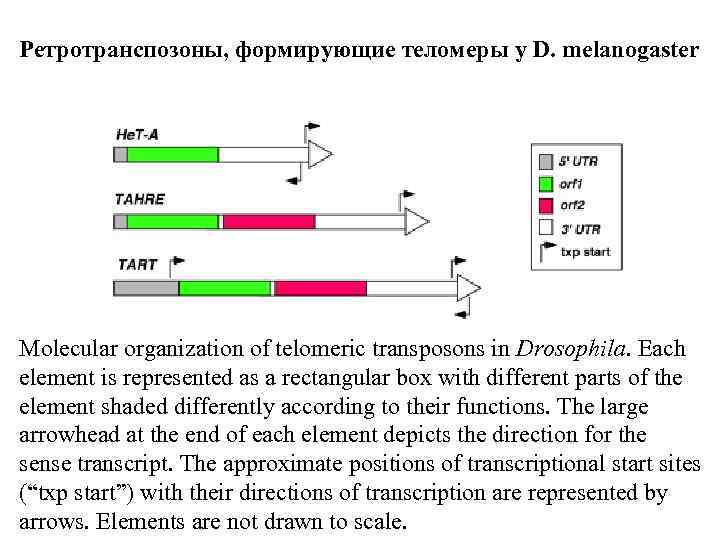
Ретротранспозоны, формирующие теломеры у D. melanogaster Molecular organization of telomeric transposons in Drosophila. Each element is represented as a rectangular box with different parts of the element shaded differently according to their functions. The large arrowhead at the end of each element depicts the direction for the sense transcript. The approximate positions of transcriptional start sites (“txp start”) with their directions of transcription are represented by arrows. Elements are not drawn to scale.
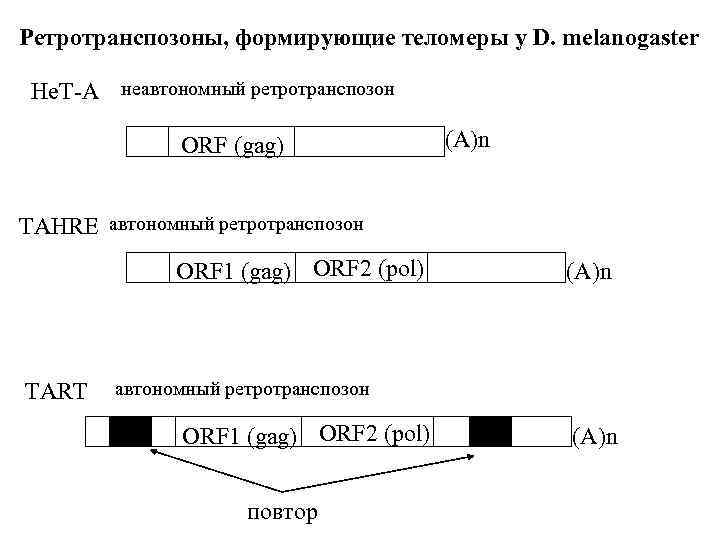
Ретротранспозоны, формирующие теломеры у D. melanogaster He. T-A неавтономный ретротранспозон ORF (gag) (A)n TAHRE автономный ретротранспозон ORF 1 (gag) ORF 2 (pol) TART (A)n автономный ретротранспозон ORF 1 (gag) ORF 2 (pol) повтор (A)n
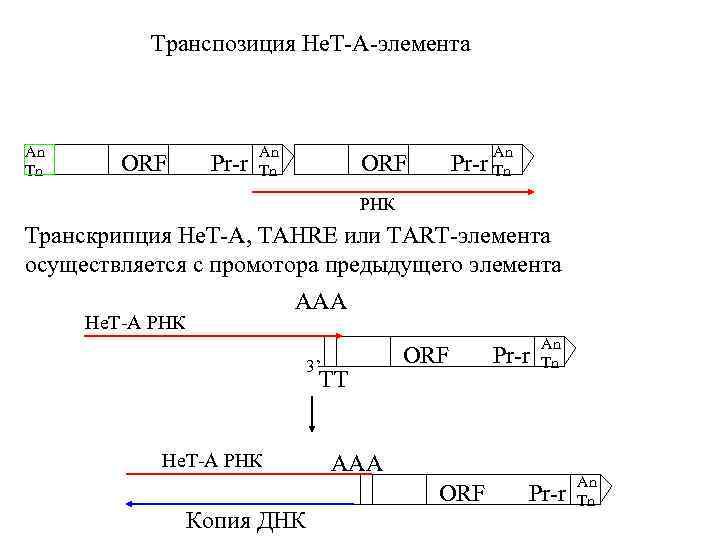
Транспозиция Нe. Т-A-элемента An Tn ORF Pr-r An Tn An ORF Pr-r Tn РНК Транскрипция Нe. Т-A, TAHRE или TART-элемента осуществляется с промотора предыдущего элемента AAA Нe. Т-A РНК 3’ TT Нe. Т-A РНК ORF An Tn AAA ORF Копия ДНК Pr-r An Tn
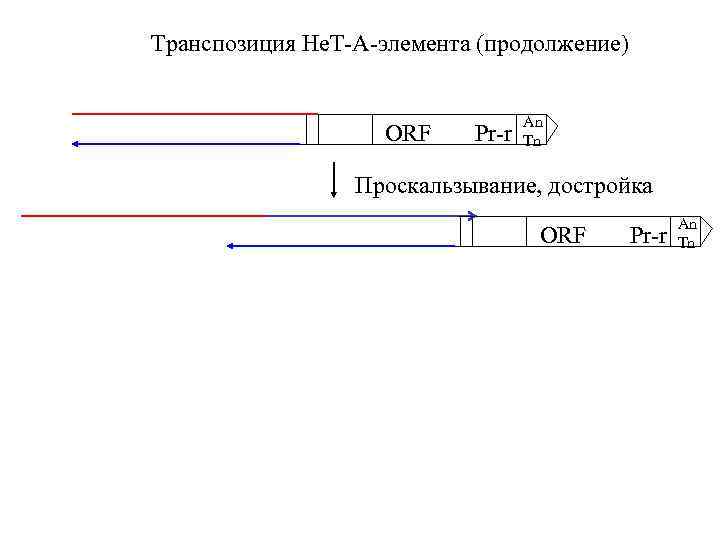
Транспозиция Нe. Т-A-элемента (продолжение) ORF Pr-r An Tn Проскальзывание, достройка ORF Pr-r An Tn
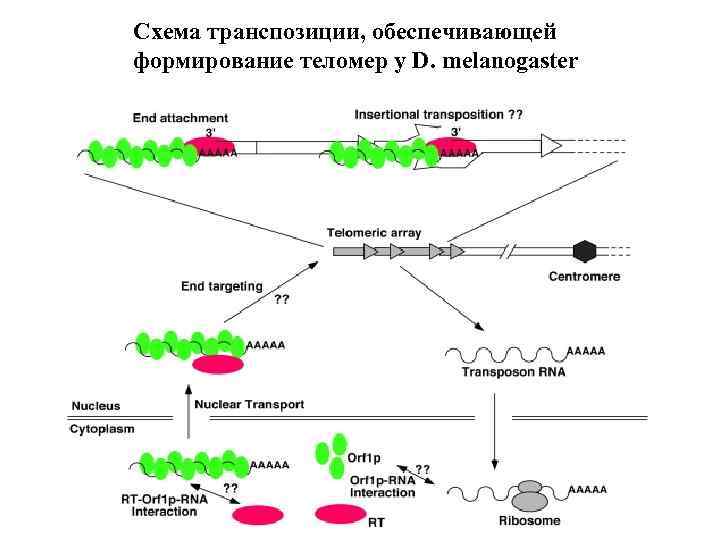
Схема транспозиции, обеспечивающей формирование теломер у D. melanogaster

A model for the life cycle of the Drosophila telomeric transposons. In the central region of the diagram is a single telomere with four transposon insertions (gray arrows). Transposon RNA (wavery line) with a poly-A tail is exported to cytoplasm for translation to produce Orf 1 p (green ovals) and RT (red oval). Orf 1 p and RT could come from different elements, e. g. Orf 1 p from a He. T-A and RT from a TAHRE. How Orf 1 p interacts with its RNA is unknown. How Orf 1 p–RNA interacts with RT is also unknown. Presumably, an RT–Orf 1 p–RNA complex is imported into the nucleus and targeted to the telomeric region of the chromosome. The mechanism for this “end targeting” process is unknown. A close-up view of the single representative telomere is given at the top. Two possible modes of transposition are given: end attachment, which has strong supporting evidence, and insertional transposition, which is speculative. During end attachment, the transposon machinery uses the 3′ end of the chromosome to prime reverse transcription. During insertional transposition, the EN activity of RT makes a nick near the junction between two arbitrarily chosen elements. The transposon machinery uses the 3′ end of this nick to prime reverse transcription.
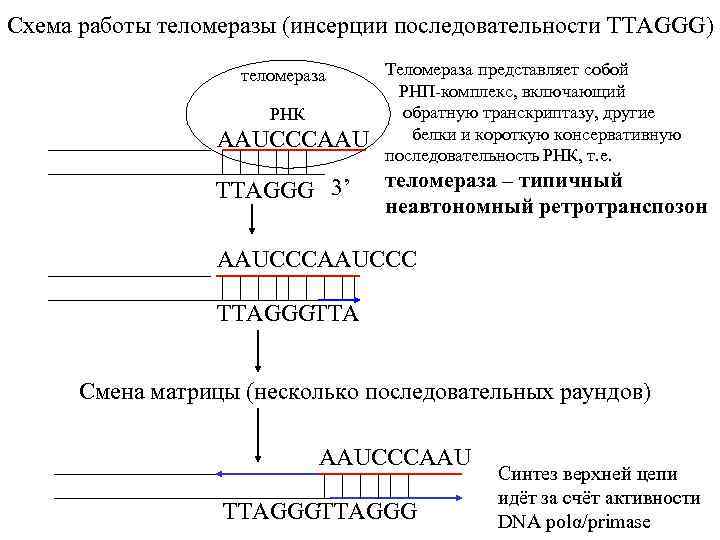
Схема работы теломеразы (инсерции последовательности TTAGGG) теломераза РНК AAUCCCAAU TTAGGG 3’ Теломераза представляет собой РНП-комплекс, включающий обратную транскриптазу, другие белки и короткую консервативную последовательность РНК, т. е. теломераза – типичный неавтономный ретротранспозон AAUCCC TTAGGGTTA Смена матрицы (несколько последовательных раундов) AAUCCCAAU TTAGGG Синтез верхней цепи идёт за счёт активности DNA polα/primase
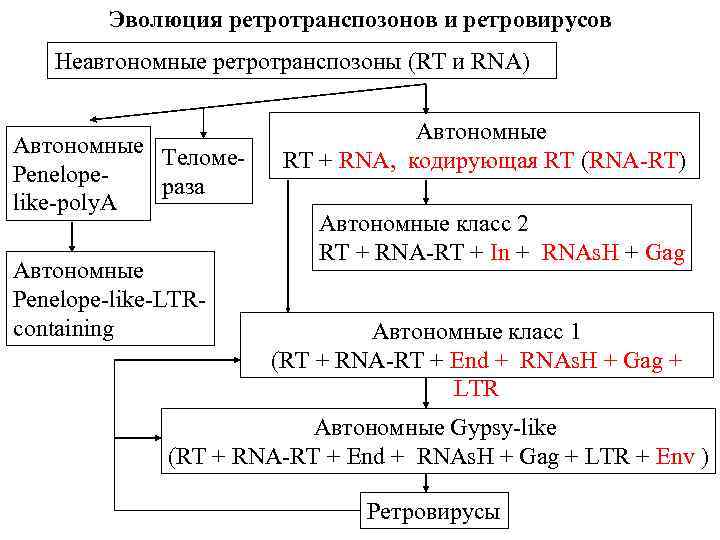
Эволюция ретротранспозонов и ретровирусов Неавтономные ретротранспозоны (RT и RNA) Автономные Теломе. Penelopeраза like-poly. A Автономные Penelope-like-LTRcontaining Автономные RT + RNA, кодирующая RT (RNA-RT) Автономные класс 2 RT + RNA-RT + In + RNAs. H + Gag Автономные класс 1 (RT + RNA-RT + End + RNAs. H + Gag + LTR Автономные Gypsy-like (RT + RNA-RT + End + RNAs. H + Gag + LTR + Env ) Ретровирусы
моб эл-ты генома.ppt