Лекция 1. ДНК, Репликация vdk.pptx
- Количество слайдов: 93
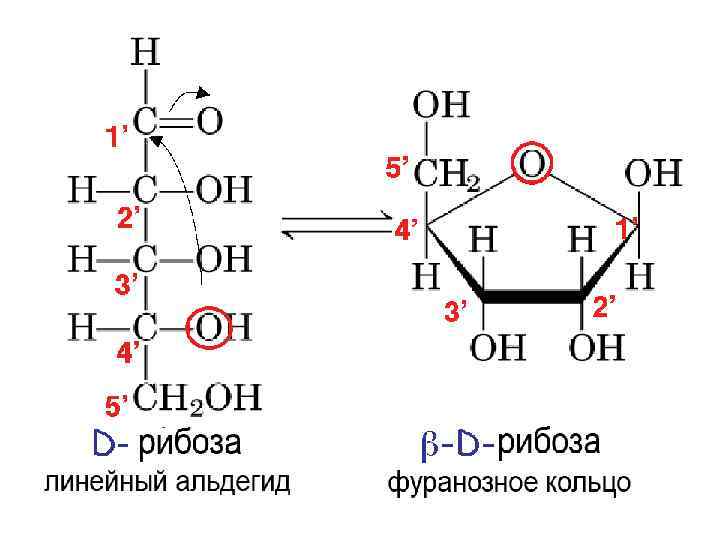
 История открытия ДНК и доказательства ее генетической роли * В 1869 году Мишер выделил ядра из белых кровяных клеток и продемонстрировал, что они содержат новое фосфорорганическое соединение ( «нуклеин» ) * К концу 19 века нуклеиновые кислоты (ДНК и РНК) были очищены и началось их изучение В 30 годы 20 века было показано, что нуклеиновые кислоты состоят из сахара (рибозы или дезоксирибозы) и четырех (пяти) типов азотсодержащих оснований. Было также показано, что основания ковалентно связаны с остатком сахара В 1953 году
История открытия ДНК и доказательства ее генетической роли * В 1869 году Мишер выделил ядра из белых кровяных клеток и продемонстрировал, что они содержат новое фосфорорганическое соединение ( «нуклеин» ) * К концу 19 века нуклеиновые кислоты (ДНК и РНК) были очищены и началось их изучение В 30 годы 20 века было показано, что нуклеиновые кислоты состоят из сахара (рибозы или дезоксирибозы) и четырех (пяти) типов азотсодержащих оснований. Было также показано, что основания ковалентно связаны с остатком сахара В 1953 году
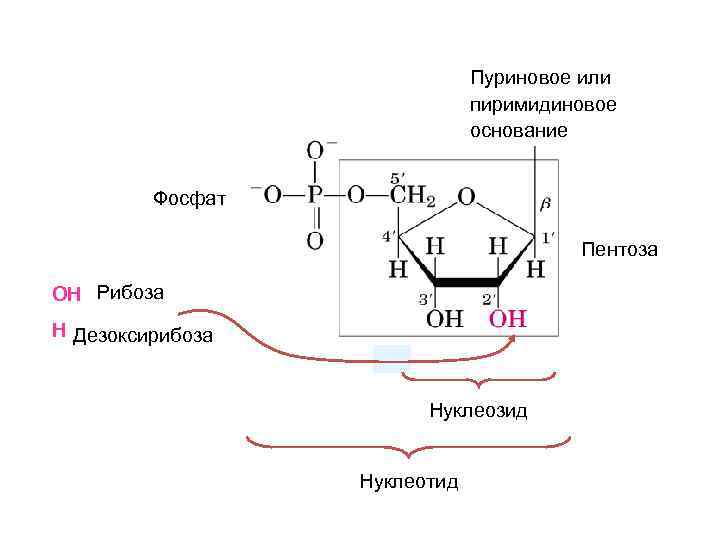 Пуриновое или пиримидиновое основание Фосфат Пентоза OH Рибоза H Дезоксирибоза Нуклеозид Нуклеотид
Пуриновое или пиримидиновое основание Фосфат Пентоза OH Рибоза H Дезоксирибоза Нуклеозид Нуклеотид
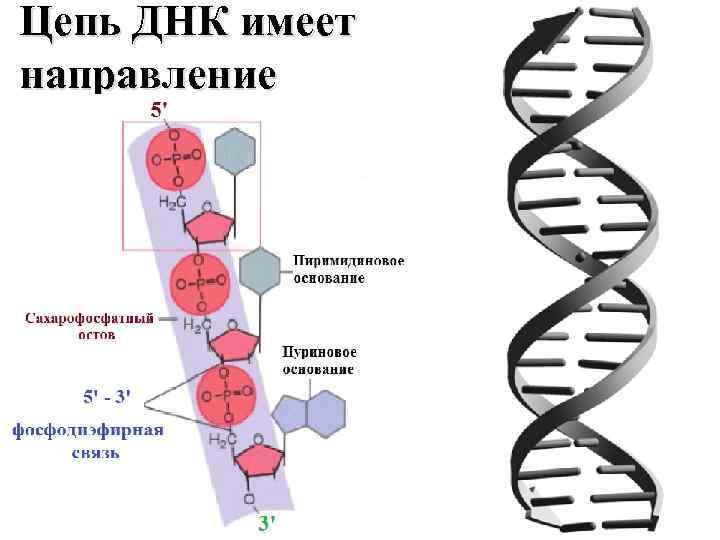 Цепь ДНК имеет направление
Цепь ДНК имеет направление
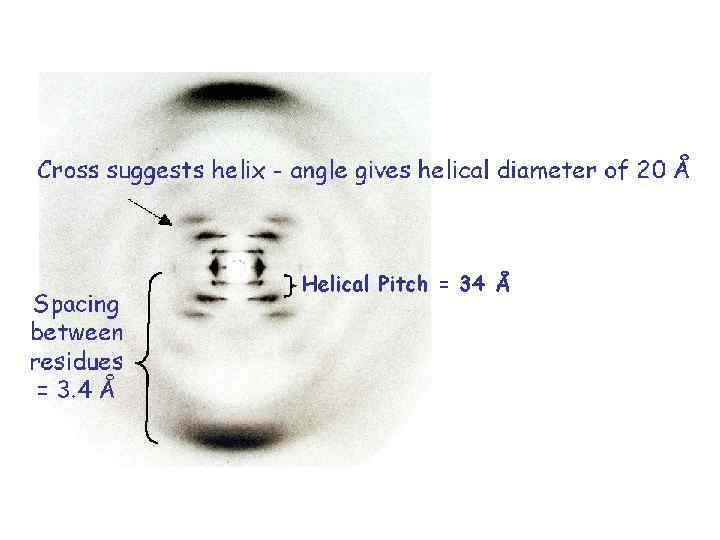
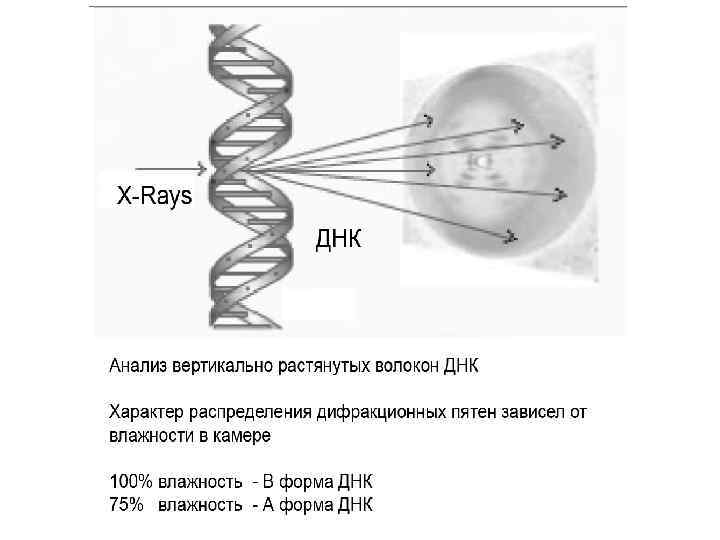
 Толщина спирали позволяла предположить, что она состоит из двух или трех цепей ДНК
Толщина спирали позволяла предположить, что она состоит из двух или трех цепей ДНК
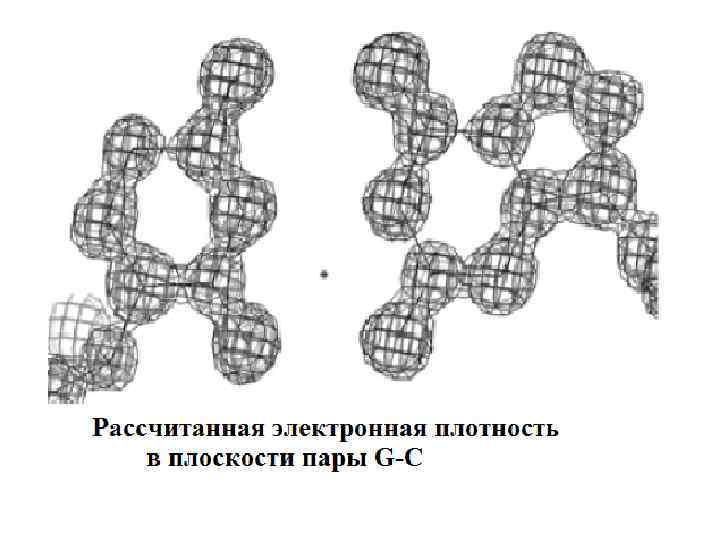
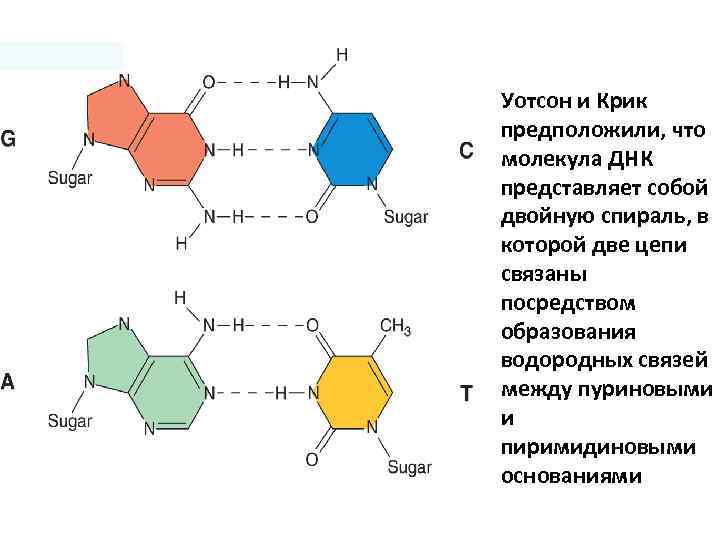 Уотсон и Крик предположили, что молекула ДНК представляет собой двойную спираль, в которой две цепи связаны посредством образования водородных связей между пуриновыми и пиримидиновыми основаниями
Уотсон и Крик предположили, что молекула ДНК представляет собой двойную спираль, в которой две цепи связаны посредством образования водородных связей между пуриновыми и пиримидиновыми основаниями

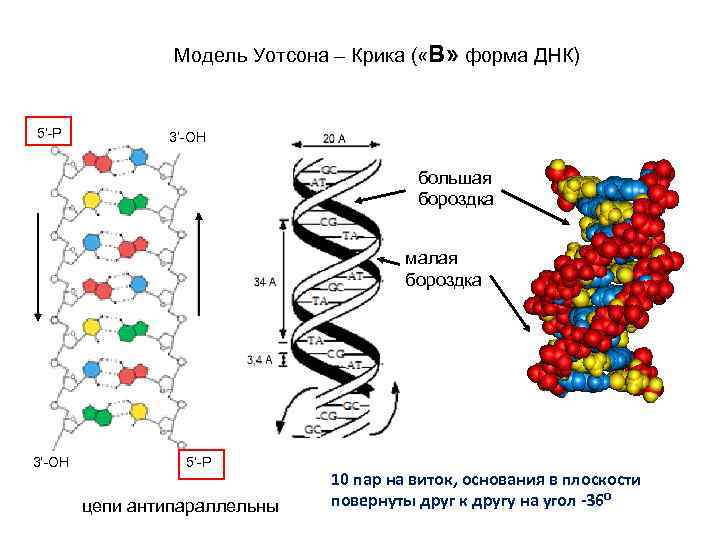 Модель Уотсона – Крика ( «B» форма ДНК) 5’-P 3’-OH большая бороздка малая бороздка 3’-OH 5’-P цепи антипараллельны 10 пар на виток, основания в плоскости повернуты друг к другу на угол -36⁰
Модель Уотсона – Крика ( «B» форма ДНК) 5’-P 3’-OH большая бороздка малая бороздка 3’-OH 5’-P цепи антипараллельны 10 пар на виток, основания в плоскости повернуты друг к другу на угол -36⁰
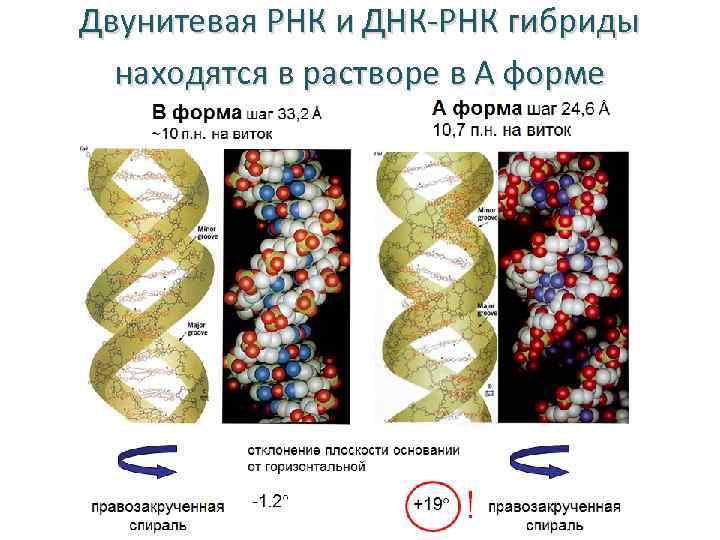 Двунитевая РНК и ДНК-РНК гибриды находятся в растворе в А форме
Двунитевая РНК и ДНК-РНК гибриды находятся в растворе в А форме
 Z форму при определенных условиях принимают поли d. G/d. C последовательности GCGCGCG Последовательности, способные образовывать Z форму, присутствуют в промоторах некоторых генов. Можно получить антитела к Z ДНК. С использованием этих антител было продемонстрировано, что Z ДНК может существовать в живой клетке. Однако, существуют опасения, что сами по себе антитела стабилизируют короткоживущую Z форму
Z форму при определенных условиях принимают поли d. G/d. C последовательности GCGCGCG Последовательности, способные образовывать Z форму, присутствуют в промоторах некоторых генов. Можно получить антитела к Z ДНК. С использованием этих антител было продемонстрировано, что Z ДНК может существовать в живой клетке. Однако, существуют опасения, что сами по себе антитела стабилизируют короткоживущую Z форму
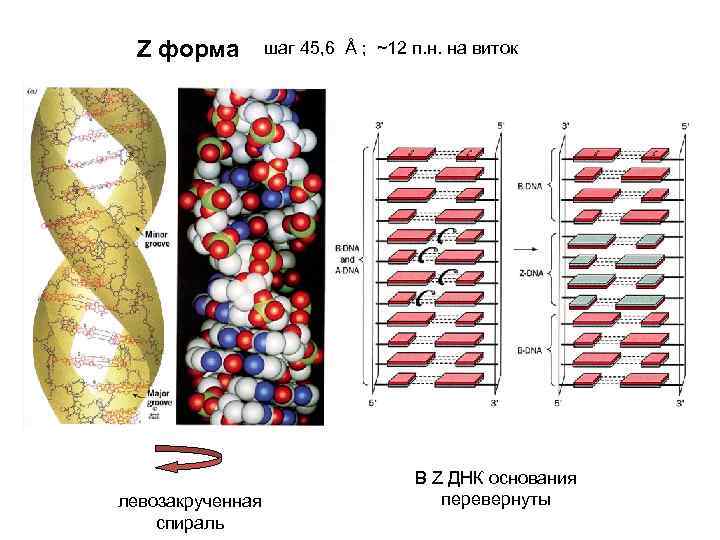 Z форма левозакрученная спираль шаг 45, 6 Å ; ~12 п. н. на виток В Z ДНК основания перевернуты
Z форма левозакрученная спираль шаг 45, 6 Å ; ~12 п. н. на виток В Z ДНК основания перевернуты
 А форма В форма Z форма
А форма В форма Z форма
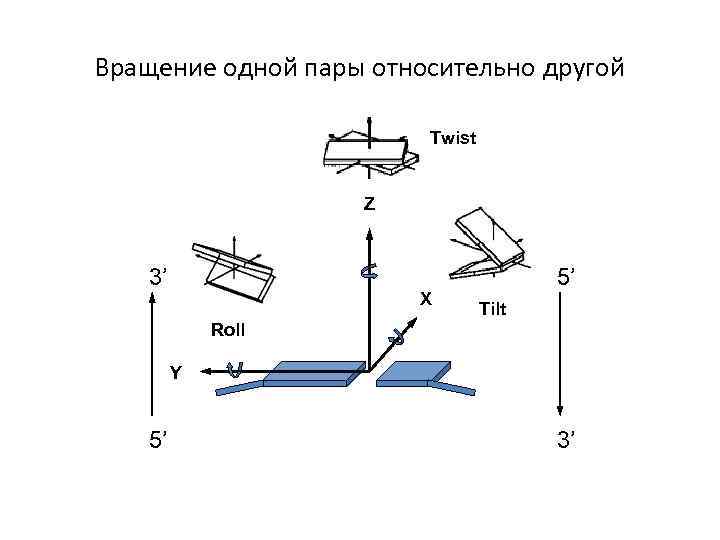 Вращение одной пары относительно другой Twist Z 3’ X 5’ Tilt Roll Y 5’ 3’
Вращение одной пары относительно другой Twist Z 3’ X 5’ Tilt Roll Y 5’ 3’
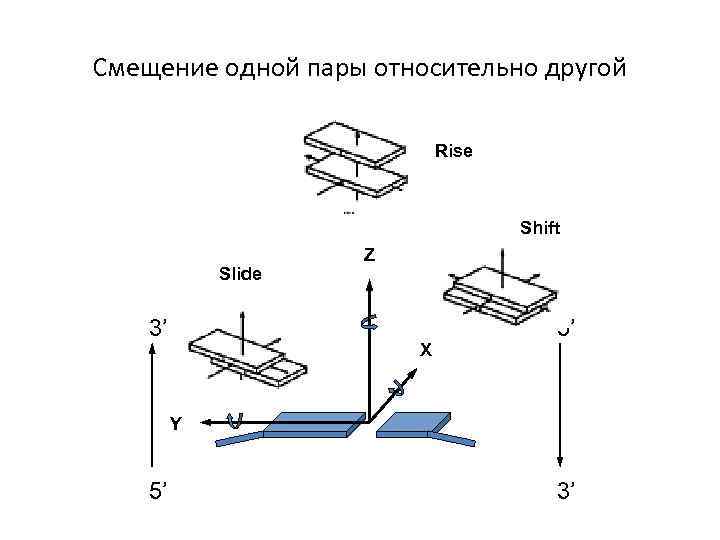 Смещение одной пары относительно другой Rise Shift Slide 3’ Z X 5’ Y 5’ 3’
Смещение одной пары относительно другой Rise Shift Slide 3’ Z X 5’ Y 5’ 3’
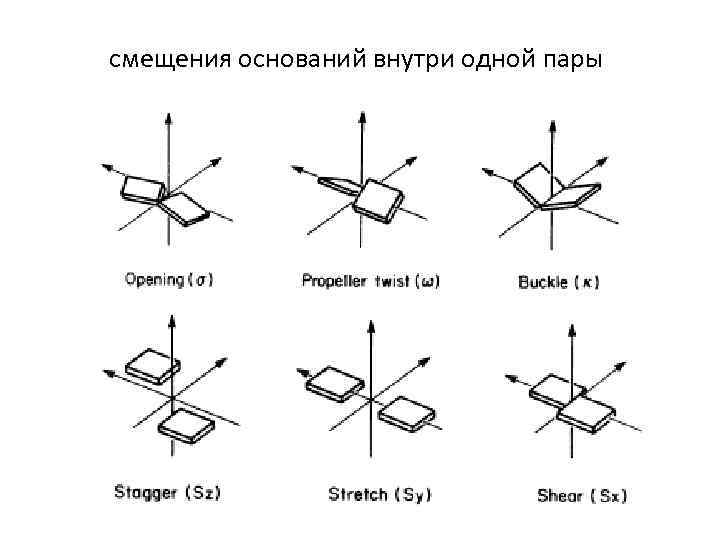 смещения оснований внутри одной пары
смещения оснований внутри одной пары
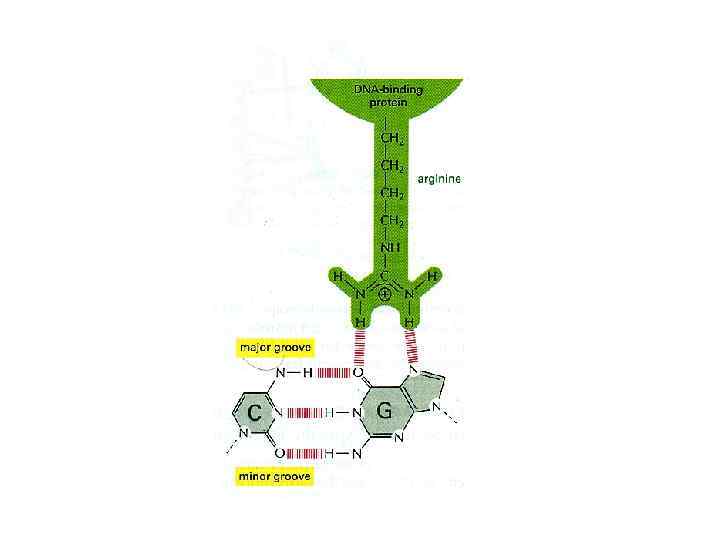
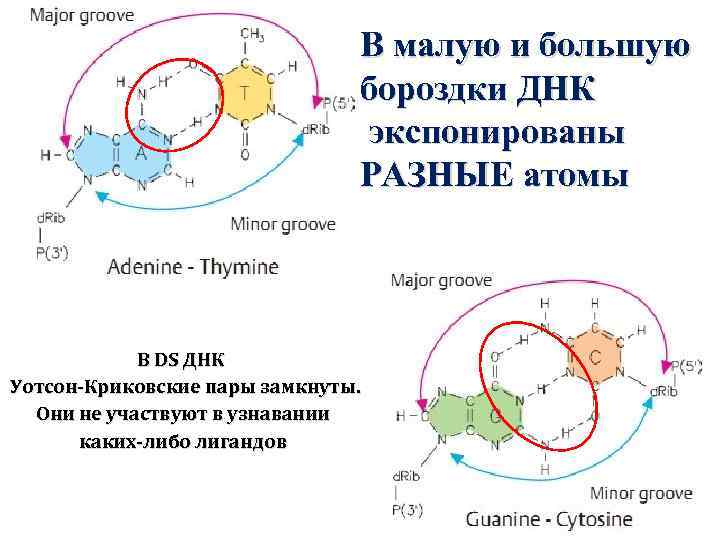 В малую и большую бороздки ДНК экспонированы РАЗНЫЕ атомы В DS ДНК Уотсон-Криковские пары замкнуты. Они не участвуют в узнавании каких-либо лигандов
В малую и большую бороздки ДНК экспонированы РАЗНЫЕ атомы В DS ДНК Уотсон-Криковские пары замкнуты. Они не участвуют в узнавании каких-либо лигандов
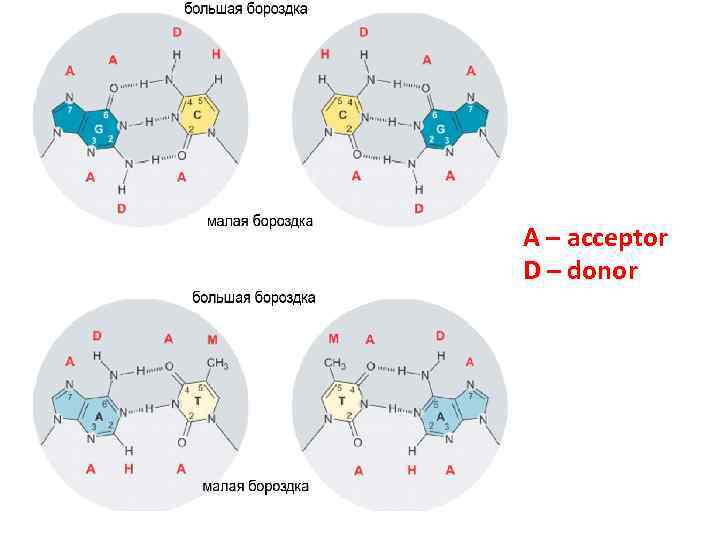 A – acceptor D – donor
A – acceptor D – donor
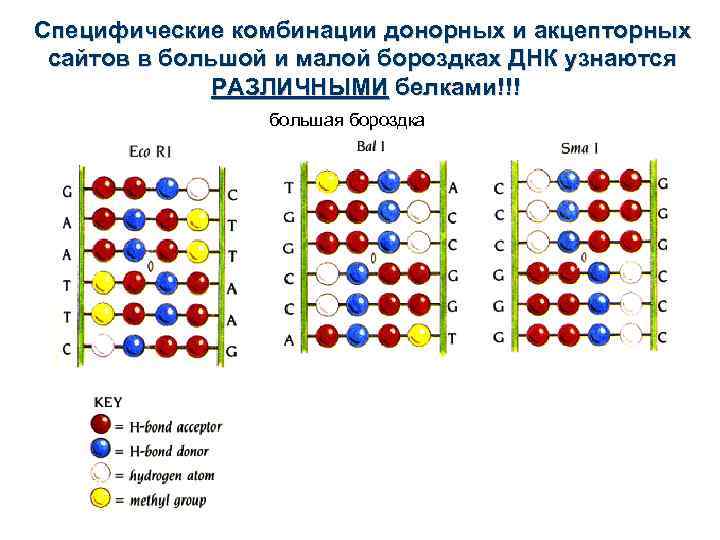 Специфические комбинации донорных и акцепторных сайтов в большой и малой бороздках ДНК узнаются РАЗЛИЧНЫМИ белками!!! большая бороздка
Специфические комбинации донорных и акцепторных сайтов в большой и малой бороздках ДНК узнаются РАЗЛИЧНЫМИ белками!!! большая бороздка
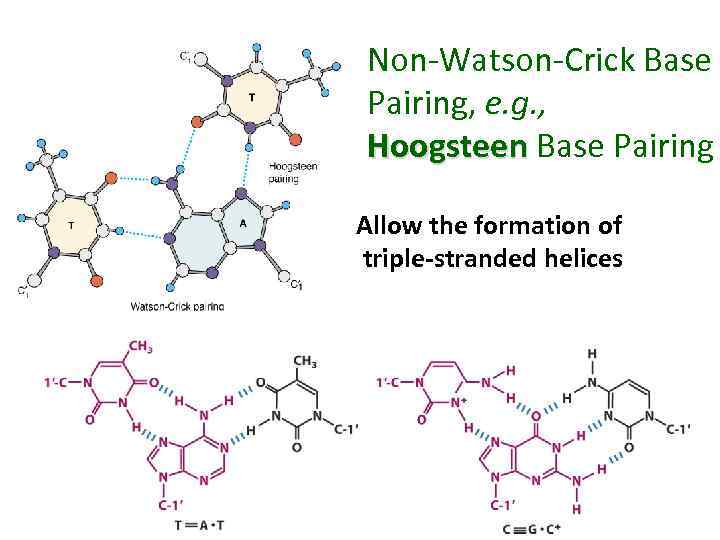 Non-Watson-Crick Base Pairing, e. g. , Hoogsteen Base Pairing Allow the formation of triple-stranded helices
Non-Watson-Crick Base Pairing, e. g. , Hoogsteen Base Pairing Allow the formation of triple-stranded helices
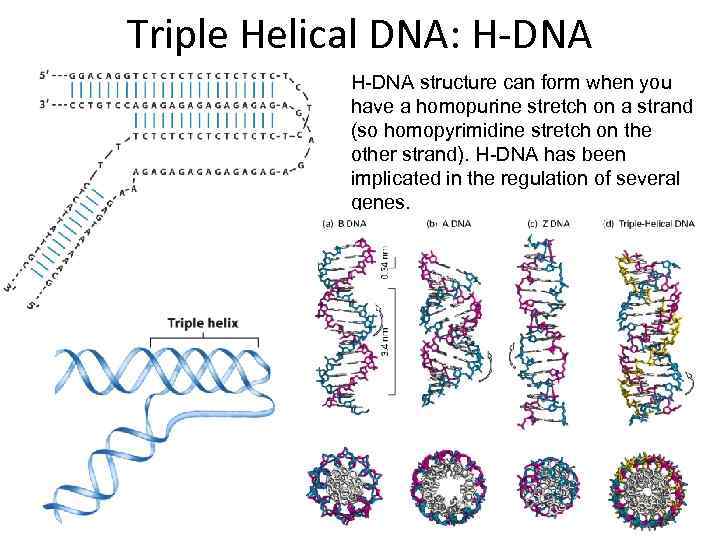 Triple Helical DNA: H-DNA structure can form when you have a homopurine stretch on a strand (so homopyrimidine stretch on the other strand). H-DNA has been implicated in the regulation of several genes.
Triple Helical DNA: H-DNA structure can form when you have a homopurine stretch on a strand (so homopyrimidine stretch on the other strand). H-DNA has been implicated in the regulation of several genes.
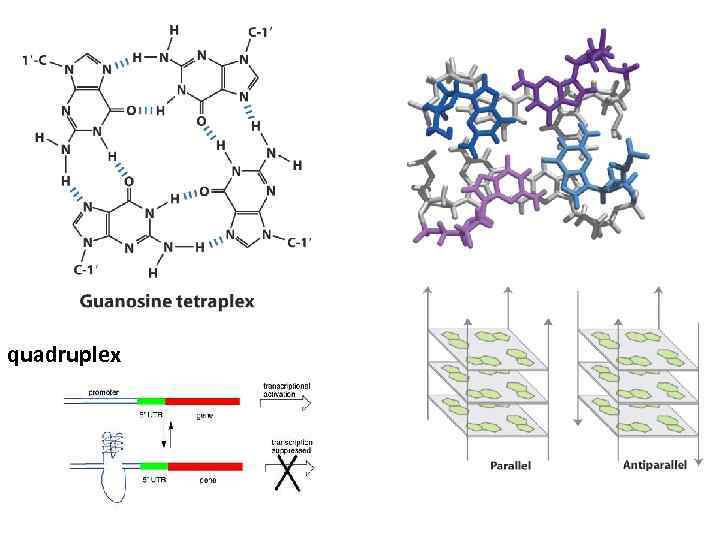 quadruplex
quadruplex
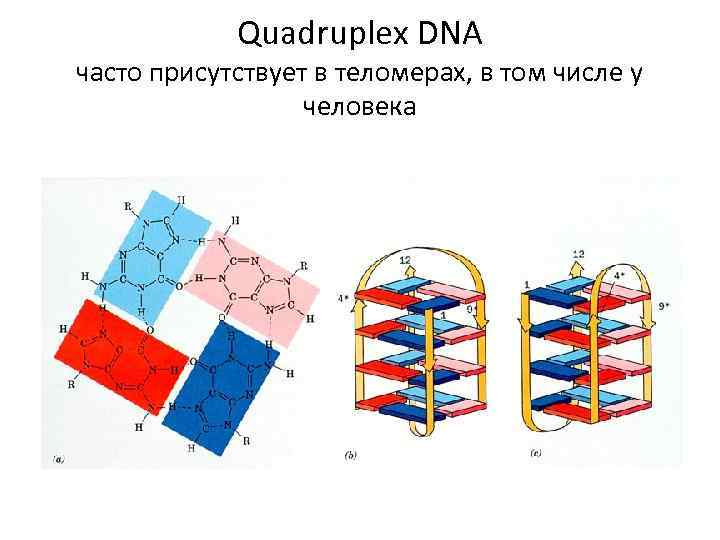 Quadruplex DNA часто присутствует в теломерах, в том числе у человека
Quadruplex DNA часто присутствует в теломерах, в том числе у человека
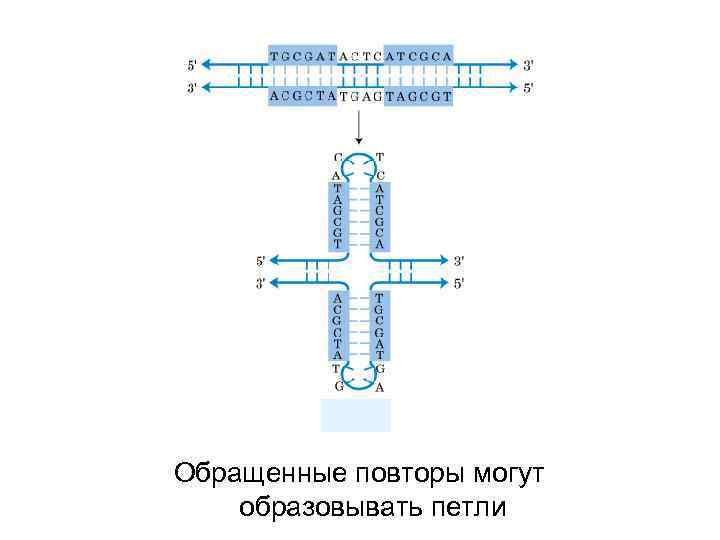 Обращенные повторы могут образовывать петли
Обращенные повторы могут образовывать петли
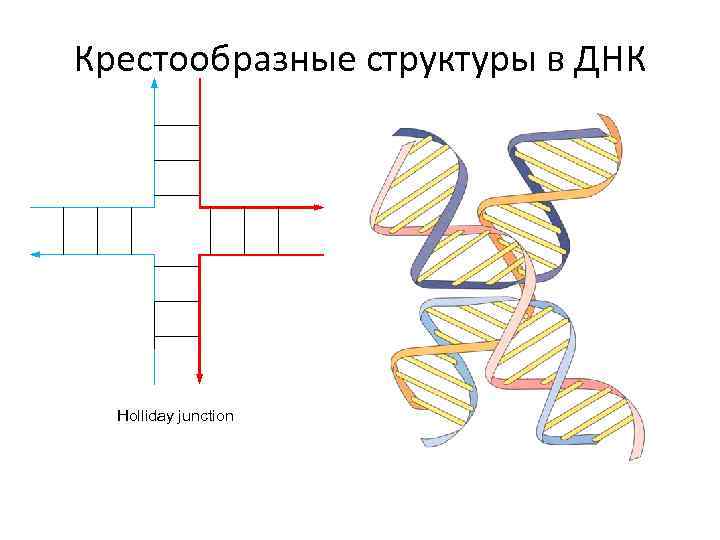 Крестообразные структуры в ДНК Holliday junction
Крестообразные структуры в ДНК Holliday junction
 Плоскости соседних пар оснований не строго параллельны. Каждая комплементарная пара оснований является как бы клином, отклоняющим ось спирали в одном или в другом направлении. Наибольший ≪крен≫ наблюдается тогда, когда два соседних аденина в одной цепи спарены с двумя тиминами другой. В этом месте происходит локальное искривление спирали. Если такие пары встречаются с периодичностью примерно один раз на 10 пар (т. е. один раз на каждый виток спирали), то молекула ДНК приобретает заметно искривленную форму.
Плоскости соседних пар оснований не строго параллельны. Каждая комплементарная пара оснований является как бы клином, отклоняющим ось спирали в одном или в другом направлении. Наибольший ≪крен≫ наблюдается тогда, когда два соседних аденина в одной цепи спарены с двумя тиминами другой. В этом месте происходит локальное искривление спирали. Если такие пары встречаются с периодичностью примерно один раз на 10 пар (т. е. один раз на каждый виток спирали), то молекула ДНК приобретает заметно искривленную форму.
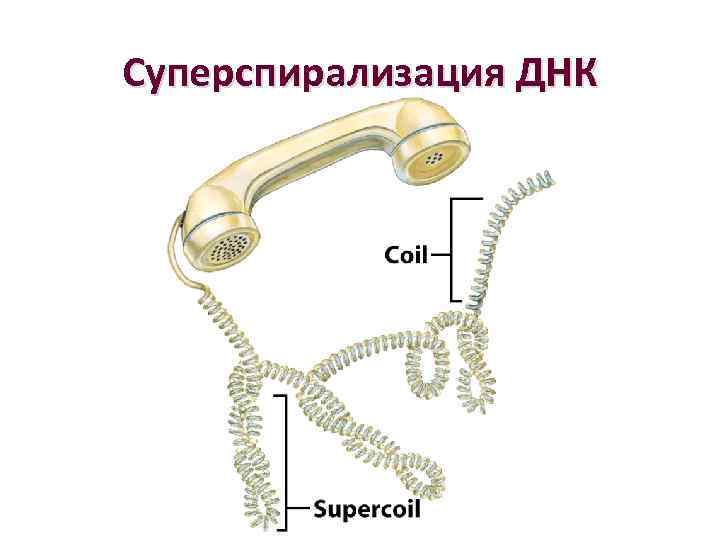 Суперспирализация ДНК
Суперспирализация ДНК
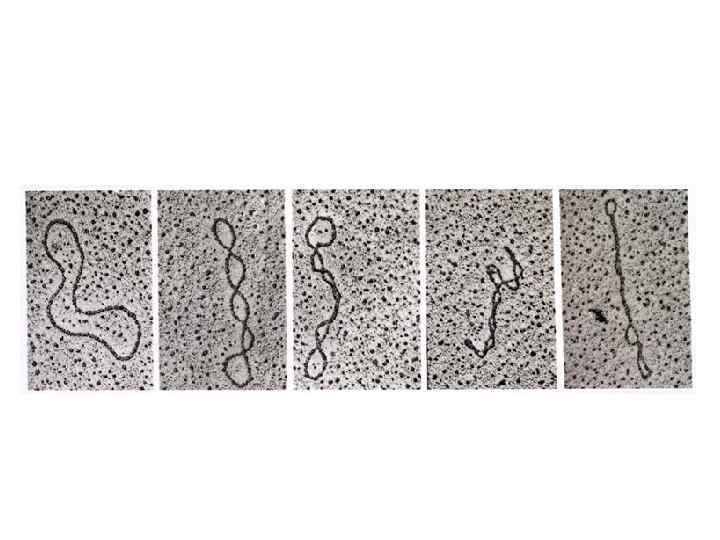
 DNA supercoiling L = linking number = число перекрещивания цепей (число раз, когда одна цепь пересекает другую) T = twist = число витков двойной спирали W = writhe = число супервитков L=T+W s = supercoiling density = (L – L 0) / L 0 = DL / L 0 обычно s ~ -0. 06 (меньше одного супервитка на 17 витков ДНК)
DNA supercoiling L = linking number = число перекрещивания цепей (число раз, когда одна цепь пересекает другую) T = twist = число витков двойной спирали W = writhe = число супервитков L=T+W s = supercoiling density = (L – L 0) / L 0 = DL / L 0 обычно s ~ -0. 06 (меньше одного супервитка на 17 витков ДНК)
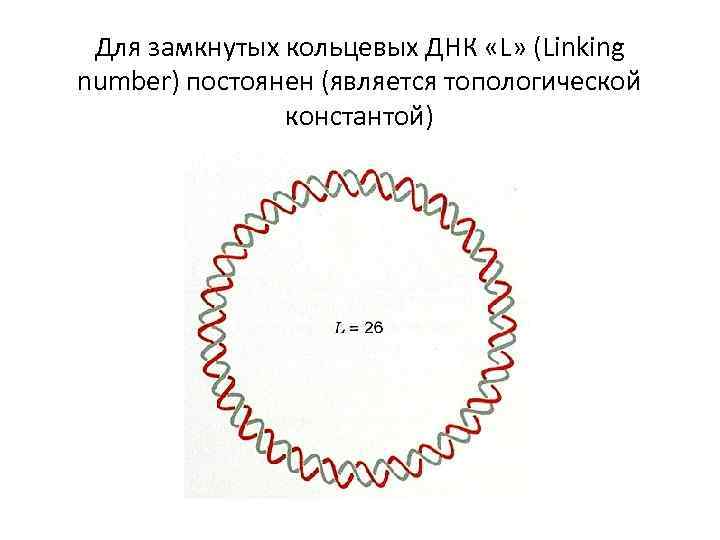 Для замкнутых кольцевых ДНК «L» (Linking number) постоянен (является топологической константой)
Для замкнутых кольцевых ДНК «L» (Linking number) постоянен (является топологической константой)
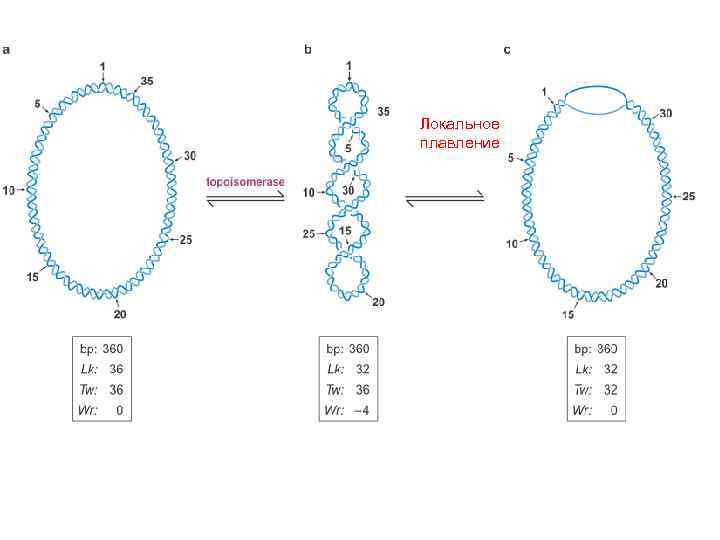 Локальное плавление
Локальное плавление
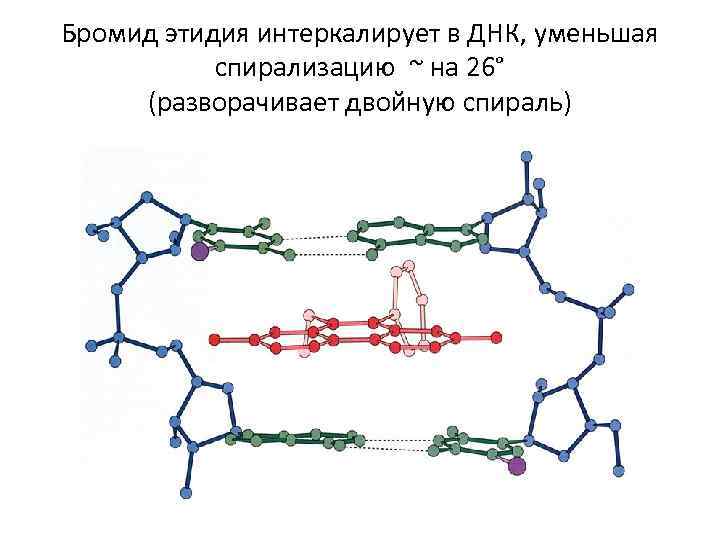 Бромид этидия интеркалирует в ДНК, уменьшая спирализацию ~ на 26° (разворачивает двойную спираль)
Бромид этидия интеркалирует в ДНК, уменьшая спирализацию ~ на 26° (разворачивает двойную спираль)
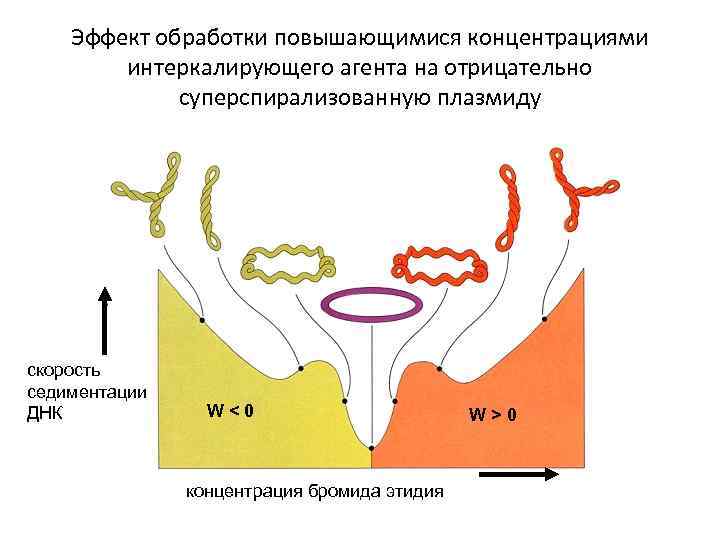 Эффект обработки повышающимися концентрациями интеркалирующего агента на отрицательно суперспирализованную плазмиду скорость седиментации ДНК W<0 концентрация бромида этидия W>0
Эффект обработки повышающимися концентрациями интеркалирующего агента на отрицательно суперспирализованную плазмиду скорость седиментации ДНК W<0 концентрация бромида этидия W>0
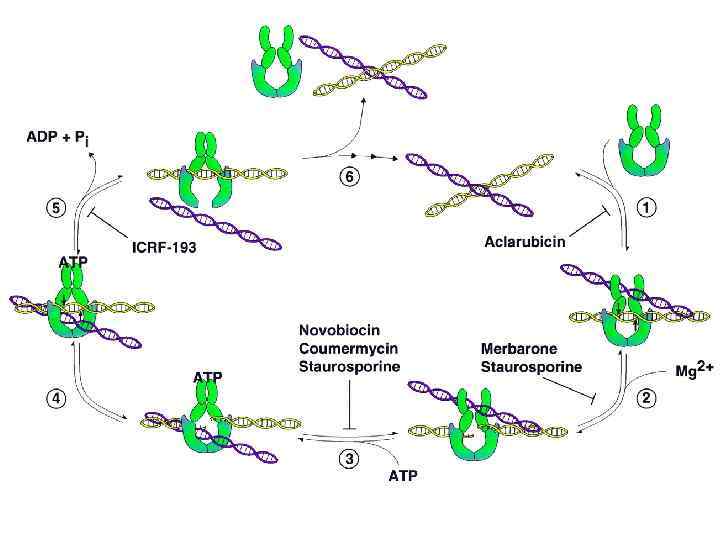
 Топоизомеразы регулируют топологию ДНК Топоизомераза I - вносит временные (eukaryotes, prokaryotes) однонитевые разрывы - релаксирует супервитки Топоизомераза II - вносит временные двунитевые (eukaryotes) разрывы - релаксирует супервитки, разделяет катенаны Гираза - генерирует негативные супервитки, используя энергию АТФ (prokaryotes) Обратная гираза (thermophiles) - генерирует позитивные супервитки
Топоизомеразы регулируют топологию ДНК Топоизомераза I - вносит временные (eukaryotes, prokaryotes) однонитевые разрывы - релаксирует супервитки Топоизомераза II - вносит временные двунитевые (eukaryotes) разрывы - релаксирует супервитки, разделяет катенаны Гираза - генерирует негативные супервитки, используя энергию АТФ (prokaryotes) Обратная гираза (thermophiles) - генерирует позитивные супервитки
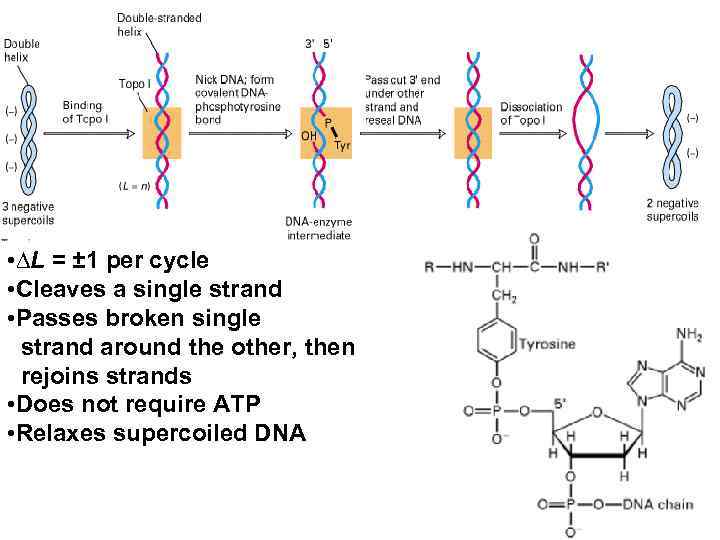 • ∆L = ± 1 per cycle • Cleaves a single strand • Passes broken single strand around the other, then rejoins strands • Does not require ATP • Relaxes supercoiled DNA
• ∆L = ± 1 per cycle • Cleaves a single strand • Passes broken single strand around the other, then rejoins strands • Does not require ATP • Relaxes supercoiled DNA
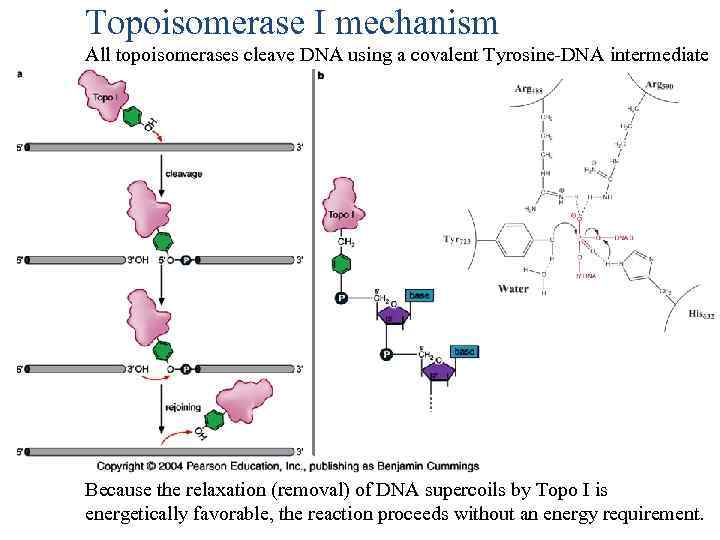 Topoisomerase I mechanism All topoisomerases cleave DNA using a covalent Tyrosine-DNA intermediate Because the relaxation (removal) of DNA supercoils by Topo I is energetically favorable, the reaction proceeds without an energy requirement.
Topoisomerase I mechanism All topoisomerases cleave DNA using a covalent Tyrosine-DNA intermediate Because the relaxation (removal) of DNA supercoils by Topo I is energetically favorable, the reaction proceeds without an energy requirement.
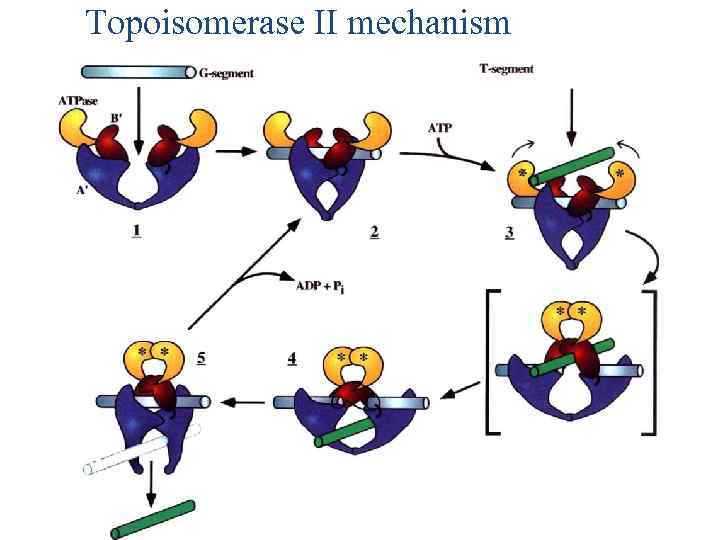 Topoisomerase II mechanism
Topoisomerase II mechanism
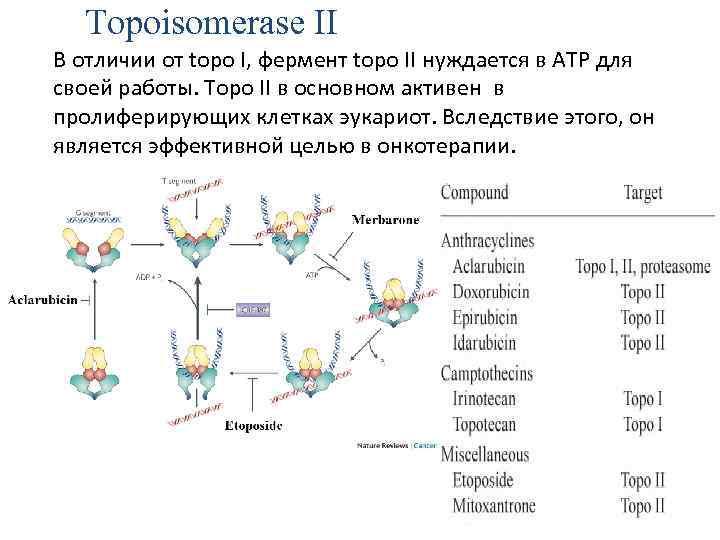 Topoisomerase II В отличии от topo I, фермент topo II нуждается в ATP для своей работы. Topo II в основном активен в пролиферирующих клетках эукариот. Вследствие этого, он является эффективной целью в онкотерапии.
Topoisomerase II В отличии от topo I, фермент topo II нуждается в ATP для своей работы. Topo II в основном активен в пролиферирующих клетках эукариот. Вследствие этого, он является эффективной целью в онкотерапии.
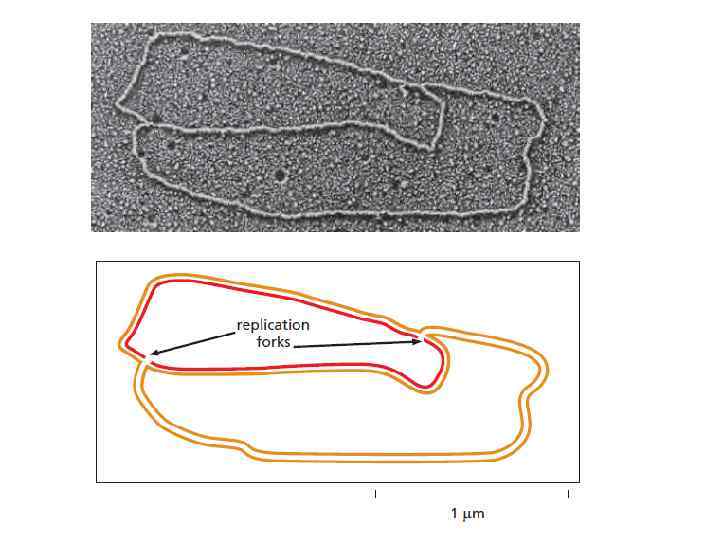
 Репликация ДНК 1. Репликация ДНК осуществляется согласно полуконсервативной модели 2. Репликация ДНК происходит двунаправленно
Репликация ДНК 1. Репликация ДНК осуществляется согласно полуконсервативной модели 2. Репликация ДНК происходит двунаправленно
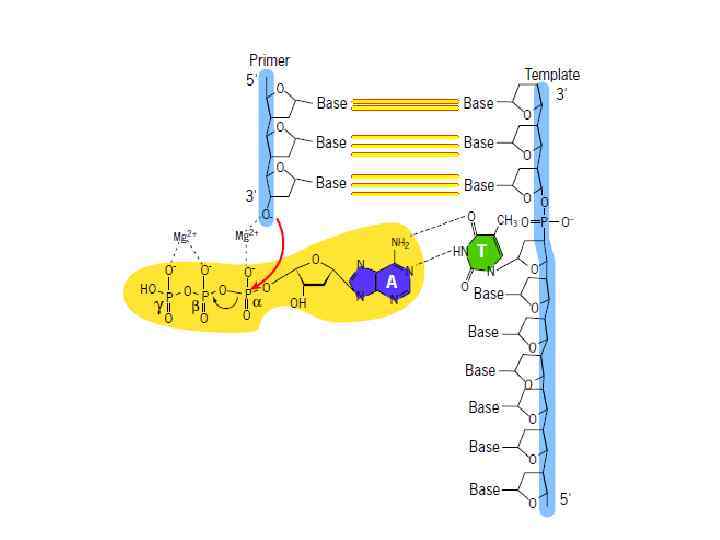
 DNA Polymerase-palm domain Содержит два активных центра, один осуществляет полимеризацию, другой – удаление ошибочно включенных нуклеотидов. Полимеразный домен осуществляет контроль правильности спаривания посредством контактов с основаниями в области малой бороздки
DNA Polymerase-palm domain Содержит два активных центра, один осуществляет полимеризацию, другой – удаление ошибочно включенных нуклеотидов. Полимеразный домен осуществляет контроль правильности спаривания посредством контактов с основаниями в области малой бороздки
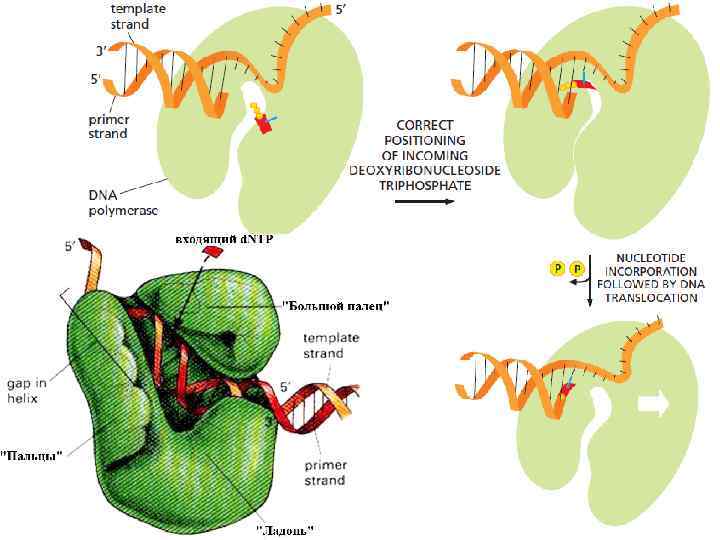
 DNA Polymerase-thumb domain Прямо не участвует в катализе Взаимодействует с матрицей и синтезирующейся ДНК с тем, чтобы обеспечить правильную позицию праймера относительно активного центра, одновременно препятствуя «соскакиванию» полимеразы с ДНК
DNA Polymerase-thumb domain Прямо не участвует в катализе Взаимодействует с матрицей и синтезирующейся ДНК с тем, чтобы обеспечить правильную позицию праймера относительно активного центра, одновременно препятствуя «соскакиванию» полимеразы с ДНК
 DNA Polymerase-finger domain Связывает d. NTP, и помещает правильно спаренные d. NTP в каталитический центр Связывает матрицу и обеспечивает экспонирование основания, с которым должен спариваться очередной d. NTP
DNA Polymerase-finger domain Связывает d. NTP, и помещает правильно спаренные d. NTP в каталитический центр Связывает матрицу и обеспечивает экспонирование основания, с которым должен спариваться очередной d. NTP
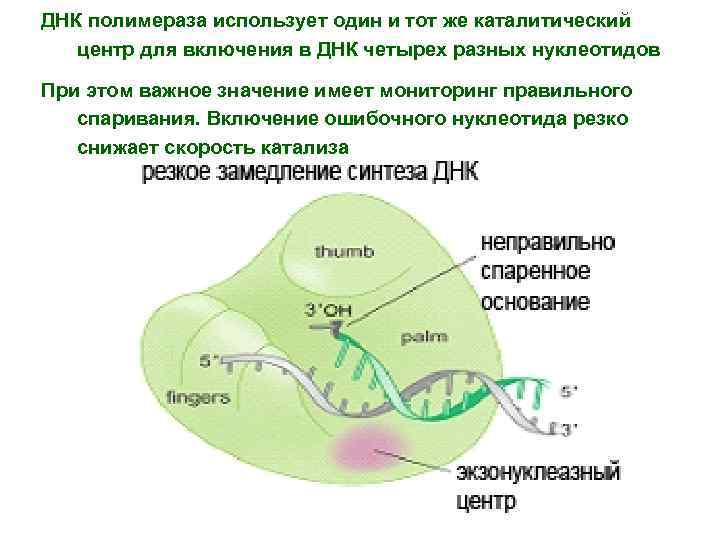 ДНК полимераза использует один и тот же каталитический центр для включения в ДНК четырех разных нуклеотидов При этом важное значение имеет мониторинг правильного спаривания. Включение ошибочного нуклеотида резко снижает скорость катализа
ДНК полимераза использует один и тот же каталитический центр для включения в ДНК четырех разных нуклеотидов При этом важное значение имеет мониторинг правильного спаривания. Включение ошибочного нуклеотида резко снижает скорость катализа
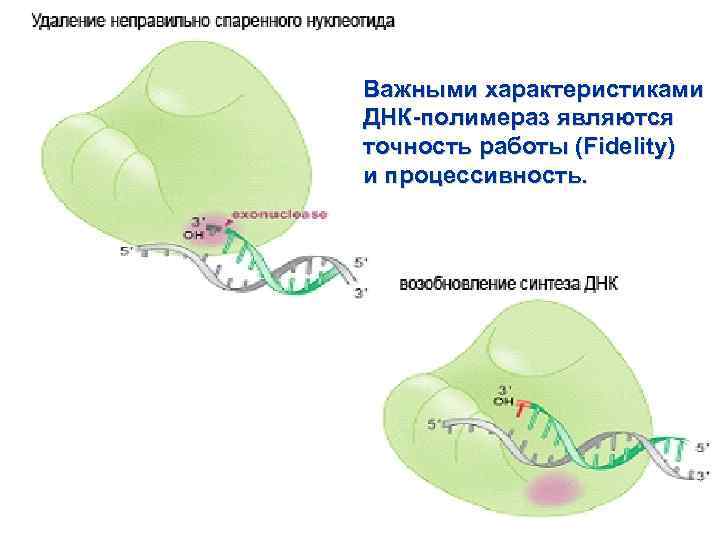 Важными характеристиками ДНК-полимераз являются точность работы (Fidelity) и процессивность.
Важными характеристиками ДНК-полимераз являются точность работы (Fidelity) и процессивность.
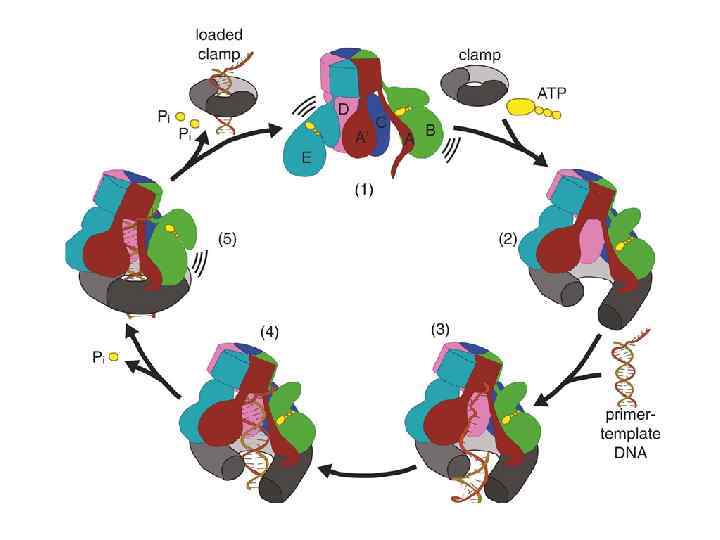
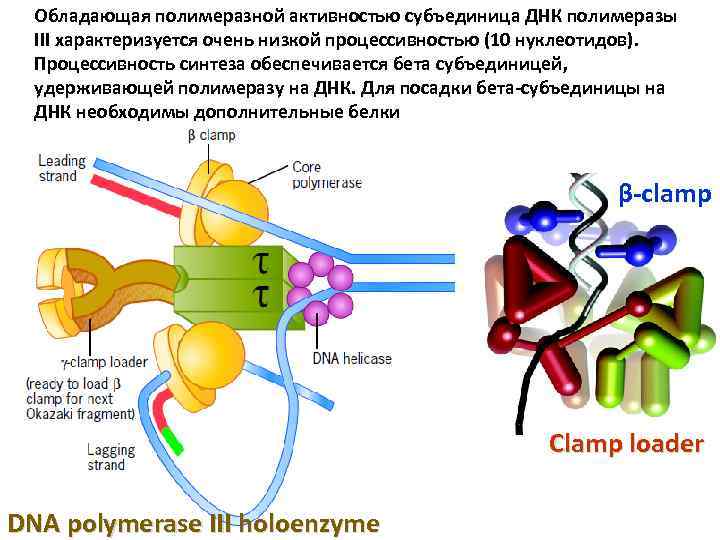 Обладающая полимеразной активностью субъединица ДНК полимеразы III характеризуется очень низкой процессивностью (10 нуклеотидов). Процессивность синтеза обеспечивается бета субъединицей, удерживающей полимеразу на ДНК. Для посадки бета-субъединицы на ДНК необходимы дополнительные белки β-clamp Clamp loader DNA polymerase III holoenzyme
Обладающая полимеразной активностью субъединица ДНК полимеразы III характеризуется очень низкой процессивностью (10 нуклеотидов). Процессивность синтеза обеспечивается бета субъединицей, удерживающей полимеразу на ДНК. Для посадки бета-субъединицы на ДНК необходимы дополнительные белки β-clamp Clamp loader DNA polymerase III holoenzyme
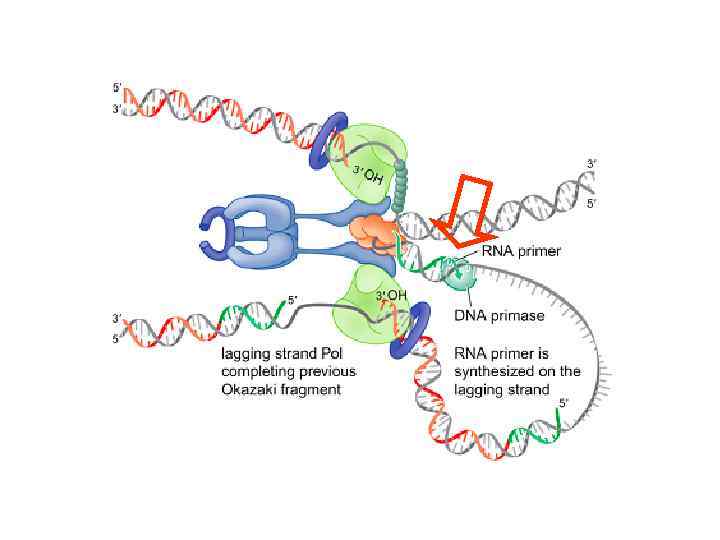
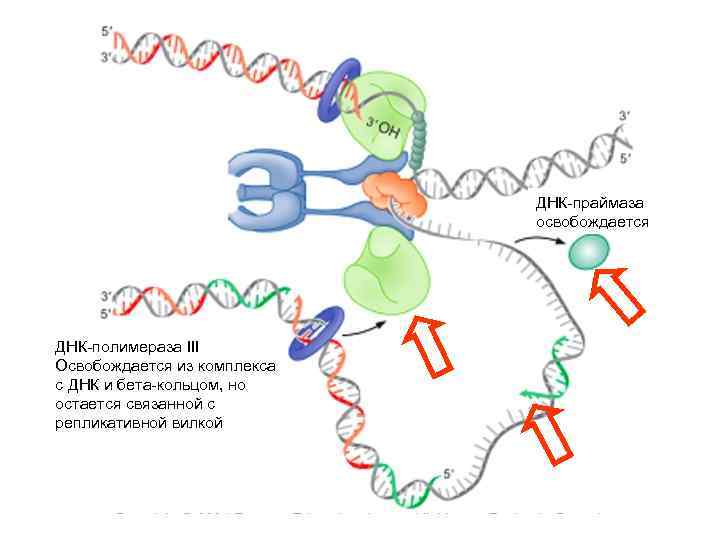 ДНК-праймаза освобождается ДНК-полимераза III Освобождается из комплекса с ДНК и бета-кольцом, но остается связанной с репликативной вилкой
ДНК-праймаза освобождается ДНК-полимераза III Освобождается из комплекса с ДНК и бета-кольцом, но остается связанной с репликативной вилкой
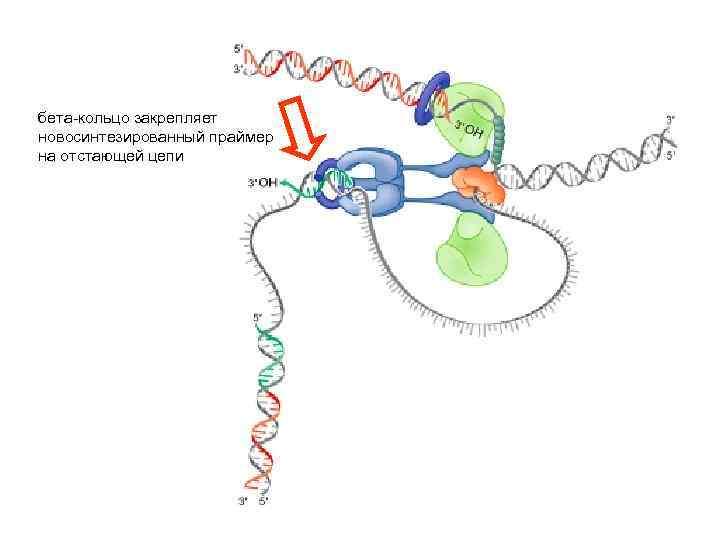 бета-кольцо закрепляет новосинтезированный праймер на отстающей цепи
бета-кольцо закрепляет новосинтезированный праймер на отстающей цепи
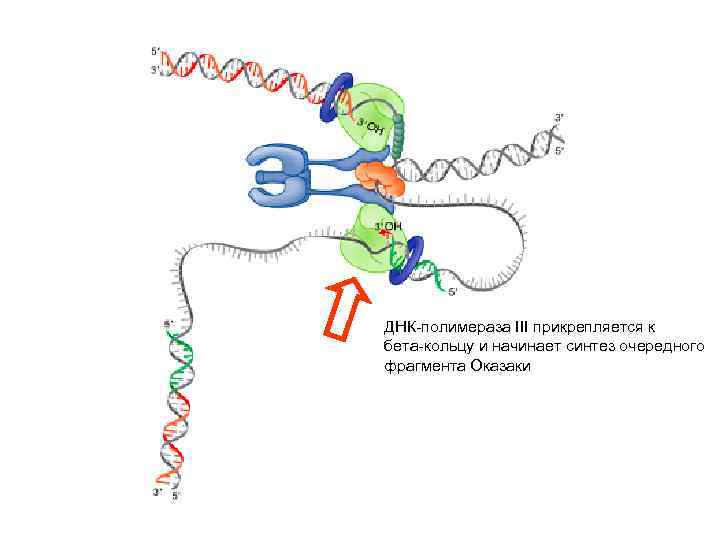 ДНК-полимераза III прикрепляется к бета-кольцу и начинает синтез очередного фрагмента Оказаки
ДНК-полимераза III прикрепляется к бета-кольцу и начинает синтез очередного фрагмента Оказаки
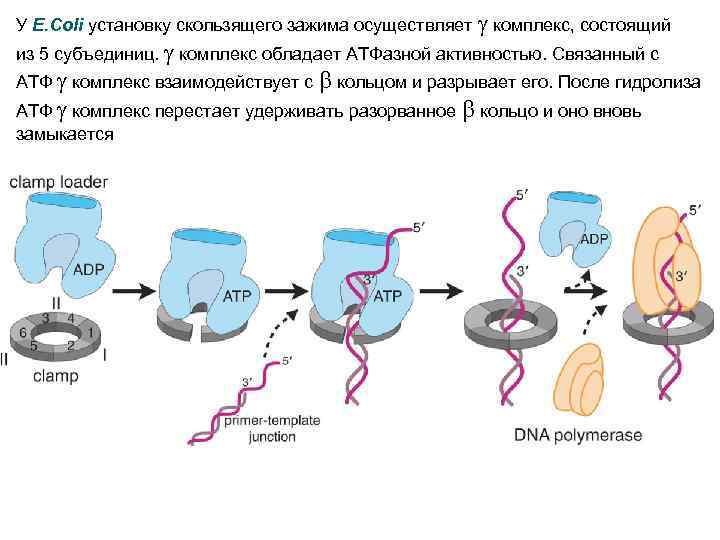 У E. Coli установку скользящего зажима осуществляет g комплекс, состоящий из 5 субъединиц. g комплекс обладает АТФазной активностью. Связанный с АТФ g комплекс взаимодействует с b кольцом и разрывает его. После гидролиза АТФ g комплекс перестает удерживать разорванное b кольцо и оно вновь замыкается
У E. Coli установку скользящего зажима осуществляет g комплекс, состоящий из 5 субъединиц. g комплекс обладает АТФазной активностью. Связанный с АТФ g комплекс взаимодействует с b кольцом и разрывает его. После гидролиза АТФ g комплекс перестает удерживать разорванное b кольцо и оно вновь замыкается
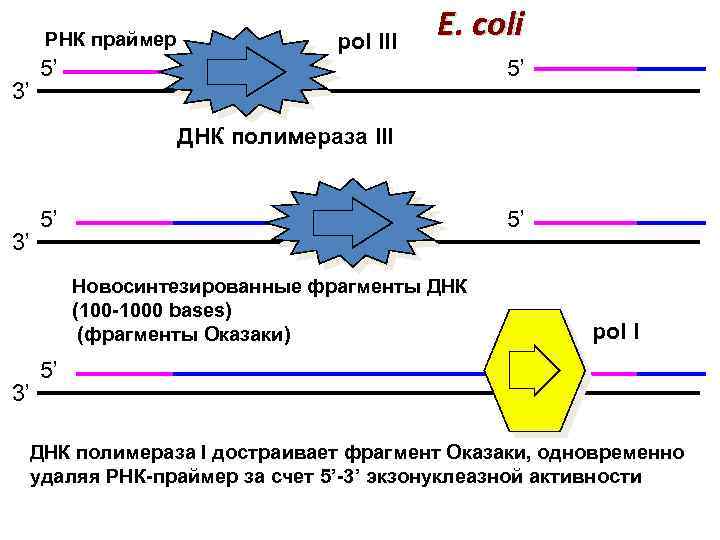 РНК праймер 3’ pol III E. coli 5’ 5’ ДНК полимераза III 3’ 5’ 5’ Новосинтезированные фрагменты ДНК (100 -1000 bases) (фрагменты Оказаки) 3’ pol I 5’ ДНК полимераза I достраивает фрагмент Оказаки, одновременно удаляя РНК-праймер за счет 5’-3’ экзонуклеазной активности
РНК праймер 3’ pol III E. coli 5’ 5’ ДНК полимераза III 3’ 5’ 5’ Новосинтезированные фрагменты ДНК (100 -1000 bases) (фрагменты Оказаки) 3’ pol I 5’ ДНК полимераза I достраивает фрагмент Оказаки, одновременно удаляя РНК-праймер за счет 5’-3’ экзонуклеазной активности
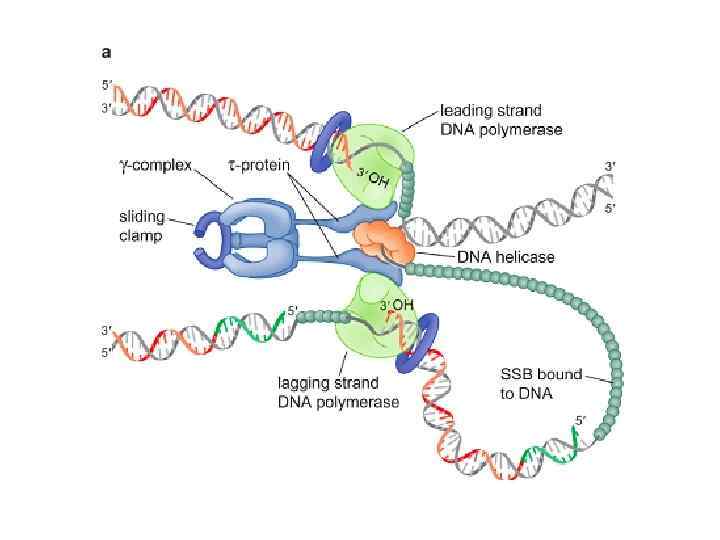
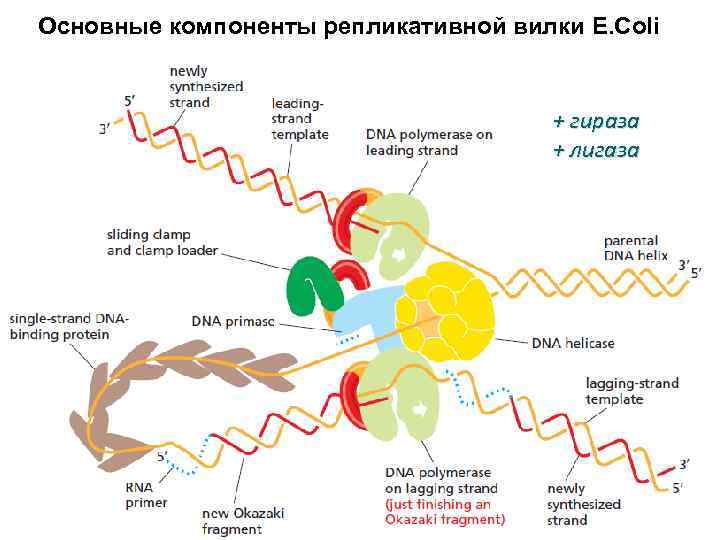 Основные компоненты репликативной вилки E. Coli + гираза + лигаза
Основные компоненты репликативной вилки E. Coli + гираза + лигаза
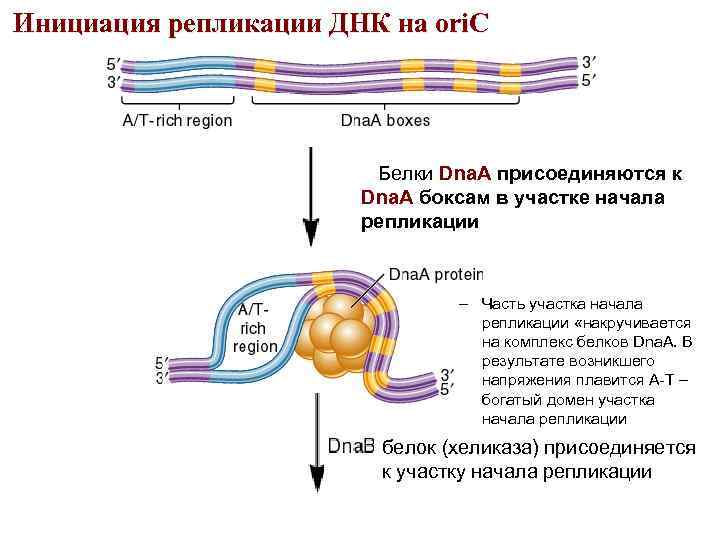 Инициация репликации ДНК на ori. C Белки Dna. A присоединяются к Dna. A боксам в участке начала репликации – Часть участка начала репликации «накручивается на комплекс белков Dna. A. В результате возникшего напряжения плавится А-Т – богатый домен участка начала репликации белок (хеликаза) присоединяется к участку начала репликации
Инициация репликации ДНК на ori. C Белки Dna. A присоединяются к Dna. A боксам в участке начала репликации – Часть участка начала репликации «накручивается на комплекс белков Dna. A. В результате возникшего напряжения плавится А-Т – богатый домен участка начала репликации белок (хеликаза) присоединяется к участку начала репликации
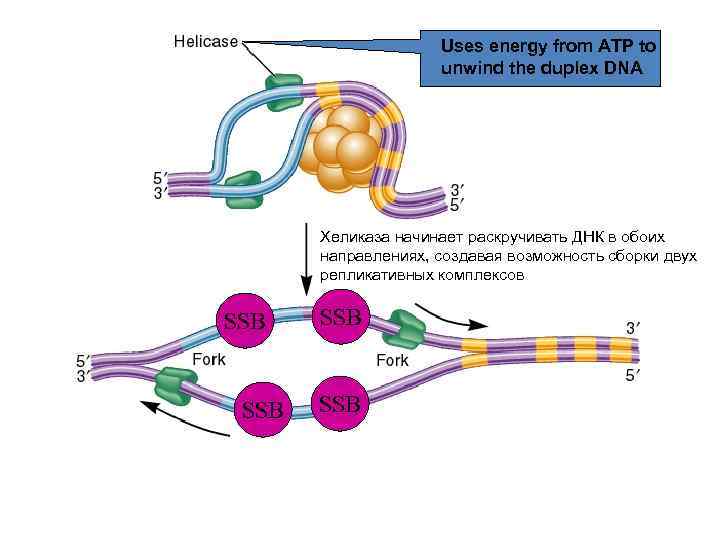 Uses energy from ATP to unwind the duplex DNA Хеликаза начинает раскручивать ДНК в обоих направлениях, создавая возможность сборки двух репликативных комплексов SSB SSB
Uses energy from ATP to unwind the duplex DNA Хеликаза начинает раскручивать ДНК в обоих направлениях, создавая возможность сборки двух репликативных комплексов SSB SSB
 Репликация ДНК у эукариот Общие черты репликации ДНК у прокариот и эукариот 1. Полуконсервативный синтез ДНК 2. Двунаправленный синтез ДНК 3 Синтез в направлении 5’-3’. Соответственно, хотя бы одна цепь синтезируется в виде фрагментов Оказаки 4. Инициация синтеза ДНК посредством РНК-праймеров
Репликация ДНК у эукариот Общие черты репликации ДНК у прокариот и эукариот 1. Полуконсервативный синтез ДНК 2. Двунаправленный синтез ДНК 3 Синтез в направлении 5’-3’. Соответственно, хотя бы одна цепь синтезируется в виде фрагментов Оказаки 4. Инициация синтеза ДНК посредством РНК-праймеров
 Pol - PRIMASE DNA Pol (I) (primase complex) - bifunctional Heterotetrameric phosphoprotein 48 k. Da PRIMASE- initiates RNA-primer synthesis (makes complementary RNA primer) 58 k. Da protein- tethers primase to 180 k. Da subunit 180 k. Da polymerase A subunit- extends RNA primer 70 k. Da subunit- no known catalytic function (may recruit pol : primase to the replication fork)
Pol - PRIMASE DNA Pol (I) (primase complex) - bifunctional Heterotetrameric phosphoprotein 48 k. Da PRIMASE- initiates RNA-primer synthesis (makes complementary RNA primer) 58 k. Da protein- tethers primase to 180 k. Da subunit 180 k. Da polymerase A subunit- extends RNA primer 70 k. Da subunit- no known catalytic function (may recruit pol : primase to the replication fork)
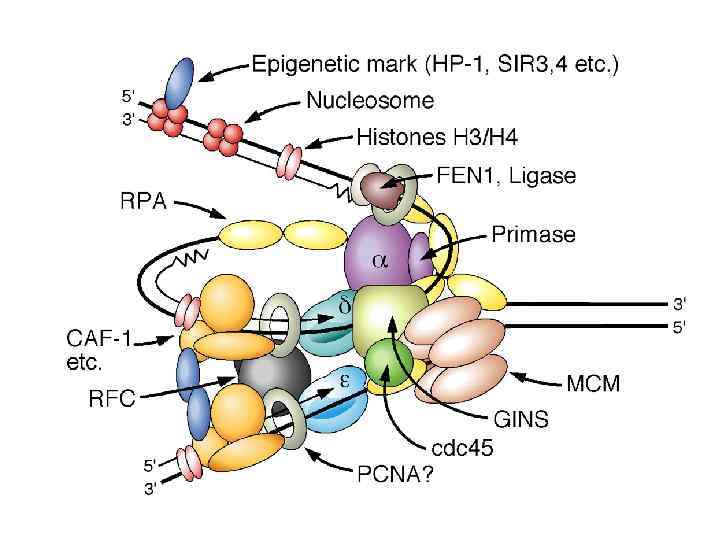
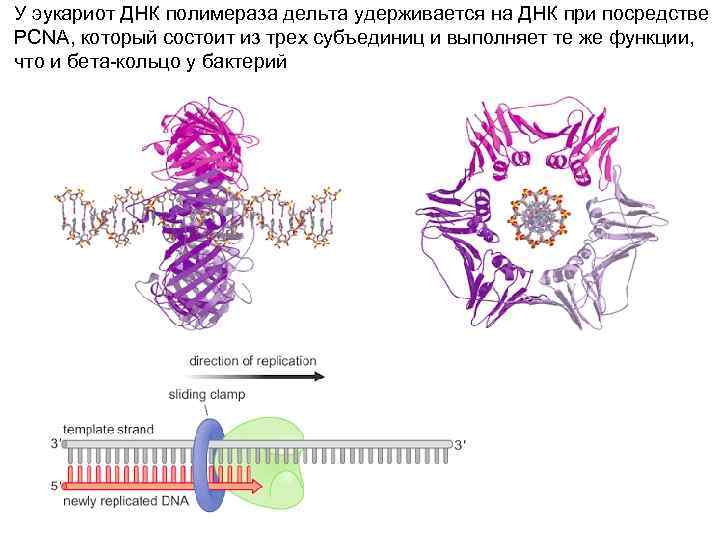 У эукариот ДНК полимераза дельта удерживается на ДНК при посредстве PCNA, который состоит из трех субъединиц и выполняет те же функции, что и бета-кольцо у бактерий
У эукариот ДНК полимераза дельта удерживается на ДНК при посредстве PCNA, который состоит из трех субъединиц и выполняет те же функции, что и бета-кольцо у бактерий
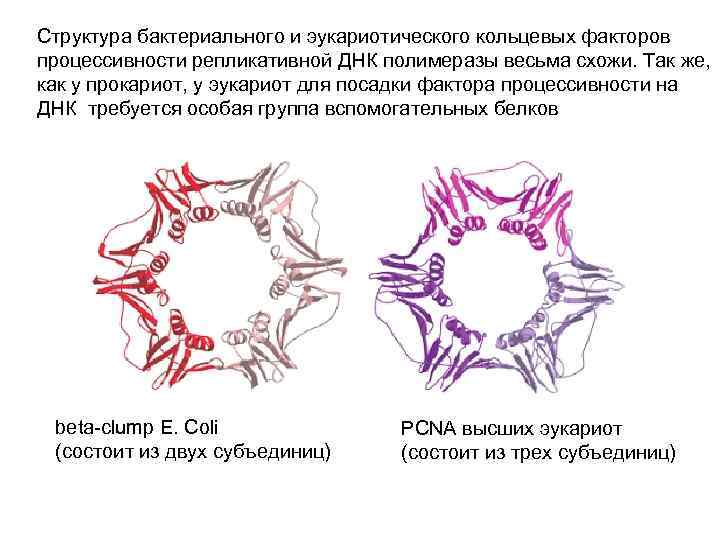 Структура бактериального и эукариотического кольцевых факторов процессивности репликативной ДНК полимеразы весьма схожи. Так же, как у прокариот, у эукариот для посадки фактора процессивности на ДНК требуется особая группа вспомогательных белков beta-clump E. Coli (состоит из двух субъединиц) PCNA высших эукариот (состоит из трех субъединиц)
Структура бактериального и эукариотического кольцевых факторов процессивности репликативной ДНК полимеразы весьма схожи. Так же, как у прокариот, у эукариот для посадки фактора процессивности на ДНК требуется особая группа вспомогательных белков beta-clump E. Coli (состоит из двух субъединиц) PCNA высших эукариот (состоит из трех субъединиц)
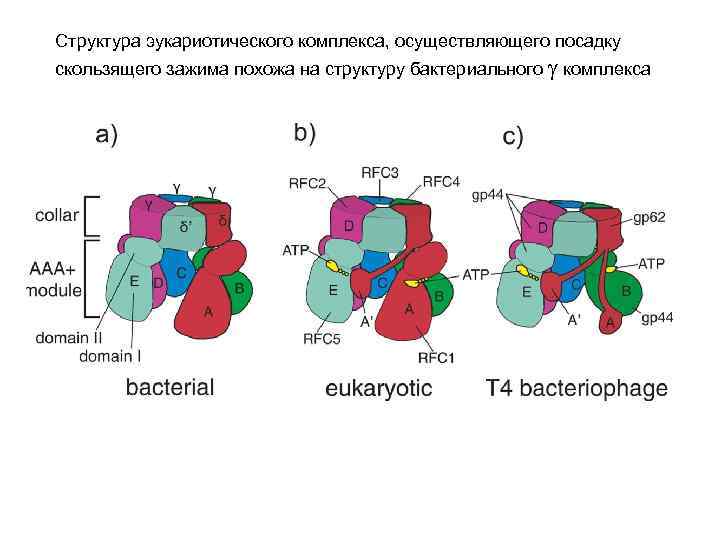 Структура эукариотического комплекса, осуществляющего посадку скользящего зажима похожа на структуру бактериального g комплекса
Структура эукариотического комплекса, осуществляющего посадку скользящего зажима похожа на структуру бактериального g комплекса
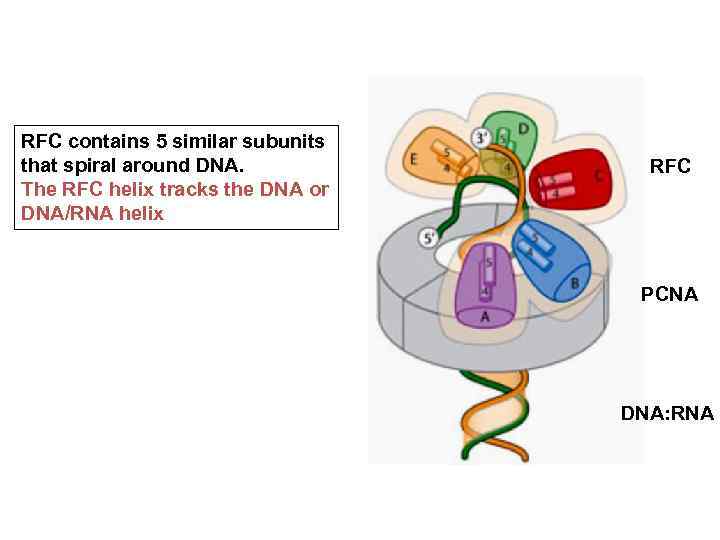 RFC contains 5 similar subunits that spiral around DNA. The RFC helix tracks the DNA or DNA/RNA helix RFC PCNA DNA: RNA
RFC contains 5 similar subunits that spiral around DNA. The RFC helix tracks the DNA or DNA/RNA helix RFC PCNA DNA: RNA
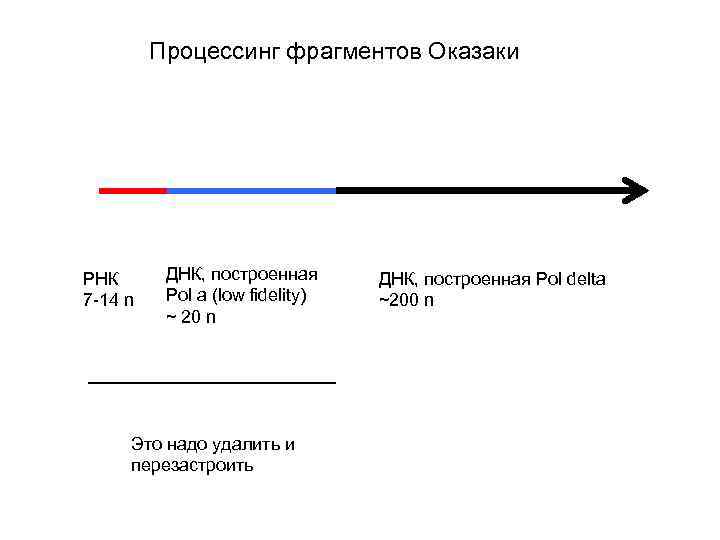 Процессинг фрагментов Оказаки РНК 7 -14 n ДНК, построенная Pol a (low fidelity) ~ 20 n Это надо удалить и перезастроить ДНК, построенная Pol delta ~200 n
Процессинг фрагментов Оказаки РНК 7 -14 n ДНК, построенная Pol a (low fidelity) ~ 20 n Это надо удалить и перезастроить ДНК, построенная Pol delta ~200 n
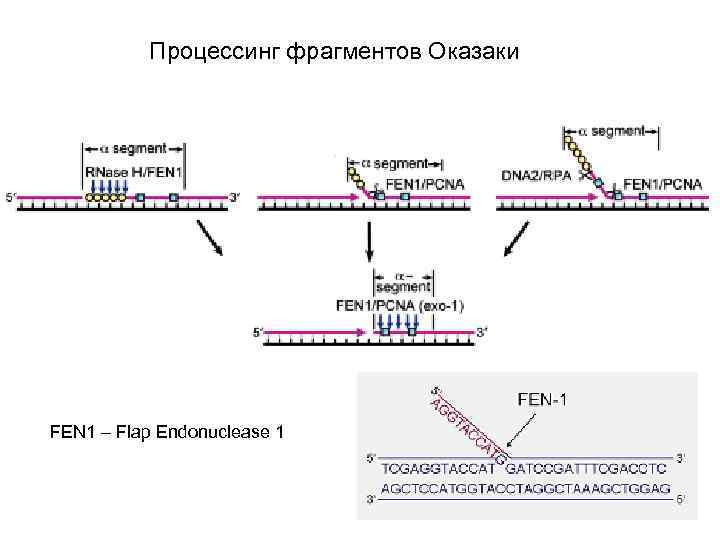 Процессинг фрагментов Оказаки FEN 1 – Flap Endonuclease 1
Процессинг фрагментов Оказаки FEN 1 – Flap Endonuclease 1
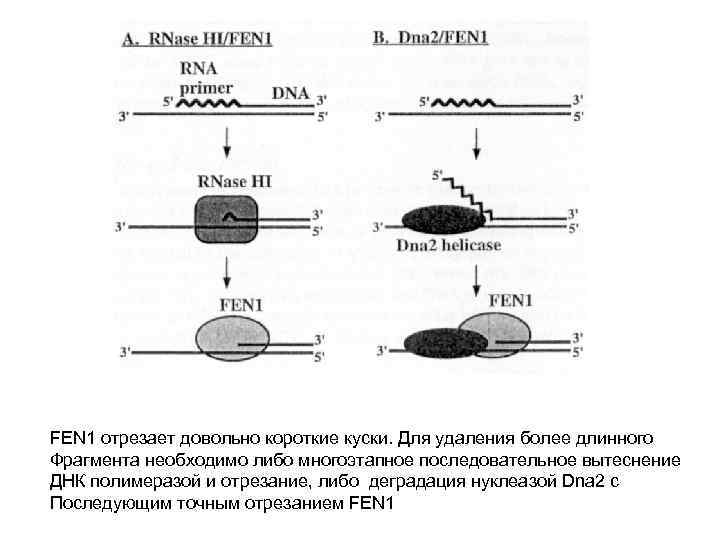 FEN 1 отрезает довольно короткие куски. Для удаления более длинного Фрагмента необходимо либо многоэтапное последовательное вытеснение ДНК полимеразой и отрезание, либо деградация нуклеазой Dna 2 с Последующим точным отрезанием FEN 1
FEN 1 отрезает довольно короткие куски. Для удаления более длинного Фрагмента необходимо либо многоэтапное последовательное вытеснение ДНК полимеразой и отрезание, либо деградация нуклеазой Dna 2 с Последующим точным отрезанием FEN 1
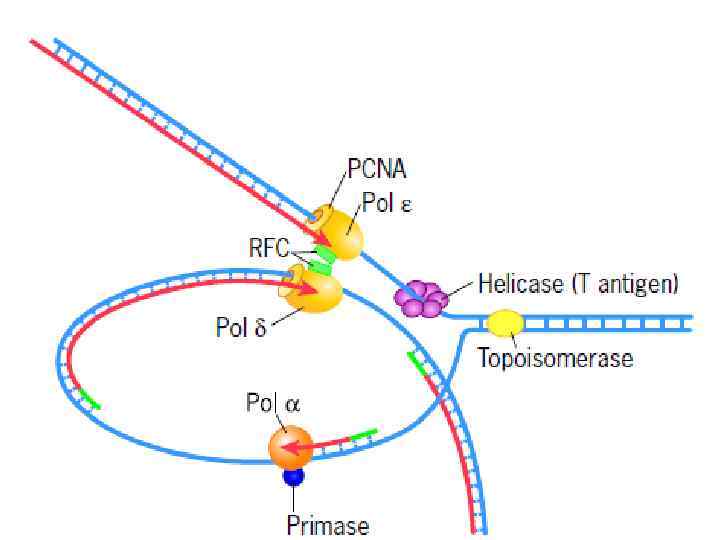
 Компоненты репликационной машины эукариот 1. DNA pol + primase: синтезирует праймеры для синтеза ведущей и отстающей цепей. 2. DNA pol : элонгация отстающей цепи DNA pol e : элонгация ведущей цепи 3. PCNA: (proliferating cell nuclear antigen) фактор процессивности pol 4. Факторы репликации C (RF-C; посадка зажима, ATPase) and RF-A (связывание однонитевой ДНК). 5. Topoisomerases I & II: регулируют топологию ДНК.
Компоненты репликационной машины эукариот 1. DNA pol + primase: синтезирует праймеры для синтеза ведущей и отстающей цепей. 2. DNA pol : элонгация отстающей цепи DNA pol e : элонгация ведущей цепи 3. PCNA: (proliferating cell nuclear antigen) фактор процессивности pol 4. Факторы репликации C (RF-C; посадка зажима, ATPase) and RF-A (связывание однонитевой ДНК). 5. Topoisomerases I & II: регулируют топологию ДНК.
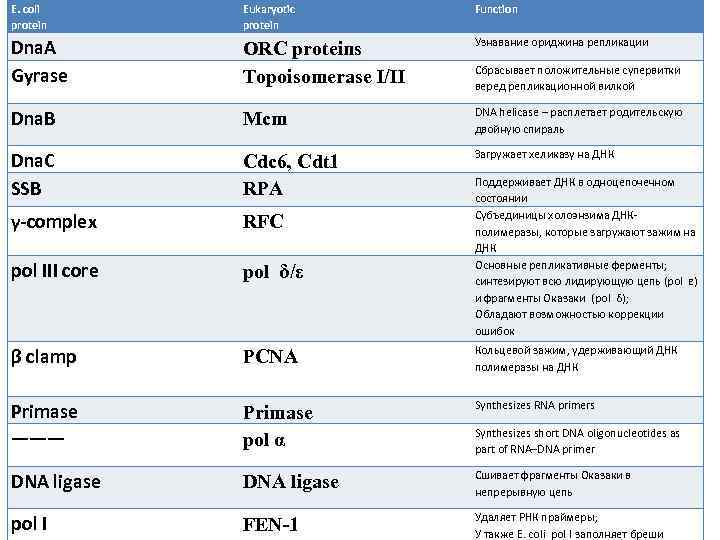 E. coli protein Eukaryotic protein Function Dna. A Gyrase ORC proteins Topoisomerase I/II Узнавание ориджина репликации Dna. B Mcm DNA helicase – расплетает родительскую двойную спираль Dna. C SSB Cdc 6, Cdt 1 RPA Загружает хеликазу на ДНК γ-complex RFC pol III core pol δ/ε β clamp Primase ——— PCNA Кольцевой зажим, удерживающий ДНК полимеразы на ДНК Primase pol α Synthesizes RNA primers DNA ligase Сшивает фрагменты Оказаки в непрерывную цепь pol I FEN-1 Удаляет РНК праймеры; У также E. coli pol I заполняет бреши Сбрасывает положительные супервитки веред репликационной вилкой Поддерживает ДНК в одноцепочечном состоянии Субъединицы холоэнзима ДНКполимеразы, которые загружают зажим на ДНК Основные репликативные ферменты; синтезируют всю лидирующую цепь (pol ε) и фрагменты Оказаки (pol δ); Обладают возможностью коррекции ошибок Synthesizes short DNA oligonucleotides as part of RNA–DNA primer
E. coli protein Eukaryotic protein Function Dna. A Gyrase ORC proteins Topoisomerase I/II Узнавание ориджина репликации Dna. B Mcm DNA helicase – расплетает родительскую двойную спираль Dna. C SSB Cdc 6, Cdt 1 RPA Загружает хеликазу на ДНК γ-complex RFC pol III core pol δ/ε β clamp Primase ——— PCNA Кольцевой зажим, удерживающий ДНК полимеразы на ДНК Primase pol α Synthesizes RNA primers DNA ligase Сшивает фрагменты Оказаки в непрерывную цепь pol I FEN-1 Удаляет РНК праймеры; У также E. coli pol I заполняет бреши Сбрасывает положительные супервитки веред репликационной вилкой Поддерживает ДНК в одноцепочечном состоянии Субъединицы холоэнзима ДНКполимеразы, которые загружают зажим на ДНК Основные репликативные ферменты; синтезируют всю лидирующую цепь (pol ε) и фрагменты Оказаки (pol δ); Обладают возможностью коррекции ошибок Synthesizes short DNA oligonucleotides as part of RNA–DNA primer
 Геном человека кодирует по меньшей мере 15 ДНК полимераз, которые по структурной гомологии можно подразделить на 4 группы: A (Pols γ, ν and θ (Theta)) B (Pols α, δ, ε and ζ) X (Pols β, λ, μ, and terminal deoxyribonucleotidyltransferase) Y (Pols η (eta), ι, κ (σ), and Rev 1) γ репликация митохондриальной ДНК θ (Theta) репликация через апуриновые и апиримидиновые сайты – включение G/C ζ (zeta) репликация через различные повреждения λ, μ участвуют в процессинге концов ДНК в ходе NHEJ η репликация через тимидиновые димеры и G-G димеры, сшитые цис-платином κ (Trf 4/Pol σ ) необходима для соединения (когезии) хроматид
Геном человека кодирует по меньшей мере 15 ДНК полимераз, которые по структурной гомологии можно подразделить на 4 группы: A (Pols γ, ν and θ (Theta)) B (Pols α, δ, ε and ζ) X (Pols β, λ, μ, and terminal deoxyribonucleotidyltransferase) Y (Pols η (eta), ι, κ (σ), and Rev 1) γ репликация митохондриальной ДНК θ (Theta) репликация через апуриновые и апиримидиновые сайты – включение G/C ζ (zeta) репликация через различные повреждения λ, μ участвуют в процессинге концов ДНК в ходе NHEJ η репликация через тимидиновые димеры и G-G димеры, сшитые цис-платином κ (Trf 4/Pol σ ) необходима для соединения (когезии) хроматид
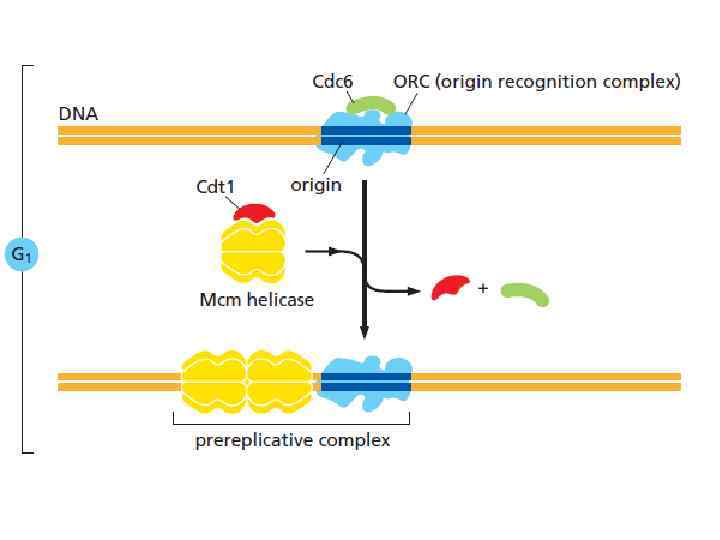
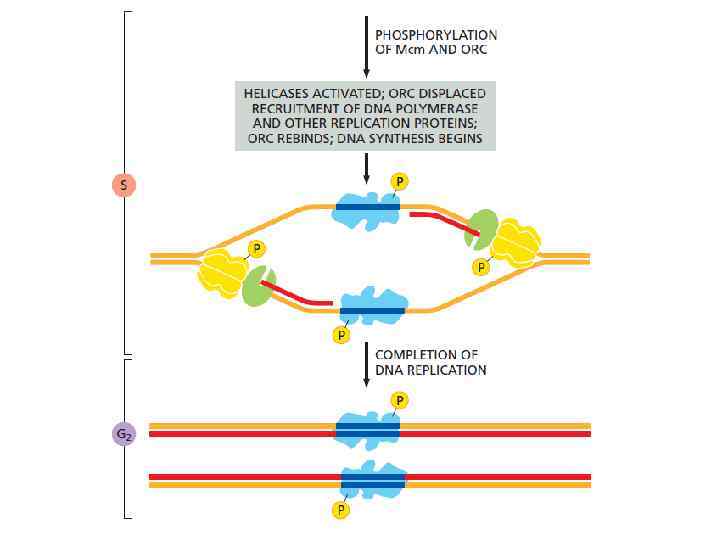
 Инициация репликации у эукариот
Инициация репликации у эукариот
 У млекопитающих размер генома составляет ~2 х10 9 п. н. средняя скорость движения репликативных вилок у эукариот составляет ~ 2 т. п. н. /мин (для сравнения, у E. Coli ~ 60 т. п. н. /мин Таким образом, для репликации всего генома человека одной парой вилок потребовалось бы 1000000 мин = 16600 часов В клетках эукариот одновременно активируется много участков начала репликации Сегмент хромосомы, реплицирующийся с одного участка начала репликации, называется репликоном
У млекопитающих размер генома составляет ~2 х10 9 п. н. средняя скорость движения репликативных вилок у эукариот составляет ~ 2 т. п. н. /мин (для сравнения, у E. Coli ~ 60 т. п. н. /мин Таким образом, для репликации всего генома человека одной парой вилок потребовалось бы 1000000 мин = 16600 часов В клетках эукариот одновременно активируется много участков начала репликации Сегмент хромосомы, реплицирующийся с одного участка начала репликации, называется репликоном
 Инициация репликации у эукариот Начинается ли репликация в строго определенных участках генома? Основные вопросы Существуют ли специфические последовательности ДНК, служащие местами инициации репликации? Какие белковые факторы участвуют в инициации репликации? Как именно происходит инициация репликации? (последовательность событий)
Инициация репликации у эукариот Начинается ли репликация в строго определенных участках генома? Основные вопросы Существуют ли специфические последовательности ДНК, служащие местами инициации репликации? Какие белковые факторы участвуют в инициации репликации? Как именно происходит инициация репликации? (последовательность событий)
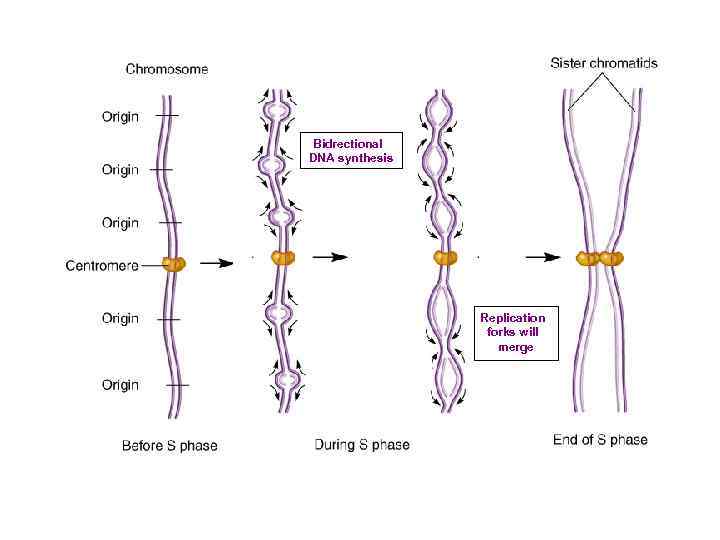 Bidrectional DNA synthesis Replication forks will merge
Bidrectional DNA synthesis Replication forks will merge
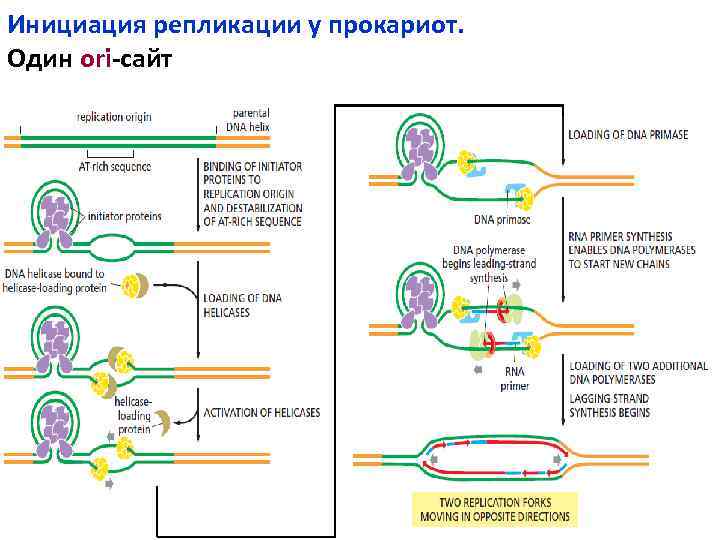 Инициация репликации у прокариот. Один ori-сайт
Инициация репликации у прокариот. Один ori-сайт
 В клетках высших эукариот инициация репликации может происходить где угодно. В эмбриональных клетках (до стадии средней бластулы) и в экстрактах из клеток ксенопуса инициация происходит через короткие интервалы (на молекуле ДНК) и без всякой специфичности в отношении последовательностей. В неэмбриональных клетках сушествуют предпочтительные места инициации, которые могут быть достаточно короткими (репликатор домена бета-глобиновых генов) и достаточно протяженными (зона инициации репликации локуса DHFR) Короткие репликаторы характеризуются наличием ряда необходимых элементов (АТ-богатые последовательности, полупуриновые/полипиримидиновые блоки, изогнутые последовательности ДНК). Набор и сочетание этих элементов могут быть разными в разных репликаторах.
В клетках высших эукариот инициация репликации может происходить где угодно. В эмбриональных клетках (до стадии средней бластулы) и в экстрактах из клеток ксенопуса инициация происходит через короткие интервалы (на молекуле ДНК) и без всякой специфичности в отношении последовательностей. В неэмбриональных клетках сушествуют предпочтительные места инициации, которые могут быть достаточно короткими (репликатор домена бета-глобиновых генов) и достаточно протяженными (зона инициации репликации локуса DHFR) Короткие репликаторы характеризуются наличием ряда необходимых элементов (АТ-богатые последовательности, полупуриновые/полипиримидиновые блоки, изогнутые последовательности ДНК). Набор и сочетание этих элементов могут быть разными в разных репликаторах.
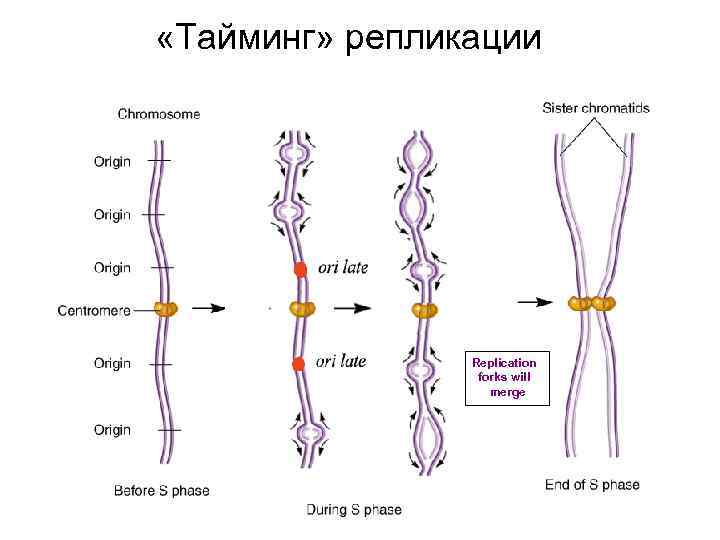 «Тайминг» репликации Replication forks will merge
«Тайминг» репликации Replication forks will merge
 активные гены обычно являются раннереплицирующимися (реплицируются в начале S фазы) молчащие гены обычно являются позднереплицирующимися (реплицируются в конце S фазы)
активные гены обычно являются раннереплицирующимися (реплицируются в начале S фазы) молчащие гены обычно являются позднереплицирующимися (реплицируются в конце S фазы)
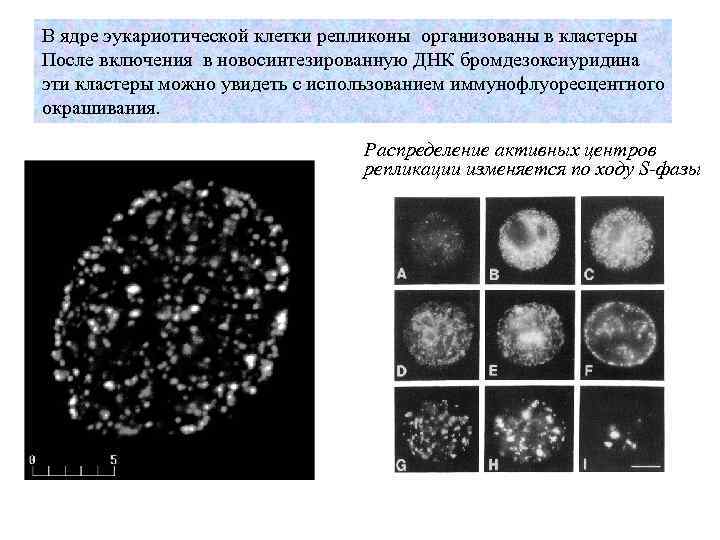 В ядре эукариотической клетки репликоны организованы в кластеры После включения в новосинтезированную ДНК бромдезоксиуридина эти кластеры можно увидеть с использованием иммунофлуоресцентного окрашивания. Распределение активных центров репликации изменяется по ходу S-фазы
В ядре эукариотической клетки репликоны организованы в кластеры После включения в новосинтезированную ДНК бромдезоксиуридина эти кластеры можно увидеть с использованием иммунофлуоресцентного окрашивания. Распределение активных центров репликации изменяется по ходу S-фазы
 В начале S-фазы в клетках присутствует несколько сотен фокусов репликации
В начале S-фазы в клетках присутствует несколько сотен фокусов репликации
 * Хромосомы построены из раннереплицирующихся и позднереплицирующихся областей * Минимальный размер раннереплицирующихся и позднереплицирующихся областей составляет 400 -800 Kb, хотя есть и исключения * Раннереплицирующиеся области предпочтительно содержат активные гены и соответствуют R-бэндам, выявляемых при окраске хромосом по Гимза * Позднереплицирующиеся области содержат преимущественно неактивные гены и соответствуют G-бэндам * Разделение хромосом на раннереплицирующиеся области и поздереплицирующиеся области эволюционно консервативно. (хромосомный пэйнтинг)
* Хромосомы построены из раннереплицирующихся и позднереплицирующихся областей * Минимальный размер раннереплицирующихся и позднереплицирующихся областей составляет 400 -800 Kb, хотя есть и исключения * Раннереплицирующиеся области предпочтительно содержат активные гены и соответствуют R-бэндам, выявляемых при окраске хромосом по Гимза * Позднереплицирующиеся области содержат преимущественно неактивные гены и соответствуют G-бэндам * Разделение хромосом на раннереплицирующиеся области и поздереплицирующиеся области эволюционно консервативно. (хромосомный пэйнтинг)
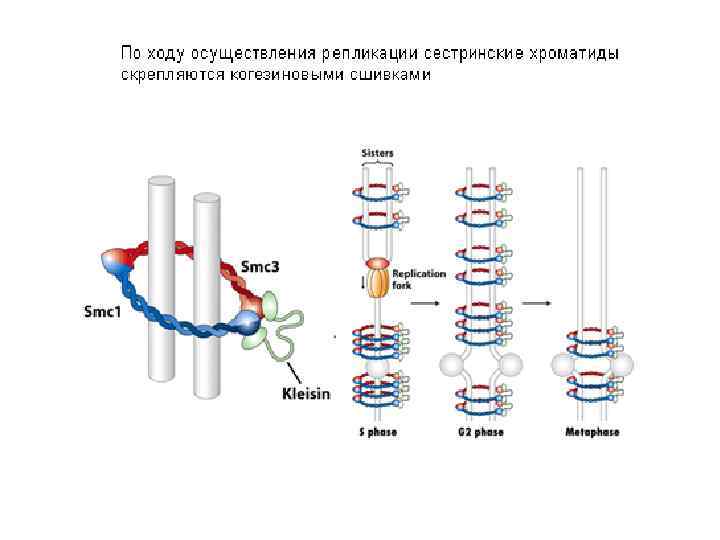
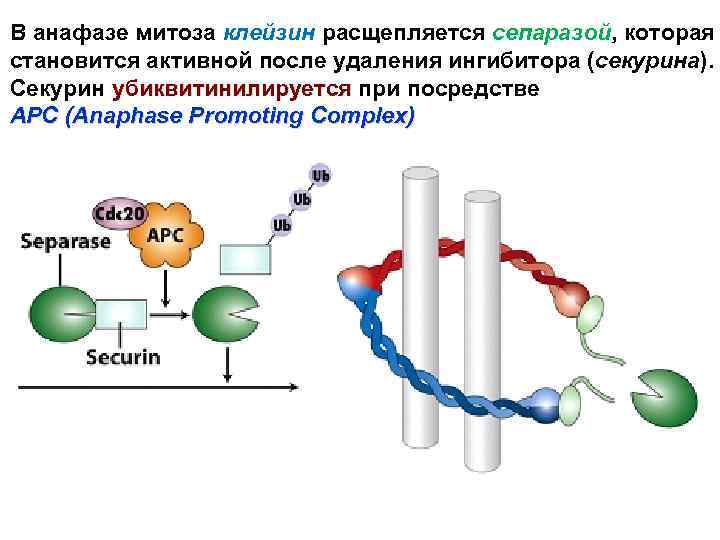 В анафазе митоза клейзин расщепляется сепаразой, которая становится активной после удаления ингибитора (секурина). Секурин убиквитинилируется при посредстве APC (Anaphase Promoting Complex)
В анафазе митоза клейзин расщепляется сепаразой, которая становится активной после удаления ингибитора (секурина). Секурин убиквитинилируется при посредстве APC (Anaphase Promoting Complex)


