1f54e10bfff835290ce07a6b2f513e5c.ppt
- Количество слайдов: 110

Introducció a la Bioinformàtica: la recerca biomèdica in silico © 2006 Plataforma Bioinformàtica de la UAB
Alineamiento de secuencias © 2006 Plataforma Bioinformàtica de la UAB

Alineamiento de secuencias Alineamiento Comparar dos (alineación a pares) o más (alineación múltiple) secuencias para buscar una serie de caracteres o patrón de caracteres que están en el mismo orden en las secuencias Secuencia 1: ATGCGACTGACG |||||| Secuencia 2: ATGCGACTGACG Significado de un alineamiento Estadístico Por azar (alineamiento de secuencias al azar pueden producir un 20% identidad) Biológico Comparten un ancestro común. Implica una información funcional, estructural y evolutiva? © 2006 Plataforma Bioinformàtica de la UAB

Alineamiento de secuencias Un alineamiento compara permitiendo: • Identificar genes homólogos/similares • Asignar funciones biológicas (reales, posibles? ) • Predecir estructura • Encontrar patrones • Reconstruir relaciones evolutivas • …… © 2006 Plataforma Bioinformàtica de la UAB

Homología vs similitud Homólogo, similar, idéntico • Homología – dos secuencias son homólogas sólo si derivan de una ancestro común – implica una herencia compartida – cualitativo – se es homólogo o no se es • Similitud – medida cuantitativa – se puede usar una medida de similitud para inferir homología © 2006 Plataforma Bioinformàtica de la UAB

Global vs Local Global: Intentamos alinear todos los caracteres de las secuencias optimizando el número máximo de identidades Local: Alineamos segmentos de las secuencias donde la densidad de identidades es mayor, generamos subalinemientos © 2006 Plataforma Bioinformàtica de la UAB
Alineamiento de secuencias Un proceso de alineamiento debe efectuar una búsqueda activa del mejor alineamiento posible y debe considerar los cambios que sufren las secuencias: • Identidades -> Emparejamientos (match) • Sustituciones -> Desemparejamientos (mismatch) • Deleciones e inserciones (indel) -> Huecos (gaps) © 2006 Plataforma Bioinformàtica de la UAB

Alineamiento de secuencias Cómo decidir cuál es el mejor? • Respuesta: el más significativo desde el punto de vista biológico • Pero: necesitamos una medida objetiva • Sistemas de puntuación (scoring) – reglas para asignar puntos – el más simple: match, mismatch, gap © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero

Valoración de un alineamiento Valoración (score) de un alineamiento Máxima puntuación = Alineamiento óptimo Puntuación = F (puntuación emparejamiento idéntico, puntuación emparejamiento similar, puntuación huecos –gap-) © 2006 Plataforma Bioinformàtica de la UAB

Valoración de un alineamiento Como valorar un alineamiento? Ejemplo Identidad = 1 Missmatch = 0 Gap = -1 Score = 10 - 4 = 6 © 2006 Plataforma Bioinformàtica de la UAB

Matriz para DNA Una matriz no es otra cosa que un sistema de scoring que permite asignar puntuaciones individuales a cada una de las letras del alfabeto en uso Fernán Agüero © 2006 Plataforma Bioinformàtica de la UAB

Matriz para DNA A G C T A +1 – 3 -3 G – 3 +1 – 3 -3 C – 3 +1 -3 T – 3 – 3 +1 CAGGTAGCAAGCTTGCATGTCA || |||||| CACGTAGCAAGCTTG-GTGTCA Score Match: + 1 Mismatch: - 3 raw score = 19 -9 = 10 Score Match: + 1 Mismatch: -10000 © 2006 Plataforma Bioinformàtica de la UAB
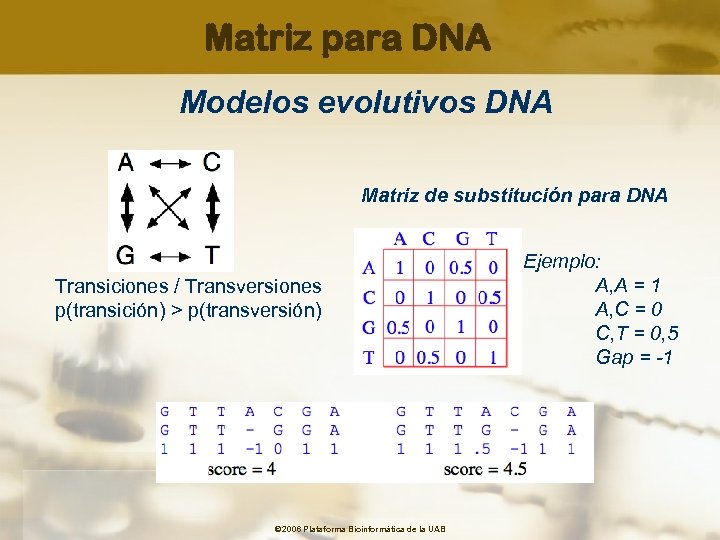
Matriz para DNA Modelos evolutivos DNA Matriz de substitución para DNA Transiciones / Transversiones p(transición) > p(transversión) © 2006 Plataforma Bioinformàtica de la UAB Ejemplo: A, A = 1 A, C = 0 C, T = 0, 5 Gap = -1

Matriz para proteínas Matrices de puntuación para proteínas Matriz de identidad Secuencia A Tyr Cys Asp Ala Met Secuencia B Phe Met Glu Gly Met 0 0 1 Puntuación total del alineamiento: 0+0+1 = 1 Matrices de susbtitución Secuencia A Tyr Cys Asp Ala Secuencia B Phe Met Glu Gly 3 -1 2 0 Puntuación total del alineamiento: 3 -1+2+0 = 4 © 2006 Plataforma Bioinformàtica de la UAB

Matriz para proteínas Modelos evolutivos Proteínas Mutaciones (código genético) Substituciones conservativas Matriz de substitución para proteínas • PAM 60, 120, 250 (Dayhoff) Extrapolación desde PAM 1 • BLOSUM 80, 62, 40 Basadas en secuencias © 2006 Plataforma Bioinformàtica de la UAB BLOCKS de

Matrices PAM (Percent Accepted Mutation) Evolutionary model • Creadas partiendo de un grupo de secuencias homólogas con un porcentage de similitud igual o superior al 85 %. • Proporcionan los cambios esperados entre proteínas homólogas a lo largo de un determinado periodo evolutivo • Generan una matriz 20 x 20 • Todas calculadas a partir de PAM 1 (identidades aprox 99%) • Se pueden extrapolar los cambios esperados en periodos cortos a los esperados en periodos largos simplemente multiplicando la matriz original n veces. • La más utilizada: PAM 250 (identidades aprox 20%) © 2006 Plataforma Bioinformàtica de la UAB

Matrices BLOSUM (BLOck SUbstitution Matrices) • Creadas utilizando un gran número de secuencias NO homólogas pertenecientes a distintas familias • Se evaluan las tasas de sustitución en patrones de residuos altamente conservados (BLOCKS) • Generan matrices 20 x 20 • Las distintas matrices se generan usando agrupaciones de bloques con mayor o menor grado de similitud • La matriz por defecto: BLOSUM 62 © 2006 Plataforma Bioinformàtica de la UAB
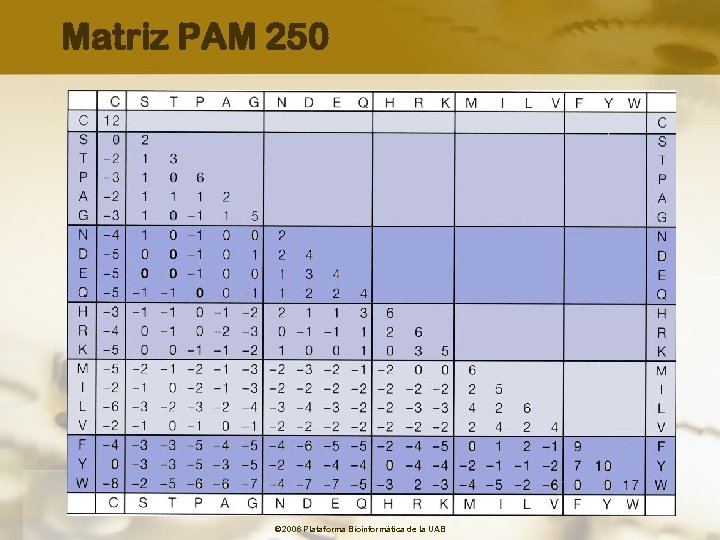
Matriz PAM 250 © 2006 Plataforma Bioinformàtica de la UAB
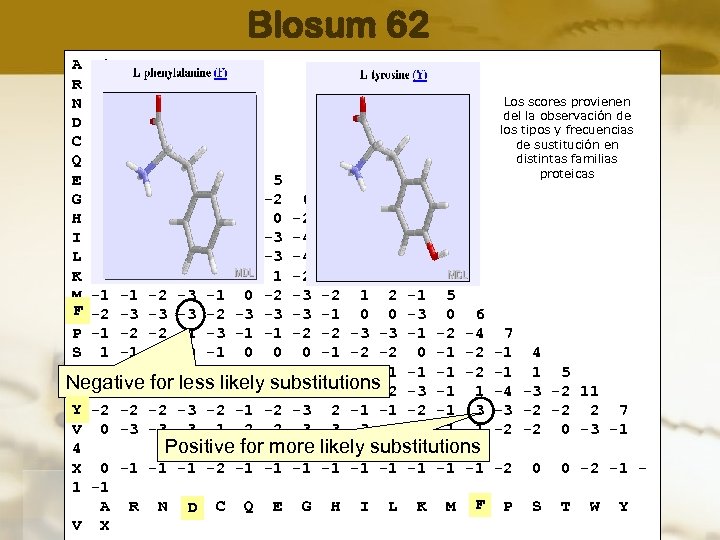
Blosum 62 A 4 R -1 5 Los scores provienen N -2 0 6 del la observación de D -2 -2 1 6 los tipos y frecuencias C 0 -3 -3 -3 9 de sustitución en distintas familias Q -1 1 0 0 -3 5 proteicas E -1 0 0 2 -4 2 5 G 0 -2 0 -1 -3 -2 -2 6 H -2 0 1 -1 -3 0 0 -2 8 I -1 -3 -3 -3 -1 -3 -3 -4 -3 4 L -1 -2 -3 -4 -3 2 4 K -1 2 0 -1 -3 1 1 -2 -1 -3 -2 5 M -1 -1 -2 -3 -1 0 -2 -3 -2 1 2 -1 5 F F -2 -3 -3 -3 -1 0 0 -3 0 6 P -1 -2 -2 -1 -3 -1 -1 -2 -2 -3 -3 -1 -2 -4 7 S 1 -1 1 0 -1 0 0 0 -1 -2 -2 0 -1 -2 -1 4 T 0 -1 -1 -2 -2 -1 -1 -2 -1 1 5 Negative for less likely substitutions W -3 -3 -4 -4 -2 -2 -3 -1 1 -4 -3 -2 11 Y Y -2 -2 -2 -3 -2 -1 -2 -3 2 -1 -1 -2 -1 3 -3 -2 -2 2 7 V 0 -3 -3 -3 -1 -2 -2 -3 -3 3 1 -2 1 -1 -2 -2 0 -3 -1 Positive for more likely substitutions 4 X 0 -1 -1 -1 -2 -1 -1 -1 -2 0 0 -2 -1 1 -1 F A R N D C Q E G H I L K M F P S T W Y D V X © 2006 Plataforma Bioinformàtica de la UAB

Equivalencia PAM - BLOSUM PAM 100 ==> Blosum 90 PAM 120 ==> Blosum 80 PAM 160 ==> Blosum 60 PAM 200 ==> Blosum 52 PAM 250 ==> Blosum 45 BLOSUM: “mejor” para la detección alineamientos locales BLOSUM 62, detección similitudes débiles BLOSUM 45, detección alineamientos largos y débiles © 2006 Plataforma Bioinformàtica de la UAB

PAM vs BLOSUM • PAM asume un modelo de evolución markoviano (todos los cambios independientes) • BLOSUM no supone ningún modelo evolutivo explícito • PAM considera todas las posiciones amino acídicas • BLOSUM considera sólo los cambios en posiciones dentro de bloques conservados • PAM asume que la distribución de aa de las secuencias que la generaron es representativa de todas las familias de proteínas • BLOSUM se generó usando un mayor número de secuencias y de familias distintas • PAM requiere un conocimiento previo de la distancia evolutiva Conclusión: PAM es más adecuada para trazar el origen evolutivo de proteínas BLOSUM es más apropiada para hallar dominios conservados © 2006 Plataforma Bioinformàtica de la UAB

Valoración de un alineamiento: gaps Gap penalty W = g - r(x-1) W es la puntuación x la longitud del gap g la penalización de abrir un gap r la penalizaciónde extender un gap • Muchos programas de alineamiento sugieren los valores por defecto • Penalización al final del alineamiento • Sí en secuencias homólogas misma longitud • No en secuencias desconocidas o longitud diferente © 2006 Plataforma Bioinformàtica de la UAB

Valoración de un alineamiento: gaps Valores de penalización de gaps Programas de alineamiento de DNA Programa Tipo de Match Mismatch Penalización alineamiento score apertura gaps extensión gaps FASTA local 5 - 4 -16 -4 Programas de alineamiento de Proteínas Programa Tipo de Match y Mismatch Penalización alineamiento score apertura gaps extensión gaps FASTA local Valores BLOSUM 50 - 16 - 4 -12 -2 BLAST local Valores BLOSUM 62 - 6 - 4 - 8 - 2 © 2006 Plataforma Bioinformàtica de la UAB

Valoración de un alineamiento: gaps Efecto de la variación en la penalización de huecos (gap) (Vingron & Waterman 1994) • Si se penaliza alto (relativamente) no aparecerán en el alineamiento • Si bajo, gaps serán ubicuos • Si la penalización gaps y desemparejamientos (mismatch) es alta habrá un alineamiento local con emparejamientos exactos El alineamiento de proteínas relacionadas muestran que gaps > 5 nunca ocurren © 2006 Plataforma Bioinformàtica de la UAB

Métodos de alineamiento de dos secuencias • Dot Matrix (Matriz de puntos) Rápida identificación de indels / No alineamientos óptimos • Dynamic Programming (Programación dinámica) Garantiza alineamiento óptimo / Computacionalmente costoso • Heuristic Searches (Búsquedas heurísticas) Búsquedas rápidas en bases de datos grandes / alineamientos locales y no siempre óptimos © 2006 Plataforma Bioinformàtica de la UAB
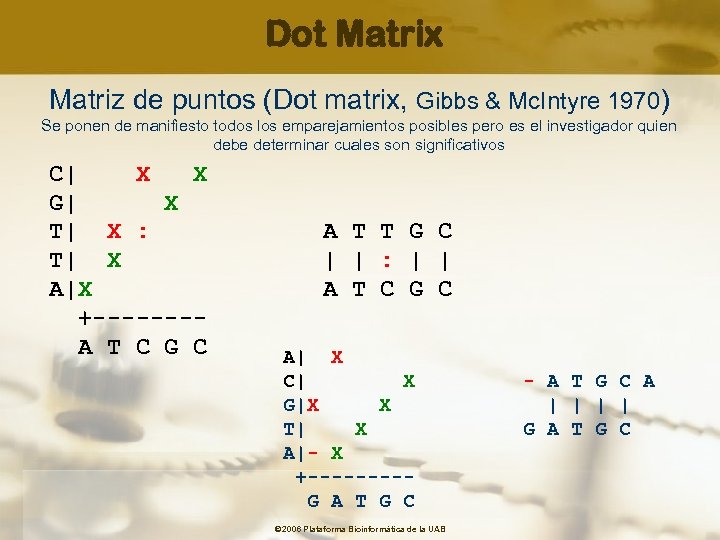
Dot Matrix Matriz de puntos (Dot matrix, Gibbs & Mc. Intyre 1970) Se ponen de manifiesto todos los emparejamientos posibles pero es el investigador quien debe determinar cuales son significativos C| X X G| X T| X : A T T G C T| X | | : | | A|X A T C G C +------- A T C G C A| X C| X - A T G C A G|X X | | T| X G A T G C A|- X +---- G A T G C © 2006 Plataforma Bioinformàtica de la UAB

Alineamiento: matriz de puntos Matriz de puntos (Dot matrix) A|X X X T| X X G| X . T| X . A T C A C T G T A C| X . | | | | A|X X A T C A - - G T A C| X T| X X A|X X +------ A T C A G T A Detección de indels: inserciones / deleciones © 2006 Plataforma Bioinformàtica de la UAB

Alineamiento: matriz de puntos Matriz de puntos de dos proteínas represoras en fagos, λ c. I y P 22 c 2 • Nucleótidos • Ventana = 11 (longitud del bloque) • Astringencia = 7 (Mínima coincidencia admitida) • 11/7 ó 15/11 Aminoácidos • Ventana = 1 • Astringencia = 1 • 1/1 excepto búsquedas pequeños dominios 15/5 © 2006 Plataforma Bioinformàtica de la UAB

Alineamiento: matriz de puntos Matriz de puntos de la proteína receptora humana LDL con ella misma. • Ventana = 23 • Astringencia = 7 • Ventana = 1 • Astringencia = 1 • © 2006 Plataforma Bioinformàtica de la UAB

Alineamiento: matriz de puntos Dominios EGF conservados en la urokinse plasminogen activator (PLAU) y el tissue plasminogen activator (PLAT) 137 23 90 90 137 23 Dot plots sugieren caminos (paths) a través del espacio de alineamientos posibles. 72 Cada path es un alineamiento único. 72 Path graphs son representaciones más explícitas de un alineamiento. PLAU 90 EPKKVKDHCSKHSPCQKGGTCVNMP--SGPH-CLCPQHLTGNHCQKEK---CFE 137 PLAT 23 ELHQVPSNCD----CLNGGTCVSNKYFSNIHWCNCPKKFGGQHCEIDKSKTCYE 72 © 2006 Plataforma Bioinformàtica de la UAB

Programación dinámica Algoritmos de programación dinámica Métodos computacionales que comparan cada pareja de caracteres y los posicionan de forma que el número de emparejamientos idénticos o relacionados sea el máximo posible Alineamiento global de Needlman-Wunsh (1970) Alineamiento local de Smith-Waterman (1981) © 2006 Plataforma Bioinformàtica de la UAB

Programación dinámica Evalúa y puntúa todos los posibles emparejamientos para cada caracter y lo posiciona en función de dicha puntuación y de la puntuación total de los caracteres ya alineados, construyendo una “ruta” hacia el alineamiento óptimo o de mayor puntuación Uso de un algoritmo recursivo que añade residuos en una posición sobre el alineamiento mejor hasta esa posición. Una ruta óptima que termina en un nodo debe pasar por uno de los tres nodos previos S(i-1, j-1) + c(i, j) S(i, j) = max S(i-1, j) + c(i, -) S(i, j-1) + c(-, j) © 2006 Plataforma Bioinformàtica de la UAB
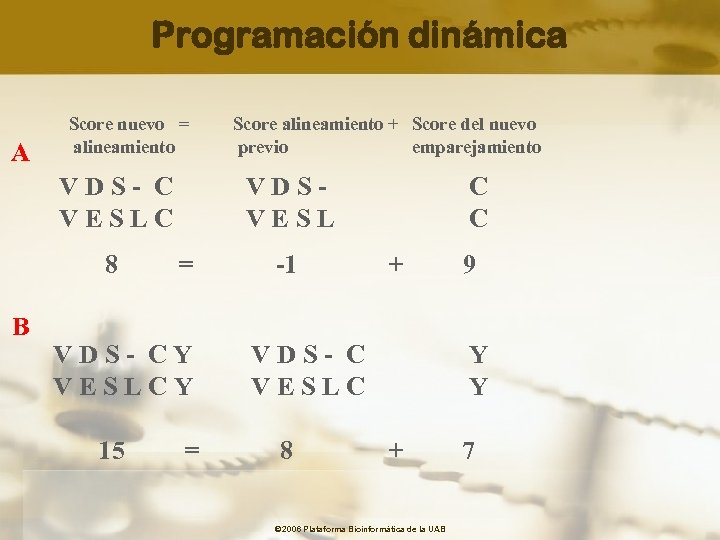
Programación dinámica A Score nuevo = alineamiento VDS- C VESLC 8 B VDSVESL = VDS- CY VESLCY 15 Score alineamiento + Score del nuevo previo emparejamiento = -1 C C + VDS- C VESLC 8 9 Y Y + © 2006 Plataforma Bioinformàtica de la UAB 7
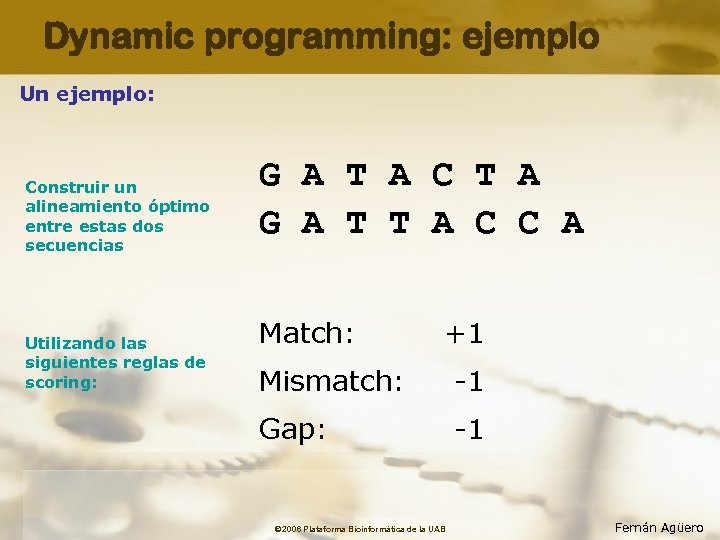
Dynamic programming: ejemplo Un ejemplo: Construir un alineamiento óptimo entre estas dos secuencias Utilizando las siguientes reglas de scoring: G A T A C T A G A T T A C C A Match: +1 Mismatch: -1 Gap: -1 © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero

Dynamic programming: ejemplo Ordenar las dos secuencias en una matriz bidimensional Los vértices de cada celda se encuentran entre letras (bases). Needleman & Wunsch (1970) G A T A C T A G A T T A C C A © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero
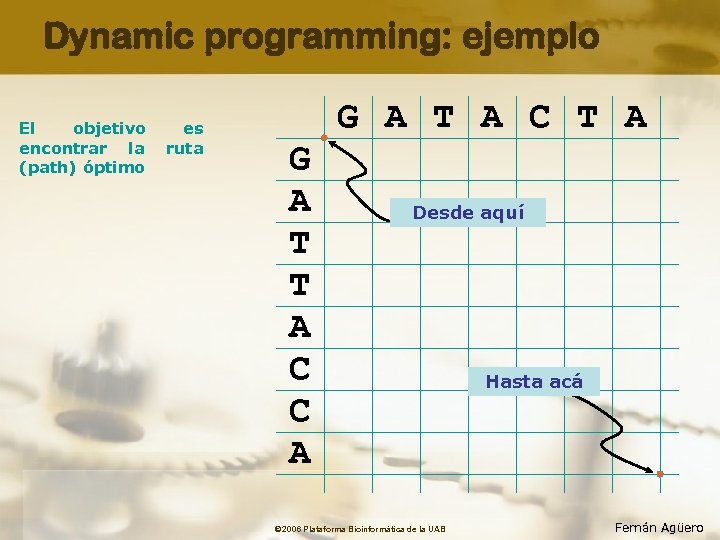
Dynamic programming: ejemplo El objetivo encontrar la (path) óptimo es ruta G A T A C T A G A T T A C C A Desde aquí © 2006 Plataforma Bioinformàtica de la UAB Hasta acá Fernán Agüero

Dynamic programming: ejemplo Cada path corresponde a un alineamiento único G A T A C T A G A T T A C C A Cuál es el óptimo? © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero
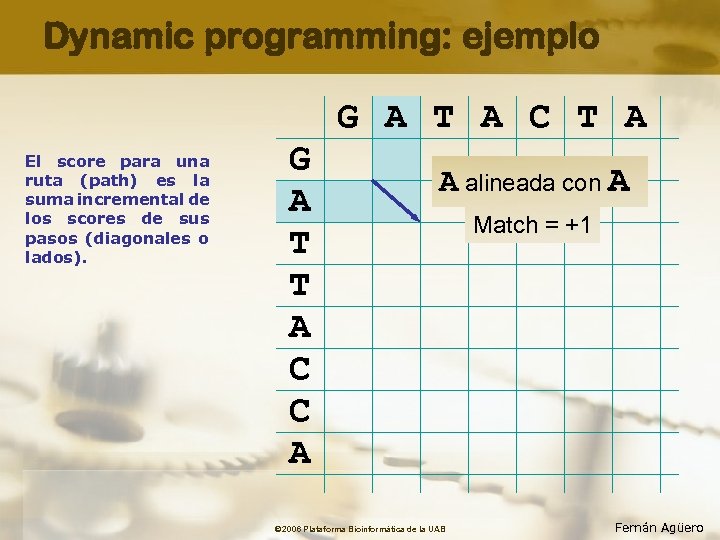
Dynamic programming: ejemplo G A T A C T A El score para una ruta (path) es la suma incremental de los scores de sus pasos (diagonales o lados). G A T T A C C A A alineada con A © 2006 Plataforma Bioinformàtica de la UAB Match = +1 Fernán Agüero
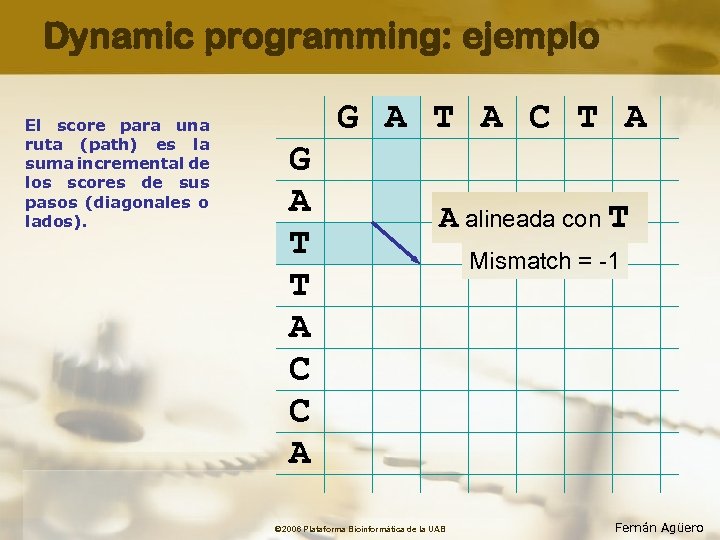
Dynamic programming: ejemplo El score para una ruta (path) es la suma incremental de los scores de sus pasos (diagonales o lados). G A T A C T A G A T T A C C A A alineada con T © 2006 Plataforma Bioinformàtica de la UAB Mismatch = -1 Fernán Agüero
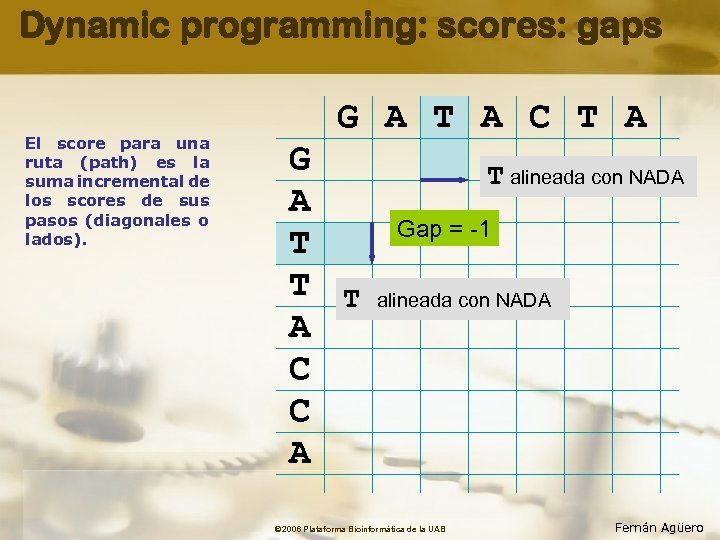
Dynamic programming: scores: gaps El score para una ruta (path) es la suma incremental de los scores de sus pasos (diagonales o lados). G A T A C T A G A T T A C C A T alineada con NADA Gap = -1 T alineada con NADA © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero
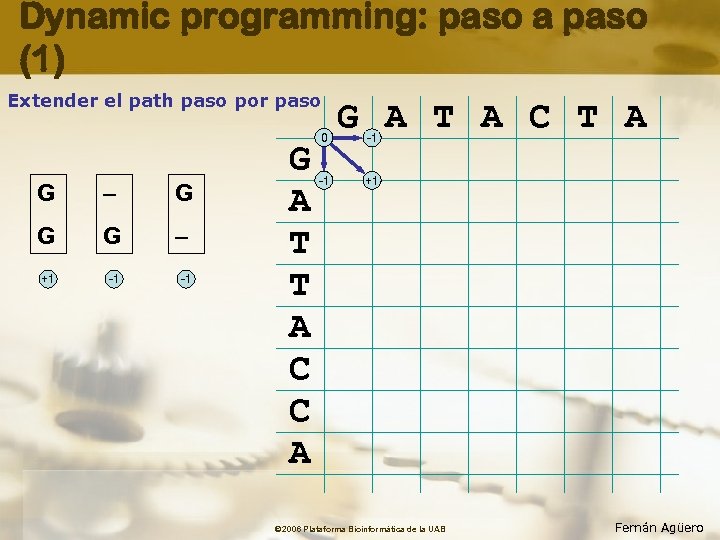
Dynamic programming: paso a paso (1) Extender el path paso por paso G – G G G – +1 -1 -1 G A T T A C C A 0 -1 G A T A C T A -1 +1 © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero
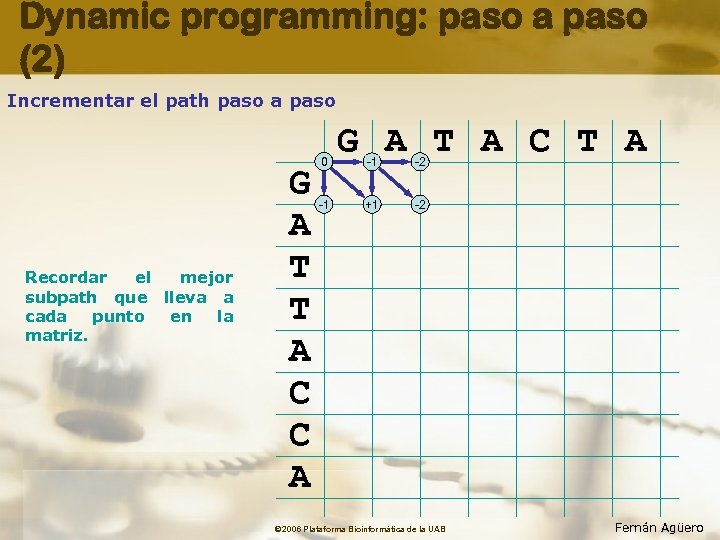
Dynamic programming: paso a paso (2) Incrementar el path paso a paso Recordar el mejor subpath que lleva a cada punto en la matriz. G A T T A C C A 0 -1 G A T A C T A -1 -2 +1 -2 © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero

Dynamic programming: paso a paso (3) Incrementar el path paso a paso Recordar el mejor subpath que lleva a cada punto en la matriz. G A T T A C C A 0 -1 G A T A C T A -1 -2 +1 -2 0 0 +2 © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero

Dynamic programming: paso a paso (4) Incrementar el path paso a paso Recordar el mejor subpath que lleva a cada punto en la matriz. G A T T A C C A 0 G A T A C T A -1 -2 -1 +1 -2 0 +2 © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero
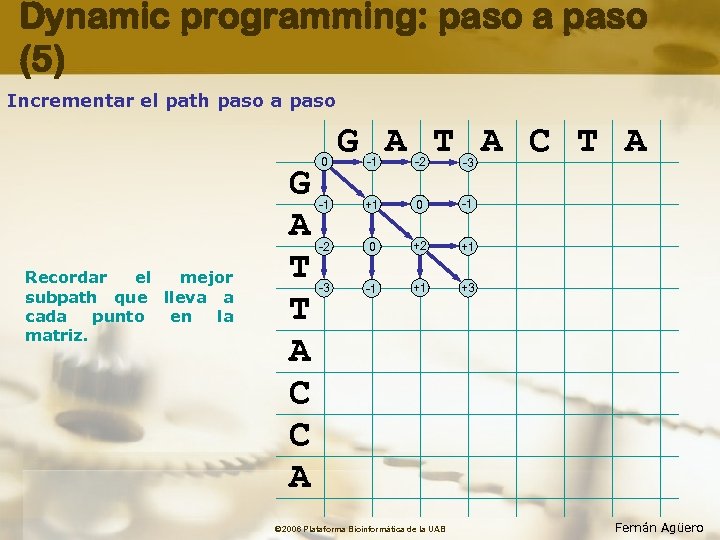
Dynamic programming: paso a paso (5) Incrementar el path paso a paso Recordar el mejor subpath que lleva a cada punto en la matriz. G A T T A C C A 0 G A T A C T A -1 -2 -3 -1 +1 -2 0 -1 -2 0 +2 +1 -3 -1 +1 +3 © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero

Dynamic programming: paso a paso (6) Incrementar el path paso a paso Recordar el mejor subpath que lleva a cada punto en la matriz. G A T T A C C A 0 G A T A C T A -1 -2 -3 -4 -5 -1 +1 0 -1 -2 -3 -2 0 +2 +1 0 -1 -3 -1 +1 +3 +2 +1 -4 -2 0 +2 +2 +1 -5 -3 -1 +1 +3 +2 © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero
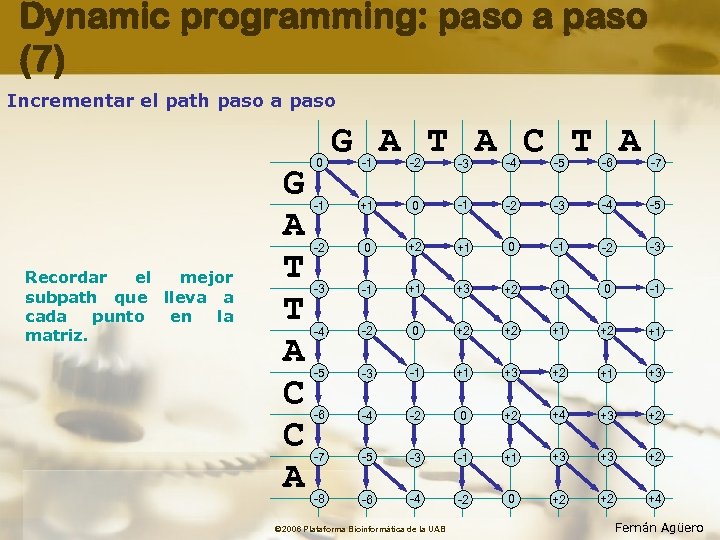
Dynamic programming: paso a paso (7) Incrementar el path paso a paso Recordar el mejor subpath que lleva a cada punto en la matriz. G A T T A C C A 0 G A T A C T A -1 -2 -3 -4 -5 -6 -7 -1 +1 0 -1 -2 -3 -4 -5 -2 0 +2 +1 0 -1 -2 -3 -3 -1 +1 +3 +2 +1 0 -1 -4 -2 0 +2 +2 +1 -5 -3 -1 +1 +3 +2 +1 +3 -6 -4 -2 0 +2 +4 +3 +2 -7 -5 -3 -1 +1 +3 +3 +2 -8 -6 -4 -2 0 +2 +2 +4 © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero
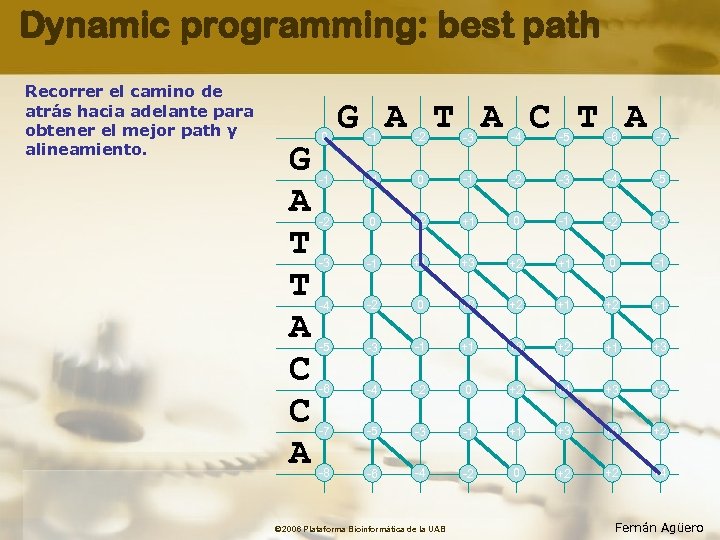
Dynamic programming: best path Recorrer el camino de atrás hacia adelante para obtener el mejor path y alineamiento. G A T T A C C A 0 G A T A C T A -1 -2 -3 -4 -5 -6 -7 -1 +1 0 -1 -2 -3 -4 -5 -2 0 +2 +1 0 -1 -2 -3 -3 -1 +1 +3 +2 +1 0 -1 -4 -2 0 +2 +2 +1 -5 -3 -1 +1 +3 +2 +1 +3 -6 -4 -2 0 +2 +4 +3 +2 -7 -5 -3 -1 +1 +3 +3 +2 -8 -6 -4 -2 0 +2 +2 +4 © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero

Dynamic programming: alineamiento obtenido G A T A C T A Imprimir el alineamiento G A - T A CT A G A T T A CC A G A T T A C C A © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero

Dynamic programming: Smith. Waterman • El método fue modificado (Smith-Waterman) para obtener alineamientos locales • El método garantiza la obtención de un alineamiento óptimo (cuyo score no puede ser mejorado) • La complejidad es proporcional al producto de las longitudes de las secuencias a alinear © 2006 Plataforma Bioinformàtica de la UAB Fernán Agüero
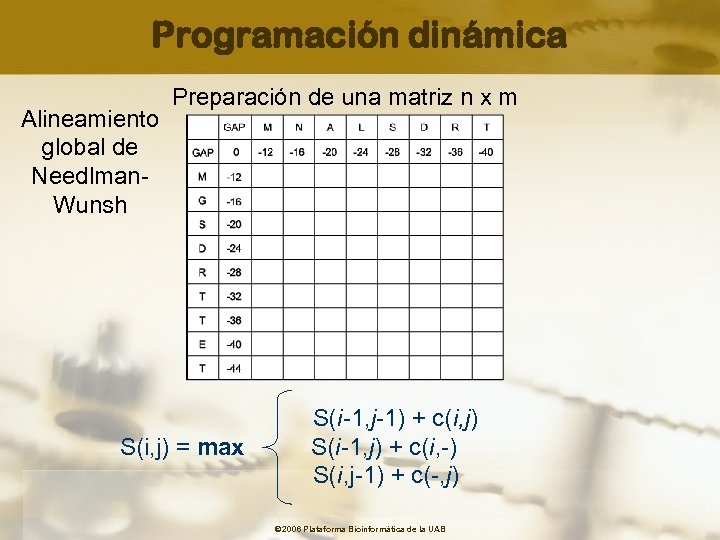
Programación dinámica Alineamiento global de Needlman. Wunsh Preparación de una matriz n x m S(i-1, j-1) + c(i, j) S(i, j) = max S(i-1, j) + c(i, -) S(i, j-1) + c(-, j) © 2006 Plataforma Bioinformàtica de la UAB

Ejemplo on-line: alineamiento global vs local Preparación de una matriz n x m © 2006 Plataforma Bioinformàtica de la UAB

Ejemplo on-line: alineamiento global vs local M N A L S D RT - - | | | M G - - S D RTT E T © 2006 Plataforma Bioinformàtica de la UAB

Ejemplo on-line: alineamiento global vs local M N A L S D R - T - | | | M G - - S D RTT E T © 2006 Plataforma Bioinformàtica de la UAB

Ejemplo on-line: alineamiento global vs local M N A L S D R - - - T | | | M G - - S D RTT E T © 2006 Plataforma Bioinformàtica de la UAB

Ejemplo on-line: alineamiento global vs local M N A L S D R - - - T | | | M - - G S D RTT E T © 2006 Plataforma Bioinformàtica de la UAB

Ejemplo on-line: alineamiento global vs local M N A L S D R - T - | | | M - - G S D RTT E T © 2006 Plataforma Bioinformàtica de la UAB

Ejemplo on-line: alineamiento global vs local M N A L S D RT - - | | | M - - G S D RTT E T © 2006 Plataforma Bioinformàtica de la UAB
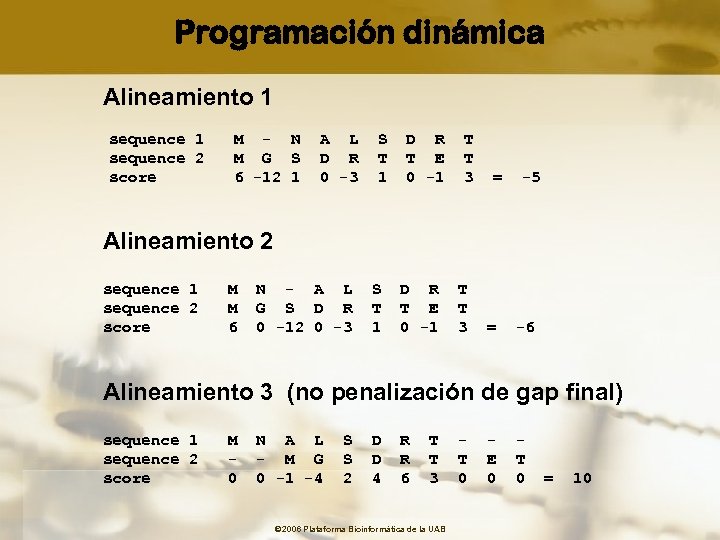
Programación dinámica Alineamiento 1 sequence 1 M - N A L S D R T sequence 2 M G S D R T T E T score 6 -12 1 0 -3 1 0 -1 3 = -5 Alineamiento 2 sequence 1 M N - A L S D R T sequence 2 M G S D R T T E T score 6 0 -12 0 -3 1 0 -1 3 = -6 Alineamiento 3 (no penalización de gap final) sequence 1 M N A L S D R T - - sequence 2 - - M G S D R T T E T score 0 0 -1 -4 2 4 6 3 0 0 0 = 10 © 2006 Plataforma Bioinformàtica de la UAB

Programación dinámica Alineamiento local con el algoritmo de Smith-Waterman • Debe haber puntuación negativa para los desemparejamientos y si la puntuación de la matriz obtiene un valor negativo se pone 0 • Así el alineamiento puede empezar en cualquier punto y se acaba cuando la puntuación alcanza el valor de 0 secuencia 1 secuencia 2 score S D R T 2 4 6 3 = 15 © 2006 Plataforma Bioinformàtica de la UAB
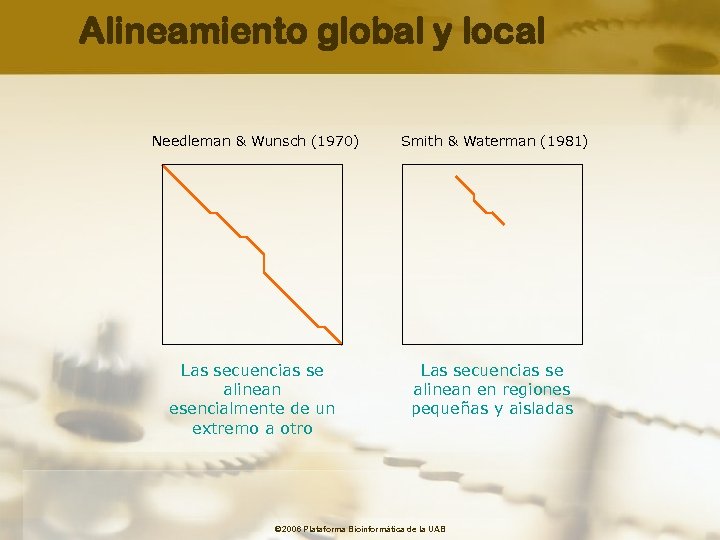
Alineamiento global y local Needleman & Wunsch (1970) Smith & Waterman (1981) Las secuencias se alinean esencialmente de un extremo a otro Las secuencias se alinean en regiones pequeñas y aisladas © 2006 Plataforma Bioinformàtica de la UAB

Heuristic searches MPKRSEYRQGTPNWVDLQTTDQSAAKKFYTLFGWGYDDNPV PGGGGVYSMATLNGEAVAAIAPMPPGAPEGMPPIWNTYIAVDD VDAVVDKVVPGGGQVMMPAFDIGDAGRMSFITDPTGAAVGWQ ANRHIGATLVNETGTLIWNELLTDKPDLALAFYEAVVGLTHSSM EIAAGQNY ¿Hay en la base de datos alguna secuencia similar a mi secuencia problema? Búsquedas por similitud Resultados üSimilar to ………… üUnknown but similar to sevral hypothetical proteins from… üPutative hypothetical protein……. . © 2006 Plataforma Bioinformàtica de la UAB

Búsqueda de similares en una base de datos Objetivo: comparar una secuencia frente a una base de datos, comprar doss base de datos, . . • Algoritmos Exactos: Smith-Waterman (sssearch, lalign, Heurísticos: BLAST (búsqueda de “words” similares) FASTA (búsqueda de “k-tuplos” idénticos) © 2006 Plataforma Bioinformàtica de la UAB . . )

Métodos heurísticos Método heurístico: prueba y error Suele encontrar secuencias relacionadas pero nunca hay garantía absoluta. Ventaja: 50 veces más rápido que programas dinámicos Usos: búsquedas sobre bases de datos de gran tamaño BLAST (Basic Local Alignment Search Tool) (Altschul, et al, 1990, J Mol Biol, 215: 403 -10) Concentra la búsqueda en patrones cortos más significativos, (palabra, word size: 3 aa / 11 nt). Rápido, menos sensible? FASTA (Lipman y Pearson, 1985; Pearson y Lipman, 1988) El algoritmo busca patrones cortos consecutivos (palabras o k-tuplos, k= 1 -2 aa / 4 -6 nt) entre la secuencia problema y las de la base de datos. © 2006 Plataforma Bioinformàtica de la UAB

Secuencias problema üBúsquedas con secuencias de DNA o aa FASTA, BLAST >Seq. DNA_Prob 1. seq ATGAAGGACTTAGTCGATACCACAGAGATGTACTTGCGTACTATC TATGAGCTGGAAGAAGAGGGAGTCACCCCTCTTCGCGCTAGGAT CGCTGAGCG üBúsquedas con perfiles Archivos con alineamientos D-HQSNGA ESHQ-YTM EAHQSN-L EGVQSYSL ü Búsquedas con Blocks DAHQS N ESHQSY EAHQS N EGVQS Y Motivos alineados sin gaps (PSSM position-specific scoring matrix) üBúsquedas de patrones y motivos PROSITE, INTERPRO, PFAM, . . DAHQS N © 2006 Plataforma Bioinformàtica de la UAB

Significación - Valoración de los alineamientos 1. Valoración mediante matrices 2. Penalización por disimilitud (aparición y extensión de gaps). Este valor es la Puntuación bruta (raw score, Sraw). 3. Puntuación de bits (Sbit). Permite comparar resultados obtenidos por diferentes sistemas. Se introducen parámetros utilizados por el programa de alineamiento. 4. E value alineamiento debido al azar. Los valores más cercanos a cero indican una mayor relevancia. Se puede considerar relevante cuando E < 0. 05. © 2006 Plataforma Bioinformàtica de la UAB
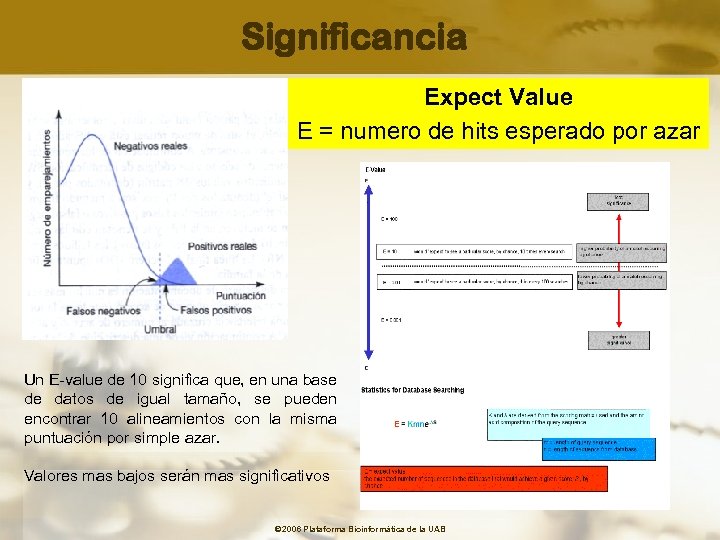
Significancia Expect Value E = numero de hits esperado por azar Un E-value de 10 significa que, en una base de datos de igual tamaño, se pueden encontrar 10 alineamientos con la misma puntuación por simple azar. Valores mas bajos serán mas significativos © 2006 Plataforma Bioinformàtica de la UAB

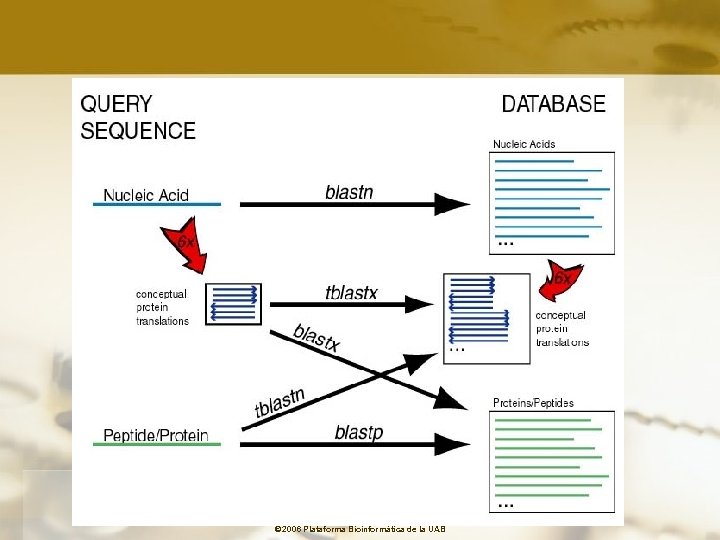
(Basic Local Alignment Search Tool) Heurístico Altschul, et al, 1990, J Mol Biol, 215: 403 -10 BLAST intenta encontrar muchos matches sacrificando la especificidad por la velocidad. Se pueden perder apareamientos. Estrategia BLAST üBúsqueda de proteínas mediante criterios de semejanza (no identidad). Se utilizan matrices de semejanza üTrabaja con segmentos pequeños que permiten acelerar el proceso (High-scoring segment pair) üExtensión de los match hacia los lados üRápido y sensible Usos Glossary übúsquedas en las bases de datos üalineamiento de pares de secuencias © 2006 Plataforma Bioinformàtica de la UAB

Esquema BLAST © 2006 Plataforma Bioinformàtica de la UAB

Nucleótidos Query: GTACTGGACATGGACCCTACAGGAA Word Size = 11 GTACTGGACAT TACTGGACATG ACTGGACATGG CTGGACATGGAC GGACATGGACC GACATGGACCC ACATGGACCCT . . . Minimum word size =7 blastn default = 11 megablast default = 28 © 2006 Plataforma Bioinformàtica de la UAB

Proteínas Query: GTQITVEDLFYNIATRRKALKN Word Size = 3 GTQ TQI Similares QIT LTV, MTV, ISV, LSV, etc. ITV TVE VED EDL DLF . . . © 2006 Plataforma Bioinformàtica de la UAB
© 2006 Plataforma Bioinformàtica de la UAB

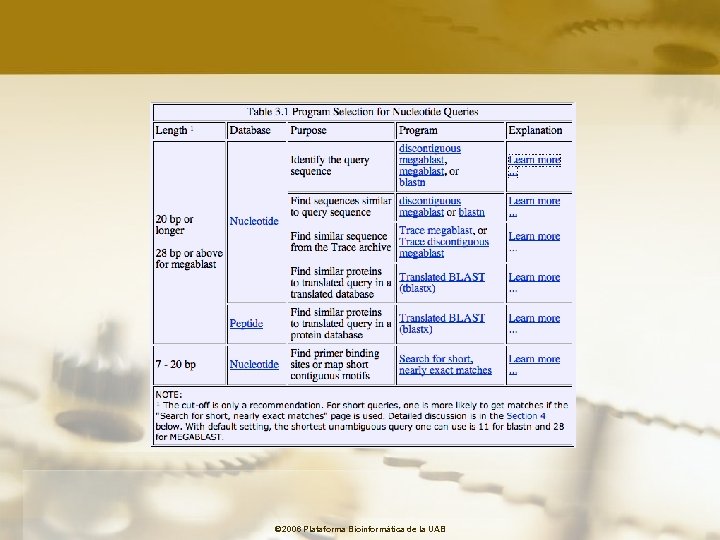
Selección del programa BLAST Help © 2006 Plataforma Bioinformàtica de la UAB
© 2006 Plataforma Bioinformàtica de la UAB

© 2006 Plataforma Bioinformàtica de la UAB

© 2006 Plataforma Bioinformàtica de la UAB

Opciones Conserved Domain Database © 2006 Plataforma Bioinformàtica de la UAB

Introducción de la Secuencia FASTA FORMAT © 2006 Plataforma Bioinformàtica de la UAB

Otras opciones Expect: 10, máximo de 10 al azar. Valores inferiores son mas restrictivos. Word Size: medida de los fragmentos (k-tup FASTA) © 2006 Plataforma Bioinformàtica de la UAB
Formatos © 2006 Plataforma Bioinformàtica de la UAB

Práctica Objetivo general Buscar secuencias similares a las secuencias problema. BLAST DNA Copiar la Secuencia PROBLEMA i someterla a BLAST de DNA >Seq. DNA_Prob 1. seq ATGAAGGACTTAGTCGATACCACAGAGATGTACTTGCGTACTATGAGCTGGAAGAAGAG GGAGTCA CCCCTCTTCGCGCTAGGATCGCTGAGCGTCTGGAACAATCTGGACCTACAGTTAGCCAAACCG TTGCCCG TATGGAGCGCGATGGACTTGTCGCCTCAGACCGCAGTCTACAAATGACACCGACAGG CCGCACT TTAGCGACTGCAGTTATGCGTAAACATCGCTTAGCTGAGCGCCTTCTTACCGATATCATTGGTC TAGATA TCAATAAAGTTCACGATGAAGCCTGCCGCTGGGAACACGTTATGAGTGACGAAGTTGAACGCA GGCTCGT GAAAGTATTGAAAGATGTCAGTCGGTCCCCCTTCGGAAACCCAATTCCAGGTCTCGACGAACT CGGCGTA GGCAATTCTGACGCGGCAGCCCCCGGAACTCGCGTTATTGACGCTGCCACCAGCATGCCCCG CAAAGTAC GCATTGTTCAGATTAACGAAATCTTTCAAGTTGAAACGGATCAGTTTACACAGCTCCTCGATGC TGACAT CCGTGTTGGATCAGAAGTCGAAATTGTAGAGACGGCCACATCACGTTGAGCCACAATGG AAAAGAT GTCGAACTCCTCGATGATCTGGCTCACACTATTCGTATCGAAGAACTCTAA © 2006 Plataforma Bioinformàtica de la UAB

Práctica BLAST Proteína Copiar la secuencia PROBLEMA i someterla a BLAST de Proteína >Seq. Prot-Prob 1. pep MENRIDRIKKQLHSSSYKLTPQREATVRVLLENEEDHLSAEDVYLLVKEKSPEIGLATVY RTLELLTELKVVDKINFGDGVSRYDLRKEGAAHFHHHLVCMEFGAVDEIEGDLLEDVEEI IERDWKFKIKDHRLTFHGICHRCNGKETE Iniciar una sesión BLAST Protein Limitar la búsqueda a Blast de Bacteria Ejecutar Blast © 2006 Plataforma Bioinformàtica de la UAB

Práctica t n Proteína x DNA traducido a Proteína Objetivo Pretendemos encontrar secuencias bacterianas en diferentes genomas de microorganismos. Realizar una búsqueda t. BLASTn sobre genomas microbianos utilizando la secuencia: >Proteína problema MPKRSEYRQGTPNWVDLQTTDQSAAKKFYTSLFGWGYDDNPVPGGGGVYSMATLNGEAVAAIAPMPPGA P EGMPPIWNTYIAVDDVDAVVDKVVPGGGQVMMPAFDIGDAGRMSFITDPTGAAVGLWQANRHIGATLVNE TGTLIWNELLTDKPDLALAFYEAVVGLTHSSMEIAAGQNYRVLKAGDAEVGGCMEPPMPGVPNHWHVYFA VDDADATAAKAAAAGGQVIAEPADIPSVGRFAVLSDPQGAIFSVLKPAPQQ © 2006 Plataforma Bioinformàtica de la UAB

Práctica X DNA traducido a Proteína x Proteína Objetivo Detectar similares a la secuencia problema y determinar si hay errores. Realizar una búsqueda con BLASTx usando: >DNA desconocido ATGCCCAAGAGAAGCGAATACAGGCACGCCGAACTGGGTCGACCTTCAGACCACCGATCAGTCCG CCGCCAAAAAGTTCTACACATCGTTggt. GTTCGGCTGGGGTTACGACGACCCGGTCCCCGGAGGCGGTGG GGTCTATTCCATGGCCACGCTGAACGGCGAAGCCGTGGCCGCCATCGCACCGATGCCCCCGGGTGCACCG GAGGGGATGCCGCCGATCTGGAACACCTATATCGCGGTGGACGACGTCGATGCGGTGGTGGACAAGGTGG TGCCCGGGGGCAGGTGATGATGCCGGCCTTCGACATCGGCGATGCCGGATGTCGTTCATCAC CGATCCGACCGGCGCTGCCGTGGGCCTATGGCAGGCCAATCGGCACATCGGAGCGACGTTGGTCAACGAG ACGGGCACGCTCATCTGGAACTGCTCACGGAttg. GCCGGATTTGGCGCTAGCGTTCTACGAGGCTG TGGTTGGCCTCACCCACTCGAGCATGGAGATAGCTGCGGGCCAGAACTATCGGGTGCTCAAGGCCGGCGA CGCGGAAGTCGGCGGCTGTATGGAACCGCCGATGCCCGGCGTGCCGAATCATTGGCACGTCTACTTTGCG GTGGATGACGCCGACccc. ACGGCGGCCAAAGCCGCCGCAGCGGCCAGGTCATTGCGGAACCGGCTG ACATTCCGTCGGTGGGCCGGTTCGCCGTGTTGTCCGATCCGCAGGGCGCGATCTTCAGTGTGTTGAAGCC CGCACCGCAGCAATAG © 2006 Plataforma Bioinformàtica de la UAB
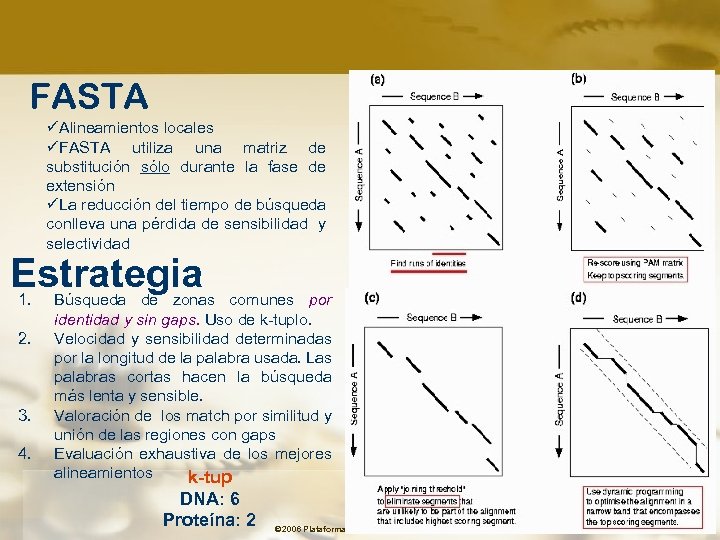
FASTA üAlineamientos locales üFASTA utiliza una matriz de substitución sólo durante la fase de extensión üLa reducción del tiempo de búsqueda conlleva una pérdida de sensibilidad y selectividad Estrategia 1. 2. 3. 4. Búsqueda de zonas comunes por identidad y sin gaps. Uso de k-tuplo. Velocidad y sensibilidad determinadas por la longitud de la palabra usada. Las palabras cortas hacen la búsqueda más lenta y sensible. Valoración de los match por similitud y unión de las regiones con gaps Evaluación exhaustiva de los mejores alineamientos k-tup DNA: 6 Proteína: 2 © 2006 Plataforma Bioinformàtica de la UAB

Programas FASTA Programa Funciones fasta 3 tfasta 3* DNA, proteína frente bd DNA y bd Prot (fasta) y proteínas frente bd DNA traducido (tfasta), no admite frameshifts ssearch* DNA, proteína frente bd DNA y bd Prot mediante el algoritmo Smith-Waterman Muy lento <10 fasta pero muy sensible. fastx/y 3 DNA (traducido 3 frames) frente bd proteínas. Admite gaps y frameshifts Velocidad x > y tfastx/y 3 Proteína frente bd DNA traducido 6 frames fasts 3 tfasts 3* Mezcla de péptidos pequeños relacionados (mass-spec) frente bd proteína (fasts) o bd DNA (tfasts)* fastf 3 tfastf 3* >mgstm 1 MILG, MLLEYTD, MGDAP Mezcla de péptidos (obtenida por Edman o CNBr) frente bd proteína (fastf) o bd DNA (tfastf)* >mgstm 1 MGCEN, MIDYP, MLLAY, MLLGY © 2006 Plataforma Bioinformàtica de la UAB

Opciones FASTA MATRIX Matriz de búsqueda GAP PENALTIES GAPOPEN: Penalización por apertura gap (-12 para proteínas, -16 para DNA). GAPEXT: Penalización extensión del gap (-2 para proteínas, -4 para DNA). HISTOGRAM Muestra el histograma con las frecuencias de las coincidencias por azar. SCORES Puntuación de los alineamientos ALIGNMENTS Número de alineamientos que se visualizaran KTUP Proteínas: 2 DNA: 6 STRAND Cadena de DNA EXPECTATION VALUE Límite superior para la presentación de un alineamiento. Valores por defecto son 10. 0 para Prot/Prot; 5. 0 para proteínas frente Prot/Prot de DNA y 2. 0 para búsquedas DNA/DNA. EXPECTATION VALUE THRESHOLD Límite inferior para la presentación de un alineamiento. © 2006 Plataforma Bioinformàtica de la UAB
© 2006 Plataforma Bioinformàtica de la UAB

Job FASTA © 2006 Plataforma Bioinformàtica de la UAB
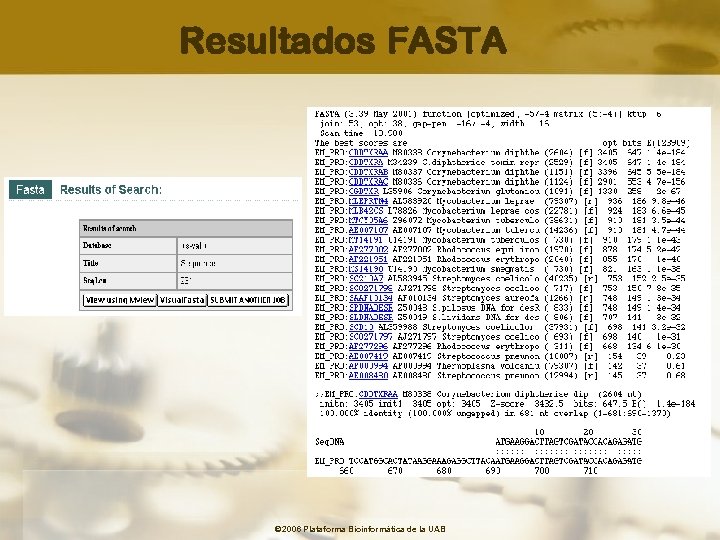
Resultados FASTA © 2006 Plataforma Bioinformàtica de la UAB

Mview Results FASTA © 2006 Plataforma Bioinformàtica de la UAB
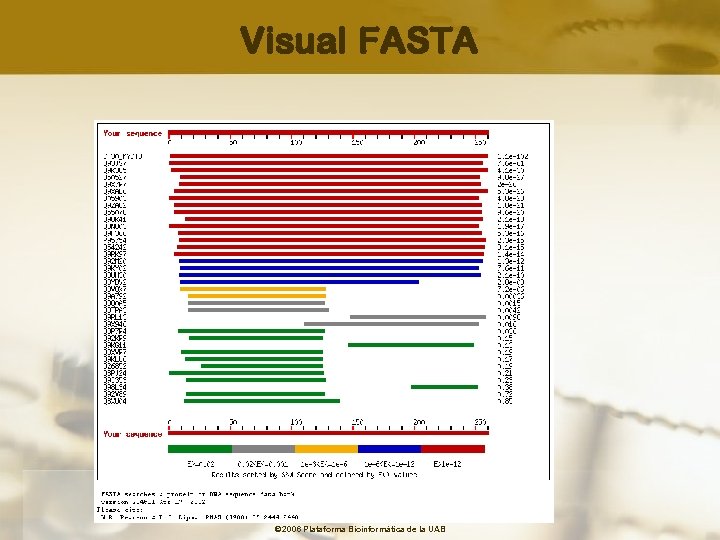
Visual FASTA © 2006 Plataforma Bioinformàtica de la UAB

Práctica FASTA Búsqueda por similitud • Iniciar una sesión FASTA • Seleccionar el programa y fijar los parámetros de búsqueda en función de la naturaleza de la secuencia problema. • Introducir la secuencia problema • Someter la búsqueda • Visualizar y comentar los resultados • Diferencias frente a BLAST © 2006 Plataforma Bioinformàtica de la UAB
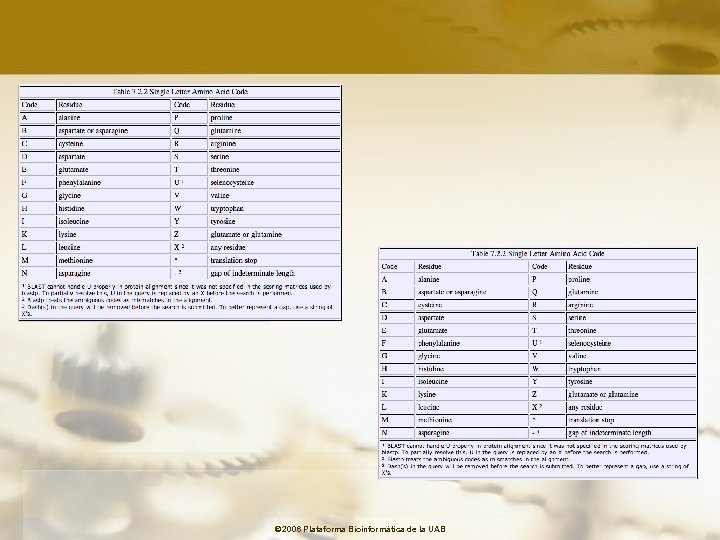
© 2006 Plataforma Bioinformàtica de la UAB

Comparación múltiple de secuencias 1 3 4 5 6 7 8 9 10 I Y D G G A V - E A L II Y D G G - - - E A L III F E G G I L V E A L IV F D - G I L V Q A V V Y E G G A V V Q A L Cons Alineamiento múltiple = Tabla 2 D 2 y d G G A/I V/L V e A l • Identificación de regiones conservadas • Predicción de estructuras y funciones • Diseño de experimentos para probar y modificar funciones de proteínas concretas • Identificación de nuevos miembros de una familia de proteínas © 2006 Plataforma Bioinformàtica de la UAB

Comparación múltiple de secuencias FHIT_HUMAN ------MS-F RFGQHLIKP-SVVFL KTELSFALVNRKPVV PGHVLV. . . APH 1_SCHPO ------MPKQ LYFSKFPVG-SQVFY RTKLSAAFVNLKPIL PGHVLV. . . HNT 2_YEAST MILSKTKKPKSMNKP IYFSKFLVT-EQVFY KSKYTYALVNLKPIV PGHVLI. . . Y 866_METJA ------MCIF CKIINGEIP-AKVVY EDEHVLAFLDINPRN KGHTLV. . . Un método de alineamiento múltiple verdadero, debería alinear todas las secuencias al mismo tiempo. Pero no existe un método computacional que pueda realizar esto en tiempo razonable © 2006 Plataforma Bioinformàtica de la UAB
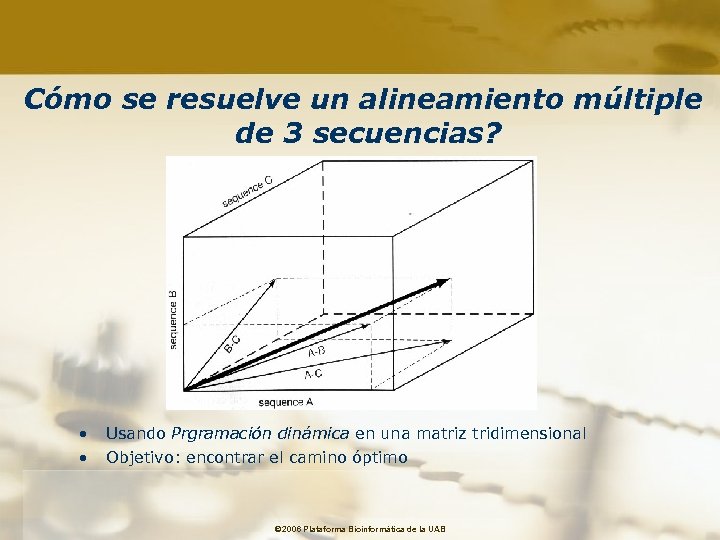
Cómo se resuelve un alineamiento múltiple de 3 secuencias? • • Usando Prgramación dinámica en una matriz tridimensional Objetivo: encontrar el camino óptimo © 2006 Plataforma Bioinformàtica de la UAB

Complejidad del algoritmo de Programación Dinámica (PD) • El número de comparaciones que el PS tiene debe realizar para llenar la matriz (sin usar heurísticas y excluyendo gaps) es el producto de las longitudes de las dos secuencias (N x M) • La complejidad del algoritmo crece en forma exponencial con el número de secuencias Alinear dos secuencias de 300 nt implica realizar 300 x 300 = 90, 000 comparaciones Alinear tres secuencias de 300 nt implica realizar 300 x 300 = 27, 000 comparaciones!! © 2006 Plataforma Bioinformàtica de la UAB

Aproximaciones al algoritmo de Programación Dinámica • Alinear todas las secuencias por pares • Usar los scores para construir un árbol • Alinear progresivamente (siguiendo el orden que sugiere el árbol) todas las secuencias para producir un Alineamiento Múltiple No es un verdadero Alineamiento Múltiple Las secuencias se alinean por pares © 2006 Plataforma Bioinformàtica de la UAB
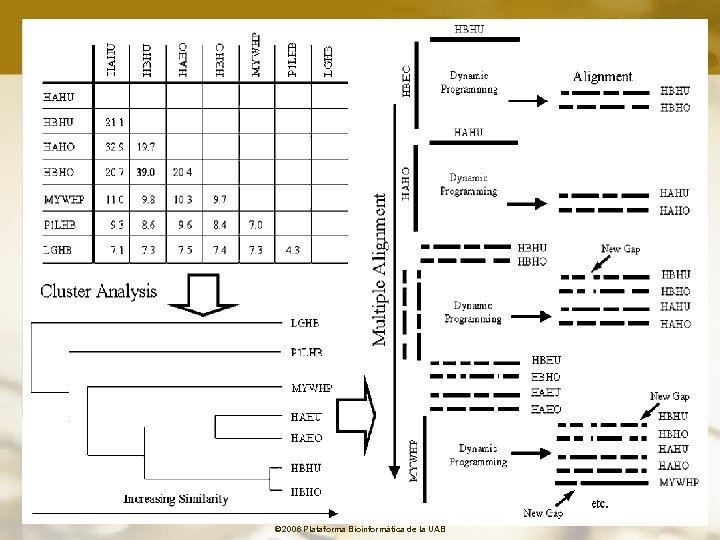
© 2006 Plataforma Bioinformàtica de la UAB

Clustal W Thompson J. D. , Higgins D. G. , Gibson T. J. (1994) "CLUSTAL W: Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. "; Nucleic Acids Res. 22: 4673 -4680 Programa de alineamiento múltiple Alineamiento progresivo © 2006 Plataforma Bioinformàtica de la UAB

Clustal W Thompson J. D. , Higgins D. G. , Gibson T. J. (1994) "CLUSTAL W: Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. "; Nucleic Acids Res. 22: 4673 -4680 Estrategia general üAlineamiento rápido • obtención de las mejores parejas • análisis de clusters • creación de un árbol guía üAlineamiento múltiple • se utiliza el árbol guía anterior • optimización alineamiento de los pares más próximos • introducción de gaps para mejorar el alineamiento • alineamiento de las parejas • optimización mediante inclusión de nuevos gaps © 2006 Plataforma Bioinformàtica de la UAB

Opciones Generales Clustal W YOUR EMAIL SEARCH TITLE CPU MODE clustalw_mp - multiprocessor SGI systems. clustalw - genérico (CPU simple) ALIGNMENT Permite realizar alineamientos completos utilizando algoritmos restrictivos que generan un árbol guía o algoritmos más rápidos. OUTPUT Formato del resultado (ALN, GCG, PHYLIP, PIR and GDE) OUTORDER Orden de las secuencias COLOR Muestra el alineamiento en colores (solo en formatos ALN or GCG) AVFPMI LW RED Small (small+ hydrophobic (incl. aromatic -Y)) DE BLUE Acidic RHK MAGENTA Basic • ": " = sustituciones conservadas STYHC NGQ GREEN Hydroxyl + Amine + Basic Q • ". " = sustituciones semi-conservadas. Others Gray Línea consenso • "*" = residuos idénticos o conservados en todas las secuencias © 2006 Plataforma Bioinformàtica de la UAB

Clustal W (EBI) © 2006 Plataforma Bioinformàtica de la UAB

Formato secuencias para Clustal W Formato FASTA >FOSB_HUMAN P 53539 homo sapiens (human). fosb protein MFQAFPGDYDSGSRCSSSPSAESQYLSSVDSFGSPPTAAASQECAGLGEMPGSFVPTVT A ITTSQDLQWLVQPTLISSMAQSQGQPLASQPPVVDPYDMPGTSYSTPGMSGYSSGGASG S Eliminar GGPSTSGTTSGPGPARPARARPRRPREETLTPEEEEKRRVRRERNKLAAAKCRNRRRELT espacios entre DRLQAETDQLEEEKAELESEIAELQKEKERLEFVLVAHKPGCKIPYEEGPGPGPLAEVRD secuencias LPGSAPAKEDGFSWLLPPPPPPPLPFQTSQDAPPNLTASLFTHSEVQVLGDPFPVVNPSY TSSFVLTCPEVSAFAGAQRTSGSDQPSDPLNSPSLLAL >FOSB_MOUSE P 13346 musculus (mouse). fosb protein. MFQAFPGDYDSGSRCSSSPSAESQYLSSVDSFGSPPTAAASQECAGLGEMPGSFVPTVT A ITTSQDLQWLVQPTLISSMAQSQGQPLASQPPAVDPYDMPGTSYSTPGLSAYSTGGASGS GGPSTSTTTSGPVSARPARARPRRPREETLTPEEEEKRRVRRERNKLAAAKCRNRRRELT DRLQAETDQLEEEKAELESEIAELQKEKERLEFVLVAHKPGCKIPYEEGPGPGPLAEVRD LPGSTSAKEDGFGWLLPPPPPPPLPFQSSRDAPPNLTASLFTHSEVQVLGDPFPVVSPSY TSSFVLTCPEVSAFAGAQRTSGSEQPSDPLNSPSLLAL © 2006 Plataforma Bioinformàtica de la UAB
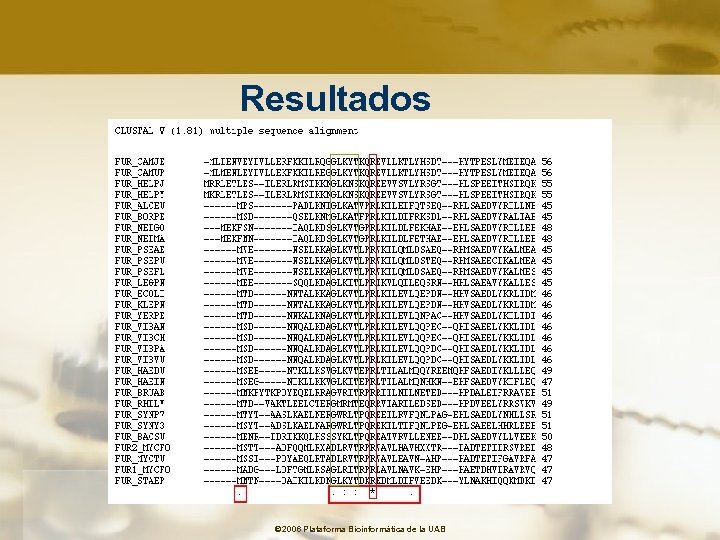
Resultados © 2006 Plataforma Bioinformàtica de la UAB

Resultados Jal. View © 2006 Plataforma Bioinformàtica de la UAB
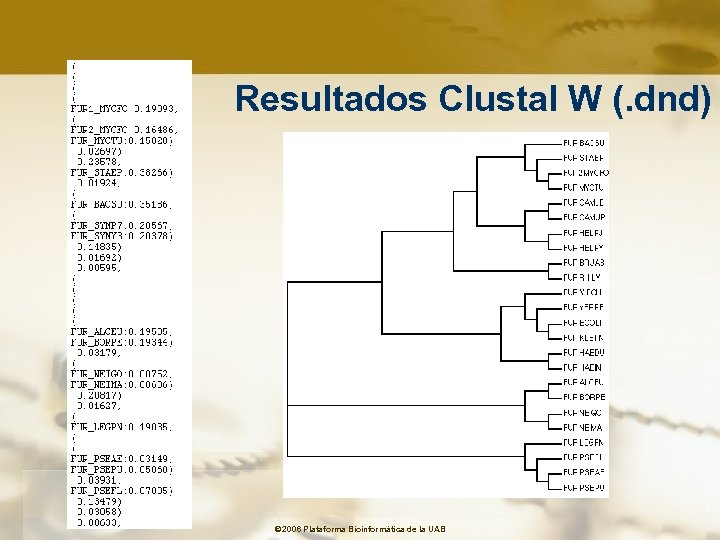
Resultados Clustal W (. dnd) © 2006 Plataforma Bioinformàtica de la UAB

Árboles Phylodendron Phylogenetic tree printer © 2006 Plataforma Bioinformàtica de la UAB

Práctica Clustal. W - Primers Comparación múltiple • Abrir una sesión Clustal W • Fija los parámetros e introduce las secuencias usando el archivo múltiple al que hemos añadido todas las secuencias a alinear. • Ejecutar Clustal W • Visualizar los resultados • Identificar y almacenar les regiones conservadas (primers) Diseño primers • Realizar una traducción reversa de los primers en la Sequence Manipulation Suite de la Univ de Alberta • Mediante las tablas de uso de codones, disminuir la degeneración de los primers adaptándolos al uso del Microorganismo problema Árboles • Visualizar el archivo. dnd con Phylodendron (o Tree. View local) • Variar la topología del árbol enraizándolo a un outgroup arbitrario © 2006 Plataforma Bioinformàtica de la UAB
1f54e10bfff835290ce07a6b2f513e5c.ppt