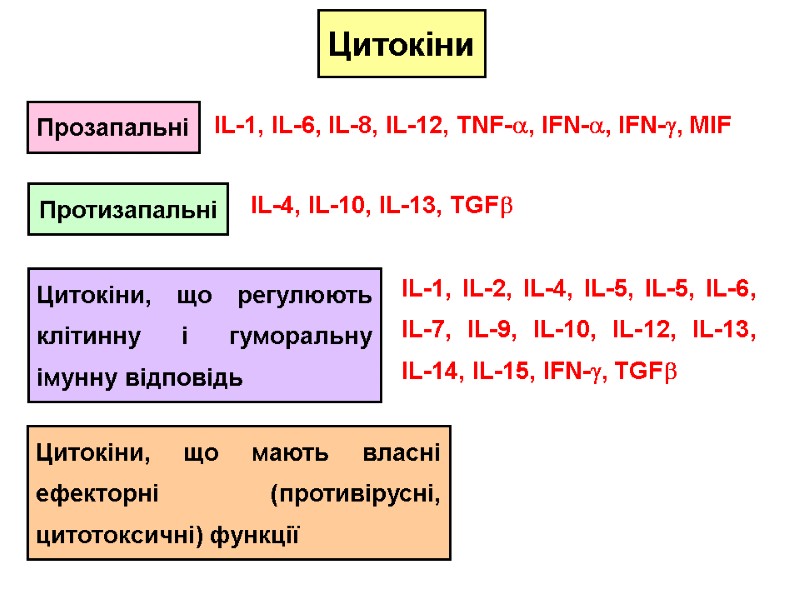
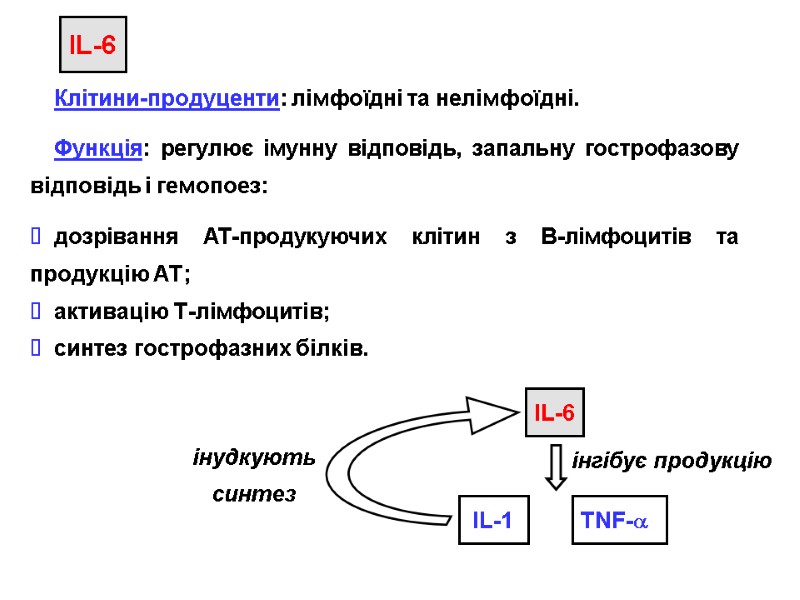
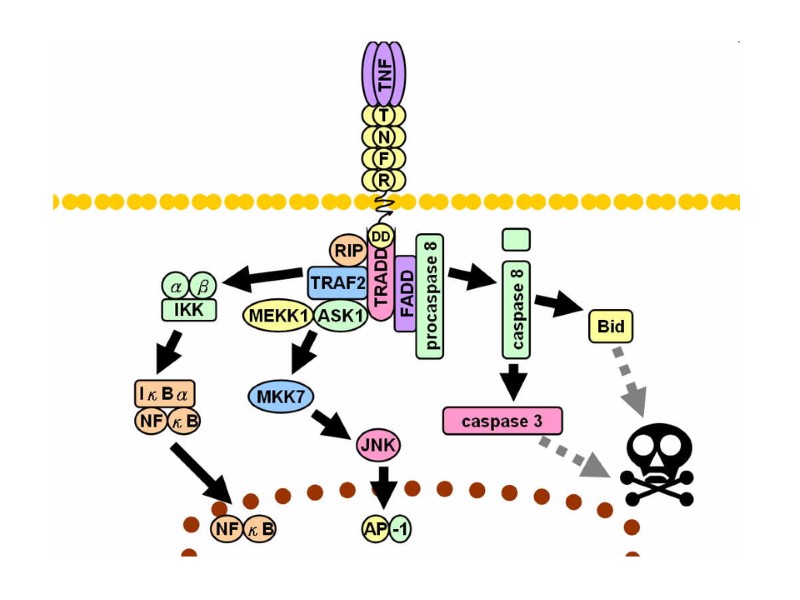
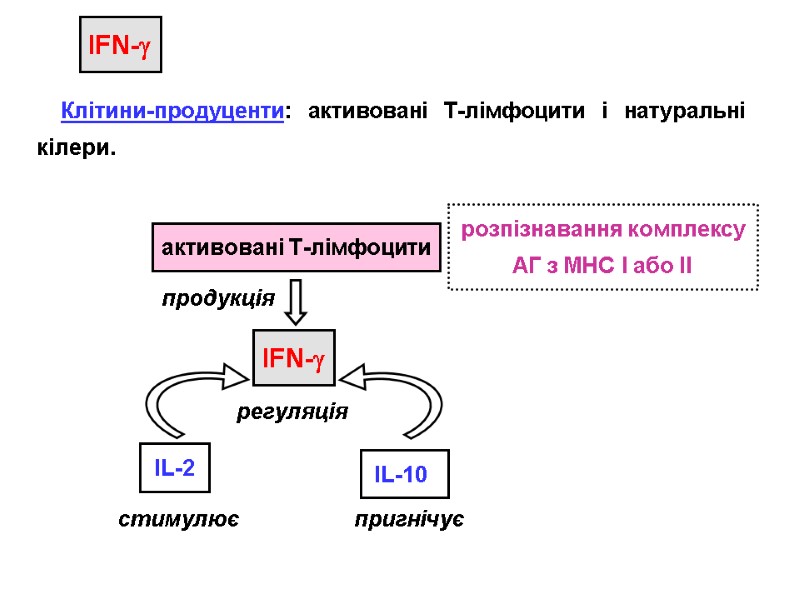
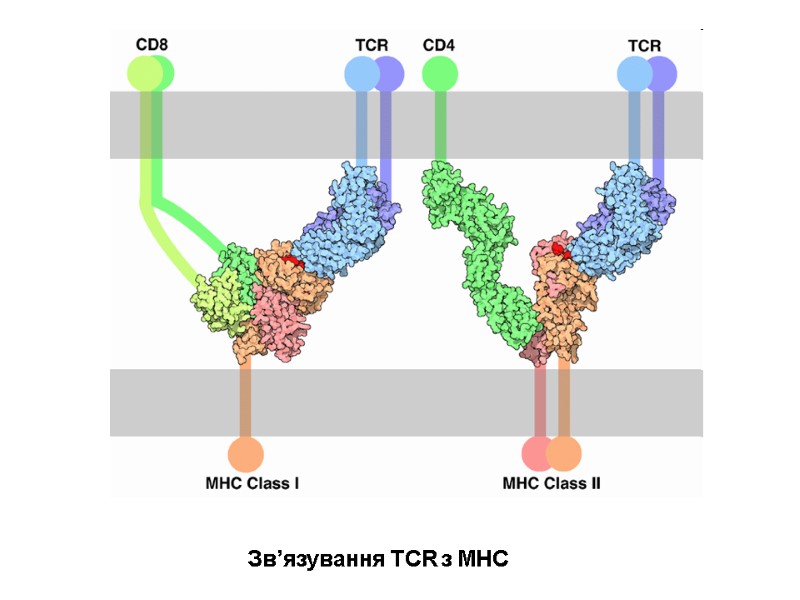
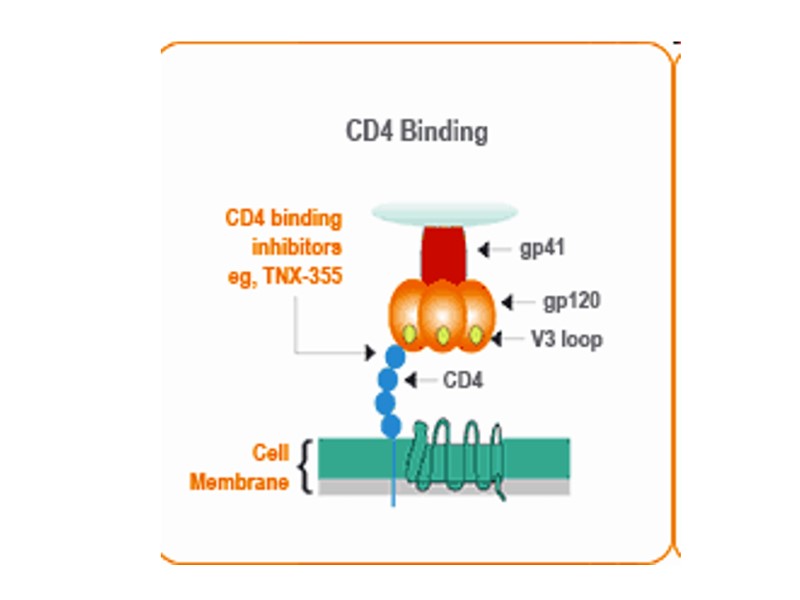
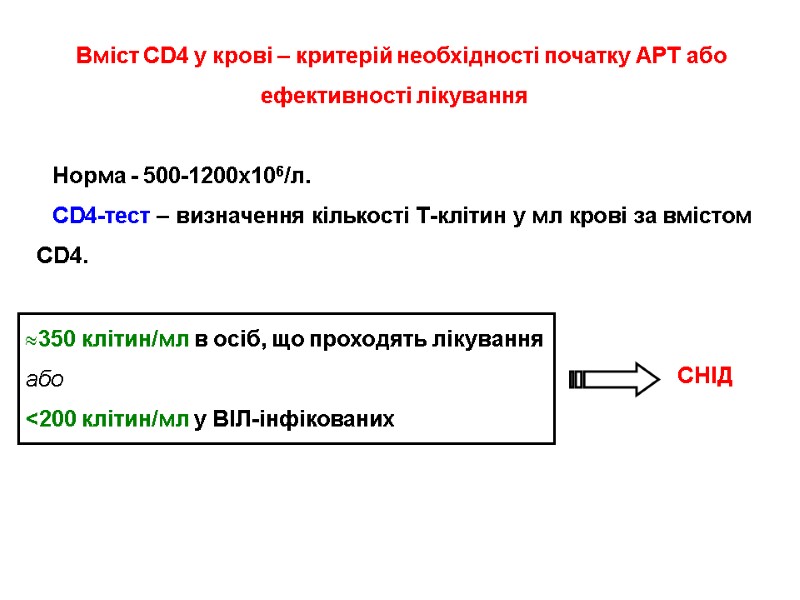
HIV Structure The “Enemy” HIV Structure SIV HIV

















































































































































7958-prezentation_2_hiv_structure_andlifecycle.ppt
- Количество слайдов: 328
 HIV Structure
HIV Structure
 The “Enemy”
The “Enemy”
 HIV Structure SIV HIV
HIV Structure SIV HIV


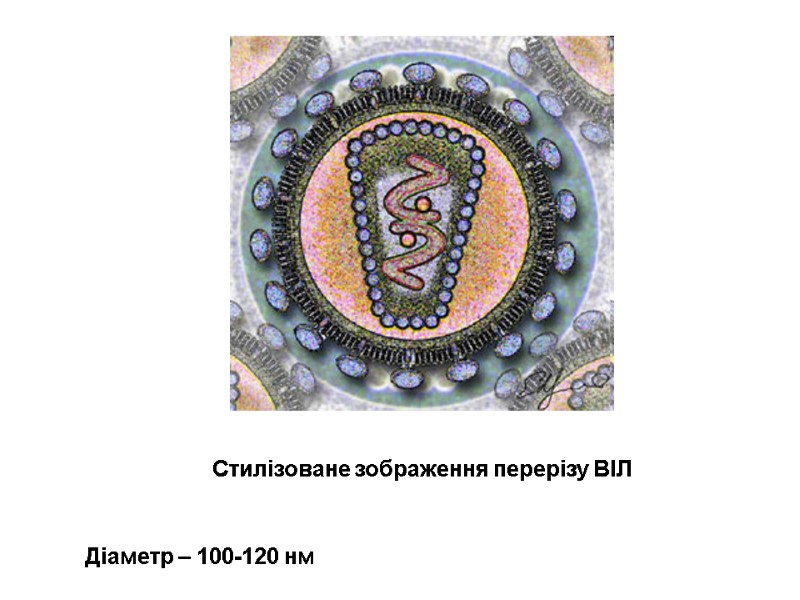
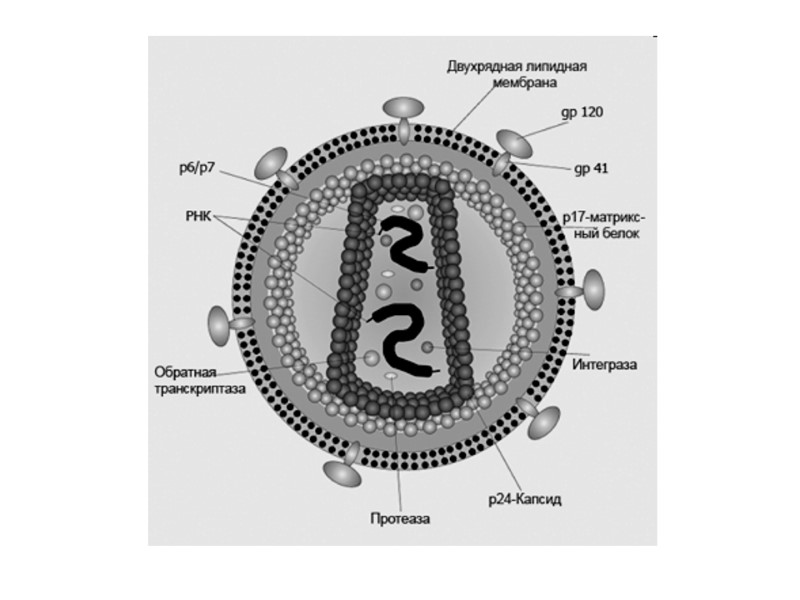
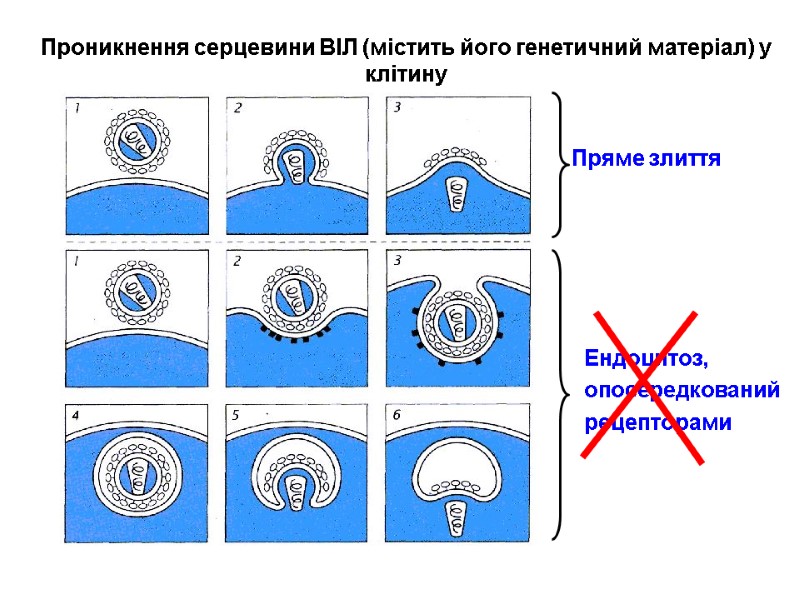
 Стилізоване зображення перерізу ВІЛ Діаметр – 100-120 нм
Стилізоване зображення перерізу ВІЛ Діаметр – 100-120 нм

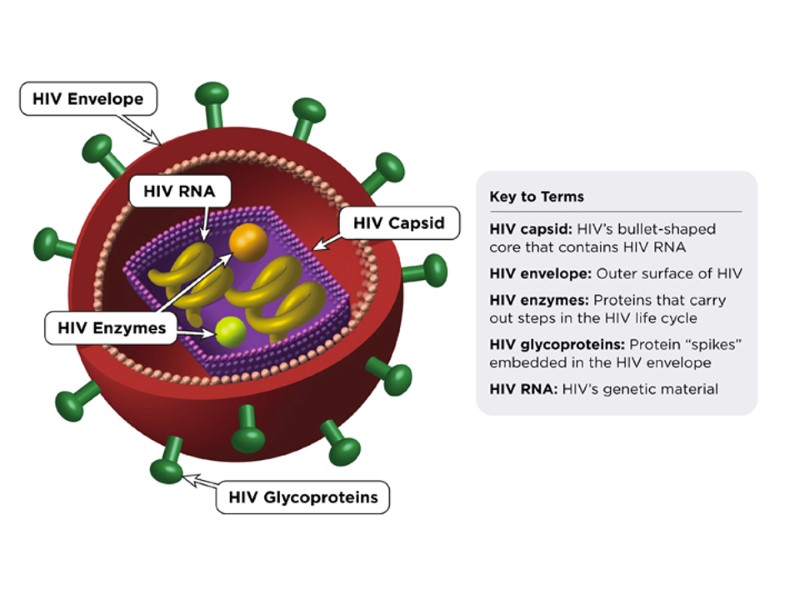
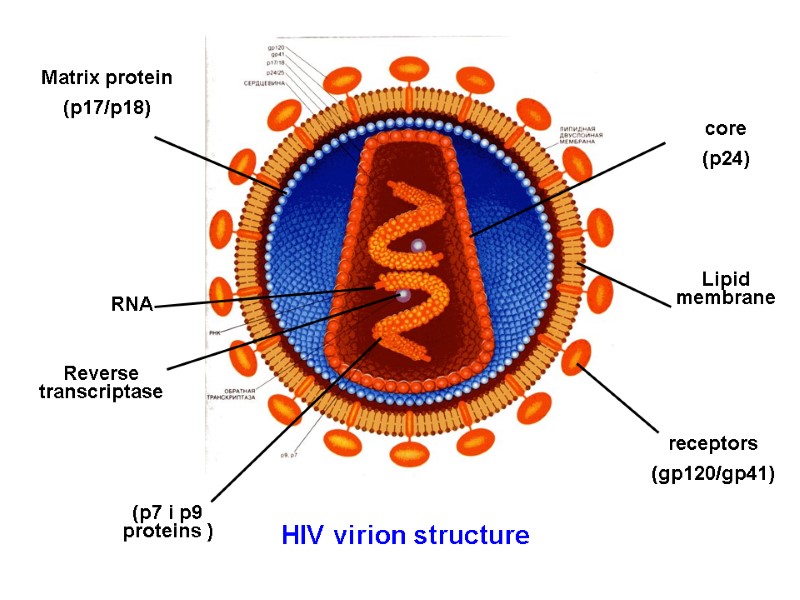
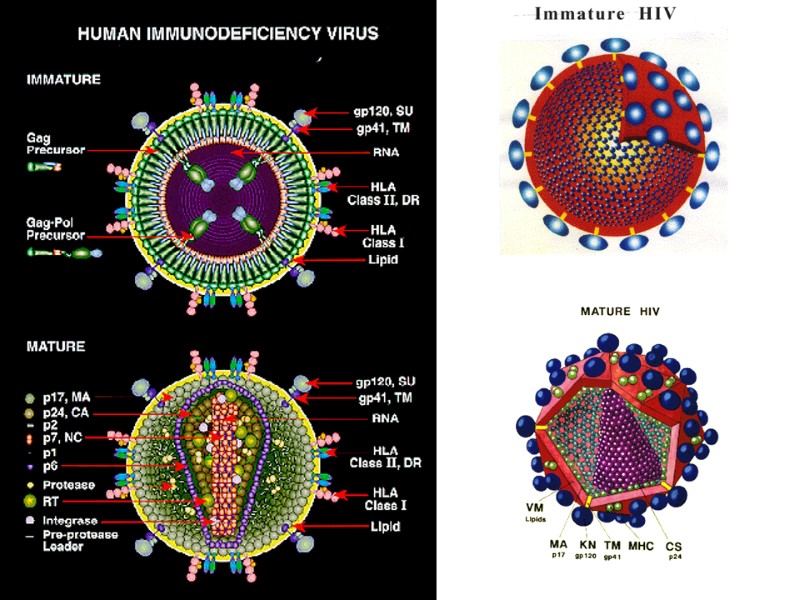
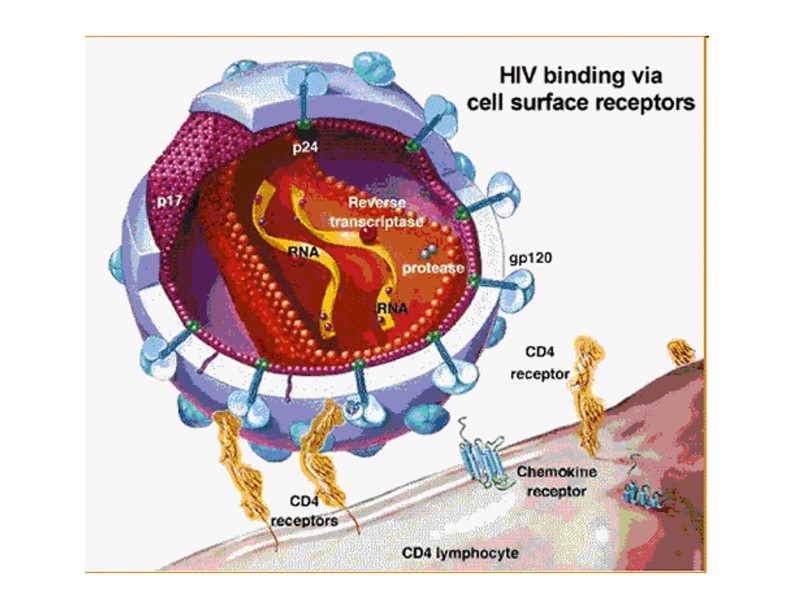
 HIV virion structure core (р24) Matrix protein (р17/р18) (р7 і р9 proteins ) Reverse transcriptase RNA Lipid membrane receptors (gр120/gр41)
HIV virion structure core (р24) Matrix protein (р17/р18) (р7 і р9 proteins ) Reverse transcriptase RNA Lipid membrane receptors (gр120/gр41)


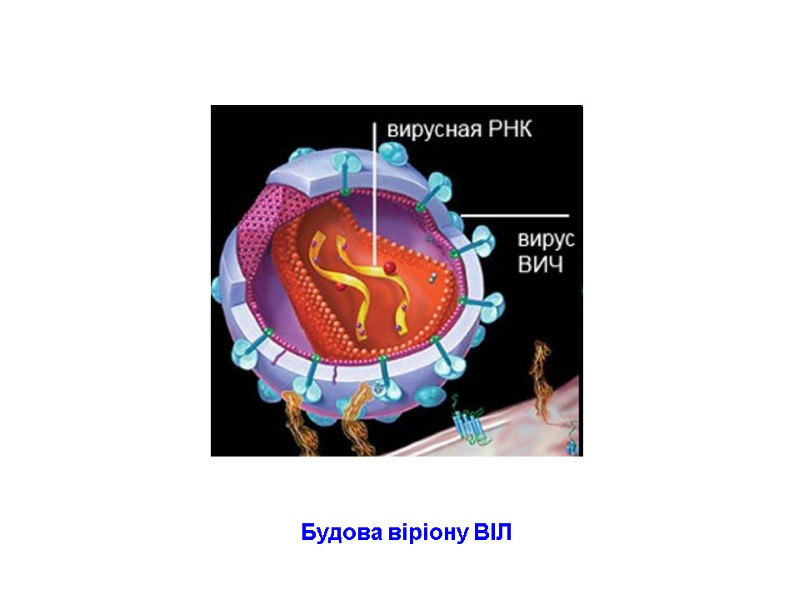
 Будова віріону ВІЛ
Будова віріону ВІЛ
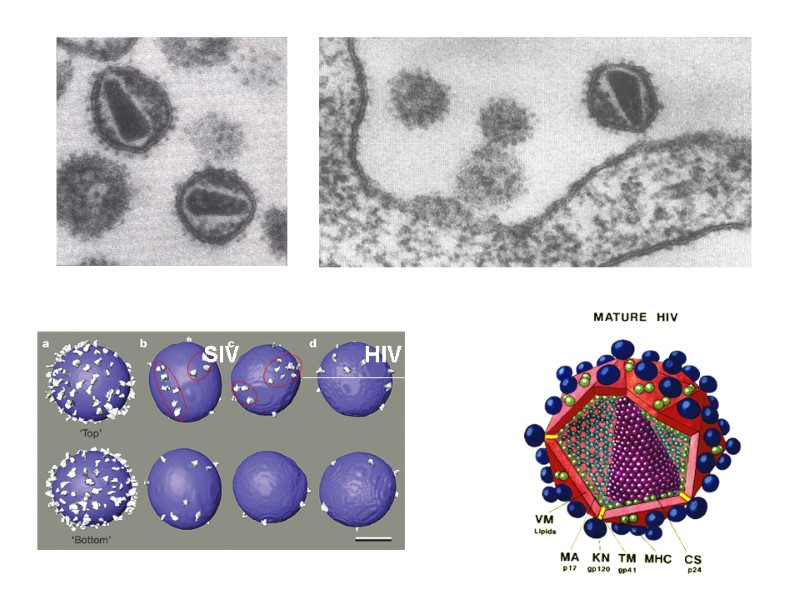
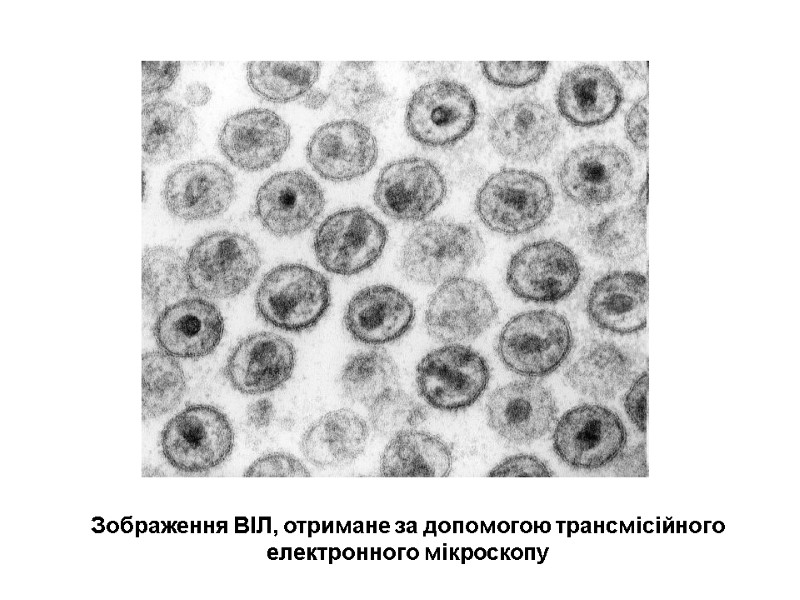
 Зображення ВІЛ, отримане за допомогою трансмісійного електронного мікроскопу
Зображення ВІЛ, отримане за допомогою трансмісійного електронного мікроскопу
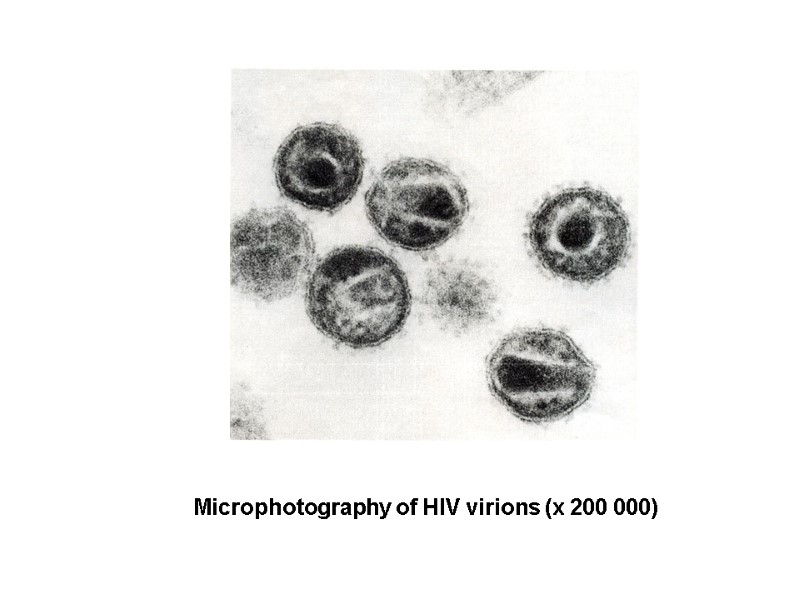
 Microphotography of HIV virions (х 200 000)
Microphotography of HIV virions (х 200 000)
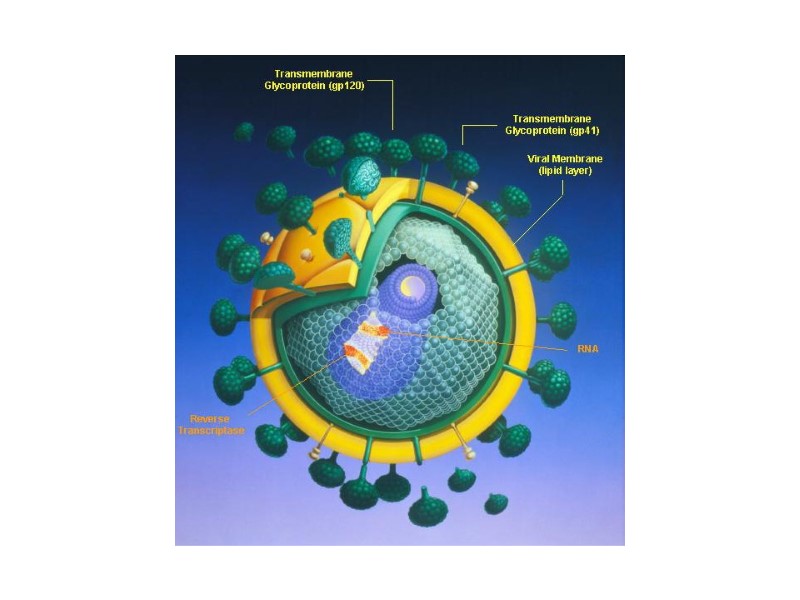
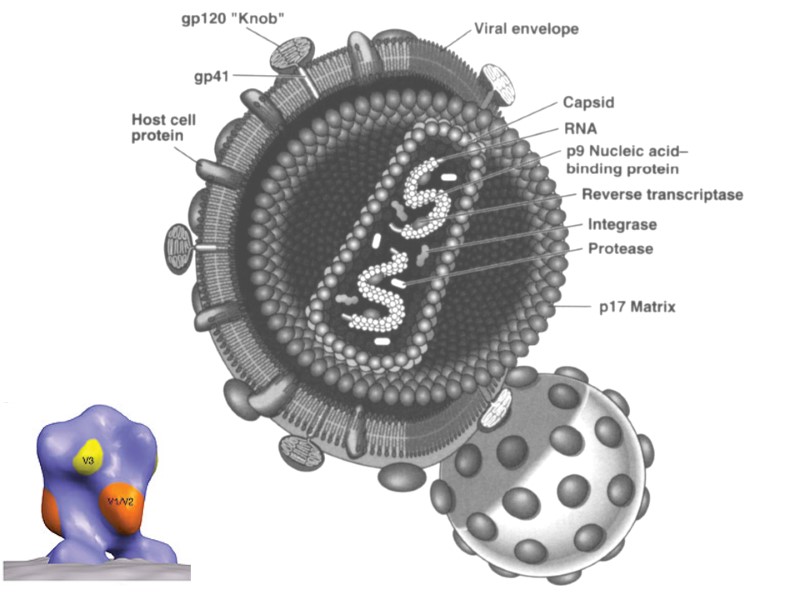
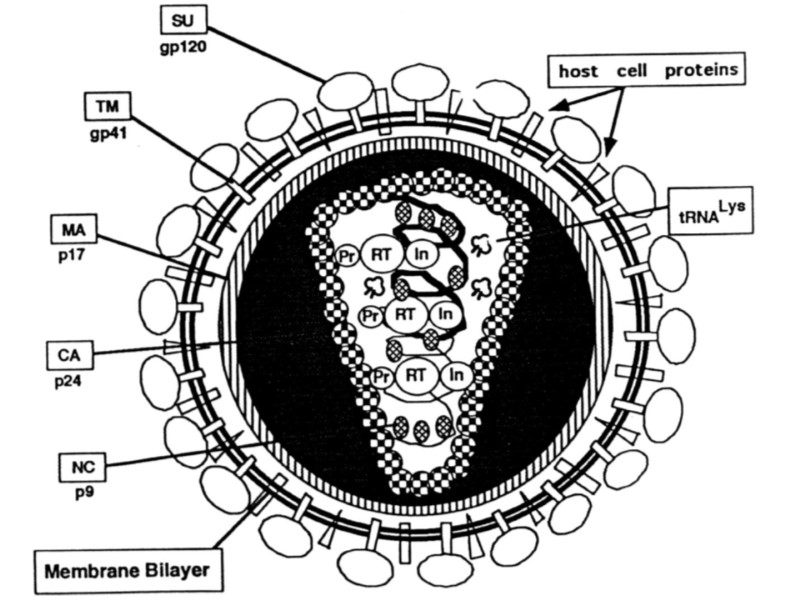
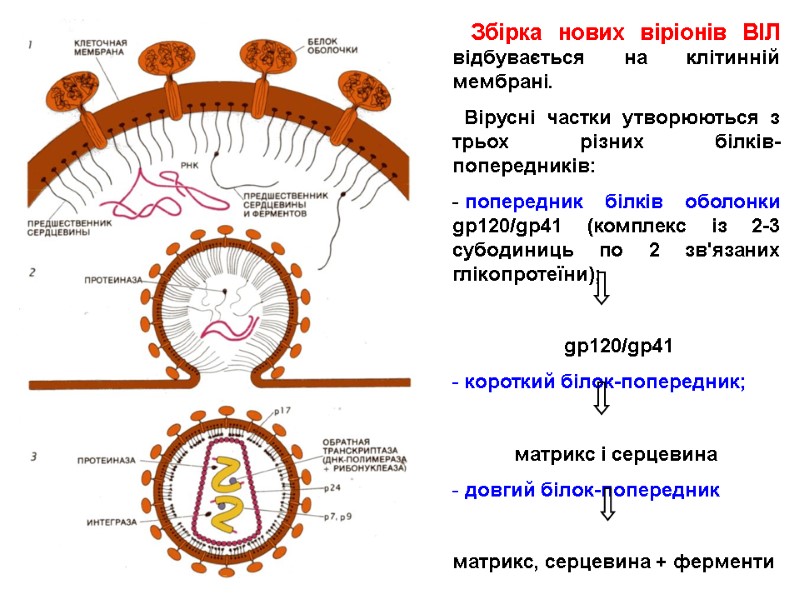
 The structure of HIV virion Supercapsid (“envelope”): Host-derived cell membrane - two layers of hospholipids (newly formed virus particle buds from the cell) Two envelope glycoproteins, gp120 (SU, surface)and gp41 (TM, transmembrane) - are required for attachment and entry of the virus into host cells. gp120 exist as a trimeric complex with gp41 on the virion surface. 2. Matrix proteins (consisting of the p17 protein), lie between the envelope and core. 3. Core (nucleocapsid) - contains the viral capsule protein p24 (CA, capsid) and surrounds: Genome - 2 single-stranded RNA copies are associated with low-molecular weight proteins р9 і р7 (NC, nucleocapsid). Enzymes needed for HIV replication: р66, RT – reverse transcriptase, р34, IN – integrase, p22, PR – protease. Переклад на наступному слайді
The structure of HIV virion Supercapsid (“envelope”): Host-derived cell membrane - two layers of hospholipids (newly formed virus particle buds from the cell) Two envelope glycoproteins, gp120 (SU, surface)and gp41 (TM, transmembrane) - are required for attachment and entry of the virus into host cells. gp120 exist as a trimeric complex with gp41 on the virion surface. 2. Matrix proteins (consisting of the p17 protein), lie between the envelope and core. 3. Core (nucleocapsid) - contains the viral capsule protein p24 (CA, capsid) and surrounds: Genome - 2 single-stranded RNA copies are associated with low-molecular weight proteins р9 і р7 (NC, nucleocapsid). Enzymes needed for HIV replication: р66, RT – reverse transcriptase, р34, IN – integrase, p22, PR – protease. Переклад на наступному слайді
 Структура частки ВІЛ Суперкапсид або зовнішня оболонка вірусу (“конверт”) - бімолекулярний шар ліпідів, походить з клітинної мембрани хазяїна, із вбудованими рецепторами - глікопротеїнами gp120 (SU, surface) та gp41 (TM, transmembrane), 1 gp41 + 3-5 gp120. 2. Матриксний білок р17 (MA, matrix) – між зовнішньою вірусною мембраною і серцевиною вірусу, ~ 2 тис. молекул білка утворюють шар товщиною 5-7 нм. 3. Нуклеокапсид або серцевина вірусу (кор) – під зовнішньою оболонкою, має форму зрізаного конусу й утворена білком р24 (CA, capsid) . 4. Геном - дві молекули позитивно-смислової (positive-sense, або (+)) РНК, з’єднані з низькомолекулярними білками р9 і р7 (NC, nucleocapsid) основного характеру - всередині серцевини. 5. Білки-ферменти - у серцевині віріону: р66, RT – зворотна транскриптаза, р34, IN – ендонуклеаза або інтеграза, Р22, PR – протеаза.
Структура частки ВІЛ Суперкапсид або зовнішня оболонка вірусу (“конверт”) - бімолекулярний шар ліпідів, походить з клітинної мембрани хазяїна, із вбудованими рецепторами - глікопротеїнами gp120 (SU, surface) та gp41 (TM, transmembrane), 1 gp41 + 3-5 gp120. 2. Матриксний білок р17 (MA, matrix) – між зовнішньою вірусною мембраною і серцевиною вірусу, ~ 2 тис. молекул білка утворюють шар товщиною 5-7 нм. 3. Нуклеокапсид або серцевина вірусу (кор) – під зовнішньою оболонкою, має форму зрізаного конусу й утворена білком р24 (CA, capsid) . 4. Геном - дві молекули позитивно-смислової (positive-sense, або (+)) РНК, з’єднані з низькомолекулярними білками р9 і р7 (NC, nucleocapsid) основного характеру - всередині серцевини. 5. Білки-ферменти - у серцевині віріону: р66, RT – зворотна транскриптаза, р34, IN – ендонуклеаза або інтеграза, Р22, PR – протеаза.
 Three proteins are associated with HIV capsid : - nef; - vif (7-20 molecules per virion). Protein vpr is located inside of the virion. HIV membrane (envelope) also contains host cell proteins: Human Leucocyte Antigens (HLA) I and II; Cellular adhesion molecules. Protein p6 Precise location in virion unknown, not generally present in other retroviruses, may aid in incorporation of Vpr into virion Переклад на наступному слайді
Three proteins are associated with HIV capsid : - nef; - vif (7-20 molecules per virion). Protein vpr is located inside of the virion. HIV membrane (envelope) also contains host cell proteins: Human Leucocyte Antigens (HLA) I and II; Cellular adhesion molecules. Protein p6 Precise location in virion unknown, not generally present in other retroviruses, may aid in incorporation of Vpr into virion Переклад на наступному слайді
 Protein p6 Локалізація у віріоні невідома, може сприяти введенню білку Vpr у віріон. Всередині віріону (можливо, за межами капсиду) виявлений білок vpr. У ліпідній мембрані ВІЛ також знаходяться мембранні білки клітин: лейкоцитарні антигени людини (HLA) класів I і II; молекули адгезії. З капсидом асоційовані білки: - nef; - vif (7-20 молекул vif на віріон).
Protein p6 Локалізація у віріоні невідома, може сприяти введенню білку Vpr у віріон. Всередині віріону (можливо, за межами капсиду) виявлений білок vpr. У ліпідній мембрані ВІЛ також знаходяться мембранні білки клітин: лейкоцитарні антигени людини (HLA) класів I і II; молекули адгезії. З капсидом асоційовані білки: - nef; - vif (7-20 молекул vif на віріон).
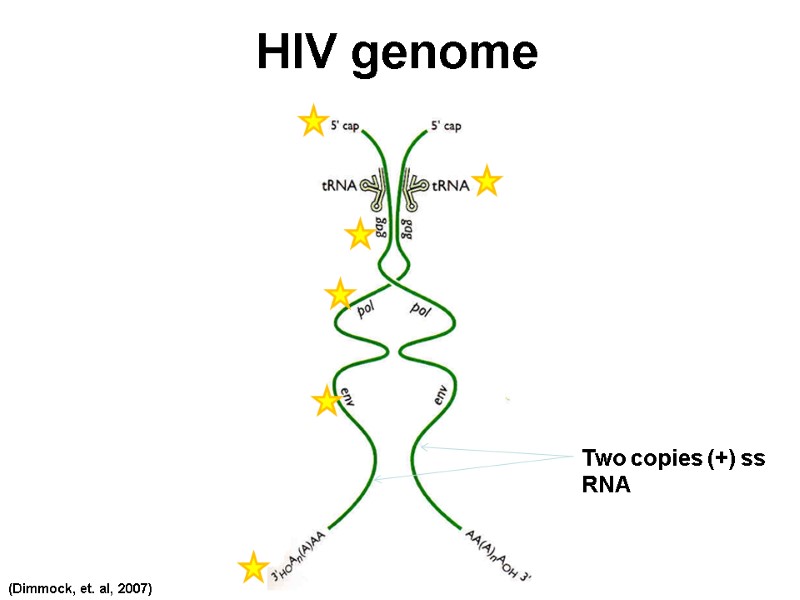
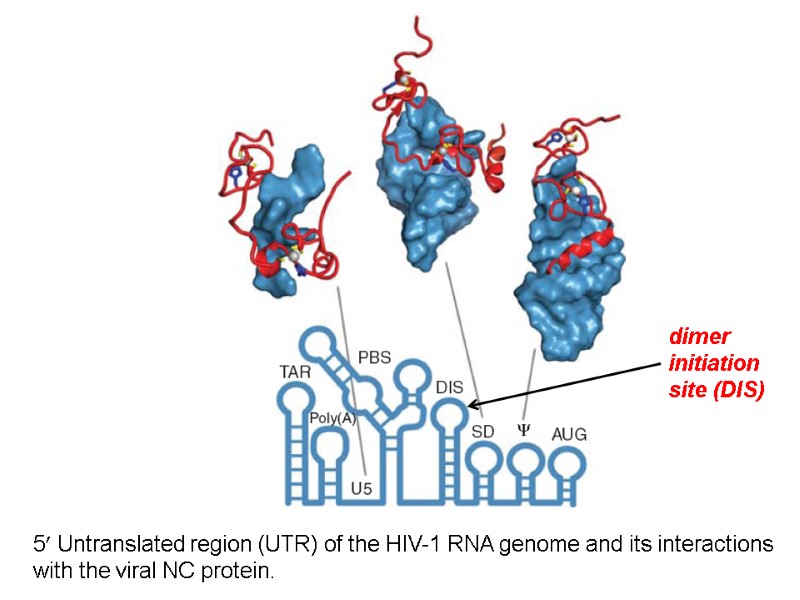
 HIV genome Two copies (+) ss RNA (Dimmock, et. al, 2007)
HIV genome Two copies (+) ss RNA (Dimmock, et. al, 2007)

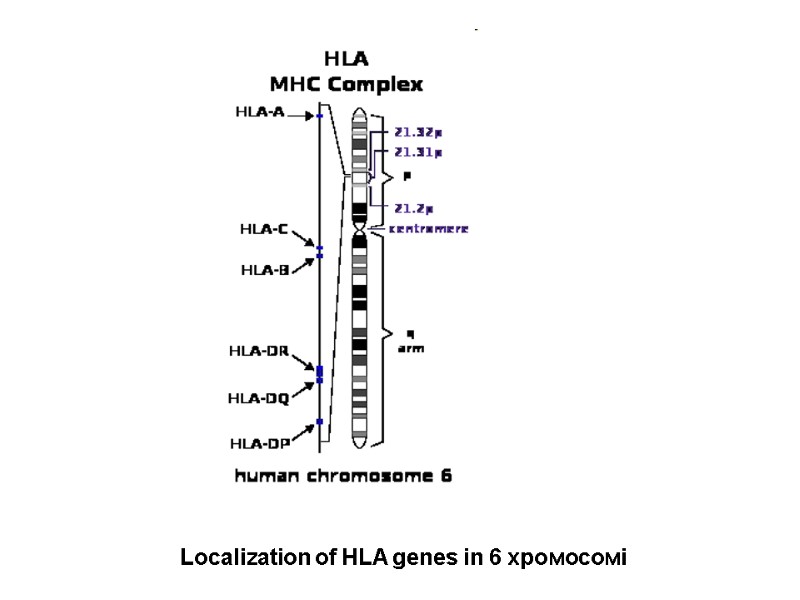
 Localization of HLA genes in 6 хромосомі
Localization of HLA genes in 6 хромосомі
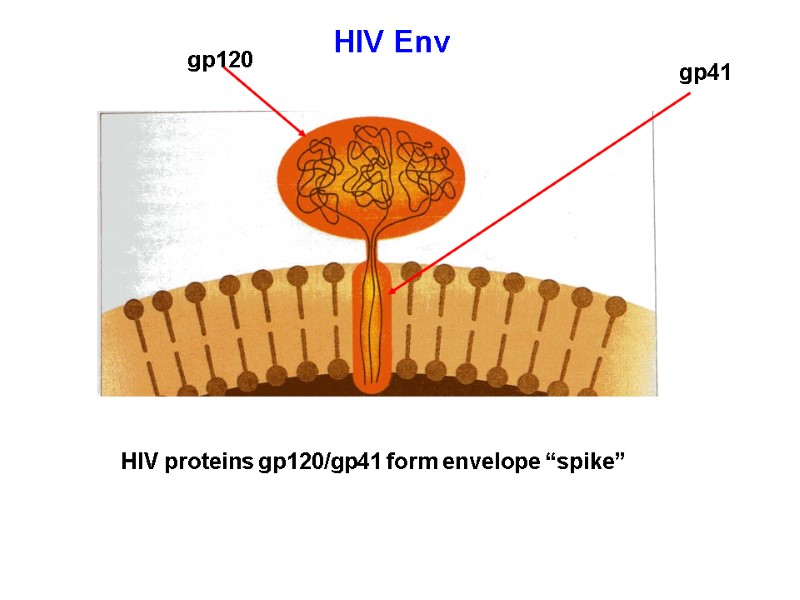
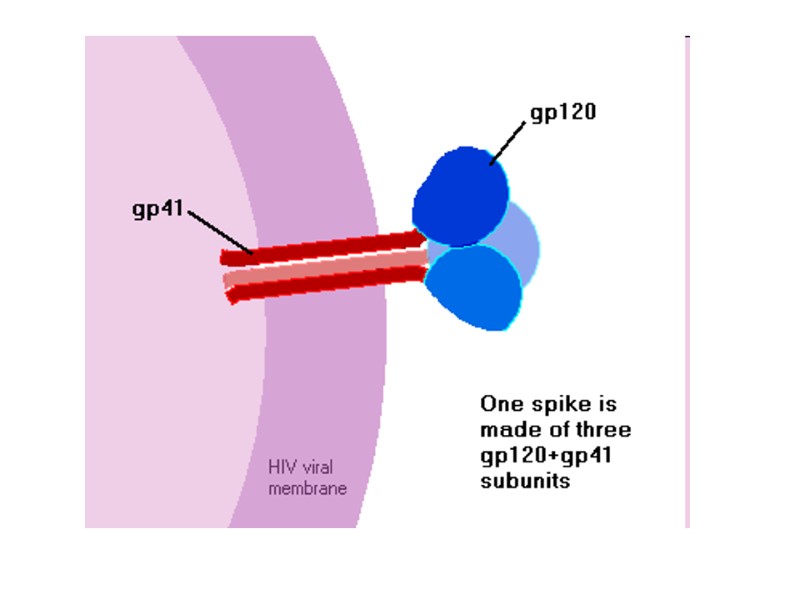
 HIV proteins gp120/gp41 form envelope “spike” HIV Env
HIV proteins gp120/gp41 form envelope “spike” HIV Env

 HIV Env Env itself consists of trimers of noncovalently bound gp120 and gp41 subunits. During replication, the integrated prophage is transcribed, producing Env mRNA that is read by endoplasmic reticulum ribosomes to produce an 845-870 amino acid precursor polypeptide. This precursor is modified with the addition of asparagine linked, high mannose content sugar chains, yielding the intermediate glycoprotein gp160. Переклад на наступному слайді
HIV Env Env itself consists of trimers of noncovalently bound gp120 and gp41 subunits. During replication, the integrated prophage is transcribed, producing Env mRNA that is read by endoplasmic reticulum ribosomes to produce an 845-870 amino acid precursor polypeptide. This precursor is modified with the addition of asparagine linked, high mannose content sugar chains, yielding the intermediate glycoprotein gp160. Переклад на наступному слайді
 HIV Env Env складається з тримерів нековалентно зв’язаних gp120 і gp41. Під час реплікації транскрибується mRNA гену Env, на клітинниз прибосомах синтезується попередник 845-870 а.к. Він глікозилюється за залишком Асп – приєднуються поліцукри, що складаються з манози, утворюється білок gp160.
HIV Env Env складається з тримерів нековалентно зв’язаних gp120 і gp41. Під час реплікації транскрибується mRNA гену Env, на клітинниз прибосомах синтезується попередник 845-870 а.к. Він глікозилюється за залишком Асп – приєднуються поліцукри, що складаються з манози, утворюється білок gp160.
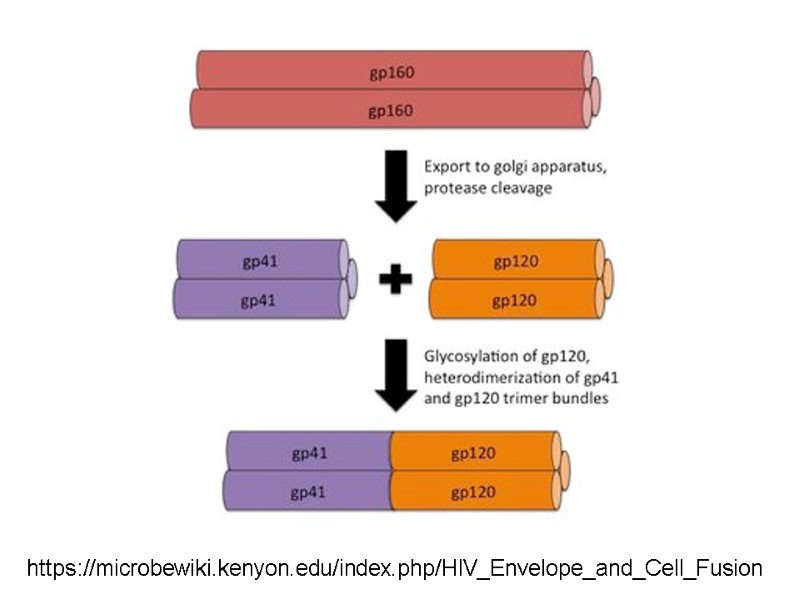
 HIV Env The gp160 glycoprotein forms homotrimers before being exported to the golgi apparatus, where host proteases digest the glycoprotein complex, yielding gp120 and gp41 subunits which remain as homotrimers. The gp120 and gp41 trimer bundles are further modified through N-glycosylation. This glycosylation step alone contributes a great deal to the variability of the Env protein structure. gp120 has around 24 potential N-glycosylation sites allowing for a wide variety of possible N-glycosylation combinations. Переклад на наступному слайді
HIV Env The gp160 glycoprotein forms homotrimers before being exported to the golgi apparatus, where host proteases digest the glycoprotein complex, yielding gp120 and gp41 subunits which remain as homotrimers. The gp120 and gp41 trimer bundles are further modified through N-glycosylation. This glycosylation step alone contributes a great deal to the variability of the Env protein structure. gp120 has around 24 potential N-glycosylation sites allowing for a wide variety of possible N-glycosylation combinations. Переклад на наступному слайді
 HIV Env gp160 утворює гомотример, експортується в апарат Гольджі, розщеплюється клітинною протеазою фурином, утворюються гомотримери gp120 і gp41. gp120 і gp41 далі підлягають N-глікозилюванню Білок gp120 глікозилюється в різних місцях (має 24 потенційних сайтів глікозилювання). Це глікозилювання (окрім мутацій гену Env), другою є причиною утворення різних варіантів структури gp120. Ці нові варіанти білка gp120 не впізнаються антитілами проти попередньо утворених варіантів цього білка.
HIV Env gp160 утворює гомотример, експортується в апарат Гольджі, розщеплюється клітинною протеазою фурином, утворюються гомотримери gp120 і gp41. gp120 і gp41 далі підлягають N-глікозилюванню Білок gp120 глікозилюється в різних місцях (має 24 потенційних сайтів глікозилювання). Це глікозилювання (окрім мутацій гену Env), другою є причиною утворення різних варіантів структури gp120. Ці нові варіанти білка gp120 не впізнаються антитілами проти попередньо утворених варіантів цього білка.
 HIV Env After N-glycosylation, the gp120 trimer bundles form noncovalent bonds with gp41 trimer bundles, yielding gp41-gp120 bundle heterodimers. Finally, the mature Env proteins are sent to the cell membrane and are embedded in virions budding from the cell. The high mutation rate of HIV means that mutations to these Env components are fairly common, resulting in a large number of progeny virions with Env glycoprotein complexes that fail to mature properly or simply fall out of the viral envelope. Переклад на наступному слайді
HIV Env After N-glycosylation, the gp120 trimer bundles form noncovalent bonds with gp41 trimer bundles, yielding gp41-gp120 bundle heterodimers. Finally, the mature Env proteins are sent to the cell membrane and are embedded in virions budding from the cell. The high mutation rate of HIV means that mutations to these Env components are fairly common, resulting in a large number of progeny virions with Env glycoprotein complexes that fail to mature properly or simply fall out of the viral envelope. Переклад на наступному слайді
 HIV Env Після N-глікозилювання тример gp120 нековалентно зв’язується з тримером gp41 з утворенням гетеродимеру gp41-gp120. Дозрілі білки Env надходять до мембрани клітини, розщеплюються клітинними ферментами до на gp120 і gp41 і включаються до мембрани віріону. Gp120 знаходиться на зовнішній поверхні мембрани, а gp41 заякорений у ліпідний бішар. Після цього віріон відбуньковується від клітини-хазяїна. Серед мутацій ВІЛ найчастіші саме мутації гену Env. Тому утворюються нащадки вірусу з порушеною структурою глікопротеїнових комплексів. Вони не можуть дозріти і навіть “випадають” з оболонки віріону.
HIV Env Після N-глікозилювання тример gp120 нековалентно зв’язується з тримером gp41 з утворенням гетеродимеру gp41-gp120. Дозрілі білки Env надходять до мембрани клітини, розщеплюються клітинними ферментами до на gp120 і gp41 і включаються до мембрани віріону. Gp120 знаходиться на зовнішній поверхні мембрани, а gp41 заякорений у ліпідний бішар. Після цього віріон відбуньковується від клітини-хазяїна. Серед мутацій ВІЛ найчастіші саме мутації гену Env. Тому утворюються нащадки вірусу з порушеною структурою глікопротеїнових комплексів. Вони не можуть дозріти і навіть “випадають” з оболонки віріону.
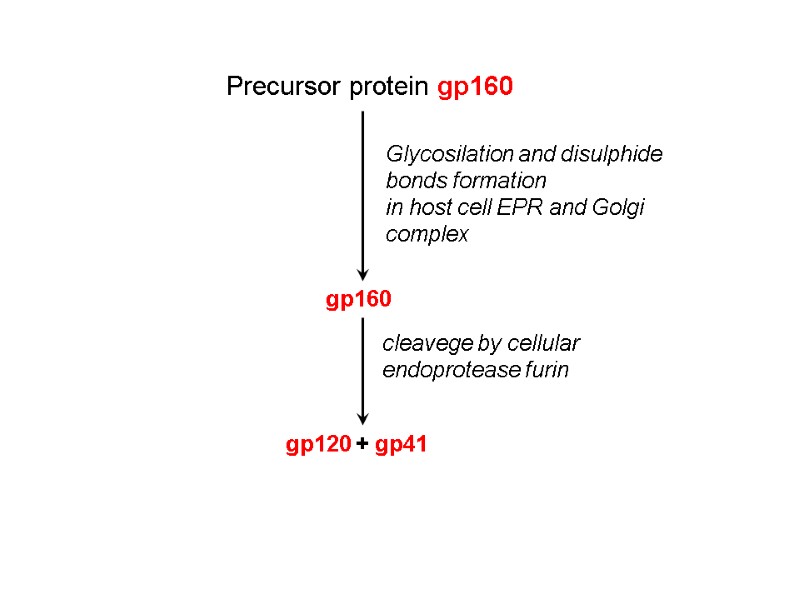
 Precursor protein gp160 Glycosilation and disulphide bonds formation in host cell EPR and Golgi complex gp120 + gp41 cleavege by cellular endoprotease furin gp160
Precursor protein gp160 Glycosilation and disulphide bonds formation in host cell EPR and Golgi complex gp120 + gp41 cleavege by cellular endoprotease furin gp160
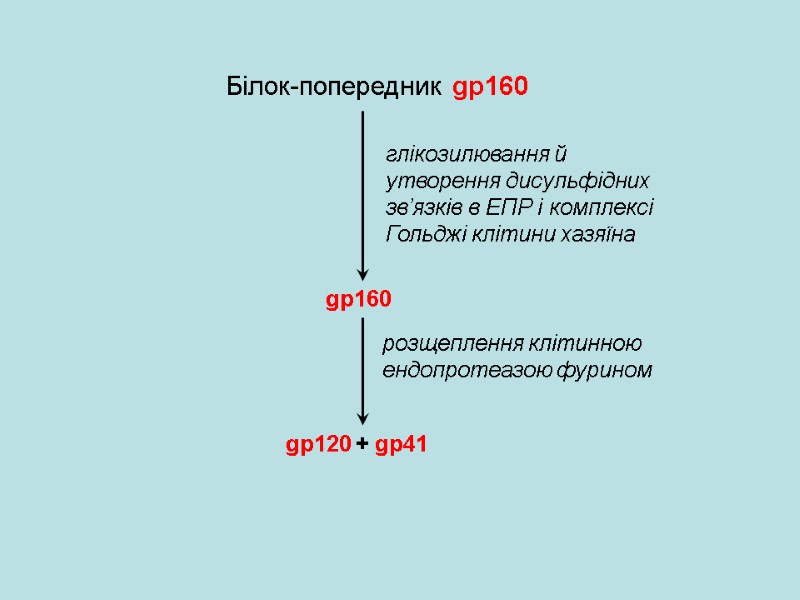
 Білок-попередник gp160 глікозилювання й утворення дисульфідних зв’язків в ЕПР і комплексі Гольджі клітини хазяїна gp120 + gp41 розщеплення клітинною ендопротеазою фурином gp160
Білок-попередник gp160 глікозилювання й утворення дисульфідних зв’язків в ЕПР і комплексі Гольджі клітини хазяїна gp120 + gp41 розщеплення клітинною ендопротеазою фурином gp160
 https://microbewiki.kenyon.edu/index.php/HIV_Envelope_and_Cell_Fusion
https://microbewiki.kenyon.edu/index.php/HIV_Envelope_and_Cell_Fusion
 When gp160 is synthesised in the cell, cellular enzymes add complex carbohydrates and turn it from a protein into a glycoprotein – hence the name “gp160″ rather than “p160″.) gp160 travels to the cell surface, where cellular enzymes again attack it, this time chopping into two pieces – gp120, and gp41. When new virus particles bud off from the host cell, gp120, and gp41 lie on opposite sides of the virus membrane. gp120 sits on the outside of the virus particle, forming the virus’s spikes, while gp41 sits just on the inside of the membrane – each gp41 being anchored to a gp120 through the membrane.
When gp160 is synthesised in the cell, cellular enzymes add complex carbohydrates and turn it from a protein into a glycoprotein – hence the name “gp160″ rather than “p160″.) gp160 travels to the cell surface, where cellular enzymes again attack it, this time chopping into two pieces – gp120, and gp41. When new virus particles bud off from the host cell, gp120, and gp41 lie on opposite sides of the virus membrane. gp120 sits on the outside of the virus particle, forming the virus’s spikes, while gp41 sits just on the inside of the membrane – each gp41 being anchored to a gp120 through the membrane.
 Коли gp160 синтезується в клітині, він надходить в ЕПР, де зазнає посттрансляційних модифікацій. Клітинний фермент приєднує вуглеводи (залишки манози) до нього з утворенням глікопротеїну (тому він називається не “p160″, “gp160″). Ендопротеаза фурин розщеплює gp160 на два білки gp120 and gp41. Вони надходять до мембрани клітини і вбудовуються в неї (gp120 знаходиться на поверхні, gp41 – заякорюється в мембрану).
Коли gp160 синтезується в клітині, він надходить в ЕПР, де зазнає посттрансляційних модифікацій. Клітинний фермент приєднує вуглеводи (залишки манози) до нього з утворенням глікопротеїну (тому він називається не “p160″, “gp160″). Ендопротеаза фурин розщеплює gp160 на два білки gp120 and gp41. Вони надходять до мембрани клітини і вбудовуються в неї (gp120 знаходиться на поверхні, gp41 – заякорюється в мембрану).
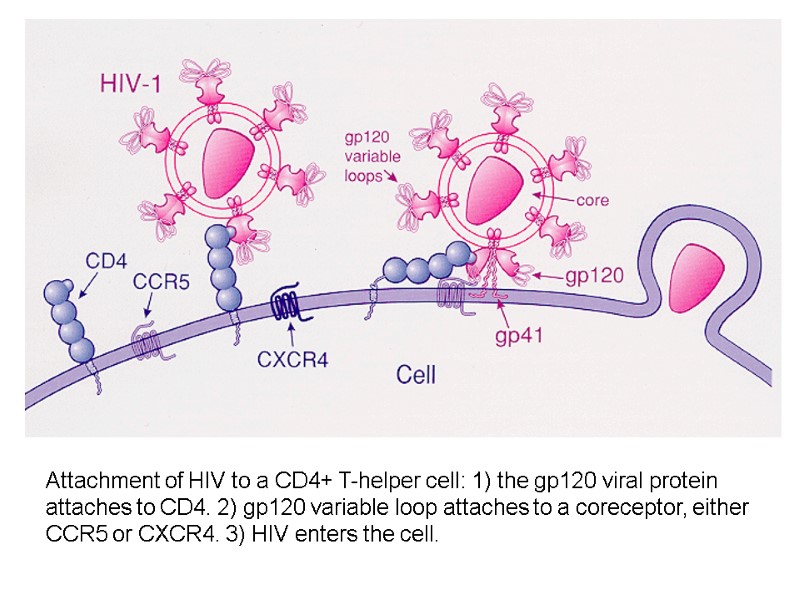
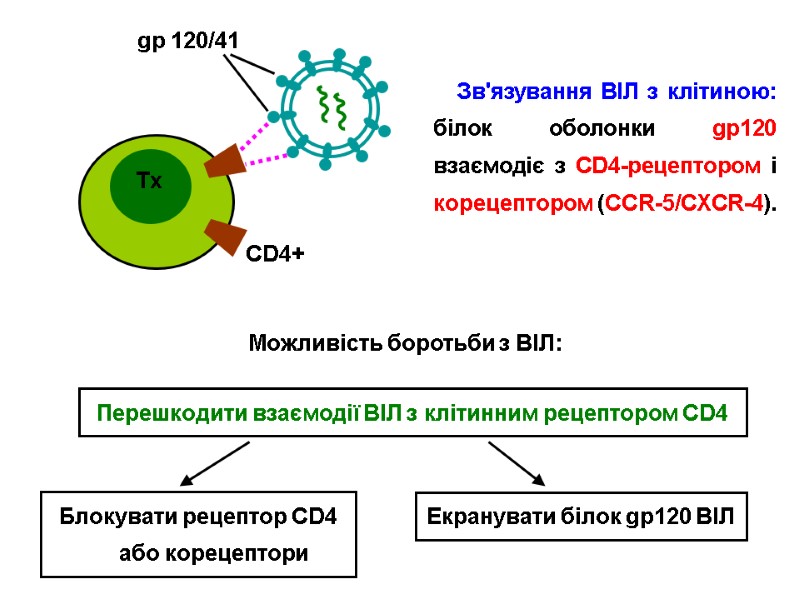
 HIV gp120 role: to seek receptors suitable for viral entry, to fix the viral particle to the cell, to assist and direct the injection of viral material. Переклад на наступному слайді
HIV gp120 role: to seek receptors suitable for viral entry, to fix the viral particle to the cell, to assist and direct the injection of viral material. Переклад на наступному слайді
 Роль HIV gp120 : взаємодія з рецепторами на лімфоцитах і макрофагах для проникнення в них ВІЛ, Фіксація вірусної частки на клітині, Сприяє проникненню вірусу в клітину.
Роль HIV gp120 : взаємодія з рецепторами на лімфоцитах і макрофагах для проникнення в них ВІЛ, Фіксація вірусної частки на клітині, Сприяє проникненню вірусу в клітину.

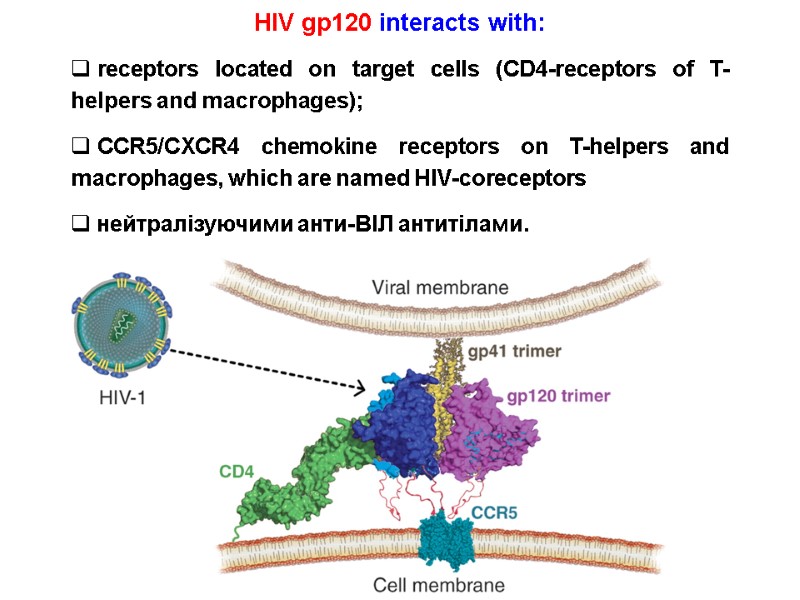
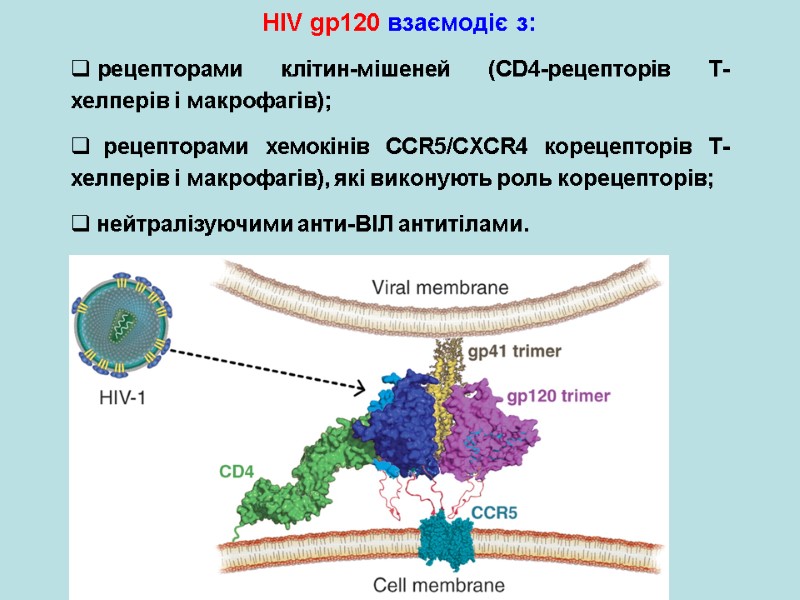
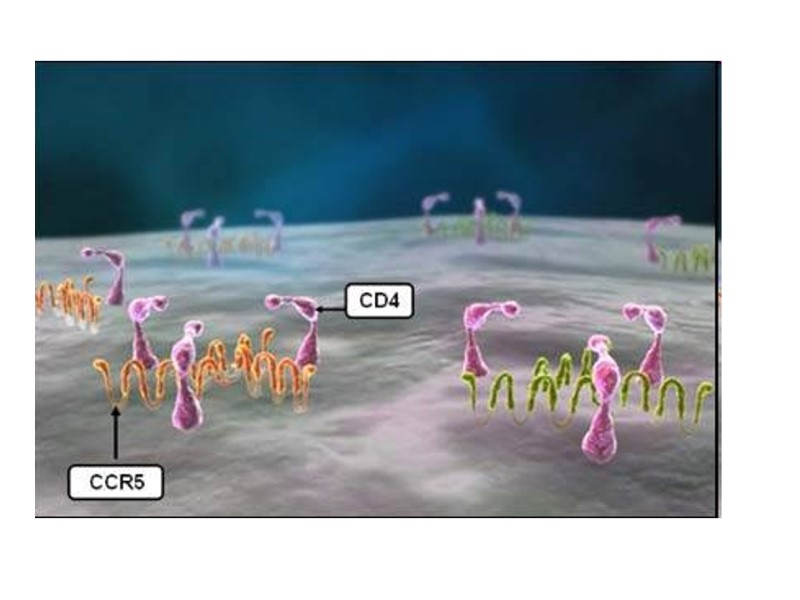
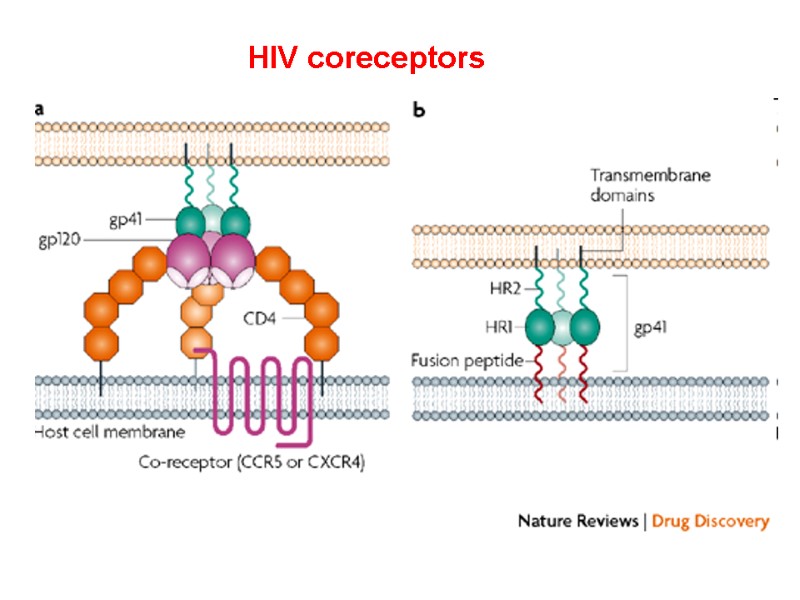
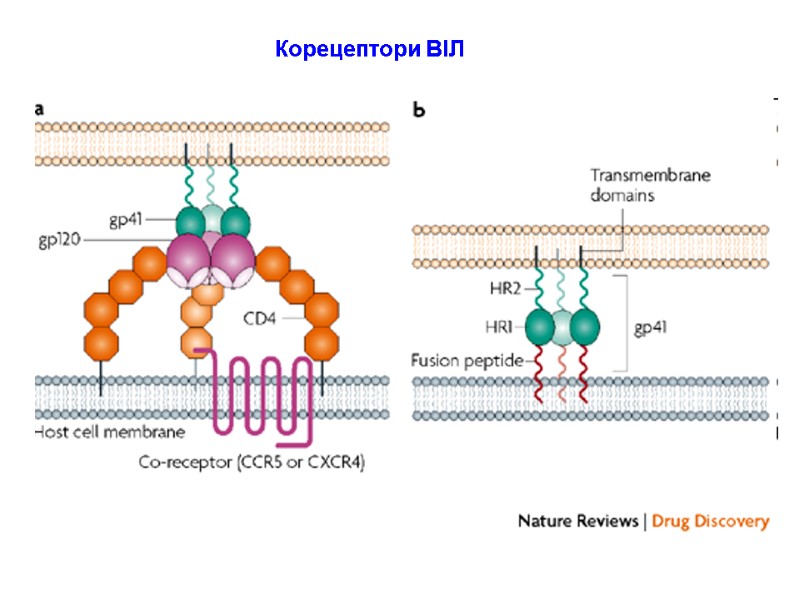
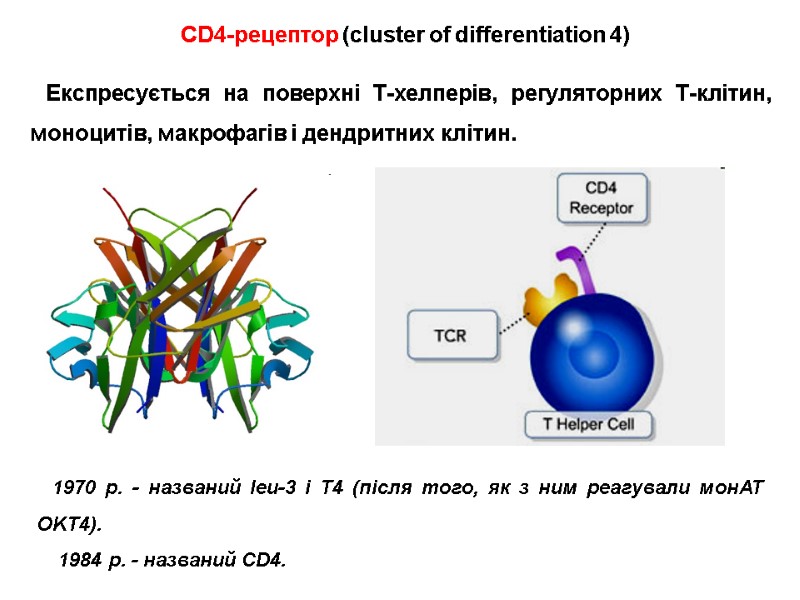
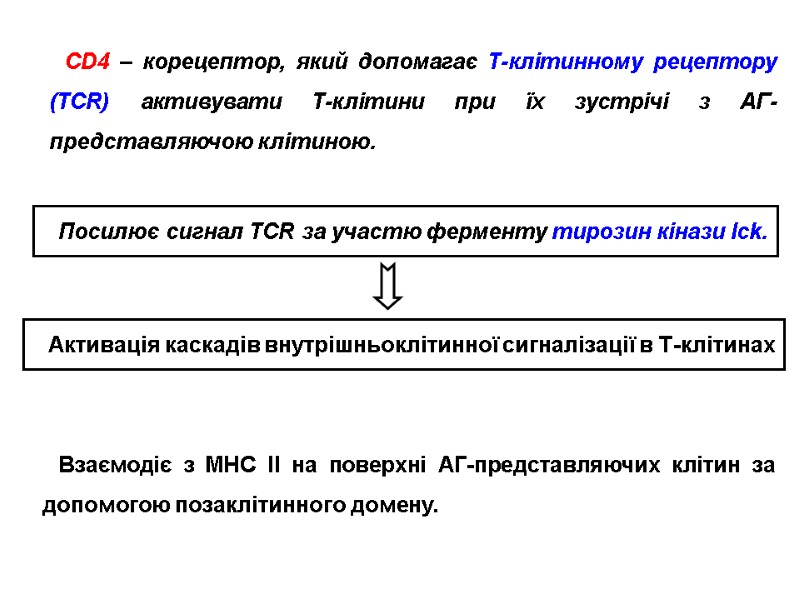
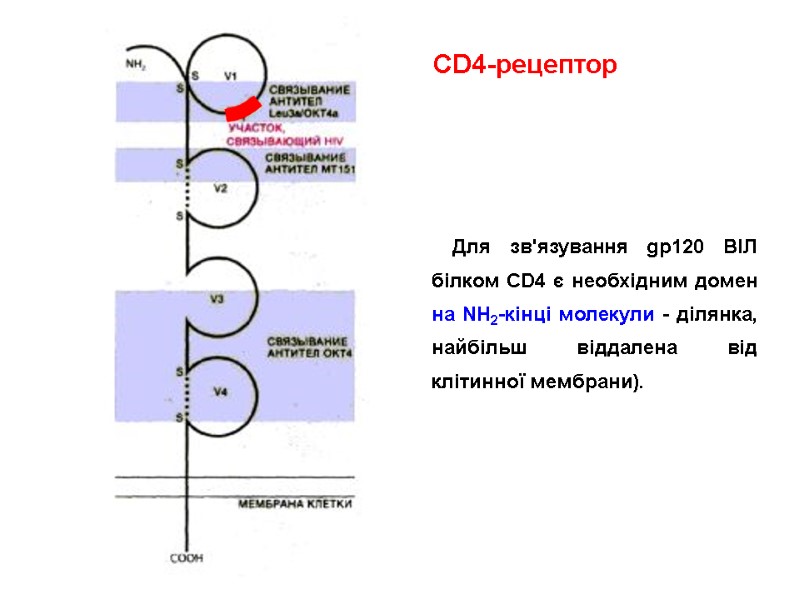
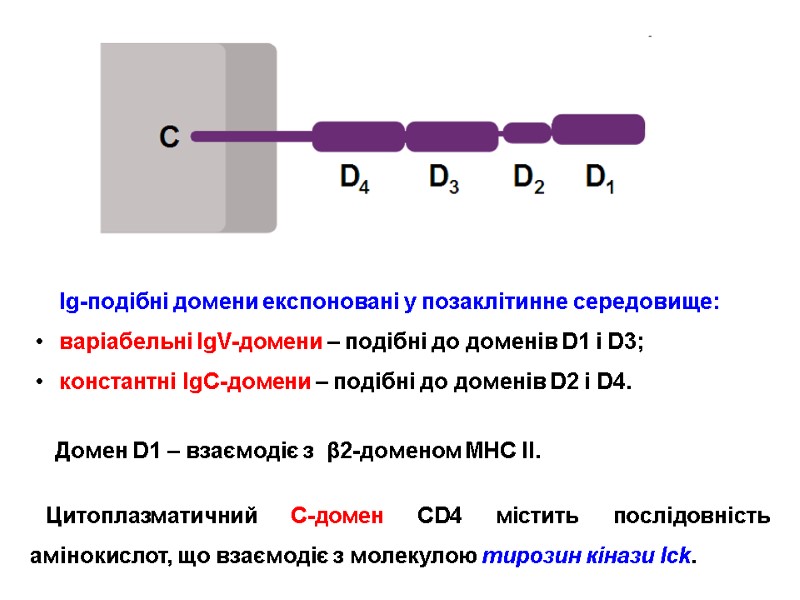
 HIV gp120 interacts with: receptors located on target cells (CD4-receptors of T-helpers and macrophages); CCR5/CXCR4 chemokine receptors on T-helpers and macrophages, which are named HIV-coreceptors нейтралізуючими анти-ВІЛ антитілами.
HIV gp120 interacts with: receptors located on target cells (CD4-receptors of T-helpers and macrophages); CCR5/CXCR4 chemokine receptors on T-helpers and macrophages, which are named HIV-coreceptors нейтралізуючими анти-ВІЛ антитілами.
 HIV gp120 взаємодіє з: рецепторами клітин-мішеней (CD4-рецепторів Т-хелперів і макрофагів); рецепторами хемокінів CCR5/CXCR4 корецепторів Т-хелперів і макрофагів), які виконують роль корецепторів; нейтралізуючими анти-ВІЛ антитілами.
HIV gp120 взаємодіє з: рецепторами клітин-мішеней (CD4-рецепторів Т-хелперів і макрофагів); рецепторами хемокінів CCR5/CXCR4 корецепторів Т-хелперів і макрофагів), які виконують роль корецепторів; нейтралізуючими анти-ВІЛ антитілами.
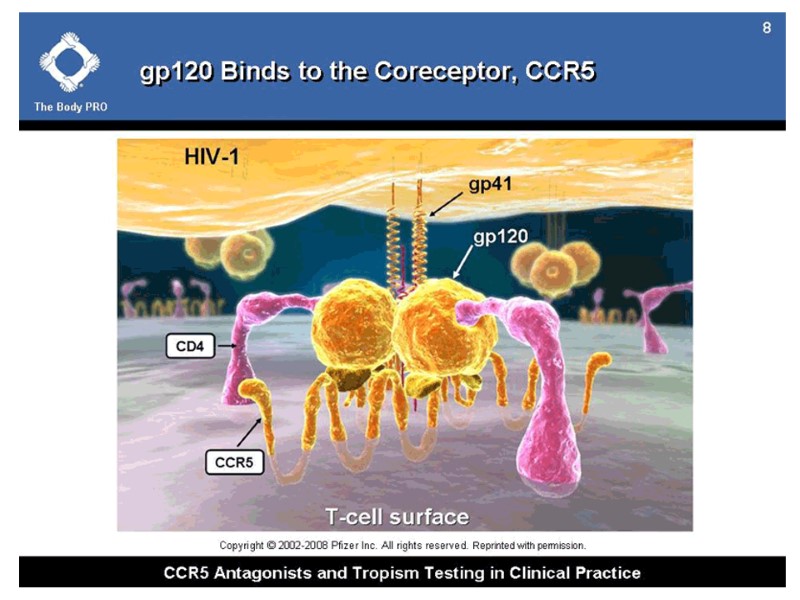
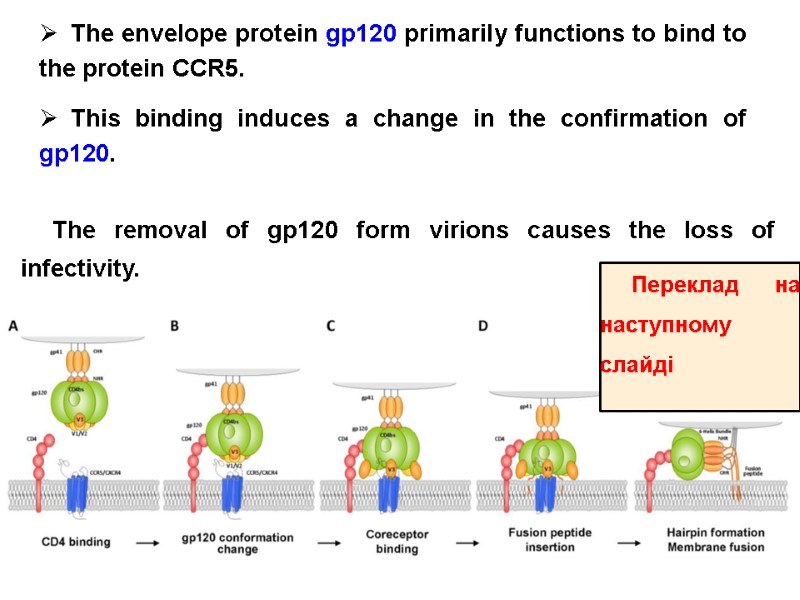
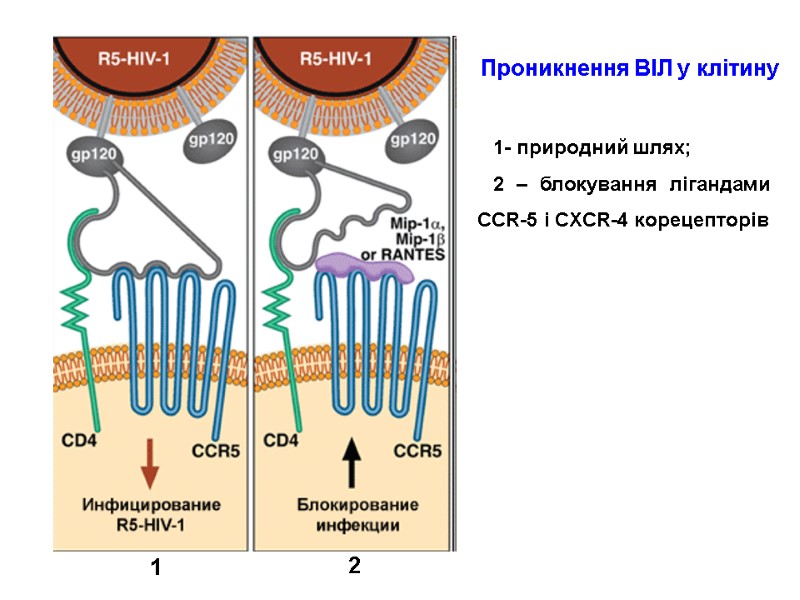
 The envelope protein gp120 primarily functions to bind to the protein CCR5. This binding induces a change in the confirmation of gp120. The removal of gp120 form virions causes the loss of infectivity. Переклад на наступному слайді
The envelope protein gp120 primarily functions to bind to the protein CCR5. This binding induces a change in the confirmation of gp120. The removal of gp120 form virions causes the loss of infectivity. Переклад на наступному слайді
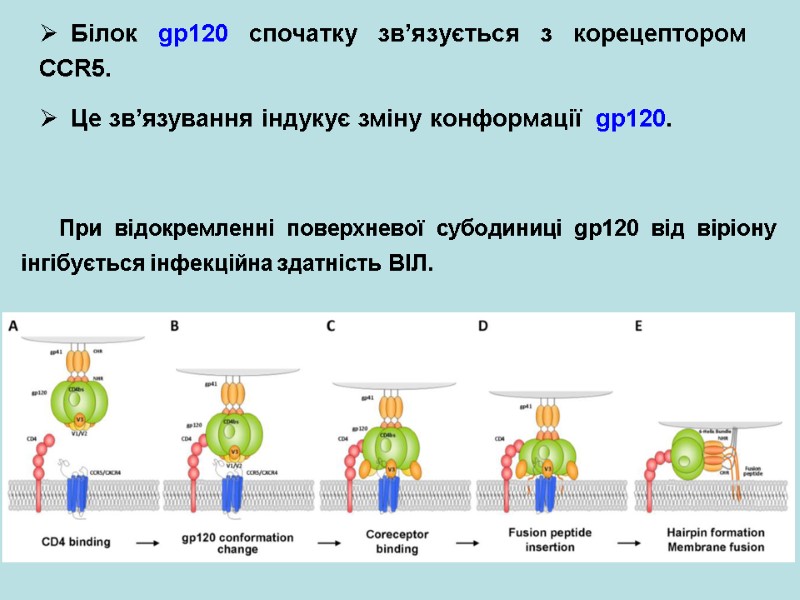
 Білок gp120 спочатку зв’язується з корецептором CCR5. Це зв’язування індукує зміну конформації gp120. При відокремленні поверхневої субодиниці gp120 від віріону інгібується інфекційна здатність ВІЛ.
Білок gp120 спочатку зв’язується з корецептором CCR5. Це зв’язування індукує зміну конформації gp120. При відокремленні поверхневої субодиниці gp120 від віріону інгібується інфекційна здатність ВІЛ.
 gp120 glycosylation HIV gp120 is covered with a number of glycosylation sites. The oligosaccharides are attached to them, which prevents detection by the immune system. Glycans at binding sites. Locations of CD4 and FAB X5 by fitting 2B4C to glycosylated gp120 model.
gp120 glycosylation HIV gp120 is covered with a number of glycosylation sites. The oligosaccharides are attached to them, which prevents detection by the immune system. Glycans at binding sites. Locations of CD4 and FAB X5 by fitting 2B4C to glycosylated gp120 model.
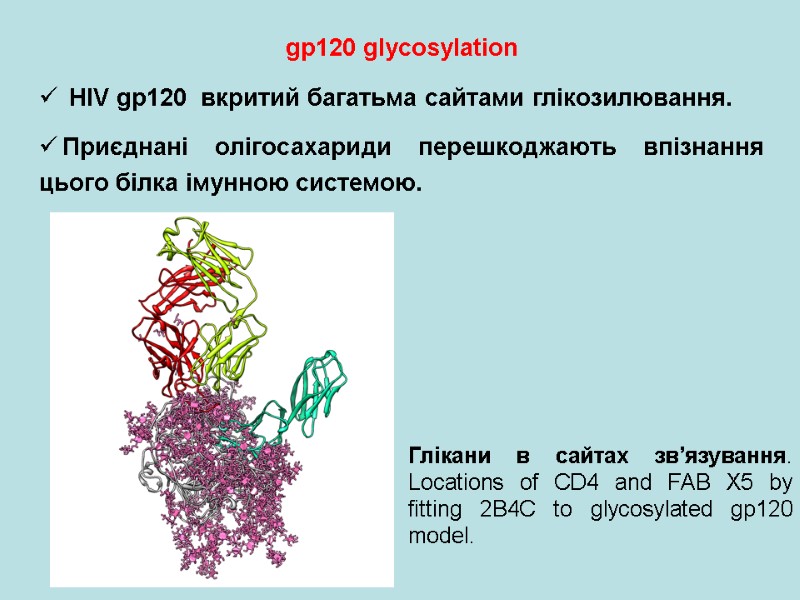
 gp120 glycosylation HIV gp120 вкритий багатьма сайтами глікозилювання. Приєднані олігосахариди перешкоджають впізнання цього білка імунною системою. Глікани в сайтах зв’язування. Locations of CD4 and FAB X5 by fitting 2B4C to glycosylated gp120 model.
gp120 glycosylation HIV gp120 вкритий багатьма сайтами глікозилювання. Приєднані олігосахариди перешкоджають впізнання цього білка імунною системою. Глікани в сайтах зв’язування. Locations of CD4 and FAB X5 by fitting 2B4C to glycosylated gp120 model.
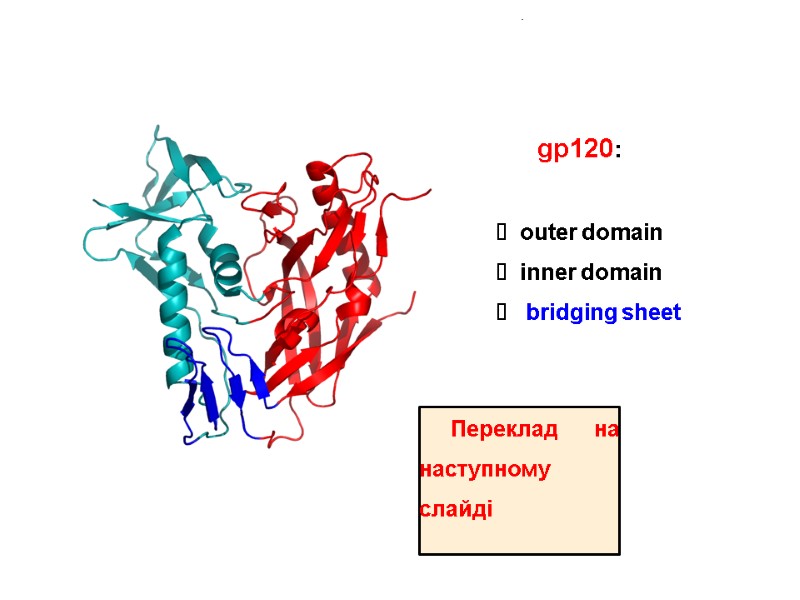
 gp120: outer domain inner domain bridging sheet Переклад на наступному слайді
gp120: outer domain inner domain bridging sheet Переклад на наступному слайді
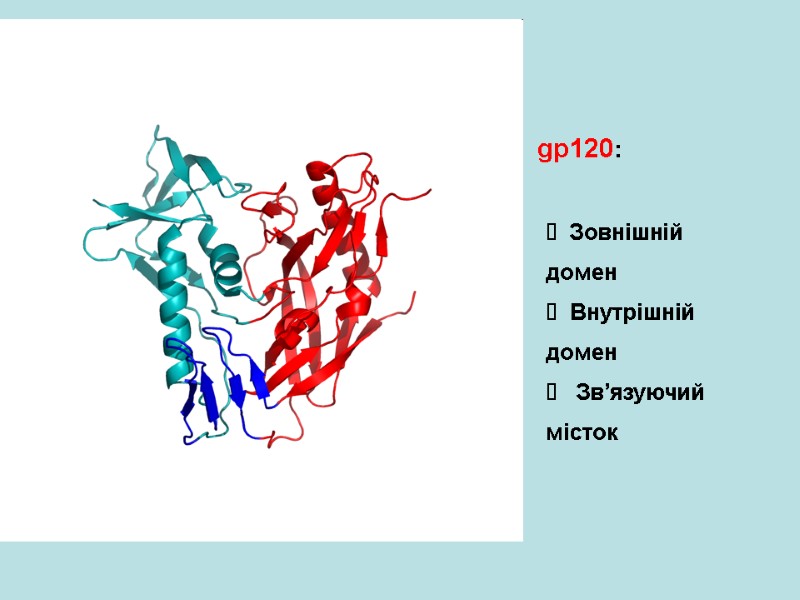
 gp120: Зовнішній домен Внутрішній домен Зв’язуючий місток
gp120: Зовнішній домен Внутрішній домен Зв’язуючий місток
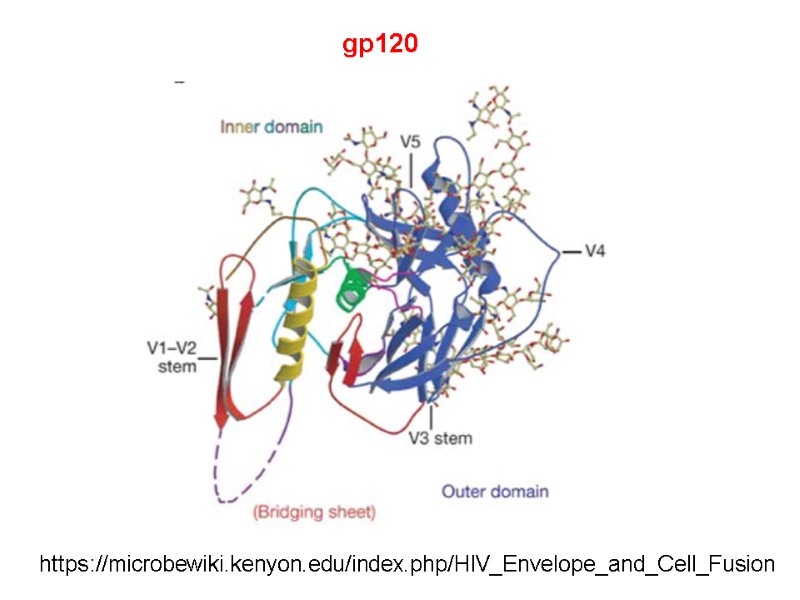
 gp120 https://microbewiki.kenyon.edu/index.php/HIV_Envelope_and_Cell_Fusion
gp120 https://microbewiki.kenyon.edu/index.php/HIV_Envelope_and_Cell_Fusion
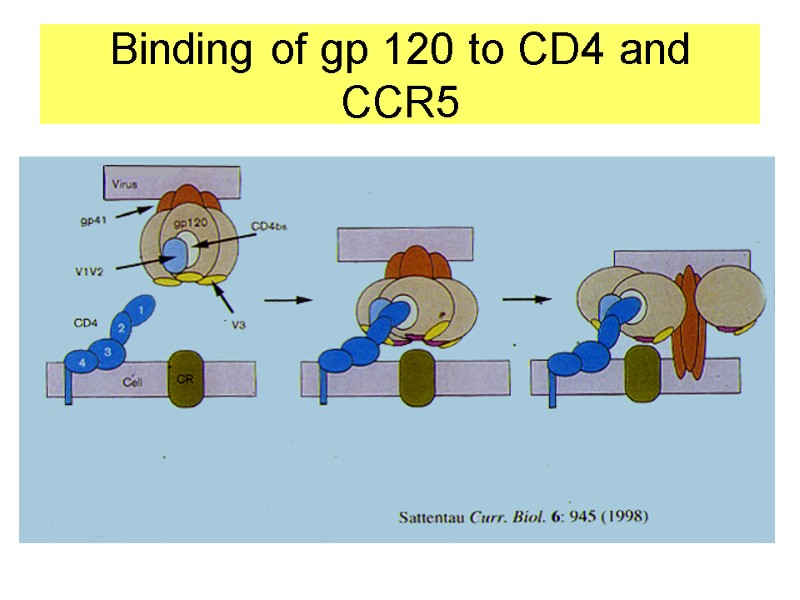
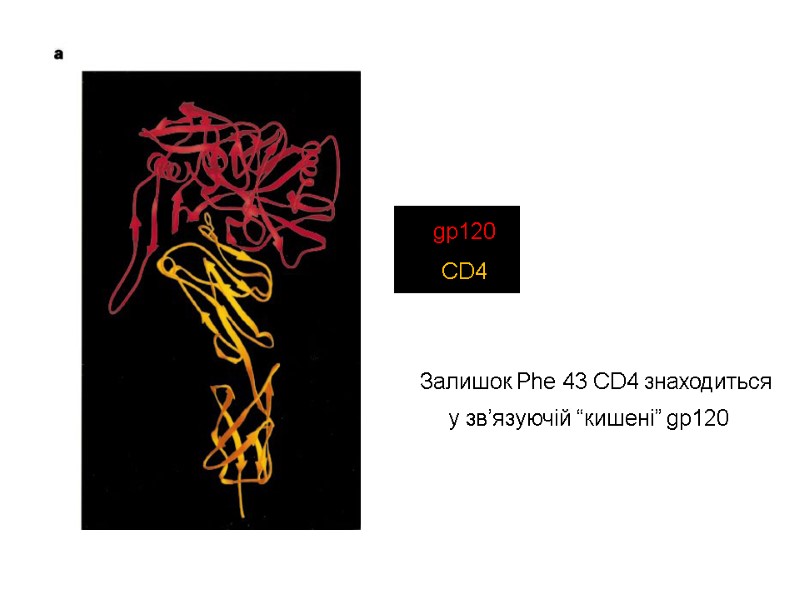
 Env gp120 serves as the target cell recognition and binding mechanism The outer domain of gp120 binds to the transmembrane protein CD4. This interaction occurs within a largely hydrophobic binding pocket composed of β-ribbon components β20-β21, the CD4 binding loop, and the α-helices of the outer domain. Binding to CD4 causes a series of conformational changes in the inner domain of gp120. That bring together the β-sheets of the so-called "bridging sheet", which connects the outer and inner domains. The compacted bridging sheet serves as a binding site for the chemokine receptor CCR5, or less frequently, CXCR4. These interactions serve to bring the virion closer to the target cell and cause the gp120 subunit to be shed from the viral envelope, exposing the gp41 subunit. Переклад на наступному слайді
Env gp120 serves as the target cell recognition and binding mechanism The outer domain of gp120 binds to the transmembrane protein CD4. This interaction occurs within a largely hydrophobic binding pocket composed of β-ribbon components β20-β21, the CD4 binding loop, and the α-helices of the outer domain. Binding to CD4 causes a series of conformational changes in the inner domain of gp120. That bring together the β-sheets of the so-called "bridging sheet", which connects the outer and inner domains. The compacted bridging sheet serves as a binding site for the chemokine receptor CCR5, or less frequently, CXCR4. These interactions serve to bring the virion closer to the target cell and cause the gp120 subunit to be shed from the viral envelope, exposing the gp41 subunit. Переклад на наступному слайді
 Env gp120 необхідний для впізнавання клітин-мішеней та зв’язування ВІЛ з ними Зовнійшній домен gp120 взаємодіє з CD4-рецептором. This interaction occurs within a largely hydrophobic binding pocket composed of β-ribbon components β20-β21, the CD4 binding loop, and the α-helices of the outer domain. Це викликає конформаційні зміни внутрішнього домену gp120. That bring together the β-sheets of the so-called "bridging sheet", which connects the outer and inner domains. Компактний зв’язуючий місток gp120 є сайтом зв’язування хемокінового рецептора CCR5 або рідше CXCR4. Завдяки цій взаємодії віріон приєднується до клітини-мішені й експонується білок gp41.
Env gp120 необхідний для впізнавання клітин-мішеней та зв’язування ВІЛ з ними Зовнійшній домен gp120 взаємодіє з CD4-рецептором. This interaction occurs within a largely hydrophobic binding pocket composed of β-ribbon components β20-β21, the CD4 binding loop, and the α-helices of the outer domain. Це викликає конформаційні зміни внутрішнього домену gp120. That bring together the β-sheets of the so-called "bridging sheet", which connects the outer and inner domains. Компактний зв’язуючий місток gp120 є сайтом зв’язування хемокінового рецептора CCR5 або рідше CXCR4. Завдяки цій взаємодії віріон приєднується до клітини-мішені й експонується білок gp41.
 gp120 structure: variable loop regions (V1-V5) - directly interact with target cell membrane proteins important for productive infection. conserved regions (С1-С5) 2 receptor sites of gp120: for CD4 receptor for coreceptors CCR5 and CXCR4 Переклад на наступному слайді
gp120 structure: variable loop regions (V1-V5) - directly interact with target cell membrane proteins important for productive infection. conserved regions (С1-С5) 2 receptor sites of gp120: for CD4 receptor for coreceptors CCR5 and CXCR4 Переклад на наступному слайді
 Структура gp120 : Варіабельні ділянки – петлі (V1-V5) – прямо взаємодіють з білками мембрани клітини-мішені. Консервативні ділянки (С1-С5) 2 рецепторні сайти gp120: for CD4 рецептор Корецептори CCR5 і CXCR4
Структура gp120 : Варіабельні ділянки – петлі (V1-V5) – прямо взаємодіють з білками мембрани клітини-мішені. Консервативні ділянки (С1-С5) 2 рецепторні сайти gp120: for CD4 рецептор Корецептори CCR5 і CXCR4

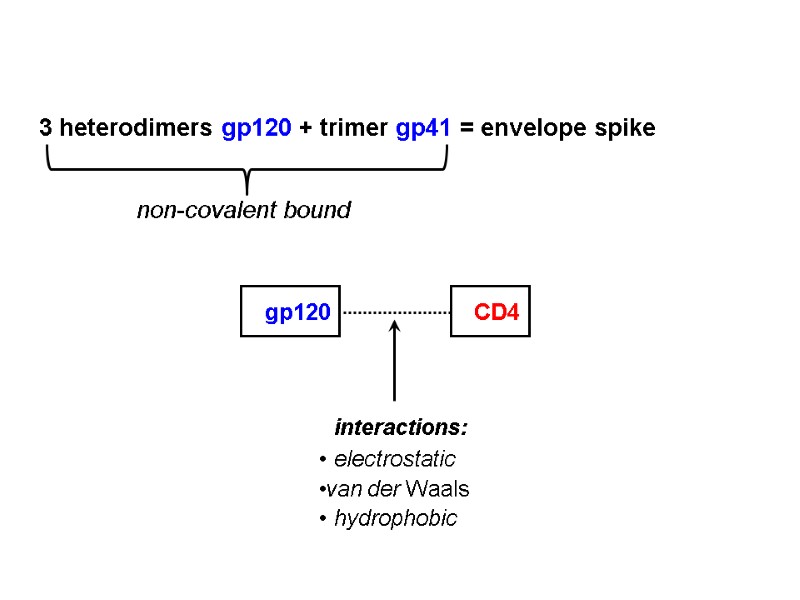
 3 heterodimers gp120 + trimer gp41 = envelope spike non-covalent bound gp120 CD4 interactions: electrostatic van der Waals hydrophobic
3 heterodimers gp120 + trimer gp41 = envelope spike non-covalent bound gp120 CD4 interactions: electrostatic van der Waals hydrophobic
 High mutation rate of HIV: The length of gp120 variable loops fluctuates greatly. The N-glycosylation state of the variable loops depends largely on mutations that add or remove potential N-glycosylation sites (PNGs). Together, the high variability in length and extent of glycosylation of the variable loops can have a profound impact on virion-target cell binding efficiency and specificity. Переклад на наступному слайді
High mutation rate of HIV: The length of gp120 variable loops fluctuates greatly. The N-glycosylation state of the variable loops depends largely on mutations that add or remove potential N-glycosylation sites (PNGs). Together, the high variability in length and extent of glycosylation of the variable loops can have a profound impact on virion-target cell binding efficiency and specificity. Переклад на наступному слайді
 Висока ступінь мітацій HIV визначається такими факторами: Варіації довжини варіабельних петель gp120. Ступінь N-глікозилювання варіабельних петель gp120 визначається мутаціями (додаються чи видаляються ділянки – потенційні місця N-глікозилювання). Отже, висока варіабельність довжини та ступеню N-глікозилювання варіабельних петель gp120 випливають на специфічність ВІЛ щодо зв’язування з клітинами-мішенями та їх інфікування.
Висока ступінь мітацій HIV визначається такими факторами: Варіації довжини варіабельних петель gp120. Ступінь N-глікозилювання варіабельних петель gp120 визначається мутаціями (додаються чи видаляються ділянки – потенційні місця N-глікозилювання). Отже, висока варіабельність довжини та ступеню N-глікозилювання варіабельних петель gp120 випливають на специфічність ВІЛ щодо зв’язування з клітинами-мішенями та їх інфікування.
 Examples of gp120 HIV mutations: mutations to V3 can cause the virion to prefer the co-receptor CXCR4 to CCR5; artificial deletion of V3 renders the virion incapable of binding to CCR5. Переклад на наступному слайді
Examples of gp120 HIV mutations: mutations to V3 can cause the virion to prefer the co-receptor CXCR4 to CCR5; artificial deletion of V3 renders the virion incapable of binding to CCR5. Переклад на наступному слайді
 Механізм, за яким імунна система не впізає варіабельні петлі gp120 Хоча петлі V1 і V2 є мішенню анти-ВІЛ антитіл, які продукуються організмом у відповідь на ВІЛ-інфекцію, висока ступінь мутацій gp120 призводить до того, що нові покоління вірусу із зміненими петлями gp120 не впізнаються наявними на той момент в організмі анти-ВІЛ антитілами. Явище “конформаційного маскування”.
Механізм, за яким імунна система не впізає варіабельні петлі gp120 Хоча петлі V1 і V2 є мішенню анти-ВІЛ антитіл, які продукуються організмом у відповідь на ВІЛ-інфекцію, висока ступінь мутацій gp120 призводить до того, що нові покоління вірусу із зміненими петлями gp120 не впізнаються наявними на той момент в організмі анти-ВІЛ антитілами. Явище “конформаційного маскування”.
 There are two mechanisms of conformational masking: additional N-glycosylation of the V1 and V2 loops; rearrangements of the existing V1 and V2 potential N-glycosylation sites (PNGs). These changes to the N-glycosylation pattern of V1 and V2 mask the viral Env protein, preventing recognition by neutralizing antibodies, which are produced targeting old V1/V2 sequences. Переклад на наступному слайді
There are two mechanisms of conformational masking: additional N-glycosylation of the V1 and V2 loops; rearrangements of the existing V1 and V2 potential N-glycosylation sites (PNGs). These changes to the N-glycosylation pattern of V1 and V2 mask the viral Env protein, preventing recognition by neutralizing antibodies, which are produced targeting old V1/V2 sequences. Переклад на наступному слайді
 Два механізми “конформаційного маскування”. : Додаткове N-глікозилювання петель V1 і V2; Перегрупування вже існуючих потенційних сайтів N-глікозилювання (potential N-glycosylation sites, PNGs) у петлях V1 and V2. Ці зміни у N-глікозилюванні петель V1 і V2 маскують білок Env (gp120), й антитіла проти послідовностей у петлях V1 і V2 «старих» (попередніх) штамів ВІЛ не впізнають їх.
Два механізми “конформаційного маскування”. : Додаткове N-глікозилювання петель V1 і V2; Перегрупування вже існуючих потенційних сайтів N-глікозилювання (potential N-glycosylation sites, PNGs) у петлях V1 and V2. Ці зміни у N-глікозилюванні петель V1 і V2 маскують білок Env (gp120), й антитіла проти послідовностей у петлях V1 і V2 «старих» (попередніх) штамів ВІЛ не впізнають їх.
 Кор gp120 екранований вуглеводами. Неекранована частина gp120 взаємодіє з gp41. Консервативний сайт зв’язування з CD4 «заглиблений» і важко доступний для антитіл.
Кор gp120 екранований вуглеводами. Неекранована частина gp120 взаємодіє з gp41. Консервативний сайт зв’язування з CD4 «заглиблений» і важко доступний для антитіл.
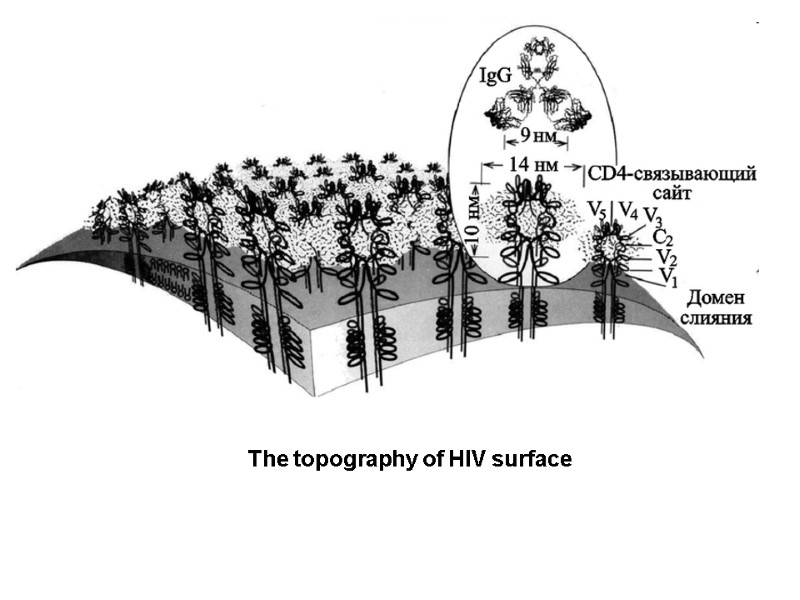
 The topography of HIV surface
The topography of HIV surface
 HIV infection of CD4+ T cells in the gut
HIV infection of CD4+ T cells in the gut
 HIV infection of CD4+ T cells in the gut HIV virions able to interact with α4β7 integrin on CD4+ T cells in the gut. HIV therefore have a better chance of binding to CD4+ T target cells. The natural ligand for α4β7 integrin is mucosal addressin cell adhesion molecule-1 (MAdCAM-1). α4β7 causes T-cell migration to the gut-associated lymphoid tissue (GALT), an important T-cell production center in the human gastrointestinal tract. The high density of CD4+ T-cells in this region allows for relatively easy intercellular transmission of the virus, making the GALT a hotbed of HIV activity. Переклад на наступному слайді
HIV infection of CD4+ T cells in the gut HIV virions able to interact with α4β7 integrin on CD4+ T cells in the gut. HIV therefore have a better chance of binding to CD4+ T target cells. The natural ligand for α4β7 integrin is mucosal addressin cell adhesion molecule-1 (MAdCAM-1). α4β7 causes T-cell migration to the gut-associated lymphoid tissue (GALT), an important T-cell production center in the human gastrointestinal tract. The high density of CD4+ T-cells in this region allows for relatively easy intercellular transmission of the virus, making the GALT a hotbed of HIV activity. Переклад на наступному слайді
 Інфікування ВІЛ CD4+ T клітин у кишечнику HIV віріони можуть взаємодіяти з α4β7 інтегрином на поверхні CD4+ T клітин у кишечнику. Тому ВІЛ має кращі шанси зв’язатися та інфікувати ці CD4+ T клітини. Природний ліганд α4β7 інтегрину є молекули MAdCAM-1 (mucosal addressin cell adhesion molecule-1). α4β7 інтегрин призводить до міграції Т-клітин у лімфоїдну тканину, асоційовану з кишечником (gut-associated lymphoid tissue, GALT), що є важливим центром продукції Т-клітин у шлунково-кишковому тракті. Висока щільність CD4+ T клітин у цій ділянці сприяє швидкому поширенню ВІЛ, і тому кишечник стає “гарячою точкою” розмноження ВІЛ.
Інфікування ВІЛ CD4+ T клітин у кишечнику HIV віріони можуть взаємодіяти з α4β7 інтегрином на поверхні CD4+ T клітин у кишечнику. Тому ВІЛ має кращі шанси зв’язатися та інфікувати ці CD4+ T клітини. Природний ліганд α4β7 інтегрину є молекули MAdCAM-1 (mucosal addressin cell adhesion molecule-1). α4β7 інтегрин призводить до міграції Т-клітин у лімфоїдну тканину, асоційовану з кишечником (gut-associated lymphoid tissue, GALT), що є важливим центром продукції Т-клітин у шлунково-кишковому тракті. Висока щільність CD4+ T клітин у цій ділянці сприяє швидкому поширенню ВІЛ, і тому кишечник стає “гарячою точкою” розмноження ВІЛ.
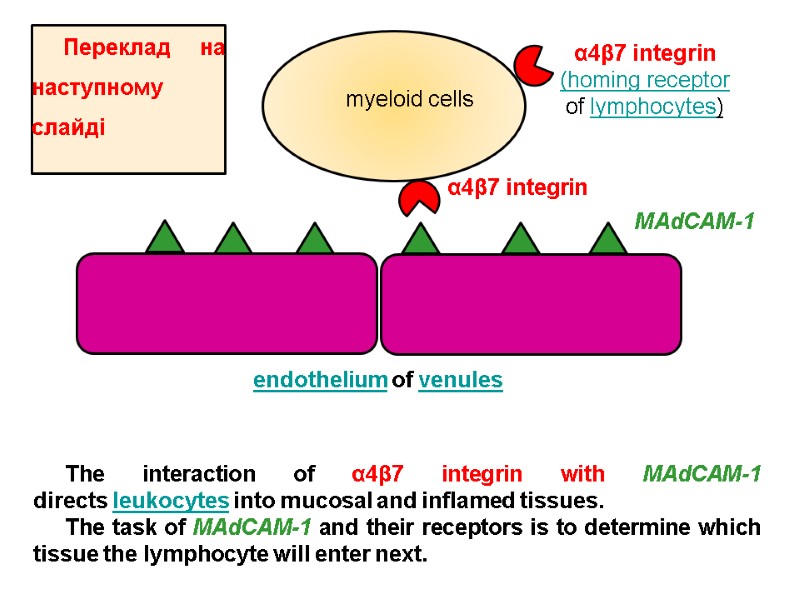
 α4β7 integrin MAdCAM-1 myeloid cells endothelium of venules α4β7 integrin (homing receptor of lymphocytes) The interaction of α4β7 integrin with MAdCAM-1 directs leukocytes into mucosal and inflamed tissues. The task of MAdCAM-1 and their receptors is to determine which tissue the lymphocyte will enter next. Переклад на наступному слайді
α4β7 integrin MAdCAM-1 myeloid cells endothelium of venules α4β7 integrin (homing receptor of lymphocytes) The interaction of α4β7 integrin with MAdCAM-1 directs leukocytes into mucosal and inflamed tissues. The task of MAdCAM-1 and their receptors is to determine which tissue the lymphocyte will enter next. Переклад на наступному слайді
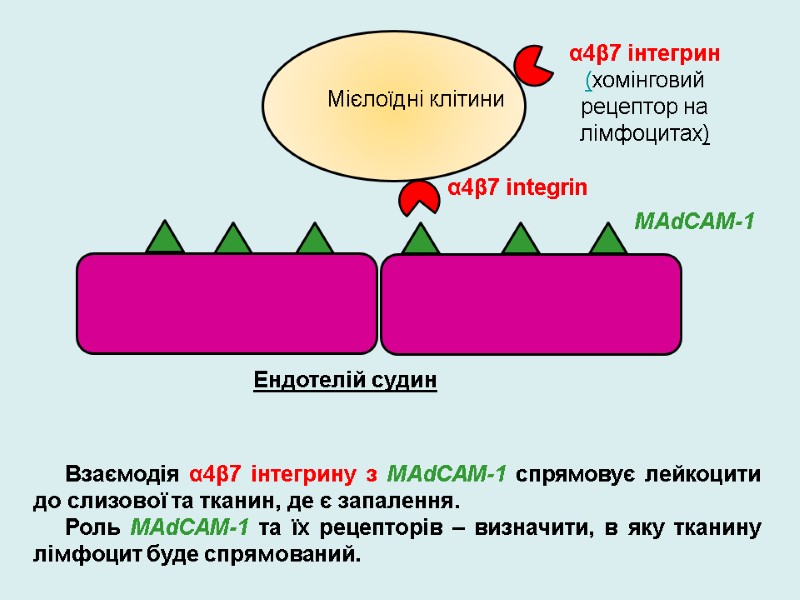
 α4β7 integrin MAdCAM-1 Мієлоїдні клітини Ендотелій судин α4β7 інтегрин (хомінговий рецептор на лімфоцитах) Взаємодія α4β7 інтегрину з MAdCAM-1 спрямовує лейкоцити до слизової та тканин, де є запалення. Роль MAdCAM-1 та їх рецепторів – визначити, в яку тканину лімфоцит буде спрямований.
α4β7 integrin MAdCAM-1 Мієлоїдні клітини Ендотелій судин α4β7 інтегрин (хомінговий рецептор на лімфоцитах) Взаємодія α4β7 інтегрину з MAdCAM-1 спрямовує лейкоцити до слизової та тканин, де є запалення. Роль MAdCAM-1 та їх рецепторів – визначити, в яку тканину лімфоцит буде спрямований.
 HIV infection of CD4+ T cells in the gut gp120 binds and signal through α4β7 integrin by means of a tripeptide motif in the V2 loop. The V2 loop mimics structures present in the natural ligands for α4β7 integrin, suggesting that α4β7 may facilitate HIV-1 infection of CD4+ T cells in the gut. J Virol. 2014 Sep;88(18):10767-77. Envelope glycoprotein binding to the integrin α4β7 is not a general property of most HIV-1 strains. Perez LG, Chen H, Liao HX, Montefiori DC. Переклад на наступному слайді
HIV infection of CD4+ T cells in the gut gp120 binds and signal through α4β7 integrin by means of a tripeptide motif in the V2 loop. The V2 loop mimics structures present in the natural ligands for α4β7 integrin, suggesting that α4β7 may facilitate HIV-1 infection of CD4+ T cells in the gut. J Virol. 2014 Sep;88(18):10767-77. Envelope glycoprotein binding to the integrin α4β7 is not a general property of most HIV-1 strains. Perez LG, Chen H, Liao HX, Montefiori DC. Переклад на наступному слайді
 Інфікування ВІЛ CD4+ T клітин у кишечнику gp120 зв’язується з α4β7 інтегрином за допомогою трипептидної послідовності у петлі V2. V2 петля подібна до структур, які містяться у природних лігандах інтегринів. Тому α4β7 може сприяти розповсюдженню ВІЛ-інфекції в кишечнику. J Virol. 2014 Sep;88(18):10767-77. Envelope glycoprotein binding to the integrin α4β7 is not a general property of most HIV-1 strains. Perez LG, Chen H, Liao HX, Montefiori DC.
Інфікування ВІЛ CD4+ T клітин у кишечнику gp120 зв’язується з α4β7 інтегрином за допомогою трипептидної послідовності у петлі V2. V2 петля подібна до структур, які містяться у природних лігандах інтегринів. Тому α4β7 може сприяти розповсюдженню ВІЛ-інфекції в кишечнику. J Virol. 2014 Sep;88(18):10767-77. Envelope glycoprotein binding to the integrin α4β7 is not a general property of most HIV-1 strains. Perez LG, Chen H, Liao HX, Montefiori DC.
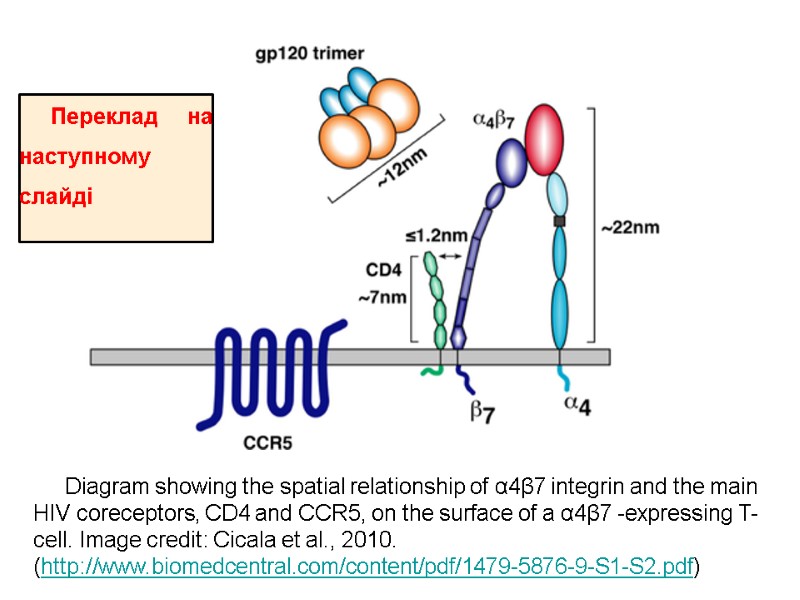
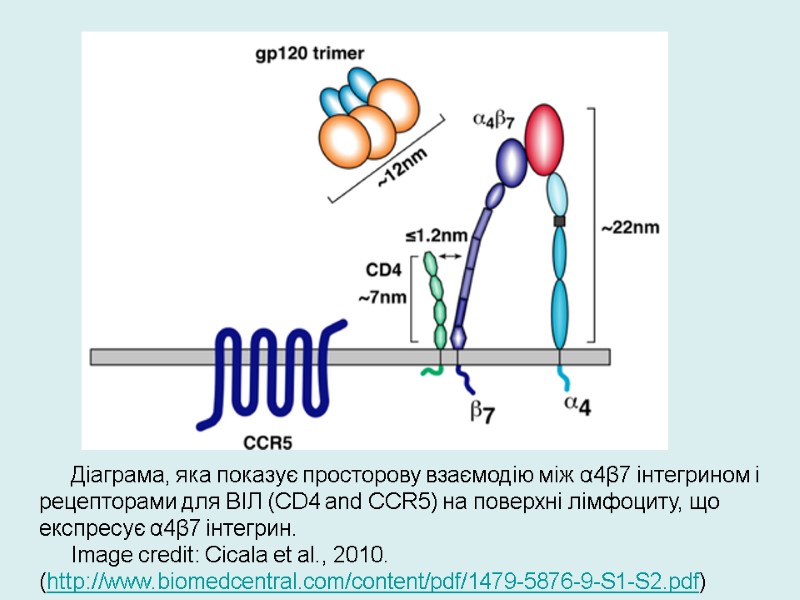
 Diagram showing the spatial relationship of α4β7 integrin and the main HIV coreceptors, CD4 and CCR5, on the surface of a α4β7 -expressing T-cell. Image credit: Cicala et al., 2010. (http://www.biomedcentral.com/content/pdf/1479-5876-9-S1-S2.pdf) Переклад на наступному слайді
Diagram showing the spatial relationship of α4β7 integrin and the main HIV coreceptors, CD4 and CCR5, on the surface of a α4β7 -expressing T-cell. Image credit: Cicala et al., 2010. (http://www.biomedcentral.com/content/pdf/1479-5876-9-S1-S2.pdf) Переклад на наступному слайді
 Діаграма, яка показує просторову взаємодію між α4β7 інтегрином і рецепторами для ВІЛ (CD4 and CCR5) на поверхні лімфоциту, що експресує α4β7 інтегрин. Image credit: Cicala et al., 2010. (http://www.biomedcentral.com/content/pdf/1479-5876-9-S1-S2.pdf)
Діаграма, яка показує просторову взаємодію між α4β7 інтегрином і рецепторами для ВІЛ (CD4 and CCR5) на поверхні лімфоциту, що експресує α4β7 інтегрин. Image credit: Cicala et al., 2010. (http://www.biomedcentral.com/content/pdf/1479-5876-9-S1-S2.pdf)
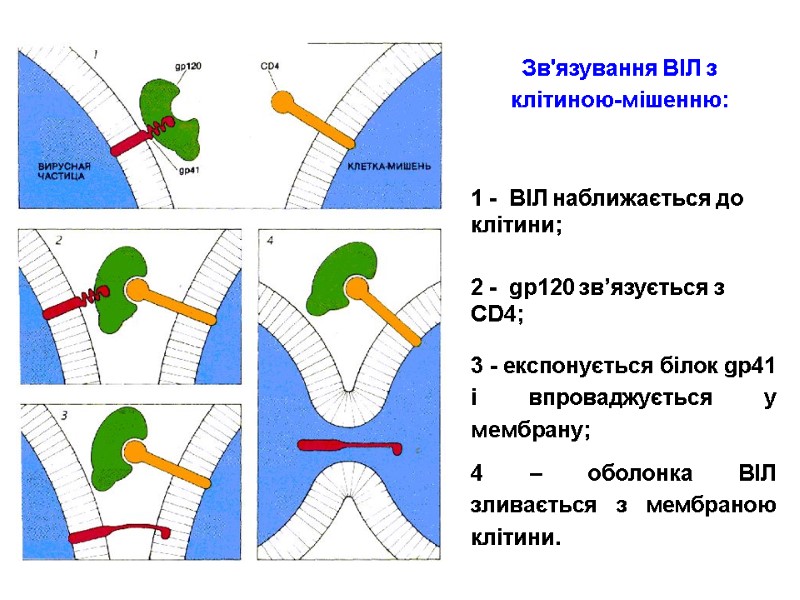
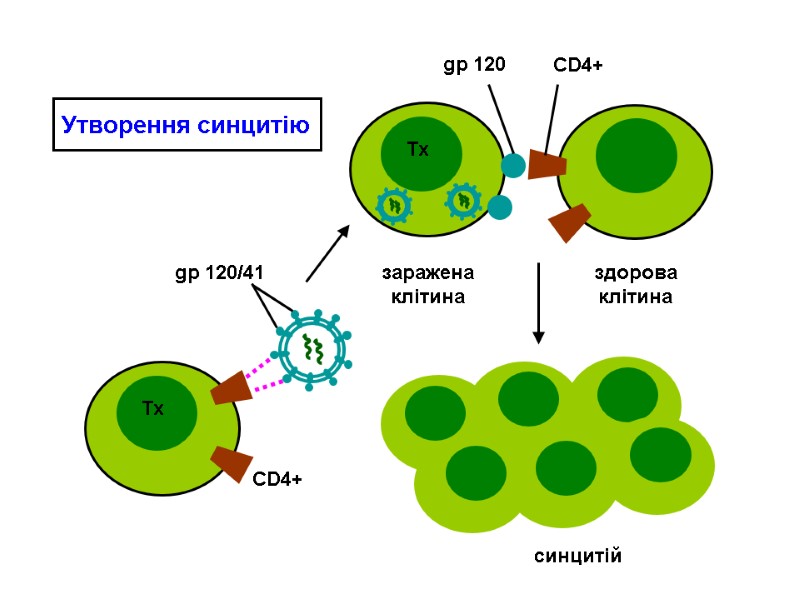
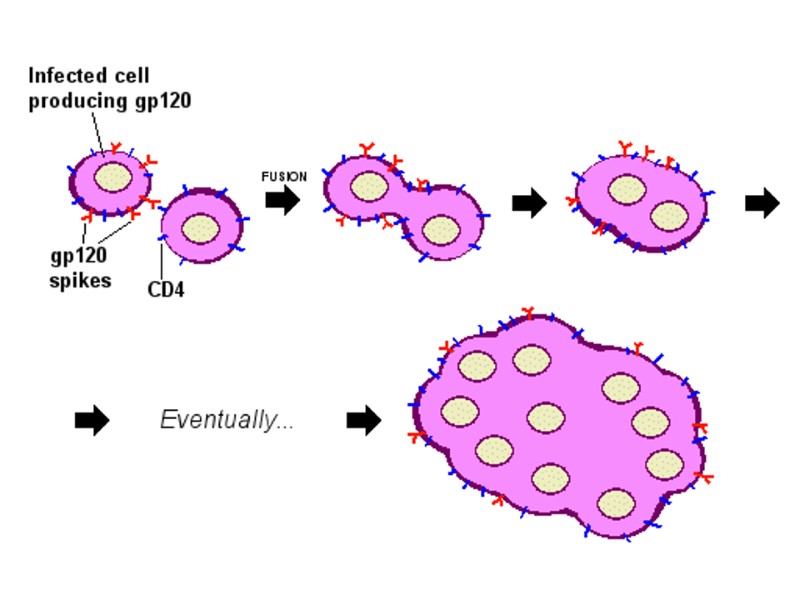
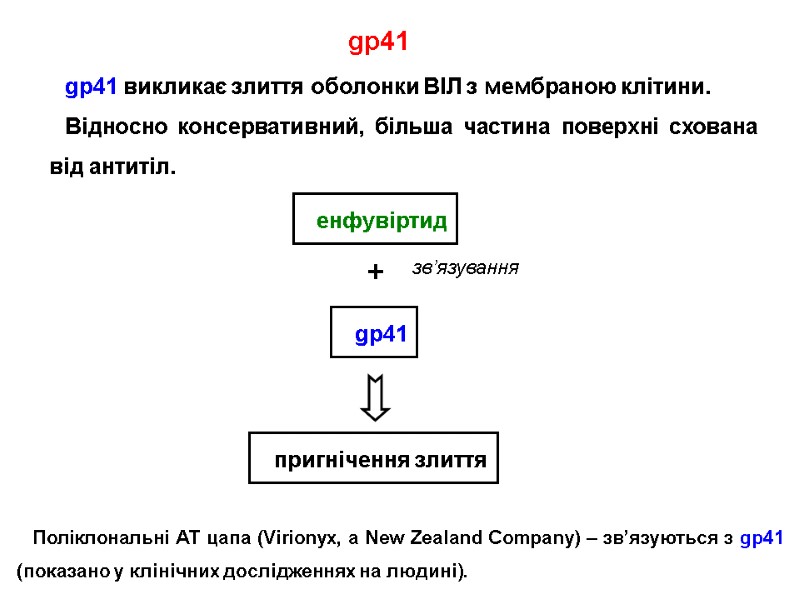
 gp41 gp41 is a glycoprotein non-covalently-bound to gp120. It is anchored within the viral envelope. gp41 provides the second step by which HIV enters the cell: gp120 binds to a CD4, gp120 changes its conformation, causing gp41 to become exposed, gp41 can assist in fusion with the host cell. Besides serving as the cell fusion initiator, gp41 also mediates the processing of the gp160 intermediate, as well as the gp120/gp41 complex. Переклад на наступному слайді
gp41 gp41 is a glycoprotein non-covalently-bound to gp120. It is anchored within the viral envelope. gp41 provides the second step by which HIV enters the cell: gp120 binds to a CD4, gp120 changes its conformation, causing gp41 to become exposed, gp41 can assist in fusion with the host cell. Besides serving as the cell fusion initiator, gp41 also mediates the processing of the gp160 intermediate, as well as the gp120/gp41 complex. Переклад на наступному слайді
 gp41 gp41 – глікопротеїн, який нековалентно зв’язаний з gp120 і заякорений у мембрану ВІЛ. gp41 забезпечує другий етап проникнення ВІЛ у лімфоцит: gp120 зв’язується з CD4-рецептором, gp120 змінює конформацію, що призводить до gp41 експонування gp41. Викликає злиття мембран ВІЛ і лімфоцита.
gp41 gp41 – глікопротеїн, який нековалентно зв’язаний з gp120 і заякорений у мембрану ВІЛ. gp41 забезпечує другий етап проникнення ВІЛ у лімфоцит: gp120 зв’язується з CD4-рецептором, gp120 змінює конформацію, що призводить до gp41 експонування gp41. Викликає злиття мембран ВІЛ і лімфоцита.
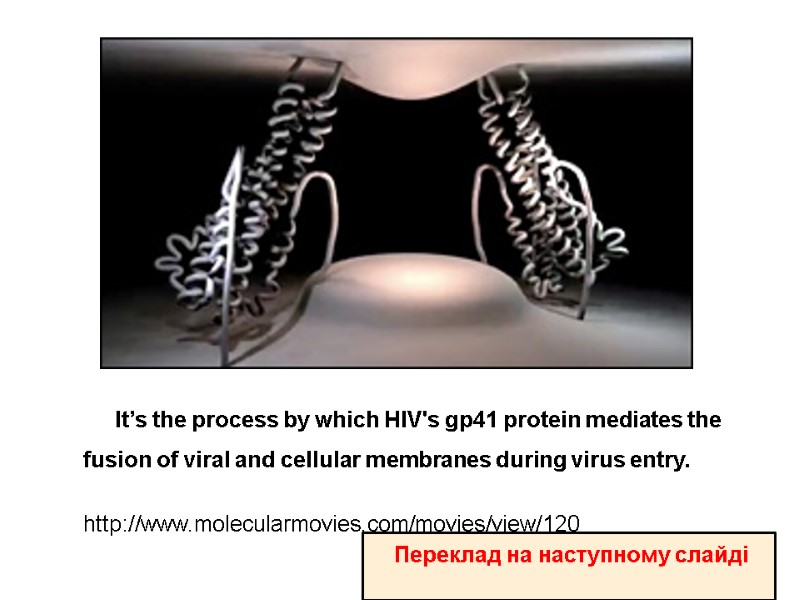
 It’s the process by which HIV's gp41 protein mediates the fusion of viral and cellular membranes during virus entry. http://www.molecularmovies.com/movies/view/120 Переклад на наступному слайді
It’s the process by which HIV's gp41 protein mediates the fusion of viral and cellular membranes during virus entry. http://www.molecularmovies.com/movies/view/120 Переклад на наступному слайді
 Процес, під час якого gp41 опосередковує злиття мембран ВІЛ і клітини під час інфікування http://www.molecularmovies.com/movies/view/120
Процес, під час якого gp41 опосередковує злиття мембран ВІЛ і клітини під час інфікування http://www.molecularmovies.com/movies/view/120
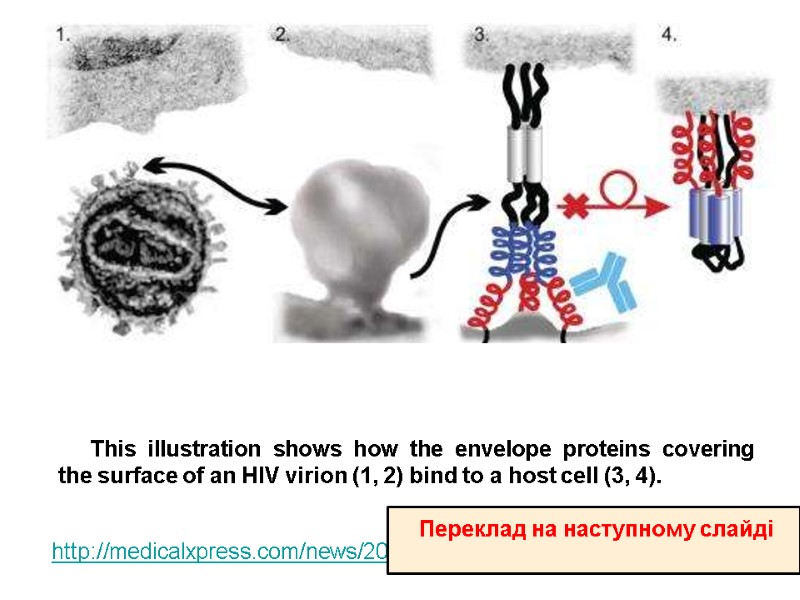
 This illustration shows how the envelope proteins covering the surface of an HIV virion (1, 2) bind to a host cell (3, 4). http://medicalxpress.com/news/2014-01-tricky-protein-hiv-vaccine.html Переклад на наступному слайді
This illustration shows how the envelope proteins covering the surface of an HIV virion (1, 2) bind to a host cell (3, 4). http://medicalxpress.com/news/2014-01-tricky-protein-hiv-vaccine.html Переклад на наступному слайді
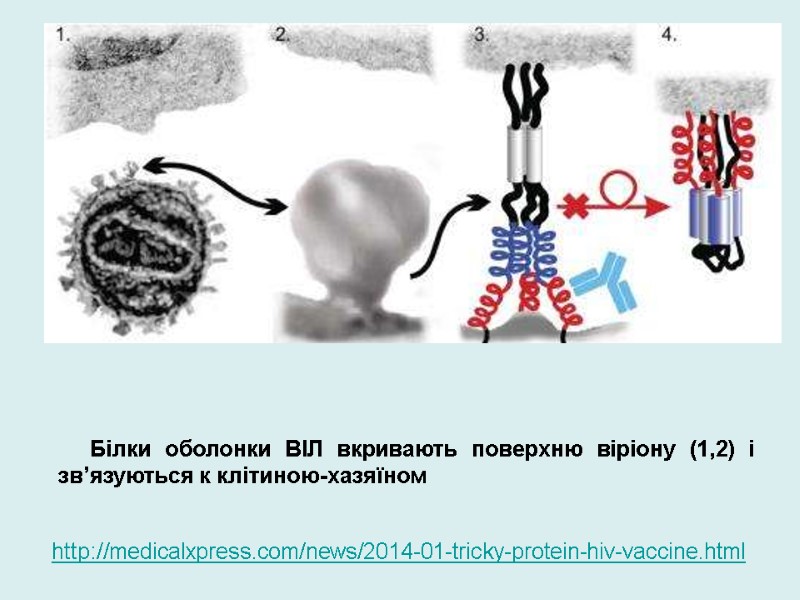
 Білки оболонки ВІЛ вкривають поверхню віріону (1,2) і зв’язуються к клітиною-хазяїном http://medicalxpress.com/news/2014-01-tricky-protein-hiv-vaccine.html
Білки оболонки ВІЛ вкривають поверхню віріону (1,2) і зв’язуються к клітиною-хазяїном http://medicalxpress.com/news/2014-01-tricky-protein-hiv-vaccine.html
 gp41 structure includes of 3 domains: an ectodomain, a transmembrane domain, a cytoplasmic domain The gp41 trimer ectodomain consists of 3 parallel N-terminal α-helices in a coiled coil conformation, cradled by three antiparallel C-terminal α-helices connected by flexible loop linkers. The coiled coil ectodomain connects to the hydrophobic transmembrane domain, followed by the hydrophilic cytoplasmic domain. Переклад на наступному слайді
gp41 structure includes of 3 domains: an ectodomain, a transmembrane domain, a cytoplasmic domain The gp41 trimer ectodomain consists of 3 parallel N-terminal α-helices in a coiled coil conformation, cradled by three antiparallel C-terminal α-helices connected by flexible loop linkers. The coiled coil ectodomain connects to the hydrophobic transmembrane domain, followed by the hydrophilic cytoplasmic domain. Переклад на наступному слайді
 У складі gp41 є 3 домени: ектодомен, трансмембранний домен, цитоплазматичний домен Тример ектодоменів gp41 складається з 3-х паралельних N –кінцевих α-спіралей у біспіральній конформації, які оточені 3-ма антипаралельними С-кінцевими α-спіралями, зв’язаними одна з одною за допомогою гнучких петлевих ділянок. Біспіральний ектодомен зв’язаний з гідрофільним трансмембранним доменом, за яким знаходиться гідрофільний цитоплазматичний домен.
У складі gp41 є 3 домени: ектодомен, трансмембранний домен, цитоплазматичний домен Тример ектодоменів gp41 складається з 3-х паралельних N –кінцевих α-спіралей у біспіральній конформації, які оточені 3-ма антипаралельними С-кінцевими α-спіралями, зв’язаними одна з одною за допомогою гнучких петлевих ділянок. Біспіральний ектодомен зв’язаний з гідрофільним трансмембранним доменом, за яким знаходиться гідрофільний цитоплазматичний домен.

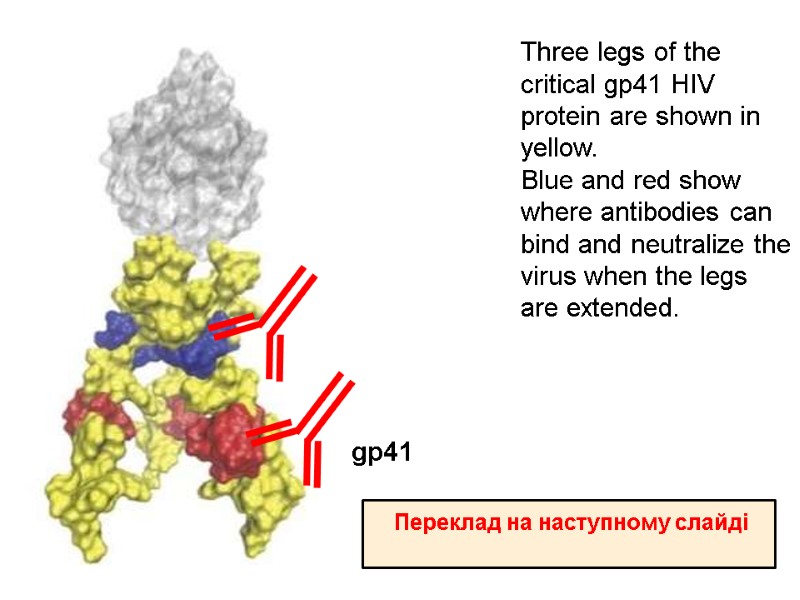
 Three legs of the critical gp41 HIV protein are shown in yellow. Blue and red show where antibodies can bind and neutralize the virus when the legs are extended. gp41 Переклад на наступному слайді
Three legs of the critical gp41 HIV protein are shown in yellow. Blue and red show where antibodies can bind and neutralize the virus when the legs are extended. gp41 Переклад на наступному слайді
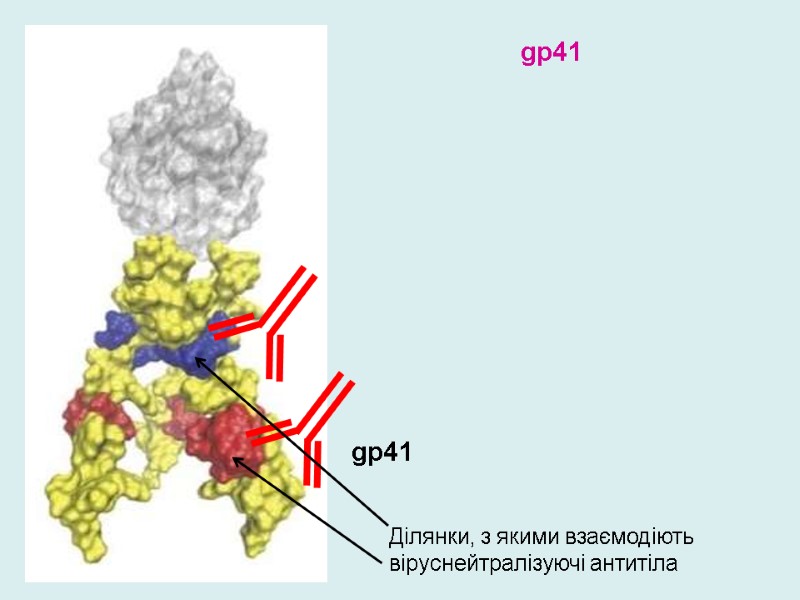
 gp41 gp41 Ділянки, з якими взаємодіють віруснейтралізуючі антитіла
gp41 gp41 Ділянки, з якими взаємодіють віруснейтралізуючі антитіла
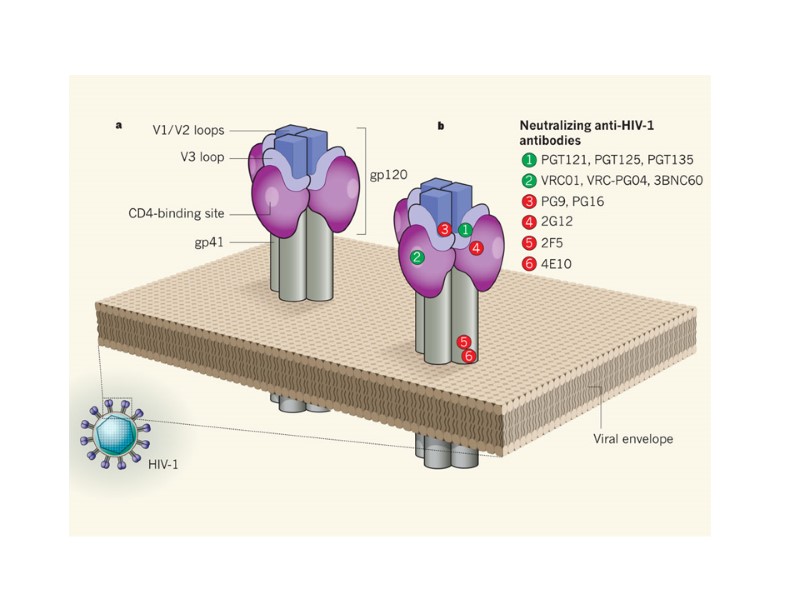
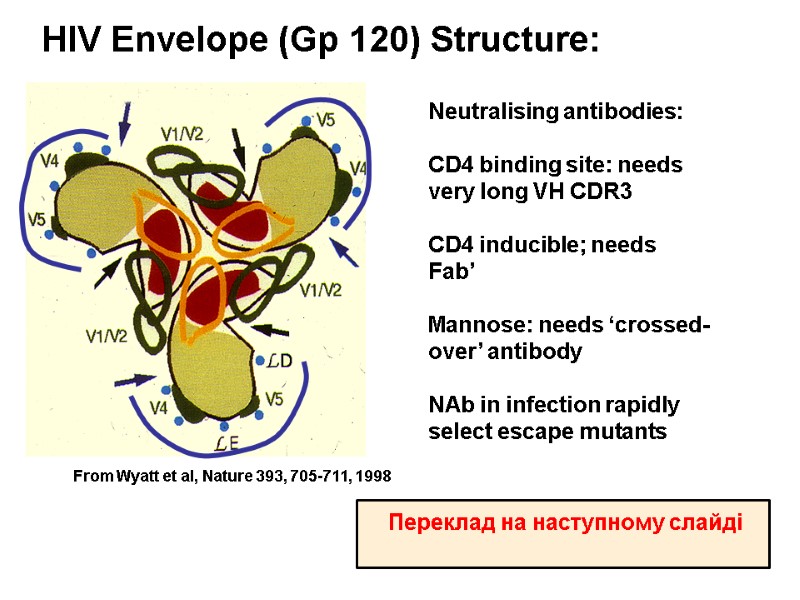
 HIV Envelope (Gp 120) Structure: Neutralising antibodies: CD4 binding site: needs very long VH CDR3 CD4 inducible; needs Fab’ Mannose: needs ‘crossed- over’ antibody NAb in infection rapidly select escape mutants Переклад на наступному слайді
HIV Envelope (Gp 120) Structure: Neutralising antibodies: CD4 binding site: needs very long VH CDR3 CD4 inducible; needs Fab’ Mannose: needs ‘crossed- over’ antibody NAb in infection rapidly select escape mutants Переклад на наступному слайді
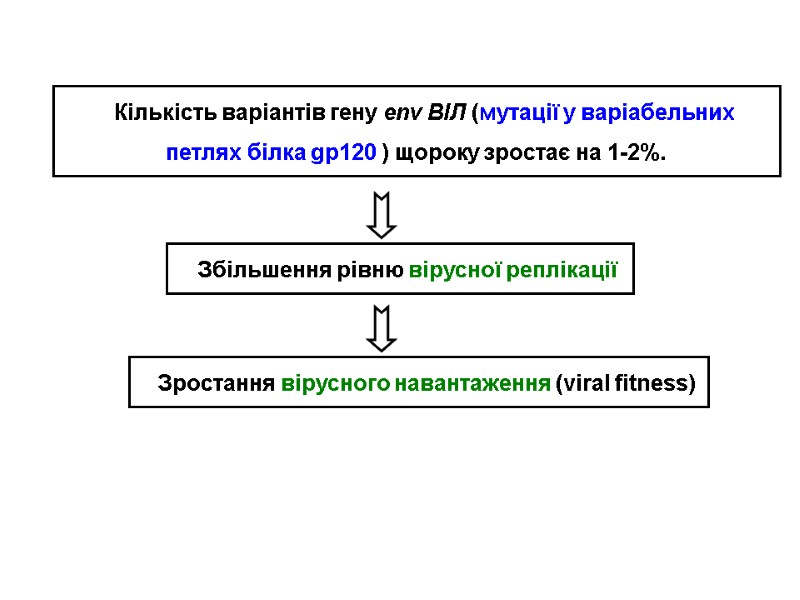
 Кількість варіантів гену env ВІЛ (мутації у варіабельних петлях білка gp120 ) щороку зростає на 1-2%. Збільшення рівню вірусної реплікації Зростання вірусного навантаження (viral fitness)
Кількість варіантів гену env ВІЛ (мутації у варіабельних петлях білка gp120 ) щороку зростає на 1-2%. Збільшення рівню вірусної реплікації Зростання вірусного навантаження (viral fitness)
 * - Wood, N., Bhattacharya, T., Keele, B.,Giorgi, E., Liu, M., Gaschen, B., Daniels, M., Ferrari, G., Haynes, B., McMichael, A., Shaw, G., Hahn, B., Korber, B., and Seoighe, C. (2009). "HIV evolution in early infection: selection pressures, patterns of insertion and deletion, and the impact of APOBEC". PLOS Pathogens 5: 1–16. Варіабельність у потенційних місцях N-глікозилювання gp120 – PNGSs (potential N-linked glycosylation sites) вірусного навантаження чутливості до нейтралізуючих АТ Гіпотеза Wood N.*: Великі вуглеводні ланцюги, зв’язані з gp120, можуть екранувати ділянки зв’язування з АТ.
* - Wood, N., Bhattacharya, T., Keele, B.,Giorgi, E., Liu, M., Gaschen, B., Daniels, M., Ferrari, G., Haynes, B., McMichael, A., Shaw, G., Hahn, B., Korber, B., and Seoighe, C. (2009). "HIV evolution in early infection: selection pressures, patterns of insertion and deletion, and the impact of APOBEC". PLOS Pathogens 5: 1–16. Варіабельність у потенційних місцях N-глікозилювання gp120 – PNGSs (potential N-linked glycosylation sites) вірусного навантаження чутливості до нейтралізуючих АТ Гіпотеза Wood N.*: Великі вуглеводні ланцюги, зв’язані з gp120, можуть екранувати ділянки зв’язування з АТ.
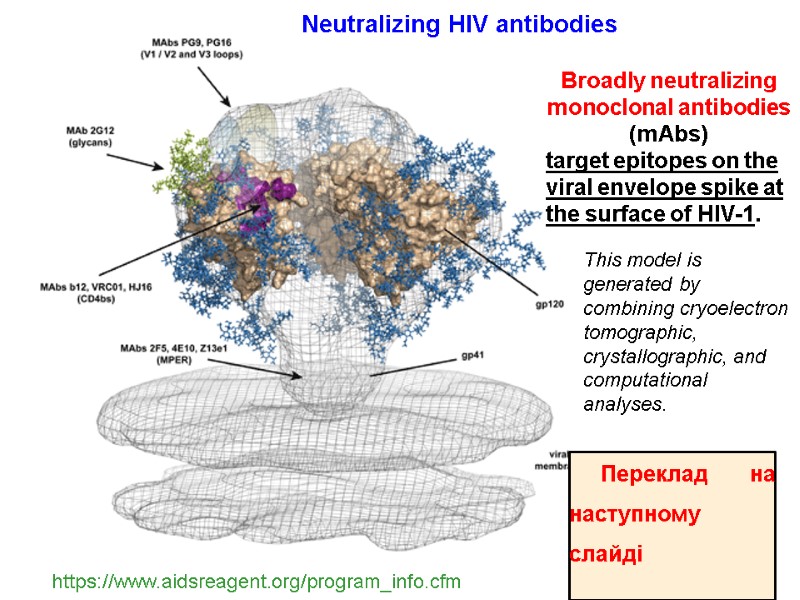
 Broadly neutralizing monoclonal antibodies (mAbs) target epitopes on the viral envelope spike at the surface of HIV-1. https://www.aidsreagent.org/program_info.cfm Neutralizing HIV antibodies This model is generated by combining cryoelectron tomographic, crystallographic, and computational analyses. Переклад на наступному слайді
Broadly neutralizing monoclonal antibodies (mAbs) target epitopes on the viral envelope spike at the surface of HIV-1. https://www.aidsreagent.org/program_info.cfm Neutralizing HIV antibodies This model is generated by combining cryoelectron tomographic, crystallographic, and computational analyses. Переклад на наступному слайді
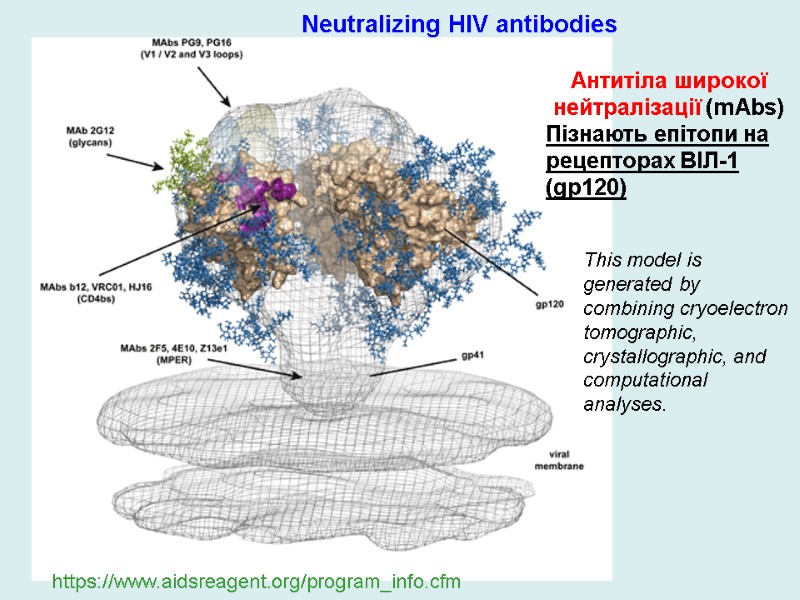
 Антитіла широкої нейтралізації (mAbs) Пізнають епітопи на рецепторах ВІЛ-1 (gp120) https://www.aidsreagent.org/program_info.cfm Neutralizing HIV antibodies This model is generated by combining cryoelectron tomographic, crystallographic, and computational analyses.
Антитіла широкої нейтралізації (mAbs) Пізнають епітопи на рецепторах ВІЛ-1 (gp120) https://www.aidsreagent.org/program_info.cfm Neutralizing HIV antibodies This model is generated by combining cryoelectron tomographic, crystallographic, and computational analyses.
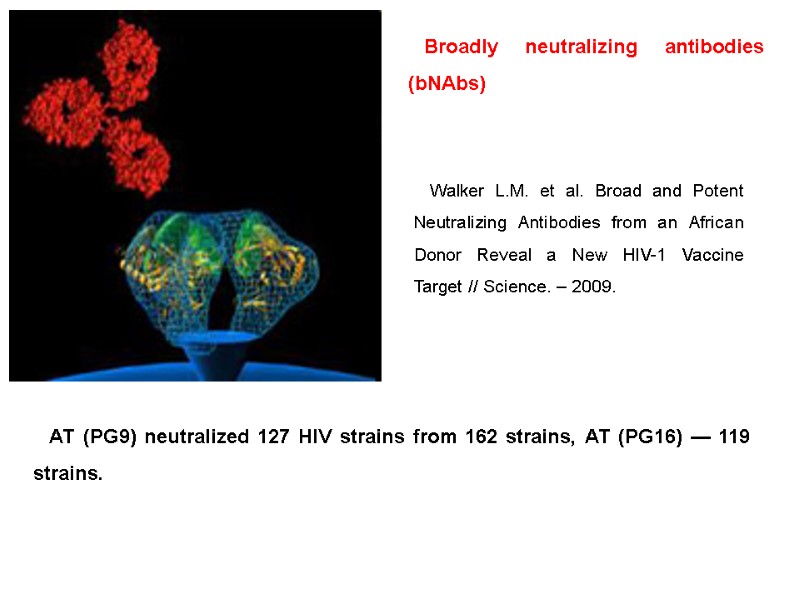
 Walker L.M. et al. Broad and Potent Neutralizing Antibodies from an African Donor Reveal a New HIV-1 Vaccine Target // Science. – 2009. Broadly neutralizing antibodies (bNAbs) АТ (PG9) neutralized 127 HIV strains from 162 strains, АТ (PG16) — 119 strains.
Walker L.M. et al. Broad and Potent Neutralizing Antibodies from an African Donor Reveal a New HIV-1 Vaccine Target // Science. – 2009. Broadly neutralizing antibodies (bNAbs) АТ (PG9) neutralized 127 HIV strains from 162 strains, АТ (PG16) — 119 strains.
 A number of highly potent and broadly neutralizing HIV-specific monoclonal antibodies have recently been isolated from B cells of infected individuals. Two to three years after infection, a fraction of HIV-1–infected individuals develop serologic activity that neutralizes most viral isolates. Broadly neutralizing antibodies that recognize the HIV-1 envelope protein have been isolated from these patients. Several HIV-specific monoclonal antibodies — in particular, PGT121, VRC01, and VRC03 — potently inhibited entry into CD4+ T cells of HIV isolated from the latent viral reservoir of infected individuals whose plasma viremia was well controlled by ART. Broad neutralization by a combination of antibodies recognizing the CD4 binding site and a new conformational epitope on the HIV-1 envelope protein Florian Klein, Christian Gaebler, Hugo Mouquet, D. Noah Sather, Clara Lehmann, Johannes F. Scheid, Zane Kraft, Yan Liu, John Pietzsch, Arlene Hurley, Pascal Poignard, Ten Feizi, Lynn Morris, Bruce D. Walker, Gerd Fätkenheuer, Michael S. Seaman, Leonidas Stamatatos, Michel C. Nussenzweig J Exp Med. 2012 July 30; 209(8): 1469–1479. Proc Natl Acad Sci U S A. 2014 Sep 9;111(36):13151-6. Broadly neutralizing antibodies suppress HIV in the persistent viral reservoir. Chun TW1, Murray D2, Justement JS2, Blazkova J2, Hallahan CW3, Fankuchen O2, Gittens K4, Benko E5, Kovacs C6, Moir S2, Fauci AS1. Переклад на наступному слайді
A number of highly potent and broadly neutralizing HIV-specific monoclonal antibodies have recently been isolated from B cells of infected individuals. Two to three years after infection, a fraction of HIV-1–infected individuals develop serologic activity that neutralizes most viral isolates. Broadly neutralizing antibodies that recognize the HIV-1 envelope protein have been isolated from these patients. Several HIV-specific monoclonal antibodies — in particular, PGT121, VRC01, and VRC03 — potently inhibited entry into CD4+ T cells of HIV isolated from the latent viral reservoir of infected individuals whose plasma viremia was well controlled by ART. Broad neutralization by a combination of antibodies recognizing the CD4 binding site and a new conformational epitope on the HIV-1 envelope protein Florian Klein, Christian Gaebler, Hugo Mouquet, D. Noah Sather, Clara Lehmann, Johannes F. Scheid, Zane Kraft, Yan Liu, John Pietzsch, Arlene Hurley, Pascal Poignard, Ten Feizi, Lynn Morris, Bruce D. Walker, Gerd Fätkenheuer, Michael S. Seaman, Leonidas Stamatatos, Michel C. Nussenzweig J Exp Med. 2012 July 30; 209(8): 1469–1479. Proc Natl Acad Sci U S A. 2014 Sep 9;111(36):13151-6. Broadly neutralizing antibodies suppress HIV in the persistent viral reservoir. Chun TW1, Murray D2, Justement JS2, Blazkova J2, Hallahan CW3, Fankuchen O2, Gittens K4, Benko E5, Kovacs C6, Moir S2, Fauci AS1. Переклад на наступному слайді
 Високоефективні ВІЛ-специфічні антитіла широкої нейтралізації було ізольовано з В-клітин інфікованих людей. Two to three years after infection, a fraction of HIV-1–infected individuals develop serologic activity that neutralizes most viral isolates. Broadly neutralizing antibodies that recognize the HIV-1 envelope protein have been isolated from these patients. Several HIV-specific monoclonal antibodies — in particular, PGT121, VRC01, and VRC03 — potently inhibited entry into CD4+ T cells of HIV isolated from the latent viral reservoir of infected individuals whose plasma viremia was well controlled by ART. Broad neutralization by a combination of antibodies recognizing the CD4 binding site and a new conformational epitope on the HIV-1 envelope protein Florian Klein, Christian Gaebler, Hugo Mouquet, D. Noah Sather, Clara Lehmann, Johannes F. Scheid, Zane Kraft, Yan Liu, John Pietzsch, Arlene Hurley, Pascal Poignard, Ten Feizi, Lynn Morris, Bruce D. Walker, Gerd Fätkenheuer, Michael S. Seaman, Leonidas Stamatatos, Michel C. Nussenzweig J Exp Med. 2012 July 30; 209(8): 1469–1479. Proc Natl Acad Sci U S A. 2014 Sep 9;111(36):13151-6. Broadly neutralizing antibodies suppress HIV in the persistent viral reservoir. Chun TW1, Murray D2, Justement JS2, Blazkova J2, Hallahan CW3, Fankuchen O2, Gittens K4, Benko E5, Kovacs C6, Moir S2, Fauci AS1.
Високоефективні ВІЛ-специфічні антитіла широкої нейтралізації було ізольовано з В-клітин інфікованих людей. Two to three years after infection, a fraction of HIV-1–infected individuals develop serologic activity that neutralizes most viral isolates. Broadly neutralizing antibodies that recognize the HIV-1 envelope protein have been isolated from these patients. Several HIV-specific monoclonal antibodies — in particular, PGT121, VRC01, and VRC03 — potently inhibited entry into CD4+ T cells of HIV isolated from the latent viral reservoir of infected individuals whose plasma viremia was well controlled by ART. Broad neutralization by a combination of antibodies recognizing the CD4 binding site and a new conformational epitope on the HIV-1 envelope protein Florian Klein, Christian Gaebler, Hugo Mouquet, D. Noah Sather, Clara Lehmann, Johannes F. Scheid, Zane Kraft, Yan Liu, John Pietzsch, Arlene Hurley, Pascal Poignard, Ten Feizi, Lynn Morris, Bruce D. Walker, Gerd Fätkenheuer, Michael S. Seaman, Leonidas Stamatatos, Michel C. Nussenzweig J Exp Med. 2012 July 30; 209(8): 1469–1479. Proc Natl Acad Sci U S A. 2014 Sep 9;111(36):13151-6. Broadly neutralizing antibodies suppress HIV in the persistent viral reservoir. Chun TW1, Murray D2, Justement JS2, Blazkova J2, Hallahan CW3, Fankuchen O2, Gittens K4, Benko E5, Kovacs C6, Moir S2, Fauci AS1.
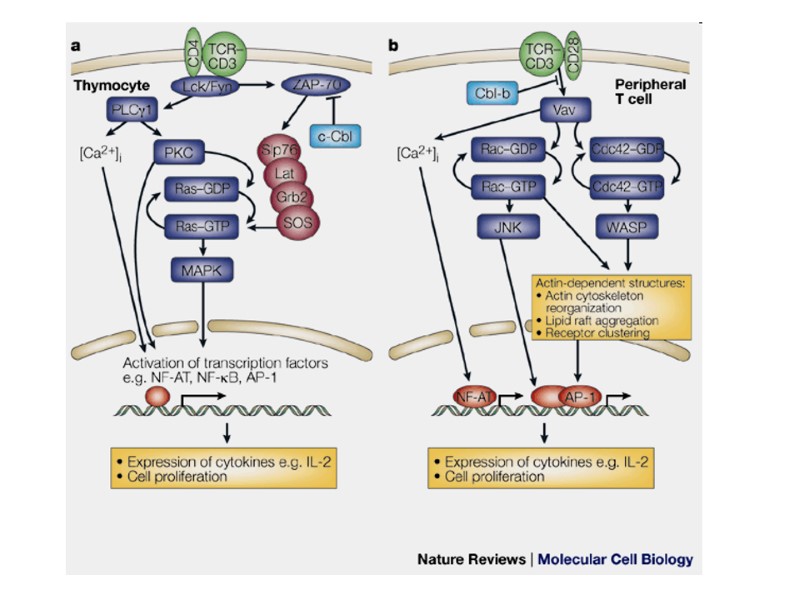
 HIV enzymes
HIV enzymes
 HIV enzymes reverse transcriptase (р66) – has three sequential biochemical activities: RNA-dependent DNA polymerase activity - synthesizes double-stranded DNA (cDNA) from HIV genomic RNA, ribonuclease H - cleaves genomic RNA after reverse transcription, and DNA-dependent DNA polymerase activity - synthesizes the second strand of HIV cDNA. integrase (р34) - inserts HIV cDNA into the host chromosomal DNA. protease (р22) – cleaves HIV precursor proteins (Gag (p55) and Gag-Pol (p160)) during virion maturation. Переклад на наступному слайді
HIV enzymes reverse transcriptase (р66) – has three sequential biochemical activities: RNA-dependent DNA polymerase activity - synthesizes double-stranded DNA (cDNA) from HIV genomic RNA, ribonuclease H - cleaves genomic RNA after reverse transcription, and DNA-dependent DNA polymerase activity - synthesizes the second strand of HIV cDNA. integrase (р34) - inserts HIV cDNA into the host chromosomal DNA. protease (р22) – cleaves HIV precursor proteins (Gag (p55) and Gag-Pol (p160)) during virion maturation. Переклад на наступному слайді
 Ферменти ВІЛ зворотна транскриптаза (р66) - складається з двох субодиниць. Має 2 ферментативні активності: ДНК-полімеразну - синтезує дволанцюгову ДНК, комплементарну вірусній РНК; рибонуклеазну - розщеплює вихідну РНК. інтеграза (р34) або ендонуклеаза - здійснює інтеграцію вірусної ДНК у геном клітини-хазяїна з утворенням провірусу; протеаза (р22) - бере участь у “нарізанні” попередників вірусних білків при дозріванні нової вірусної частки.
Ферменти ВІЛ зворотна транскриптаза (р66) - складається з двох субодиниць. Має 2 ферментативні активності: ДНК-полімеразну - синтезує дволанцюгову ДНК, комплементарну вірусній РНК; рибонуклеазну - розщеплює вихідну РНК. інтеграза (р34) або ендонуклеаза - здійснює інтеграцію вірусної ДНК у геном клітини-хазяїна з утворенням провірусу; протеаза (р22) - бере участь у “нарізанні” попередників вірусних білків при дозріванні нової вірусної частки.
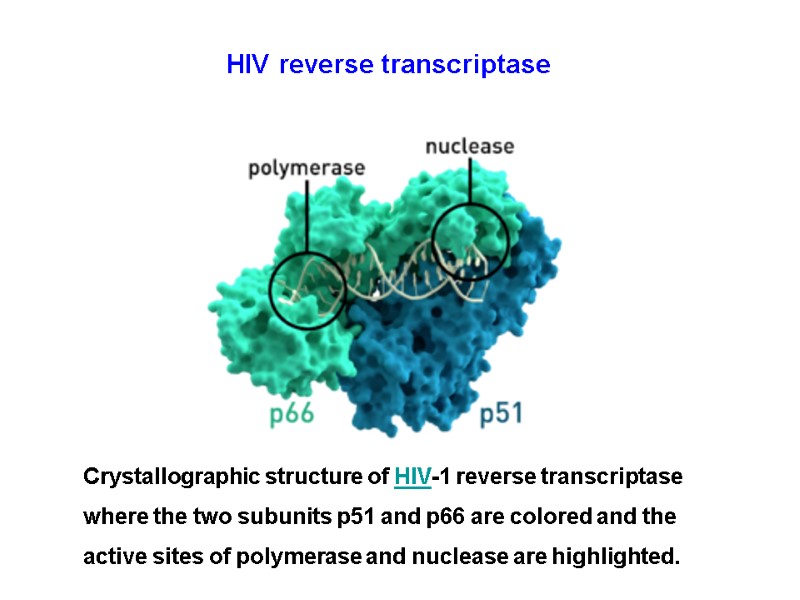
 Crystallographic structure of HIV-1 reverse transcriptase where the two subunits p51 and p66 are colored and the active sites of polymerase and nuclease are highlighted. HIV reverse transcriptase
Crystallographic structure of HIV-1 reverse transcriptase where the two subunits p51 and p66 are colored and the active sites of polymerase and nuclease are highlighted. HIV reverse transcriptase
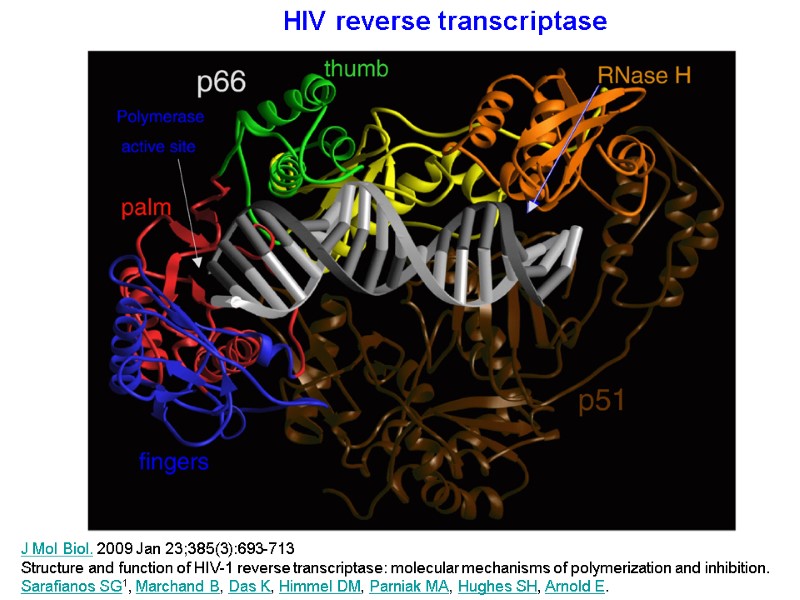
 HIV reverse transcriptase J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
HIV reverse transcriptase J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
 The structure of HIV reverse transcriptase (RT) RT of HIV-1 is an asymmetric heterodimer composed of two related subunits: p66 (560 amino acids) - contains the active sites for both of the enzymatic activities of RT (polymerase and RNase H). p51 (440 amino acids ) - plays a structural role. Both of the enzymatic functions of RT, the DNA polymerase and RNase H, are essential for the copying of the single stranded RNA genome found in virions into the double-stranded DNA that is inserted into the host genome by IN. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E. Переклад на наступному слайді
The structure of HIV reverse transcriptase (RT) RT of HIV-1 is an asymmetric heterodimer composed of two related subunits: p66 (560 amino acids) - contains the active sites for both of the enzymatic activities of RT (polymerase and RNase H). p51 (440 amino acids ) - plays a structural role. Both of the enzymatic functions of RT, the DNA polymerase and RNase H, are essential for the copying of the single stranded RNA genome found in virions into the double-stranded DNA that is inserted into the host genome by IN. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E. Переклад на наступному слайді
 Структура зворотної транскриптази (RT) RT HIV-1 асиметричний гетеродмер, що складається з двох субодиниць: p66 (560 а.к.) – містить активний центр, що відповідає за обидва ферментативні активності (полімераза і РНКаза Н) p51 (440 а.к.) – структурна роль. Обидва активності RT необхідні для синтезу кДНК ВІЛ. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
Структура зворотної транскриптази (RT) RT HIV-1 асиметричний гетеродмер, що складається з двох субодиниць: p66 (560 а.к.) – містить активний центр, що відповідає за обидва ферментативні активності (полімераза і РНКаза Н) p51 (440 а.к.) – структурна роль. Обидва активності RT необхідні для синтезу кДНК ВІЛ. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
 Error-Prone HIV Replication is a Pathogenic Determinant Error rate on order of 1-4 / 100,000 bases synthesized Replication rapid and error prone Each round of HIV replication generates numerous mutants. The ability of the mutants to replicate (viral fitness”) may vary greatly. The virus population can respond rapidly to a selective pressure. Переклад на наступному слайді
Error-Prone HIV Replication is a Pathogenic Determinant Error rate on order of 1-4 / 100,000 bases synthesized Replication rapid and error prone Each round of HIV replication generates numerous mutants. The ability of the mutants to replicate (viral fitness”) may vary greatly. The virus population can respond rapidly to a selective pressure. Переклад на наступному слайді
 Причина мутацій ВІЛ – помилки при роботі зворотної транскриптази Помилки - 1-4 / 100,000 основ Швидка реплікація, наслідок – велика кількість помилок. За кожний цикл реплікації утворюється велика кількість мутантів. Зростання “вірусного навантаження” у крові.
Причина мутацій ВІЛ – помилки при роботі зворотної транскриптази Помилки - 1-4 / 100,000 основ Швидка реплікація, наслідок – велика кількість помилок. За кожний цикл реплікації утворюється велика кількість мутантів. Зростання “вірусного навантаження” у крові.
 p66 is composed of two spatially distinct domains: polymerase RNase H. The polymerase domain of HIV RT is composed of four subdomains: fingers (пальці) (residues 1–85 and 118– 155), palm (великий палець) (residues 86–117 and 156–236), thumb (долоня) (237–318), connection (перемичка) (319–426) (5, 6).
p66 is composed of two spatially distinct domains: polymerase RNase H. The polymerase domain of HIV RT is composed of four subdomains: fingers (пальці) (residues 1–85 and 118– 155), palm (великий палець) (residues 86–117 and 156–236), thumb (долоня) (237–318), connection (перемичка) (319–426) (5, 6).
 p51 folds into the same four subdomains as the polymerase domain of p66 (fingers, palm, thumb, and connection). However the positions of the subdomains relative to each other are different in p66 and p51. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E. Переклад на наступному слайді
p51 folds into the same four subdomains as the polymerase domain of p66 (fingers, palm, thumb, and connection). However the positions of the subdomains relative to each other are different in p66 and p51. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E. Переклад на наступному слайді
 Субодиниця p51 згортається в такі ж самі субдомени , які і p66 (fingers, palm, thumb, and connection). Проте, їх положення може відрізнятися. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
Субодиниця p51 згортається в такі ж самі субдомени , які і p66 (fingers, palm, thumb, and connection). Проте, їх положення може відрізнятися. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
 The nucleic-acid binding cleft is formed primarily by the: p66 fingers, palm, thumb, connection, RNase H subdomains of p66. The binding cleft is configured so that the nucleic acid contacts both the polymerase and the RNase H active sites. Переклад на наступному слайді
The nucleic-acid binding cleft is formed primarily by the: p66 fingers, palm, thumb, connection, RNase H subdomains of p66. The binding cleft is configured so that the nucleic acid contacts both the polymerase and the RNase H active sites. Переклад на наступному слайді
 Впадина, що зв’язує нуклеїнову кислоту (nucleic-acid binding cleft ), сформована доменами: p66 fingers, palm, thumb, connection, RNase H subdomains of p66. Така конфігурації дозволяє нуклеїновій кислоті контактувати і з полімеразним, і з нуклеазним центрами ревертази.
Впадина, що зв’язує нуклеїнову кислоту (nucleic-acid binding cleft ), сформована доменами: p66 fingers, palm, thumb, connection, RNase H subdomains of p66. Така конфігурації дозволяє нуклеїновій кислоті контактувати і з полімеразним, і з нуклеазним центрами ревертази.
 The polymerase active site is composed of: three catalytic carboxylates in the palm subdomain of p66 (D110, D185, and D186). They bind two divalent ions that are required for catalysis: Mg2+ appears to be the divalent cation used in vivo; Mn2+ can support polymerization in vitro). J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E. Переклад на наступному слайді
The polymerase active site is composed of: three catalytic carboxylates in the palm subdomain of p66 (D110, D185, and D186). They bind two divalent ions that are required for catalysis: Mg2+ appears to be the divalent cation used in vivo; Mn2+ can support polymerization in vitro). J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E. Переклад на наступному слайді
 Полімеразний активний центр: три карбоксильні групи залишків аспартату у субдомені palm p66 субодиниці (D110, D185, and D186). Вони зв’язують двовалентні катіони: Mg2+ необхідний in vivo; Mn2+ необхідний для полімеризації in vitro. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
Полімеразний активний центр: три карбоксильні групи залишків аспартату у субдомені palm p66 субодиниці (D110, D185, and D186). Вони зв’язують двовалентні катіони: Mg2+ необхідний in vivo; Mn2+ необхідний для полімеризації in vitro. J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
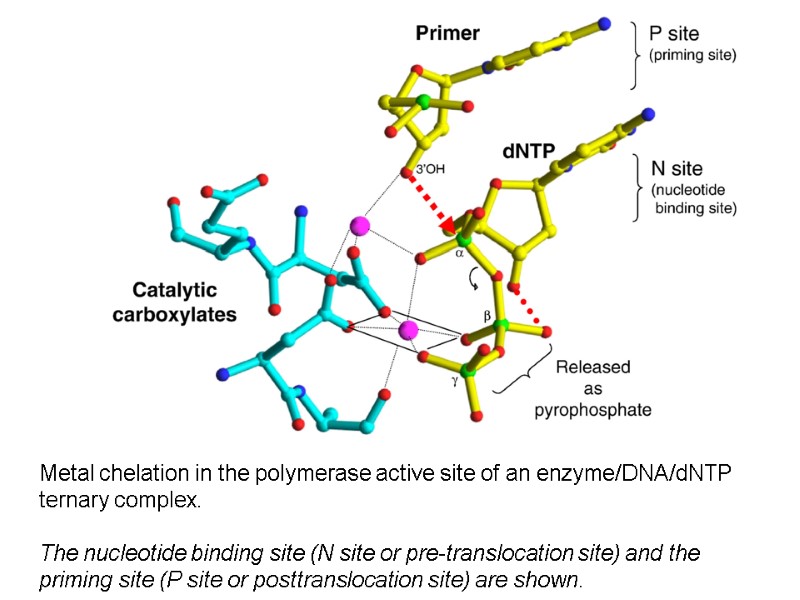
 Metal chelation in the polymerase active site of an enzyme/DNA/dNTP ternary complex. The nucleotide binding site (N site or pre-translocation site) and the priming site (P site or posttranslocation site) are shown.
Metal chelation in the polymerase active site of an enzyme/DNA/dNTP ternary complex. The nucleotide binding site (N site or pre-translocation site) and the priming site (P site or posttranslocation site) are shown.
 J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
J Mol Biol. 2009 Jan 23;385(3):693-713 Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition. Sarafianos SG1, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E.
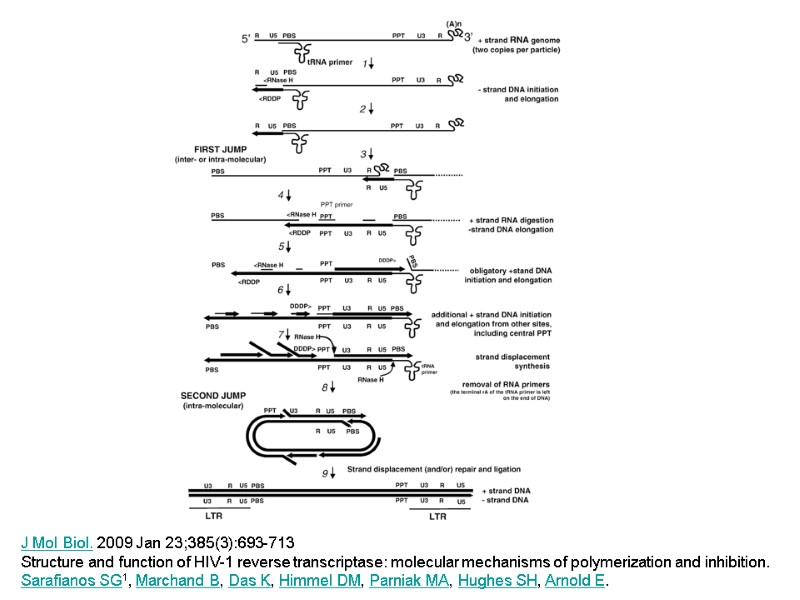
 Step 1: Synthesis proceeds to the 5’ end of the RNA genome through the U5 region, ending at the R region at the 5’end, forming the “minus-strand strong stop DNA.” Step 2: DNA synthesis is accompanied by RNase H digestion of the RNA portion of the RNA-DNA hybrid product, thus exposing the single-strand DNA product. Step 3: This exposure facilitates hybridization with the R region at the 3’ end of the same, or the second RNA genome, a strand-transfer reaction known as the “first jump”. Step 4: When minus-strand elongation passes a polypurine rich region called the polypurine tract (PPT) region, a unique plus-strand RNA primer is formed by RNase H cleavage at its borders. Plus-strand synthesis then continues back to the U5 region using the minus-strand DNA as a template. Step 5: Meanwhile, minus-strand synthesis continues through the genome using the plus-strand RNA as a template, and removing the RNA template in its wake via RNase H activity. Step 6: The RNase H digestion products formed are presumed to provide additional primers for plus-strand synthesis at a number of internal locations along the minus-strand DNA. Step 7: PPT-initiated plus-strand DNA synthesis stops after copying the annealed portion of the tRNA to generate the plus-strand DNA form o f the PBS, forming the “plus-strand strong stop” product. The tRNA is then removed by the RNase H activity of RT. Step 8: This may facilitate annealing to the PBS complement on the minus-strand DNA, providing the complementarity for the “second jump.” DNA synthesis then continues.Step 9: Strand displacement synthesis by RT to the PBS and PPT ends, and/or repair and ligation of a circular intermediate produces a linear duplex with long terminal repeats (LTRs) at both ends.
Step 1: Synthesis proceeds to the 5’ end of the RNA genome through the U5 region, ending at the R region at the 5’end, forming the “minus-strand strong stop DNA.” Step 2: DNA synthesis is accompanied by RNase H digestion of the RNA portion of the RNA-DNA hybrid product, thus exposing the single-strand DNA product. Step 3: This exposure facilitates hybridization with the R region at the 3’ end of the same, or the second RNA genome, a strand-transfer reaction known as the “first jump”. Step 4: When minus-strand elongation passes a polypurine rich region called the polypurine tract (PPT) region, a unique plus-strand RNA primer is formed by RNase H cleavage at its borders. Plus-strand synthesis then continues back to the U5 region using the minus-strand DNA as a template. Step 5: Meanwhile, minus-strand synthesis continues through the genome using the plus-strand RNA as a template, and removing the RNA template in its wake via RNase H activity. Step 6: The RNase H digestion products formed are presumed to provide additional primers for plus-strand synthesis at a number of internal locations along the minus-strand DNA. Step 7: PPT-initiated plus-strand DNA synthesis stops after copying the annealed portion of the tRNA to generate the plus-strand DNA form o f the PBS, forming the “plus-strand strong stop” product. The tRNA is then removed by the RNase H activity of RT. Step 8: This may facilitate annealing to the PBS complement on the minus-strand DNA, providing the complementarity for the “second jump.” DNA synthesis then continues.Step 9: Strand displacement synthesis by RT to the PBS and PPT ends, and/or repair and ligation of a circular intermediate produces a linear duplex with long terminal repeats (LTRs) at both ends.
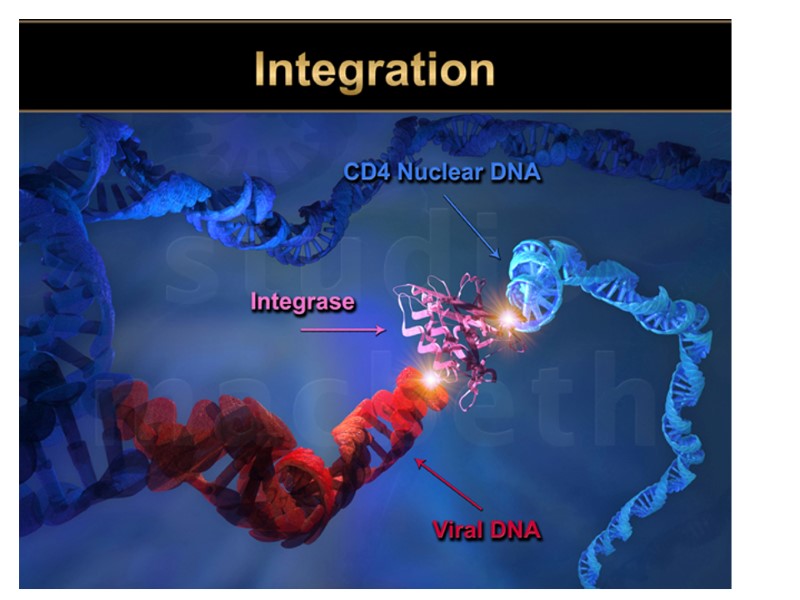
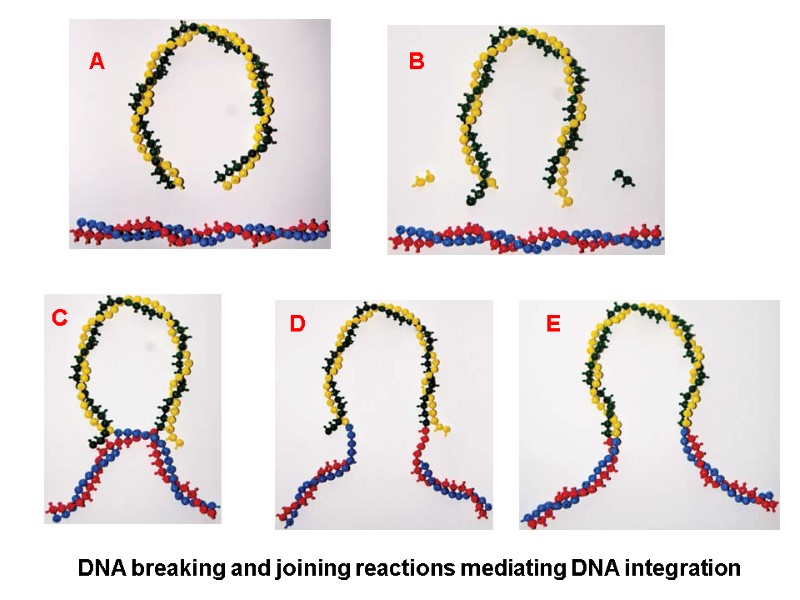
 HIV Integrase Function: inserts the viral DNA into the host chromosomal DNA, a step that is essential for HIV replication. Integration is a point of no return for the cell, which becomes a permanent carrier of the viral genome (provirus). Integration is responsible for the persistence of retroviral infections. http://cdn.intechopen.com/pdfs-wm/44028.pdf Переклад на наступному слайді
HIV Integrase Function: inserts the viral DNA into the host chromosomal DNA, a step that is essential for HIV replication. Integration is a point of no return for the cell, which becomes a permanent carrier of the viral genome (provirus). Integration is responsible for the persistence of retroviral infections. http://cdn.intechopen.com/pdfs-wm/44028.pdf Переклад на наступному слайді
 HIV інтеграза Функція: вбудовує вірусну ДНК у ДНК клітини. Після цього інфекція зберігається в клітині на все життя. Провірус – геном ВІЛ, інтегрований у геном клітини. http://cdn.intechopen.com/pdfs-wm/44028.pdf
HIV інтеграза Функція: вбудовує вірусну ДНК у ДНК клітини. Після цього інфекція зберігається в клітині на все життя. Провірус – геном ВІЛ, інтегрований у геном клітини. http://cdn.intechopen.com/pdfs-wm/44028.pdf
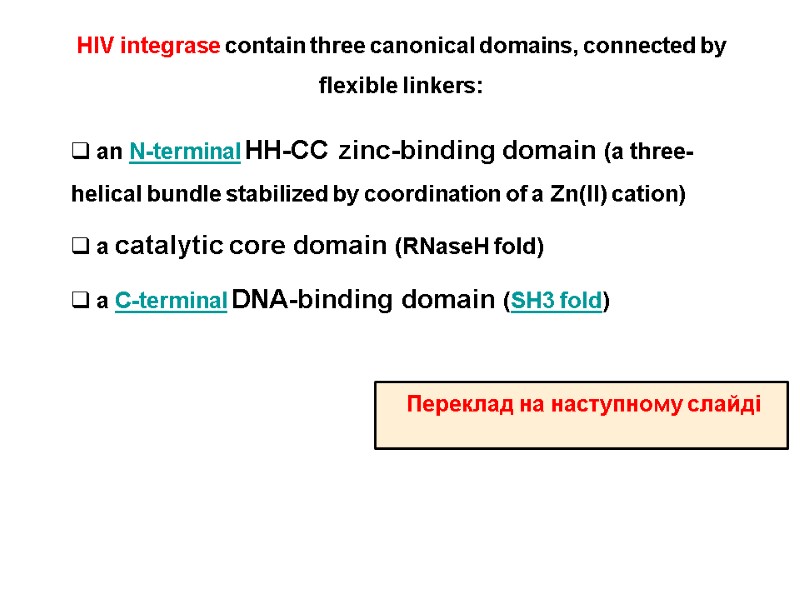
 HIV integrase contain three canonical domains, connected by flexible linkers: an N-terminal HH-CC zinc-binding domain (a three-helical bundle stabilized by coordination of a Zn(II) cation) a catalytic core domain (RNaseH fold) a C-terminal DNA-binding domain (SH3 fold) Переклад на наступному слайді
HIV integrase contain three canonical domains, connected by flexible linkers: an N-terminal HH-CC zinc-binding domain (a three-helical bundle stabilized by coordination of a Zn(II) cation) a catalytic core domain (RNaseH fold) a C-terminal DNA-binding domain (SH3 fold) Переклад на наступному слайді
 HIV integrase має три домени, зв’єднані гнучкими лінкерами: an N-кінцевий HH-CC zinc-binding domain (три спіралі, стабілізується Zn(II)) каталітичний коровий домен (RNaseH fold) a C-кінцевий DNA-зв’язуючий домен (SH3 fold)
HIV integrase має три домени, зв’єднані гнучкими лінкерами: an N-кінцевий HH-CC zinc-binding domain (три спіралі, стабілізується Zn(II)) каталітичний коровий домен (RNaseH fold) a C-кінцевий DNA-зв’язуючий домен (SH3 fold)
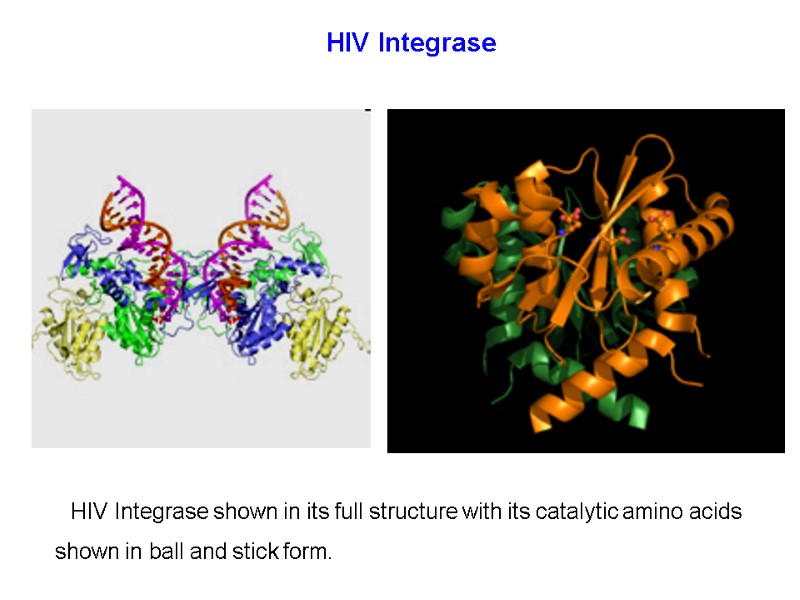
 HIV Integrase shown in its full structure with its catalytic amino acids shown in ball and stick form. HIV Integrase
HIV Integrase shown in its full structure with its catalytic amino acids shown in ball and stick form. HIV Integrase
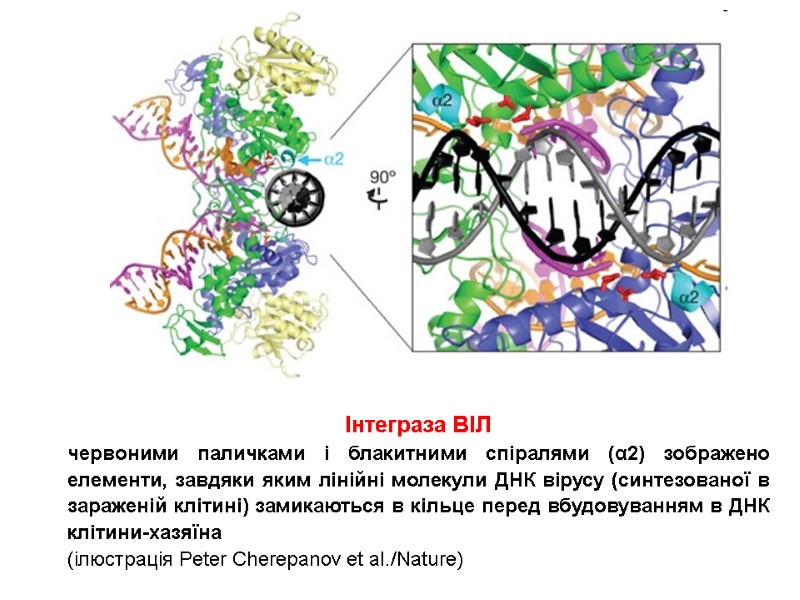
 Інтеграза ВІЛ червоними паличками і блакитними спіралями (α2) зображено елементи, завдяки яким лінійні молекули ДНК вірусу (синтезованої в зараженій клітині) замикаються в кільце перед вбудовуванням в ДНК клітини-хазяїна (ілюстрація Peter Cherepanov et al./Nature)
Інтеграза ВІЛ червоними паличками і блакитними спіралями (α2) зображено елементи, завдяки яким лінійні молекули ДНК вірусу (синтезованої в зараженій клітині) замикаються в кільце перед вбудовуванням в ДНК клітини-хазяїна (ілюстрація Peter Cherepanov et al./Nature)
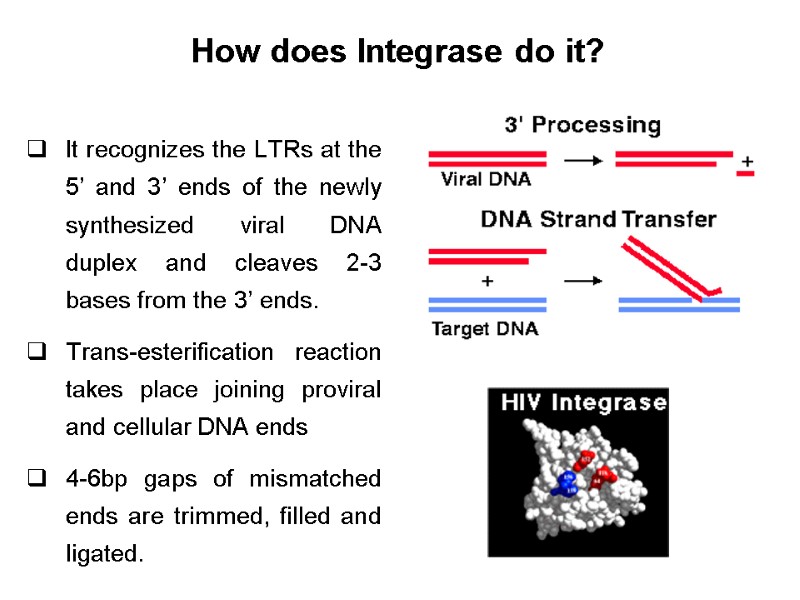
 HIV integrase catalyzes two reactions: 3'-processing, in which two or three nucleotides are removed from one or both 3' ends of the viral DNA to expose the invariant CA dinucleotides at both 3'-ends of the viral DNA. the strand transfer reaction, in which the processed 3' ends of the viral DNA are covalently ligated to the host chromosomal DNA.
HIV integrase catalyzes two reactions: 3'-processing, in which two or three nucleotides are removed from one or both 3' ends of the viral DNA to expose the invariant CA dinucleotides at both 3'-ends of the viral DNA. the strand transfer reaction, in which the processed 3' ends of the viral DNA are covalently ligated to the host chromosomal DNA.
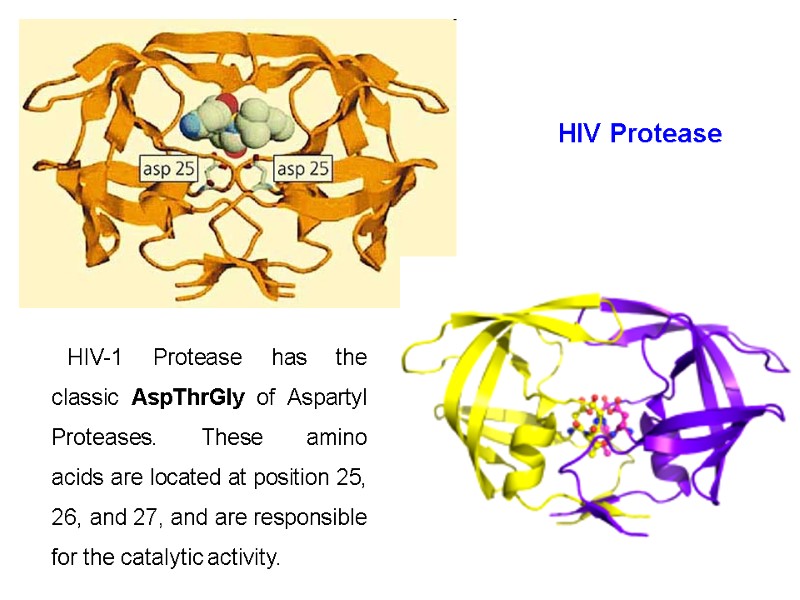
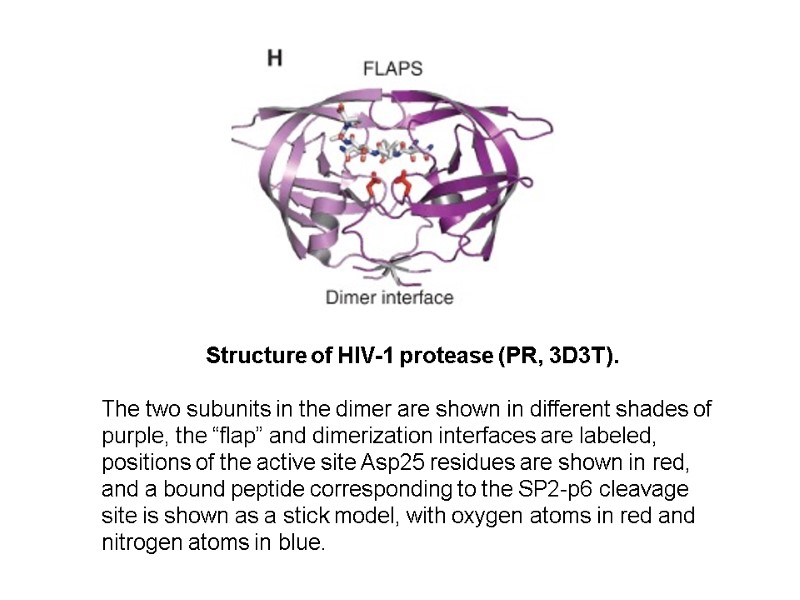
 HIV-1 protease Function A retroviral aspartyl protease. It cleaves newly synthesized polyproteins at the appropriate places to create the mature protein components of an infectious HIV virion.
HIV-1 protease Function A retroviral aspartyl protease. It cleaves newly synthesized polyproteins at the appropriate places to create the mature protein components of an infectious HIV virion.
 HIV-1 protease Structure It exists as a homodimer, with each subunit made up of 99 amino acids. The active site: lies between the identical subunits and has the characteristic Asp-Thr-Gly (Asp25, Thr26 and Gly27) sequence common to aspartic proteases. the two Asp25 residues (one from each chain) act as the catalytic residues.
HIV-1 protease Structure It exists as a homodimer, with each subunit made up of 99 amino acids. The active site: lies between the identical subunits and has the characteristic Asp-Thr-Gly (Asp25, Thr26 and Gly27) sequence common to aspartic proteases. the two Asp25 residues (one from each chain) act as the catalytic residues.
 HIV-1 Protease has the classic AspThrGly of Aspartyl Proteases. These amino acids are located at position 25, 26, and 27, and are responsible for the catalytic activity. HIV Protease
HIV-1 Protease has the classic AspThrGly of Aspartyl Proteases. These amino acids are located at position 25, 26, and 27, and are responsible for the catalytic activity. HIV Protease
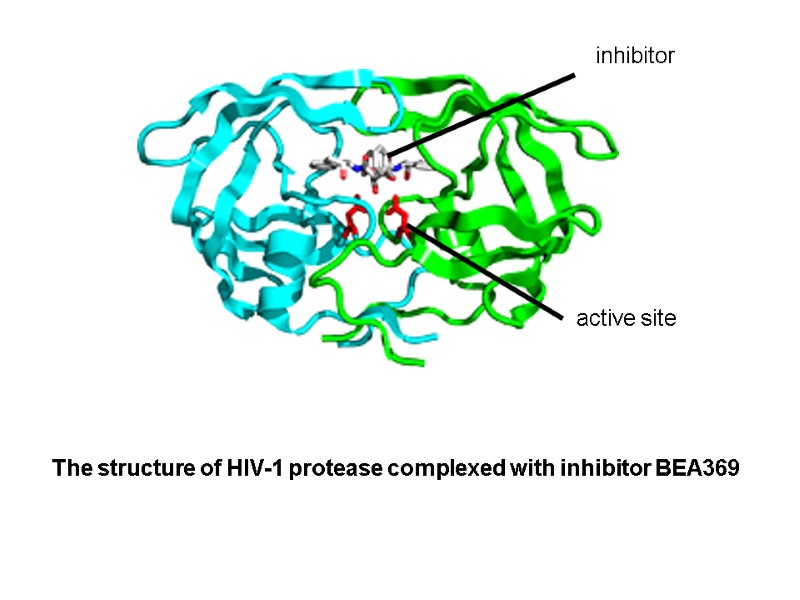
 The structure of HIV-1 protease complexed with inhibitor BEA369 active site inhibitor
The structure of HIV-1 protease complexed with inhibitor BEA369 active site inhibitor
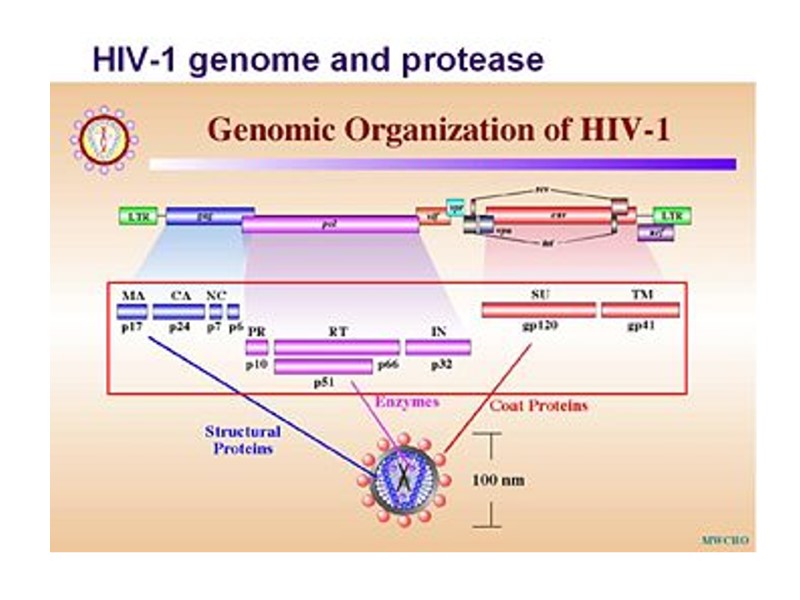
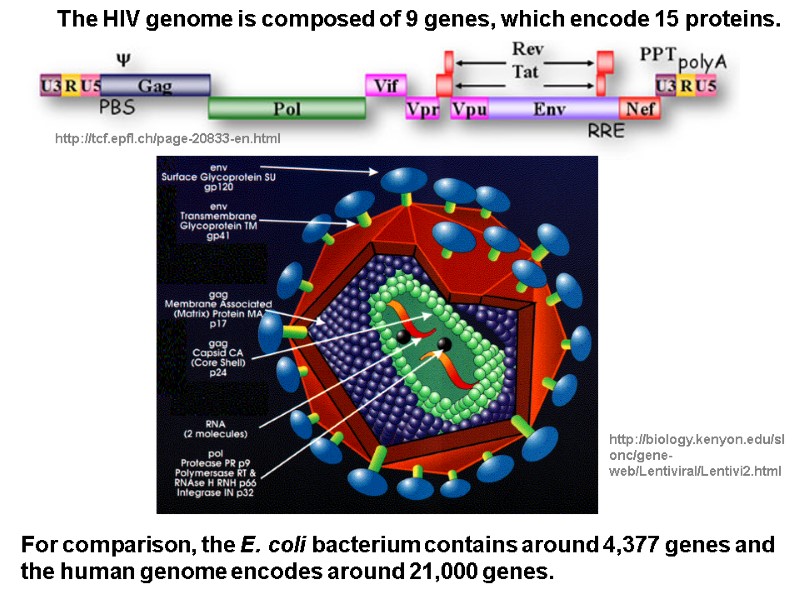
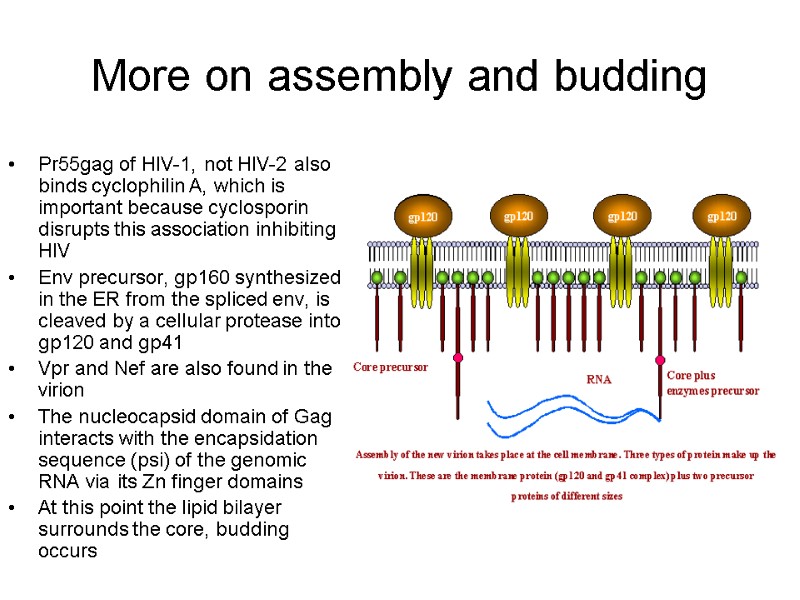
 http://tcf.epfl.ch/page-20833-en.html The HIV genome is composed of 9 genes, which encode 15 proteins. For comparison, the E. coli bacterium contains around 4,377 genes and the human genome encodes around 21,000 genes. http://biology.kenyon.edu/slonc/gene-web/Lentiviral/Lentivi2.html
http://tcf.epfl.ch/page-20833-en.html The HIV genome is composed of 9 genes, which encode 15 proteins. For comparison, the E. coli bacterium contains around 4,377 genes and the human genome encodes around 21,000 genes. http://biology.kenyon.edu/slonc/gene-web/Lentiviral/Lentivi2.html
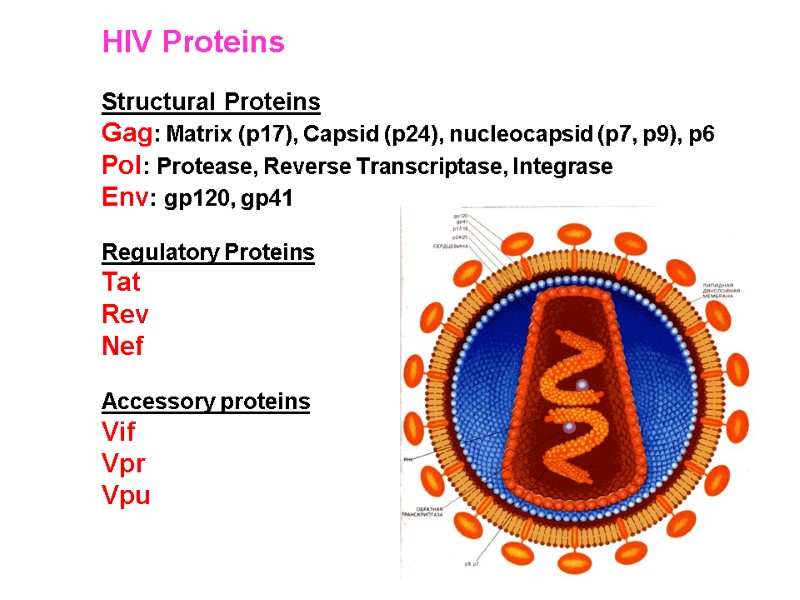
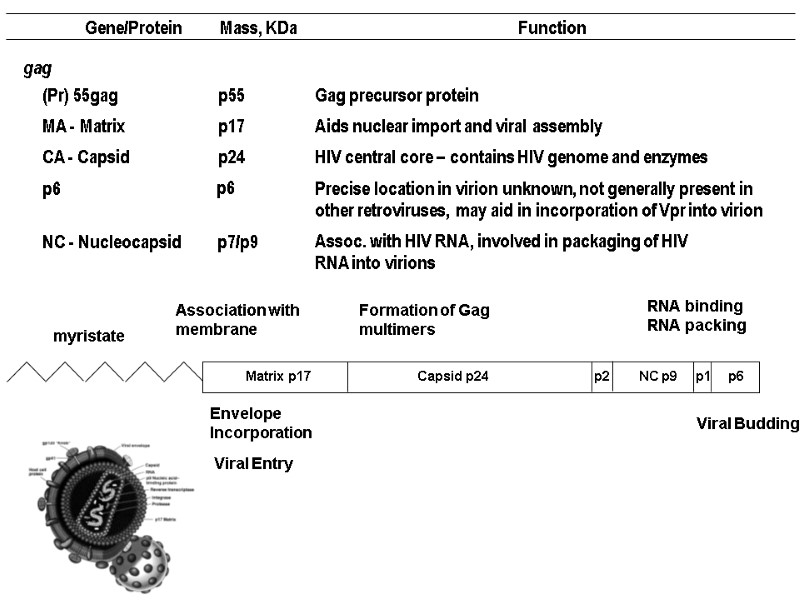
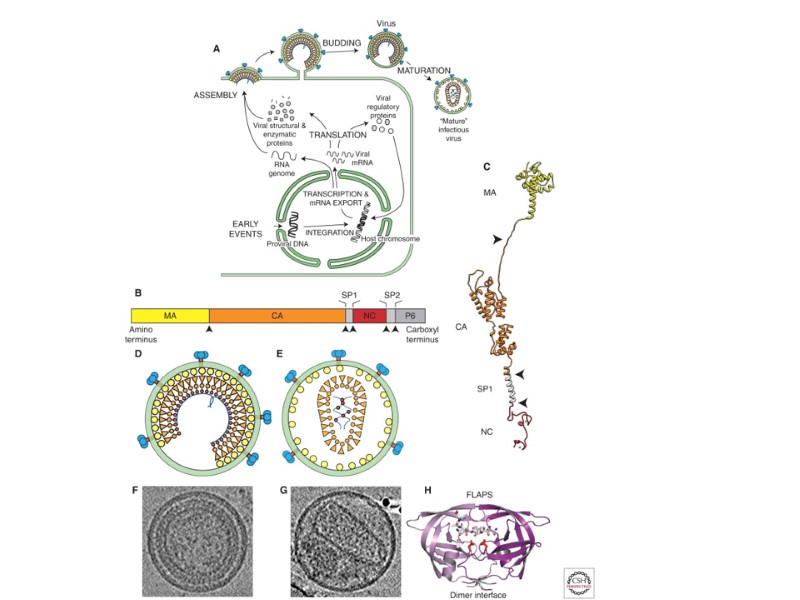
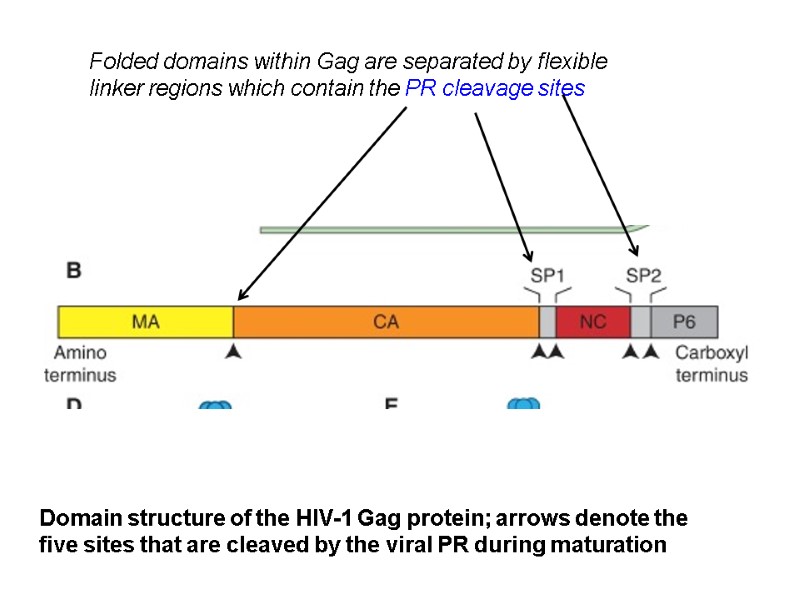
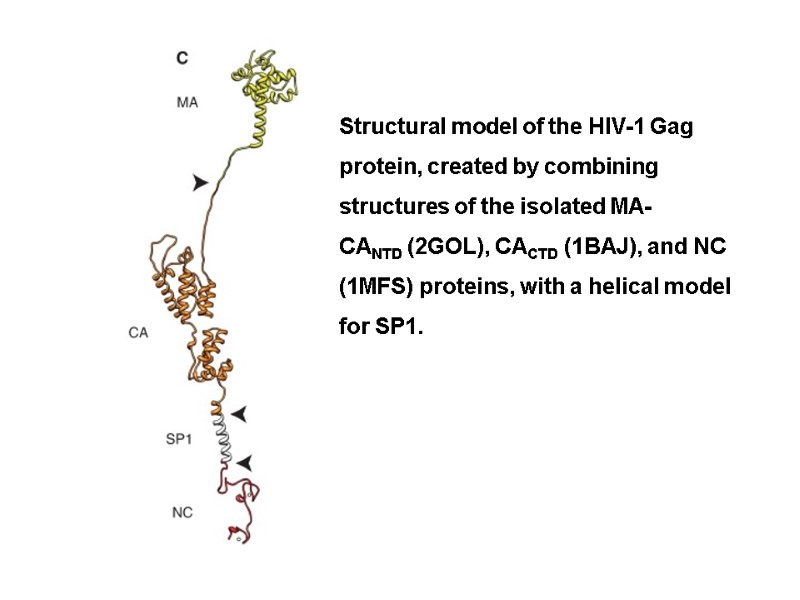
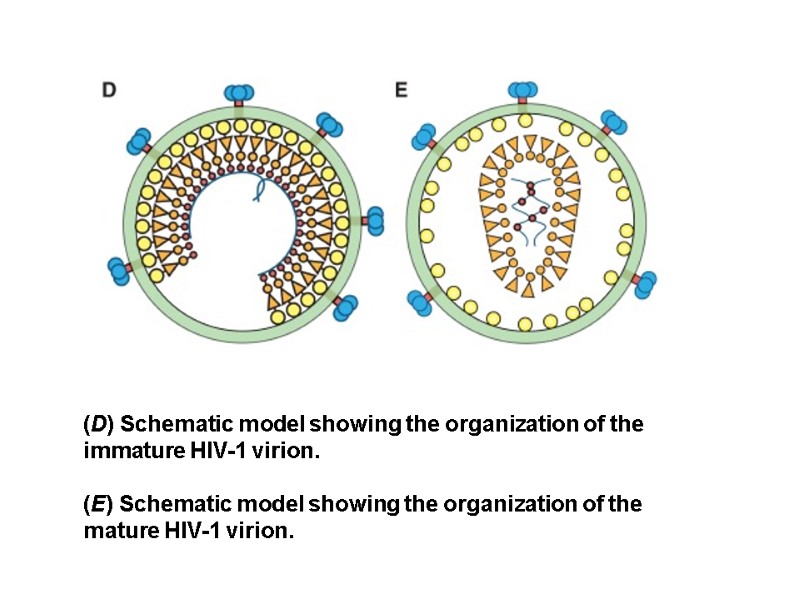
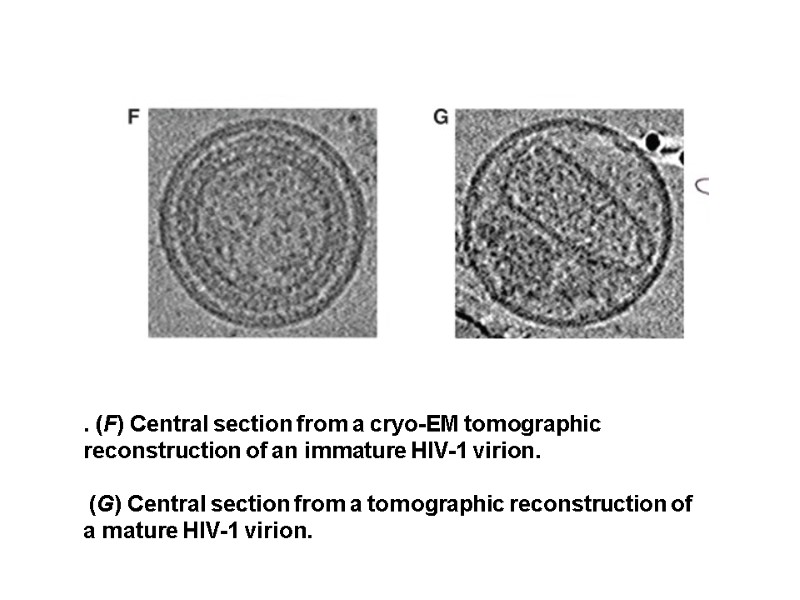
 HIV Proteins Structural Proteins Gag: Matrix (p17), Capsid (p24), nucleocapsid (p7, p9), p6 Pol: Protease, Reverse Transcriptase, Integrase Env: gp120, gp41 Regulatory Proteins Tat Rev Nef Accessory proteins Vif Vpr Vpu
HIV Proteins Structural Proteins Gag: Matrix (p17), Capsid (p24), nucleocapsid (p7, p9), p6 Pol: Protease, Reverse Transcriptase, Integrase Env: gp120, gp41 Regulatory Proteins Tat Rev Nef Accessory proteins Vif Vpr Vpu
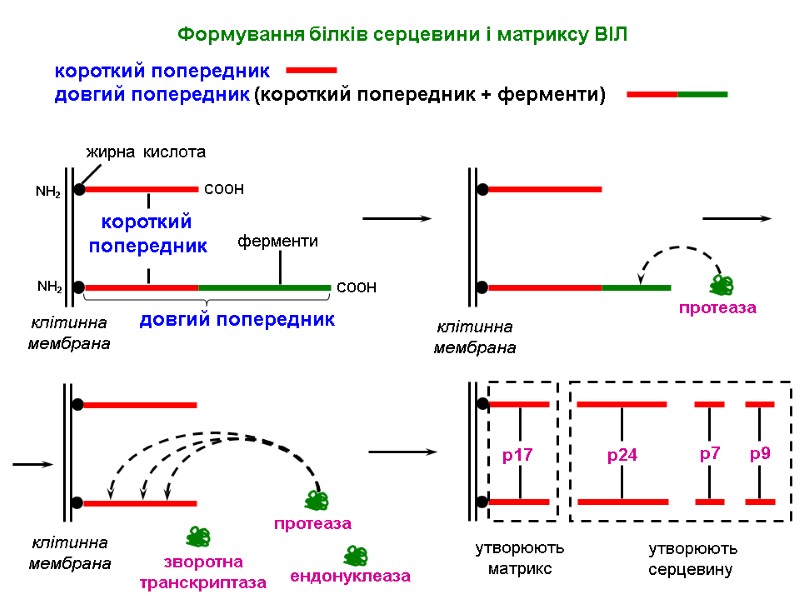
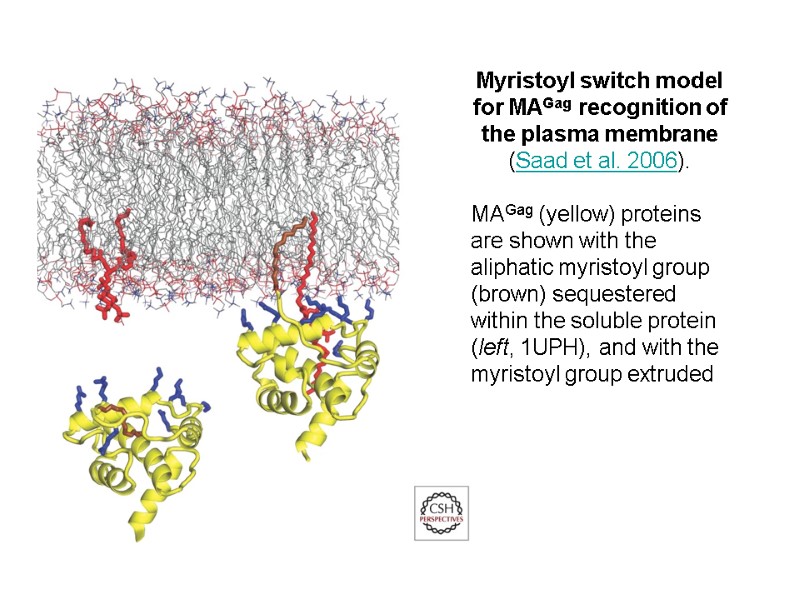
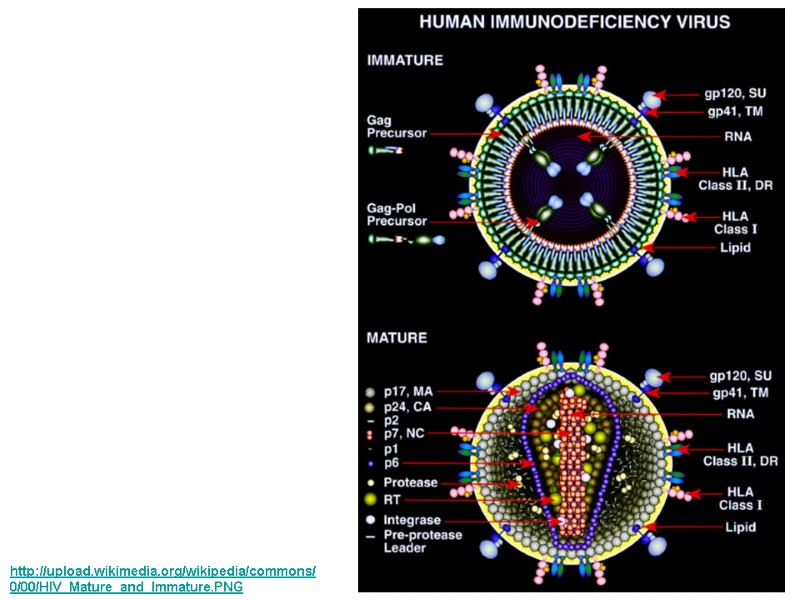
 Gene/Protein Mass, KDa Function gag (Pr) 55gag p55 Gag precursor protein MA - Matrix p17 Aids nuclear import and viral assembly CA - Capsid p24 HIV central core – contains HIV genome and enzymes Matrix p17 Capsid p24 NC p9 p6 p1 p2 myristate Association with membrane Formation of Gag multimers RNA binding RNA packing Viral Entry Envelope Incorporation Viral Budding NC - Nucleocapsid p7/p9 Precise location in virion unknown, not generally present in other retroviruses, may aid in incorporation of Vpr into virion Assoc. with HIV RNA, involved in packaging of HIV RNA into virions p6 p6
Gene/Protein Mass, KDa Function gag (Pr) 55gag p55 Gag precursor protein MA - Matrix p17 Aids nuclear import and viral assembly CA - Capsid p24 HIV central core – contains HIV genome and enzymes Matrix p17 Capsid p24 NC p9 p6 p1 p2 myristate Association with membrane Formation of Gag multimers RNA binding RNA packing Viral Entry Envelope Incorporation Viral Budding NC - Nucleocapsid p7/p9 Precise location in virion unknown, not generally present in other retroviruses, may aid in incorporation of Vpr into virion Assoc. with HIV RNA, involved in packaging of HIV RNA into virions p6 p6
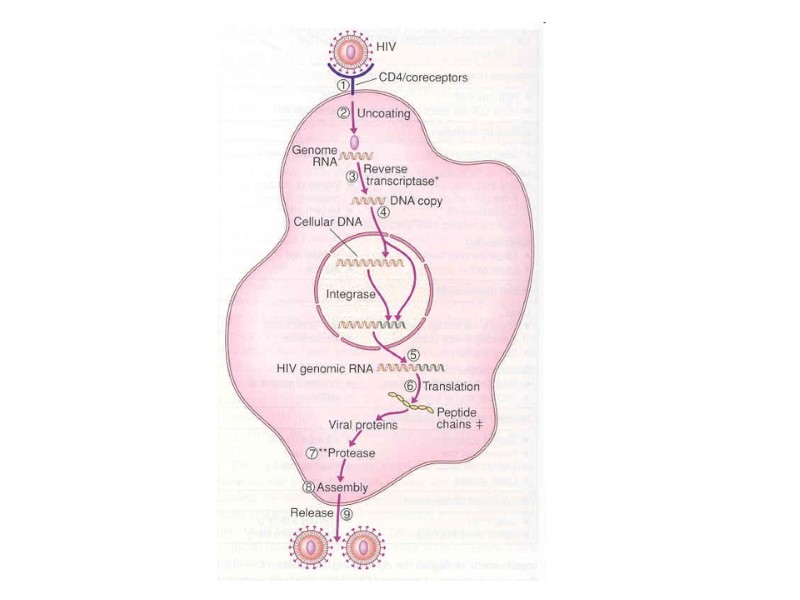
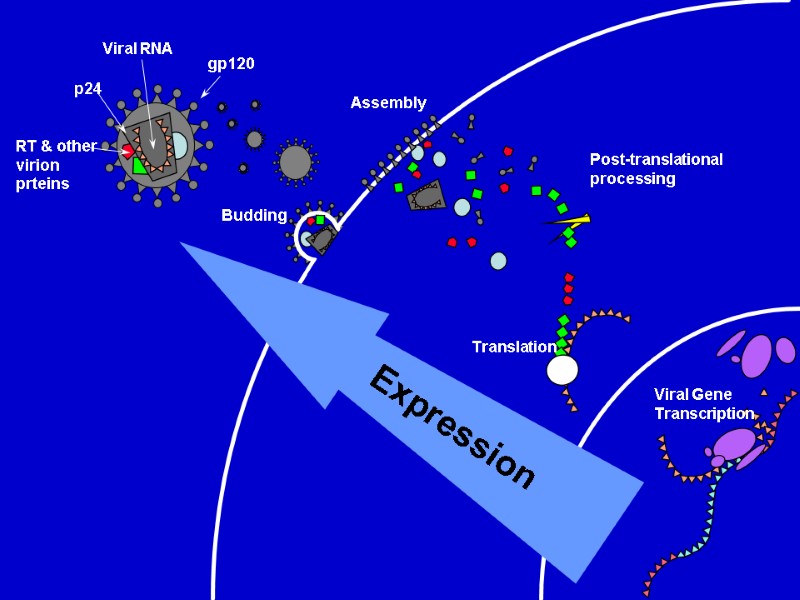
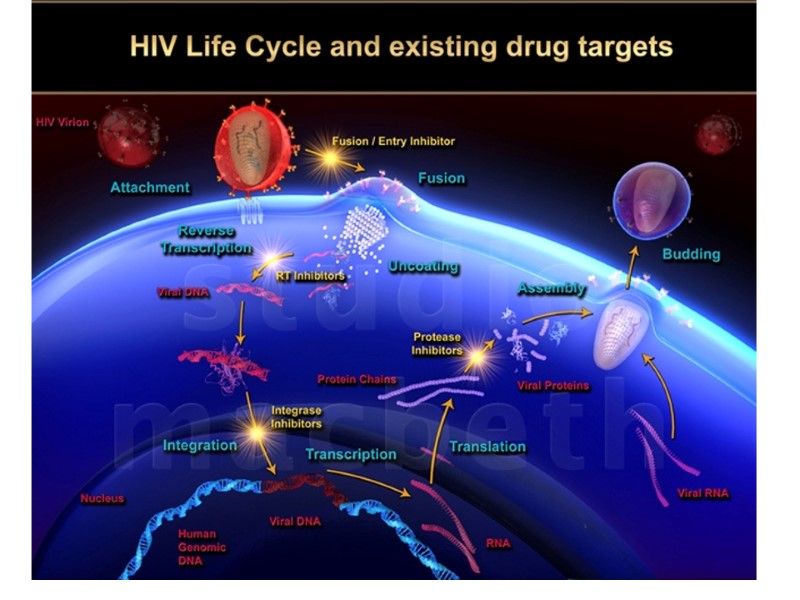
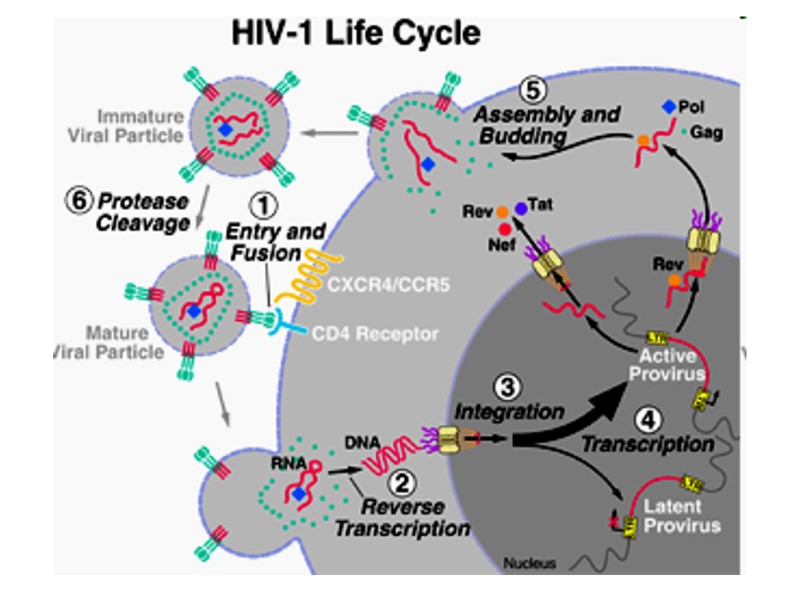
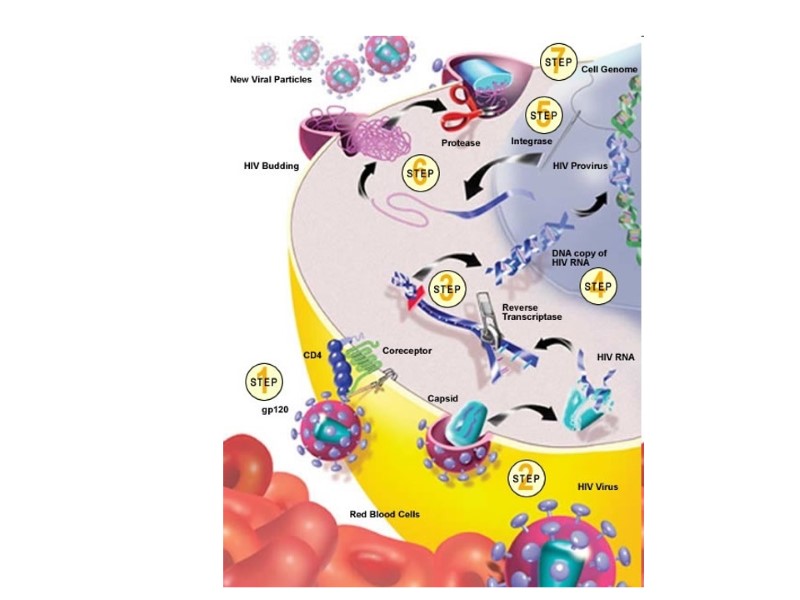
 HIV lifecycle
HIV lifecycle



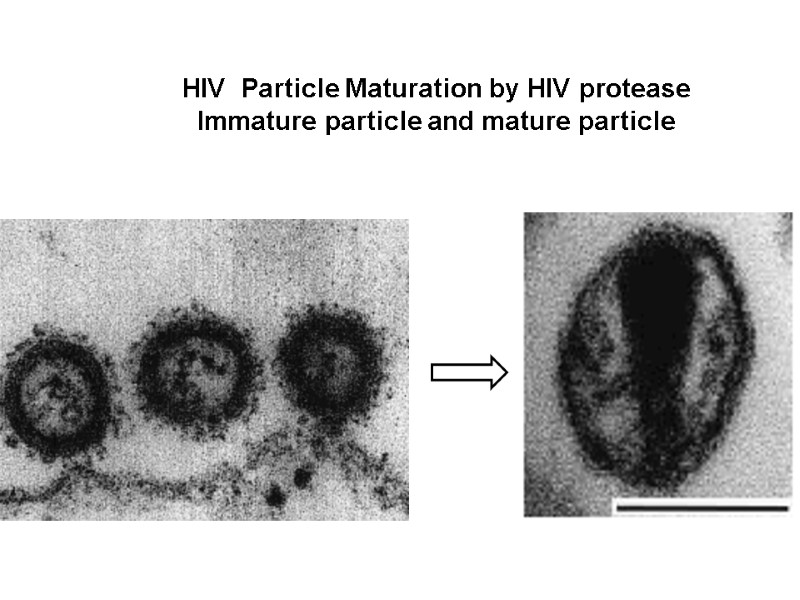
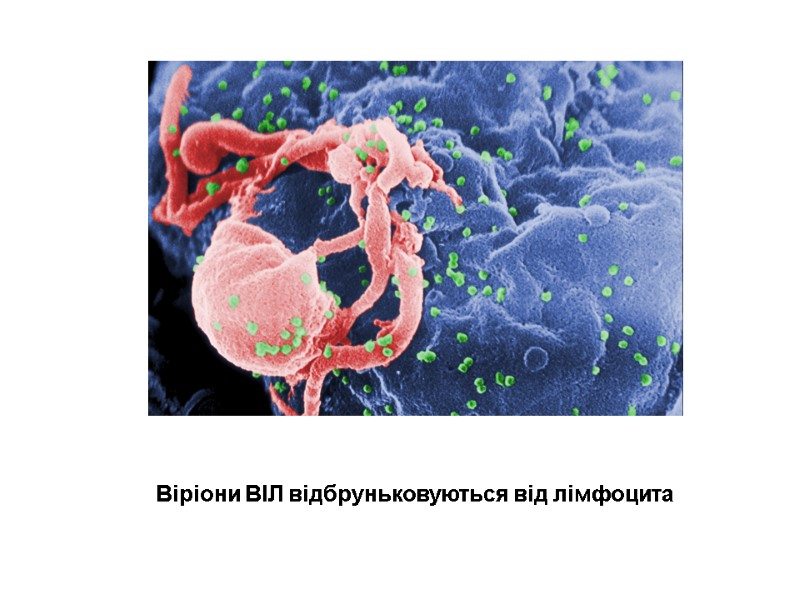
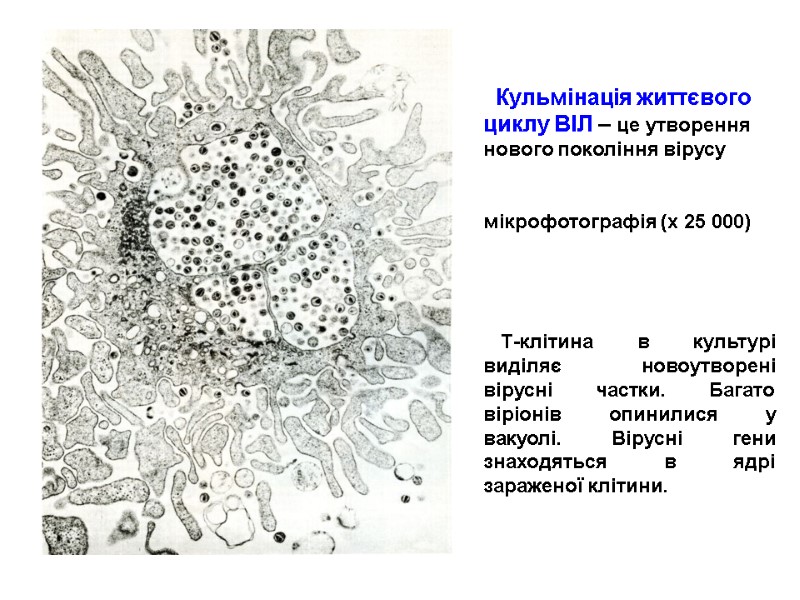
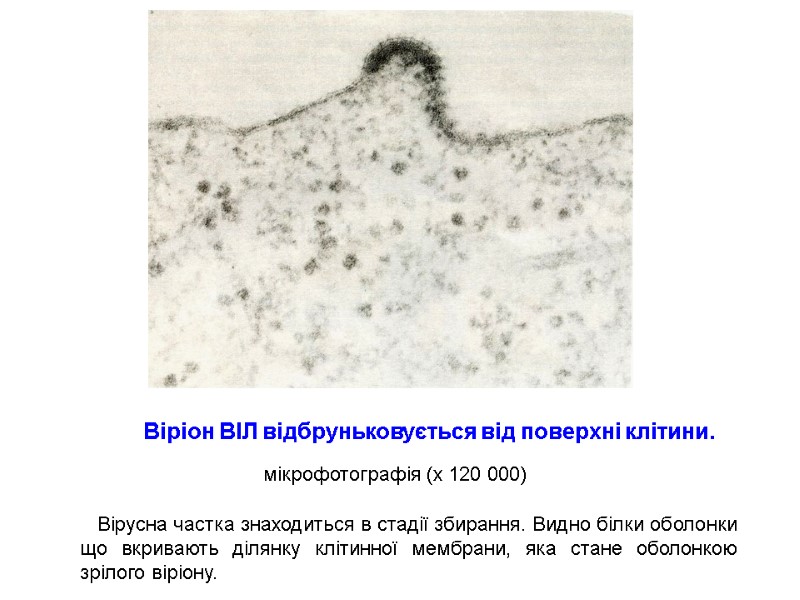
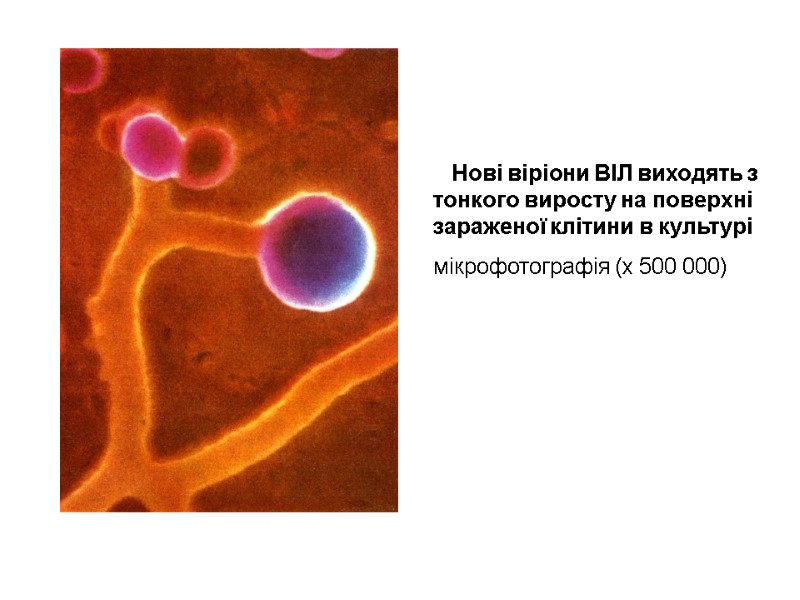
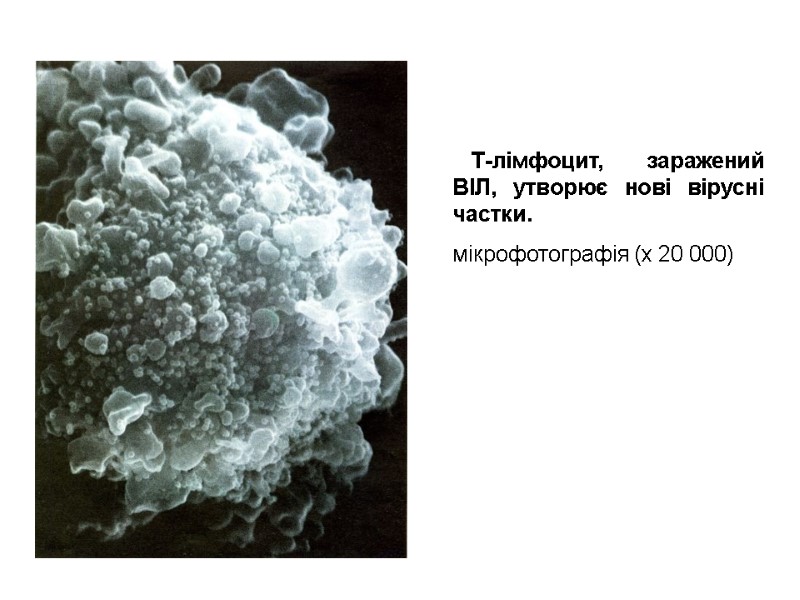
 The imprint of the surface of HIV-infected cell microphotography (х 40 000) Для отримання препарату клітину зневоднюють, заморожують, наносять на її поверхню платину і вуглець. Очищають кислотою, промивають і досліджують в електронному мікроскопі.
The imprint of the surface of HIV-infected cell microphotography (х 40 000) Для отримання препарату клітину зневоднюють, заморожують, наносять на її поверхню платину і вуглець. Очищають кислотою, промивають і досліджують в електронному мікроскопі.
 HIV Lifecycle
HIV Lifecycle
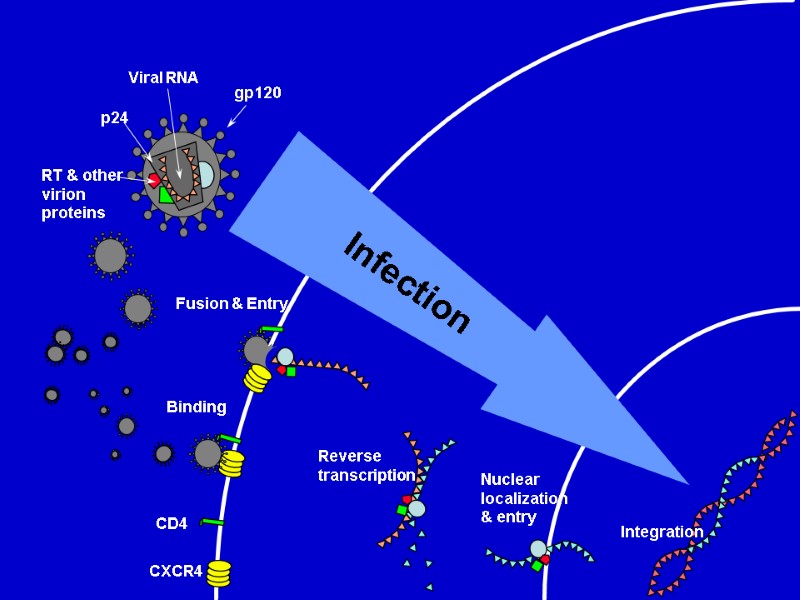
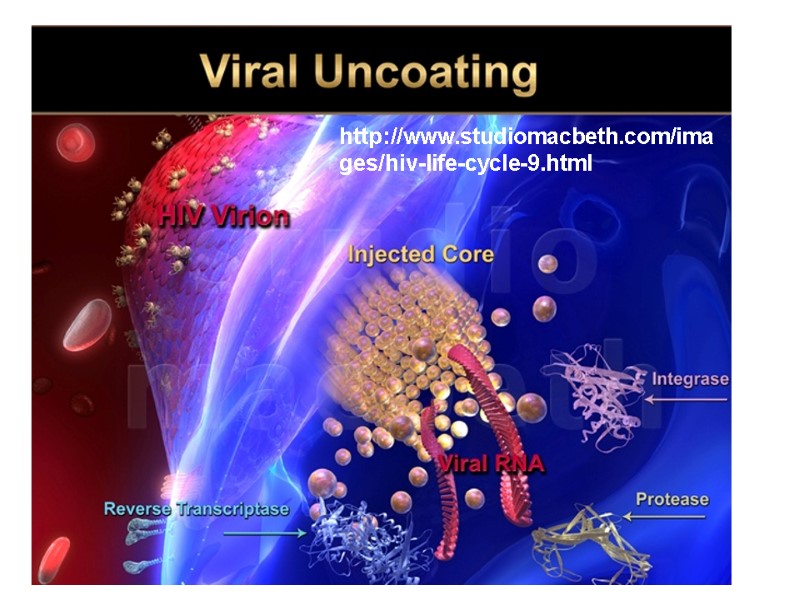
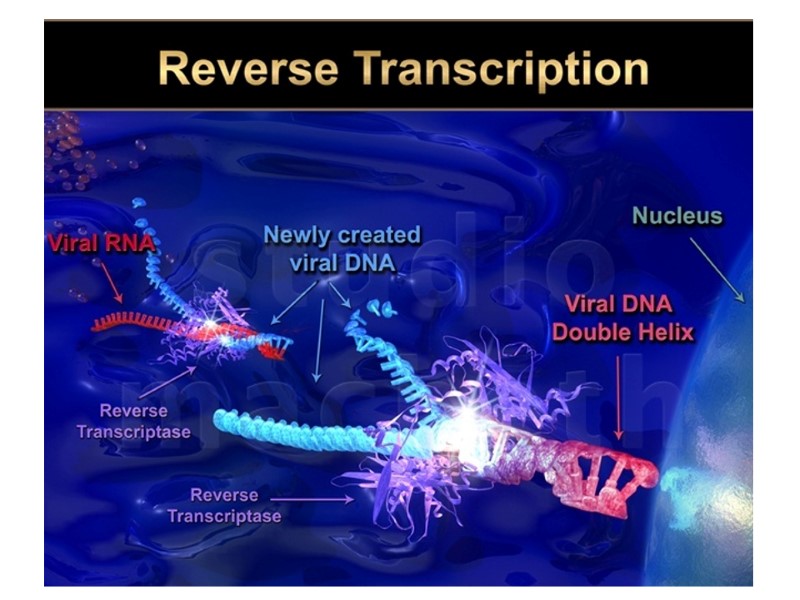
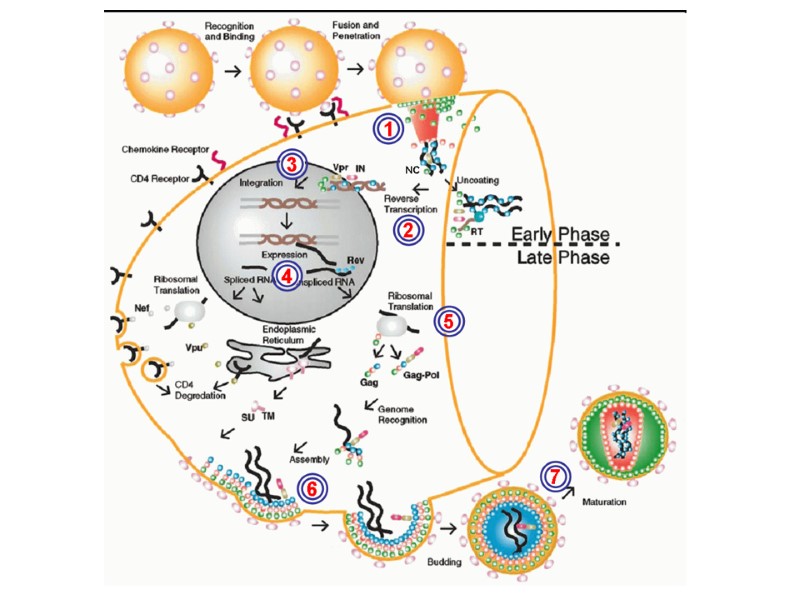
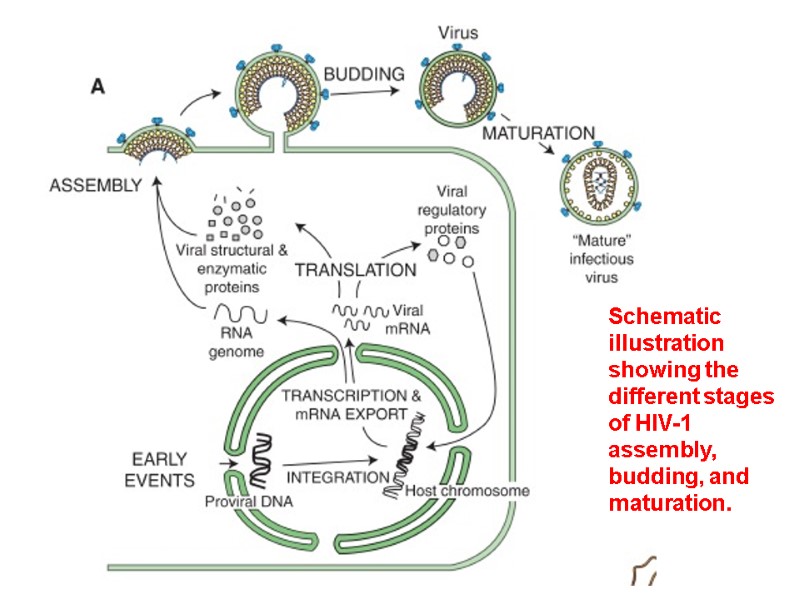
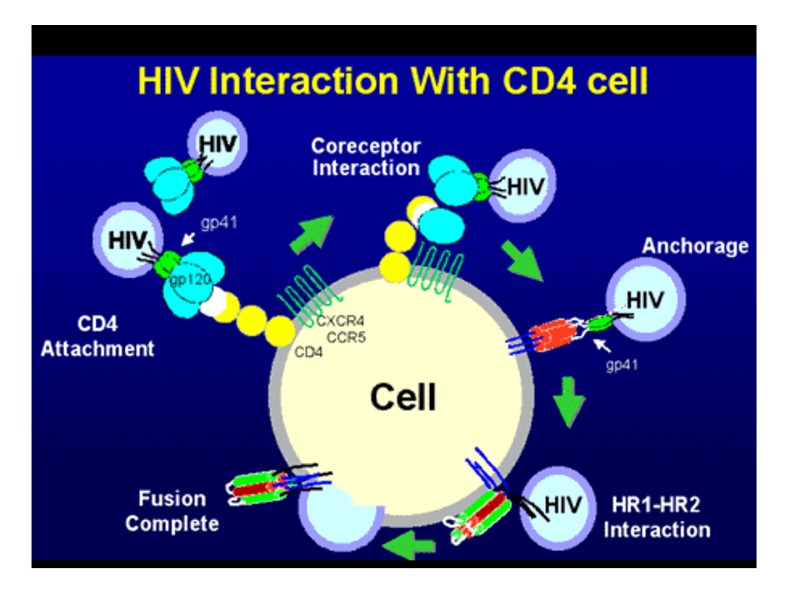
 HIV Lifecycle (simple version) HIV-1 gp120 in the viral surface binds to CD4 receptor and CCR5/CXCR4 coreceptor in T-helper or macrophage membrane. Viral capsid enters the cell. Upon entry, the virus uncoats and reverse transcribes its RNA to DNA. Viral DNA integrates into the host cell genome. Using host cell machinery, the viral DNA is transcribed and translated to produce viral protein used to replicate the virus and increase viral progeny. Newly formed virus can then bud off from the cell and are free to infect neighboring healthy cells.
HIV Lifecycle (simple version) HIV-1 gp120 in the viral surface binds to CD4 receptor and CCR5/CXCR4 coreceptor in T-helper or macrophage membrane. Viral capsid enters the cell. Upon entry, the virus uncoats and reverse transcribes its RNA to DNA. Viral DNA integrates into the host cell genome. Using host cell machinery, the viral DNA is transcribed and translated to produce viral protein used to replicate the virus and increase viral progeny. Newly formed virus can then bud off from the cell and are free to infect neighboring healthy cells.
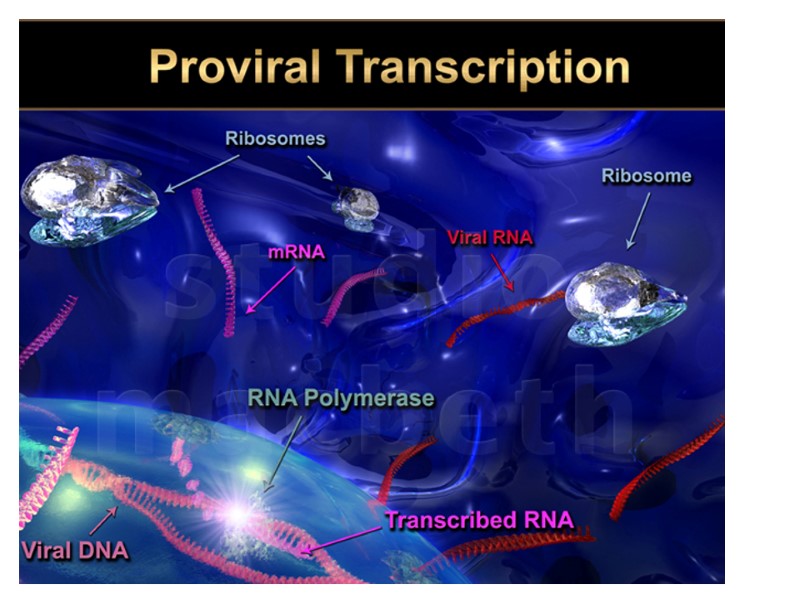
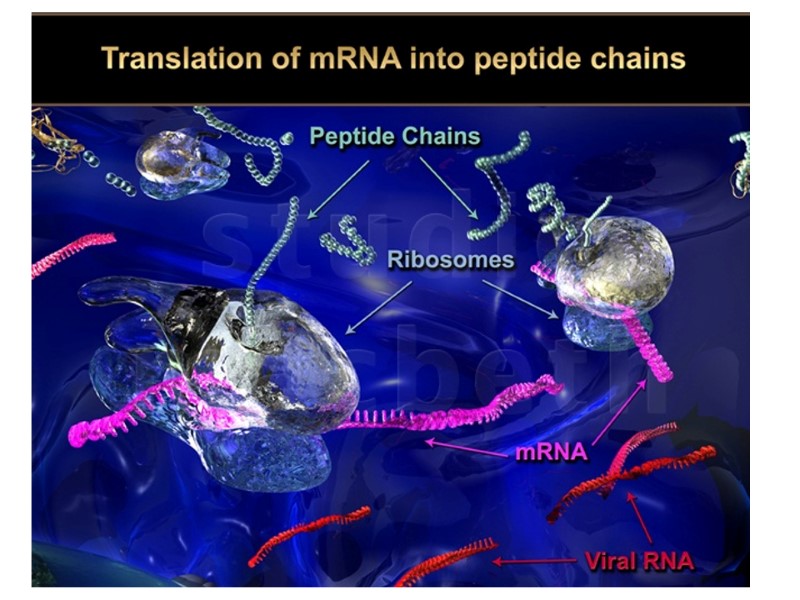
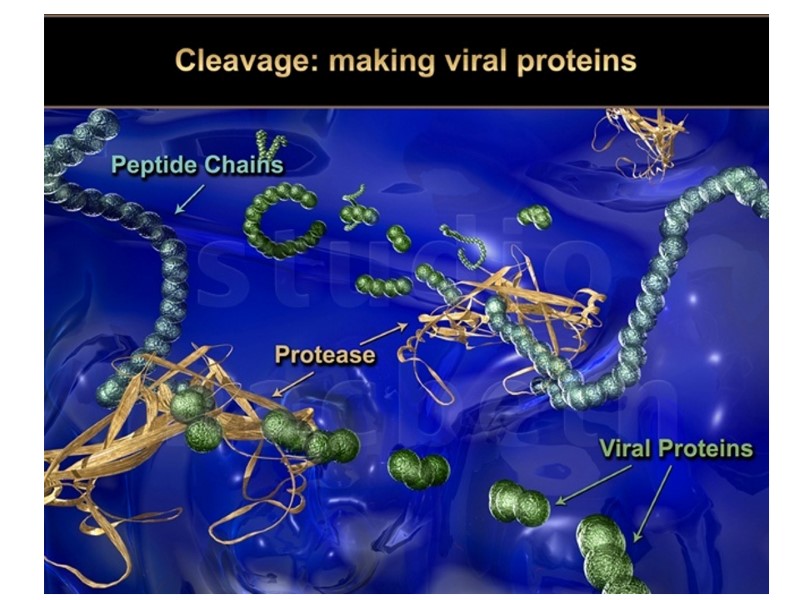
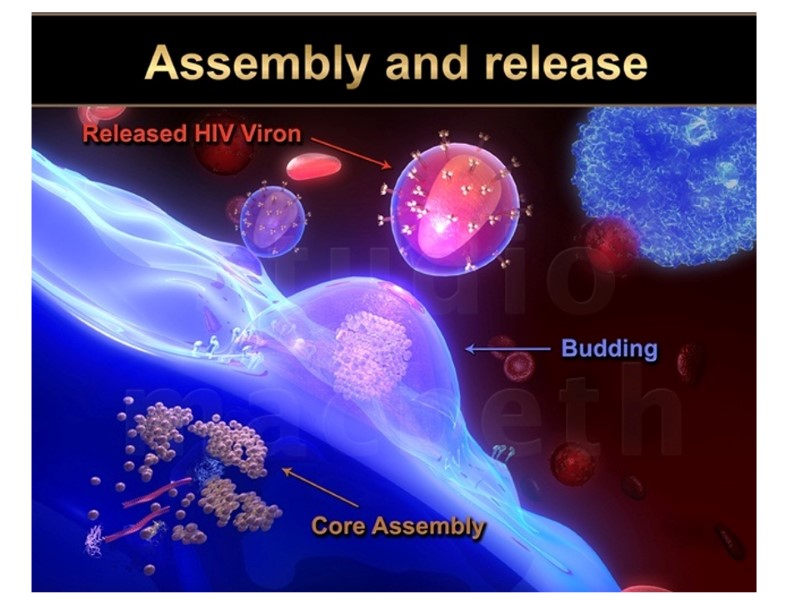
 HIV Lifecycle 1. Вірусна частка приєднується до поверхні клітини. Глікопротеїн gp120 зв’язується з CD4-рецептором і CCR5/CXCR4-корецептором на Т-клітині. 2. Оболонка вірусу і мембрана клітини зливаються, серцевина віріону (білки та РНК) проникає в цитоплазму клітини (uncoated virus). 3. Зворотна транскриптаза (знаходиться в серцевині) синтезує одноланцюгову ДНК, комплементарну вірусній РНК. Після розщеплення вірусної РНК синтезується другий ланцюг ДНК. 4. Дволанцюгова вірусна ДНК (провірус), за сприяння інтегрази, проникає в ядро клітини і вбудовується в хромосомну ДНК. Далі може бути літична або прихована (латентна) фаза. Це залежить від вірусних білків або клітинних факторів. 5. Коли провірус активується, за допомогою клітинного механізму транскрипції утворюються мРНК (РНК-копії провірусу). 6. З мРНК на рибосомах клітини синтезуються вірусні білки і надходять до клітинної мембрани. З білків і РНК-копій формуються нові вірусні частки, що залишають клітину, відбруньковуючись від її поверхні. 7. Дозрівання віріону відбувається, коли він відокремлюється від клітини: протеаза ВІЛ розщеплює білки-попередники на білки матриксу, серцевини, нуклеокапсиду та ферменти.
HIV Lifecycle 1. Вірусна частка приєднується до поверхні клітини. Глікопротеїн gp120 зв’язується з CD4-рецептором і CCR5/CXCR4-корецептором на Т-клітині. 2. Оболонка вірусу і мембрана клітини зливаються, серцевина віріону (білки та РНК) проникає в цитоплазму клітини (uncoated virus). 3. Зворотна транскриптаза (знаходиться в серцевині) синтезує одноланцюгову ДНК, комплементарну вірусній РНК. Після розщеплення вірусної РНК синтезується другий ланцюг ДНК. 4. Дволанцюгова вірусна ДНК (провірус), за сприяння інтегрази, проникає в ядро клітини і вбудовується в хромосомну ДНК. Далі може бути літична або прихована (латентна) фаза. Це залежить від вірусних білків або клітинних факторів. 5. Коли провірус активується, за допомогою клітинного механізму транскрипції утворюються мРНК (РНК-копії провірусу). 6. З мРНК на рибосомах клітини синтезуються вірусні білки і надходять до клітинної мембрани. З білків і РНК-копій формуються нові вірусні частки, що залишають клітину, відбруньковуючись від її поверхні. 7. Дозрівання віріону відбувається, коли він відокремлюється від клітини: протеаза ВІЛ розщеплює білки-попередники на білки матриксу, серцевини, нуклеокапсиду та ферменти.
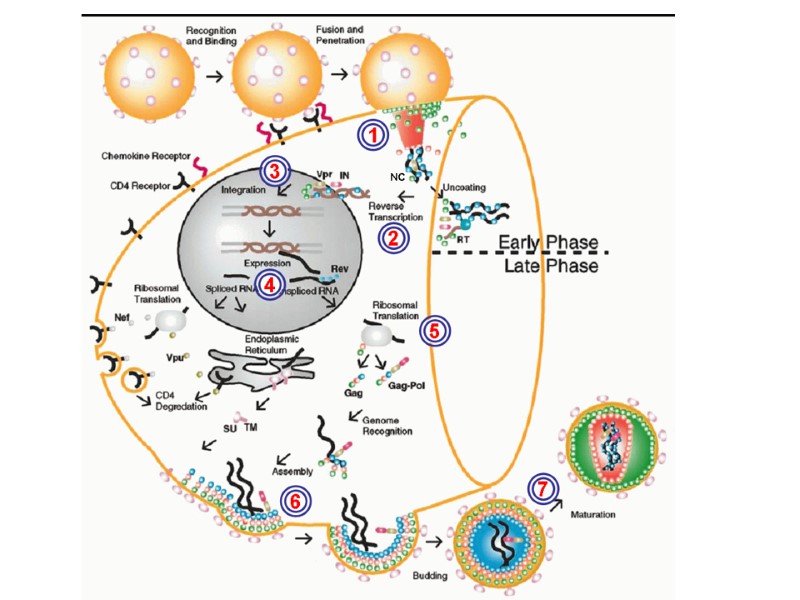
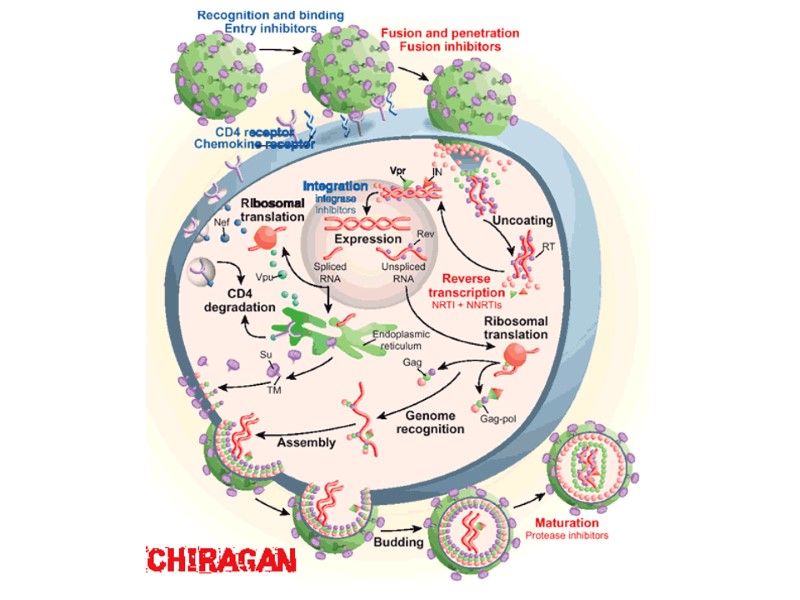
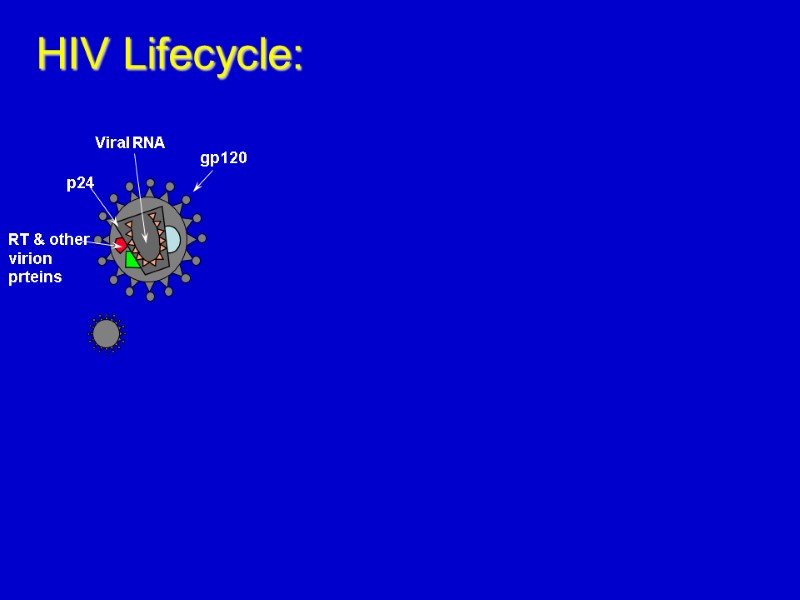
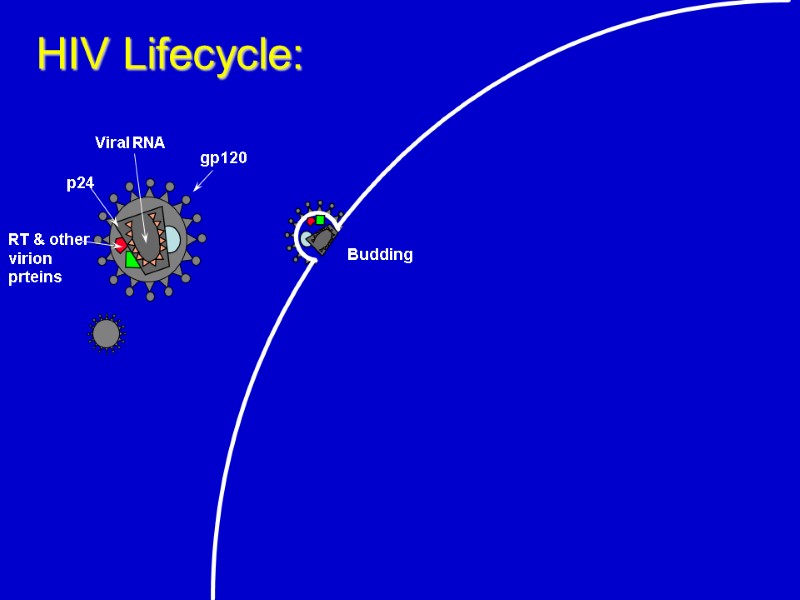
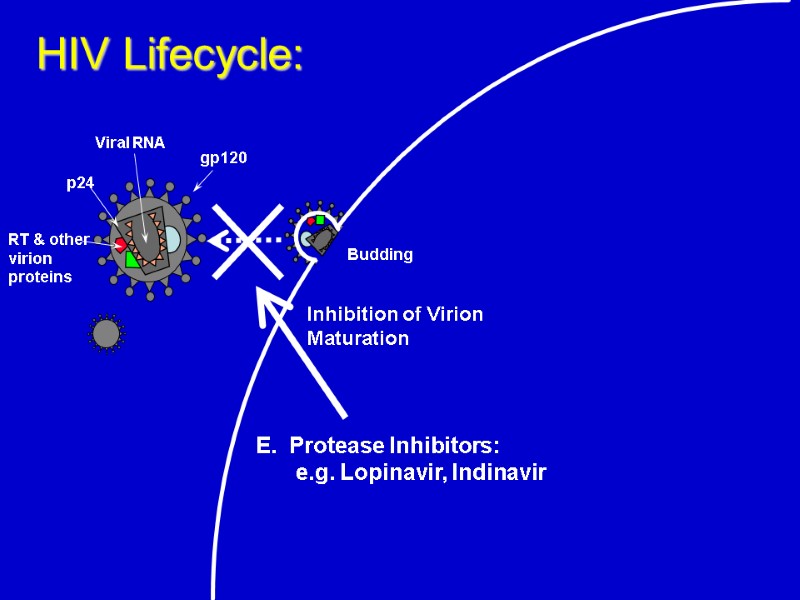
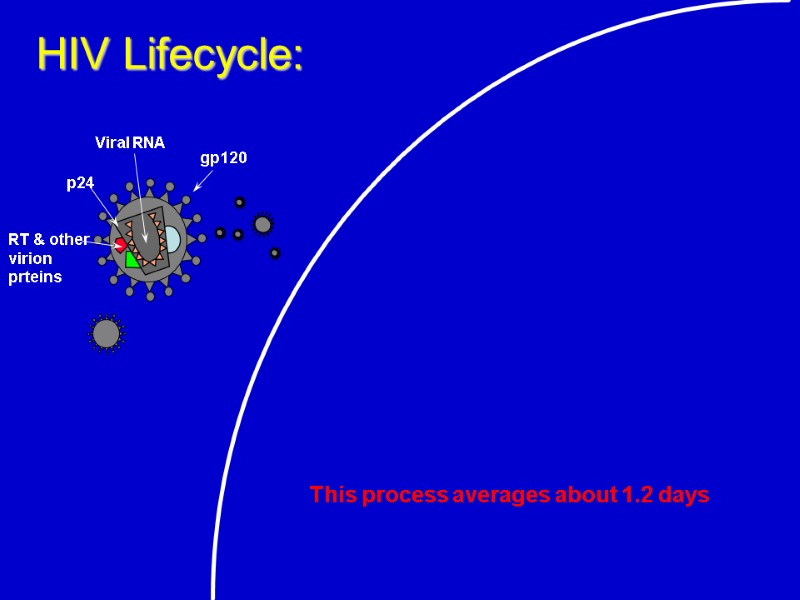
 HIV Lifecycle:
HIV Lifecycle:
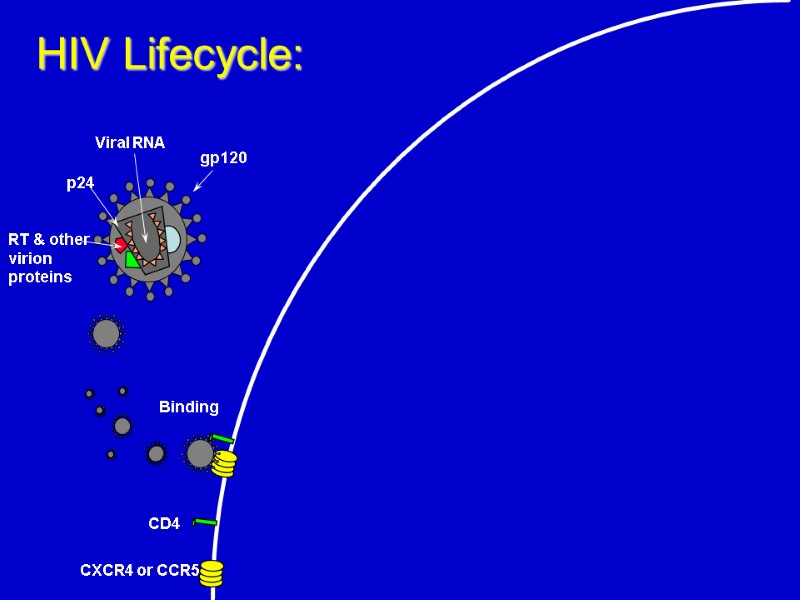
 CD4 CXCR4 or CCR5 Binding HIV Lifecycle:
CD4 CXCR4 or CCR5 Binding HIV Lifecycle:
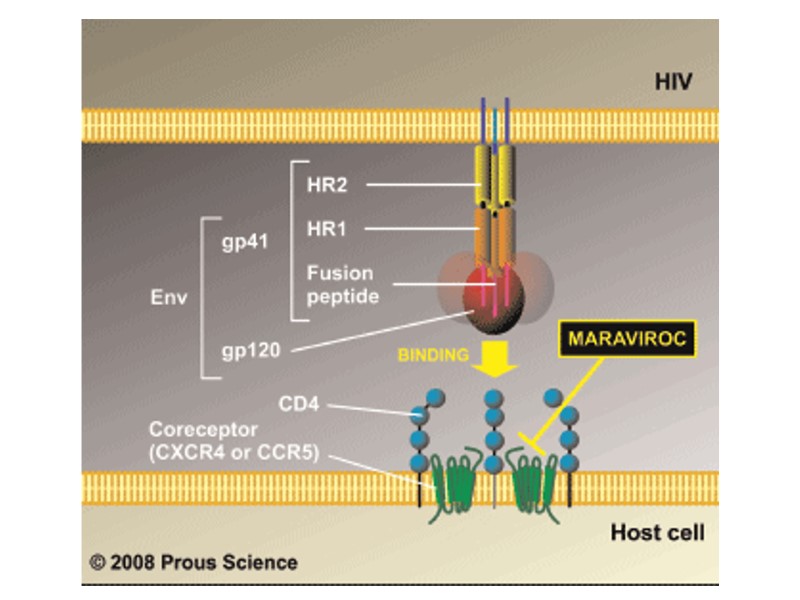
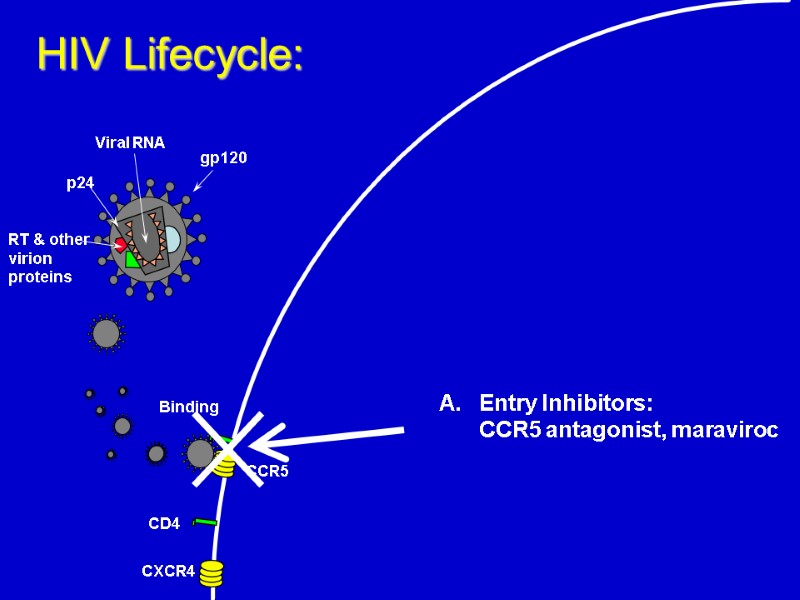
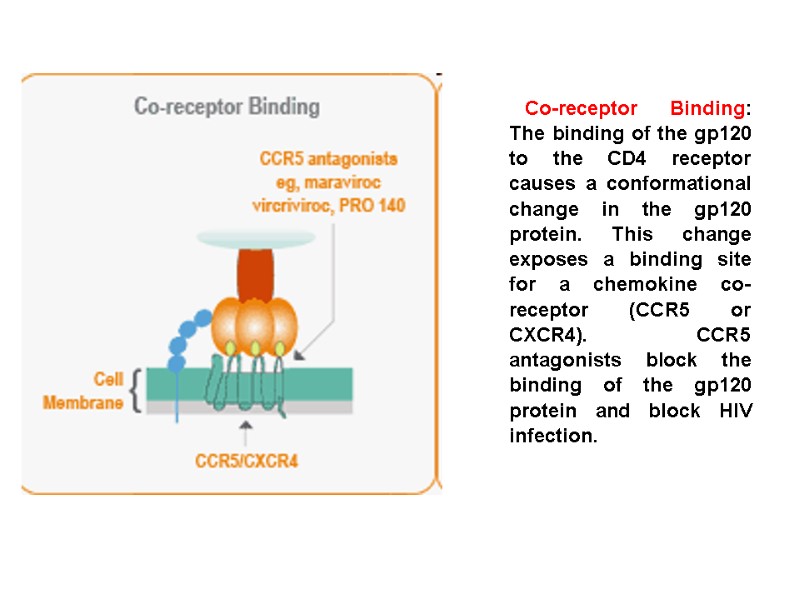
 CD4 CXCR4 Binding HIV Lifecycle: Entry Inhibitors: CCR5 antagonist, maraviroc CCR5
CD4 CXCR4 Binding HIV Lifecycle: Entry Inhibitors: CCR5 antagonist, maraviroc CCR5
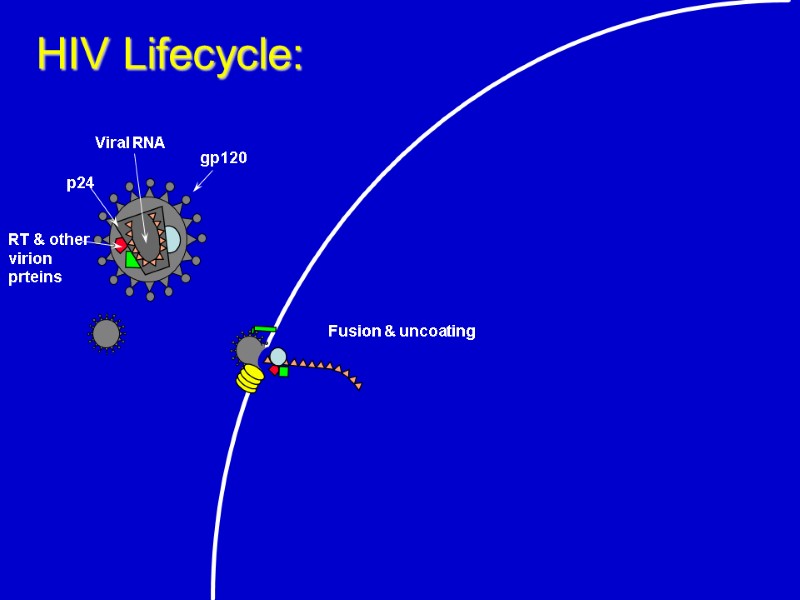
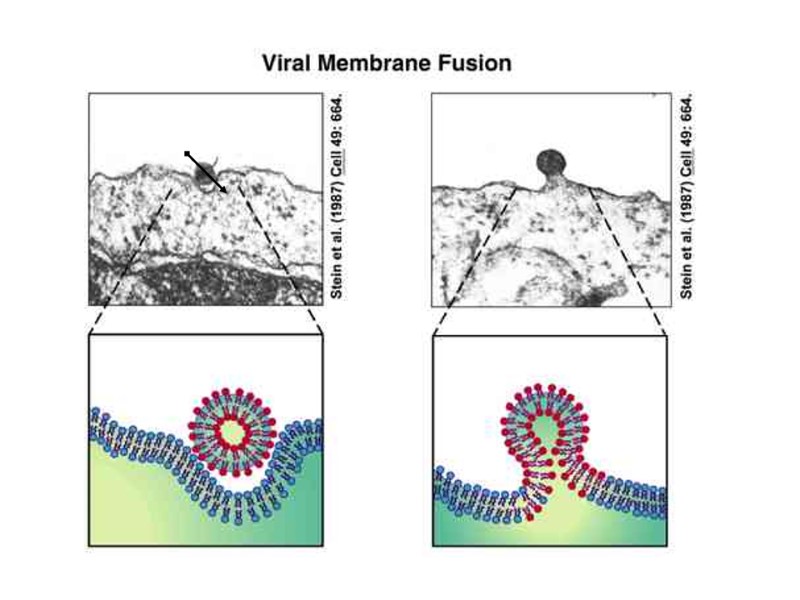
 Fusion & uncoating HIV Lifecycle:
Fusion & uncoating HIV Lifecycle:
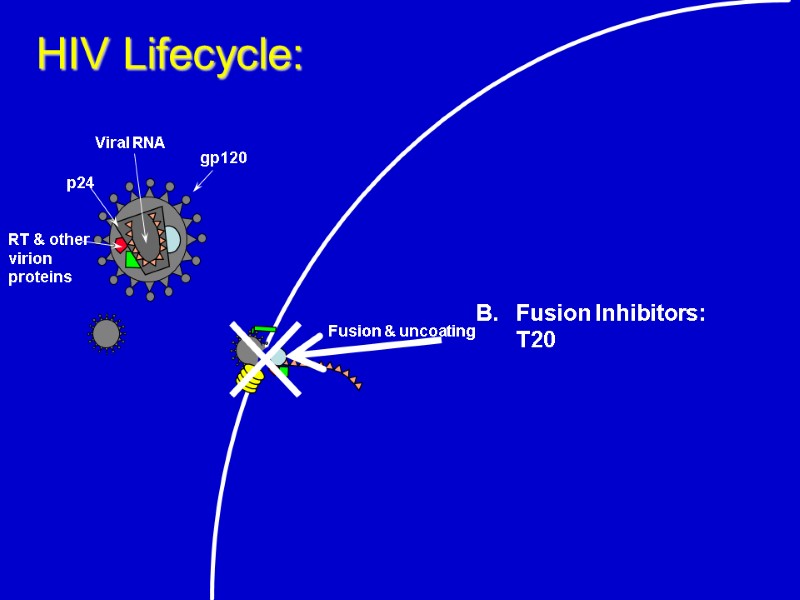
 Fusion & uncoating HIV Lifecycle: B. Fusion Inhibitors: T20
Fusion & uncoating HIV Lifecycle: B. Fusion Inhibitors: T20
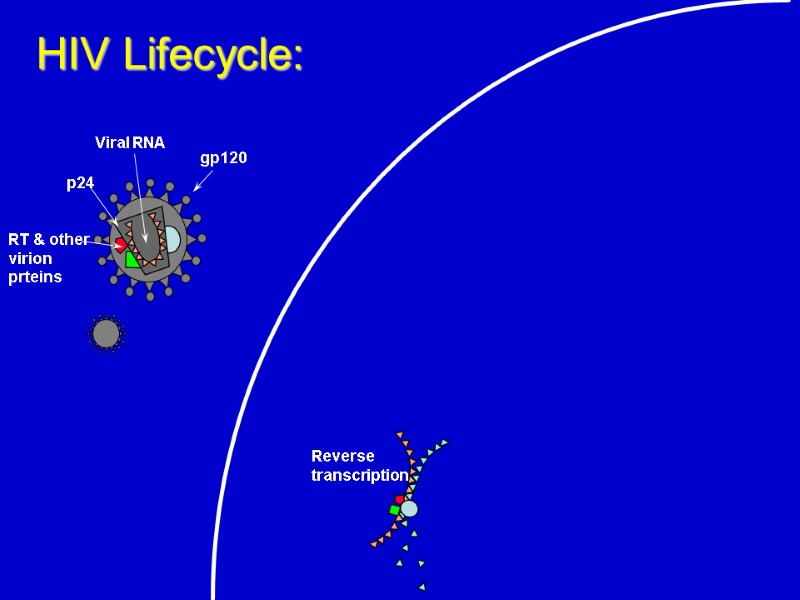
 HIV Lifecycle:
HIV Lifecycle:
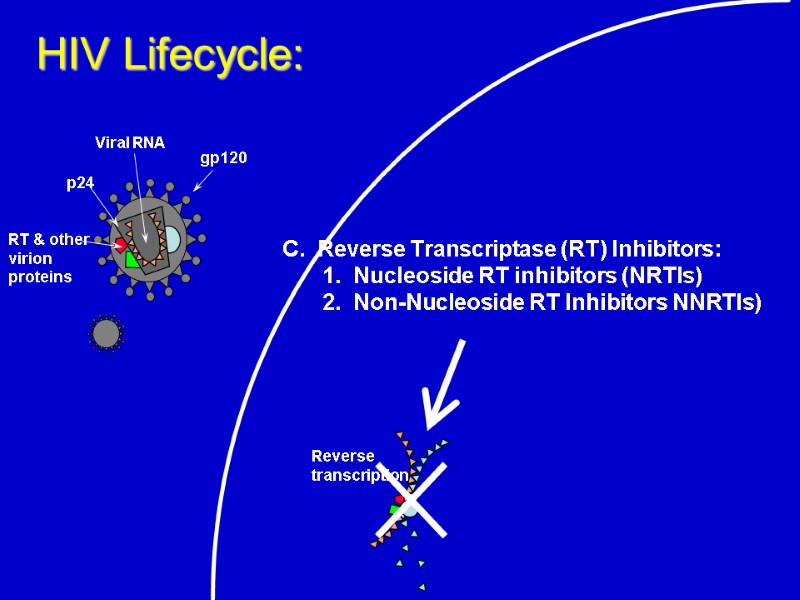
 HIV Lifecycle: C. Reverse Transcriptase (RT) Inhibitors: 1. Nucleoside RT inhibitors (NRTIs) 2. Non-Nucleoside RT Inhibitors NNRTIs)
HIV Lifecycle: C. Reverse Transcriptase (RT) Inhibitors: 1. Nucleoside RT inhibitors (NRTIs) 2. Non-Nucleoside RT Inhibitors NNRTIs)
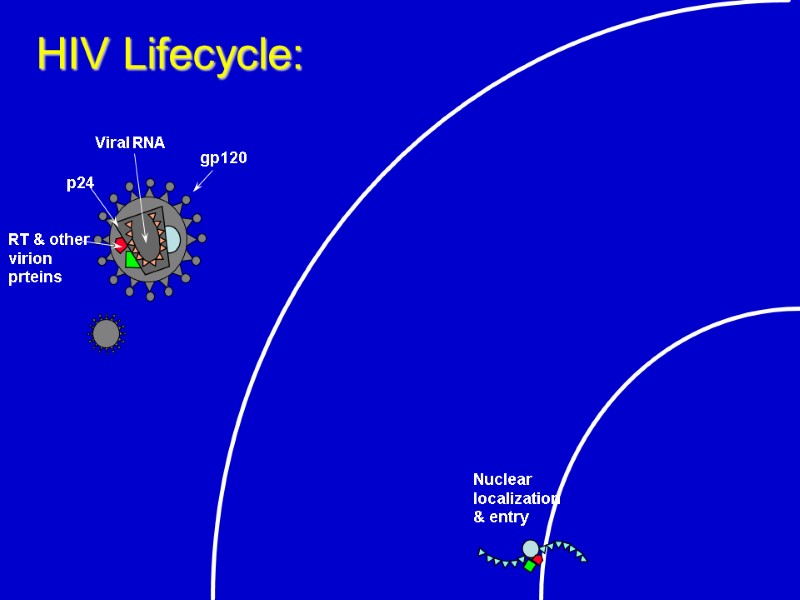
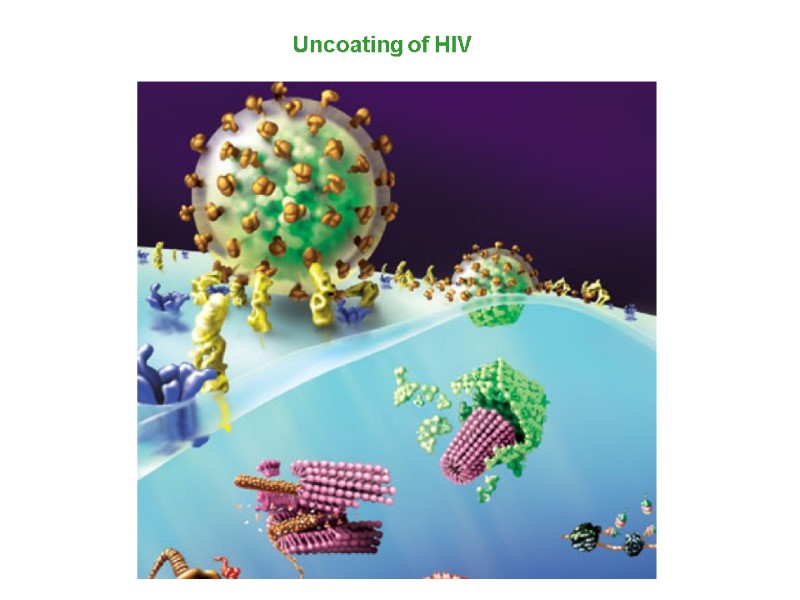
 Nuclear localization & entry HIV Lifecycle:
Nuclear localization & entry HIV Lifecycle:
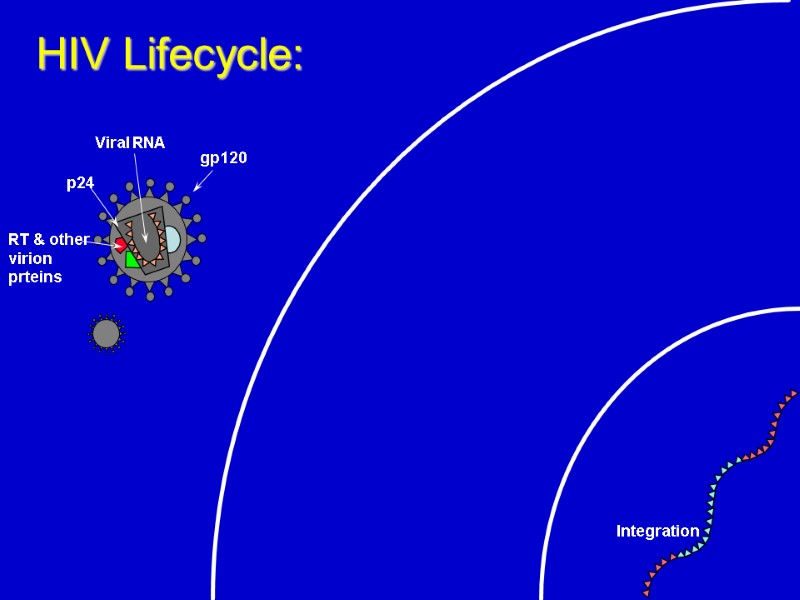
 HIV Lifecycle: Integration
HIV Lifecycle: Integration
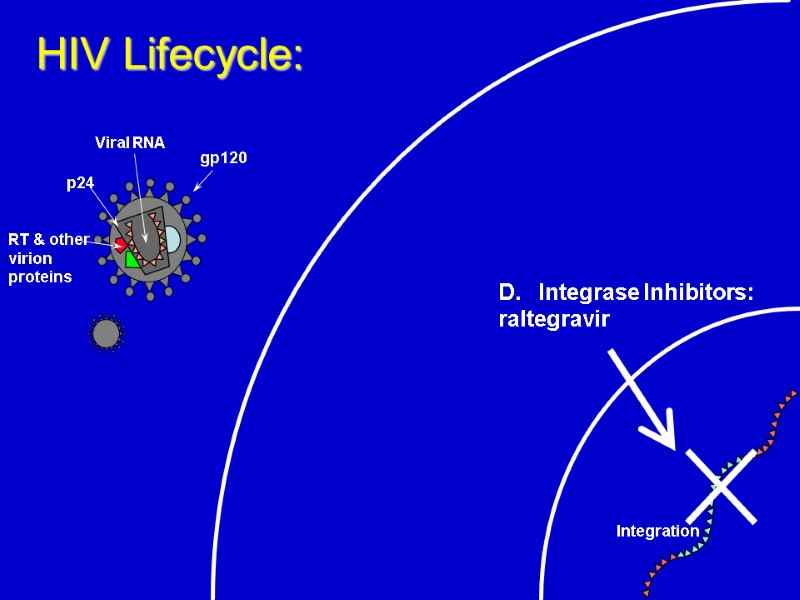
 HIV Lifecycle: Integration Integrase Inhibitors: raltegravir
HIV Lifecycle: Integration Integrase Inhibitors: raltegravir
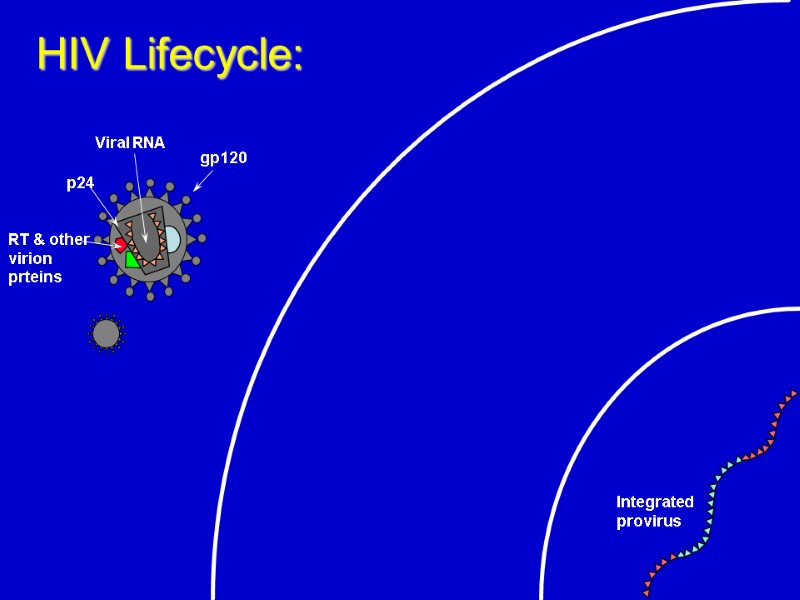
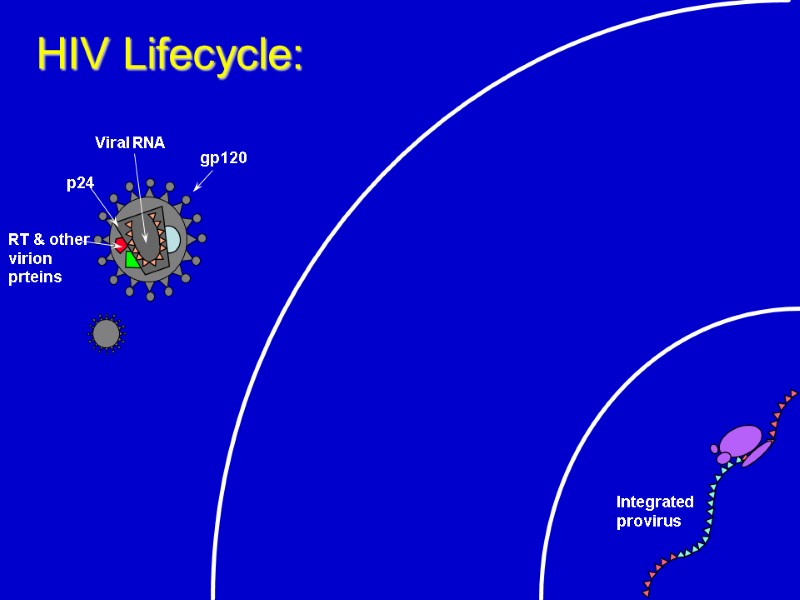
 HIV Lifecycle: Integrated provirus
HIV Lifecycle: Integrated provirus
 Integrated provirus HIV Lifecycle:
Integrated provirus HIV Lifecycle:
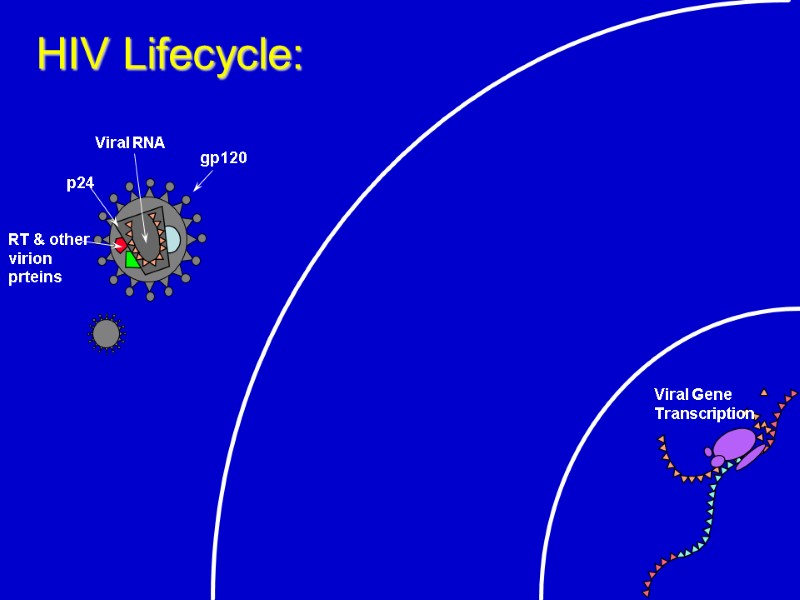
 HIV Lifecycle:
HIV Lifecycle:
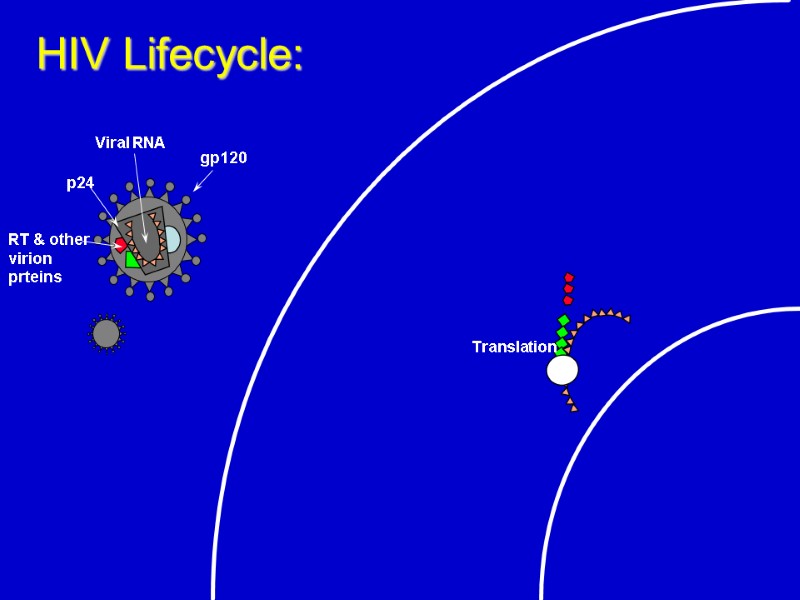
 Translation HIV Lifecycle:
Translation HIV Lifecycle:
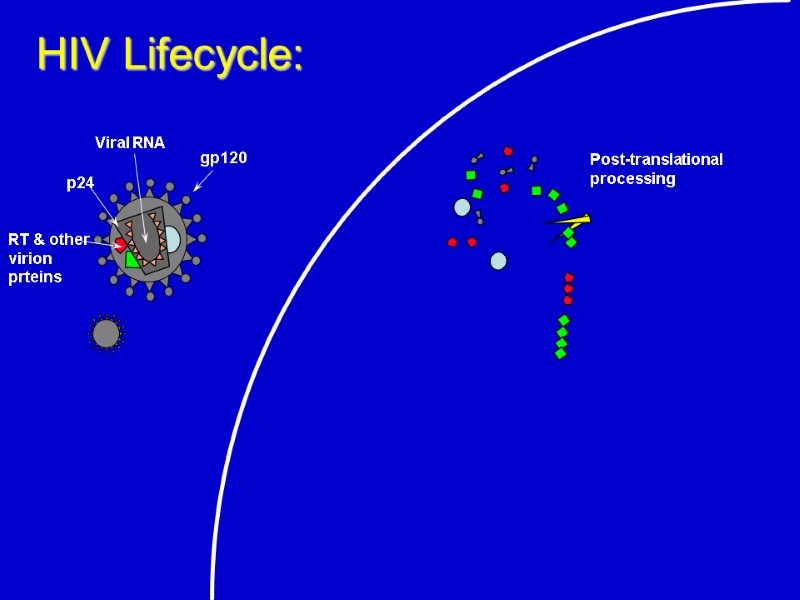
 Post-translational processing HIV Lifecycle:
Post-translational processing HIV Lifecycle:
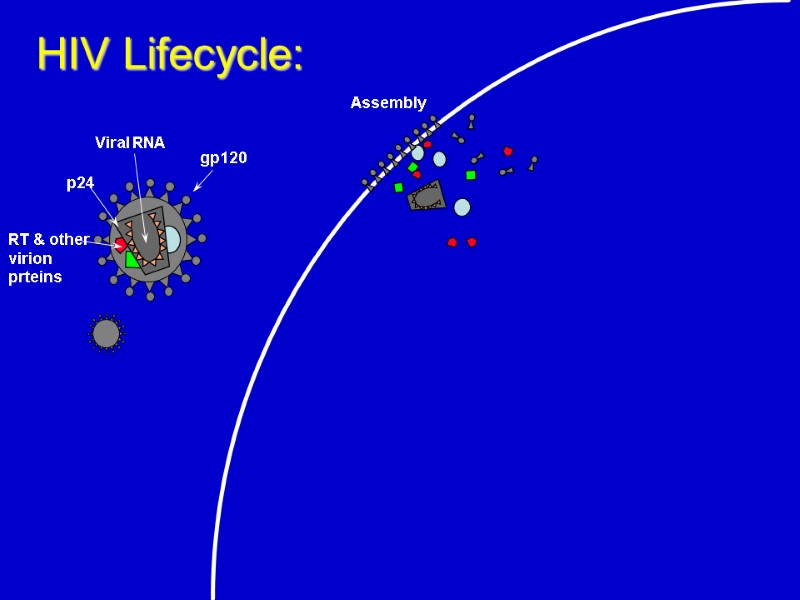
 Assembly HIV Lifecycle:
Assembly HIV Lifecycle:
 Budding HIV Lifecycle:
Budding HIV Lifecycle:
 Budding HIV Lifecycle: E. Protease Inhibitors: e.g. Lopinavir, Indinavir Inhibition of Virion Maturation
Budding HIV Lifecycle: E. Protease Inhibitors: e.g. Lopinavir, Indinavir Inhibition of Virion Maturation
 HIV Lifecycle: This process averages about 1.2 days
HIV Lifecycle: This process averages about 1.2 days
 CD4 CXCR4 Binding Fusion & Entry Nuclear localization & entry Integration Infection
CD4 CXCR4 Binding Fusion & Entry Nuclear localization & entry Integration Infection
 Expression Budding Assembly Translation Post-translational processing
Expression Budding Assembly Translation Post-translational processing
 http://www.studiomacbeth.com/images/hiv-life-cycle-9.html
http://www.studiomacbeth.com/images/hiv-life-cycle-9.html










 HIV Lifecycle (Entry to the cell) Initial interaction between gp120 and CD4. Conformational change in gp120 allows for secondary interaction with CCR5. The distal tips of gp41 are inserted into the cellular membrane. gp41 undergoes significant conformational change; folding in half and forming coiled-coils. This process pulls the viral and cellular membranes together, fusing them. The HIV virion enters macrophages and CD4+ T-cells by the adsorbtion of glycoproteins gp120 on its surface to receptors on the target cell. It is followed by fusion of the viral envelope with the cell membrane and the release of the HIV capsid into the cell.
HIV Lifecycle (Entry to the cell) Initial interaction between gp120 and CD4. Conformational change in gp120 allows for secondary interaction with CCR5. The distal tips of gp41 are inserted into the cellular membrane. gp41 undergoes significant conformational change; folding in half and forming coiled-coils. This process pulls the viral and cellular membranes together, fusing them. The HIV virion enters macrophages and CD4+ T-cells by the adsorbtion of glycoproteins gp120 on its surface to receptors on the target cell. It is followed by fusion of the viral envelope with the cell membrane and the release of the HIV capsid into the cell.

 Uncoating of HIV
Uncoating of HIV
 HIV core enters the cytoplasm of the host cell… Uncoating of the virus 1. Deproteinization of nucleocapsid and the release of genomic RNA HIV capsid dissociates within the cell in a multistep process known as uncoating. It influences completion of reverse transcription of the viral genome. Double-stranded viral DNA is imported into the nucleus for integration into the host genome (it is hallmark of retroviral infection). Reverse transcription, nuclear entry, and integration are coordinated by a capsid uncoating process that is regulated by cellular proteins.
HIV core enters the cytoplasm of the host cell… Uncoating of the virus 1. Deproteinization of nucleocapsid and the release of genomic RNA HIV capsid dissociates within the cell in a multistep process known as uncoating. It influences completion of reverse transcription of the viral genome. Double-stranded viral DNA is imported into the nucleus for integration into the host genome (it is hallmark of retroviral infection). Reverse transcription, nuclear entry, and integration are coordinated by a capsid uncoating process that is regulated by cellular proteins.
 The HIV-1 capsid likely interacts with multiple host cell factors for trafficking of the viral genome to the nucleus for proper uncoating kinetics and Virology. 2014 Apr;454-455:371-9. HIV-1 uncoating: connection to nuclear entry and regulation by host proteins. Ambrose Z1, Aiken C2.
The HIV-1 capsid likely interacts with multiple host cell factors for trafficking of the viral genome to the nucleus for proper uncoating kinetics and Virology. 2014 Apr;454-455:371-9. HIV-1 uncoating: connection to nuclear entry and regulation by host proteins. Ambrose Z1, Aiken C2.
 HIV-1 uncoating is linked to reverse transcription and nuclear import and requires host cell factors.
HIV-1 uncoating is linked to reverse transcription and nuclear import and requires host cell factors.
 Eukaryotic elongation factor 1 complex subunits are critical HIV-1 reverse transcription cofactors.Warren K, Wei T, Li D, Qin F, Warrilow D, Lin MH, Sivakumaran H, Apolloni A, Abbott CM, Jones A, Anderson JL, Harrich D Proc Natl Acad Sci U S A. 2012 Jun 12; 109(24):9587-92. The viral capsid influences HIV-1 reverse transcription Completion of HIV-1 reverse transcription may require at least partial capsid dissociation. Viral DNA synthesis requires cellular factors.
Eukaryotic elongation factor 1 complex subunits are critical HIV-1 reverse transcription cofactors.Warren K, Wei T, Li D, Qin F, Warrilow D, Lin MH, Sivakumaran H, Apolloni A, Abbott CM, Jones A, Anderson JL, Harrich D Proc Natl Acad Sci U S A. 2012 Jun 12; 109(24):9587-92. The viral capsid influences HIV-1 reverse transcription Completion of HIV-1 reverse transcription may require at least partial capsid dissociation. Viral DNA synthesis requires cellular factors.
 2 molecules of HIV RNA tRNALys 2 molecules of reverse transcriptase and integrase HIV Vpr protein Capsid and nucleocapsid proteins. It is stabilized by HIV Vif protein. 2. The formation of HIV-1 reverse transcription complex A stepwise uncoating process occur during reverse transcription.
2 molecules of HIV RNA tRNALys 2 molecules of reverse transcriptase and integrase HIV Vpr protein Capsid and nucleocapsid proteins. It is stabilized by HIV Vif protein. 2. The formation of HIV-1 reverse transcription complex A stepwise uncoating process occur during reverse transcription.
 1. Утворення комплексу зворотної транскрипції: 2 молекули РНК ВІЛ тРНКліз 2 молекули зворотної транскриптази та інтегрази білок Vpr ВІЛ матриксні та нуклеокапсидні білки ВІЛ. Асоційований з актиновими мікрофібрилами клітини. Стабілізується білком Vif ВІЛ. Поетапне роздягання вірусу відбувається під час зворотної транскрипції
1. Утворення комплексу зворотної транскрипції: 2 молекули РНК ВІЛ тРНКліз 2 молекули зворотної транскриптази та інтегрази білок Vpr ВІЛ матриксні та нуклеокапсидні білки ВІЛ. Асоційований з актиновими мікрофібрилами клітини. Стабілізується білком Vif ВІЛ. Поетапне роздягання вірусу відбувається під час зворотної транскрипції
 3. The formation of HIV-1 pre-integration complex (PIC) Double-stranded HIV cDNA integrase Matrix protein Vpr host proteins (including HMG proteins ) The complex is transported to the nucleus.
3. The formation of HIV-1 pre-integration complex (PIC) Double-stranded HIV cDNA integrase Matrix protein Vpr host proteins (including HMG proteins ) The complex is transported to the nucleus.
 2. Утворення комплексу передінтеграції: дволанцюгова кДНК ВІЛ інтеграза матриксний білок Vpr в комплексі з клітинним білком (HMGI). Комплекс переміщується до ядра.
2. Утворення комплексу передінтеграції: дволанцюгова кДНК ВІЛ інтеграза матриксний білок Vpr в комплексі з клітинним білком (HMGI). Комплекс переміщується до ядра.
 The pre-integration complex (PIC) is a nucleoprotein complex of viral genetic material and associated viral and host proteins which is capable of inserting a viral genome into a host genome. PIC forms after the Reverse Transcription Complex (RTC) has reverse transcribed the viral RNA code into DNA code. The PIC enters the cellular nucleus through the nuclear pore complex without disrupting the nuclear envelope, thus allowing HIV to replicate in non-dividing cells. Following nuclear entry, the PIC's DNA may be integrated into the host DNA as a "provirus".
The pre-integration complex (PIC) is a nucleoprotein complex of viral genetic material and associated viral and host proteins which is capable of inserting a viral genome into a host genome. PIC forms after the Reverse Transcription Complex (RTC) has reverse transcribed the viral RNA code into DNA code. The PIC enters the cellular nucleus through the nuclear pore complex without disrupting the nuclear envelope, thus allowing HIV to replicate in non-dividing cells. Following nuclear entry, the PIC's DNA may be integrated into the host DNA as a "provirus".
 High-Mobility Group (HMG) – група хромосомних білків, залучених до регуляції ДНК-залежних процесів (транскрипції, реплікації, рекомбінації, репарації). HMGA – містить AT-hook домен HMGA1 HMGA2 HMGB – містить HMG-box домен HMGB1 HMGB2 HMGB3 HMGB4 HMGN – містить нуклеосома-зв’язуючий домен HMGN1 HMGN2 HMGN3 HMGN4 Sex-Determining Region Y Protein TCF Transcription Factors Lymphoid enhancer-binding factor 1 T Cell Transcription Factor 1
High-Mobility Group (HMG) – група хромосомних білків, залучених до регуляції ДНК-залежних процесів (транскрипції, реплікації, рекомбінації, репарації). HMGA – містить AT-hook домен HMGA1 HMGA2 HMGB – містить HMG-box домен HMGB1 HMGB2 HMGB3 HMGB4 HMGN – містить нуклеосома-зв’язуючий домен HMGN1 HMGN2 HMGN3 HMGN4 Sex-Determining Region Y Protein TCF Transcription Factors Lymphoid enhancer-binding factor 1 T Cell Transcription Factor 1



 (a) The initial interactions of HMG-containing proteins (H) with DNA may disrupt the structure of nucleosomes due to their ability to introduce a bend in the backbone of the nucleic acid. (b) As the DNA unravels new protein-binding sites are exposed on the DNA and the recruitment of additional nuclear factors (X and Y) starts. (c) These factors can interact, but they may be too far apart from each other. HMG box protein (H) exerts its bending activity and forces the two factors (X and Y) closer together. Additional domains present in these HMG proteins can have activating functions or can recruit other proteins. (d) The final result is that a new remodeled region of the chromatin is formed where previously only nucleosomes existed..
(a) The initial interactions of HMG-containing proteins (H) with DNA may disrupt the structure of nucleosomes due to their ability to introduce a bend in the backbone of the nucleic acid. (b) As the DNA unravels new protein-binding sites are exposed on the DNA and the recruitment of additional nuclear factors (X and Y) starts. (c) These factors can interact, but they may be too far apart from each other. HMG box protein (H) exerts its bending activity and forces the two factors (X and Y) closer together. Additional domains present in these HMG proteins can have activating functions or can recruit other proteins. (d) The final result is that a new remodeled region of the chromatin is formed where previously only nucleosomes existed..
 Білки, що містять послідовності HMG - HMG motif proteins. HMG-box білки знайдені у багатьох еукаріоттичних організмів.
Білки, що містять послідовності HMG - HMG motif proteins. HMG-box білки знайдені у багатьох еукаріоттичних організмів.
 Функції білків HMG : підтримують структуру хроматину, захищаючи від дії нуклеаз. рекрутують транскрипційні фактори, які зв’язуються з енхансерами. Порушення генів HMG білків пов’язано з виникненням пухлин. Білок HMGB1 має позаклітинну активність – діє як хемокін, приваблює нейтрофіли й мононуклеарні клітин запалення у печінку під час інфекції.
Функції білків HMG : підтримують структуру хроматину, захищаючи від дії нуклеаз. рекрутують транскрипційні фактори, які зв’язуються з енхансерами. Порушення генів HMG білків пов’язано з виникненням пухлин. Білок HMGB1 має позаклітинну активність – діє як хемокін, приваблює нейтрофіли й мононуклеарні клітин запалення у печінку під час інфекції.
 Before integration into host DNA… Vpr (hasn’t NLS) activates cellular nuclear import machinery by connecting the pre-integration complex to protein importin-alpha. Matrix protein (p17), Integrase and Vpr, all having nucleophilic characteristics important for active transport, bring the pre-integration complex into the nucleus. Transport of HIV pre-integration complex (PIC) to the cell nucleus
Before integration into host DNA… Vpr (hasn’t NLS) activates cellular nuclear import machinery by connecting the pre-integration complex to protein importin-alpha. Matrix protein (p17), Integrase and Vpr, all having nucleophilic characteristics important for active transport, bring the pre-integration complex into the nucleus. Transport of HIV pre-integration complex (PIC) to the cell nucleus
 Проникнення в ядро Передінтеграційний комплекс вдвічі більший за розмір ядерної пори. Наслідок: вірусна ДНК повинна зазнати структурних змін. Проникненню в ядро передінтеграційного комплексу сприяють білки: матриксний білок – має сигнал ядерної локалізації, подібний до клітинних імпортинів (компонентів класичного шляху ядерного імпорту); Vpr – здатен ігнорувати імпортинову систему й ініціювати прямий контакт передінтеграційного комплексу з компонентами комплексу ядерної пори. Інтеграза – зв’язується з транспортином 3 (TNPO3)
Проникнення в ядро Передінтеграційний комплекс вдвічі більший за розмір ядерної пори. Наслідок: вірусна ДНК повинна зазнати структурних змін. Проникненню в ядро передінтеграційного комплексу сприяють білки: матриксний білок – має сигнал ядерної локалізації, подібний до клітинних імпортинів (компонентів класичного шляху ядерного імпорту); Vpr – здатен ігнорувати імпортинову систему й ініціювати прямий контакт передінтеграційного комплексу з компонентами комплексу ядерної пори. Інтеграза – зв’язується з транспортином 3 (TNPO3)
 A localization (NLS) is an amino acid sequence that 'tags' a protein for import into the cell nucleus by nuclear transport. Different nuclear localized proteins may share the same NLS. NES (nuclear export signal) targets proteins out of the nucleus. Matrix protein (MA, p17) has NLS – nuclear localization signal that is bound by cellular importins.
A localization (NLS) is an amino acid sequence that 'tags' a protein for import into the cell nucleus by nuclear transport. Different nuclear localized proteins may share the same NLS. NES (nuclear export signal) targets proteins out of the nucleus. Matrix protein (MA, p17) has NLS – nuclear localization signal that is bound by cellular importins.
 Nuclear transport Macromolecules such as RNA and proteins require association with karyopherins called importins to enter the nucleus and exportins to exit. Nuclear import Protein that must be imported to the nucleus from the cytoplasm carry nuclear localization signals (NLS) that are bound by importins. A NLS is a sequence of amino acids that acts as a tag. Nuclear export Proteins, transfer RNA, and assembled ribosomal subunits are exported from the nucleus due to association with exportins, which bind signaling sequences called nuclear export signals (NES).
Nuclear transport Macromolecules such as RNA and proteins require association with karyopherins called importins to enter the nucleus and exportins to exit. Nuclear import Protein that must be imported to the nucleus from the cytoplasm carry nuclear localization signals (NLS) that are bound by importins. A NLS is a sequence of amino acids that acts as a tag. Nuclear export Proteins, transfer RNA, and assembled ribosomal subunits are exported from the nucleus due to association with exportins, which bind signaling sequences called nuclear export signals (NES).
 The ability of both importins and exportins to transport their cargo is regulated by the small Ras related GTPase, Ran. In its GTP bound state, Ran is capable of binding karyopherins (importins and exportins). release cargo upon binding to RanGTP must bind RanGTP to form a ternary complex with their export cargo
The ability of both importins and exportins to transport their cargo is regulated by the small Ras related GTPase, Ran. In its GTP bound state, Ran is capable of binding karyopherins (importins and exportins). release cargo upon binding to RanGTP must bind RanGTP to form a ternary complex with their export cargo
 Nuclear transport importin exportin
Nuclear transport importin exportin
 Nuclear import Importin proteins bind their cargo in the cytoplasm, after which they are able to interact with the nuclear pore complex and pass through its channel. Once inside the nucleus, interaction with Ran-GTP causes aconformational change in the importin that causes it to dissociate from its cargo. The resulting complex of importin and Ran-GTP then translocates to the cytoplasm, where a protein called Ran Binding Protein (RanBP) separates Ran-GTP from importin. Separation allows access to a GTPase activating protein (GAP) that binds Ran-GTP and induces the hydrolysis of GTP to GDP. The Ran-GDP produced from this process now binds the nuclear transport factor NUTF2 which returns it to the nucleoplasm. Now in the nucleus, the Ran-GDP interacts with a guanine nucleotide exchange factor (GEF) which replaces the GDP with GTP, resulting again in Ran-GTP, and beginning the cycle anew.
Nuclear import Importin proteins bind their cargo in the cytoplasm, after which they are able to interact with the nuclear pore complex and pass through its channel. Once inside the nucleus, interaction with Ran-GTP causes aconformational change in the importin that causes it to dissociate from its cargo. The resulting complex of importin and Ran-GTP then translocates to the cytoplasm, where a protein called Ran Binding Protein (RanBP) separates Ran-GTP from importin. Separation allows access to a GTPase activating protein (GAP) that binds Ran-GTP and induces the hydrolysis of GTP to GDP. The Ran-GDP produced from this process now binds the nuclear transport factor NUTF2 which returns it to the nucleoplasm. Now in the nucleus, the Ran-GDP interacts with a guanine nucleotide exchange factor (GEF) which replaces the GDP with GTP, resulting again in Ran-GTP, and beginning the cycle anew.
 Nuclear pore
Nuclear pore
 Nuclear pore
Nuclear pore
 Схема імпорту білків у ядро. 1. Утворення комплексу вантаж-рецептор (імпортин). 2. Заякорювання комплексу на білках ядерної пори і транслокація. 3. Дисоціація комплексу вантаж-імпортин під впливом Ran-ГТФ, вивільнення вантажу, утворення комплексу Ran-ГТФ-імпортин. 4. Реекспорт утвореного комплексу в цитоплазму. 5. Гідроліз ГТФ і дисоціація комплексу.
Схема імпорту білків у ядро. 1. Утворення комплексу вантаж-рецептор (імпортин). 2. Заякорювання комплексу на білках ядерної пори і транслокація. 3. Дисоціація комплексу вантаж-імпортин під впливом Ran-ГТФ, вивільнення вантажу, утворення комплексу Ran-ГТФ-імпортин. 4. Реекспорт утвореного комплексу в цитоплазму. 5. Гідроліз ГТФ і дисоціація комплексу.
 HIV Capsid Uncoating Influences Nuclear Import Several additional cellular factors have been shown to interact with Capsid and to affect HIV-1 infection. Transportin 3 (TNPO3) is a karyopherin that binds arginine/serine (RS)-rich proteins. It is a host factors required for HIV-1 infection. Transportin 3 acts via by binding to HIV integrase and promotes trafficking of pre-integration complex to the nucleus.
HIV Capsid Uncoating Influences Nuclear Import Several additional cellular factors have been shown to interact with Capsid and to affect HIV-1 infection. Transportin 3 (TNPO3) is a karyopherin that binds arginine/serine (RS)-rich proteins. It is a host factors required for HIV-1 infection. Transportin 3 acts via by binding to HIV integrase and promotes trafficking of pre-integration complex to the nucleus.
 HIV-1 uses the nuclear pore to entry into the nucleus by a mechanism that depends on the viral capsid The nuclear pore complex includes: nuclear pore proteins NUP153 NUP358 (or RanBP2) Both of them are required for HIV-1 infection in the same screens that identified Transportin 3 .
HIV-1 uses the nuclear pore to entry into the nucleus by a mechanism that depends on the viral capsid The nuclear pore complex includes: nuclear pore proteins NUP153 NUP358 (or RanBP2) Both of them are required for HIV-1 infection in the same screens that identified Transportin 3 .
 Nuclear pore ! !
Nuclear pore ! !
 The pre-integration complex (PIC) is transported into the nucleus Now the preintegration complex is ready for integration. Provirus preferentially integrates at sites of highly bent DNA, such as nucleosomes.
The pre-integration complex (PIC) is transported into the nucleus Now the preintegration complex is ready for integration. Provirus preferentially integrates at sites of highly bent DNA, such as nucleosomes.
 HIV DNA integration HIV Integrase catalyses the covalent attachment of HIV DNA copy to host cell DNA. Host cell proteins that necessary for HIV DNA integration: HMGI proteins After the viral DNA is synthesized by reverse transcription in the cytoplasm, it stably associates with IN and other proteins as a high-molecular-weight nucleoprotein complex that is later transported to the nucleus for subsequent integration.
HIV DNA integration HIV Integrase catalyses the covalent attachment of HIV DNA copy to host cell DNA. Host cell proteins that necessary for HIV DNA integration: HMGI proteins After the viral DNA is synthesized by reverse transcription in the cytoplasm, it stably associates with IN and other proteins as a high-molecular-weight nucleoprotein complex that is later transported to the nucleus for subsequent integration.
 HIV integration is strongly favored in active transcription units, which may promote efficient viral gene expression after integration. The HIV integration machinery must also interact with many additional host factors during infection, including: nuclear trafficking and pore proteins during nuclear entry, histones during initial target capture, DNA repair proteins during completion of the DNA joining steps. HIV DNA Integration Robert Craigie, Frederic D. Bushman Cold Spring Harb Perspect Med. 2012 July; 2(7):
HIV integration is strongly favored in active transcription units, which may promote efficient viral gene expression after integration. The HIV integration machinery must also interact with many additional host factors during infection, including: nuclear trafficking and pore proteins during nuclear entry, histones during initial target capture, DNA repair proteins during completion of the DNA joining steps. HIV DNA Integration Robert Craigie, Frederic D. Bushman Cold Spring Harb Perspect Med. 2012 July; 2(7):
 How does Integrase do it? It recognizes the LTRs at the 5’ and 3’ ends of the newly synthesized viral DNA duplex and cleaves 2-3 bases from the 3’ ends. Trans-esterification reaction takes place joining proviral and cellular DNA ends 4-6bp gaps of mismatched ends are trimmed, filled and ligated.
How does Integrase do it? It recognizes the LTRs at the 5’ and 3’ ends of the newly synthesized viral DNA duplex and cleaves 2-3 bases from the 3’ ends. Trans-esterification reaction takes place joining proviral and cellular DNA ends 4-6bp gaps of mismatched ends are trimmed, filled and ligated.
 DNA breaking and joining reactions mediating DNA integration А B C D E
DNA breaking and joining reactions mediating DNA integration А B C D E
 HIV DNA integration (А) The linear blunt-ended viral DNA (green and yellow) and target DNA (blue and red). (B) 30 end processing. Two nucleotides are inmost cases removed fromeach 30 end of the viralDNA. (C) The 30 ends generated by 30 processing attack a pair of phosphodiester bonds in the target DNA. The sites of attack on the two target DNA strands are separated by five nucleotides in the case of HIV-1. The 30 ends of the viral DNA are joined to the 50 ends of the target DNA at the site of integration. The 50 ends of the viral DNA are not joined to target DNA in the intermediate. (D) Completion of provirus formation requires removal of the two unpaired bases at the 50 ends of the viral DNA, filling in the single-strand gaps between viral and target DNA and ligation of the 50 ends of the viral DNA to target DNA. IN catalyzes the 30 processing and DNA-strand transfer steps to form the integration intermediate. Subsequent steps are thought to be catalyzed by cellular enzymes. (E) The integrated provirus.
HIV DNA integration (А) The linear blunt-ended viral DNA (green and yellow) and target DNA (blue and red). (B) 30 end processing. Two nucleotides are inmost cases removed fromeach 30 end of the viralDNA. (C) The 30 ends generated by 30 processing attack a pair of phosphodiester bonds in the target DNA. The sites of attack on the two target DNA strands are separated by five nucleotides in the case of HIV-1. The 30 ends of the viral DNA are joined to the 50 ends of the target DNA at the site of integration. The 50 ends of the viral DNA are not joined to target DNA in the intermediate. (D) Completion of provirus formation requires removal of the two unpaired bases at the 50 ends of the viral DNA, filling in the single-strand gaps between viral and target DNA and ligation of the 50 ends of the viral DNA to target DNA. IN catalyzes the 30 processing and DNA-strand transfer steps to form the integration intermediate. Subsequent steps are thought to be catalyzed by cellular enzymes. (E) The integrated provirus.
 PICs may interact with specific nuclear pore proteins during nuclear transit transportin 3 (product of the TNPO3 gene) Nup358 (product of the RANBP2 gene) These nuclear pore proteins are also important not only for nuclear localization but also for efficient integration after nuclear entry. It suggests possible coupling of nuclear translocation and integration.
PICs may interact with specific nuclear pore proteins during nuclear transit transportin 3 (product of the TNPO3 gene) Nup358 (product of the RANBP2 gene) These nuclear pore proteins are also important not only for nuclear localization but also for efficient integration after nuclear entry. It suggests possible coupling of nuclear translocation and integration.
 The significance of HIV DNA integration Once integrated, the proviral DNA is replicated along with cellular DNA during cycles of cell division, as with any cellular gene. The provirus serves as the template for transcription of viral RNAs. Some viral RNAs are translated to yield the viral proteins, whereas a portion of the full-length viral RNA is recruited to serve as genomic RNA in progeny virions.
The significance of HIV DNA integration Once integrated, the proviral DNA is replicated along with cellular DNA during cycles of cell division, as with any cellular gene. The provirus serves as the template for transcription of viral RNAs. Some viral RNAs are translated to yield the viral proteins, whereas a portion of the full-length viral RNA is recruited to serve as genomic RNA in progeny virions.
 Integration Target Site Selection integration reaction was not highly sequence specific, although weakly conserved sequences at target sites were shown. Host cell sequences at the point of integration differed among proviral isolates.
Integration Target Site Selection integration reaction was not highly sequence specific, although weakly conserved sequences at target sites were shown. Host cell sequences at the point of integration differed among proviral isolates.
 Integration Target Site Selection Mapping of favored sites indicated that: sharp DNA bends in the nucleosome structure were particularly favored targets. DNA distortion can promote integration.
Integration Target Site Selection Mapping of favored sites indicated that: sharp DNA bends in the nucleosome structure were particularly favored targets. DNA distortion can promote integration.
 Integration Target Site Selection 524 sites of HIV integration were mapped after acute infection of the T-cell line. HIV favored integration within transcription units.
Integration Target Site Selection 524 sites of HIV integration were mapped after acute infection of the T-cell line. HIV favored integration within transcription units.
 HIV integration site distributions on the human chromosomes (Wang et al. 2007) The human chromosomes are numbered at the sides of the diagram. HIV integration sites (20,000 total) are shown in green, gene density is shown in blue (measured as the count of RefSeq genes in a 500-kb interval), and the G/ C content is shown in orange (measured in 500-kb intervals). The gray coloring indicates regions of centromeric repeats that have not been sequenced.
HIV integration site distributions on the human chromosomes (Wang et al. 2007) The human chromosomes are numbered at the sides of the diagram. HIV integration sites (20,000 total) are shown in green, gene density is shown in blue (measured as the count of RefSeq genes in a 500-kb interval), and the G/ C content is shown in orange (measured in 500-kb intervals). The gray coloring indicates regions of centromeric repeats that have not been sequenced.
 HIV takes advantage of host cell factors to allow efficient integration and optimize integration target site selection host cell protein LEDGF/p75 - (product of the PSIP1 gene) - boosts the efficiency of integration and mediates targeting to active transcription units. LEDGF/p75 is a transcriptional mediator protein that promoted activator-dependent transcription in vitro. LEDGF/p75 tightly binds with HIV integrase (could tether IN to chromatin).
HIV takes advantage of host cell factors to allow efficient integration and optimize integration target site selection host cell protein LEDGF/p75 - (product of the PSIP1 gene) - boosts the efficiency of integration and mediates targeting to active transcription units. LEDGF/p75 is a transcriptional mediator protein that promoted activator-dependent transcription in vitro. LEDGF/p75 tightly binds with HIV integrase (could tether IN to chromatin).
 HIV takes advantage of host cell factors to allow efficient integration and optimize integration target site selection Several cellular chromatin proteins have also been suggested to influence integration including: Ini-1, EED, SUV39H1, HP1g.
HIV takes advantage of host cell factors to allow efficient integration and optimize integration target site selection Several cellular chromatin proteins have also been suggested to influence integration including: Ini-1, EED, SUV39H1, HP1g.
 Once permanently integrated… Early phase commences: only short spliced mRNA species encoding Tat, Rev and Nef are synthesized and transported out the nucleus and translated The amount of Rev (NLS) controls the level of singly and multiply spliced messages in the cytoplasm Differential distribution of viral RNAs is due to cis-acting nuclear retention sequences (CRS) in the gag, pol and env coding regions Transport to the cytoplasm is also dependent on Rev, which binds to the RRE of nascent unspliced mRNAs and recruits the cellular nuclear shuttling protein exportin-1. The complex is then transported through the nuclear pore to the cytosol where GTP is hydrolyzed to GDP, the complex dissociates and Rev goes back
Once permanently integrated… Early phase commences: only short spliced mRNA species encoding Tat, Rev and Nef are synthesized and transported out the nucleus and translated The amount of Rev (NLS) controls the level of singly and multiply spliced messages in the cytoplasm Differential distribution of viral RNAs is due to cis-acting nuclear retention sequences (CRS) in the gag, pol and env coding regions Transport to the cytoplasm is also dependent on Rev, which binds to the RRE of nascent unspliced mRNAs and recruits the cellular nuclear shuttling protein exportin-1. The complex is then transported through the nuclear pore to the cytosol where GTP is hydrolyzed to GDP, the complex dissociates and Rev goes back

 HIV-1 Assembly
HIV-1 Assembly
 Формування віріонів ВІЛ До мембрани клітини транспортуються: попередники білків ВІЛ: Gag (матрикс, серцевина, нуклеокапсид) Gag-Pol (матрикс, серцевина, нуклеокапсид + ферменти) рецептори (gp120 і gp41) – транспортуються незалежно від Gag геномна РНК ВІЛ та tRNALys (праймер для ревертази) Матриксний білок ВІЛ (MA) заякорюється у клітинній мембрані за допомогою залишку міристинової кислоти у складі зв’язаного з ним інозитол-4,5-дифосфату (PI(4,5)P2). У клітинну мембрану вбудовуються білки оболонки gp41 і gp120 незалежно від серцевинних (корових, від англ. core — серцевина) білків. У процесі агрегації вірусних білків на клітинній мембрані вона випинається.
Формування віріонів ВІЛ До мембрани клітини транспортуються: попередники білків ВІЛ: Gag (матрикс, серцевина, нуклеокапсид) Gag-Pol (матрикс, серцевина, нуклеокапсид + ферменти) рецептори (gp120 і gp41) – транспортуються незалежно від Gag геномна РНК ВІЛ та tRNALys (праймер для ревертази) Матриксний білок ВІЛ (MA) заякорюється у клітинній мембрані за допомогою залишку міристинової кислоти у складі зв’язаного з ним інозитол-4,5-дифосфату (PI(4,5)P2). У клітинну мембрану вбудовуються білки оболонки gp41 і gp120 незалежно від серцевинних (корових, від англ. core — серцевина) білків. У процесі агрегації вірусних білків на клітинній мембрані вона випинається.
 Формування віріонів ВІЛ У щойно сформований віріон надходять два ланцюги вірусної РНК і tRNALys . Віріон відбруньковується від клітини хазяїна 3. Від довгого білка-попередника відрізається протеаза, що завершує формування віріону: вона відщеплює від довгого попередника ще молекули протеази, зворотну транскриптазу та ендорибонуклеазу. Протеаза розрізає кожний з попередників на чотири частини. Одна частина (р17) залишається зв'язаною з клітинною мембраною, що оточує віріон, а три (р24, р7 і р9) утворюють кулеподібну серцевину.
Формування віріонів ВІЛ У щойно сформований віріон надходять два ланцюги вірусної РНК і tRNALys . Віріон відбруньковується від клітини хазяїна 3. Від довгого білка-попередника відрізається протеаза, що завершує формування віріону: вона відщеплює від довгого попередника ще молекули протеази, зворотну транскриптазу та ендорибонуклеазу. Протеаза розрізає кожний з попередників на чотири частини. Одна частина (р17) залишається зв'язаною з клітинною мембраною, що оточує віріон, а три (р24, р7 і р9) утворюють кулеподібну серцевину.
 HIV-1 Assembly The assembling virion packages all of the components required for infectivity: two copies of the positive sense genomic viral RNA, cellular tRNALys,3 molecules to prime cDNA synthesis, the viral envelope (Env) protein, the Gag polyprotein, the three viral enzymes: protease (PR), reverse transcriptase (RT), and integrase (IN). The viral enzymes are packaged as domains within the Gag-Pro-Pol polyprotein, which is generated when translating ribosomes shift into the −1 reading frame at a site near the 3′ end of the gag open reading frame, and then go on to translate the pol gene.
HIV-1 Assembly The assembling virion packages all of the components required for infectivity: two copies of the positive sense genomic viral RNA, cellular tRNALys,3 molecules to prime cDNA synthesis, the viral envelope (Env) protein, the Gag polyprotein, the three viral enzymes: protease (PR), reverse transcriptase (RT), and integrase (IN). The viral enzymes are packaged as domains within the Gag-Pro-Pol polyprotein, which is generated when translating ribosomes shift into the −1 reading frame at a site near the 3′ end of the gag open reading frame, and then go on to translate the pol gene.
 Many cellular proteins has been identified in HIV-1 particles Their importance for virus assembly, budding, and/or infectivity is currently not well established in most cases. plasma membrane proteins like ICAM-1 (which may mediate virus adherence to cells) HLA-II or MHC (which may modulate immune responses) cytoplasmic proteins which may be incorporated via direct or indirect Gag interactions (e.g., actin and actin-binding proteins, cyclophilin A, ubiquitin, lysyl-tRNA-synthetase, and many RNA binding proteins)
Many cellular proteins has been identified in HIV-1 particles Their importance for virus assembly, budding, and/or infectivity is currently not well established in most cases. plasma membrane proteins like ICAM-1 (which may mediate virus adherence to cells) HLA-II or MHC (which may modulate immune responses) cytoplasmic proteins which may be incorporated via direct or indirect Gag interactions (e.g., actin and actin-binding proteins, cyclophilin A, ubiquitin, lysyl-tRNA-synthetase, and many RNA binding proteins)
 HIV-1 virion assembly occurs at the plasma membrane, within specialized membrane microdomains The HIV-1 Gag (and Gag-Pro-Pol) polyprotein itself mediates all of the essential events in virion assembly, including: binding the plasma membrane, making the protein–protein interactions necessary to create spherical particles, concentrating the viral Env protein, and packaging the genomic RNA via direct interactions with the RNA packaging sequence (termed Ψ).
HIV-1 virion assembly occurs at the plasma membrane, within specialized membrane microdomains The HIV-1 Gag (and Gag-Pro-Pol) polyprotein itself mediates all of the essential events in virion assembly, including: binding the plasma membrane, making the protein–protein interactions necessary to create spherical particles, concentrating the viral Env protein, and packaging the genomic RNA via direct interactions with the RNA packaging sequence (termed Ψ).
 HIV-1 virion assembly occur simultaneously at the plasma membrane The conformational change(s) within Gag couples: membrane binding, virion assembly, RNA packaging. Although Gag itself can bind membranes and assemble into spherical particles, the budding event that releases the virion from the plasma membrane is mediated by the host ESCRT (endosomal sorting complexes required for transport) machinery.
HIV-1 virion assembly occur simultaneously at the plasma membrane The conformational change(s) within Gag couples: membrane binding, virion assembly, RNA packaging. Although Gag itself can bind membranes and assemble into spherical particles, the budding event that releases the virion from the plasma membrane is mediated by the host ESCRT (endosomal sorting complexes required for transport) machinery.
 HIV-1 assembly, budding, and maturation. (A) Schematic illustration showing the different stages of HIV-1 assembly, budding, and maturation. (B) Domain structure of the HIV-1 Gag protein; arrows denote the five sites that are cleaved by the viral PR during maturation. (C) Structural model of the HIV-1 Gag protein, created by combining structures of the isolated MA-CANTD (2GOL), CACTD (1BAJ), and NC (1MFS) proteins, with a helical model for SP1. (D) Schematic model showing the organization of the immature HIV-1 virion. (E) Schematic model showing the organization of the mature HIV-1 virion. (F) Central section from a cryo-EM tomographic reconstruction of an immature HIV-1 virion. (G) Central section from a tomographic reconstruction of a mature HIV-1 virion. (H) Structure of HIV-1 protease (PR, 3D3T). The two subunits in the dimer are shown in different shades of purple, the “flap” and dimerization interfaces are labeled, positions of the active site Asp25 residues are shown in red, and a bound peptide corresponding to the SP2-p6 cleavage site is shown as a stick model, with oxygen atoms in red and nitrogen atoms in blue.
HIV-1 assembly, budding, and maturation. (A) Schematic illustration showing the different stages of HIV-1 assembly, budding, and maturation. (B) Domain structure of the HIV-1 Gag protein; arrows denote the five sites that are cleaved by the viral PR during maturation. (C) Structural model of the HIV-1 Gag protein, created by combining structures of the isolated MA-CANTD (2GOL), CACTD (1BAJ), and NC (1MFS) proteins, with a helical model for SP1. (D) Schematic model showing the organization of the immature HIV-1 virion. (E) Schematic model showing the organization of the mature HIV-1 virion. (F) Central section from a cryo-EM tomographic reconstruction of an immature HIV-1 virion. (G) Central section from a tomographic reconstruction of a mature HIV-1 virion. (H) Structure of HIV-1 protease (PR, 3D3T). The two subunits in the dimer are shown in different shades of purple, the “flap” and dimerization interfaces are labeled, positions of the active site Asp25 residues are shown in red, and a bound peptide corresponding to the SP2-p6 cleavage site is shown as a stick model, with oxygen atoms in red and nitrogen atoms in blue.

 Schematic illustration showing the different stages of HIV-1 assembly, budding, and maturation.
Schematic illustration showing the different stages of HIV-1 assembly, budding, and maturation.
 HIV-1 virion assembly occur simultaneously at the plasma membrane The conformational change(s) within Gag couples: membrane binding, virion assembly, RNA packaging. Although Gag itself can bind membranes and assemble into spherical particles, the budding event that releases the virion from the plasma membrane is mediated by the host ESCRT (endosomal sorting complexes required for transport) machinery.
HIV-1 virion assembly occur simultaneously at the plasma membrane The conformational change(s) within Gag couples: membrane binding, virion assembly, RNA packaging. Although Gag itself can bind membranes and assemble into spherical particles, the budding event that releases the virion from the plasma membrane is mediated by the host ESCRT (endosomal sorting complexes required for transport) machinery.
 Domain structure of the HIV-1 Gag protein; arrows denote the five sites that are cleaved by the viral PR during maturation Folded domains within Gag are separated by flexible linker regions which contain the PR cleavage sites
Domain structure of the HIV-1 Gag protein; arrows denote the five sites that are cleaved by the viral PR during maturation Folded domains within Gag are separated by flexible linker regions which contain the PR cleavage sites
 Gag domains The amino-terminal domain – MA, functions to bind the plasma membrane and to recruit the viral Env protein. The central domain - CA, it mediates the protein–protein interactions required for immature virion assembly and then creates the conical shell (capsid) of the mature viral core. The basic domain – NC, contains two copies of the retroviral zinc finger motif. NC captures the viral genome during assembly, NC also functions as a nucleic acid “chaperone” during tRNALys,3 primer annealing and reverse transcription.
Gag domains The amino-terminal domain – MA, functions to bind the plasma membrane and to recruit the viral Env protein. The central domain - CA, it mediates the protein–protein interactions required for immature virion assembly and then creates the conical shell (capsid) of the mature viral core. The basic domain – NC, contains two copies of the retroviral zinc finger motif. NC captures the viral genome during assembly, NC also functions as a nucleic acid “chaperone” during tRNALys,3 primer annealing and reverse transcription.
 Gag domains (continuation) the carboxy-terminal Gag p6 region contains binding sites for several other proteins, including the accessory viral protein Vpr. two spacer peptides, SP1 and SP2, which help to regulate the conformational changes.
Gag domains (continuation) the carboxy-terminal Gag p6 region contains binding sites for several other proteins, including the accessory viral protein Vpr. two spacer peptides, SP1 and SP2, which help to regulate the conformational changes.
 Structural model of the HIV-1 Gag protein, created by combining structures of the isolated MA-CANTD (2GOL), CACTD (1BAJ), and NC (1MFS) proteins, with a helical model for SP1.
Structural model of the HIV-1 Gag protein, created by combining structures of the isolated MA-CANTD (2GOL), CACTD (1BAJ), and NC (1MFS) proteins, with a helical model for SP1.
 Env Env is an integral membrane protein. Env is modified in Endoplasmic Reticulum: it is inserted cotranslationally* into ER membranes then travels through the cellular secretory pathway where it is glycosylated, assembled into trimeric complexes, processed into the trans-membrane (TM; gp41) and surface (SU; gp120) subunits by the cellular protease furin, delivered to the plasma membrane via vesicular transport.
Env Env is an integral membrane protein. Env is modified in Endoplasmic Reticulum: it is inserted cotranslationally* into ER membranes then travels through the cellular secretory pathway where it is glycosylated, assembled into trimeric complexes, processed into the trans-membrane (TM; gp41) and surface (SU; gp120) subunits by the cellular protease furin, delivered to the plasma membrane via vesicular transport.
 * - Co-translational translocation is the action of a protein being fed into the ER as it is synthesized by theribosome. In this scenario the protein begins to be translated normally. As the N-terminus (the first end of the protein to be synthesized) leaves the ribosome, a short sequence (normally 5-10 hydrophobic amino acids) of the nascent protein's amino acids is recognized by a protein complex called SRP, or signal recognition particle. SRP binds to this sequence of amino acids and then subsequently binds to a protein complex called the SRP receptor embedded in the ER membrane. Both the SRP complex and SRP receptor complex hydrolyze a molecule of GTP into GDP. Once this occurs the growing protein chain is moved to a membrane channel called the translocon. It is through this pore in the ER membrane that the protein passes through as it is synthesized. The result of all this is a new protein residing within the membrane of the endoplasmic reticulum.
* - Co-translational translocation is the action of a protein being fed into the ER as it is synthesized by theribosome. In this scenario the protein begins to be translated normally. As the N-terminus (the first end of the protein to be synthesized) leaves the ribosome, a short sequence (normally 5-10 hydrophobic amino acids) of the nascent protein's amino acids is recognized by a protein complex called SRP, or signal recognition particle. SRP binds to this sequence of amino acids and then subsequently binds to a protein complex called the SRP receptor embedded in the ER membrane. Both the SRP complex and SRP receptor complex hydrolyze a molecule of GTP into GDP. Once this occurs the growing protein chain is moved to a membrane channel called the translocon. It is through this pore in the ER membrane that the protein passes through as it is synthesized. The result of all this is a new protein residing within the membrane of the endoplasmic reticulum.
 HIV virion assembly The Gag polyprotein initially assembles into spherical immature particles. The membrane-bound Gag molecules project radially toward the virion interior (Fig. D,F). As the immature virion buds, HIV protease is activated and cleaves Gag into its constituent MA, CA, NC, and p6 proteins, thereby also releasing the SP1 and SP2 peptides. Proteolysis is required for conversion of the immature virion into its mature infectious form.
HIV virion assembly The Gag polyprotein initially assembles into spherical immature particles. The membrane-bound Gag molecules project radially toward the virion interior (Fig. D,F). As the immature virion buds, HIV protease is activated and cleaves Gag into its constituent MA, CA, NC, and p6 proteins, thereby also releasing the SP1 and SP2 peptides. Proteolysis is required for conversion of the immature virion into its mature infectious form.
 (D) Schematic model showing the organization of the immature HIV-1 virion. (E) Schematic model showing the organization of the mature HIV-1 virion.
(D) Schematic model showing the organization of the immature HIV-1 virion. (E) Schematic model showing the organization of the mature HIV-1 virion.
 . (F) Central section from a cryo-EM tomographic reconstruction of an immature HIV-1 virion. (G) Central section from a tomographic reconstruction of a mature HIV-1 virion.
. (F) Central section from a cryo-EM tomographic reconstruction of an immature HIV-1 virion. (G) Central section from a tomographic reconstruction of a mature HIV-1 virion.
 HIV protease Like other retroviral proteases, HIV-1 PR is a dimeric aspartic protease. PR recognizes specific sites within Gag and cleaves them in an ordered fashion. Gag proteolysis triggers major changes which include: condensing and stabilizing the dimeric RNA genome, assembling the conical capsid about the genomic RNA–NC-enzyme complex, preparing the virion to enter, replicate, and uncoat in the next host cell. The matured HIV virion can enter and replicate in a new host cell.
HIV protease Like other retroviral proteases, HIV-1 PR is a dimeric aspartic protease. PR recognizes specific sites within Gag and cleaves them in an ordered fashion. Gag proteolysis triggers major changes which include: condensing and stabilizing the dimeric RNA genome, assembling the conical capsid about the genomic RNA–NC-enzyme complex, preparing the virion to enter, replicate, and uncoat in the next host cell. The matured HIV virion can enter and replicate in a new host cell.
 Structure of HIV-1 protease (PR, 3D3T). The two subunits in the dimer are shown in different shades of purple, the “flap” and dimerization interfaces are labeled, positions of the active site Asp25 residues are shown in red, and a bound peptide corresponding to the SP2-p6 cleavage site is shown as a stick model, with oxygen atoms in red and nitrogen atoms in blue.
Structure of HIV-1 protease (PR, 3D3T). The two subunits in the dimer are shown in different shades of purple, the “flap” and dimerization interfaces are labeled, positions of the active site Asp25 residues are shown in red, and a bound peptide corresponding to the SP2-p6 cleavage site is shown as a stick model, with oxygen atoms in red and nitrogen atoms in blue.
 Збірка нових віріонів ВІЛ відбувається на клітинній мембрані. Вірусні частки утворюються з трьох різних білків-попередників: попередник білків оболонки gp120/gp41 (комплекс із 2-3 субодиниць по 2 зв'язаних глікопротеїни); gp120/gp41 короткий білок-попередник; матрикс і серцевина довгий білок-попередник матрикс, серцевина + ферменти
Збірка нових віріонів ВІЛ відбувається на клітинній мембрані. Вірусні частки утворюються з трьох різних білків-попередників: попередник білків оболонки gp120/gp41 (комплекс із 2-3 субодиниць по 2 зв'язаних глікопротеїни); gp120/gp41 короткий білок-попередник; матрикс і серцевина довгий білок-попередник матрикс, серцевина + ферменти
 СООН Формування білків серцевини і матриксу ВІЛ короткий попередник довгий попередник (короткий попередник + ферменти)
СООН Формування білків серцевини і матриксу ВІЛ короткий попередник довгий попередник (короткий попередник + ферменти)
 Gag - makes up 50% of the entire virion mass. the viral membrane lipids - account for 30% of virion mass. the main constituents of HIV-1 other viral and cellular proteins – together contribute an additional 20%. The genomic RNA and other small RNAs – amount to 2.5% of virion mass. HIV virion composition
Gag - makes up 50% of the entire virion mass. the viral membrane lipids - account for 30% of virion mass. the main constituents of HIV-1 other viral and cellular proteins – together contribute an additional 20%. The genomic RNA and other small RNAs – amount to 2.5% of virion mass. HIV virion composition
 Protein Trafficking and Virion Incorporation All virion components need to traffic from their point of synthesis to sites of assembly on the plasma membrane. Gag and Gag-Pro-Pol polyproteins (which are produced at a ratio of 20:1) are made at the proper stoichiometry. Gag then binds other virion components through direct protein–protein and protein–RNA interactions, which allows the virus to assemble all of its components using a single targeting signal.
Protein Trafficking and Virion Incorporation All virion components need to traffic from their point of synthesis to sites of assembly on the plasma membrane. Gag and Gag-Pro-Pol polyproteins (which are produced at a ratio of 20:1) are made at the proper stoichiometry. Gag then binds other virion components through direct protein–protein and protein–RNA interactions, which allows the virus to assemble all of its components using a single targeting signal.
 The HIV Gag and Gag-Pro-Pol polyproteins traffic from their sites of synthesis in the cytoplasm to the plasma membrane and then sort into detergent-resistant membrane microdomains. Virus production is cholesterol and sphingolipid dependent, and the virus is enriched in “raft”-associated proteins and lipids. Gag interacts with the cellular motor protein KIF4 and with various components of intracellular vesicle trafficking pathways.
The HIV Gag and Gag-Pro-Pol polyproteins traffic from their sites of synthesis in the cytoplasm to the plasma membrane and then sort into detergent-resistant membrane microdomains. Virus production is cholesterol and sphingolipid dependent, and the virus is enriched in “raft”-associated proteins and lipids. Gag interacts with the cellular motor protein KIF4 and with various components of intracellular vesicle trafficking pathways.
 The Gag recognition of the plasma membrane Gag molecules thus arrive at the plasma membrane as small oligomers, probably monomers or dimers. They polymerize onto nucleation sites composed of Gag–RNA complexes. Gag membrane targeting requires myristoylation MA domain of Gag binds to PI(4,5)P2*. * - plasma membrane-specific lipid phosphatidyl inositol (4,5) bisphosphate The aminoterminal myristoyl group of MA is exposed Gag stably anchores on the inner leaflet of plama membrane.
The Gag recognition of the plasma membrane Gag molecules thus arrive at the plasma membrane as small oligomers, probably monomers or dimers. They polymerize onto nucleation sites composed of Gag–RNA complexes. Gag membrane targeting requires myristoylation MA domain of Gag binds to PI(4,5)P2*. * - plasma membrane-specific lipid phosphatidyl inositol (4,5) bisphosphate The aminoterminal myristoyl group of MA is exposed Gag stably anchores on the inner leaflet of plama membrane.
 Myristoyl switch model for MAGag recognition of the plasma membrane (Saad et al. 2006). MAGag (yellow) proteins are shown with the aliphatic myristoyl group (brown) sequestered within the soluble protein (left, 1UPH), and with the myristoyl group extruded
Myristoyl switch model for MAGag recognition of the plasma membrane (Saad et al. 2006). MAGag (yellow) proteins are shown with the aliphatic myristoyl group (brown) sequestered within the soluble protein (left, 1UPH), and with the myristoyl group extruded
 Lipid Composition of viral membrane There are differences between the lipid compositions of the producer cell and HIV-1 membranes. Virion is enriched with: sphingomyelin (SM), phosphatidyl serine (PS), phosphatidyl ethanol (PE), and cholesterol, The membrane fluidity is decreased.
Lipid Composition of viral membrane There are differences between the lipid compositions of the producer cell and HIV-1 membranes. Virion is enriched with: sphingomyelin (SM), phosphatidyl serine (PS), phosphatidyl ethanol (PE), and cholesterol, The membrane fluidity is decreased.
 Lipid Composition of viral membrane strong virion enrichment of the “raft lipids”: SM, cholesterol, plasmalogen-PE, PI(4,5)P2* with an increase in saturated fatty acids Compared with the producer cell membrane The HIV-1 membrane is also enriched in PI(4,5)P2, consistent with the idea that this phosphatidylinositide plays an important role in targeting Gag to the membrane.
Lipid Composition of viral membrane strong virion enrichment of the “raft lipids”: SM, cholesterol, plasmalogen-PE, PI(4,5)P2* with an increase in saturated fatty acids Compared with the producer cell membrane The HIV-1 membrane is also enriched in PI(4,5)P2, consistent with the idea that this phosphatidylinositide plays an important role in targeting Gag to the membrane.
 RNA Trafficking and Incorporation HIV selectively incorporates two copies of the capped and polyadenylated full-length RNA genome into the virion. The two RNA strands are noncovalently dimerized in their 5-UTR. RNA dimerization initiates through formation of a “kissing-loop” structure mediated by Watson–Crick base pairing of the self-complementary sequence within the loop of the dimer initiation site (DIS).
RNA Trafficking and Incorporation HIV selectively incorporates two copies of the capped and polyadenylated full-length RNA genome into the virion. The two RNA strands are noncovalently dimerized in their 5-UTR. RNA dimerization initiates through formation of a “kissing-loop” structure mediated by Watson–Crick base pairing of the self-complementary sequence within the loop of the dimer initiation site (DIS).
 RNA Trafficking and Incorporation RNA dimerization is required for RNA packaging and viral infectivity. It involves cis-acting sequences within the 5-UTR of the viral genome, which are recognized by the NC(Gag) domain.
RNA Trafficking and Incorporation RNA dimerization is required for RNA packaging and viral infectivity. It involves cis-acting sequences within the 5-UTR of the viral genome, which are recognized by the NC(Gag) domain.
 5 Untranslated region (UTR) of the HIV-1 RNA genome and its interactions with the viral NC protein. dimer initiation site (DIS)
5 Untranslated region (UTR) of the HIV-1 RNA genome and its interactions with the viral NC protein. dimer initiation site (DIS)
 Assembly and Budding Assembly of the virus particle takes place on the inner surface of the cell membrane, in macrophages also in vacuoles Gag precursor Pr55gag, major virion component, is cleaved into 4 major proteins: matrix, capsid, nucleocapsid, and p6gag in the viral particle and associates into virion spontaneously Gag is targeted to the cell membrane after being post-translationally modified, (addition of myristyl group) Pr55gag directs incorporation of Gag-Pol precursor, Pr160gag, the envelope protein, and Vpr
Assembly and Budding Assembly of the virus particle takes place on the inner surface of the cell membrane, in macrophages also in vacuoles Gag precursor Pr55gag, major virion component, is cleaved into 4 major proteins: matrix, capsid, nucleocapsid, and p6gag in the viral particle and associates into virion spontaneously Gag is targeted to the cell membrane after being post-translationally modified, (addition of myristyl group) Pr55gag directs incorporation of Gag-Pol precursor, Pr160gag, the envelope protein, and Vpr

 HIV Particle Maturation by HIV protease Immature particle and mature particle
HIV Particle Maturation by HIV protease Immature particle and mature particle
 More on assembly and budding Pr55gag of HIV-1, not HIV-2 also binds cyclophilin A, which is important because cyclosporin disrupts this association inhibiting HIV Env precursor, gp160 synthesized in the ER from the spliced env, is cleaved by a cellular protease into gp120 and gp41 Vpr and Nef are also found in the virion The nucleocapsid domain of Gag interacts with the encapsidation sequence (psi) of the genomic RNA via its Zn finger domains At this point the lipid bilayer surrounds the core, budding occurs
More on assembly and budding Pr55gag of HIV-1, not HIV-2 also binds cyclophilin A, which is important because cyclosporin disrupts this association inhibiting HIV Env precursor, gp160 synthesized in the ER from the spliced env, is cleaved by a cellular protease into gp120 and gp41 Vpr and Nef are also found in the virion The nucleocapsid domain of Gag interacts with the encapsidation sequence (psi) of the genomic RNA via its Zn finger domains At this point the lipid bilayer surrounds the core, budding occurs
 Maturation 1200-2000 copies of Gag bud to from an immature particle with 2 copies of unspliced viral genome Proteolytic processing mediated by the viral protease separates the domains of the different polyproteins and sets the conditions for RT Structural proteins rearrange 7-100 copies of Vif required for production of infectious virions in some cell lines are packaged although not known if essential ONLY THE MATURE PARTICLES ARE COMPETENT FOR INTECTION
Maturation 1200-2000 copies of Gag bud to from an immature particle with 2 copies of unspliced viral genome Proteolytic processing mediated by the viral protease separates the domains of the different polyproteins and sets the conditions for RT Structural proteins rearrange 7-100 copies of Vif required for production of infectious virions in some cell lines are packaged although not known if essential ONLY THE MATURE PARTICLES ARE COMPETENT FOR INTECTION
 http://upload.wikimedia.org/wikipedia/commons/0/00/HIV_Mature_and_Immature.PNG
http://upload.wikimedia.org/wikipedia/commons/0/00/HIV_Mature_and_Immature.PNG
 Віріони ВІЛ відбруньковуються від лімфоцита
Віріони ВІЛ відбруньковуються від лімфоцита
 Т-клітина в культурі виділяє новоутворені вірусні частки. Багато віріонів опинилися у вакуолі. Вірусні гени знаходяться в ядрі зараженої клітини. Кульмінація життєвого циклу ВІЛ – це утворення нового покоління вірусу мікрофотографія (х 25 000)
Т-клітина в культурі виділяє новоутворені вірусні частки. Багато віріонів опинилися у вакуолі. Вірусні гени знаходяться в ядрі зараженої клітини. Кульмінація життєвого циклу ВІЛ – це утворення нового покоління вірусу мікрофотографія (х 25 000)
 Віріон ВІЛ відбруньковується від поверхні клітини. Вірусна частка знаходиться в стадії збирання. Видно білки оболонки що вкривають ділянку клітинної мембрани, яка стане оболонкою зрілого віріону. мікрофотографія (х 120 000)
Віріон ВІЛ відбруньковується від поверхні клітини. Вірусна частка знаходиться в стадії збирання. Видно білки оболонки що вкривають ділянку клітинної мембрани, яка стане оболонкою зрілого віріону. мікрофотографія (х 120 000)
 Нові віріони ВІЛ виходять з тонкого виросту на поверхні зараженої клітини в культурі мікрофотографія (х 500 000)
Нові віріони ВІЛ виходять з тонкого виросту на поверхні зараженої клітини в культурі мікрофотографія (х 500 000)
 Т-лімфоцит, заражений ВІЛ, утворює нові вірусні частки. мікрофотографія (х 20 000)
Т-лімфоцит, заражений ВІЛ, утворює нові вірусні частки. мікрофотографія (х 20 000)
 Віріони ВІЛ масовано покидають заражену клітину в культурі мікрофотографія (х 10 000)
Віріони ВІЛ масовано покидають заражену клітину в культурі мікрофотографія (х 10 000)
 Синцитій - велика кількість ядер, оточених однією клітинною мембраною – ознака зараження ВІЛ в культурі клітин. Синцитій утворюється в результаті того, що заражені клітини, які утворюють вірусний білок gp120 і несуть його на своїй поверхні, зливаються із здоровими клітинами, які мають у мембрані молекули антигену CD4. Клітини HeLa, що не мають CD4, не можуть бути зараженими ВІЛ Генетично змінені клітини HeLa, що мають CD4, заражаються ВІЛ і утворюють синцитії
Синцитій - велика кількість ядер, оточених однією клітинною мембраною – ознака зараження ВІЛ в культурі клітин. Синцитій утворюється в результаті того, що заражені клітини, які утворюють вірусний білок gp120 і несуть його на своїй поверхні, зливаються із здоровими клітинами, які мають у мембрані молекули антигену CD4. Клітини HeLa, що не мають CD4, не можуть бути зараженими ВІЛ Генетично змінені клітини HeLa, що мають CD4, заражаються ВІЛ і утворюють синцитії
 Important facts about HIV No proofreading mechanism leading to accumulation of mutations, 104 – 106 times the incidence of mutations in DNA counterparts 1 nt substitution per replication cycle leading to CD4+ lymphocytes depletion at a rate of 2 billion per day The progeny of a single virus can differ greatly in antigenic configuration (gp120 and gp160) from the parent Extremely rapid RNA virus replication Half-life of free virus is 6-8 hours
Important facts about HIV No proofreading mechanism leading to accumulation of mutations, 104 – 106 times the incidence of mutations in DNA counterparts 1 nt substitution per replication cycle leading to CD4+ lymphocytes depletion at a rate of 2 billion per day The progeny of a single virus can differ greatly in antigenic configuration (gp120 and gp160) from the parent Extremely rapid RNA virus replication Half-life of free virus is 6-8 hours
 ВЗАЄМОДІЯ ВІЛ З КЛІТИНОЮ HIV interaction of with СCD4 cell HIV coreceptors
ВЗАЄМОДІЯ ВІЛ З КЛІТИНОЮ HIV interaction of with СCD4 cell HIV coreceptors

 Attachment of HIV to a CD4+ T-helper cell: 1) the gp120 viral protein attaches to CD4. 2) gp120 variable loop attaches to a coreceptor, either CCR5 or CXCR4. 3) HIV enters the cell.
Attachment of HIV to a CD4+ T-helper cell: 1) the gp120 viral protein attaches to CD4. 2) gp120 variable loop attaches to a coreceptor, either CCR5 or CXCR4. 3) HIV enters the cell.
 З’ясування механізму проникнення ВІЛ у клітину дає основу для розробки вакцин чи захисних препаратів, здатних блокувати дію вірусу на найбільш ранніх стадіях — ще до зараження першої клітини-хазяїна. Перший крок будь-якої вірусної інфекції — зв'язування вірусної частки з компонентом мембрани клітини, що заражається. Спектр клітин і тканин організму, що заражаються ВІЛ, відповідає розподілу CD4-рецепторів.
З’ясування механізму проникнення ВІЛ у клітину дає основу для розробки вакцин чи захисних препаратів, здатних блокувати дію вірусу на найбільш ранніх стадіях — ще до зараження першої клітини-хазяїна. Перший крок будь-якої вірусної інфекції — зв'язування вірусної частки з компонентом мембрани клітини, що заражається. Спектр клітин і тканин організму, що заражаються ВІЛ, відповідає розподілу CD4-рецепторів.
 ВІЛ приєднується до глікопротеїнових рецепторів gp12/gp41 на поверхні Т-лімфоцита
ВІЛ приєднується до глікопротеїнових рецепторів gp12/gp41 на поверхні Т-лімфоцита
 Binding of gp 120 to CD4 and CCR5
Binding of gp 120 to CD4 and CCR5
 Зв'язування ВІЛ з клітиною-мішенню: 1 - ВІЛ наближається до клітини; 2 - gp120 зв’язується з CD4; 3 - експонується білок gp41 і впроваджується у мембрану; 4 – оболонка ВІЛ зливається з мембраною клітини.
Зв'язування ВІЛ з клітиною-мішенню: 1 - ВІЛ наближається до клітини; 2 - gp120 зв’язується з CD4; 3 - експонується білок gp41 і впроваджується у мембрану; 4 – оболонка ВІЛ зливається з мембраною клітини.
 Проникнення серцевини ВІЛ (містить його генетичний матеріал) у клітину Ендоцитоз, опосередкований рецепторами Пряме злиття
Проникнення серцевини ВІЛ (містить його генетичний матеріал) у клітину Ендоцитоз, опосередкований рецепторами Пряме злиття

 Syncytium formation HIV infects CD4+ T cells and makes the cell produce viral proteins (gp120) HIV gp120 receptors displayed on the surface of the T helper cell will bind to other similar lymphocytes. This makes dozens of T helper cells fuse cell membranes into a giant, nonfunctional syncytium, which allows the HIV virion to kill many T helper cells by infecting only one. One infected cell can fuse with 50 healthy cells.
Syncytium formation HIV infects CD4+ T cells and makes the cell produce viral proteins (gp120) HIV gp120 receptors displayed on the surface of the T helper cell will bind to other similar lymphocytes. This makes dozens of T helper cells fuse cell membranes into a giant, nonfunctional syncytium, which allows the HIV virion to kill many T helper cells by infecting only one. One infected cell can fuse with 50 healthy cells.
 Механізм утворення синцитію: Клітини, заражені ВІЛ, синтезують gp120 і несуть його на своїй мембрані. Заражена клітина (має gp120) зв’язується із здоровою клітиною (має CD4+), вони зливаються з утворенням синцитію. Синцитій здатний далі зливатися із здоровими клітинами. Одна заражена клітина може в результаті приєднати до 50 клітин.
Механізм утворення синцитію: Клітини, заражені ВІЛ, синтезують gp120 і несуть його на своїй мембрані. Заражена клітина (має gp120) зв’язується із здоровою клітиною (має CD4+), вони зливаються з утворенням синцитію. Синцитій здатний далі зливатися із здоровими клітинами. Одна заражена клітина може в результаті приєднати до 50 клітин.


 Syncytium
Syncytium
 HIV-infected cells with syncytium formation (left) and their fluorescent image (right upper), and an electron microscopic image of HIV virions (right lower)
HIV-infected cells with syncytium formation (left) and their fluorescent image (right upper), and an electron microscopic image of HIV virions (right lower)
 Спектр тканин, що можуть бути заражені HIV, обумовлений розподілом клітин, що мають антиген CD4. Виняток: гліальні клітини головного мозку, хромафінні клітини товстої, дванадцятипалої і прямої кишок.
Спектр тканин, що можуть бути заражені HIV, обумовлений розподілом клітин, що мають антиген CD4. Виняток: гліальні клітини головного мозку, хромафінні клітини товстої, дванадцятипалої і прямої кишок.
 gp 120/41 CD4+ Зв'язування ВІЛ з клітиною: білок оболонки gp120 взаємодіє з CD4-рецептором і корецептором (CCR-5/CXCR-4). Можливість боротьби з ВІЛ: Перешкодити взаємодії ВІЛ з клітинним рецептором CD4 Блокувати рецептор CD4 або корецептори Екранувати білок gp120 ВІЛ
gp 120/41 CD4+ Зв'язування ВІЛ з клітиною: білок оболонки gp120 взаємодіє з CD4-рецептором і корецептором (CCR-5/CXCR-4). Можливість боротьби з ВІЛ: Перешкодити взаємодії ВІЛ з клітинним рецептором CD4 Блокувати рецептор CD4 або корецептори Екранувати білок gp120 ВІЛ

 HIV coreceptors
HIV coreceptors
 Корецептори ВІЛ
Корецептори ВІЛ
 Проникнення ВІЛ у клітину 1 2 1- природний шлях; 2 – блокування лігандами CCR-5 і CXCR-4 корецепторів
Проникнення ВІЛ у клітину 1 2 1- природний шлях; 2 – блокування лігандами CCR-5 і CXCR-4 корецепторів
 Chemokine receptors (хемокінові рецептори) Their ligands are chemokines (хемокіни) or chemotactic cytokines (хемотактичні цитокіни) Chemokines: ability to induce directed chemotaxis in nearby responsive cells crucial for immune responses towards infection and inflammation
Chemokine receptors (хемокінові рецептори) Their ligands are chemokines (хемокіни) or chemotactic cytokines (хемотактичні цитокіни) Chemokines: ability to induce directed chemotaxis in nearby responsive cells crucial for immune responses towards infection and inflammation
 Хемокінові рецептори (chemokine receptors) Ліганди – хемокіни (хемотаксисні цитокіни) Здатні індукувати спрямований хемотаксис до оточуючих чутливих клітин Критичні для імунної відповіді на інфекцію та запалення
Хемокінові рецептори (chemokine receptors) Ліганди – хемокіни (хемотаксисні цитокіни) Здатні індукувати спрямований хемотаксис до оточуючих чутливих клітин Критичні для імунної відповіді на інфекцію та запалення
 β chemokines: RANTES (Regulated upon Activation, Normal T-cell Expressed, and Secreted) (CCL5) chemotactic for T cells, eosinophils, and basophils, and plays an active role in recruiting leukocytes into inflammatory sites induces the proliferation and activation of certain natural-killer (NK) cells HIV-suppressive factor released from CD8+ T cells MIP (macrophage inflammatory proteins) - MIP-1α (CCL3 ) і MIP-1β (CCL4) major factors produced by macrophages after they are stimulated with bacterial endotoxins
β chemokines: RANTES (Regulated upon Activation, Normal T-cell Expressed, and Secreted) (CCL5) chemotactic for T cells, eosinophils, and basophils, and plays an active role in recruiting leukocytes into inflammatory sites induces the proliferation and activation of certain natural-killer (NK) cells HIV-suppressive factor released from CD8+ T cells MIP (macrophage inflammatory proteins) - MIP-1α (CCL3 ) і MIP-1β (CCL4) major factors produced by macrophages after they are stimulated with bacterial endotoxins
 β-хемокіни RANTES (Regulated upon Activation, Normal T-cell Expressed, and Secreted) (CCL5) Викликає хемотаксис Т-клітин, еозинофілів та базофілів, Активна роль у рекрутуванні лейкоцитів до вогни ща запалення; Індукує проліферацію та активацію певних натуральних кілерів; ВІЛ-супресивний фактор, що вивільнюється з CD8+ -клітин. MIP (macrophage inflammatory proteins) - MIP-1α (CCL3 ) і MIP-1β (CCL4) Продукується макрофагами після їх стимуляції бактеріальними ендотксинами.
β-хемокіни RANTES (Regulated upon Activation, Normal T-cell Expressed, and Secreted) (CCL5) Викликає хемотаксис Т-клітин, еозинофілів та базофілів, Активна роль у рекрутуванні лейкоцитів до вогни ща запалення; Індукує проліферацію та активацію певних натуральних кілерів; ВІЛ-супресивний фактор, що вивільнюється з CD8+ -клітин. MIP (macrophage inflammatory proteins) - MIP-1α (CCL3 ) і MIP-1β (CCL4) Продукується макрофагами після їх стимуляції бактеріальними ендотксинами.
 19 different chemokine receptors G protein-coupled receptors containing 7 transmembrane domains that are found on the surface of leukocytes 4 families depending on the type of chemokine they bind CXCR -bind the CXC chemokines, CCR - bind the CC chemokines, CX3CR1 - binds the CX3C chemokine (CX3CL1), XCR1 - binds the two XC chemokines (XCL1 and XCL2). Signal transduction – via phosphatidylinositol (4,5)-bisphosphate (PIP2) system Responses - chemotaxis, degranulation, release of superoxide anions and changes in the avidity of cell adhesion molecules called integrins within the cell harbouring the chemokine receptor
19 different chemokine receptors G protein-coupled receptors containing 7 transmembrane domains that are found on the surface of leukocytes 4 families depending on the type of chemokine they bind CXCR -bind the CXC chemokines, CCR - bind the CC chemokines, CX3CR1 - binds the CX3C chemokine (CX3CL1), XCR1 - binds the two XC chemokines (XCL1 and XCL2). Signal transduction – via phosphatidylinositol (4,5)-bisphosphate (PIP2) system Responses - chemotaxis, degranulation, release of superoxide anions and changes in the avidity of cell adhesion molecules called integrins within the cell harbouring the chemokine receptor

 CD8+ T-лімфоцити продукують β хемокіни RANTES, MIP 1α і1β, які пригнічують HIV-1 (HIV-suppressive factors).
CD8+ T-лімфоцити продукують β хемокіни RANTES, MIP 1α і1β, які пригнічують HIV-1 (HIV-suppressive factors).
 CCR5 - -хемокіновий рецептор, є корецептором штамів ВІЛ, що уражують макрофаги. -CXCR4 - -хемокіновий рецептор, є корецептором штамів ВІЛ, що мають тропізм до Т-лімфоцитів. Різні варіанти ВІЛ використовують: або CCR-5 – штам R5-HIV-1 (M-tropic) або CXCR-4 - штам X4-HIV-1 (T-tropic) обидва корецептори відразу - штам X4R5-HIV-1 або R3-HIV-1 Корецептори ВІЛ CXCR4-корецептор – експресується на мієлоїдних клітинах, Т- і В-клітинах, макрофагах, епітеліальних, ендотеліальних і дендритних клітинах. Взаємодія з gp120 ВІЛ з хемокіновим рецептором запускає конформаційні зміни, що призводять до злиття оболонки вірусу та клітинної мембрани.
CCR5 - -хемокіновий рецептор, є корецептором штамів ВІЛ, що уражують макрофаги. -CXCR4 - -хемокіновий рецептор, є корецептором штамів ВІЛ, що мають тропізм до Т-лімфоцитів. Різні варіанти ВІЛ використовують: або CCR-5 – штам R5-HIV-1 (M-tropic) або CXCR-4 - штам X4-HIV-1 (T-tropic) обидва корецептори відразу - штам X4R5-HIV-1 або R3-HIV-1 Корецептори ВІЛ CXCR4-корецептор – експресується на мієлоїдних клітинах, Т- і В-клітинах, макрофагах, епітеліальних, ендотеліальних і дендритних клітинах. Взаємодія з gp120 ВІЛ з хемокіновим рецептором запускає конформаційні зміни, що призводять до злиття оболонки вірусу та клітинної мембрани.

 CCR5 C-C chemokine receptor type 5, also known as CCR5 or CD195. It is a protein on the surface of white blood cells that is involved in the immune system as it acts as a receptor for chemokines. This is the process by which T cells are attracted to specific tissue and organ targets. Many forms of HIV, the virus that causes AIDS, initially use CCR5 to enter and infect host cells.
CCR5 C-C chemokine receptor type 5, also known as CCR5 or CD195. It is a protein on the surface of white blood cells that is involved in the immune system as it acts as a receptor for chemokines. This is the process by which T cells are attracted to specific tissue and organ targets. Many forms of HIV, the virus that causes AIDS, initially use CCR5 to enter and infect host cells.
 CCR5 The CCR5 protein belongs to the beta chemokine receptors family of integral membrane proteins. It is a G protein–coupled receptor which functions as a chemokine receptor in the CC chemokine group. CCR5 is predominantly expressed on T cells, macrophages, dendritic cells, eosinophils and microglia. It is likely that CCR5 plays a role in inflammatory responses to infection, though its exact role in normal immune function is unclear. Regions of this protein are also crucial for chemokine ligand binding, functional response of the receptor, and HIV co-receptor activity.
CCR5 The CCR5 protein belongs to the beta chemokine receptors family of integral membrane proteins. It is a G protein–coupled receptor which functions as a chemokine receptor in the CC chemokine group. CCR5 is predominantly expressed on T cells, macrophages, dendritic cells, eosinophils and microglia. It is likely that CCR5 plays a role in inflammatory responses to infection, though its exact role in normal immune function is unclear. Regions of this protein are also crucial for chemokine ligand binding, functional response of the receptor, and HIV co-receptor activity.
 gp120 binding to CCR5 HIV most commonly uses CCR5 and/or CXCR4 as a co-receptor to enter its target cells. The tyrosine sulfated amino terminus of this co-receptor is the "essential determinant" of binding to the gp120 glycoprotein. Co-receptor recognition also include the V1-V2 region of gp120, and the bridging sheet (an antiparallel, 4-stranded β sheet that connects the inner and outer domains of gp120). The V1-V2 stem can influence "co-receptor usage through its peptide composition as well as by the degree of N-linked glycosylation."
gp120 binding to CCR5 HIV most commonly uses CCR5 and/or CXCR4 as a co-receptor to enter its target cells. The tyrosine sulfated amino terminus of this co-receptor is the "essential determinant" of binding to the gp120 glycoprotein. Co-receptor recognition also include the V1-V2 region of gp120, and the bridging sheet (an antiparallel, 4-stranded β sheet that connects the inner and outer domains of gp120). The V1-V2 stem can influence "co-receptor usage through its peptide composition as well as by the degree of N-linked glycosylation."
 CCR5 ligands The normal ligands for this receptor, RANTES, MIP-1β, and MIP-1α, are able to suppress HIV-1 infection in vitro.
CCR5 ligands The normal ligands for this receptor, RANTES, MIP-1β, and MIP-1α, are able to suppress HIV-1 infection in vitro.
 How to block CCR5 function? A number of new experimental HIV drugs, called CCR5 receptor antagonists, have been designed to interfere with the interaction between CCR5 and HIV (specifically to its envelope glycoprotein gp120), including: PRO140 (Progenics), Vicriviroc (Phase III trials were cancelled in July 2010) (Schering Plough), Aplaviroc (GW-873140) (GlaxoSmithKline) Maraviroc (UK-427857) (Pfizer). Maraviroc was approved for use by the FDA in August 2007. It is the only one thus far approved by the FDA for clinical use, thus becoming the first CCR5 inhibitor.
How to block CCR5 function? A number of new experimental HIV drugs, called CCR5 receptor antagonists, have been designed to interfere with the interaction between CCR5 and HIV (specifically to its envelope glycoprotein gp120), including: PRO140 (Progenics), Vicriviroc (Phase III trials were cancelled in July 2010) (Schering Plough), Aplaviroc (GW-873140) (GlaxoSmithKline) Maraviroc (UK-427857) (Pfizer). Maraviroc was approved for use by the FDA in August 2007. It is the only one thus far approved by the FDA for clinical use, thus becoming the first CCR5 inhibitor.
 CCR5 mutations A few individuals carry a mutation known as CCR5-Δ32 in the CCR5 gene, protecting them against these strains of HIV. CCR5-Δ32 is a deletion mutation of a gene that has a specific impact on the function of T cells. The deleted portion of the CCR5 gene consists of thirty-two base pairs that correspond to the second extracellular loop of the receptor. The mutated receptor is non-functional and does not allow M-tropic HIV-1 virus entry, thus resulting in infection resistance.
CCR5 mutations A few individuals carry a mutation known as CCR5-Δ32 in the CCR5 gene, protecting them against these strains of HIV. CCR5-Δ32 is a deletion mutation of a gene that has a specific impact on the function of T cells. The deleted portion of the CCR5 gene consists of thirty-two base pairs that correspond to the second extracellular loop of the receptor. The mutated receptor is non-functional and does not allow M-tropic HIV-1 virus entry, thus resulting in infection resistance.
 ВІЛ-інфіковані: мутація гену env CCR-5 CXCR-4 співпадає із спустошенням CD4-клітин переважний корецептор
ВІЛ-інфіковані: мутація гену env CCR-5 CXCR-4 співпадає із спустошенням CD4-клітин переважний корецептор
 Інігбування проникнення ВІЛ у клітину: стеричний ефект – конкурентна блокада рецептора шляхом прямого зв’язування з ним ліганду; інактивація рецептора після зв’язування хемокіну. Вивільнення хемокінів – рання реакція на інфікування ВІЛ. Дослідження показали: Високий рівень хемокінів (рецептор - CCR-5) пов’язаний з повільним прогресуванням ВІЛ-інфекції.
Інігбування проникнення ВІЛ у клітину: стеричний ефект – конкурентна блокада рецептора шляхом прямого зв’язування з ним ліганду; інактивація рецептора після зв’язування хемокіну. Вивільнення хемокінів – рання реакція на інфікування ВІЛ. Дослідження показали: Високий рівень хемокінів (рецептор - CCR-5) пов’язаний з повільним прогресуванням ВІЛ-інфекції.
 CD4 Binding: The gp120 viral envelope protein has an affinity for the CD4 receptor which is expressed on the cell surface of the CD4+ cell. CD4 binding inhibitors such as the experimental drug TNX-355 have been shown to inhibit the gp120 and CD4 receptor interaction and block HIV infection.
CD4 Binding: The gp120 viral envelope protein has an affinity for the CD4 receptor which is expressed on the cell surface of the CD4+ cell. CD4 binding inhibitors such as the experimental drug TNX-355 have been shown to inhibit the gp120 and CD4 receptor interaction and block HIV infection.
 Co-receptor Binding: The binding of the gp120 to the CD4 receptor causes a conformational change in the gp120 protein. This change exposes a binding site for a chemokine co-receptor (CCR5 or CXCR4). CCR5 antagonists block the binding of the gp120 protein and block HIV infection.
Co-receptor Binding: The binding of the gp120 to the CD4 receptor causes a conformational change in the gp120 protein. This change exposes a binding site for a chemokine co-receptor (CCR5 or CXCR4). CCR5 antagonists block the binding of the gp120 protein and block HIV infection.
 Exploring HIV-1 Structure Entry of HIV involves a sequential interaction of the envelope glycoprotein (gp120) CD4 glycoprotein chemokine receptor (primary receptor) CD4i (antibodies that block gp120-CD4 complexes to the chemokine receptor) CCR5 and CXCR4 for HIV-1 (secondary receptors) CD4 binding induces conformational changes in the gp120 Entry of HIV is mediated by envelope glycoproteins 5 variable regions Variable and non variable regions are glycosylated V3 loop determines specificity
Exploring HIV-1 Structure Entry of HIV involves a sequential interaction of the envelope glycoprotein (gp120) CD4 glycoprotein chemokine receptor (primary receptor) CD4i (antibodies that block gp120-CD4 complexes to the chemokine receptor) CCR5 and CXCR4 for HIV-1 (secondary receptors) CD4 binding induces conformational changes in the gp120 Entry of HIV is mediated by envelope glycoproteins 5 variable regions Variable and non variable regions are glycosylated V3 loop determines specificity
 gp120 і деменція gp120 мітохондріальні білки клітинної загибелі, подібні до каспаз індукція зміна експресії Fas апоптоз у нейронах gp120 індукція оксидативний стрес STAT1 gp120 активація індукція секреція IL-6 і IL-8 у нейронах
gp120 і деменція gp120 мітохондріальні білки клітинної загибелі, подібні до каспаз індукція зміна експресії Fas апоптоз у нейронах gp120 індукція оксидативний стрес STAT1 gp120 активація індукція секреція IL-6 і IL-8 у нейронах
 Signaling pathways of Fas
Signaling pathways of Fas
 викликає дисбаланс у системі цитокінів; підвищує концентрацію Са, індукує синтез інозитолтрифосфату; інгібує мітогенну або антигенну активацію Т-клітин; змінює продукцію цитокінів шляхом активації субпопуляцій мононуклеарних клітин периферійної крові (PBMC); впливає на активність АГ-представляючих клітин; порушує цитотоксичність і хемотаксис у відповідь на АГ; стимулює поліклональну активацію В-клітин; посилює продукцію певних цитокінів (IL6, TNF-alpha, IL1-alpha and IL1-beta, IL10 and IL8). Вплив gp120 на імунокомпетентні клітини
викликає дисбаланс у системі цитокінів; підвищує концентрацію Са, індукує синтез інозитолтрифосфату; інгібує мітогенну або антигенну активацію Т-клітин; змінює продукцію цитокінів шляхом активації субпопуляцій мононуклеарних клітин периферійної крові (PBMC); впливає на активність АГ-представляючих клітин; порушує цитотоксичність і хемотаксис у відповідь на АГ; стимулює поліклональну активацію В-клітин; посилює продукцію певних цитокінів (IL6, TNF-alpha, IL1-alpha and IL1-beta, IL10 and IL8). Вплив gp120 на імунокомпетентні клітини
 Система цитокінів
Система цитокінів
 Цитокіни Прозапальні Протизапальні Цитокіни, що регулюють клітинну і гуморальну імунну відповідь Цитокіни, що мають власні ефекторні (противірусні, цитотоксичні) функції IL-1, IL-6, IL-8, IL-12, TNF-, IFN-, IFN-, MIF IL-4, IL-10, IL-13, TGF IL-1, IL-2, IL-4, IL-5, IL-5, IL-6, IL-7, IL-9, IL-10, IL-12, IL-13, IL-14, IL-15, IFN-, TGF
Цитокіни Прозапальні Протизапальні Цитокіни, що регулюють клітинну і гуморальну імунну відповідь Цитокіни, що мають власні ефекторні (противірусні, цитотоксичні) функції IL-1, IL-6, IL-8, IL-12, TNF-, IFN-, IFN-, MIF IL-4, IL-10, IL-13, TGF IL-1, IL-2, IL-4, IL-5, IL-5, IL-6, IL-7, IL-9, IL-10, IL-12, IL-13, IL-14, IL-15, IFN-, TGF
 Прозапальні цитокіни IL-1 і IL-1 IL-1 Клітини-продуценти: гемопоетичні, епітеліальні, нервові клітини. Продукується не спонтанно, а у відповідь на: інфекцію, мікробні токсини, запальні агенти, білки комплементу, білки зсідання крові. Функція: широкий спектр прозапальної, метаболічної, фізіологічної, гемопоетичної та імунологічної активності.
Прозапальні цитокіни IL-1 і IL-1 IL-1 Клітини-продуценти: гемопоетичні, епітеліальні, нервові клітини. Продукується не спонтанно, а у відповідь на: інфекцію, мікробні токсини, запальні агенти, білки комплементу, білки зсідання крові. Функція: широкий спектр прозапальної, метаболічної, фізіологічної, гемопоетичної та імунологічної активності.
 Синергізм: IL-1 + TNF- + IL-6 – прозапальні ефекти: стимулюють Т- і В-лімфоцити, посилюють клітинну проліферацію, впливають на експресію певних генів; IL-1 – медіатор запалення: опосередковує розвиток системної гострофазної відповіді. Підвищення рівню IL-1: лихоманка, анорексія, нейтрофілія, активація ендотеліальних клітин і пдвищення експресії на них молекул адгезії, активація нейтрофілів, підвищення синтезу білків гострої фази і комплементу. IL-1 – індукує власний синтез й експресію рецепторів для IL-2.
Синергізм: IL-1 + TNF- + IL-6 – прозапальні ефекти: стимулюють Т- і В-лімфоцити, посилюють клітинну проліферацію, впливають на експресію певних генів; IL-1 – медіатор запалення: опосередковує розвиток системної гострофазної відповіді. Підвищення рівню IL-1: лихоманка, анорексія, нейтрофілія, активація ендотеліальних клітин і пдвищення експресії на них молекул адгезії, активація нейтрофілів, підвищення синтезу білків гострої фази і комплементу. IL-1 – індукує власний синтез й експресію рецепторів для IL-2.
 IL-6 Клітини-продуценти: лімфоїдні та нелімфоїдні. Функція: регулює імунну відповідь, запальну гострофазову відповідь і гемопоез: дозрівання АТ-продукуючих клітин з В-лімфоцитів та продукцію АТ; активацію Т-лімфоцитів; синтез гострофазних білків. IL-6 TNF- інгібує продукцію IL-1 інудкують синтез
IL-6 Клітини-продуценти: лімфоїдні та нелімфоїдні. Функція: регулює імунну відповідь, запальну гострофазову відповідь і гемопоез: дозрівання АТ-продукуючих клітин з В-лімфоцитів та продукцію АТ; активацію Т-лімфоцитів; синтез гострофазних білків. IL-6 TNF- інгібує продукцію IL-1 інудкують синтез
 Клітини-продуценти: моноцити і макрофаги, лімфоцити крові, натуральні кілери, гранулоцити крові, Т-лімфоцитарні клітинні лінії. Індуктори синтезу: бактеріальні ліполісахариди (ЛПС), цитокіни (IL-1, IL-2, IFN- і -). Функція: активація або інгібування експресії генів, напр., інших прозапальних цитокінів (через фактор NFB). Біологічна активність: цитотоксичність по відношенню до деяких пухлинних клітин; пригнічення ліпопротеїнкінази; участь у регуляції імунної відповіді і запалення. TNF-
Клітини-продуценти: моноцити і макрофаги, лімфоцити крові, натуральні кілери, гранулоцити крові, Т-лімфоцитарні клітинні лінії. Індуктори синтезу: бактеріальні ліполісахариди (ЛПС), цитокіни (IL-1, IL-2, IFN- і -). Функція: активація або інгібування експресії генів, напр., інших прозапальних цитокінів (через фактор NFB). Біологічна активність: цитотоксичність по відношенню до деяких пухлинних клітин; пригнічення ліпопротеїнкінази; участь у регуляції імунної відповіді і запалення. TNF-
 Запуск запалення: активує ендотелій; сприяє адгезії лейкоцитів до ендотелію через індукцію на ньому експресії молекул адгезії і міграції лейкоцитів до вогнища запалення; активує лейкоцити (гранулоцити, моноцити, лімфоцити); індукує продукцію інших прозапальних цитокінів (IL-1, IL-6, IFN-), що виявляють синергізм з TNF-. Місцева продукція TNF- у вогнищі запалення викликає: хемотаксис гранулоцитів і моноцитів, посилення фагоцитозу; дегрануляцію фагоцитів; продукцію і секрецію реактивних кисневих радикалів; збільшену цитотоксичність фагоцитів; Т-лімфоцити посилюють експресію рецепторів для IL-2 і TNF-. TNF-
Запуск запалення: активує ендотелій; сприяє адгезії лейкоцитів до ендотелію через індукцію на ньому експресії молекул адгезії і міграції лейкоцитів до вогнища запалення; активує лейкоцити (гранулоцити, моноцити, лімфоцити); індукує продукцію інших прозапальних цитокінів (IL-1, IL-6, IFN-), що виявляють синергізм з TNF-. Місцева продукція TNF- у вогнищі запалення викликає: хемотаксис гранулоцитів і моноцитів, посилення фагоцитозу; дегрануляцію фагоцитів; продукцію і секрецію реактивних кисневих радикалів; збільшену цитотоксичність фагоцитів; Т-лімфоцити посилюють експресію рецепторів для IL-2 і TNF-. TNF-

 IFN- Клітини-продуценти: активовані Т-лімфоцити і натуральні кілери. активовані Т-лімфоцити розпізнавання комплексу АГ з МНС І або ІІ продукція IFN- регуляція IL-2 IL-10 стимулює пригнічує
IFN- Клітини-продуценти: активовані Т-лімфоцити і натуральні кілери. активовані Т-лімфоцити розпізнавання комплексу АГ з МНС І або ІІ продукція IFN- регуляція IL-2 IL-10 стимулює пригнічує
 натуральні кілери взаємодія з клітинами-мішенями (пухлинними, зараженими) продукція IFN- регуляція IL-12 стимулює (продукується активованими макрофагами і Т-лімфоцитами) IFN-
натуральні кілери взаємодія з клітинами-мішенями (пухлинними, зараженими) продукція IFN- регуляція IL-12 стимулює (продукується активованими макрофагами і Т-лімфоцитами) IFN-
 Функції IFN-: активація секреторних функцій макрофагів (мікробіцидності й цитотоксичності), продукції ними цитокінів, кисневих радикалів, простагнландинів; підвищення експресії МНС І і ІІ на різних клітинах підвищення ефективності презентації АГ і їх розпізнавання Т-лімфоцитами; участь в адгезії лімфоцитів до ендотеліальних клітин у посткапілярних венах; сумісно з TNF- індукує продукцію хемокінів (напр., RANTES); збільшує функціональну активність цитотоксичних Т-лімфоцитів. IFN-
Функції IFN-: активація секреторних функцій макрофагів (мікробіцидності й цитотоксичності), продукції ними цитокінів, кисневих радикалів, простагнландинів; підвищення експресії МНС І і ІІ на різних клітинах підвищення ефективності презентації АГ і їх розпізнавання Т-лімфоцитами; участь в адгезії лімфоцитів до ендотеліальних клітин у посткапілярних венах; сумісно з TNF- індукує продукцію хемокінів (напр., RANTES); збільшує функціональну активність цитотоксичних Т-лімфоцитів. IFN-
 Протизапальні цитокіни IL-4 Клітини-продуценти: Т-хелпери 2 порядку (TH2), частково тучні клітини, базофіли, В-лімфоцити, сторомальні клітини кістковго мозку. Функція: контроль проліферації, диференціювання і функцій В-лімфоцитів (антитільної відповіді); інгібування моноцитів/макрофагів; блокування продукції прозапальних цитокінів (IL-1, IL-8, IL-8, TNF-) моноцитами і макрофагами; блокування продукції супероксидних радикалів і ПГ Е2.
Протизапальні цитокіни IL-4 Клітини-продуценти: Т-хелпери 2 порядку (TH2), частково тучні клітини, базофіли, В-лімфоцити, сторомальні клітини кістковго мозку. Функція: контроль проліферації, диференціювання і функцій В-лімфоцитів (антитільної відповіді); інгібування моноцитів/макрофагів; блокування продукції прозапальних цитокінів (IL-1, IL-8, IL-8, TNF-) моноцитами і макрофагами; блокування продукції супероксидних радикалів і ПГ Е2.
 Протизапальні цитокіни IL-13 Клітини-продуценти: активовані CD4+- (Th1 і Th2) і CD8+-Т-лімфоцити. Функція: Спільна з IL-4 дія на моноцити, макрофаги і В-лімфоцити, але не діє на Т-лімфоцити. підвищує адгезію і виживання моноцитів, підвищує експресію на моноцитах молекул адгезії, МНС ІІ, посилює їх АГ-презентуючу функцію; інгібує продукцію макрофагами цитокінів IL-1, IL-6, IL-8, IL-10, TNF-; підвищує експресію на В-лімфоцитах МНС ІІ, поверхневих Ig, сприяяє переключення синтезу з IgG4 на IgE; індукує синтез IFN- натуральними кілерами.
Протизапальні цитокіни IL-13 Клітини-продуценти: активовані CD4+- (Th1 і Th2) і CD8+-Т-лімфоцити. Функція: Спільна з IL-4 дія на моноцити, макрофаги і В-лімфоцити, але не діє на Т-лімфоцити. підвищує адгезію і виживання моноцитів, підвищує експресію на моноцитах молекул адгезії, МНС ІІ, посилює їх АГ-презентуючу функцію; інгібує продукцію макрофагами цитокінів IL-1, IL-6, IL-8, IL-10, TNF-; підвищує експресію на В-лімфоцитах МНС ІІ, поверхневих Ig, сприяяє переключення синтезу з IgG4 на IgE; індукує синтез IFN- натуральними кілерами.
 PBMC gp120 стимуляція IFN- і IFN- продукція бета-2-мікроглобулін і неоптерин експресія рецептори для IL2 екзогенний IL2 проліферація PBMC
PBMC gp120 стимуляція IFN- і IFN- продукція бета-2-мікроглобулін і неоптерин експресія рецептори для IL2 екзогенний IL2 проліферація PBMC
 Взаємодія між gp120 і TNF- - роль у нелімфоцитарній не пов’язаній з імунодефіцитом деструкції тканин (non-lymphocytic-, non-immunodeficiency-related tissue-destructive morbidity) gp120 моноцити посилення експресії TNF- надлишкове зв’язування рецепторів TNF- на сусідніх лімфоцитах, моноцитах і гліальних клітинах* посилення реплікації ВІЛ посилення зв’язування gp120 з клітинами моноцитарного ряду посилення продукції TNF- * - у гліальних клітинах ВІЛ не реплікується
Взаємодія між gp120 і TNF- - роль у нелімфоцитарній не пов’язаній з імунодефіцитом деструкції тканин (non-lymphocytic-, non-immunodeficiency-related tissue-destructive morbidity) gp120 моноцити посилення експресії TNF- надлишкове зв’язування рецепторів TNF- на сусідніх лімфоцитах, моноцитах і гліальних клітинах* посилення реплікації ВІЛ посилення зв’язування gp120 з клітинами моноцитарного ряду посилення продукції TNF- * - у гліальних клітинах ВІЛ не реплікується
 gp120 клітини глії продукція TNF- руйнування сусідніх нейронів продукція цитокінів активація
gp120 клітини глії продукція TNF- руйнування сусідніх нейронів продукція цитокінів активація
 gp41 викликає злиття оболонки ВІЛ з мембраною клітини. Відносно консервативний, більша частина поверхні схована від антитіл. gp41 gp41 енфувіртид + зв’язування пригнічення злиття Поліклональні АТ цапа (Virionyx, a New Zealand Company) – зв’язуються з gp41 (показано у клінічних дослідженнях на людині).
gp41 викликає злиття оболонки ВІЛ з мембраною клітини. Відносно консервативний, більша частина поверхні схована від антитіл. gp41 gp41 енфувіртид + зв’язування пригнічення злиття Поліклональні АТ цапа (Virionyx, a New Zealand Company) – зв’язуються з gp41 (показано у клінічних дослідженнях на людині).
 gp41
gp41
 CD4-рецептор (cluster of differentiation 4) Експресується на поверхні Т-хелперів, регуляторних Т-клітин, моноцитів, макрофагів і дендритних клітин. 1970 р. - названий leu-3 і T4 (після того, як з ним реагували монАТ OKT4). 1984 р. - названий CD4.
CD4-рецептор (cluster of differentiation 4) Експресується на поверхні Т-хелперів, регуляторних Т-клітин, моноцитів, макрофагів і дендритних клітин. 1970 р. - названий leu-3 і T4 (після того, як з ним реагували монАТ OKT4). 1984 р. - названий CD4.
 gp120 CD4 Залишок Phe 43 CD4 знаходиться у зв’язуючій “кишені” gp120
gp120 CD4 Залишок Phe 43 CD4 знаходиться у зв’язуючій “кишені” gp120
 CD4 – корецептор, який допомагає Т-клітинному рецептору (TCR) активувати Т-клітини при їх зустрічі з АГ-представляючою клітиною. Посилює сигнал TCR за участю ферменту тирозин кінази lck. Активація каскадів внутрішньоклітинної сигналізації в Т-клітинах Взаємодіє з МНС ІІ на поверхні АГ-представляючих клітин за допомогою позаклітинного домену.
CD4 – корецептор, який допомагає Т-клітинному рецептору (TCR) активувати Т-клітини при їх зустрічі з АГ-представляючою клітиною. Посилює сигнал TCR за участю ферменту тирозин кінази lck. Активація каскадів внутрішньоклітинної сигналізації в Т-клітинах Взаємодіє з МНС ІІ на поверхні АГ-представляючих клітин за допомогою позаклітинного домену.

 Для зв'язування gp120 ВІЛ білком CD4 є необхідним домен на ΝΗ2-кінці молекули - ділянка, найбільш віддалена від клітинної мембрани). CD4-рецептор
Для зв'язування gp120 ВІЛ білком CD4 є необхідним домен на ΝΗ2-кінці молекули - ділянка, найбільш віддалена від клітинної мембрани). CD4-рецептор
 Ig-подібні домени експоновані у позаклітинне середовище: варіабельні IgV-домени – подібні до доменів D1 і D3; константні IgC-домени – подібні до доменів D2 і D4. Домен D1 – взаємодіє з β2-доменом MHC II. Цитоплазматичний С-домен CD4 містить послідовність амінокислот, що взаємодіє з молекулою тирозин кінази lck.
Ig-подібні домени експоновані у позаклітинне середовище: варіабельні IgV-домени – подібні до доменів D1 і D3; константні IgC-домени – подібні до доменів D2 і D4. Домен D1 – взаємодіє з β2-доменом MHC II. Цитоплазматичний С-домен CD4 містить послідовність амінокислот, що взаємодіє з молекулою тирозин кінази lck.
 Зв’язування TCR з MHC
Зв’язування TCR з MHC


 Вміст CD4 у крові – критерій необхідності початку АРТ або ефективності лікування Норма - 500-1200x106/л. CD4-тест – визначення кількості Т-клітин у мл крові за вмістом CD4. 350 клітин/мл в осіб, що проходять лікування або <200 клітин/мл у ВІЛ-інфікованих СНІД
Вміст CD4 у крові – критерій необхідності початку АРТ або ефективності лікування Норма - 500-1200x106/л. CD4-тест – визначення кількості Т-клітин у мл крові за вмістом CD4. 350 клітин/мл в осіб, що проходять лікування або <200 клітин/мл у ВІЛ-інфікованих СНІД

