2014_Борок_Мюге_Колюшка.pptx
- Количество слайдов: 67
 ГЕНОМИКА АДАПТАЦИЙ ТРЕХИГЛОЙ КОЛЮШКИ GASTEROSTEUS ACULEATUS Н. С. Мюге Институт Биологии Развития, (ИБР РАН) Институт Рыбного Хозяйства и Океанографии (ВНИРО)
ГЕНОМИКА АДАПТАЦИЙ ТРЕХИГЛОЙ КОЛЮШКИ GASTEROSTEUS ACULEATUS Н. С. Мюге Институт Биологии Развития, (ИБР РАН) Институт Рыбного Хозяйства и Океанографии (ВНИРО)
 Three-spine stickleback (трехиглая колюшка)
Three-spine stickleback (трехиглая колюшка)
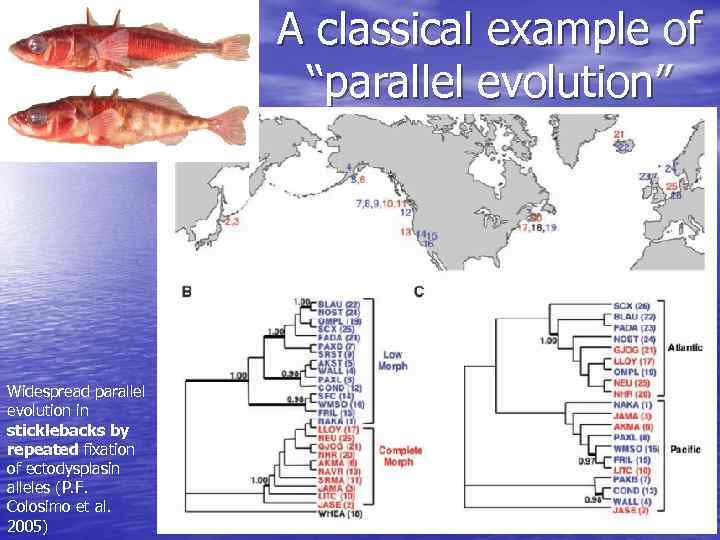 A classical example of “parallel evolution” Widespread parallel evolution in sticklebacks by repeated fixation of ectodysplasin alleles (P. F. Colosimo et al. 2005)
A classical example of “parallel evolution” Widespread parallel evolution in sticklebacks by repeated fixation of ectodysplasin alleles (P. F. Colosimo et al. 2005)
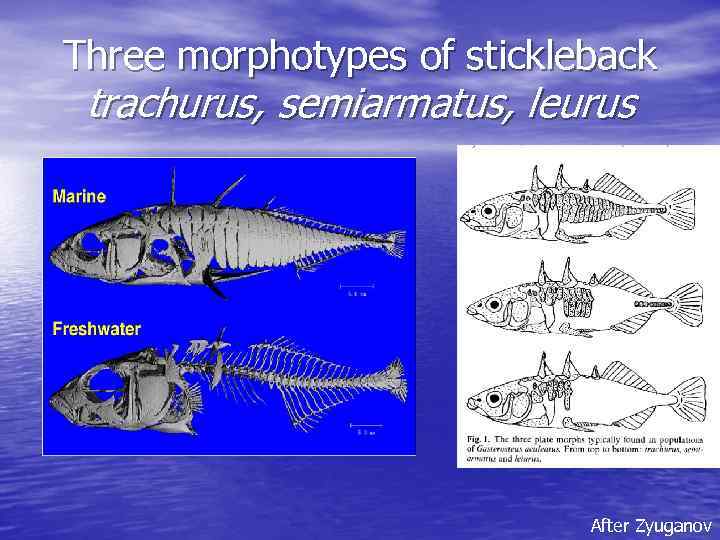 Three morphotypes of stickleback trachurus, semiarmatus, leurus After Zyuganov
Three morphotypes of stickleback trachurus, semiarmatus, leurus After Zyuganov

 Жилые пресноводные колюшки в сравнении с анадромными (морскими): • Адаптации – Соленость и температура – Пища – Состав паразитофауны – Условия зимовки • Видообразование – Ассортативное скрещивание (reproductive isolation) в некоторых изолятах
Жилые пресноводные колюшки в сравнении с анадромными (морскими): • Адаптации – Соленость и температура – Пища – Состав паразитофауны – Условия зимовки • Видообразование – Ассортативное скрещивание (reproductive isolation) в некоторых изолятах
 • Lake Azhabachie, Kamchatka River
• Lake Azhabachie, Kamchatka River
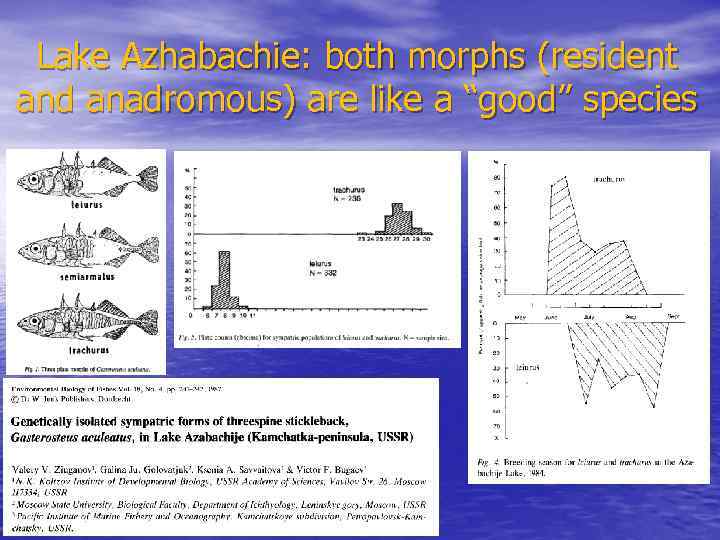 Lake Azhabachie: both morphs (resident and anadromous) are like a “good” species
Lake Azhabachie: both morphs (resident and anadromous) are like a “good” species
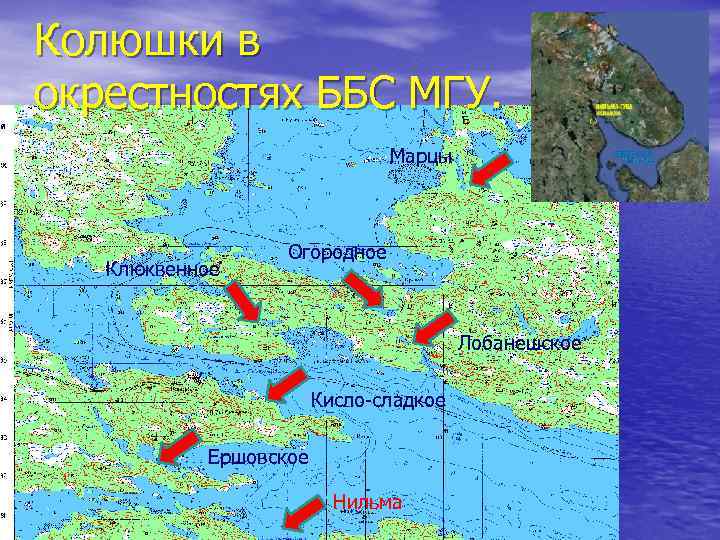 Колюшки в окрестностях ББС МГУ. Марцы Клюквенное Огородное Лобанешское Кисло-сладкое Ершовское Нильма
Колюшки в окрестностях ББС МГУ. Марцы Клюквенное Огородное Лобанешское Кисло-сладкое Ершовское Нильма
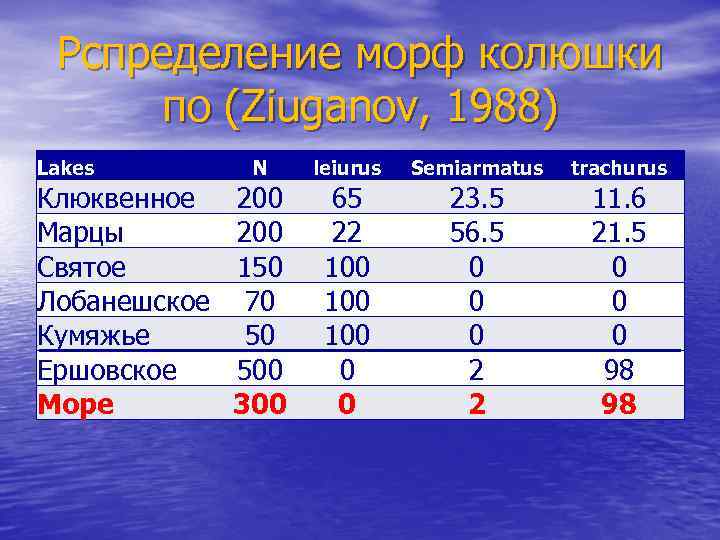 Рспределение морф колюшки по (Ziuganov, 1988) Lakes Клюквенное Марцы Святое Лобанешское Кумяжье Ершовское Море N leiurus Semiarmatus trachurus 200 150 70 50 500 300 65 22 100 100 0 0 23. 5 56. 5 0 0 0 2 2 11. 6 21. 5 0 0 0 98 98
Рспределение морф колюшки по (Ziuganov, 1988) Lakes Клюквенное Марцы Святое Лобанешское Кумяжье Ершовское Море N leiurus Semiarmatus trachurus 200 150 70 50 500 300 65 22 100 100 0 0 23. 5 56. 5 0 0 0 2 2 11. 6 21. 5 0 0 0 98 98
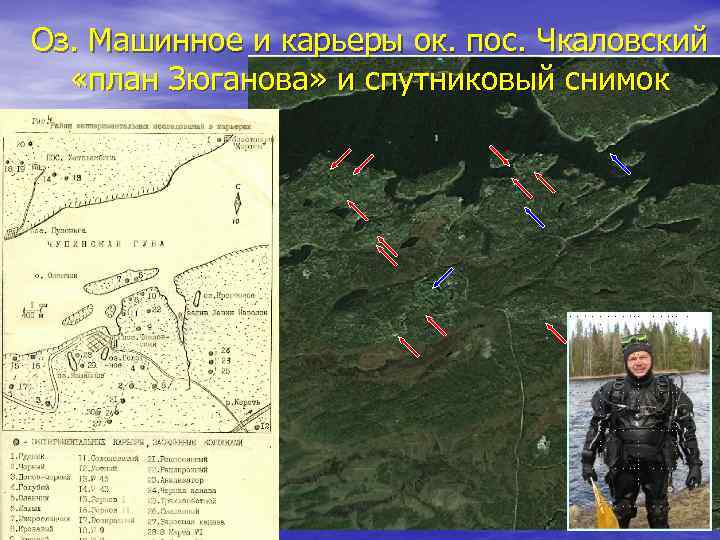 Оз. Машинное и карьеры ок. пос. Чкаловский «план Зюганова» и спутниковый снимок
Оз. Машинное и карьеры ок. пос. Чкаловский «план Зюганова» и спутниковый снимок

 Экспериментальные скрещивания В. В. Зюганова(1983) карьер “Малыш” 1 + 1 карьерy “Голубой” 20 + 20
Экспериментальные скрещивания В. В. Зюганова(1983) карьер “Малыш” 1 + 1 карьерy “Голубой” 20 + 20
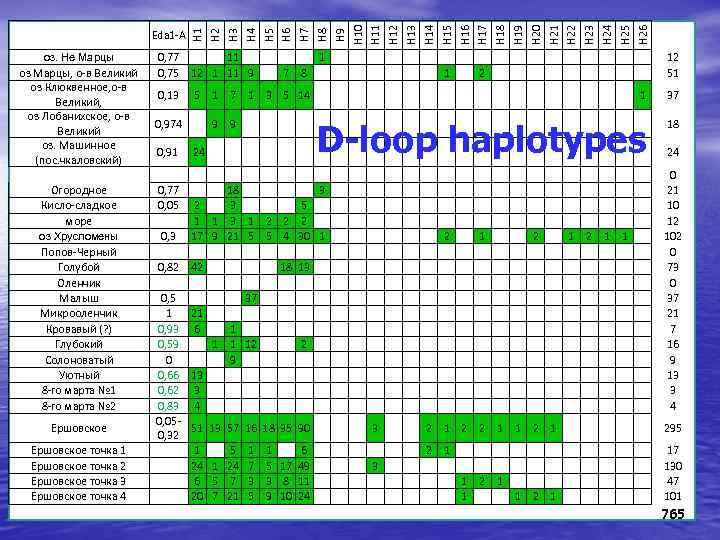 оз. Не Марцы оз Марцы, о-в Великий оз Клюквенное, о-в Великий, оз Лобанихское, о-в Великий оз. Машинное (пос. чкаловский) Огородное Кисло-сладкое море оз Хрусломены Попов-Черный Голубой Оленчик Малыш Микрооленчик Кровавый (? ) Глубокий Солоноватый Уютный 8 -го марта № 1 8 -го марта № 2 Ершовское точка 1 Ершовское точка 2 Ершовское точка 3 Ершовское точка 4 Eda 1 -A H 1 H 2 H 3 H 4 H 5 H 6 H 7 H 8 H 9 H 10 H 11 H 12 H 13 H 14 H 15 H 16 H 17 H 18 H 19 H 20 H 21 H 22 H 23 H 24 H 25 H 26 0, 77 11 0, 75 12 1 11 9 1 7 8 1 2 12 51 0, 13 5 1 7 1 3 5 14 1 37 0, 974 D-loop haplotypes 9 9 18 24 3 5 2 2 2 5 4 30 1 18 13 2 2 1 2 1 2 1 1 0 21 10 12 102 0 73 0 37 21 7 16 9 13 3 4 51 13 57 16 18 35 90 3 2 1 2 2 1 1 2 1 295 1 24 6 20 3 2 1 1 2 1 1 1 2 1 17 130 47 101 0, 91 24 0, 77 0, 05 0, 3 0, 82 0, 5 1 0, 93 0, 59 0 0, 66 0, 62 0, 83 0, 050, 32 2 1 17 42 21 6 13 3 4 1 9 1 1 5 7 18 3 3 21 1 1 9 1 5 37 12 5 24 7 21 1 7 3 5 1 6 5 17 49 3 8 11 9 10 24 765
оз. Не Марцы оз Марцы, о-в Великий оз Клюквенное, о-в Великий, оз Лобанихское, о-в Великий оз. Машинное (пос. чкаловский) Огородное Кисло-сладкое море оз Хрусломены Попов-Черный Голубой Оленчик Малыш Микрооленчик Кровавый (? ) Глубокий Солоноватый Уютный 8 -го марта № 1 8 -го марта № 2 Ершовское точка 1 Ершовское точка 2 Ершовское точка 3 Ершовское точка 4 Eda 1 -A H 1 H 2 H 3 H 4 H 5 H 6 H 7 H 8 H 9 H 10 H 11 H 12 H 13 H 14 H 15 H 16 H 17 H 18 H 19 H 20 H 21 H 22 H 23 H 24 H 25 H 26 0, 77 11 0, 75 12 1 11 9 1 7 8 1 2 12 51 0, 13 5 1 7 1 3 5 14 1 37 0, 974 D-loop haplotypes 9 9 18 24 3 5 2 2 2 5 4 30 1 18 13 2 2 1 2 1 2 1 1 0 21 10 12 102 0 73 0 37 21 7 16 9 13 3 4 51 13 57 16 18 35 90 3 2 1 2 2 1 1 2 1 295 1 24 6 20 3 2 1 1 2 1 1 1 2 1 17 130 47 101 0, 91 24 0, 77 0, 05 0, 3 0, 82 0, 5 1 0, 93 0, 59 0 0, 66 0, 62 0, 83 0, 050, 32 2 1 17 42 21 6 13 3 4 1 9 1 1 5 7 18 3 3 21 1 1 9 1 5 37 12 5 24 7 21 1 7 3 5 1 6 5 17 49 3 8 11 9 10 24 765
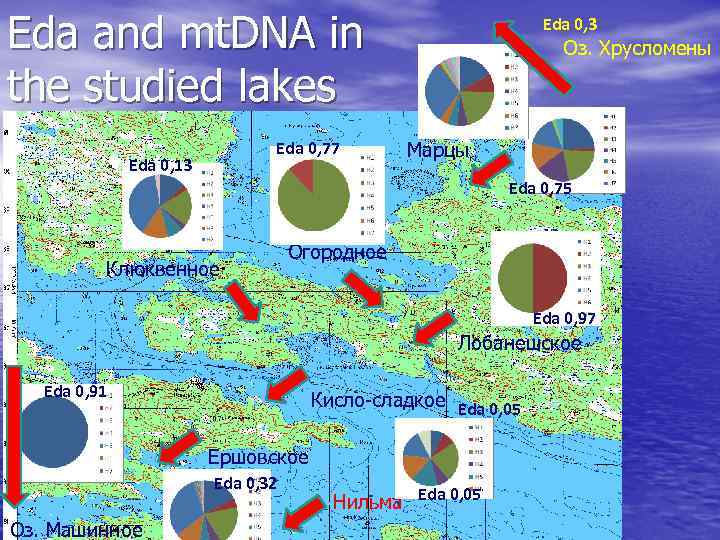 Eda and mt. DNA in the studied lakes Eda 0, 77 Eda 0, 13 Eda 0, 3 Оз. Хрусломены Марцы Eda 0, 75 Клюквенное Огородное 0, 05 Eda 0, 97 Лобанешское Eda 0, 91 Кисло-сладкое Eda 0, 05 Ершовское Eda 0, 32 Оз. Машинное Нильма Eda 0, 05
Eda and mt. DNA in the studied lakes Eda 0, 77 Eda 0, 13 Eda 0, 3 Оз. Хрусломены Марцы Eda 0, 75 Клюквенное Огородное 0, 05 Eda 0, 97 Лобанешское Eda 0, 91 Кисло-сладкое Eda 0, 05 Ершовское Eda 0, 32 Оз. Машинное Нильма Eda 0, 05
 ЗАДАЧИ ГЕНОМНОЙ ФАЗЫ ПРОЕКТА • Выявить геномные участки, ответственные за формирование пресноводных фор колюшки • Оценить естественный отбор в экспериментальных карьерах Зюганова и в молодых озерах • Изучить генетически основы адаптации
ЗАДАЧИ ГЕНОМНОЙ ФАЗЫ ПРОЕКТА • Выявить геномные участки, ответственные за формирование пресноводных фор колюшки • Оценить естественный отбор в экспериментальных карьерах Зюганова и в молодых озерах • Изучить генетически основы адаптации
 Полногеномное секвенирование Illumina Hi. Seq 2000 Location Nilma (Sea) Lake Mashinnoe Lake Lobaneshskoe Lake Martzi Lake Ershovskoe Quarry Goluboy Quarry Malysh N 16 10 8 10 12 20 20 Sea spec. from Erchovsoe 12 Lake Azabache (Kamchatka) 18 Kamchatka, Sea sample 16 Секвенирование – М. Логачева, Биоинформатика – Н. Тереханова Reads# 389. 034. 906 385. 531. 658 386. 779. 485 385. 781. 053 389. 937. 880 390. 536. 519
Полногеномное секвенирование Illumina Hi. Seq 2000 Location Nilma (Sea) Lake Mashinnoe Lake Lobaneshskoe Lake Martzi Lake Ershovskoe Quarry Goluboy Quarry Malysh N 16 10 8 10 12 20 20 Sea spec. from Erchovsoe 12 Lake Azabache (Kamchatka) 18 Kamchatka, Sea sample 16 Секвенирование – М. Логачева, Биоинформатика – Н. Тереханова Reads# 389. 034. 906 385. 531. 658 386. 779. 485 385. 781. 053 389. 937. 880 390. 536. 519

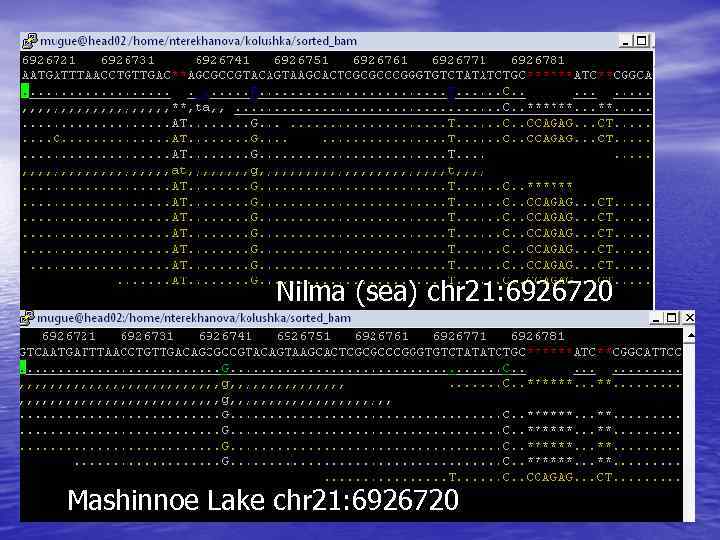 Nilma (sea) chr 21: 6926720 Mashinnoe Lake chr 21: 6926720
Nilma (sea) chr 21: 6926720 Mashinnoe Lake chr 21: 6926720
 Определения: • SNP as selective MARKER: – 0 -20% в двух морских выборках (Nilma, Sersh) – 80 -100% в двух старых пресноводных популяциях (о. Лобанешское, о. Машинное). – Покрытие не менее 5 ридов (20 for Nilma) • Soft MARKER - 50 -100% в озерах (с целью • выявления слабого или балансирующего отбора) CLUMP (сгусток, скопление - КЛАМП): непрерывный участок с >5 Markers в рамке т. н.
Определения: • SNP as selective MARKER: – 0 -20% в двух морских выборках (Nilma, Sersh) – 80 -100% в двух старых пресноводных популяциях (о. Лобанешское, о. Машинное). – Покрытие не менее 5 ридов (20 for Nilma) • Soft MARKER - 50 -100% в озерах (с целью • выявления слабого или балансирующего отбора) CLUMP (сгусток, скопление - КЛАМП): непрерывный участок с >5 Markers в рамке т. н.
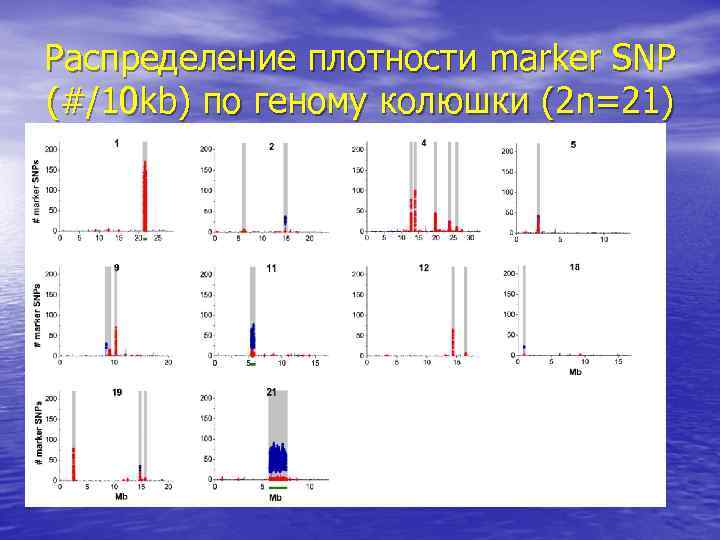 Распределение плотности marker SNP (#/10 kb) по геному колюшки (2 n=21)
Распределение плотности marker SNP (#/10 kb) по геному колюшки (2 n=21)
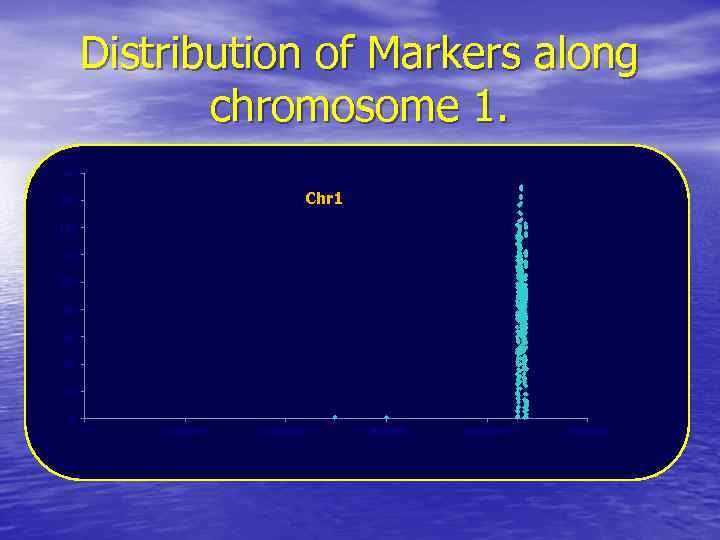 Distribution of Markers along chromosome 1. 180 Chr 1 160 140 120 100 80 60 40 20 0 0 5000000 10000000 15000000 20000000 25000000
Distribution of Markers along chromosome 1. 180 Chr 1 160 140 120 100 80 60 40 20 0 0 5000000 10000000 15000000 20000000 25000000
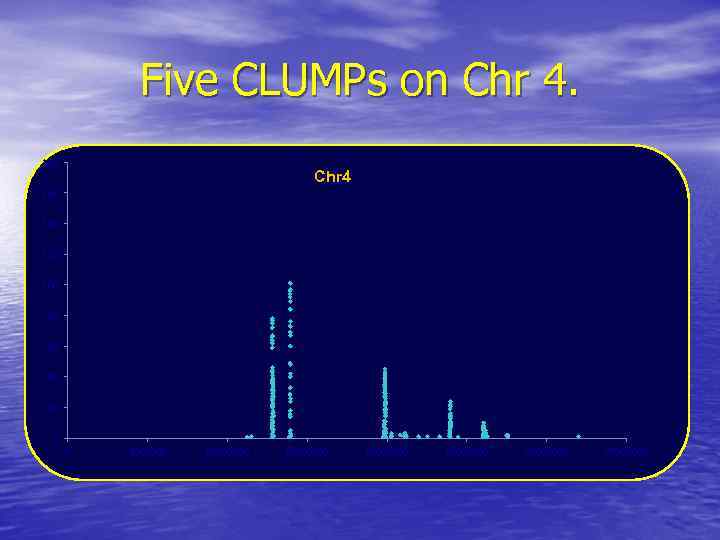 Five CLUMPs on Chr 4. 180 Chr 4 160 140 120 100 80 60 40 20 0 0 5000000 10000000 15000000 20000000 25000000 30000000 35000000
Five CLUMPs on Chr 4. 180 Chr 4 160 140 120 100 80 60 40 20 0 0 5000000 10000000 15000000 20000000 25000000 30000000 35000000
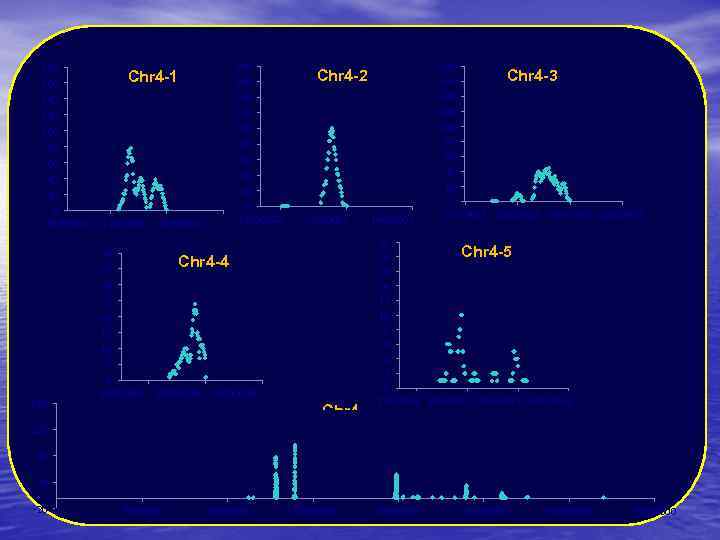 180 Chr 4 -1 160 180 Chr 4 -2 Chr 4 -3 160 140 140 120 120 100 100 80 80 80 60 60 60 40 40 20 20 0 12703801 12803801 0 13830002 12903801 40 40 20 13930002 30 25 20 15 10 5 180 0 23833246 23933246 20 18 16 14 12 10 8 6 4 2 0 25922621 26022621 26122621 26222621 Chr 4 -5 Chr 4 -4 35 14030002 0 19713419 19813419 19913419 20013419 24033246 Chr 4 130 80 30 -20 0 5000000 10000000 15000000 20000000 25000000 30000000 35000000
180 Chr 4 -1 160 180 Chr 4 -2 Chr 4 -3 160 140 140 120 120 100 100 80 80 80 60 60 60 40 40 20 20 0 12703801 12803801 0 13830002 12903801 40 40 20 13930002 30 25 20 15 10 5 180 0 23833246 23933246 20 18 16 14 12 10 8 6 4 2 0 25922621 26022621 26122621 26222621 Chr 4 -5 Chr 4 -4 35 14030002 0 19713419 19813419 19913419 20013419 24033246 Chr 4 130 80 30 -20 0 5000000 10000000 15000000 20000000 25000000 30000000 35000000
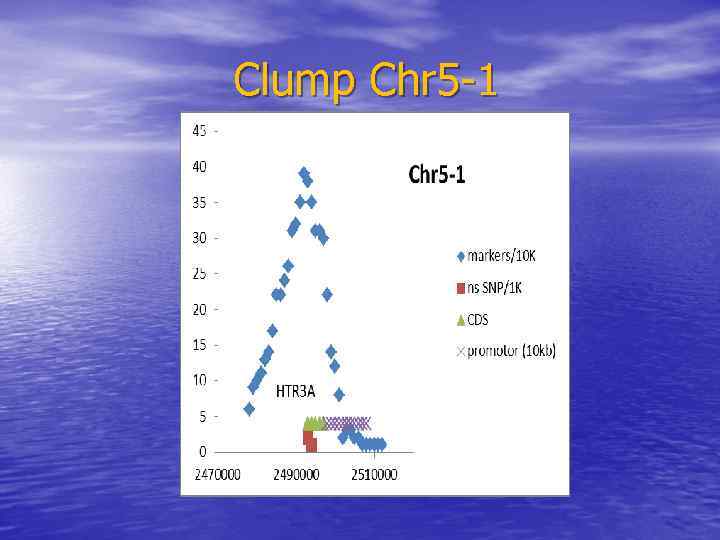 Clump Chr 5 -1
Clump Chr 5 -1
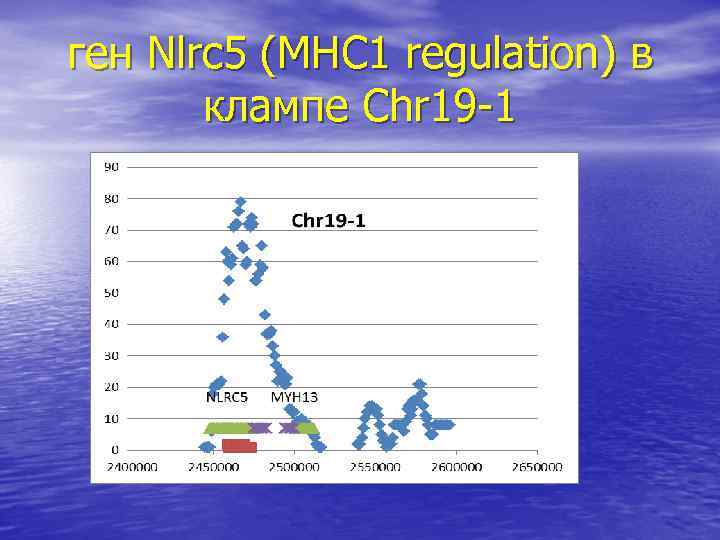 ген Nlrc 5 (MHC 1 regulation) в клампе Chr 19 -1
ген Nlrc 5 (MHC 1 regulation) в клампе Chr 19 -1
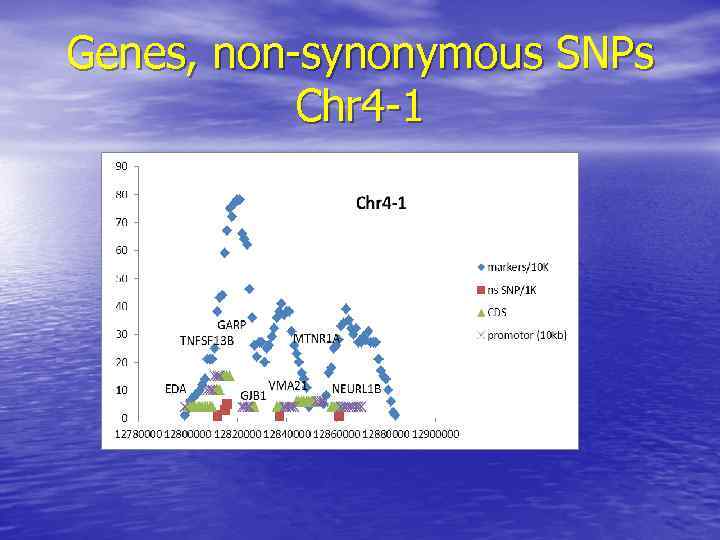 Genes, non-synonymous SNPs Chr 4 -1
Genes, non-synonymous SNPs Chr 4 -1
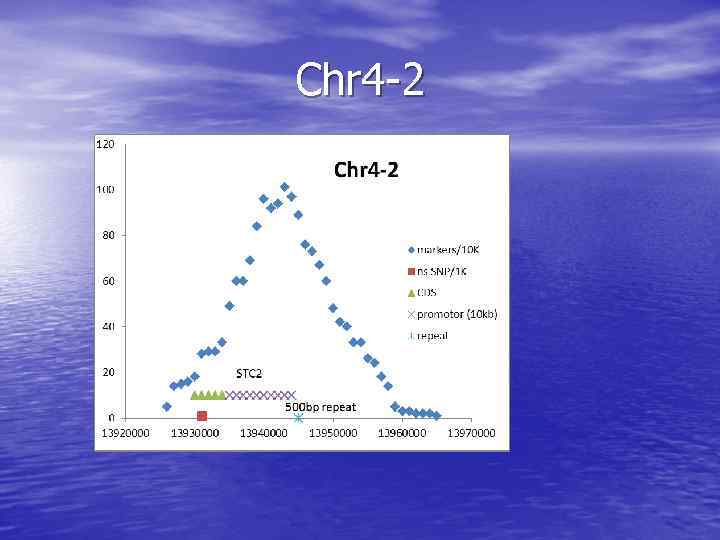 Chr 4 -2
Chr 4 -2
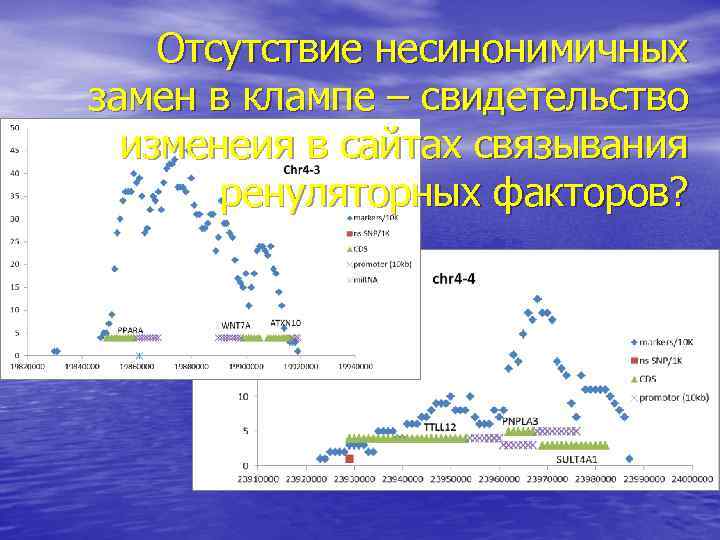 Отсутствие несинонимичных замен в клампе – свидетельство изменеия в сайтах связывания ренуляторных факторов? Many clumps with no NS SNPs
Отсутствие несинонимичных замен в клампе – свидетельство изменеия в сайтах связывания ренуляторных факторов? Many clumps with no NS SNPs

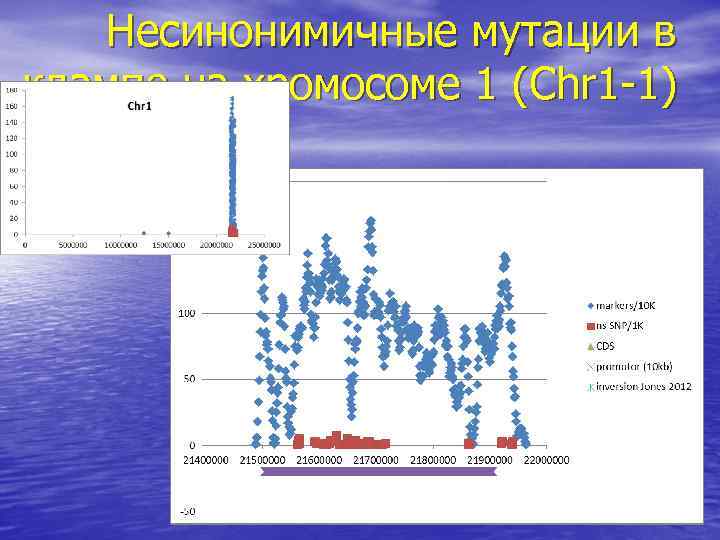 Несинонимичные мутации в клампе на хромосоме 1 (Chr 1 -1)
Несинонимичные мутации в клампе на хромосоме 1 (Chr 1 -1)
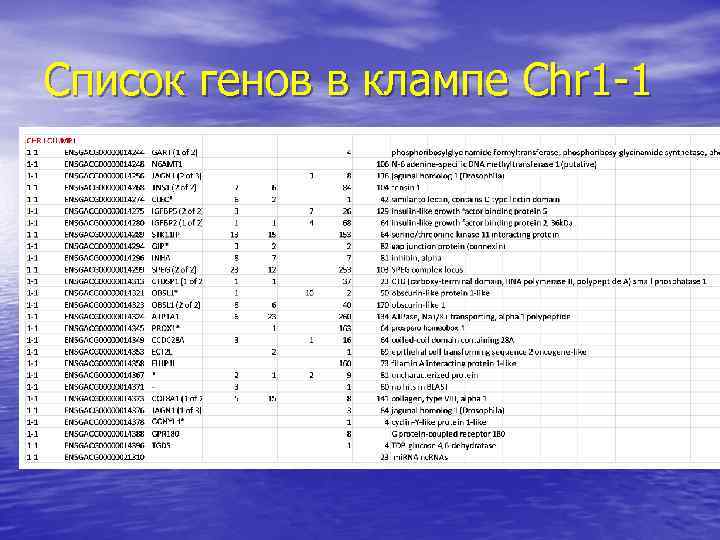 Список генов в клампе Chr 1 -1
Список генов в клампе Chr 1 -1
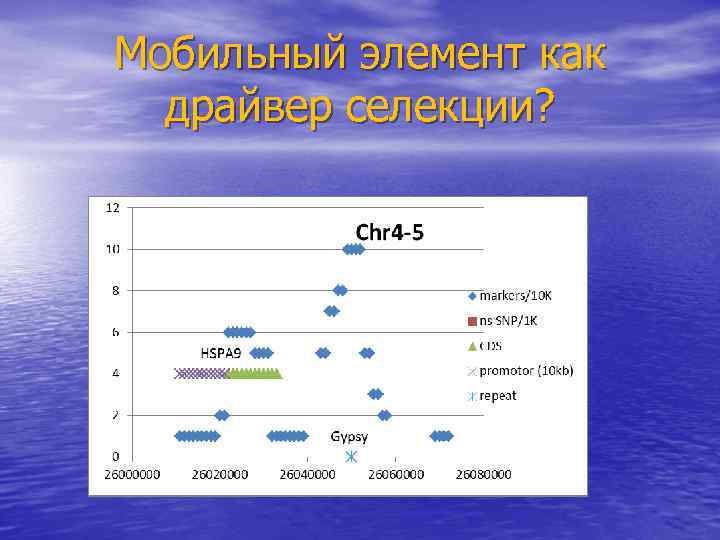 Мобильный элемент как драйвер селекции?
Мобильный элемент как драйвер селекции?
 Type value Candidate genes Comments inversion 100 27 genes inc. : ATP 1 A 1 pole 6 Mobile element peak 35 zfand 3 AN 1 -type zinc finger 3 -peaks 80 -35 -33 EDA+GJP 1, LRR 32, NEURL 1 B Chr 4 -2 peak 26262 94 STC 2 Osmos Chr 4 -3 100866 40 PPARA regulator of lipid metabolism Chr 4 -4 87586 5 MUC 19 Spiggin! Chr 4 -5 54792 22 PNPLA 3 adiponutrin Chr 4 -6 163077 7 HSPA 9 heat shock 70 k. Da protein Chr 5 -1 14546 39 HTR 3 A Serotonin receptor Chr 9 -1 13891 15 LRRTM 1 leucine-rich repeat transmembrane neuronal protein 1 precursor Chr 9 -2 33757 70 МЕТ, RPTOR HGF receptor, energy metabolism Chr 11 -1_>50 412587 inversion 40 21 genes inc. FZD 2, STAT 3, ATP 6 V 0 A 1 etc. Chr 12 -1 23289 61 PTDSS 1 CDP-diacylglycerol--serine Ophosphatidyltransferase chr 12 -2 16650 6 OVGP 1 mucin-9 Chr 19 -1 138325 80 NLRC 5 regulates MHC class I antigen presentation (intracellular pathogens ) Chr 19 -2 39345 18 Empty Chr 21 -1_>50 1722698 inversion 45 45 genes Chr 1 -1 Chr 2 -2_>50 Chr 4 -1 Span 471490 3559 48797 75854 Гены в клампах
Type value Candidate genes Comments inversion 100 27 genes inc. : ATP 1 A 1 pole 6 Mobile element peak 35 zfand 3 AN 1 -type zinc finger 3 -peaks 80 -35 -33 EDA+GJP 1, LRR 32, NEURL 1 B Chr 4 -2 peak 26262 94 STC 2 Osmos Chr 4 -3 100866 40 PPARA regulator of lipid metabolism Chr 4 -4 87586 5 MUC 19 Spiggin! Chr 4 -5 54792 22 PNPLA 3 adiponutrin Chr 4 -6 163077 7 HSPA 9 heat shock 70 k. Da protein Chr 5 -1 14546 39 HTR 3 A Serotonin receptor Chr 9 -1 13891 15 LRRTM 1 leucine-rich repeat transmembrane neuronal protein 1 precursor Chr 9 -2 33757 70 МЕТ, RPTOR HGF receptor, energy metabolism Chr 11 -1_>50 412587 inversion 40 21 genes inc. FZD 2, STAT 3, ATP 6 V 0 A 1 etc. Chr 12 -1 23289 61 PTDSS 1 CDP-diacylglycerol--serine Ophosphatidyltransferase chr 12 -2 16650 6 OVGP 1 mucin-9 Chr 19 -1 138325 80 NLRC 5 regulates MHC class I antigen presentation (intracellular pathogens ) Chr 19 -2 39345 18 Empty Chr 21 -1_>50 1722698 inversion 45 45 genes Chr 1 -1 Chr 2 -2_>50 Chr 4 -1 Span 471490 3559 48797 75854 Гены в клампах
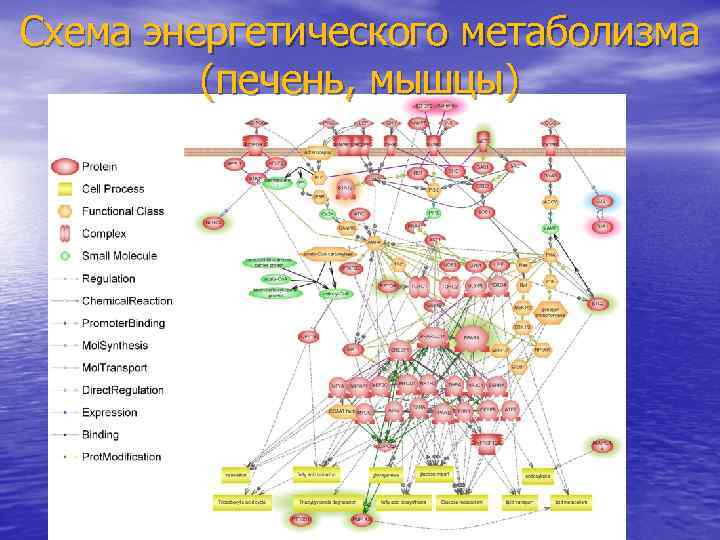 Схема энергетического метаболизма (печень, мышцы)
Схема энергетического метаболизма (печень, мышцы)

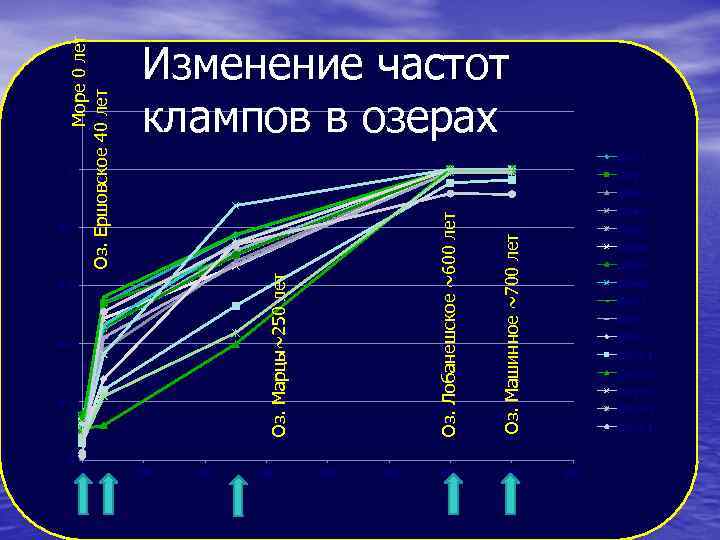 Море 0 лет Оз. Ершовское 40 лет 1. 2 Изменение частот клампов в озерах Chr 1 -1 1 Chr 2 -1 Chr 4 -1 Оз. Лобанешское ~600 лет 0. 4 0. 2 Оз. Машинное ~700 лет Оз. Марцы~250 лет 0. 8 0. 6 Chr 4 -2 600 700 Chr 4 -3 Chr 4 -4 Chr 4 -5 Chr 4 -6 Chr 5 -1 Chr 9 -2 Chr 11 -1 Chr 12 -1 Chr 19 -2 Chr 21 -1 0 0 100 200 300 400 500 800
Море 0 лет Оз. Ершовское 40 лет 1. 2 Изменение частот клампов в озерах Chr 1 -1 1 Chr 2 -1 Chr 4 -1 Оз. Лобанешское ~600 лет 0. 4 0. 2 Оз. Машинное ~700 лет Оз. Марцы~250 лет 0. 8 0. 6 Chr 4 -2 600 700 Chr 4 -3 Chr 4 -4 Chr 4 -5 Chr 4 -6 Chr 5 -1 Chr 9 -2 Chr 11 -1 Chr 12 -1 Chr 19 -2 Chr 21 -1 0 0 100 200 300 400 500 800
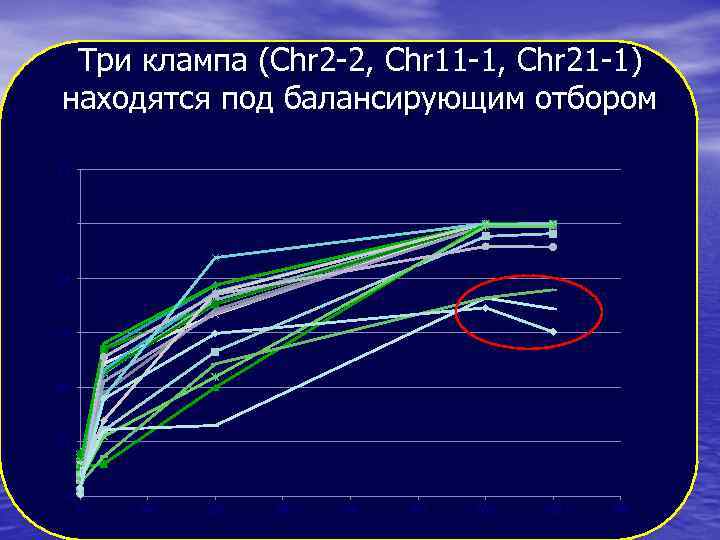 Три клампа (Chr 2 -2, Chr 11 -1, Chr 21 -1) находятся под балансирующим отбором 1. 2 1 0. 8 0. 6 0. 4 0. 2 0 0 100 200 300 400 500 600 700 800
Три клампа (Chr 2 -2, Chr 11 -1, Chr 21 -1) находятся под балансирующим отбором 1. 2 1 0. 8 0. 6 0. 4 0. 2 0 0 100 200 300 400 500 600 700 800
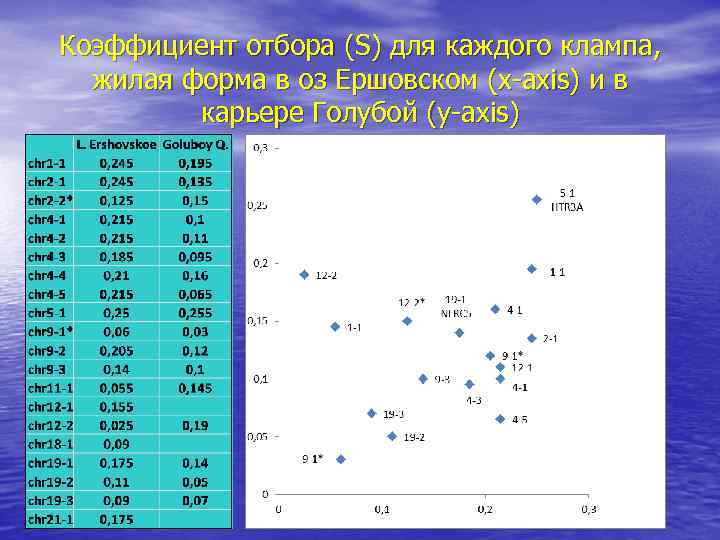 Коэффициент отбора (S) для каждого клампа, жилая форма в оз Ершовском (x-axis) и в карьере Голубой (y-axis)
Коэффициент отбора (S) для каждого клампа, жилая форма в оз Ершовском (x-axis) и в карьере Голубой (y-axis)
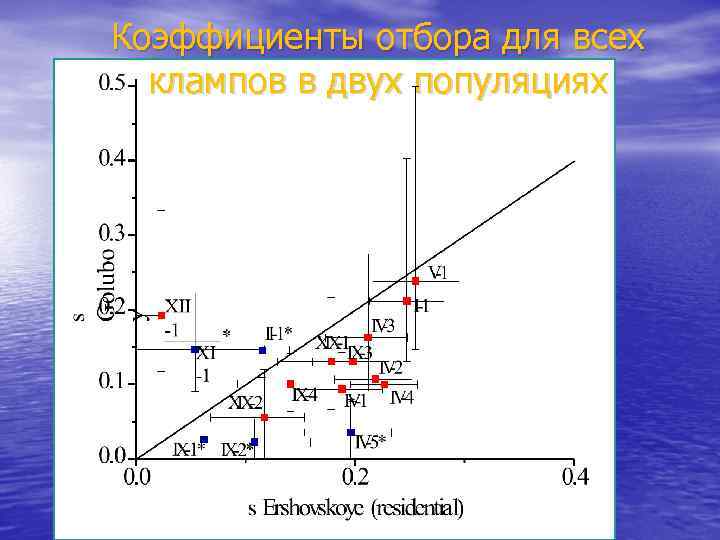 Коэффициенты отбора для всех клампов в двух популяциях
Коэффициенты отбора для всех клампов в двух популяциях
 Как формируются клампы? Only young selective sweeps can usually be detected. time But fresh- and saltwater adaptive genomic regions accumulate neutral mutations separately and rarely meet and recombine during “sea phase” SEA LAKE
Как формируются клампы? Only young selective sweeps can usually be detected. time But fresh- and saltwater adaptive genomic regions accumulate neutral mutations separately and rarely meet and recombine during “sea phase” SEA LAKE
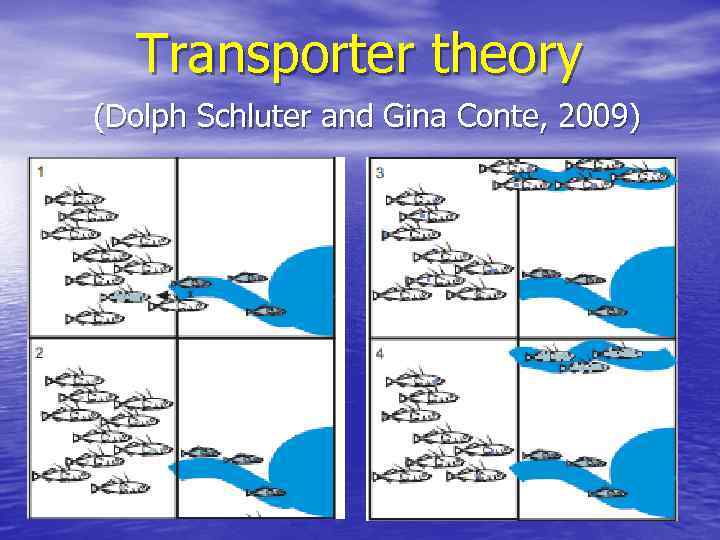 Transporter theory (Dolph Schluter and Gina Conte, 2009)
Transporter theory (Dolph Schluter and Gina Conte, 2009)
 Design of allele-specific primers and genotyping of individuals for all adaptive loci (clumps) in large set of lakes.
Design of allele-specific primers and genotyping of individuals for all adaptive loci (clumps) in large set of lakes.
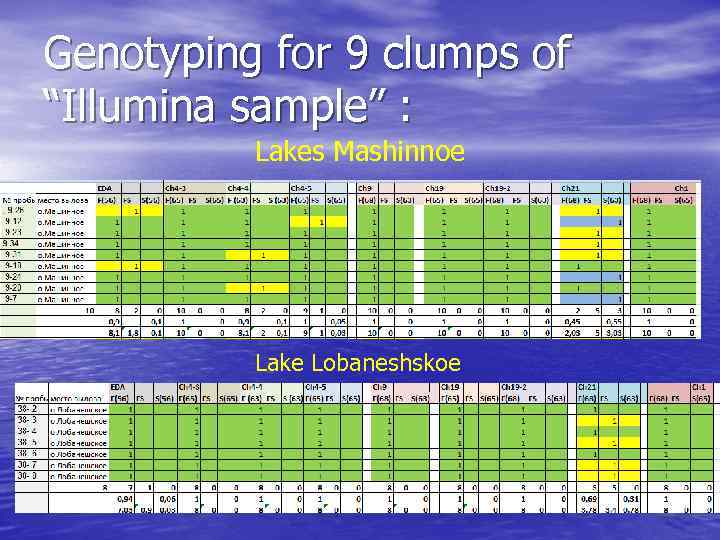 Genotyping for 9 clumps of “Illumina sample” : Lakes Mashinnoe Lake Lobaneshskoe
Genotyping for 9 clumps of “Illumina sample” : Lakes Mashinnoe Lake Lobaneshskoe
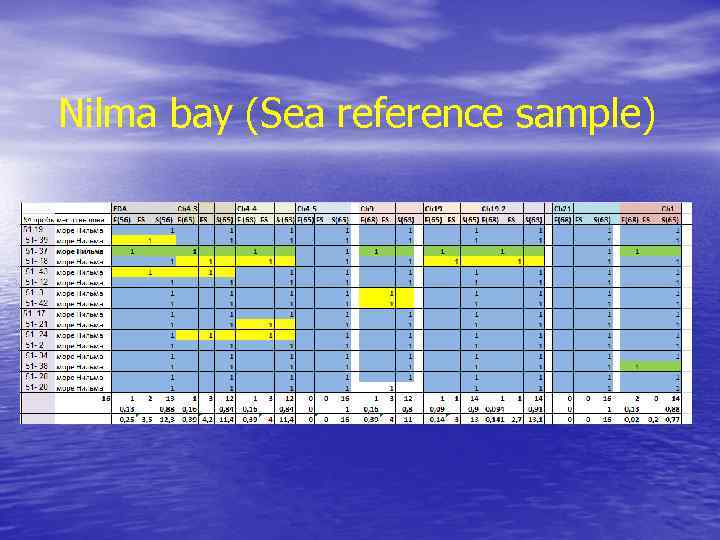 Nilma bay (Sea reference sample)
Nilma bay (Sea reference sample)
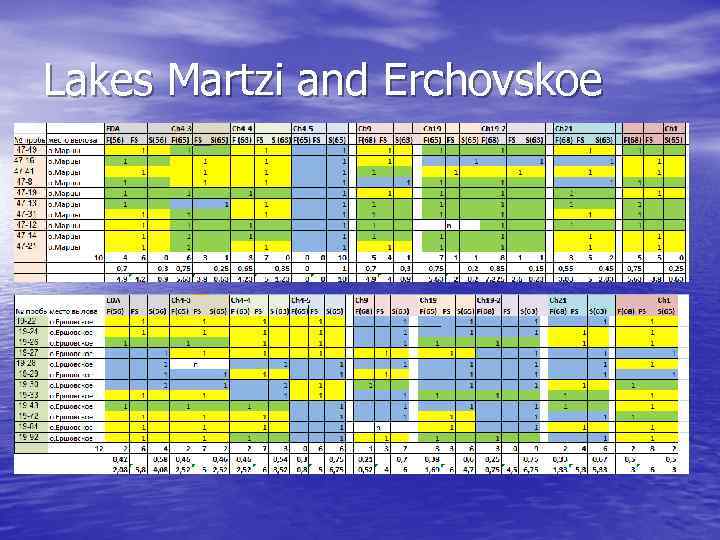 Lakes Martzi and Erchovskoe
Lakes Martzi and Erchovskoe
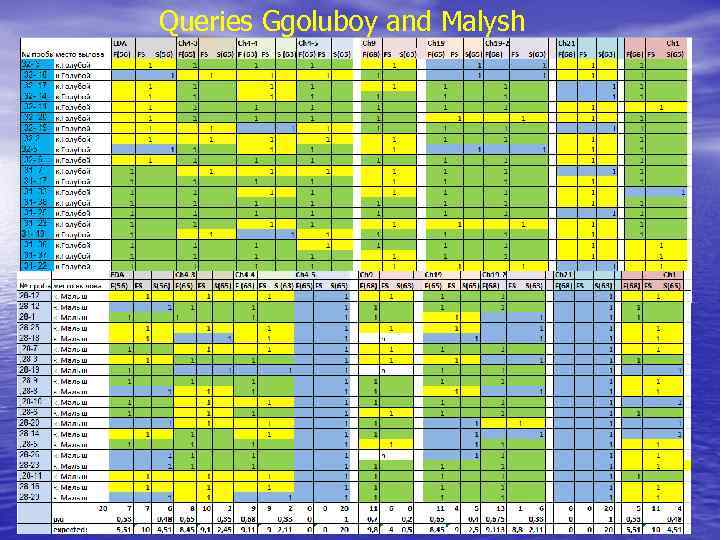 Queries Ggoluboy and Malysh
Queries Ggoluboy and Malysh
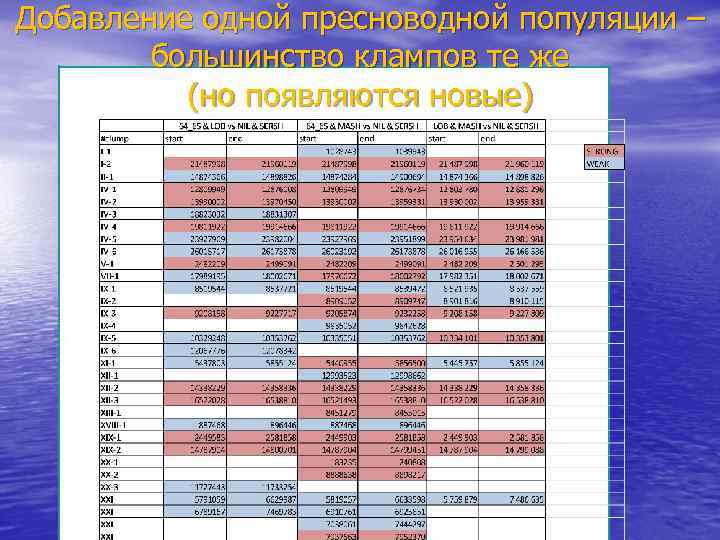 Добавление одной пресноводной популяции – большинство клампов те же (но появляются новые)
Добавление одной пресноводной популяции – большинство клампов те же (но появляются новые)
 Есть ли между формами колюшки репродуктивная изоляция?
Есть ли между формами колюшки репродуктивная изоляция?
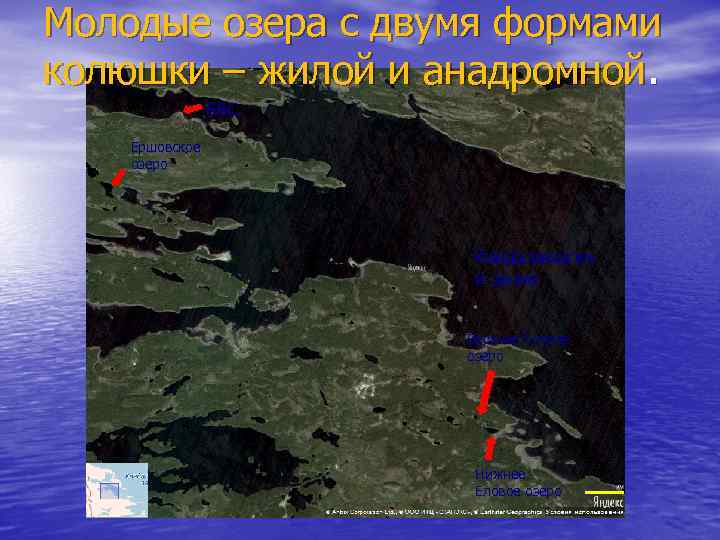 Молодые озера с двумя формами колюшки – жилой и анадромной. ББС Ершовское озеро Кандалакшски й залив Верхнее Еловое озеро Нижнее Еловое озеро
Молодые озера с двумя формами колюшки – жилой и анадромной. ББС Ершовское озеро Кандалакшски й залив Верхнее Еловое озеро Нижнее Еловое озеро
 Кламп-специфичные праймеры. Название Stn 382_F Stn 382_R Ch 1_FS_F Ch 1_F_R Ch 1_S_Rnew Ch 4 -1 -1 -FF Ch 4 -1 -1 -SF Ch 4 -1 -1 -FSR Ch 4 -6 n_S_R Ch 4 -6 n_F_R Ch 4 -6 n. FS_F Ch 5 -1 -FSR Ch 5 -1 -SF Chr 9_F_F Chr 9_FS_R Chr 9_S_F Chr 21_FS_R Chr 21_S_F Последовательность 5` CCCTTAGAGAATTTCCTAGCAG CTTGTCCCGGATCATACGC AGGCCTTTTCACAGGCAGCCTC AGTCACTTACAAAACCAAGAAGCTTTC TCAACTTACAAAACCAAGAAGCGG GACCGATCAACACAGCAGATGA GACCGATCAACACAGCAGATAC AGGCGGTTGCCAGATATGTT GATTTGCAAAACACTGTGTGTAA GATTTGCAAAATACTGTGTTGAG TGGTGCTGTTGGTCTGAA GAGGTTCTCACAAGTCACCAAAAAG AACCAATGCTGACACGTCCT GGTTCTCATAAGTCACCACACC AGCGCTTCCAAAGGAGTTCTGAG TGTGCCAACCCGCCATCATA CAGCGCTTCCAAAGGAGTTCTGTA CAATGATTTAACCTGTTGACAGC CAGTAATGGTGGGTCAGTGTACGT GTCAATGATTTAACCTGTTGACATA 3`
Кламп-специфичные праймеры. Название Stn 382_F Stn 382_R Ch 1_FS_F Ch 1_F_R Ch 1_S_Rnew Ch 4 -1 -1 -FF Ch 4 -1 -1 -SF Ch 4 -1 -1 -FSR Ch 4 -6 n_S_R Ch 4 -6 n_F_R Ch 4 -6 n. FS_F Ch 5 -1 -FSR Ch 5 -1 -SF Chr 9_F_F Chr 9_FS_R Chr 9_S_F Chr 21_FS_R Chr 21_S_F Последовательность 5` CCCTTAGAGAATTTCCTAGCAG CTTGTCCCGGATCATACGC AGGCCTTTTCACAGGCAGCCTC AGTCACTTACAAAACCAAGAAGCTTTC TCAACTTACAAAACCAAGAAGCGG GACCGATCAACACAGCAGATGA GACCGATCAACACAGCAGATAC AGGCGGTTGCCAGATATGTT GATTTGCAAAACACTGTGTGTAA GATTTGCAAAATACTGTGTTGAG TGGTGCTGTTGGTCTGAA GAGGTTCTCACAAGTCACCAAAAAG AACCAATGCTGACACGTCCT GGTTCTCATAAGTCACCACACC AGCGCTTCCAAAGGAGTTCTGAG TGTGCCAACCCGCCATCATA CAGCGCTTCCAAAGGAGTTCTGTA CAATGATTTAACCTGTTGACAGC CAGTAATGGTGGGTCAGTGTACGT GTCAATGATTTAACCTGTTGACATA 3`
 Пример агарозного фореза с локусом EDA (локус Stn 382, участок генома 4 -1) • Ген Ectodisplasin (EDA) отвечает за формирование кожных структур (в частности, ногтей) у человека, у колюшки отвечает за число боковых пластин.
Пример агарозного фореза с локусом EDA (локус Stn 382, участок генома 4 -1) • Ген Ectodisplasin (EDA) отвечает за формирование кожных структур (в частности, ногтей) у человека, у колюшки отвечает за число боковых пластин.
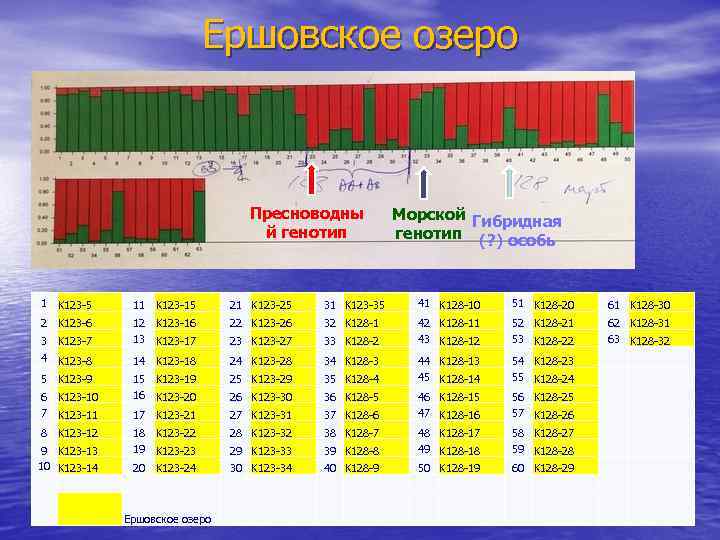 Ершовское озеро Пресный аллель Пресноводны й генотип Гетрозигот а Морской Гибридная аллель генотип (? ) особь 1 K 123 -5 11 K 123 -15 21 K 123 -25 31 K 123 -35 41 K 128 -10 51 K 128 -20 61 K 128 -30 2 K 123 -6 22 K 123 -26 32 K 128 -1 3 K 123 -7 12 K 123 -16 13 K 123 -17 23 K 123 -27 33 K 128 -2 42 K 128 -11 43 K 128 -12 52 K 128 -21 53 K 128 -22 62 K 128 -31 63 K 128 -32 4 K 123 -8 14 K 123 -18 24 K 123 -28 34 K 128 -3 5 K 123 -9 15 K 123 -19 16 K 123 -20 25 K 123 -29 35 K 128 -4 44 K 128 -13 45 K 128 -14 54 K 128 -23 55 K 128 -24 26 K 123 -30 36 K 128 -5 17 K 123 -21 27 K 123 -31 37 K 128 -6 46 K 128 -15 47 K 128 -16 56 K 128 -25 57 K 128 -26 18 K 123 -22 19 K 123 -23 28 K 123 -32 38 K 128 -7 29 K 123 -33 39 K 128 -8 48 K 128 -17 49 K 128 -18 58 K 128 -27 59 K 128 -28 20 K 123 -24 30 K 123 -34 40 K 128 -9 50 K 128 -19 60 K 128 -29 6 K 123 -10 7 K 123 -11 8 K 123 -12 9 K 123 -13 10 K 123 -14 Ершовское озеро
Ершовское озеро Пресный аллель Пресноводны й генотип Гетрозигот а Морской Гибридная аллель генотип (? ) особь 1 K 123 -5 11 K 123 -15 21 K 123 -25 31 K 123 -35 41 K 128 -10 51 K 128 -20 61 K 128 -30 2 K 123 -6 22 K 123 -26 32 K 128 -1 3 K 123 -7 12 K 123 -16 13 K 123 -17 23 K 123 -27 33 K 128 -2 42 K 128 -11 43 K 128 -12 52 K 128 -21 53 K 128 -22 62 K 128 -31 63 K 128 -32 4 K 123 -8 14 K 123 -18 24 K 123 -28 34 K 128 -3 5 K 123 -9 15 K 123 -19 16 K 123 -20 25 K 123 -29 35 K 128 -4 44 K 128 -13 45 K 128 -14 54 K 128 -23 55 K 128 -24 26 K 123 -30 36 K 128 -5 17 K 123 -21 27 K 123 -31 37 K 128 -6 46 K 128 -15 47 K 128 -16 56 K 128 -25 57 K 128 -26 18 K 123 -22 19 K 123 -23 28 K 123 -32 38 K 128 -7 29 K 123 -33 39 K 128 -8 48 K 128 -17 49 K 128 -18 58 K 128 -27 59 K 128 -28 20 K 123 -24 30 K 123 -34 40 K 128 -9 50 K 128 -19 60 K 128 -29 6 K 123 -10 7 K 123 -11 8 K 123 -12 9 K 123 -13 10 K 123 -14 Ершовское озеро
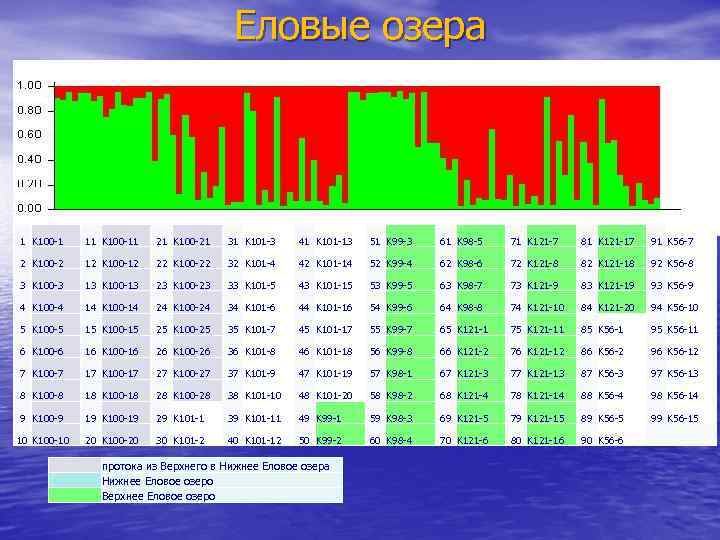 Еловые озера 1 K 100 -1 11 K 100 -11 21 K 100 -21 31 K 101 -3 41 K 101 -13 51 K 99 -3 61 K 98 -5 71 K 121 -7 81 K 121 -17 91 K 56 -7 2 K 100 -2 12 K 100 -12 22 K 100 -22 32 K 101 -4 42 K 101 -14 52 K 99 -4 62 K 98 -6 72 K 121 -8 82 K 121 -18 92 K 56 -8 3 K 100 -3 13 K 100 -13 23 K 100 -23 33 K 101 -5 43 K 101 -15 53 K 99 -5 63 K 98 -7 73 K 121 -9 83 K 121 -19 93 K 56 -9 4 K 100 -4 14 K 100 -14 24 K 100 -24 34 K 101 -6 44 K 101 -16 54 K 99 -6 64 K 98 -8 74 K 121 -10 84 K 121 -20 94 K 56 -10 5 K 100 -5 15 K 100 -15 25 K 100 -25 35 K 101 -7 45 K 101 -17 55 K 99 -7 65 K 121 -1 75 K 121 -11 85 K 56 -1 95 K 56 -11 6 K 100 -6 16 K 100 -16 26 K 100 -26 36 K 101 -8 46 K 101 -18 56 K 99 -8 66 K 121 -2 76 K 121 -12 86 K 56 -2 96 K 56 -12 7 K 100 -7 17 K 100 -17 27 K 100 -27 37 K 101 -9 47 K 101 -19 57 K 98 -1 67 K 121 -3 77 K 121 -13 87 K 56 -3 97 K 56 -13 8 K 100 -8 18 K 100 -18 28 K 100 -28 38 K 101 -10 48 K 101 -20 58 K 98 -2 68 K 121 -4 78 K 121 -14 88 K 56 -4 98 K 56 -14 9 K 100 -9 19 K 100 -19 29 K 101 -1 39 K 101 -11 49 K 99 -1 59 K 98 -3 69 K 121 -5 79 K 121 -15 89 K 56 -5 99 K 56 -15 10 K 100 -10 20 K 100 -20 30 K 101 -2 40 K 101 -12 50 K 99 -2 60 K 98 -4 70 K 121 -6 80 K 121 -16 90 K 56 -6 протока из Верхнего в Нижнее Еловое озера Нижнее Еловое озеро Верхнее Еловое озеро
Еловые озера 1 K 100 -1 11 K 100 -11 21 K 100 -21 31 K 101 -3 41 K 101 -13 51 K 99 -3 61 K 98 -5 71 K 121 -7 81 K 121 -17 91 K 56 -7 2 K 100 -2 12 K 100 -12 22 K 100 -22 32 K 101 -4 42 K 101 -14 52 K 99 -4 62 K 98 -6 72 K 121 -8 82 K 121 -18 92 K 56 -8 3 K 100 -3 13 K 100 -13 23 K 100 -23 33 K 101 -5 43 K 101 -15 53 K 99 -5 63 K 98 -7 73 K 121 -9 83 K 121 -19 93 K 56 -9 4 K 100 -4 14 K 100 -14 24 K 100 -24 34 K 101 -6 44 K 101 -16 54 K 99 -6 64 K 98 -8 74 K 121 -10 84 K 121 -20 94 K 56 -10 5 K 100 -5 15 K 100 -15 25 K 100 -25 35 K 101 -7 45 K 101 -17 55 K 99 -7 65 K 121 -1 75 K 121 -11 85 K 56 -1 95 K 56 -11 6 K 100 -6 16 K 100 -16 26 K 100 -26 36 K 101 -8 46 K 101 -18 56 K 99 -8 66 K 121 -2 76 K 121 -12 86 K 56 -2 96 K 56 -12 7 K 100 -7 17 K 100 -17 27 K 100 -27 37 K 101 -9 47 K 101 -19 57 K 98 -1 67 K 121 -3 77 K 121 -13 87 K 56 -3 97 K 56 -13 8 K 100 -8 18 K 100 -18 28 K 100 -28 38 K 101 -10 48 K 101 -20 58 K 98 -2 68 K 121 -4 78 K 121 -14 88 K 56 -4 98 K 56 -14 9 K 100 -9 19 K 100 -19 29 K 101 -1 39 K 101 -11 49 K 99 -1 59 K 98 -3 69 K 121 -5 79 K 121 -15 89 K 56 -5 99 K 56 -15 10 K 100 -10 20 K 100 -20 30 K 101 -2 40 K 101 -12 50 K 99 -2 60 K 98 -4 70 K 121 -6 80 K 121 -16 90 K 56 -6 протока из Верхнего в Нижнее Еловое озера Нижнее Еловое озеро Верхнее Еловое озеро
 Генотипирование самцов на гнезде и потомства в оз Ершовском
Генотипирование самцов на гнезде и потомства в оз Ершовском
 Генотипирование самцов на гнезде и потомства в оз Ершовском При выборе полового партнера самки преимущественно выбирают самца той же формы – свидетельство наличия ассортативного скрещивания.
Генотипирование самцов на гнезде и потомства в оз Ершовском При выборе полового партнера самки преимущественно выбирают самца той же формы – свидетельство наличия ассортативного скрещивания.
 Выводы: • 1. Адаптация на геномном уровне – это не один • • • локус, а набор несцепленных локусов 2. При адаптации к новым условиям, отбор идет по существующим минорным аллелям, присутствующим в исходной популяции, а не за счет новых мутаций 3. Единицей отбора является не хромосома и не SNP, а участок хромосомы. Часто сцепление генов обеспечивается инверсией. 4. Репродуктивная изоляция выгодна только при существенном потоке генов между соседними адаптивными ландшафтами и приводит к ускорению фиксации по адаптивным аллелям.
Выводы: • 1. Адаптация на геномном уровне – это не один • • • локус, а набор несцепленных локусов 2. При адаптации к новым условиям, отбор идет по существующим минорным аллелям, присутствующим в исходной популяции, а не за счет новых мутаций 3. Единицей отбора является не хромосома и не SNP, а участок хромосомы. Часто сцепление генов обеспечивается инверсией. 4. Репродуктивная изоляция выгодна только при существенном потоке генов между соседними адаптивными ландшафтами и приводит к ускорению фиксации по адаптивным аллелям.
 Что получилось: • Найдено 20 регионов, находящийся под сильным положительным отбором • Выявлено около 100 генов в этих зонах • Показано несколько генов, с большой вероятностью являющиеся драйверами отбора • Показана роль генов энергетического обмена
Что получилось: • Найдено 20 регионов, находящийся под сильным положительным отбором • Выявлено около 100 генов в этих зонах • Показано несколько генов, с большой вероятностью являющиеся драйверами отбора • Показана роль генов энергетического обмена
 Что не получилось • Не приблизились к выявлению механизмов репродуктивной изоляции • Для трети районов не найдено потенциальных субъектов отбора • Большинство генов находятся в инверсиях – нет возможности картирования гена под отбором • Выявленный набор генов – минимальный общий или достаточный?
Что не получилось • Не приблизились к выявлению механизмов репродуктивной изоляции • Для трети районов не найдено потенциальных субъектов отбора • Большинство генов находятся в инверсиях – нет возможности картирования гена под отбором • Выявленный набор генов – минимальный общий или достаточный?
 • Plosgenetics. org
• Plosgenetics. org
 • Plosgenetics. org
• Plosgenetics. org
 В работе принимали участие: Мария Логачева, ФББ МГУ Надя Тереханова, ФББ МГУ, ИППИ РАН
В работе принимали участие: Мария Логачева, ФББ МГУ Надя Тереханова, ФББ МГУ, ИППИ РАН
 Алексей Кондрашов
Алексей Кондрашов
 Татьяна Неретина, ББС МГУ Анна Барминцева, ВНИРО
Татьяна Неретина, ББС МГУ Анна Барминцева, ВНИРО
 Студенты летних практик (каф. зоологии б/п, иммунологии, ф-т Биоинформатики и биоинженерии, международных летних школ по молекулярной зоологии на ББС МГУ
Студенты летних практик (каф. зоологии б/п, иммунологии, ф-т Биоинформатики и биоинженерии, международных летних школ по молекулярной зоологии на ББС МГУ
 Лаборатория молекулярной генетики ВНИРО
Лаборатория молекулярной генетики ВНИРО
 БЛАГОДАРЮ ЗА ВНИМАНИЕ!
БЛАГОДАРЮ ЗА ВНИМАНИЕ!


