Эпигенетика Эпигенетические метки – митотически наследуемые






epigenetika.pptx
- Размер: 12.1 Мб
- Автор:
- Количество слайдов: 53
Описание презентации Эпигенетика Эпигенетические метки – митотически наследуемые по слайдам
 Эпигенетика
Эпигенетика
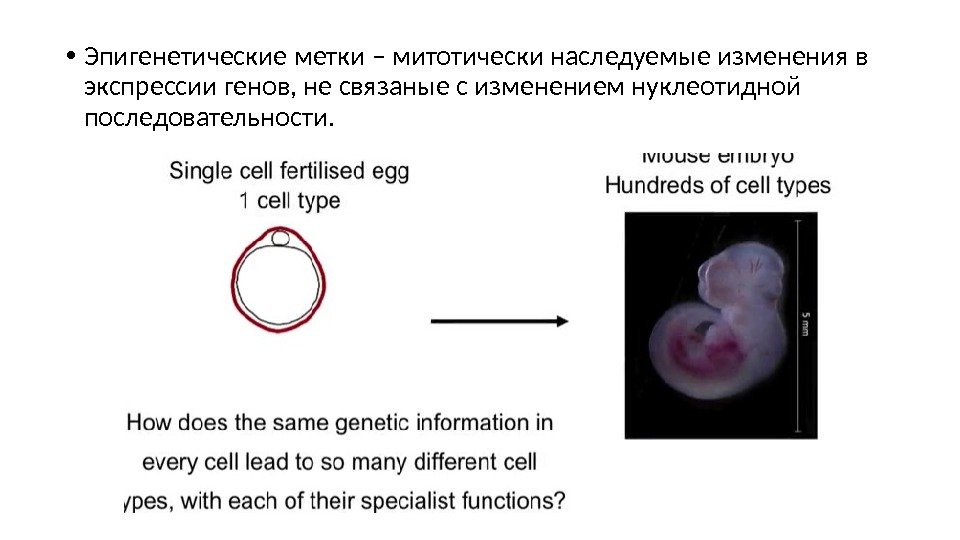
 • Эпигенетические метки – митотически наследуемые изменения в экспрессии генов, не связаные с изменением нуклеотидной последовательности.
• Эпигенетические метки – митотически наследуемые изменения в экспрессии генов, не связаные с изменением нуклеотидной последовательности.
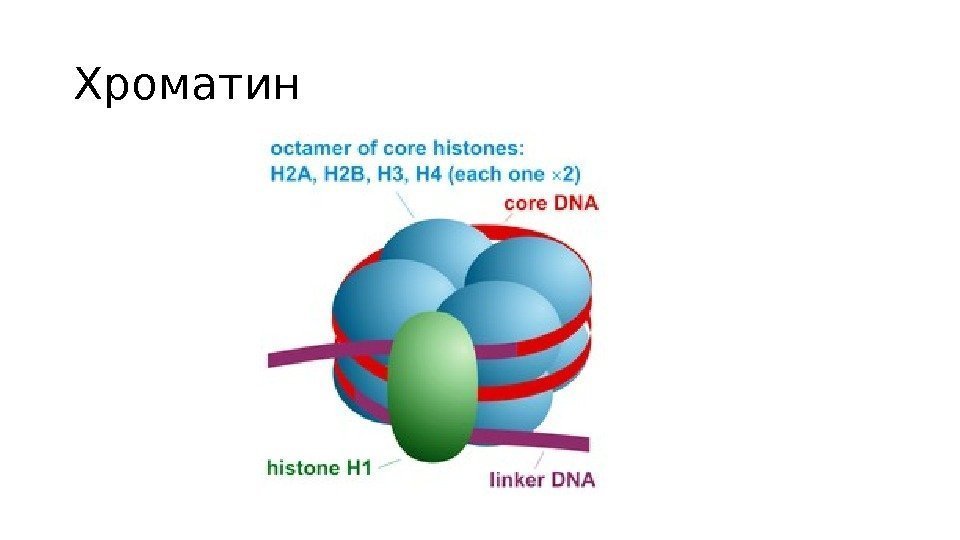
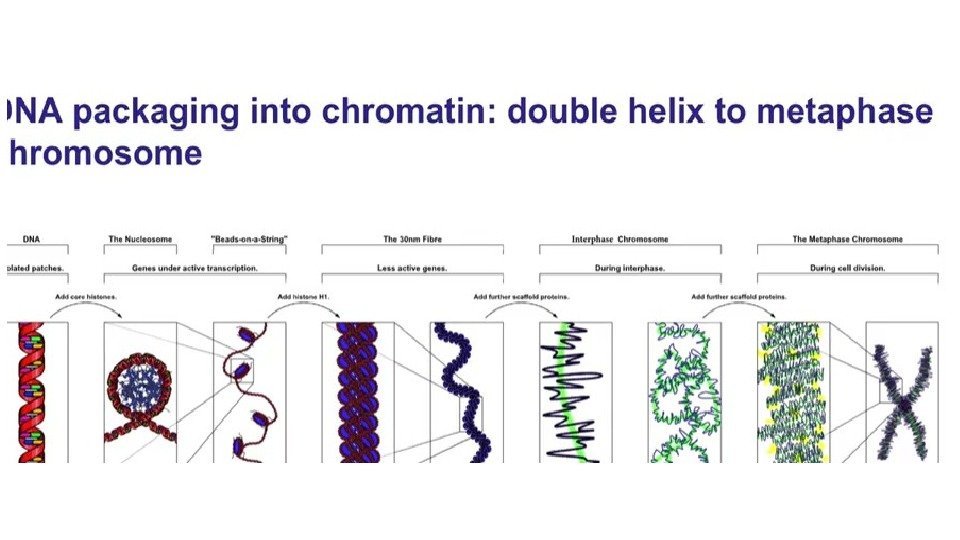
 Хроматин
Хроматин

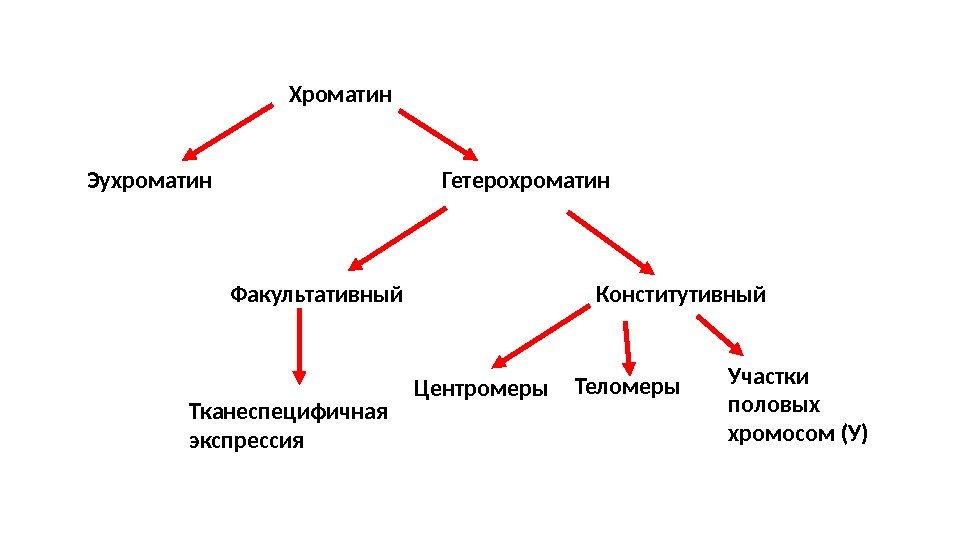
 Эухроматин Гетерохроматин. Хроматин Факультативный Конститутивный Центромеры Теломеры Участки половых хромосом (У)Тканеспецифичная экспрессия
Эухроматин Гетерохроматин. Хроматин Факультативный Конститутивный Центромеры Теломеры Участки половых хромосом (У)Тканеспецифичная экспрессия


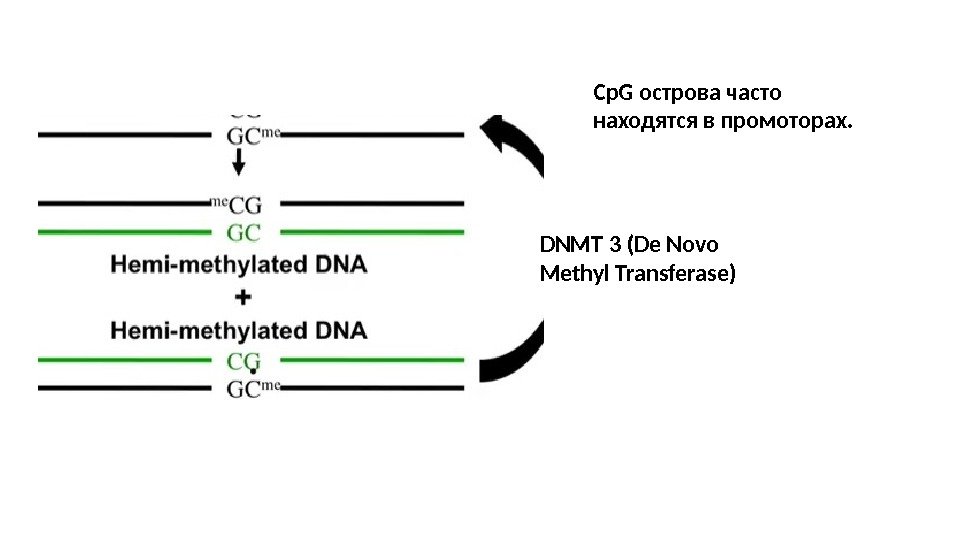
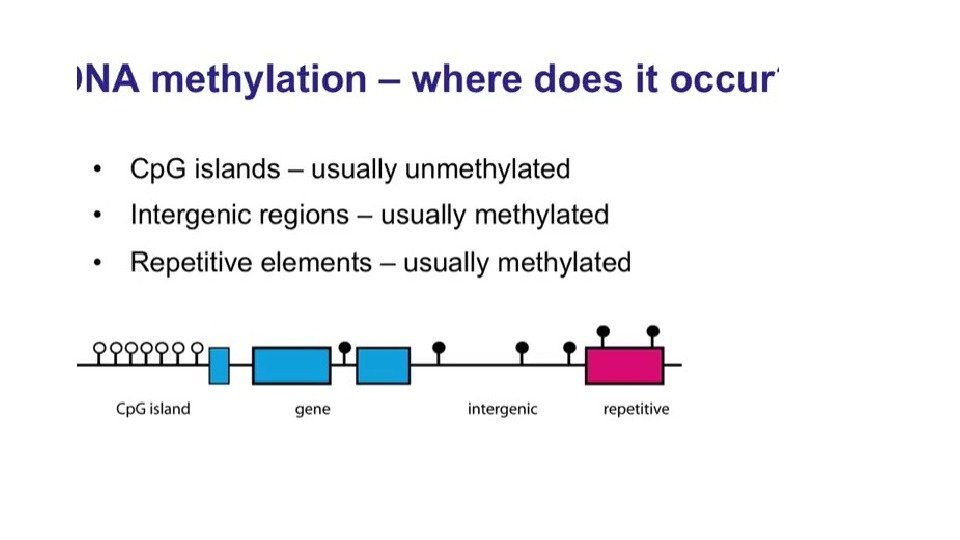
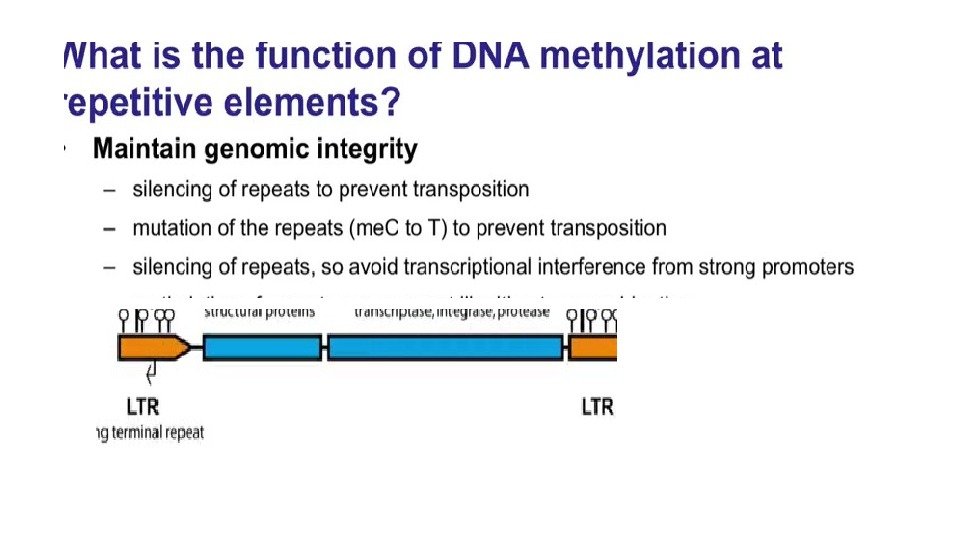
 DNMT 3 (De Novo Methyl Transferase) Cp. G острова часто находятся в промоторах.
DNMT 3 (De Novo Methyl Transferase) Cp. G острова часто находятся в промоторах.



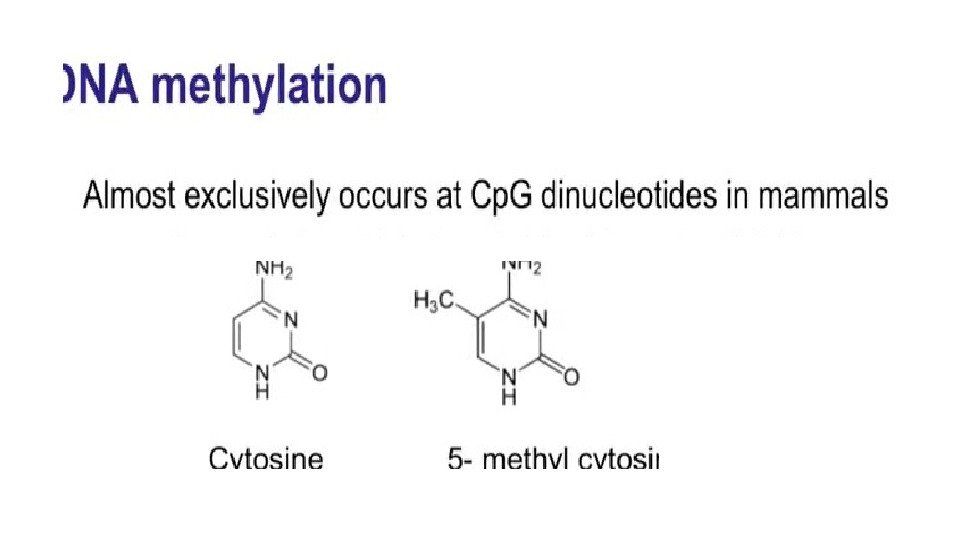
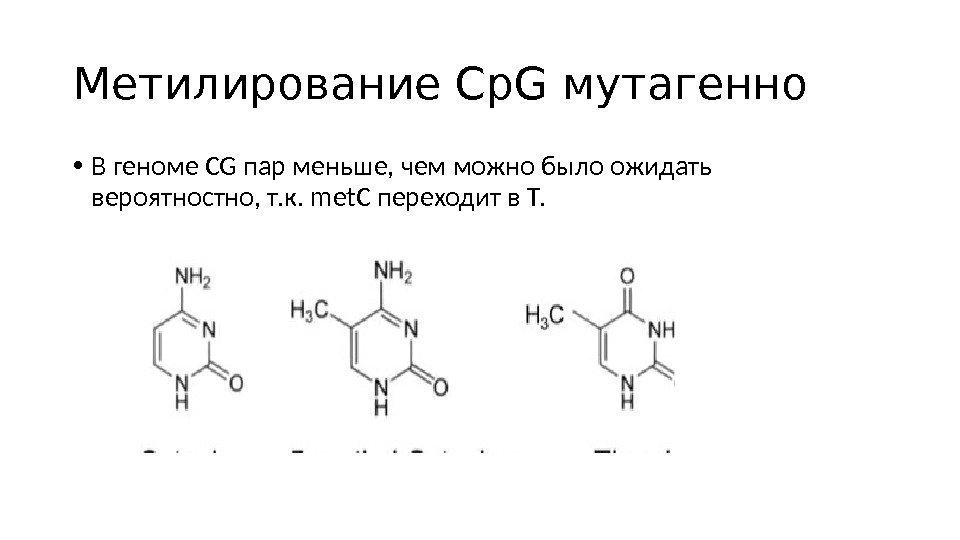
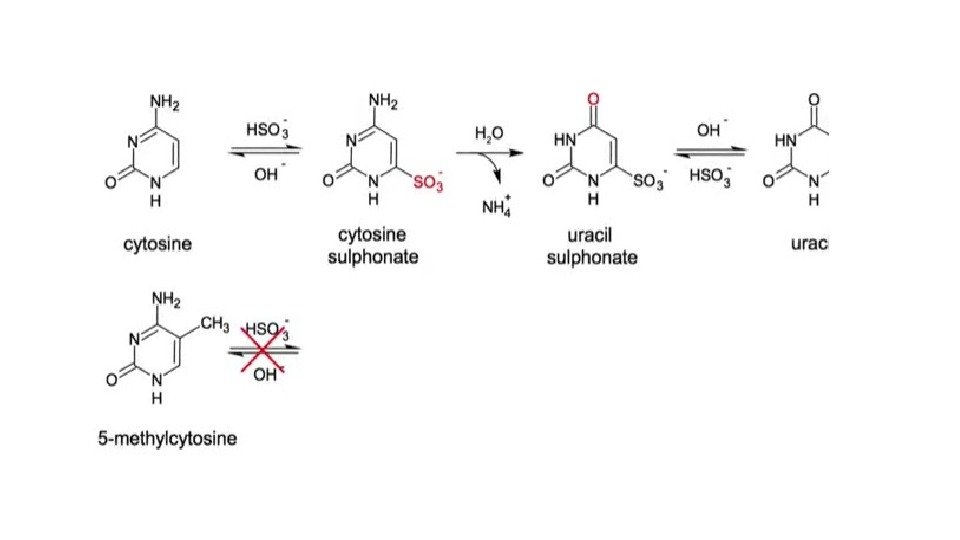
 Метилирование Ср. G мутагенно • В геноме CG пар меньше, чем можно было ожидать вероятностно, т. к. met. C переходит в Т.
Метилирование Ср. G мутагенно • В геноме CG пар меньше, чем можно было ожидать вероятностно, т. к. met. C переходит в Т.


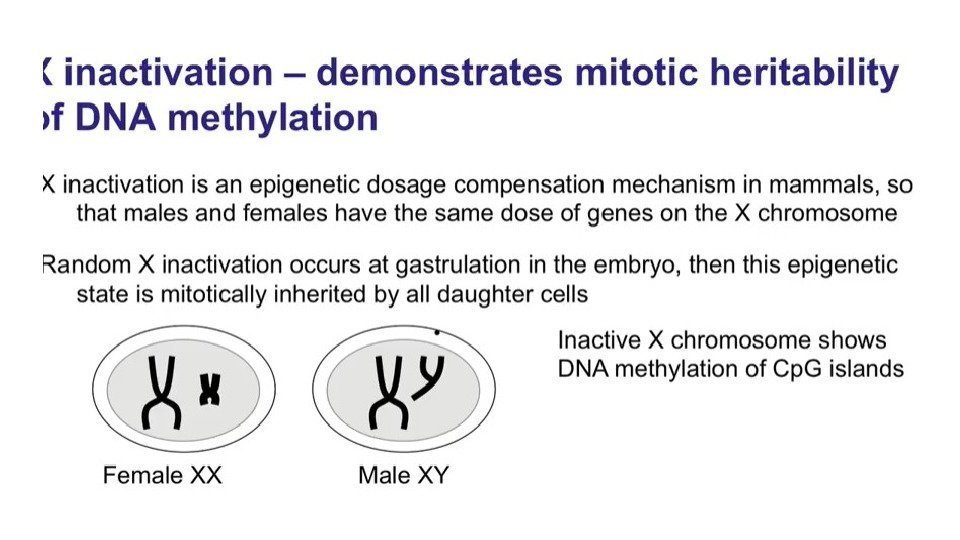
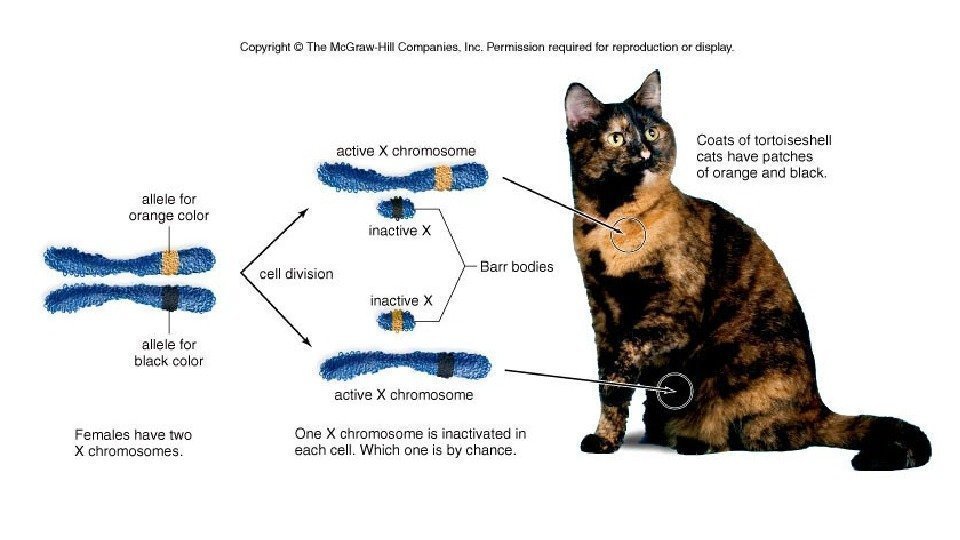
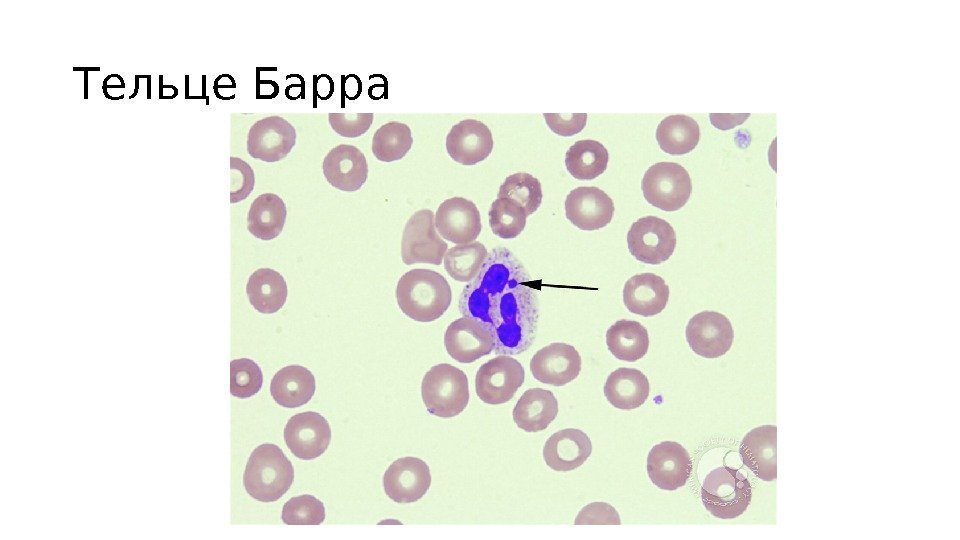
 Тельце Барра
Тельце Барра
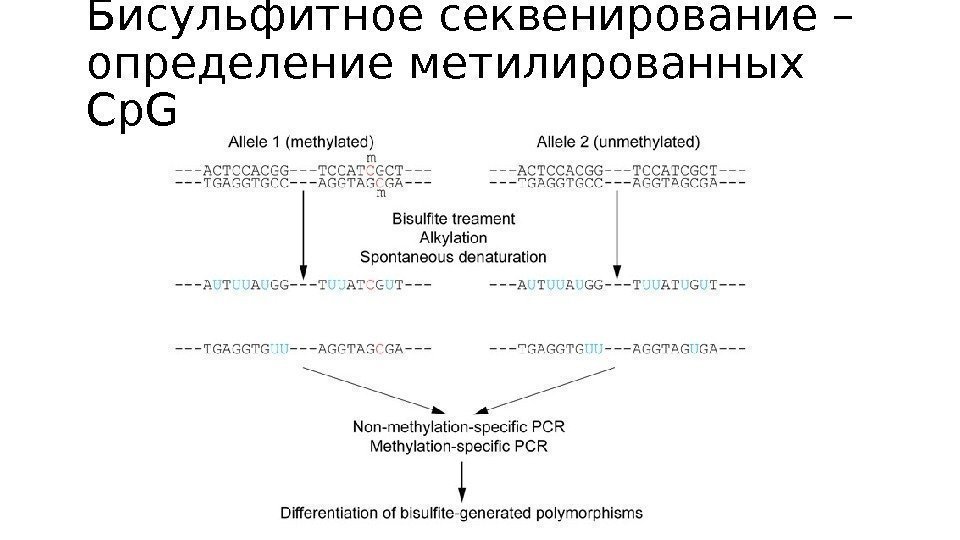
 Бисульфитное секвенирование – определение метилированных Cp. G
Бисульфитное секвенирование – определение метилированных Cp. G

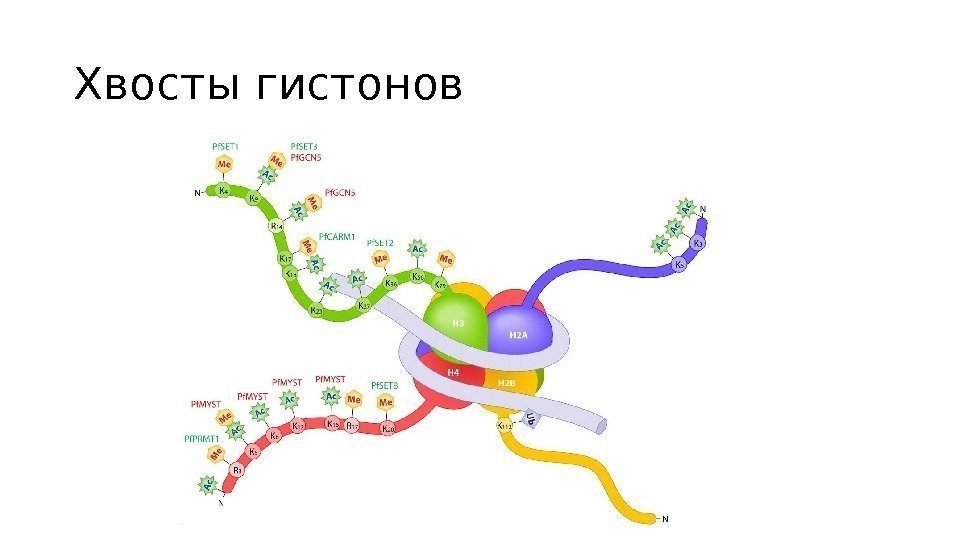
 Хвосты гистонов
Хвосты гистонов

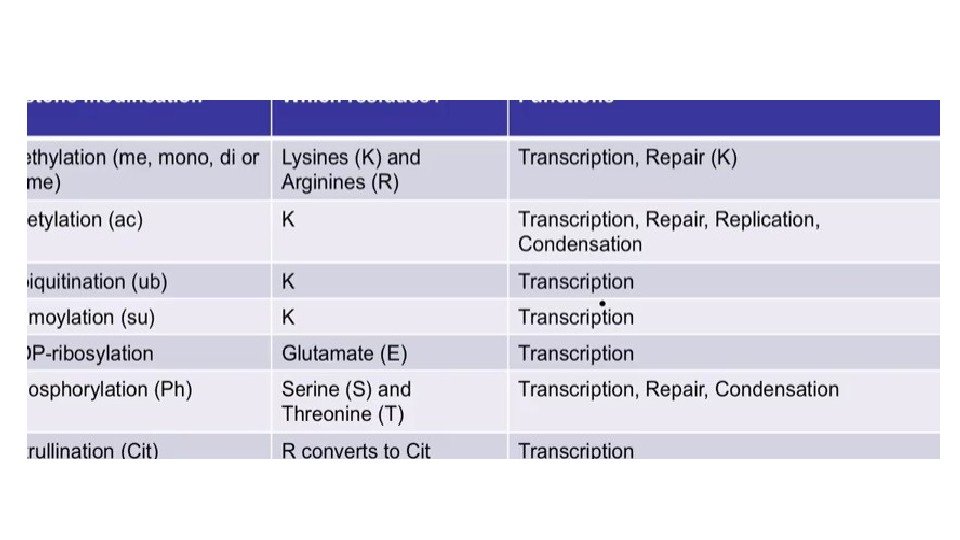
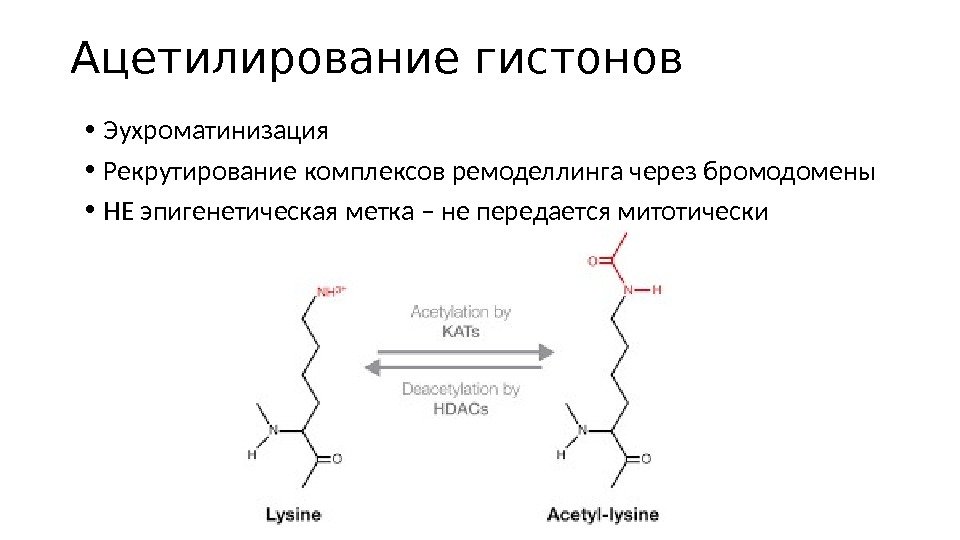
 Ацетилирование гистонов • Эухроматинизация • Рекрутирование комплексов ремоделлинга через бромодомены • НЕ эпигенетическая метка – не передается митотически
Ацетилирование гистонов • Эухроматинизация • Рекрутирование комплексов ремоделлинга через бромодомены • НЕ эпигенетическая метка – не передается митотически
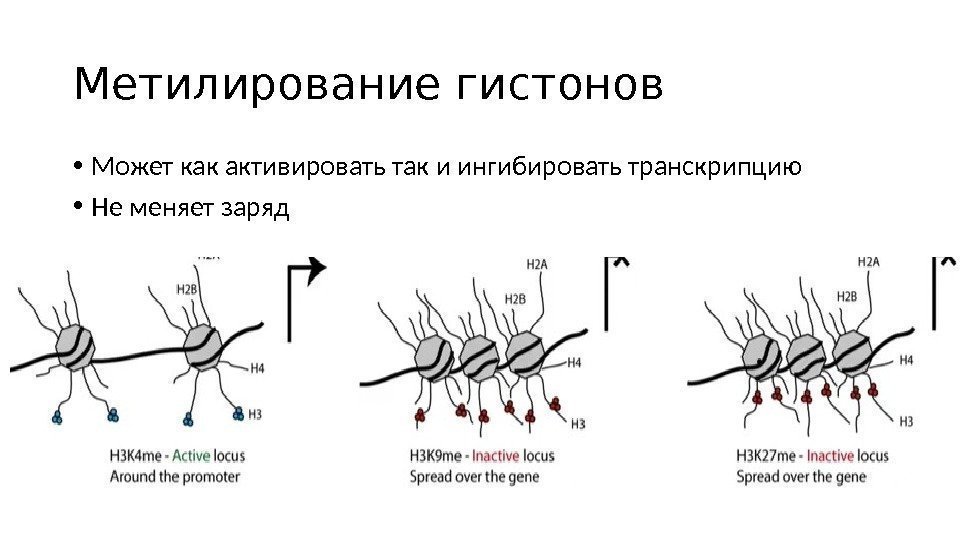
 Метилирование гистонов • Может как активировать так и ингибировать транскрипцию • Не меняет заряд
Метилирование гистонов • Может как активировать так и ингибировать транскрипцию • Не меняет заряд

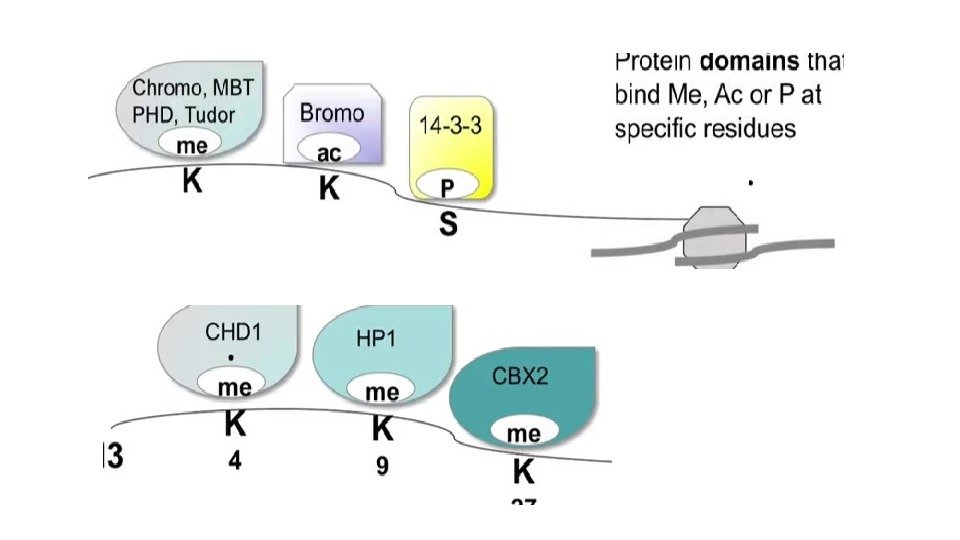
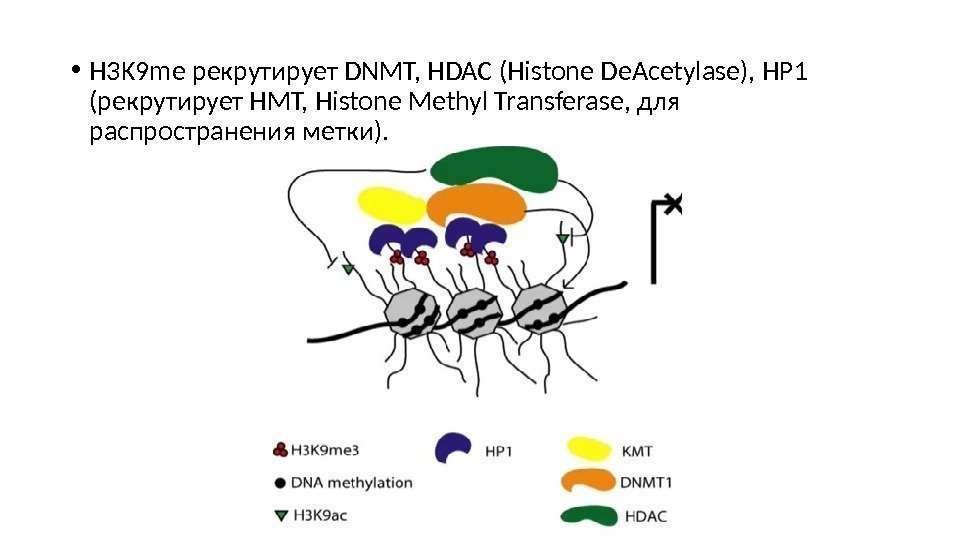
 • H 3 K 9 me рекрутирует DNMT, HDAC (Histone De. Acetylase), HP 1 (рекрутирует HMT, Histone Methyl Transferase, для распространения метки).
• H 3 K 9 me рекрутирует DNMT, HDAC (Histone De. Acetylase), HP 1 (рекрутирует HMT, Histone Methyl Transferase, для распространения метки).
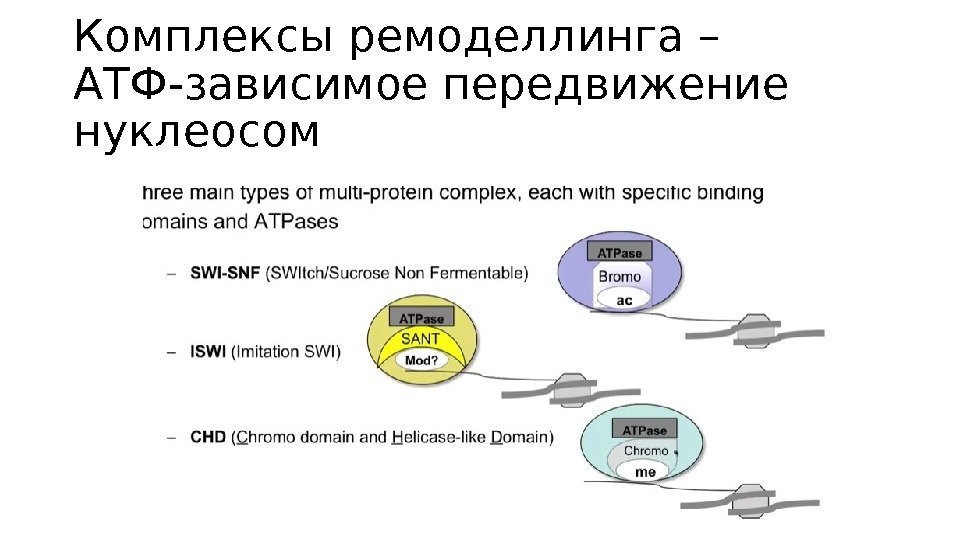
 Комплексы ремоделлинга – АТФ-зависимое передвижение нуклеосом
Комплексы ремоделлинга – АТФ-зависимое передвижение нуклеосом
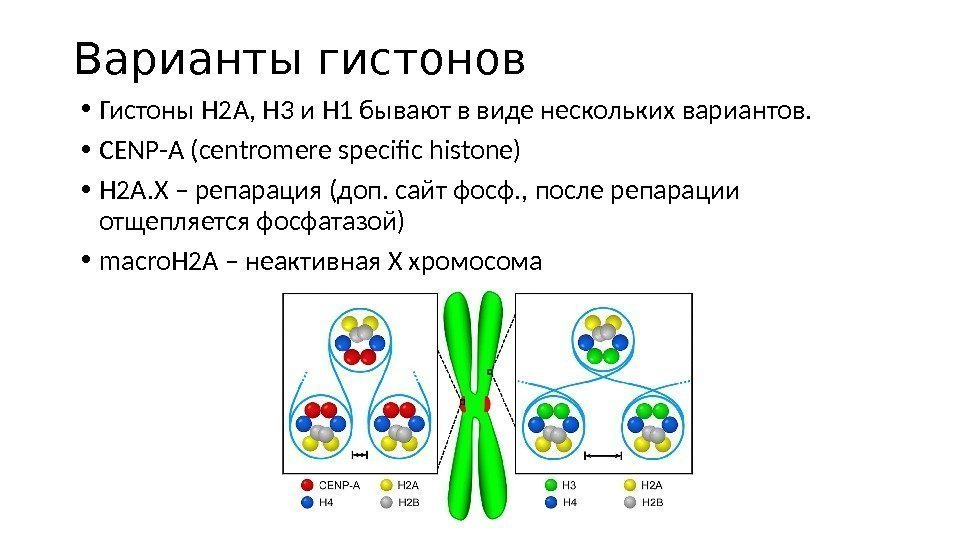
 Варианты гистонов • Гистоны Н 2 А, Н 3 и Н 1 бывают в виде нескольких вариантов. • CENP-A (centromere specific histone) • H 2 A. X – репарация (доп. сайт фосф. , после репарации отщепляется фосфатазой) • macro. H 2 A – неактивная Х хромосома
Варианты гистонов • Гистоны Н 2 А, Н 3 и Н 1 бывают в виде нескольких вариантов. • CENP-A (centromere specific histone) • H 2 A. X – репарация (доп. сайт фосф. , после репарации отщепляется фосфатазой) • macro. H 2 A – неактивная Х хромосома
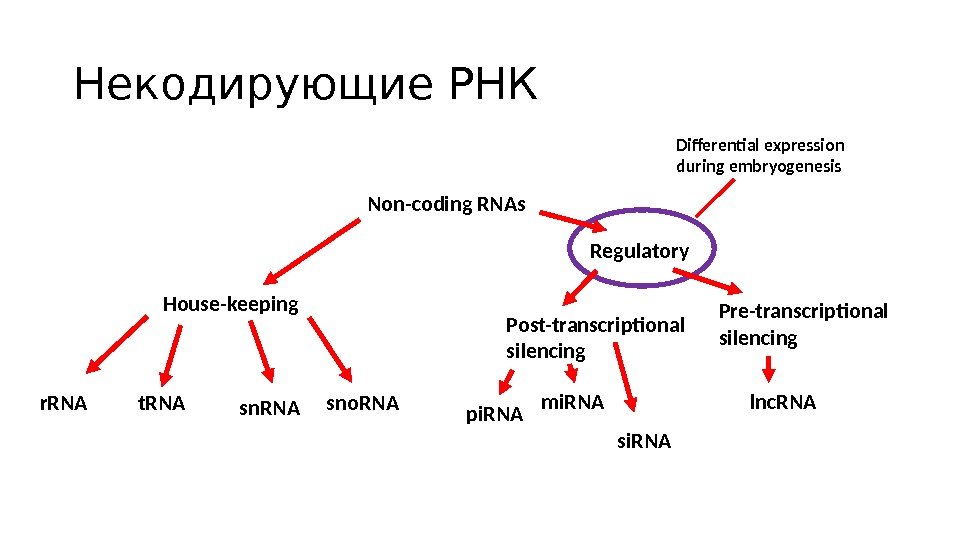
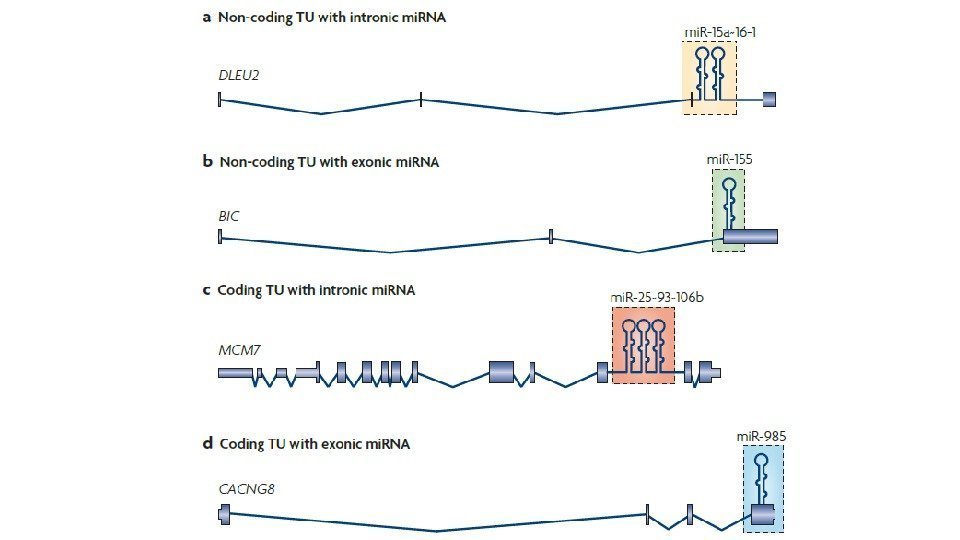
 Некодирующие РНК Non-coding RNAs House-keeping Regulatory r. RNA t. RNA sno. RNA Post-transcriptional silencing Pre-transcriptional silencing pi. RNA mi. RNA si. RNA lnc. RNADifferential expression during embryogenesis
Некодирующие РНК Non-coding RNAs House-keeping Regulatory r. RNA t. RNA sno. RNA Post-transcriptional silencing Pre-transcriptional silencing pi. RNA mi. RNA si. RNA lnc. RNADifferential expression during embryogenesis
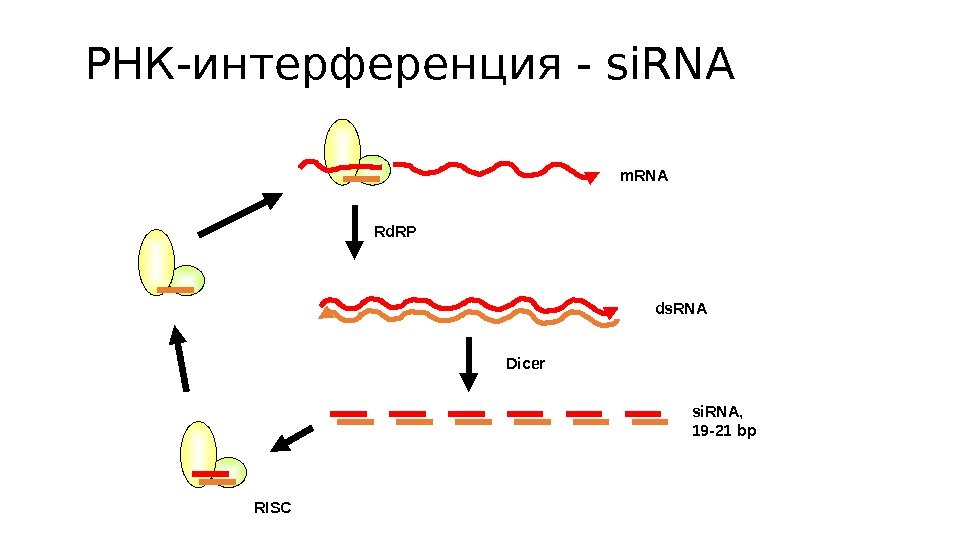
 РНК-интерференция — si. RNA ds. RNA si. RNA, 19 -21 bp. Dicer RISC m. RNA Rd. RP
РНК-интерференция — si. RNA ds. RNA si. RNA, 19 -21 bp. Dicer RISC m. RNA Rd. RP
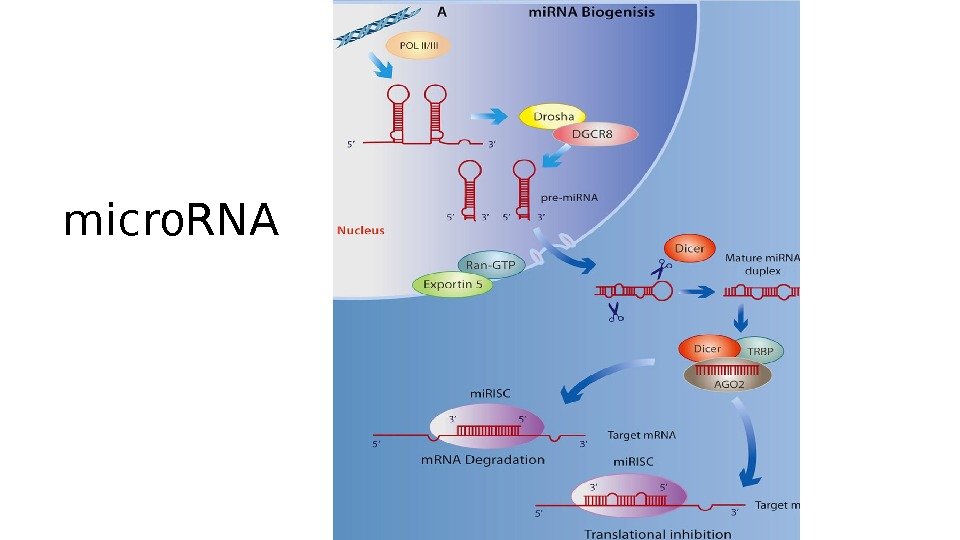
 micro. RN
micro. RN

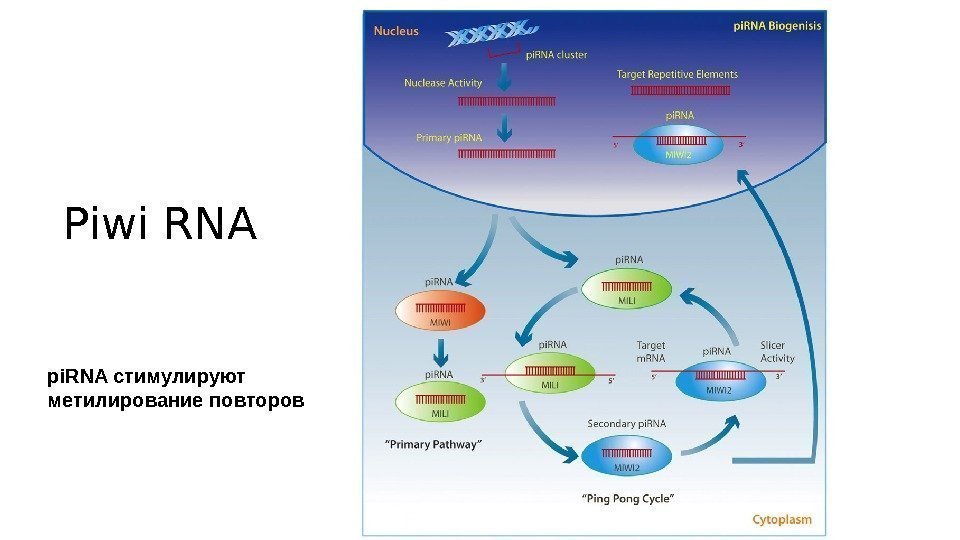
 Piwi RNA pi. RNA стимулируют метилирование повторов
Piwi RNA pi. RNA стимулируют метилирование повторов

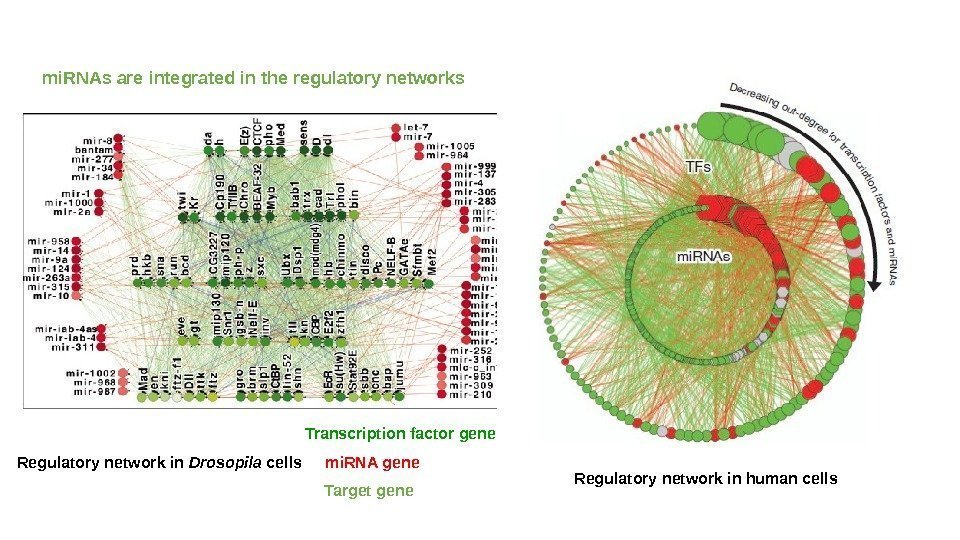
 mi. RNAs are integrated in the regulatory networks Transcription factor gene mi. RNA gene Target gene. Regulatory network in Drosopila cells Regulatory network in human cells
mi. RNAs are integrated in the regulatory networks Transcription factor gene mi. RNA gene Target gene. Regulatory network in Drosopila cells Regulatory network in human cells
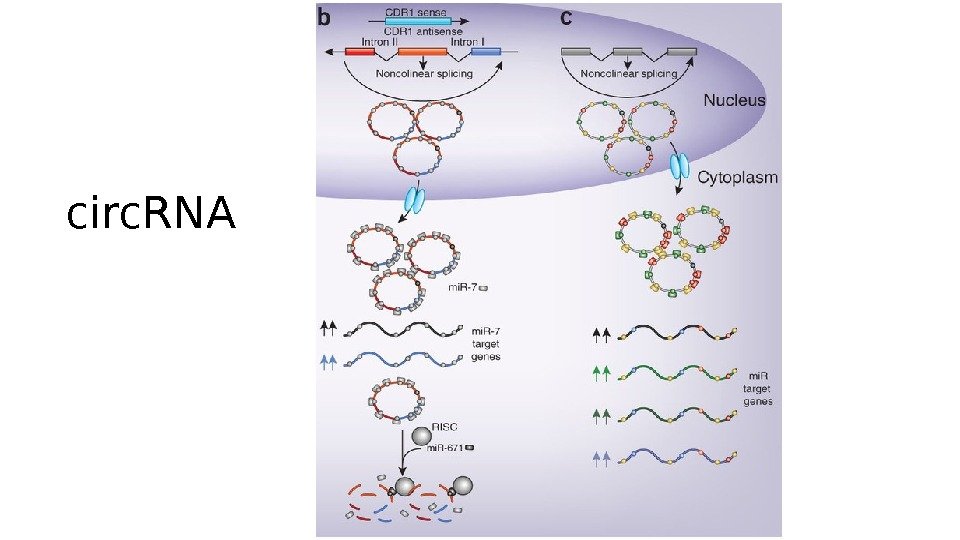
 circ. RN
circ. RN

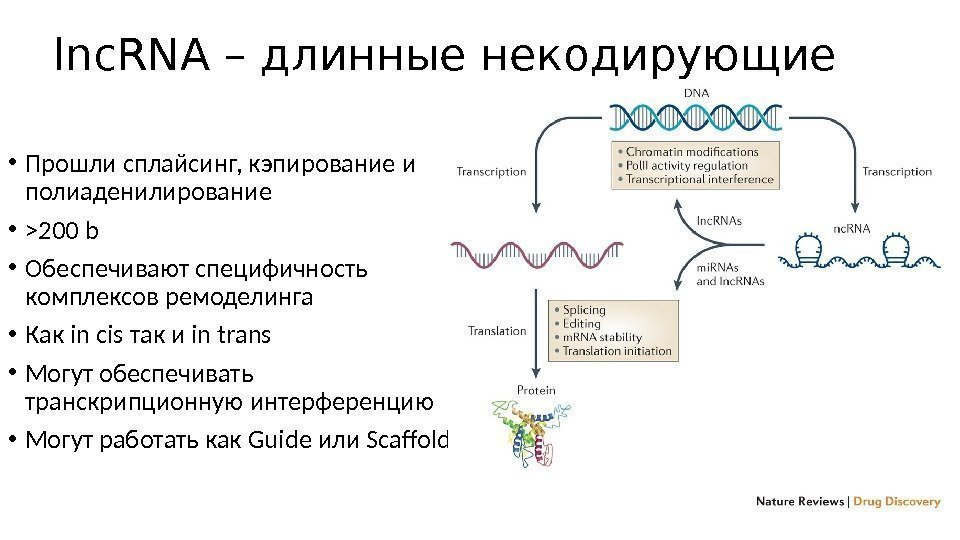
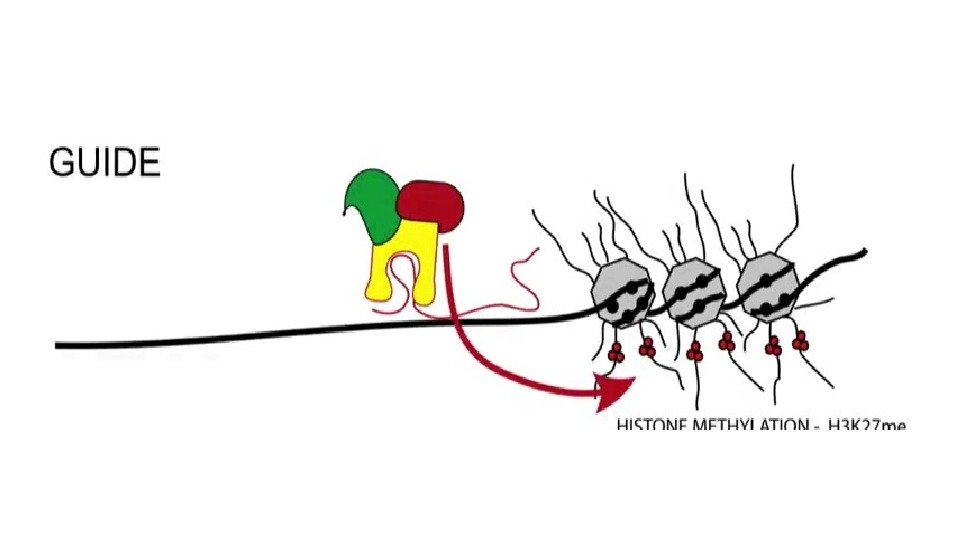
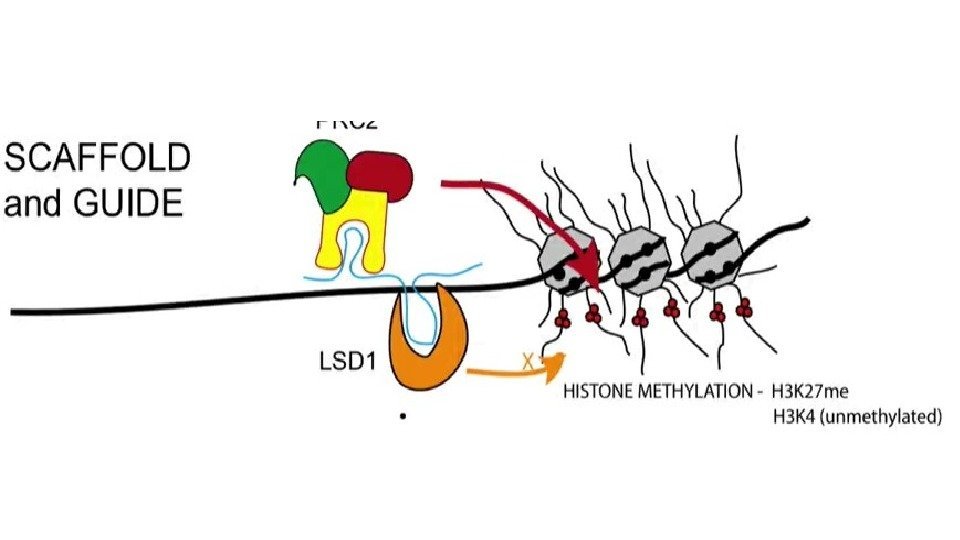
 lnc. RNA – длинные некодирующие • Прошли сплайсинг, кэпирование и полиаденилирование • >200 b • Обеспечивают специфичность комплексов ремоделинга • Как іn cis так и in trans • Могут обеспечивать транскрипционную интерференцию • Могут работать как Guide или Scaffold
lnc. RNA – длинные некодирующие • Прошли сплайсинг, кэпирование и полиаденилирование • >200 b • Обеспечивают специфичность комплексов ремоделинга • Как іn cis так и in trans • Могут обеспечивать транскрипционную интерференцию • Могут работать как Guide или Scaffold

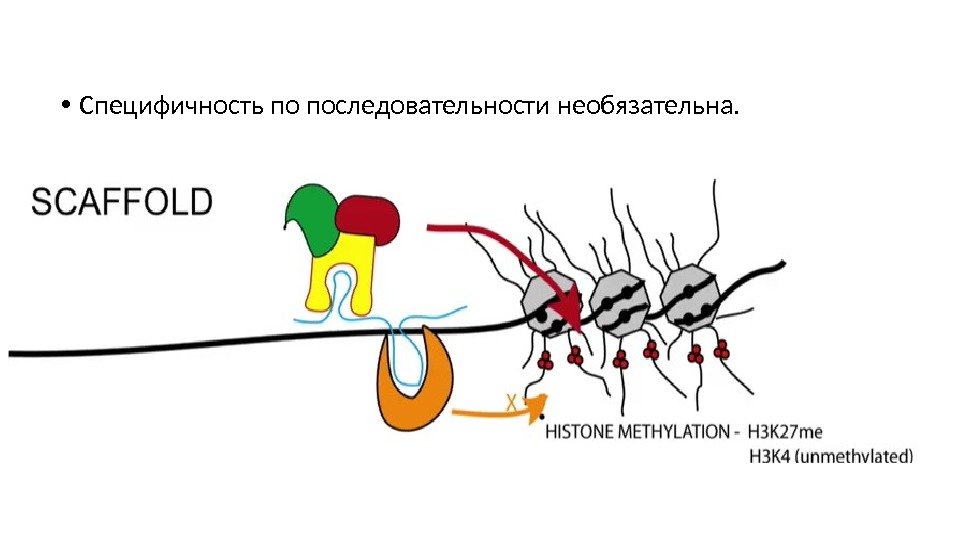
 • Специфичность по последовательности необязательна.
• Специфичность по последовательности необязательна.
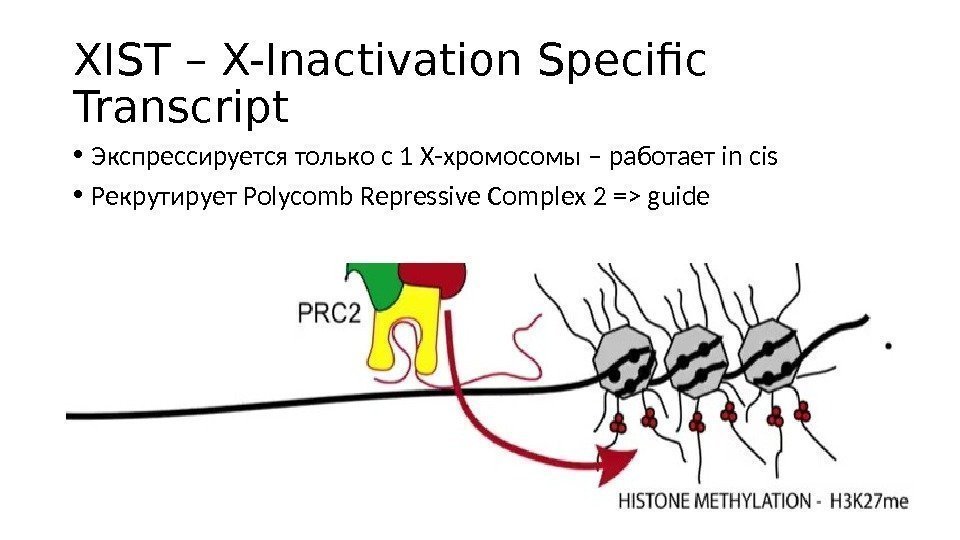
 XIST – X-Inactivation Specific Transcript • Экспрессируется только с 1 Х-хромосомы – работает in cis • Рекрутирует Polycomb Repressive Complex 2 => guide
XIST – X-Inactivation Specific Transcript • Экспрессируется только с 1 Х-хромосомы – работает in cis • Рекрутирует Polycomb Repressive Complex 2 => guide
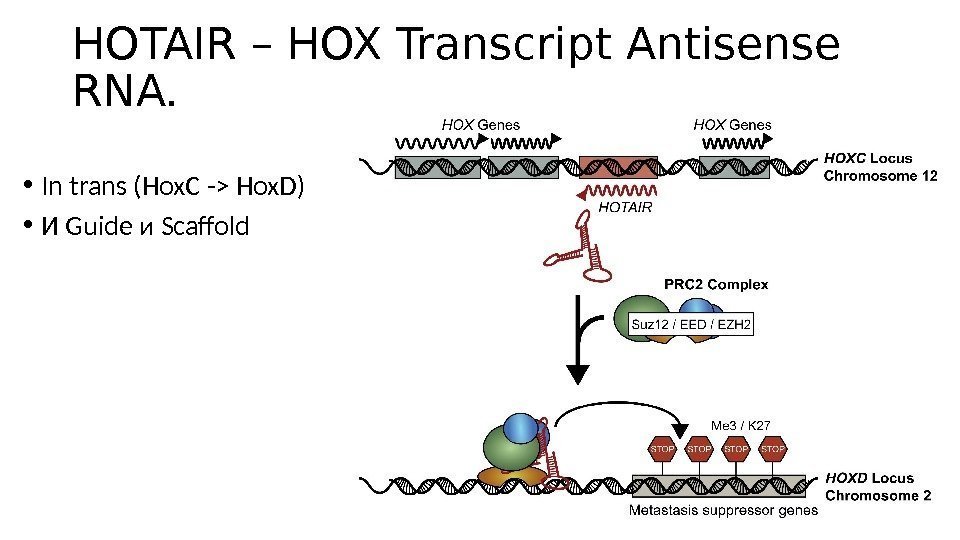
 HOTAIR – HOX Transcript Antisense RNA. • In trans (Hox. C -> Hox. D) • И Guide и Scaffold
HOTAIR – HOX Transcript Antisense RNA. • In trans (Hox. C -> Hox. D) • И Guide и Scaffold

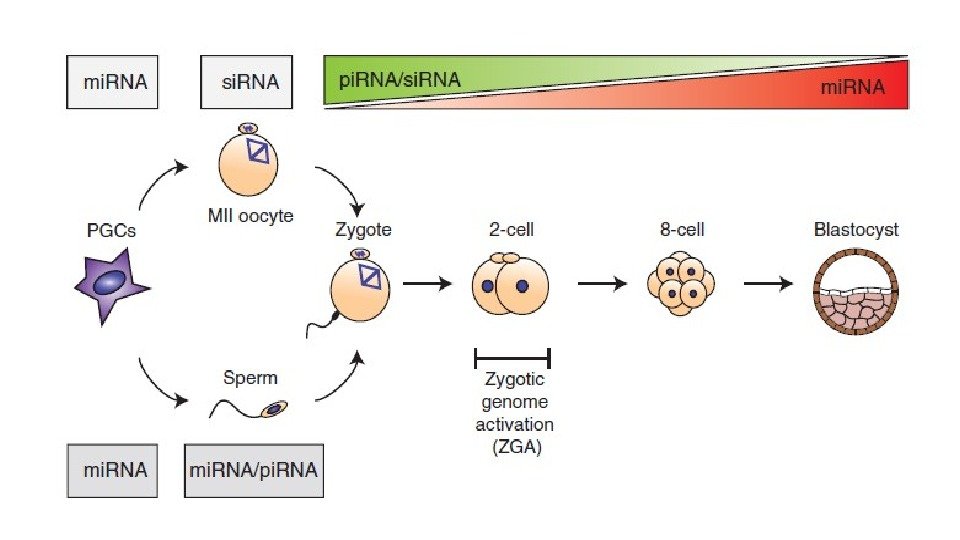
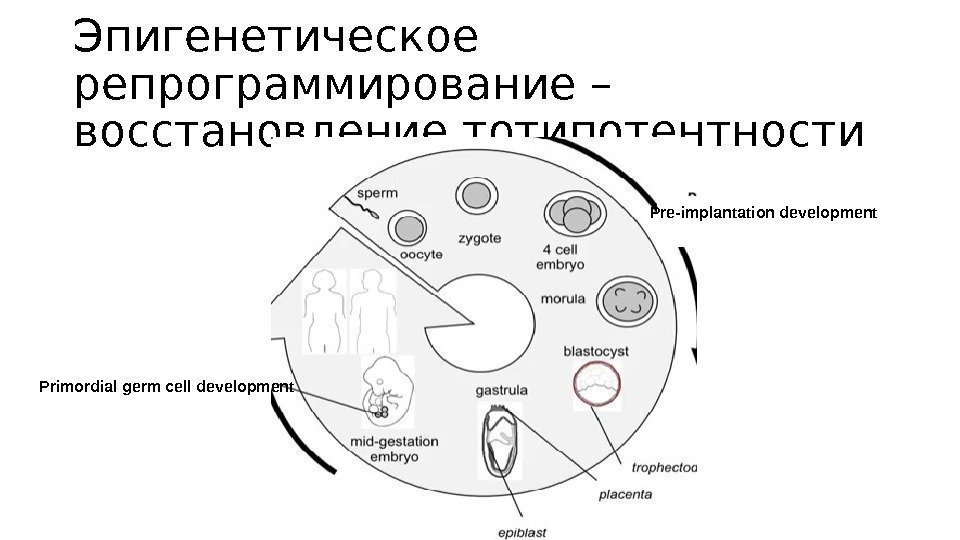
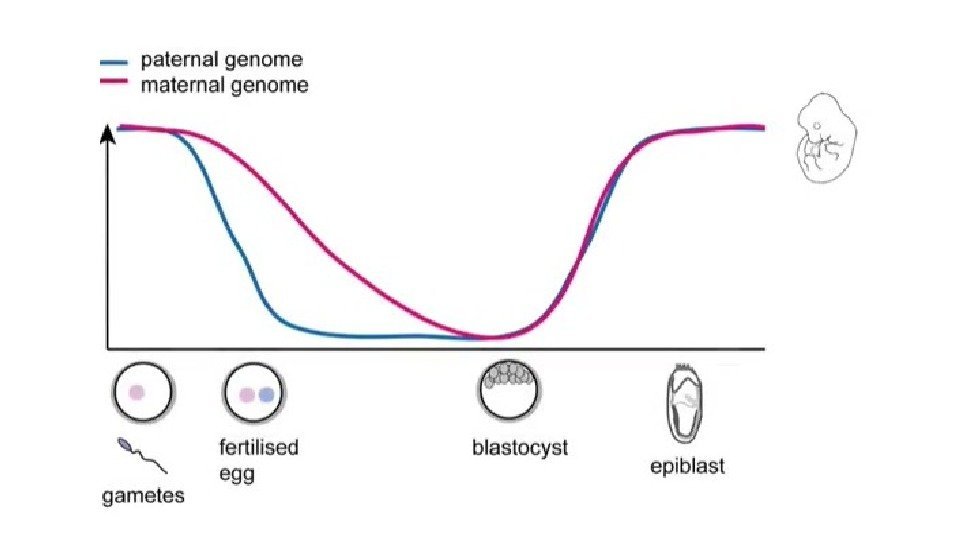
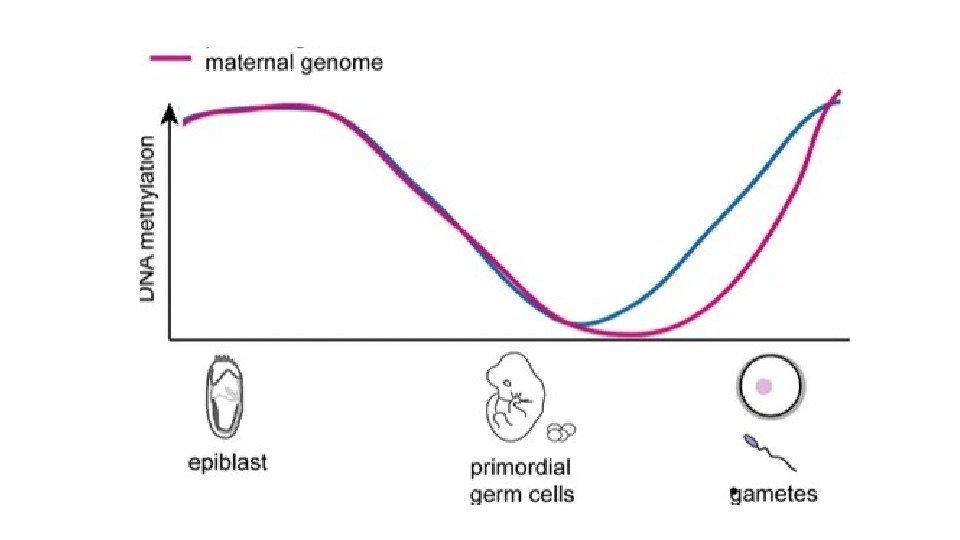
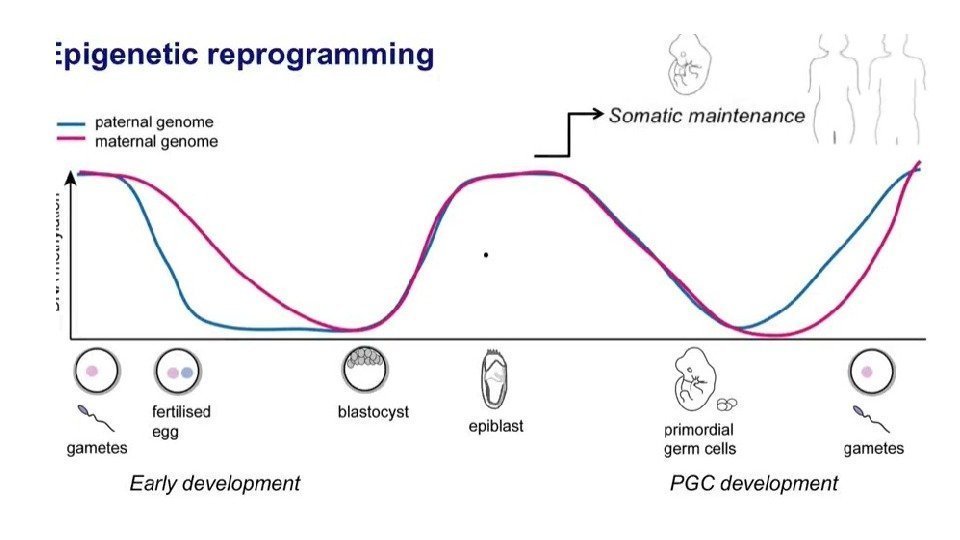
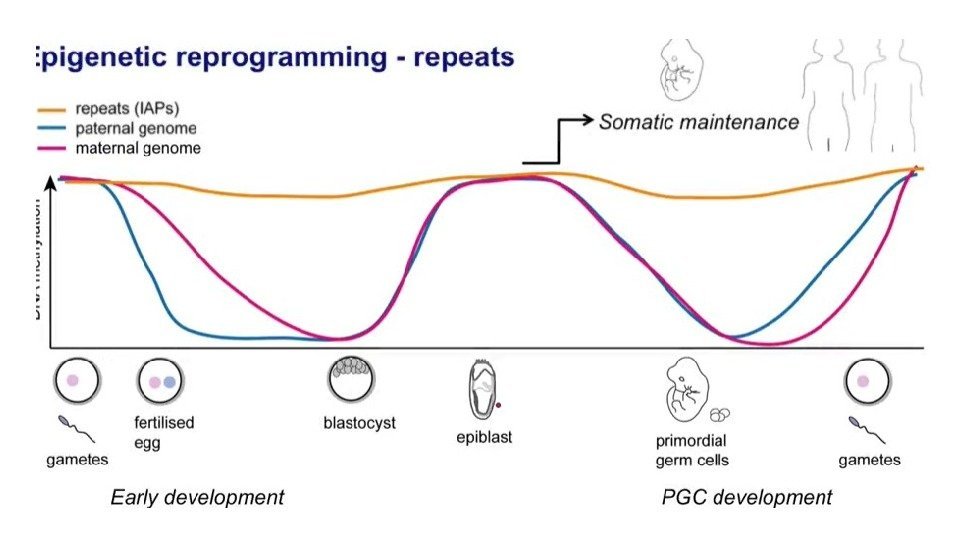
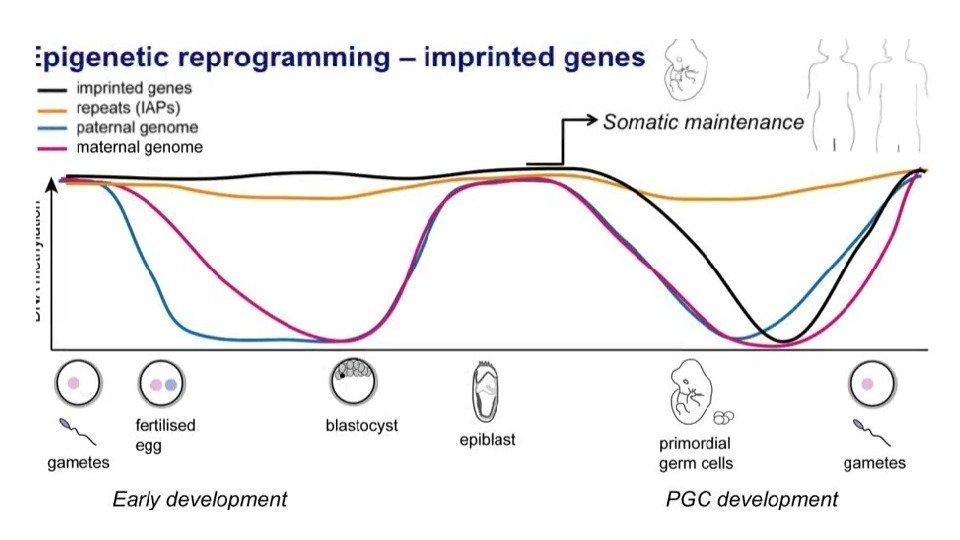
 Эпигенетическое репрограммирование – восстановление тотипотентности Primordial germ cell development Pre-implantation development
Эпигенетическое репрограммирование – восстановление тотипотентности Primordial germ cell development Pre-implantation development




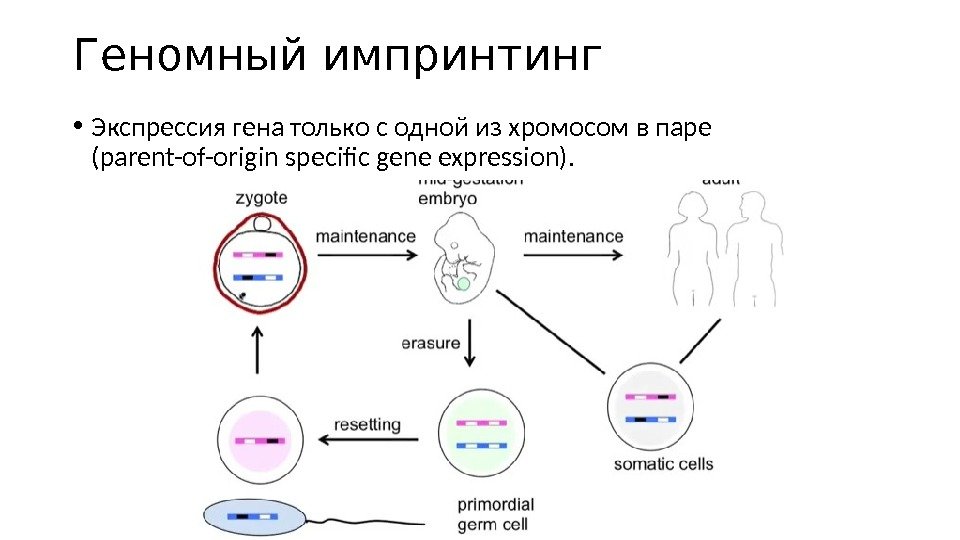
 Геномный импринтинг • Экспрессия гена только с одной из хромосом в паре (parent-of-origin specific gene expression).
Геномный импринтинг • Экспрессия гена только с одной из хромосом в паре (parent-of-origin specific gene expression).

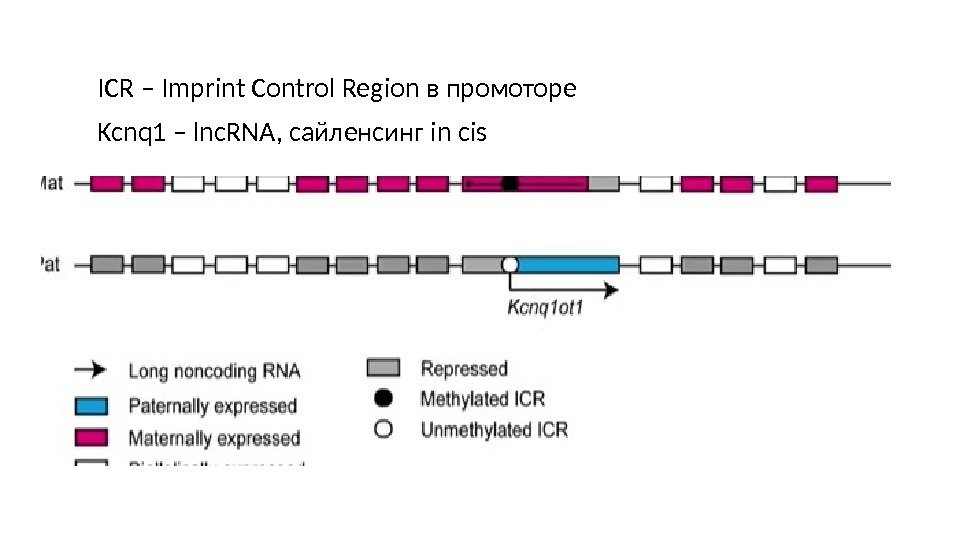
 ICR – Imprint Control Region в промоторе Kcnq 1 – lnc. RNA, сайленсинг in cis
ICR – Imprint Control Region в промоторе Kcnq 1 – lnc. RNA, сайленсинг in cis
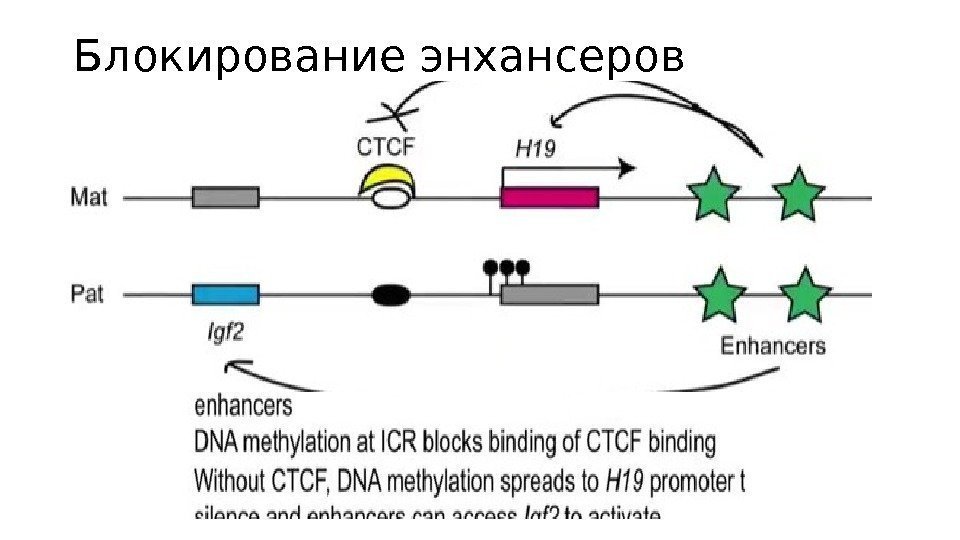
 Блокирование энхансеров
Блокирование энхансеров
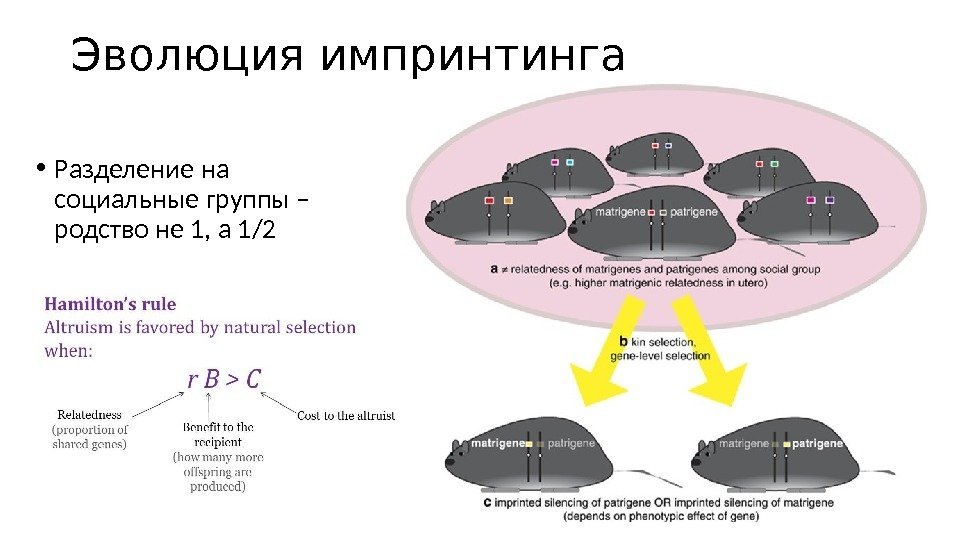
 Эволюция импринтинга • Разделение на социальные группы – родство не 1, а 1/
Эволюция импринтинга • Разделение на социальные группы – родство не 1, а 1/
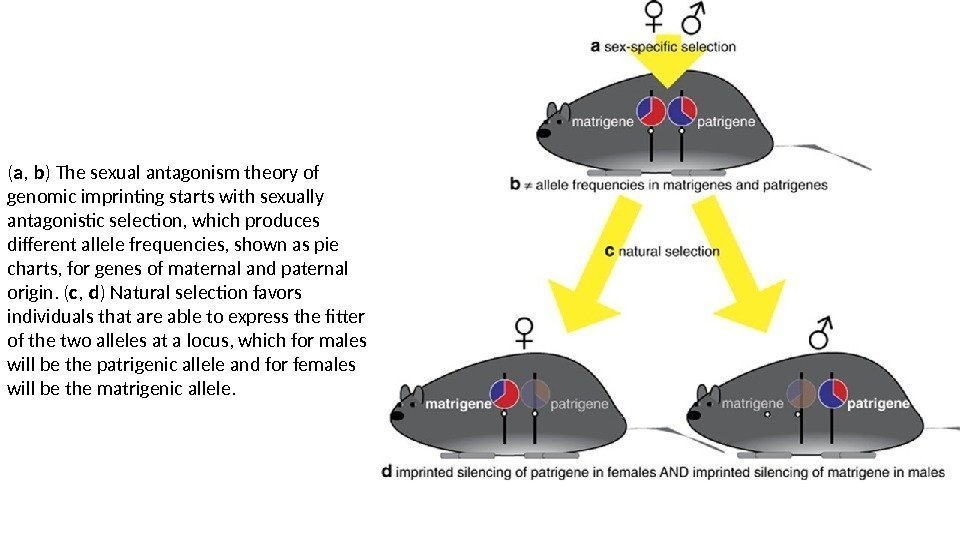
 ( a , b ) The sexual antagonism theory of genomic imprinting starts with sexually antagonistic selection, which produces different allele frequencies, shown as pie charts, for genes of maternal and paternal origin. ( c , d ) Natural selection favors individuals that are able to express the fitter of the two alleles at a locus, which for males will be the patrigenic allele and for females will be the matrigenic allele.
( a , b ) The sexual antagonism theory of genomic imprinting starts with sexually antagonistic selection, which produces different allele frequencies, shown as pie charts, for genes of maternal and paternal origin. ( c , d ) Natural selection favors individuals that are able to express the fitter of the two alleles at a locus, which for males will be the patrigenic allele and for females will be the matrigenic allele.
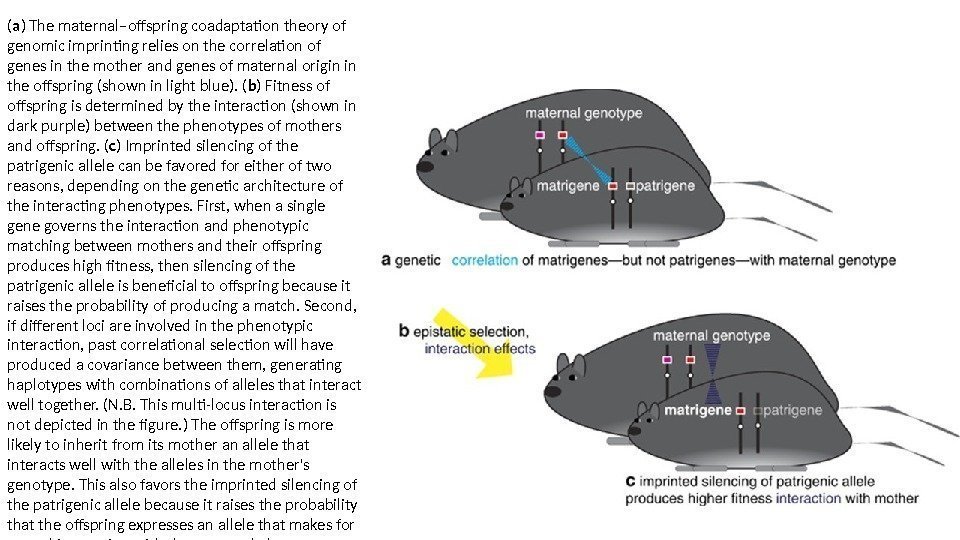
 ( a ) The maternal–offspring coadaptation theory of genomic imprinting relies on the correlation of genes in the mother and genes of maternal origin in the offspring (shown in light blue). ( b ) Fitness of offspring is determined by the interaction (shown in dark purple) between the phenotypes of mothers and offspring. ( c ) Imprinted silencing of the patrigenic allele can be favored for either of two reasons, depending on the genetic architecture of the interacting phenotypes. First, when a single gene governs the interaction and phenotypic matching between mothers and their offspring produces high fitness, then silencing of the patrigenic allele is beneficial to offspring because it raises the probability of producing a match. Second, if different loci are involved in the phenotypic interaction, past correlational selection will have produced a covariance between them, generating haplotypes with combinations of alleles that interact well together. (N. B. This multi-locus interaction is not depicted in the figure. ) The offspring is more likely to inherit from its mother an allele that interacts well with the alleles in the mother’s genotype. This also favors the imprinted silencing of the patrigenic allele because it raises the probability that the offspring expresses an allele that makes for a good interaction with the maternal phenotype.
( a ) The maternal–offspring coadaptation theory of genomic imprinting relies on the correlation of genes in the mother and genes of maternal origin in the offspring (shown in light blue). ( b ) Fitness of offspring is determined by the interaction (shown in dark purple) between the phenotypes of mothers and offspring. ( c ) Imprinted silencing of the patrigenic allele can be favored for either of two reasons, depending on the genetic architecture of the interacting phenotypes. First, when a single gene governs the interaction and phenotypic matching between mothers and their offspring produces high fitness, then silencing of the patrigenic allele is beneficial to offspring because it raises the probability of producing a match. Second, if different loci are involved in the phenotypic interaction, past correlational selection will have produced a covariance between them, generating haplotypes with combinations of alleles that interact well together. (N. B. This multi-locus interaction is not depicted in the figure. ) The offspring is more likely to inherit from its mother an allele that interacts well with the alleles in the mother’s genotype. This also favors the imprinted silencing of the patrigenic allele because it raises the probability that the offspring expresses an allele that makes for a good interaction with the maternal phenotype.
 Спасибо за внимание!
Спасибо за внимание!

