6eb6ae8720e03f1a61a31580ec74bed7.ppt
- Количество слайдов: 53
 Д. А. Равчеев Регуляция транскрипции в прокариотах Изучение методами сравнительной геномики Факультет Биоинженерии и Биоинформатики, Московский Государственный Университет им. М. В. Ломоносова Учебно-научный центр “Биоинформатика” Институт Проблем Передачи Информации РАН
Д. А. Равчеев Регуляция транскрипции в прокариотах Изучение методами сравнительной геномики Факультет Биоинженерии и Биоинформатики, Московский Государственный Университет им. М. В. Ломоносова Учебно-научный центр “Биоинформатика” Институт Проблем Передачи Информации РАН
 Изучение регуляции методами сравнительной геномики v Поиск потенциальных регуляторных последовательностей ● Пример: сайты связывания факторов транскрипции v Что дает изучение регуляции с помощью сравнительной геномики ● Полезно для функциональной аннотации и метаболической реконструкции ● Интересно само по себе n Исследование эволюции
Изучение регуляции методами сравнительной геномики v Поиск потенциальных регуляторных последовательностей ● Пример: сайты связывания факторов транскрипции v Что дает изучение регуляции с помощью сравнительной геномики ● Полезно для функциональной аннотации и метаболической реконструкции ● Интересно само по себе n Исследование эволюции
 Поиск потенциальных регуляторных сайтов Поиск по консенсусу Обучающая выборка – набор сайтов (обычно экспериментально определенных), на основании которого строится представление о структуре сигнала. Пример: Ген Обучающая выборка : pur. L cvp. A pur. C pur. H pur. R prs. A cod. B pur. E pur. M Консенсусная последовательность : Сайт tcc. ACGCAAACGGTTTCGTcag cct. ACGCAAACGTTTTCTTttt gat. ACGCAAACGTGTGCGTctg gtt. GCGCAAACGTTTTCGTtac taa. AGGCAAACGTTTACCTtgc cgc. AAGAAAACGTTTTCGCgag ccc. ACGAAAACGATTGCTTttt gcc. ACGCAACCGTTTTCCTtgc gtc. TCGCAAACGTTTGCTTtcc ACGCAAACGTTTGCGT Поиск потенциального сайта в геномной последовательности : car. A ACGCAAACGTTTGCGT atgcaatcttcttgctg. CGCAAg. CGTTTt. Ccagaacaggttagatgatctttttgtcgctt 11 нуклеотидов из 16 совпадают с консенсусом
Поиск потенциальных регуляторных сайтов Поиск по консенсусу Обучающая выборка – набор сайтов (обычно экспериментально определенных), на основании которого строится представление о структуре сигнала. Пример: Ген Обучающая выборка : pur. L cvp. A pur. C pur. H pur. R prs. A cod. B pur. E pur. M Консенсусная последовательность : Сайт tcc. ACGCAAACGGTTTCGTcag cct. ACGCAAACGTTTTCTTttt gat. ACGCAAACGTGTGCGTctg gtt. GCGCAAACGTTTTCGTtac taa. AGGCAAACGTTTACCTtgc cgc. AAGAAAACGTTTTCGCgag ccc. ACGAAAACGATTGCTTttt gcc. ACGCAACCGTTTTCCTtgc gtc. TCGCAAACGTTTGCTTtcc ACGCAAACGTTTGCGT Поиск потенциального сайта в геномной последовательности : car. A ACGCAAACGTTTGCGT atgcaatcttcttgctg. CGCAAg. CGTTTt. Ccagaacaggttagatgatctttttgtcgctt 11 нуклеотидов из 16 совпадают с консенсусом
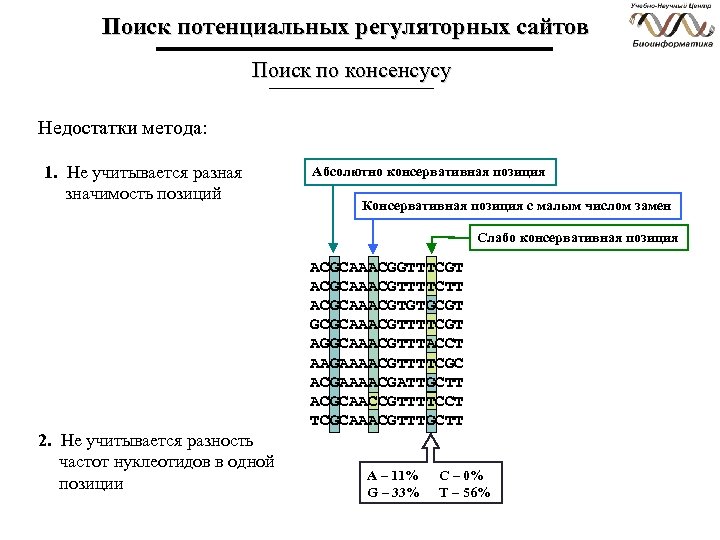 Поиск потенциальных регуляторных сайтов Поиск по консенсусу Недостатки метода: 1. Не учитывается разная значимость позиций Абсолютно консервативная позиция Консервативная позиция с малым числом замен Слабо консервативная позиция 2. Не учитывается разность частот нуклеотидов в одной позиции ACGCAAACGGTTTCGT ACGCAAACGTTTTCTT ACGCAAACGTGTGCGT GCGCAAACGTTTTCGT AGGCAAACGTTTACCT AAGAAAACGTTTTCGC ACGAAAACGATTGCTT ACGCAACCGTTTTCCT TCGCAAACGTTTGCTT А – 11% G – 33% С – 0% T – 56%
Поиск потенциальных регуляторных сайтов Поиск по консенсусу Недостатки метода: 1. Не учитывается разная значимость позиций Абсолютно консервативная позиция Консервативная позиция с малым числом замен Слабо консервативная позиция 2. Не учитывается разность частот нуклеотидов в одной позиции ACGCAAACGGTTTCGT ACGCAAACGTTTTCTT ACGCAAACGTGTGCGT GCGCAAACGTTTTCGT AGGCAAACGTTTACCT AAGAAAACGTTTTCGC ACGAAAACGATTGCTT ACGCAACCGTTTTCCT TCGCAAACGTTTGCTT А – 11% G – 33% С – 0% T – 56%
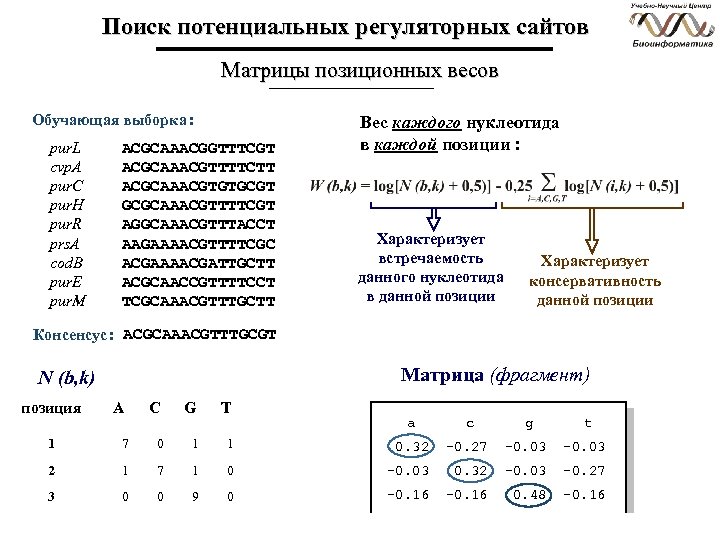 Поиск потенциальных регуляторных сайтов Матрицы позиционных весов Обучающая выборка : pur. L cvp. A pur. C pur. H pur. R prs. A cod. B pur. E pur. M ACGCAAACGGTTTCGT ACGCAAACGTTTTCTT ACGCAAACGTGTGCGT GCGCAAACGTTTTCGT AGGCAAACGTTTACCT AAGAAAACGTTTTCGC ACGAAAACGATTGCTT ACGCAACCGTTTTCCT TCGCAAACGTTTGCTT Вес каждого нуклеотида в каждой позиции : Характеризует встречаемость данного нуклеотида в данной позиции Характеризует консервативность данной позиции Консенсус : ACGCAAACGTTTGCGT Матрица (фрагмент) N (b, k) позиция A C G T 1 7 0 1 2 1 7 3 0 0 a c g t 1 0. 32 -0. 27 -0. 03 1 0 -0. 03 0. 32 -0. 03 -0. 27 9 0 -0. 16 0. 48 -0. 16
Поиск потенциальных регуляторных сайтов Матрицы позиционных весов Обучающая выборка : pur. L cvp. A pur. C pur. H pur. R prs. A cod. B pur. E pur. M ACGCAAACGGTTTCGT ACGCAAACGTTTTCTT ACGCAAACGTGTGCGT GCGCAAACGTTTTCGT AGGCAAACGTTTACCT AAGAAAACGTTTTCGC ACGAAAACGATTGCTT ACGCAACCGTTTTCCT TCGCAAACGTTTGCTT Вес каждого нуклеотида в каждой позиции : Характеризует встречаемость данного нуклеотида в данной позиции Характеризует консервативность данной позиции Консенсус : ACGCAAACGTTTGCGT Матрица (фрагмент) N (b, k) позиция A C G T 1 7 0 1 2 1 7 3 0 0 a c g t 1 0. 32 -0. 27 -0. 03 1 0 -0. 03 0. 32 -0. 03 -0. 27 9 0 -0. 16 0. 48 -0. 16
 Поиск потенциальных регуляторных сайтов Матрицы позиционных весов Поиск потенциального сайта в геномной последовательности : позиция car. A atgcaatcttcttgct. GCGCAAGCGTTTTCCAgaacaggttagatgatctttttgtcgctt a 0. 32 -0. 03 -0. 16 0. 12 0. 48 0. 40 -0. 16 -0. 03 -0. 21 -0. 16 -0. 06 -0. 16 -0. 31 -0. 21 c -0. 27 0. 32 -0. 16 0. 35 -0. 16 0. 03 0. 48 -0. 16 -0. 27 -0. 21 -0. 16 -0. 30 0. 48 0. 04 0. 03 g -0. 03 0. 48 -0. 24 -0. 16 -0. 21 -0. 16 0. 48 -0. 03 -0. 16 0. 13 -0. 16 0. 17 -0. 21 t -0. 03 -0. 27 -0. 16 -0. 24 -0. 16 -0. 21 -0. 16 0. 32 0. 40 0. 48 0. 23 -0. 16 0. 11 0. 40 Вес сайта : Вес найденного сайта : 4, 57 Недостаток метода : q ложные предсказания сайтов, вызванные случайным сходством последовательностей
Поиск потенциальных регуляторных сайтов Матрицы позиционных весов Поиск потенциального сайта в геномной последовательности : позиция car. A atgcaatcttcttgct. GCGCAAGCGTTTTCCAgaacaggttagatgatctttttgtcgctt a 0. 32 -0. 03 -0. 16 0. 12 0. 48 0. 40 -0. 16 -0. 03 -0. 21 -0. 16 -0. 06 -0. 16 -0. 31 -0. 21 c -0. 27 0. 32 -0. 16 0. 35 -0. 16 0. 03 0. 48 -0. 16 -0. 27 -0. 21 -0. 16 -0. 30 0. 48 0. 04 0. 03 g -0. 03 0. 48 -0. 24 -0. 16 -0. 21 -0. 16 0. 48 -0. 03 -0. 16 0. 13 -0. 16 0. 17 -0. 21 t -0. 03 -0. 27 -0. 16 -0. 24 -0. 16 -0. 21 -0. 16 0. 32 0. 40 0. 48 0. 23 -0. 16 0. 11 0. 40 Вес сайта : Вес найденного сайта : 4, 57 Недостаток метода : q ложные предсказания сайтов, вызванные случайным сходством последовательностей
 Поиск потенциальных регуляторных сайтов Распознающее правило 1. Матрица позиционных весов 2. Область поиска сайтов как привило, в потенциальной реуляторной области гена: - 400 … +100 п. н. от старта трансляции. 3. Пороговое значение для веса сайта а) Минимальный вес сайтов из обучающей выборки Сайты из обучающей выборки, оцененные по матрице весов Ген Сайт Вес cvp. A pur. L pur. H pur. C pur. M pur. E cod. B pur. R prs. A ACGCAAACGTTTTCTT ACGCAAACGGTTTCGT GCGCAAACGTTTTCGT ACGCAAACGTGTGCGT TCGCAAACGTTTGCTT ACGCAACCGTTTTCCT ACGAAAACGATTGCTT AGGCAAACGTTTACCT AAGAAAACGTTTTCGC 6, 23 5, 94 5, 81 5, 79 5, 78 5, 55 5, 53 5, 32 б) В соответствии с ожидаемым размером регулона пороговое значение
Поиск потенциальных регуляторных сайтов Распознающее правило 1. Матрица позиционных весов 2. Область поиска сайтов как привило, в потенциальной реуляторной области гена: - 400 … +100 п. н. от старта трансляции. 3. Пороговое значение для веса сайта а) Минимальный вес сайтов из обучающей выборки Сайты из обучающей выборки, оцененные по матрице весов Ген Сайт Вес cvp. A pur. L pur. H pur. C pur. M pur. E cod. B pur. R prs. A ACGCAAACGTTTTCTT ACGCAAACGGTTTCGT GCGCAAACGTTTTCGT ACGCAAACGTGTGCGT TCGCAAACGTTTGCTT ACGCAACCGTTTTCCT ACGAAAACGATTGCTT AGGCAAACGTTTACCT AAGAAAACGTTTTCGC 6, 23 5, 94 5, 81 5, 79 5, 78 5, 55 5, 53 5, 32 б) В соответствии с ожидаемым размером регулона пороговое значение
 Сравнительный подход к изучению регуляции Метод проверки соответствия Базовый геном гены сайты
Сравнительный подход к изучению регуляции Метод проверки соответствия Базовый геном гены сайты
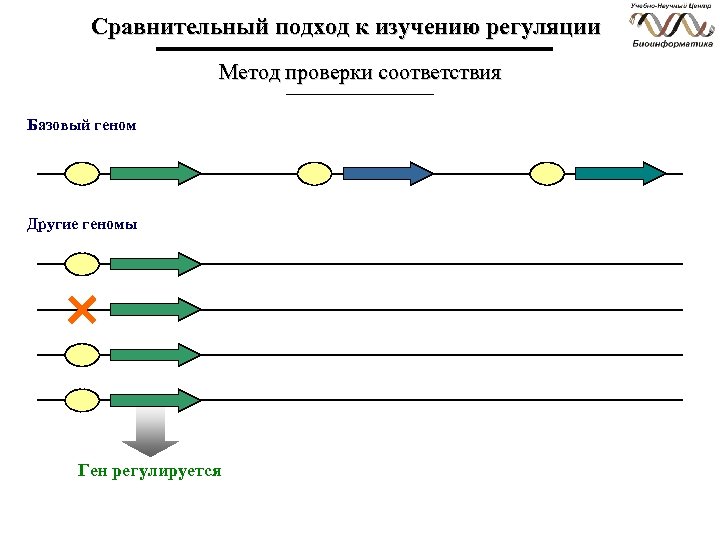 Сравнительный подход к изучению регуляции Метод проверки соответствия Базовый геном Другие геномы Ген регулируется
Сравнительный подход к изучению регуляции Метод проверки соответствия Базовый геном Другие геномы Ген регулируется
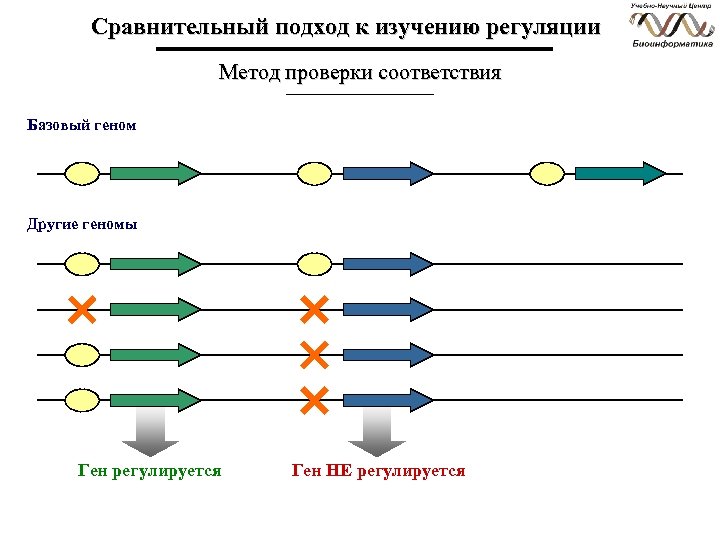 Сравнительный подход к изучению регуляции Метод проверки соответствия Базовый геном Другие геномы Ген регулируется Ген НЕ регулируется
Сравнительный подход к изучению регуляции Метод проверки соответствия Базовый геном Другие геномы Ген регулируется Ген НЕ регулируется
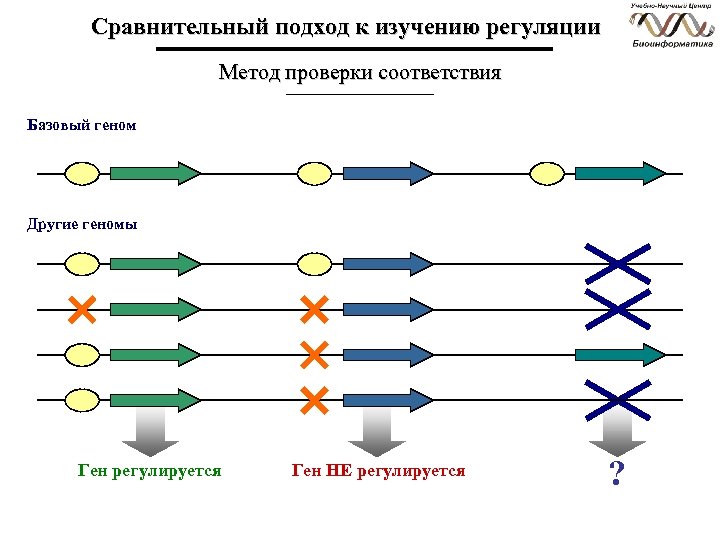 Сравнительный подход к изучению регуляции Метод проверки соответствия Базовый геном Другие геномы Ген регулируется Ген НЕ регулируется ?
Сравнительный подход к изучению регуляции Метод проверки соответствия Базовый геном Другие геномы Ген регулируется Ген НЕ регулируется ?
 Сравнительный подход к изучению регуляции Ключевые понятия Регулон (Regulon) — группа генов, непосредственно регулирующихся одной регуляторной системой в одном организме Обобщенный регулон (Regulog) — группа генов, непосредственно регулирующихся одной регуляторной системой в группе родственных организмов
Сравнительный подход к изучению регуляции Ключевые понятия Регулон (Regulon) — группа генов, непосредственно регулирующихся одной регуляторной системой в одном организме Обобщенный регулон (Regulog) — группа генов, непосредственно регулирующихся одной регуляторной системой в группе родственных организмов
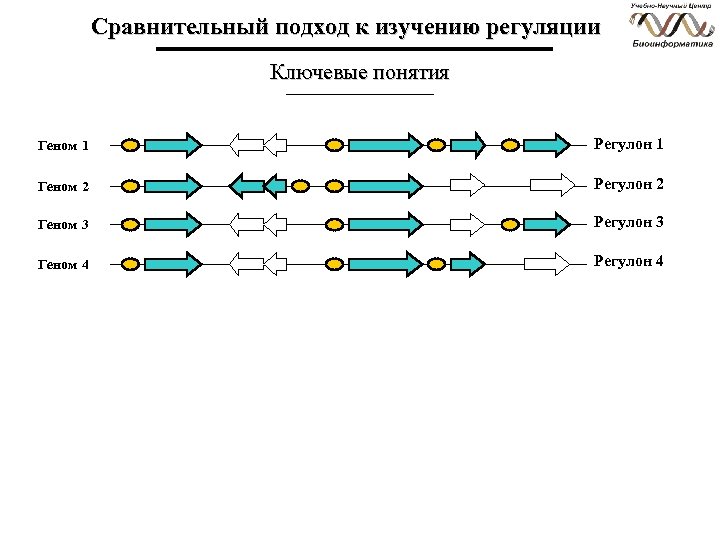 Сравнительный подход к изучению регуляции Ключевые понятия Геном 1 Регулон 1 Геном 2 Регулон 2 Геном 3 Регулон 3 Геном 4 Регулон 4
Сравнительный подход к изучению регуляции Ключевые понятия Геном 1 Регулон 1 Геном 2 Регулон 2 Геном 3 Регулон 3 Геном 4 Регулон 4
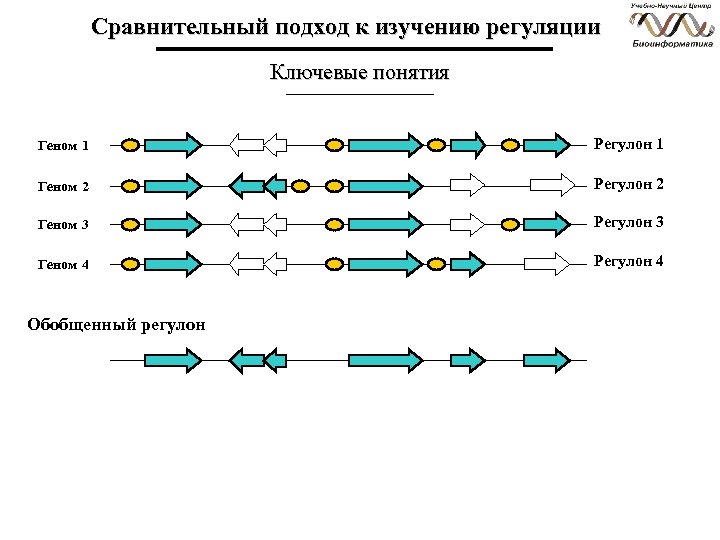 Сравнительный подход к изучению регуляции Ключевые понятия Геном 1 Регулон 1 Геном 2 Регулон 2 Геном 3 Регулон 3 Геном 4 Регулон 4 Обобщенный регулон
Сравнительный подход к изучению регуляции Ключевые понятия Геном 1 Регулон 1 Геном 2 Регулон 2 Геном 3 Регулон 3 Геном 4 Регулон 4 Обобщенный регулон
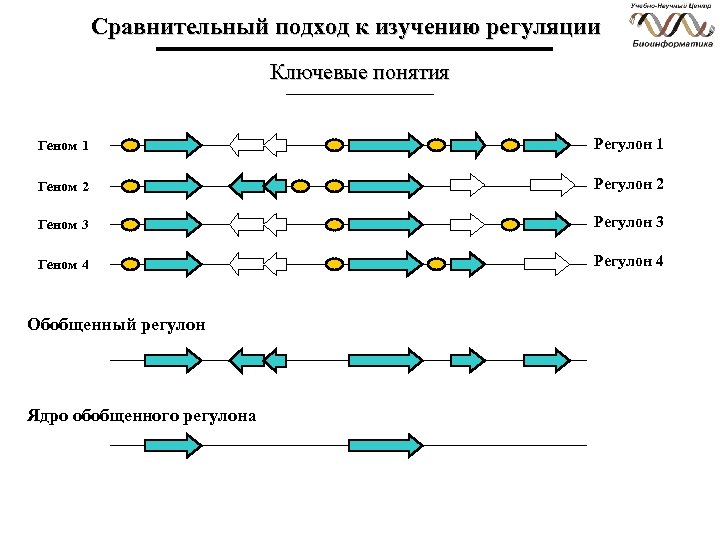 Сравнительный подход к изучению регуляции Ключевые понятия Геном 1 Регулон 1 Геном 2 Регулон 2 Геном 3 Регулон 3 Геном 4 Регулон 4 Обобщенный регулон Ядро обобщенного регулона
Сравнительный подход к изучению регуляции Ключевые понятия Геном 1 Регулон 1 Геном 2 Регулон 2 Геном 3 Регулон 3 Геном 4 Регулон 4 Обобщенный регулон Ядро обобщенного регулона
 Открытие Bio. Y, транспортера биотина Исследование обобщенного Bir. A-регулона v Биотин (витамин Н) – кофактор ферментов карбоксилаз и декарбоксилаз, служит переносчиком карбоксильных групп v Bir. A – фактор транскрипции, репрессор, регулятор генов биосинтеза биотина Rodionov D. A. , Mironov A. A. , Gelfand M. S. (2002) Conservation of the biotin regulon and the Bir. A regulatory signal in Eubacteria and Archaea. Genome Res. 12 (10). 1507 -1516. Исследование Bir. A-зависимой регуляции в прокариотах : ● ● Бациллы ● ● Клостридии ● ● Актинобактерии ● ● Зеленые серные бактерии ● Цианобактерии ● Археи
Открытие Bio. Y, транспортера биотина Исследование обобщенного Bir. A-регулона v Биотин (витамин Н) – кофактор ферментов карбоксилаз и декарбоксилаз, служит переносчиком карбоксильных групп v Bir. A – фактор транскрипции, репрессор, регулятор генов биосинтеза биотина Rodionov D. A. , Mironov A. A. , Gelfand M. S. (2002) Conservation of the biotin regulon and the Bir. A regulatory signal in Eubacteria and Archaea. Genome Res. 12 (10). 1507 -1516. Исследование Bir. A-зависимой регуляции в прокариотах : ● ● Бациллы ● ● Клостридии ● ● Актинобактерии ● ● Зеленые серные бактерии ● Цианобактерии ● Археи
 Открытие Bio. Y, транспортера биотина Исследование обобщенного Bir. A-регулона bio. Y – новый член обобщенного Bir. A-регулона ● имеет консервативные сайты связывания Bir. A во всех исследованных геномах ● не имеет гомологов с известной функцией ● в белках семейства Bio. Y обнаружено шесть потенциальных трансмембранных сегментов
Открытие Bio. Y, транспортера биотина Исследование обобщенного Bir. A-регулона bio. Y – новый член обобщенного Bir. A-регулона ● имеет консервативные сайты связывания Bir. A во всех исследованных геномах ● не имеет гомологов с известной функцией ● в белках семейства Bio. Y обнаружено шесть потенциальных трансмембранных сегментов
 Открытие Bio. Y, транспортера биотина Дополнительные соображения Хромосомные кластеры v гены располагаются в одном опероне или просто рядом на хромосоме v такое расположение сохраняестя в различных геномах bio. Y кластеризуется с : 1. cbi. O-cbi. Q – предполагаемые компоненты ABC-транспортной системы ( Указывает на то, что Bio. Y может выполнять транспортную функцию ) 2. Гены биосинтеза жирных кислот. ( Биотин является кофактором карбоксилаз, ферментов данного метаболического пути ) 3. bir. A.
Открытие Bio. Y, транспортера биотина Дополнительные соображения Хромосомные кластеры v гены располагаются в одном опероне или просто рядом на хромосоме v такое расположение сохраняестя в различных геномах bio. Y кластеризуется с : 1. cbi. O-cbi. Q – предполагаемые компоненты ABC-транспортной системы ( Указывает на то, что Bio. Y может выполнять транспортную функцию ) 2. Гены биосинтеза жирных кислот. ( Биотин является кофактором карбоксилаз, ферментов данного метаболического пути ) 3. bir. A.
 Цинк-зависимая регуляция паралогов рибосомных белков Паралоги рибосомных белков Makarova K. S. , Ponomarev V. A. , Koonin E. V. (2001) Two C or not two C: recurrent disruption of Zn-ribbons, gene duplication, lineage-specific gene loss, and horizontal gene transfer in evolution of bacterial ribosomal proteins. Genome Biology. 2 (9). RESEARCH 0033. q L 36, L 33, L 31, S 14 – единственные рибосомные белки, дуплицированные более, чем в одном геноме q L 36, L 33, L 31, S 14 – четыре из семи рибосомных белков, содержащих мотив цинковой ленты: четыре эволюционно консервативных цистеина (в некоторых случаях один из них может быть заменен на гистидин) q В случае наличия нескольких паралогов, одна из копий L 36, L 33, L 31, S 14 содержит мотив цинковой ленты, тогда как другие – нет
Цинк-зависимая регуляция паралогов рибосомных белков Паралоги рибосомных белков Makarova K. S. , Ponomarev V. A. , Koonin E. V. (2001) Two C or not two C: recurrent disruption of Zn-ribbons, gene duplication, lineage-specific gene loss, and horizontal gene transfer in evolution of bacterial ribosomal proteins. Genome Biology. 2 (9). RESEARCH 0033. q L 36, L 33, L 31, S 14 – единственные рибосомные белки, дуплицированные более, чем в одном геноме q L 36, L 33, L 31, S 14 – четыре из семи рибосомных белков, содержащих мотив цинковой ленты: четыре эволюционно консервативных цистеина (в некоторых случаях один из них может быть заменен на гистидин) q В случае наличия нескольких паралогов, одна из копий L 36, L 33, L 31, S 14 содержит мотив цинковой ленты, тогда как другие – нет
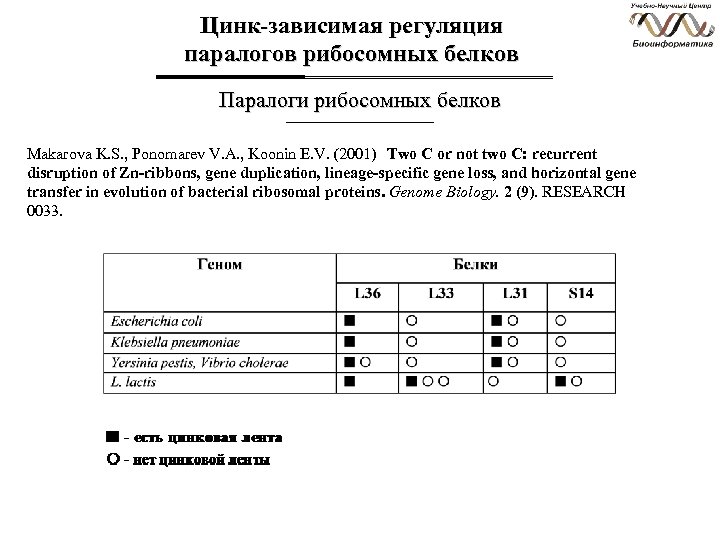 Цинк-зависимая регуляция паралогов рибосомных белков Паралоги рибосомных белков Makarova K. S. , Ponomarev V. A. , Koonin E. V. (2001) Two C or not two C: recurrent disruption of Zn-ribbons, gene duplication, lineage-specific gene loss, and horizontal gene transfer in evolution of bacterial ribosomal proteins. Genome Biology. 2 (9). RESEARCH 0033.
Цинк-зависимая регуляция паралогов рибосомных белков Паралоги рибосомных белков Makarova K. S. , Ponomarev V. A. , Koonin E. V. (2001) Two C or not two C: recurrent disruption of Zn-ribbons, gene duplication, lineage-specific gene loss, and horizontal gene transfer in evolution of bacterial ribosomal proteins. Genome Biology. 2 (9). RESEARCH 0033.
 Цинк-зависимая регуляция паралогов рибосомных белков Panina E. M. , Mironov A. A. , Gelfand M. S. (2003) Comparative genomics of bacterial zinc regulons: enhanced ion transport, pathogenesis, and rearrangement of ribosomal proteins. Proc. Natl. Acad. Sci. U. S. A. 100 (17). 9912 -9917 Исследование цинк-зависимой регуляции:
Цинк-зависимая регуляция паралогов рибосомных белков Panina E. M. , Mironov A. A. , Gelfand M. S. (2003) Comparative genomics of bacterial zinc regulons: enhanced ion transport, pathogenesis, and rearrangement of ribosomal proteins. Proc. Natl. Acad. Sci. U. S. A. 100 (17). 9912 -9917 Исследование цинк-зависимой регуляции:
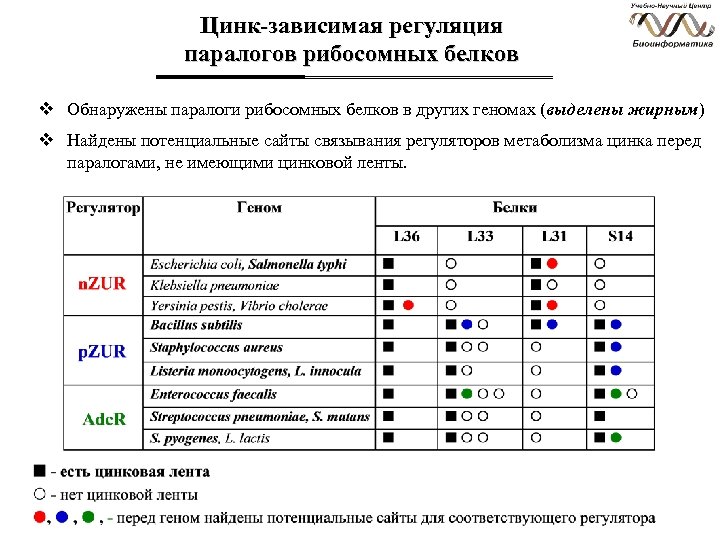 Цинк-зависимая регуляция паралогов рибосомных белков v Обнаружены паралоги рибосомных белков в других геномах (выделены жирным) v Найдены потенциальные сайты связывания регуляторов метаболизма цинка перед паралогами, не имеющими цинковой ленты.
Цинк-зависимая регуляция паралогов рибосомных белков v Обнаружены паралоги рибосомных белков в других геномах (выделены жирным) v Найдены потенциальные сайты связывания регуляторов метаболизма цинка перед паралогами, не имеющими цинковой ленты.
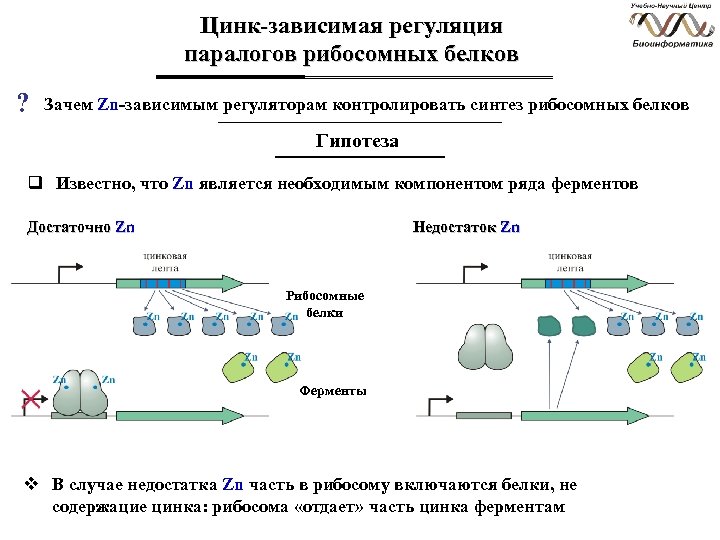 Цинк-зависимая регуляция паралогов рибосомных белков ? Зачем Zn-зависимым регуляторам контролировать синтез рибосомных белков Гипотеза q Известно, что Zn является необходимым компонентом ряда ферментов Достаточно Zn Недостаток Zn Рибосомные белки Ферменты v В случае недостатка Zn часть в рибосому включаются белки, не содержацие цинка: рибосома «отдает» часть цинка ферментам
Цинк-зависимая регуляция паралогов рибосомных белков ? Зачем Zn-зависимым регуляторам контролировать синтез рибосомных белков Гипотеза q Известно, что Zn является необходимым компонентом ряда ферментов Достаточно Zn Недостаток Zn Рибосомные белки Ферменты v В случае недостатка Zn часть в рибосому включаются белки, не содержацие цинка: рибосома «отдает» часть цинка ферментам
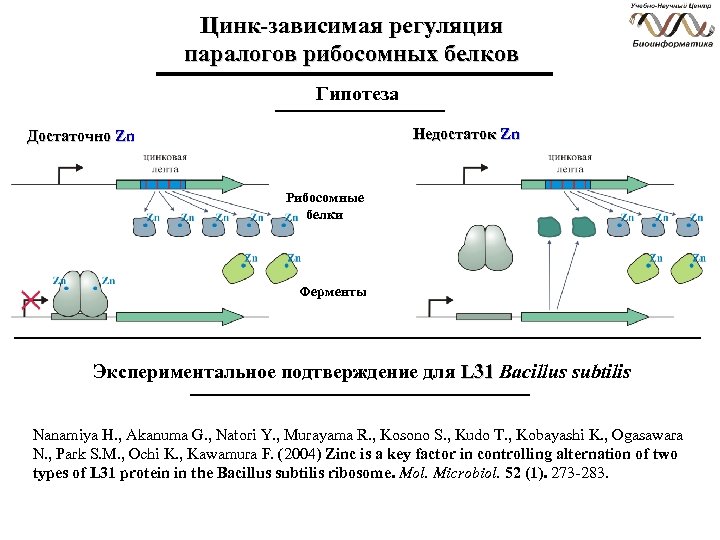 Цинк-зависимая регуляция паралогов рибосомных белков Гипотеза Недостаток Zn Достаточно Zn Рибосомные белки Ферменты Экспериментальное подтверждение для L 31 Bacillus subtilis Nanamiya H. , Akanuma G. , Natori Y. , Murayama R. , Kosono S. , Kudo T. , Kobayashi K. , Ogasawara N. , Park S. M. , Ochi K. , Kawamura F. (2004) Zinc is a key factor in controlling alternation of two types of L 31 protein in the Bacillus subtilis ribosome. Mol. Microbiol. 52 (1). 273 -283.
Цинк-зависимая регуляция паралогов рибосомных белков Гипотеза Недостаток Zn Достаточно Zn Рибосомные белки Ферменты Экспериментальное подтверждение для L 31 Bacillus subtilis Nanamiya H. , Akanuma G. , Natori Y. , Murayama R. , Kosono S. , Kudo T. , Kobayashi K. , Ogasawara N. , Park S. M. , Ochi K. , Kawamura F. (2004) Zinc is a key factor in controlling alternation of two types of L 31 protein in the Bacillus subtilis ribosome. Mol. Microbiol. 52 (1). 273 -283.
 Биосинтез лейцина в дрожжах v Начинается в митохондрии, заканчивается в цитоплазме v Транспортер промежуточного продукта, изопропилмалата, не известен Кандидат: YOR 271 cp q Локализован в митохондрии q Предсказаны 4 трансмембранных сегмента q Имеется консервативный сайт связывания лейцинового регулятора Leu 3 p q Регуляторная область YOR 271 c связывает Leu 3 p в Ch. IP-chip (хотя чувствительность и специфичность эксперимента ~50%, других кандидатов с эксперименте консервативными сайтами нет) q Гомологичен транспортеру трикарбоксилатов крысы
Биосинтез лейцина в дрожжах v Начинается в митохондрии, заканчивается в цитоплазме v Транспортер промежуточного продукта, изопропилмалата, не известен Кандидат: YOR 271 cp q Локализован в митохондрии q Предсказаны 4 трансмембранных сегмента q Имеется консервативный сайт связывания лейцинового регулятора Leu 3 p q Регуляторная область YOR 271 c связывает Leu 3 p в Ch. IP-chip (хотя чувствительность и специфичность эксперимента ~50%, других кандидатов с эксперименте консервативными сайтами нет) q Гомологичен транспортеру трикарбоксилатов крысы
 Изучение эволюции регуляции Историк - это вспять обращенный пророк Г. Гегель Однажды Гегель ненароком И, вероятно, наугад Назвал историка пророком, Предсказывающим назад Б. Пастернак v Предсказание новых членов регулона способствует пониманию устройства прокариотической клетки v Изучение эволюции регуляторных систем
Изучение эволюции регуляции Историк - это вспять обращенный пророк Г. Гегель Однажды Гегель ненароком И, вероятно, наугад Назвал историка пророком, Предсказывающим назад Б. Пастернак v Предсказание новых членов регулона способствует пониманию устройства прокариотической клетки v Изучение эволюции регуляторных систем
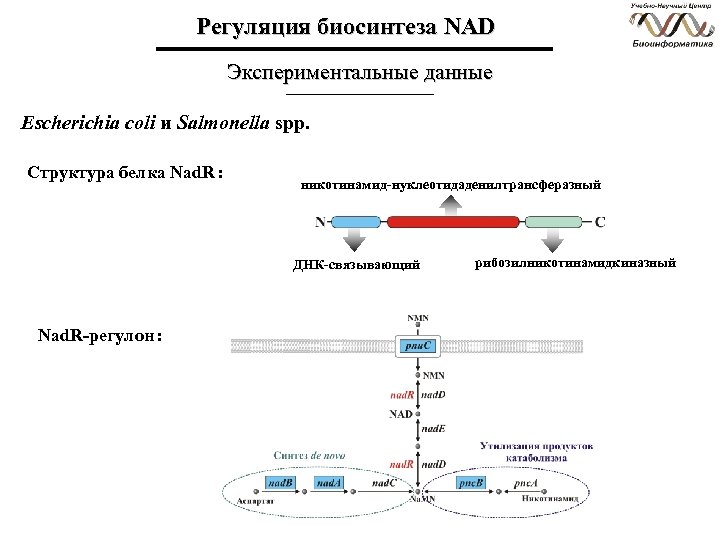 Регуляция биосинтеза NAD Экспериментальные данные Escherichia coli и Salmonella spp. Структура белка Nad. R : никотинамид-нуклеотидаденилтрансферазный ДНК-связывающий Nad. R-регулон : рибозилникотинамидкиназный
Регуляция биосинтеза NAD Экспериментальные данные Escherichia coli и Salmonella spp. Структура белка Nad. R : никотинамид-нуклеотидаденилтрансферазный ДНК-связывающий Nad. R-регулон : рибозилникотинамидкиназный
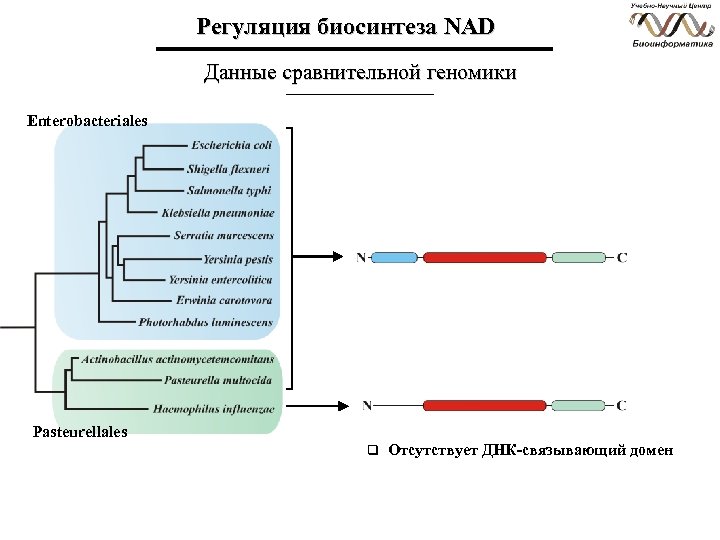 Регуляция биосинтеза NAD Данные сравнительной геномики Enterobacteriales Pasteurellales q Отсутствует ДНК-связывающий домен
Регуляция биосинтеза NAD Данные сравнительной геномики Enterobacteriales Pasteurellales q Отсутствует ДНК-связывающий домен
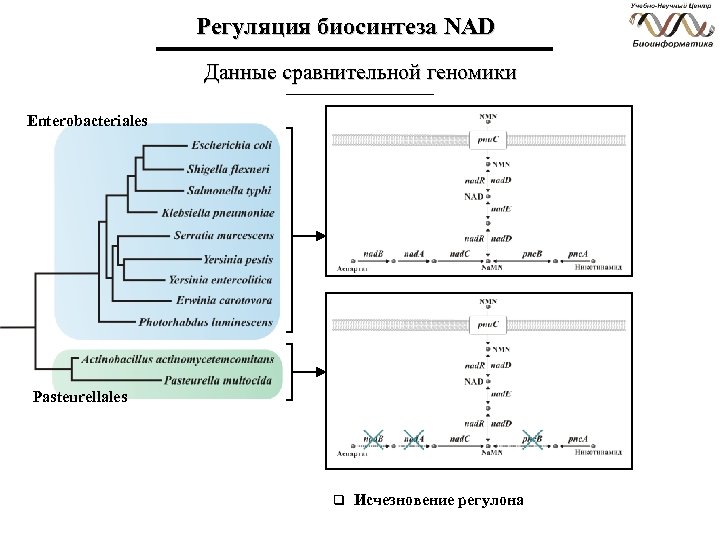 Регуляция биосинтеза NAD Данные сравнительной геномики Enterobacteriales Pasteurellales q Исчезновение регулона
Регуляция биосинтеза NAD Данные сравнительной геномики Enterobacteriales Pasteurellales q Исчезновение регулона
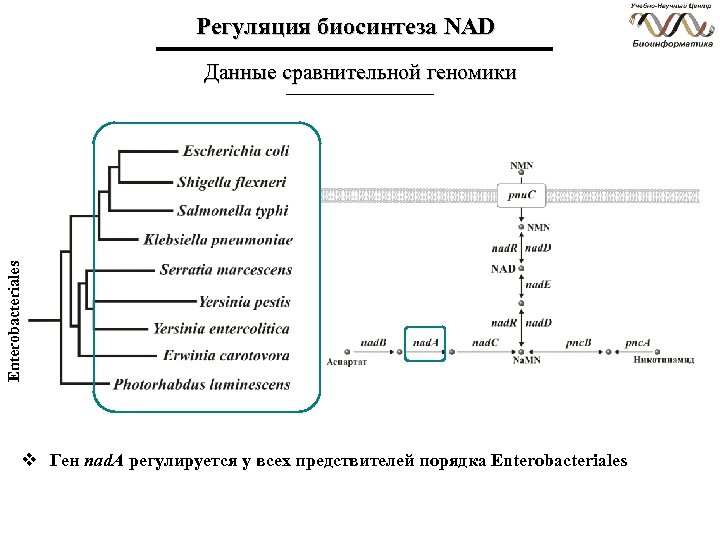 Регуляция биосинтеза NAD Enterobacteriales Данные сравнительной геномики v Ген nad. A регулируется у всех предствителей порядка Enterobacteriales
Регуляция биосинтеза NAD Enterobacteriales Данные сравнительной геномики v Ген nad. A регулируется у всех предствителей порядка Enterobacteriales
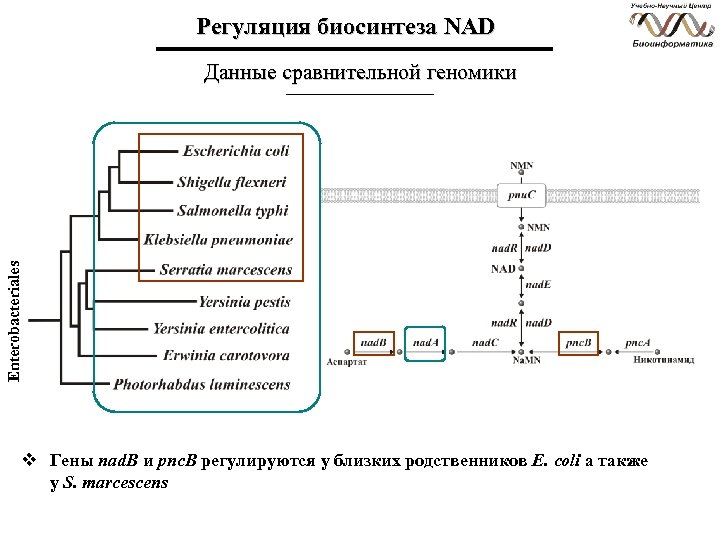 Регуляция биосинтеза NAD Enterobacteriales Данные сравнительной геномики v Гены nad. B и pnc. B регулируются у близких родственников E. coli а также у S. marcescens
Регуляция биосинтеза NAD Enterobacteriales Данные сравнительной геномики v Гены nad. B и pnc. B регулируются у близких родственников E. coli а также у S. marcescens
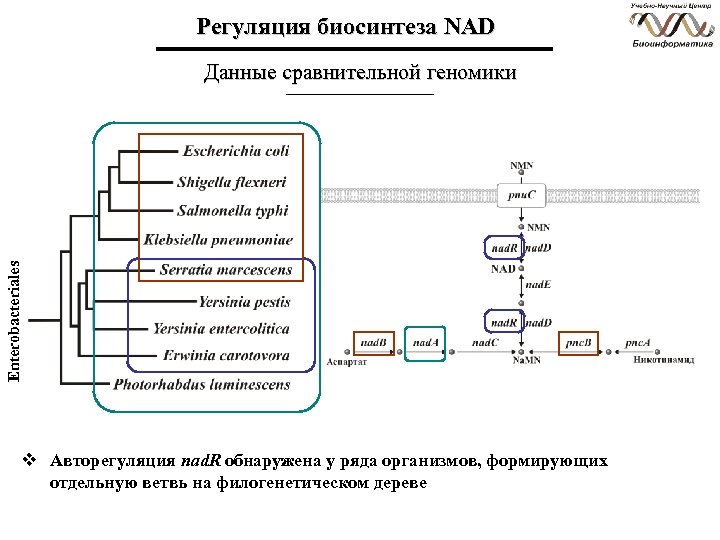 Регуляция биосинтеза NAD Enterobacteriales Данные сравнительной геномики v Авторегуляция nad. R обнаружена у ряда организмов, формирующих отдельную ветвь на филогенетическом дереве
Регуляция биосинтеза NAD Enterobacteriales Данные сравнительной геномики v Авторегуляция nad. R обнаружена у ряда организмов, формирующих отдельную ветвь на филогенетическом дереве
 Регуляция биосинтеза NAD Модель эволюции I. Появление нового регулятора 1. Pasteurellales – регулон отсутствует, иногда белок не имеет ДНКсвязывающего домена 2. Enterobacteriales – белок используется в качестве фактора транскрипции a) Регуляция гена nad. A b 1) Регуляция генов nad. B и pnc. B b 2) Авторегуляция гена nad. R v Регуляторные взаимодействия могут менятся даже в близкородственных геномах и даже в случае регуляции важных метаболических путей Gerasimova A. V. , Gelfand M. S. (2005) Evolution of the Nad. R regulon in Enterobacteriaceae. J. Bioinform. Comput. Biol. 3(4). 1007 -1019.
Регуляция биосинтеза NAD Модель эволюции I. Появление нового регулятора 1. Pasteurellales – регулон отсутствует, иногда белок не имеет ДНКсвязывающего домена 2. Enterobacteriales – белок используется в качестве фактора транскрипции a) Регуляция гена nad. A b 1) Регуляция генов nad. B и pnc. B b 2) Авторегуляция гена nad. R v Регуляторные взаимодействия могут менятся даже в близкородственных геномах и даже в случае регуляции важных метаболических путей Gerasimova A. V. , Gelfand M. S. (2005) Evolution of the Nad. R regulon in Enterobacteriaceae. J. Bioinform. Comput. Biol. 3(4). 1007 -1019.
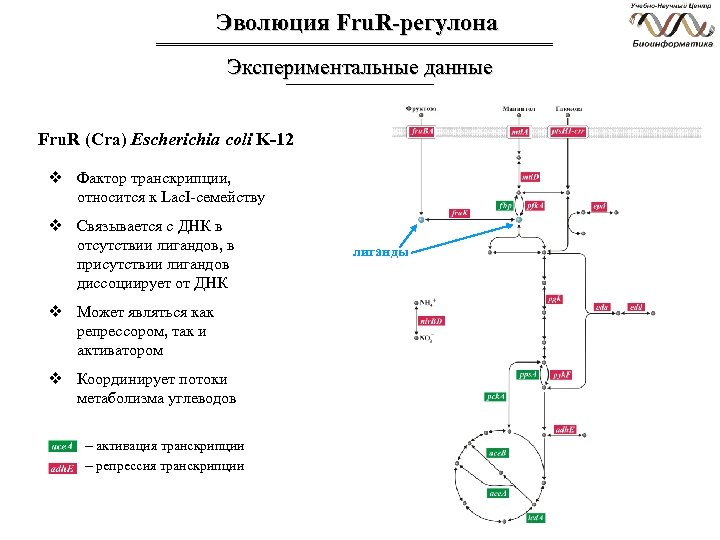 Эволюция Fru. R-регулона Экспериментальные данные Fru. R (Cra) Escherichia coli K-12 v Фактор транскрипции, относится к Lac. I-семейству v Связывается с ДНК в отсутствии лигандов, в присутствии лигандов диссоциирует от ДНК v Может являться как репрессором, так и активатором v Координирует потоки метаболизма углеводов - активация транскрипции - репрессия транскрипции лиганды
Эволюция Fru. R-регулона Экспериментальные данные Fru. R (Cra) Escherichia coli K-12 v Фактор транскрипции, относится к Lac. I-семейству v Связывается с ДНК в отсутствии лигандов, в присутствии лигандов диссоциирует от ДНК v Может являться как репрессором, так и активатором v Координирует потоки метаболизма углеводов - активация транскрипции - репрессия транскрипции лиганды
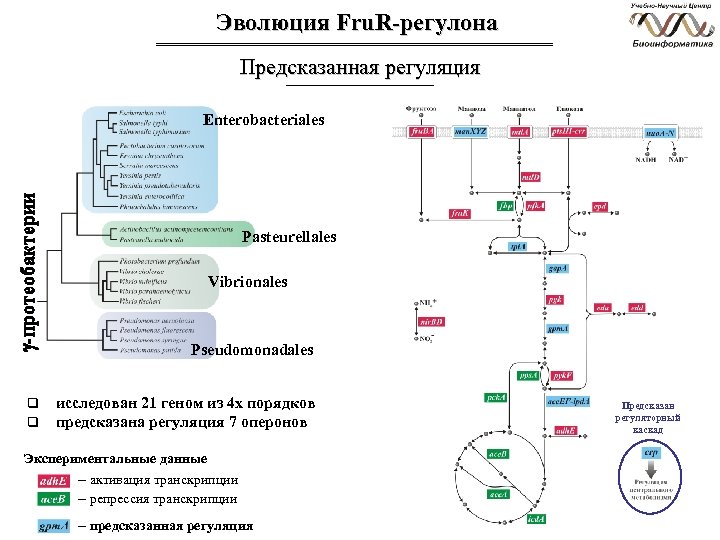 Эволюция Fru. R-регулона Предсказанная регуляция Enterobacteriales Pasteurellales Vibrionales Pseudomonadales q q исследован 21 геном из 4 х порядков предсказана регуляция 7 оперонов Экспериментальные данные - активация транскрипции - репрессия транскрипции - предсказанная регуляция Предсказан регуляторный каскад
Эволюция Fru. R-регулона Предсказанная регуляция Enterobacteriales Pasteurellales Vibrionales Pseudomonadales q q исследован 21 геном из 4 х порядков предсказана регуляция 7 оперонов Экспериментальные данные - активация транскрипции - репрессия транскрипции - предсказанная регуляция Предсказан регуляторный каскад
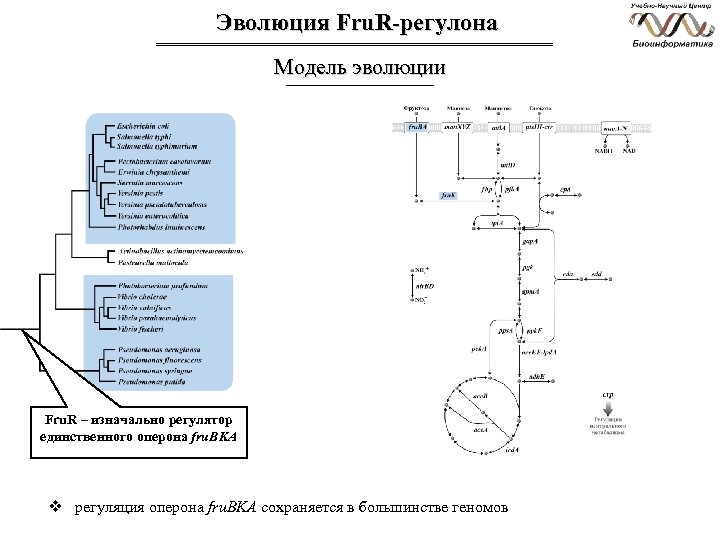 Эволюция Fru. R-регулона Модель эволюции Fru. R – изначально регулятор единственного оперона fru. BKA v регуляция оперона fru. BKA сохраняется в большинстве геномов
Эволюция Fru. R-регулона Модель эволюции Fru. R – изначально регулятор единственного оперона fru. BKA v регуляция оперона fru. BKA сохраняется в большинстве геномов
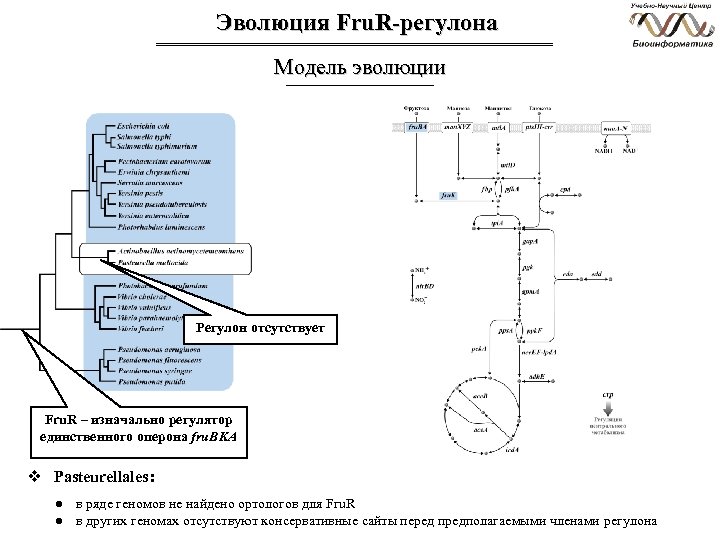 Эволюция Fru. R-регулона Модель эволюции Регулон отсутствует Fru. R – изначально регулятор единственного оперона fru. BKA v Pasteurellales : ● в ряде геномов не найдено ортологов для Fru. R ● в других геномах отсутствуют консервативные сайты перед предполагаемыми членами регулона
Эволюция Fru. R-регулона Модель эволюции Регулон отсутствует Fru. R – изначально регулятор единственного оперона fru. BKA v Pasteurellales : ● в ряде геномов не найдено ортологов для Fru. R ● в других геномах отсутствуют консервативные сайты перед предполагаемыми членами регулона
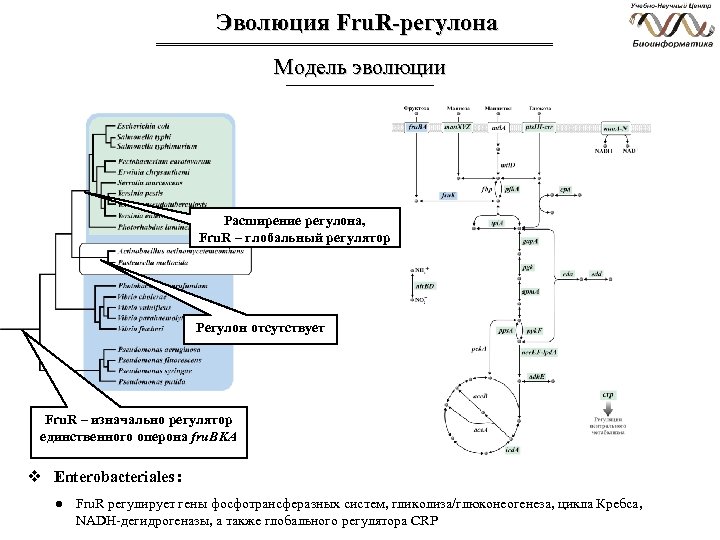 Эволюция Fru. R-регулона Модель эволюции Расширение регулона, Fru. R – глобальный регулятор Регулон отсутствует Fru. R – изначально регулятор единственного оперона fru. BKA v Enterobacteriales : ● Fru. R регулирует гены фосфотрансферазных систем, гликолиза/глюконеогенеза, цикла Кребса, NADH-дегидрогеназы, а также глобального регулятора CRP
Эволюция Fru. R-регулона Модель эволюции Расширение регулона, Fru. R – глобальный регулятор Регулон отсутствует Fru. R – изначально регулятор единственного оперона fru. BKA v Enterobacteriales : ● Fru. R регулирует гены фосфотрансферазных систем, гликолиза/глюконеогенеза, цикла Кребса, NADH-дегидрогеназы, а также глобального регулятора CRP
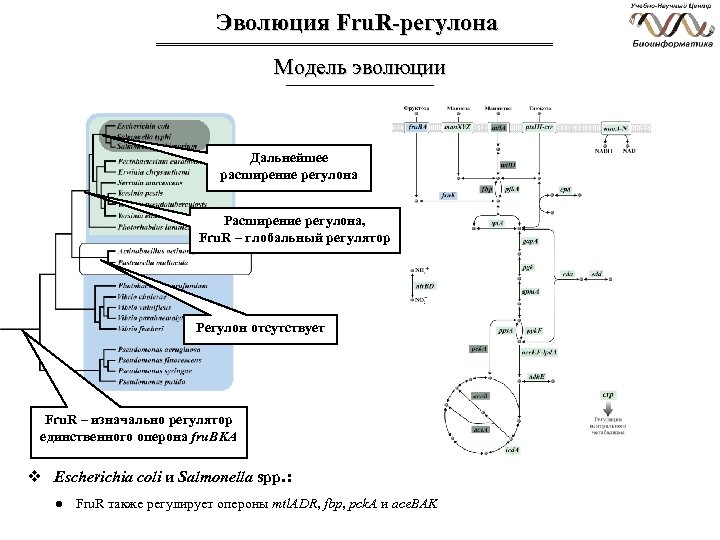 Эволюция Fru. R-регулона Модель эволюции Дальнейшее расширение регулона Расширение регулона, Fru. R – глобальный регулятор Регулон отсутствует Fru. R – изначально регулятор единственного оперона fru. BKA v Escherichia coli и Salmonella spp. : ● Fru. R также регулирует опероны mtl. ADR, fbp, pck. A и ace. BAK
Эволюция Fru. R-регулона Модель эволюции Дальнейшее расширение регулона Расширение регулона, Fru. R – глобальный регулятор Регулон отсутствует Fru. R – изначально регулятор единственного оперона fru. BKA v Escherichia coli и Salmonella spp. : ● Fru. R также регулирует опероны mtl. ADR, fbp, pck. A и ace. BAK
 Новые тенденции в сравнительной геномике 1. Исследование нескольких функционально связанных регуляторных систем Ø Восстановление оксидов азота в различных группах бактерий Rodionov D. A. , Dubchak I. L. , Arkin A. P. , Alm E. J. , Gelfand M. S. (2005) Dissimilatory metabolism of nitrogen oxides in bacteria: comparative reconstruction of transcriptional networks. PLo. S Comput. Biol. 1. e 55. Ravcheev D. A. , Gerasimova A. V. , Mironov A. A, Gelfand M. S. Complex comparative genomic analysis of respiration regulation in gamma-proteobacteria. Genome Biol. [in press] 2. Изучение таксон-специфической регуляции v Сравнение геномов организмов, относящихся к одной таксономической группе v Развитию методики способствует увеличение количества полных геномных последовательностей
Новые тенденции в сравнительной геномике 1. Исследование нескольких функционально связанных регуляторных систем Ø Восстановление оксидов азота в различных группах бактерий Rodionov D. A. , Dubchak I. L. , Arkin A. P. , Alm E. J. , Gelfand M. S. (2005) Dissimilatory metabolism of nitrogen oxides in bacteria: comparative reconstruction of transcriptional networks. PLo. S Comput. Biol. 1. e 55. Ravcheev D. A. , Gerasimova A. V. , Mironov A. A, Gelfand M. S. Complex comparative genomic analysis of respiration regulation in gamma-proteobacteria. Genome Biol. [in press] 2. Изучение таксон-специфической регуляции v Сравнение геномов организмов, относящихся к одной таксономической группе v Развитию методики способствует увеличение количества полных геномных последовательностей
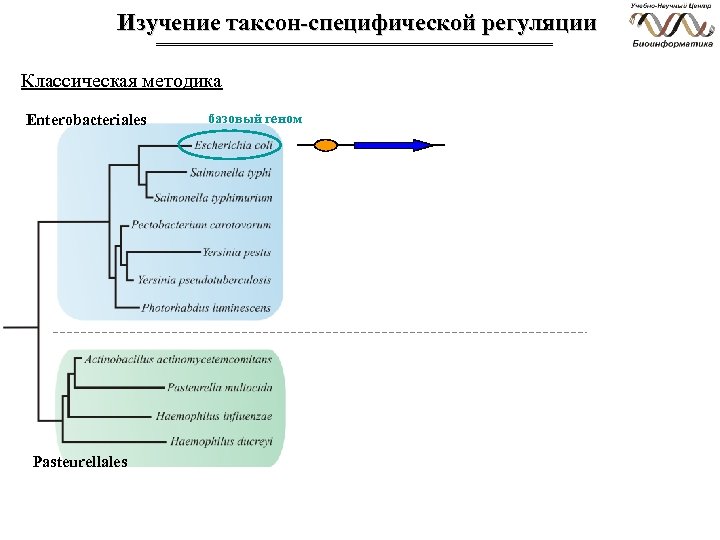 Изучение таксон-специфической регуляции Классическая методика Enterobacteriales Pasteurellales базовый геном
Изучение таксон-специфической регуляции Классическая методика Enterobacteriales Pasteurellales базовый геном
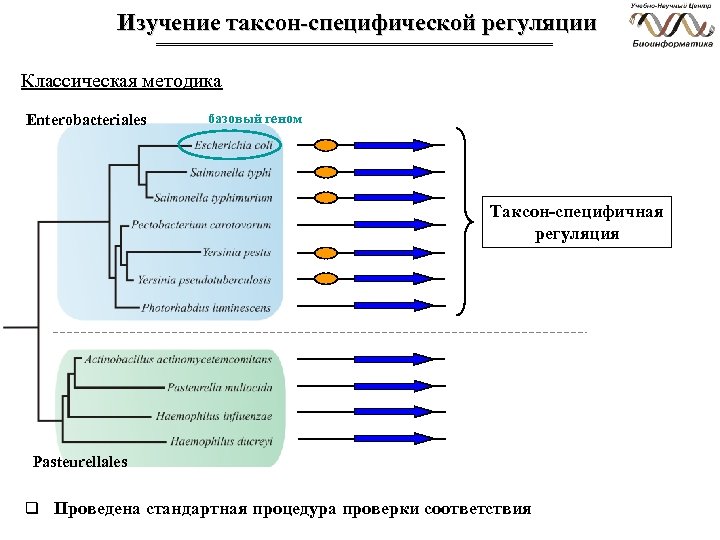 Изучение таксон-специфической регуляции Классическая методика Enterobacteriales базовый геном Таксон-специфичная регуляция Pasteurellales q Проведена стандартная процедура проверки соответствия
Изучение таксон-специфической регуляции Классическая методика Enterobacteriales базовый геном Таксон-специфичная регуляция Pasteurellales q Проведена стандартная процедура проверки соответствия
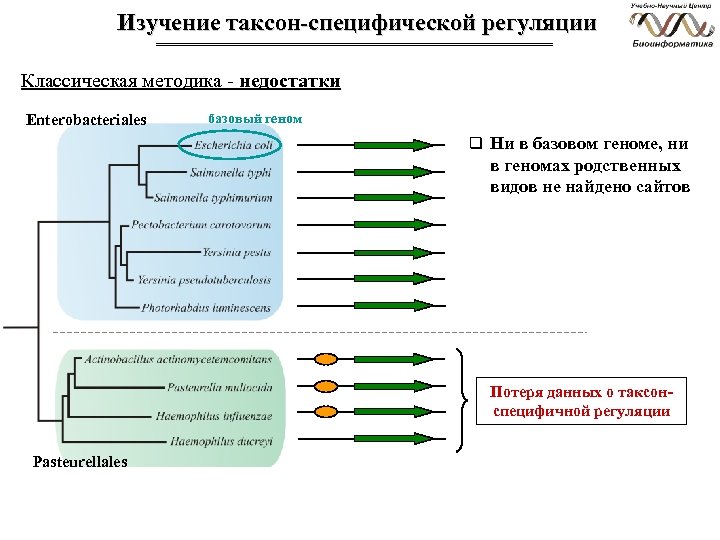 Изучение таксон-специфической регуляции Классическая методика - недостатки Enterobacteriales базовый геном q Ни в базовом геноме, ни в геномах родственных видов не найдено сайтов Потеря данных о таксонспецифичной регуляции Pasteurellales
Изучение таксон-специфической регуляции Классическая методика - недостатки Enterobacteriales базовый геном q Ни в базовом геноме, ни в геномах родственных видов не найдено сайтов Потеря данных о таксонспецифичной регуляции Pasteurellales
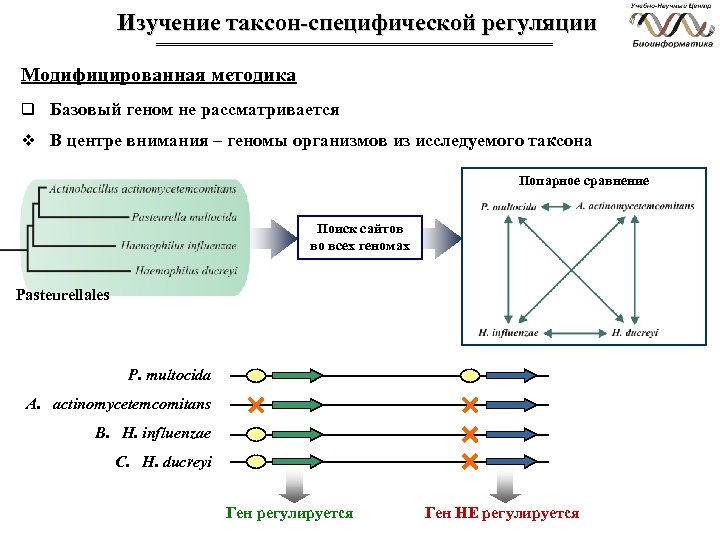 Изучение таксон-специфической регуляции Модифицированная методика q Базовый геном не рассматривается v В центре внимания – геномы организмов из исследуемого таксона Попарное сравнение Поиск сайтов во всех геномах Pasteurellales P. multocida A. actinomycetemcomitans B. H. influenzae C. H. ducreyi Ген регулируется Ген НЕ регулируется
Изучение таксон-специфической регуляции Модифицированная методика q Базовый геном не рассматривается v В центре внимания – геномы организмов из исследуемого таксона Попарное сравнение Поиск сайтов во всех геномах Pasteurellales P. multocida A. actinomycetemcomitans B. H. influenzae C. H. ducreyi Ген регулируется Ген НЕ регулируется
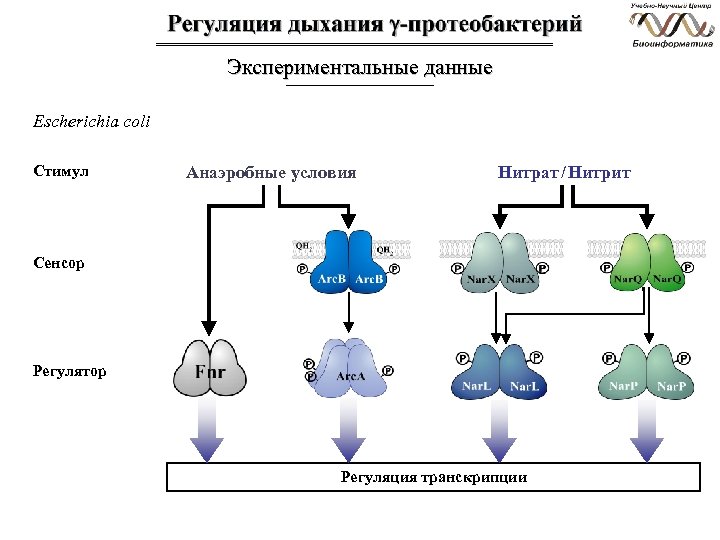 Экспериментальные данные Escherichia coli Стимул Анаэробные условия Нитрат / Нитрит Сенсор Регулятор Регуляция транскрипции
Экспериментальные данные Escherichia coli Стимул Анаэробные условия Нитрат / Нитрит Сенсор Регулятор Регуляция транскрипции
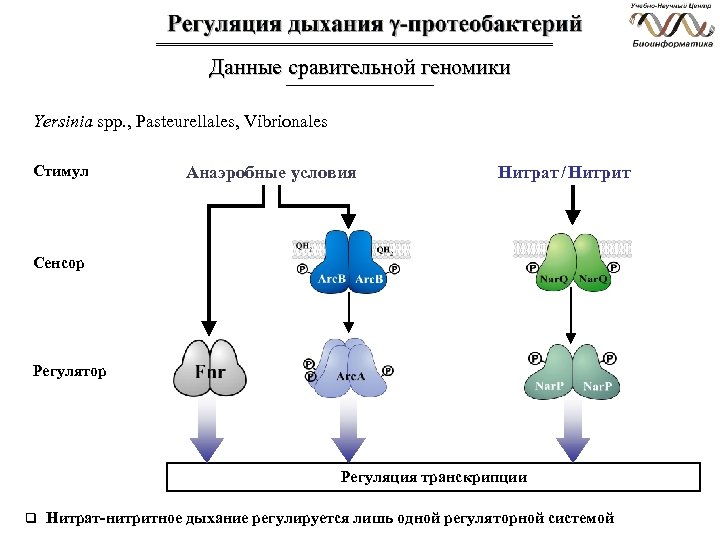 Данные сравительной геномики Yersinia spp. , Pasteurellales, Vibrionales Стимул Анаэробные условия Нитрат / Нитрит Сенсор Регулятор Регуляция транскрипции q Нитрат-нитритное дыхание регулируется лишь одной регуляторной системой
Данные сравительной геномики Yersinia spp. , Pasteurellales, Vibrionales Стимул Анаэробные условия Нитрат / Нитрит Сенсор Регулятор Регуляция транскрипции q Нитрат-нитритное дыхание регулируется лишь одной регуляторной системой
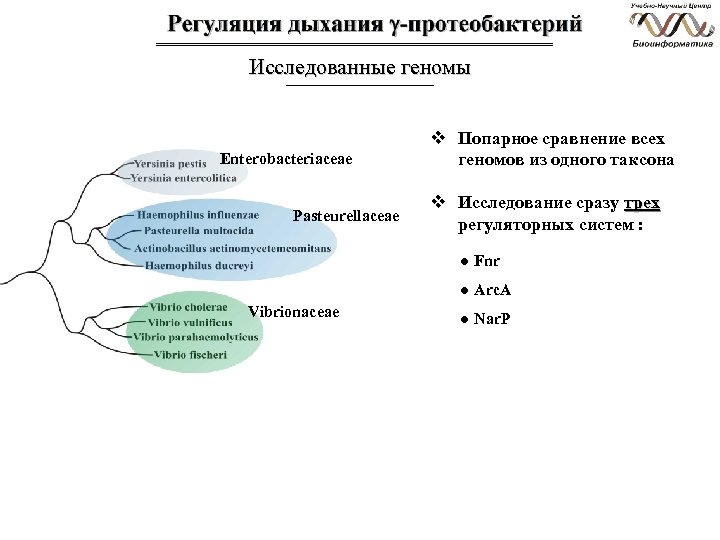 Исследованные геномы Enterobacteriaceae Pasteurellaceae v Попарное сравнение всех геномов из одного таксона v Исследование сразу трех регуляторных систем : ● Fnr ● Arc. A Vibrionaceae ● Nar. P
Исследованные геномы Enterobacteriaceae Pasteurellaceae v Попарное сравнение всех геномов из одного таксона v Исследование сразу трех регуляторных систем : ● Fnr ● Arc. A Vibrionaceae ● Nar. P
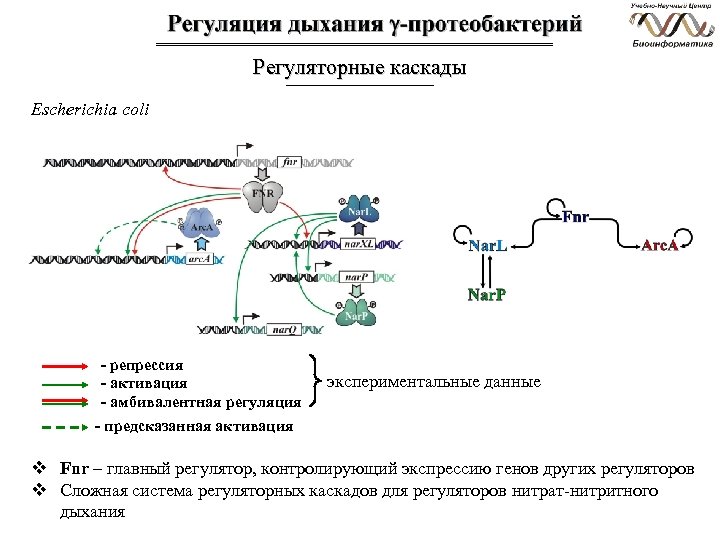 Регуляторные каскады Escherichia coli - репрессия - активация - амбивалентная регуляция экспериментальные данные - предсказанная активация v Fnr – главный регулятор, контролирующий экспрессию генов других регуляторов v Сложная система регуляторных каскадов для регуляторов нитрат-нитритного дыхания
Регуляторные каскады Escherichia coli - репрессия - активация - амбивалентная регуляция экспериментальные данные - предсказанная активация v Fnr – главный регулятор, контролирующий экспрессию генов других регуляторов v Сложная система регуляторных каскадов для регуляторов нитрат-нитритного дыхания
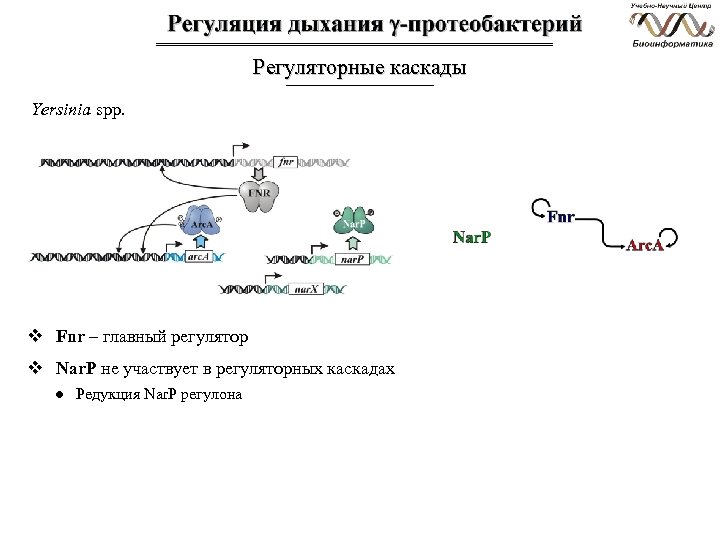 Регуляторные каскады Yersinia spp. v Fnr – главный регулятор v Nar. P не участвует в регуляторных каскадах ● Редукция Nar. P регулона
Регуляторные каскады Yersinia spp. v Fnr – главный регулятор v Nar. P не участвует в регуляторных каскадах ● Редукция Nar. P регулона
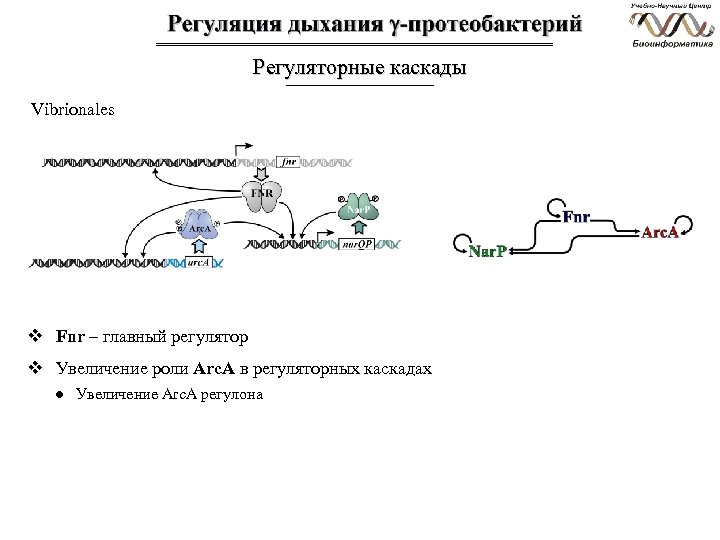 Регуляторные каскады Vibrionales v Fnr – главный регулятор v Увеличение роли Arc. A в регуляторных каскадах ● Увеличение Arc. A регулона
Регуляторные каскады Vibrionales v Fnr – главный регулятор v Увеличение роли Arc. A в регуляторных каскадах ● Увеличение Arc. A регулона
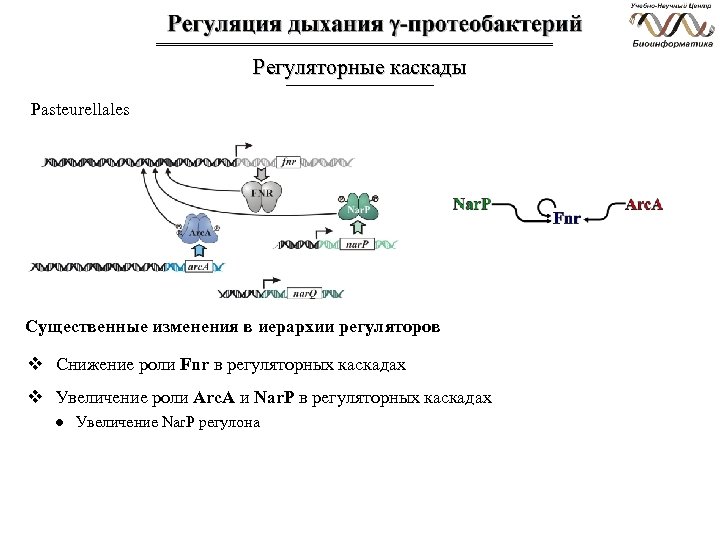 Регуляторные каскады Pasteurellales Существенные изменения в иерархии регуляторов v Снижение роли Fnr в регуляторных каскадах v Увеличение роли Arc. A и Nar. P в регуляторных каскадах ● Увеличение Nar. P регулона
Регуляторные каскады Pasteurellales Существенные изменения в иерархии регуляторов v Снижение роли Fnr в регуляторных каскадах v Увеличение роли Arc. A и Nar. P в регуляторных каскадах ● Увеличение Nar. P регулона
 Выводы 1. В ходе эволюции регуляторные взаимодействия могут существенно изменяться 2. Данные изменения носят таксон-специфичный характер 3. Изменения в структуре регуляторных каскадов коррелируют с изменениями в структуре регулонов
Выводы 1. В ходе эволюции регуляторные взаимодействия могут существенно изменяться 2. Данные изменения носят таксон-специфичный характер 3. Изменения в структуре регуляторных каскадов коррелируют с изменениями в структуре регулонов
 Благодарности Гельфанд М. С. Герасимова А. В. Миронов А. А. Родионов Д. А. Рахманинова А. Б Панина Е. М. Закирзянова В. Спасибо за внимание !
Благодарности Гельфанд М. С. Герасимова А. В. Миронов А. А. Родионов Д. А. Рахманинова А. Б Панина Е. М. Закирзянова В. Спасибо за внимание !


