 Chapter 9 / Глава 9 Structural Genomics / Структурная геномика Determining the three-dimensional structure of proteins and RNA/ определение пространственной (3 D) структуры белков и РНК © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Chapter 9 / Глава 9 Structural Genomics / Структурная геномика Determining the three-dimensional structure of proteins and RNA/ определение пространственной (3 D) структуры белков и РНК © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Contents/ Содержание · The biology, chemistry, and physics of proteins/ Биология, химия и физика белков · Experimental methods for determining protein structure/ Экспериментальные методы определения белковой структуры · Computer modeling techniques for predicting the structure of proteins/ Техники омпьютерного моделирования для прогнозирования белковой структуры · High-throughput methods for structural genomics/ Высокопропускные методы структуральной геномики · RNA structural genomics/ Структура РНК © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Contents/ Содержание · The biology, chemistry, and physics of proteins/ Биология, химия и физика белков · Experimental methods for determining protein structure/ Экспериментальные методы определения белковой структуры · Computer modeling techniques for predicting the structure of proteins/ Техники омпьютерного моделирования для прогнозирования белковой структуры · High-throughput methods for structural genomics/ Высокопропускные методы структуральной геномики · RNA structural genomics/ Структура РНК © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Aim of structural genomics/ Цель структурной геномики · To match the structure of protein and RNA molecules to their respective DNA sequences/ изучить структуру белков и молекул РНК · Presently, all major efforts are concentrating on proteins because the structure of RNA molecules is poorly understood/ На сегодняшний день, преимущественные усилия направлены на белки, поскольку структура молекул РНК плохо изучена · Initial efforts will focus mostly on soluble proteins/ Прежде всего изучаются растворимые белки © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Aim of structural genomics/ Цель структурной геномики · To match the structure of protein and RNA molecules to their respective DNA sequences/ изучить структуру белков и молекул РНК · Presently, all major efforts are concentrating on proteins because the structure of RNA molecules is poorly understood/ На сегодняшний день, преимущественные усилия направлены на белки, поскольку структура молекул РНК плохо изучена · Initial efforts will focus mostly on soluble proteins/ Прежде всего изучаются растворимые белки © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 The importance of protein structure/ Значимость белковой структуры · Structure implies function/ Структура определяет функцию · A membrane-bound ion channel looks like a pore/ Белки йонных каналов, связанные с мембраной, имеют вид поры. · Match between ligand size and ligand-binding site/ Соотношение размера лиганда с сайтом его связывания · Practical implications/ Практическое применение · Drug design / «Дизайн» лекарств (имеется в виду, создание оптимально подходящих лекарств) The amprenavir protease inhibitor binding to the HIV protease © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The importance of protein structure/ Значимость белковой структуры · Structure implies function/ Структура определяет функцию · A membrane-bound ion channel looks like a pore/ Белки йонных каналов, связанные с мембраной, имеют вид поры. · Match between ligand size and ligand-binding site/ Соотношение размера лиганда с сайтом его связывания · Practical implications/ Практическое применение · Drug design / «Дизайн» лекарств (имеется в виду, создание оптимально подходящих лекарств) The amprenavir protease inhibitor binding to the HIV protease © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
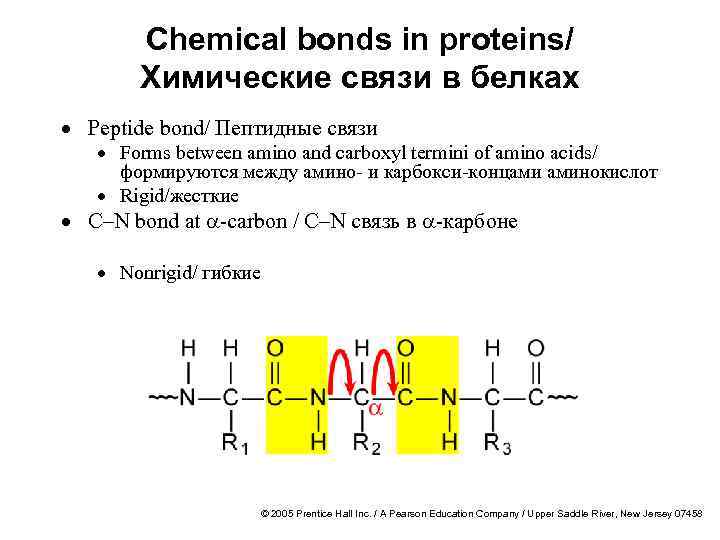 Chemical bonds in proteins/ Химические связи в белках · Peptide bond/ Пептидные связи · Forms between amino and carboxyl termini of amino acids/ формируются между амино- и карбокси-концами аминокислот · Rigid/жесткие · C–N bond at a-carbon / C–N связь в a-карбоне · Nonrigid/ гибкие © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Chemical bonds in proteins/ Химические связи в белках · Peptide bond/ Пептидные связи · Forms between amino and carboxyl termini of amino acids/ формируются между амино- и карбокси-концами аминокислот · Rigid/жесткие · C–N bond at a-carbon / C–N связь в a-карбоне · Nonrigid/ гибкие © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
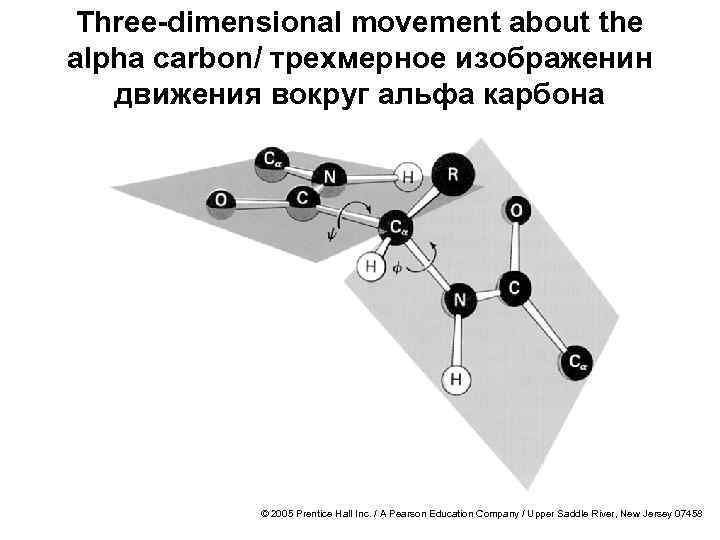 Three-dimensional movement about the alpha carbon/ трехмерное изображенин движения вокруг альфа карбона © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Three-dimensional movement about the alpha carbon/ трехмерное изображенин движения вокруг альфа карбона © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 The physics of proteins/ Физика белков · External forces acting on proteins/ внешние силы, которые действуют на белки · Solvation/ Сольватации (оно же гидратации, кстати) · Membrane components for membrane-embedded proteins/ Действие мембранных компонентов на белки, связанные с мембраной · Internal forces/ Внутренние силы: · Disulfide bridging/ Дисульфидные мостики · Hydrogen bonding / Связывание водорода · Dipolar interactions/ Диполярные взаимодействия · Van der Waals attraction/ Ван-дер-Ваальсовые взаимодействия · Protein folding/ Укладка белка · Affected by all of the foregoing forces/ определяется вышеперечисленными воздействиями © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The physics of proteins/ Физика белков · External forces acting on proteins/ внешние силы, которые действуют на белки · Solvation/ Сольватации (оно же гидратации, кстати) · Membrane components for membrane-embedded proteins/ Действие мембранных компонентов на белки, связанные с мембраной · Internal forces/ Внутренние силы: · Disulfide bridging/ Дисульфидные мостики · Hydrogen bonding / Связывание водорода · Dipolar interactions/ Диполярные взаимодействия · Van der Waals attraction/ Ван-дер-Ваальсовые взаимодействия · Protein folding/ Укладка белка · Affected by all of the foregoing forces/ определяется вышеперечисленными воздействиями © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
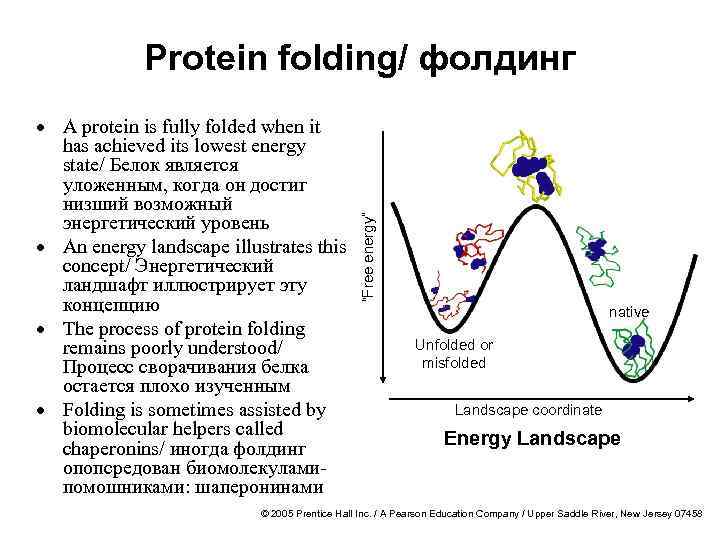 · A protein is fully folded when it has achieved its lowest energy state/ Белок является уложенным, когда он достиг низший возможный энергетический уровень · An energy landscape illustrates this concept/ Энергетический ландшафт иллюстрирует эту концепцию · The process of protein folding remains poorly understood/ Процесс сворачивания белка остается плохо изученным · Folding is sometimes assisted by biomolecular helpers called chaperonins/ иногда фолдинг опопсредован биомолекуламипомошниками: шаперонинами “Free energy” Protein folding/ фолдинг native Unfolded or misfolded Landscape coordinate Energy Landscape © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
· A protein is fully folded when it has achieved its lowest energy state/ Белок является уложенным, когда он достиг низший возможный энергетический уровень · An energy landscape illustrates this concept/ Энергетический ландшафт иллюстрирует эту концепцию · The process of protein folding remains poorly understood/ Процесс сворачивания белка остается плохо изученным · Folding is sometimes assisted by biomolecular helpers called chaperonins/ иногда фолдинг опопсредован биомолекуламипомошниками: шаперонинами “Free energy” Protein folding/ фолдинг native Unfolded or misfolded Landscape coordinate Energy Landscape © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
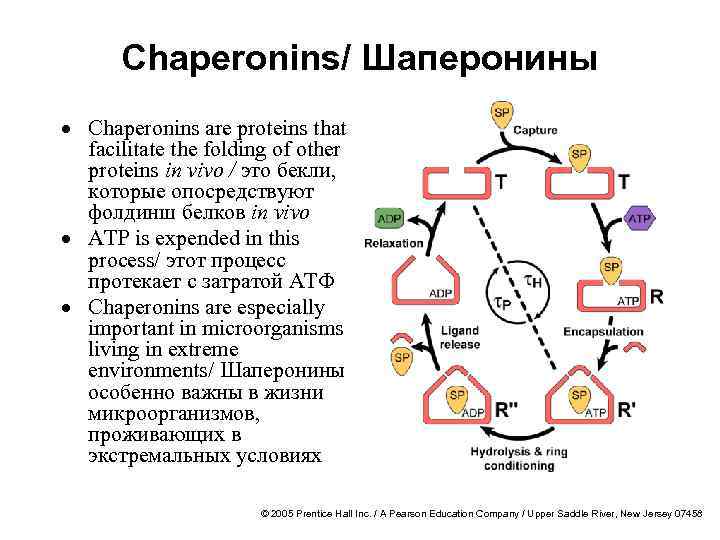 Chaperonins/ Шаперонины · Chaperonins are proteins that facilitate the folding of other proteins in vivo / это бекли, которые опосредствуют фолдинш белков in vivo · ATP is expended in this process/ этот процесс протекает с затратой АТФ · Chaperonins are especially important in microorganisms living in extreme environments/ Шаперонины особенно важны в жизни микроорганизмов, проживающих в экстремальных условиях © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Chaperonins/ Шаперонины · Chaperonins are proteins that facilitate the folding of other proteins in vivo / это бекли, которые опосредствуют фолдинш белков in vivo · ATP is expended in this process/ этот процесс протекает с затратой АТФ · Chaperonins are especially important in microorganisms living in extreme environments/ Шаперонины особенно важны в жизни микроорганизмов, проживающих в экстремальных условиях © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 The structure of proteins/ Структура белков · Primary/ Первичная · A protein’s amino acid sequence/ Белок представляет собой аминокислотную последовательность · Secondary/ Вторичная · Simple 3 -D structures/ простая пространственная укладка · Alpha helices/ альфа-спирали · Beta-pleated sheets/бета-складчастые слои · Tertiary / Третичная · Complete 3 -D structure of a protein molecule/ Образует более сложную пространственную молекулу · Quaternary / Четвертичная · Multi-subunit structure of a protein complex consisting of two or more individual molecules/ Мультикомплекс, состоящий из двух и более молекул © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The structure of proteins/ Структура белков · Primary/ Первичная · A protein’s amino acid sequence/ Белок представляет собой аминокислотную последовательность · Secondary/ Вторичная · Simple 3 -D structures/ простая пространственная укладка · Alpha helices/ альфа-спирали · Beta-pleated sheets/бета-складчастые слои · Tertiary / Третичная · Complete 3 -D structure of a protein molecule/ Образует более сложную пространственную молекулу · Quaternary / Четвертичная · Multi-subunit structure of a protein complex consisting of two or more individual molecules/ Мультикомплекс, состоящий из двух и более молекул © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Determining protein structure/ Определение белковой структуры · Direct measurement of structure/ прямое определение · X-ray crystallography/ Рентгеновская кристаллография · NMR spectroscopy/ЯМР-спектроскопия · Cryoelectron microscopy/ Криоэлектронная микросопия · Site-directed mutagenesis/ сайт-направленный мутагенез · Computer modeling/ компьютерное моделирование · Ab initio prediction of structure/ прогнозирование структуры Ab initio · Comparative protein-structure modeling/ Моделирование белковой структуры на основе сравнения © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Determining protein structure/ Определение белковой структуры · Direct measurement of structure/ прямое определение · X-ray crystallography/ Рентгеновская кристаллография · NMR spectroscopy/ЯМР-спектроскопия · Cryoelectron microscopy/ Криоэлектронная микросопия · Site-directed mutagenesis/ сайт-направленный мутагенез · Computer modeling/ компьютерное моделирование · Ab initio prediction of structure/ прогнозирование структуры Ab initio · Comparative protein-structure modeling/ Моделирование белковой структуры на основе сравнения © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 X-ray crystallography/ Рентгеновская кристаллография · Concept/ Суть · X-rays are scattered by electron clouds surrounding atoms/ рентгеновские лучи рассеиваются электронными облаками вокрук атомов · Pattern of scattered X-rays used to determine position of atoms in molecule/ Структуру такого рассеянного облучения используют для обнаржуения позиций атомов в молекуле · History/ история · 1959: first protein structures described by X-ray crystallography/ 1959 год: впервые был описан белок, с помощью этого метода Model of myoglobin / модель миоглобина © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
X-ray crystallography/ Рентгеновская кристаллография · Concept/ Суть · X-rays are scattered by electron clouds surrounding atoms/ рентгеновские лучи рассеиваются электронными облаками вокрук атомов · Pattern of scattered X-rays used to determine position of atoms in molecule/ Структуру такого рассеянного облучения используют для обнаржуения позиций атомов в молекуле · History/ история · 1959: first protein structures described by X-ray crystallography/ 1959 год: впервые был описан белок, с помощью этого метода Model of myoglobin / модель миоглобина © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Preparing the sample/ Приготовление образца · Single, very high quality protein crystals are required for X-ray crystallography/ этот метод требует выскокачественных белковых кристалов · Crystallizing protein is difficult/ кристализация белков является затруднительным процессом · Sample is mounted in front of an X-ray source/ Образцы фиксируют напротив источника излучения · Diffraction pattern is measured by CCD/ дифракционная картина улавливается сенсором Protein Crystals CCD detector © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Preparing the sample/ Приготовление образца · Single, very high quality protein crystals are required for X-ray crystallography/ этот метод требует выскокачественных белковых кристалов · Crystallizing protein is difficult/ кристализация белков является затруднительным процессом · Sample is mounted in front of an X-ray source/ Образцы фиксируют напротив источника излучения · Diffraction pattern is measured by CCD/ дифракционная картина улавливается сенсором Protein Crystals CCD detector © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Synchrotrons/ синхротроны · · Synchrotrons/ синхротроны · Provide high-intensity X-ray source/ излучают интенсивные рентгеновские лучи · 1, 000 times more intense than traditional “in-house” sources/ тысячекратно интенсивнее традиционных излучателей · Allow collection of high-quality diffraction data in hours/ в считанные часы получают дифракционные картины высокого качества Synchrotron at Brookhaven National Laboratory Limitations/ ограничения · Very expensive/ оооочень дорогие $$$ · Shared facility/ крупное оборудование (общего пользования ☺) Beamlines in purple © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Synchrotrons/ синхротроны · · Synchrotrons/ синхротроны · Provide high-intensity X-ray source/ излучают интенсивные рентгеновские лучи · 1, 000 times more intense than traditional “in-house” sources/ тысячекратно интенсивнее традиционных излучателей · Allow collection of high-quality diffraction data in hours/ в считанные часы получают дифракционные картины высокого качества Synchrotron at Brookhaven National Laboratory Limitations/ ограничения · Very expensive/ оооочень дорогие $$$ · Shared facility/ крупное оборудование (общего пользования ☺) Beamlines in purple © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
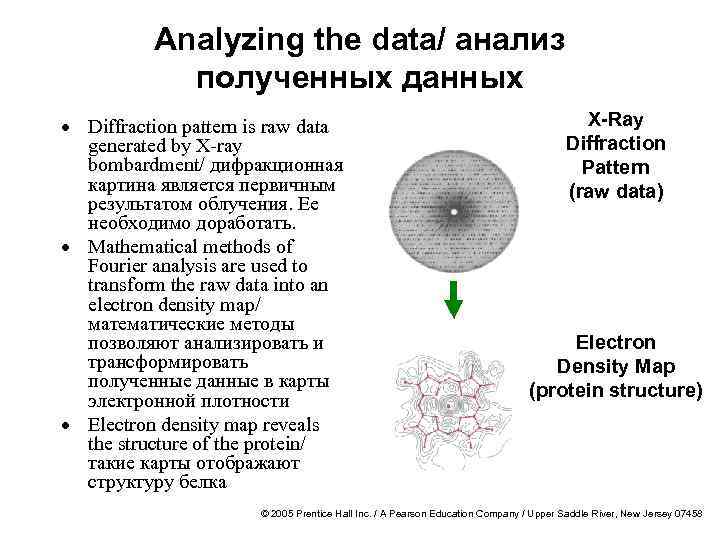 Analyzing the data/ анализ полученных данных · Diffraction pattern is raw data generated by X-ray bombardment/ дифракционная картина является первичным результатом облучения. Ее необходимо доработать. · Mathematical methods of Fourier analysis are used to transform the raw data into an electron density map/ математические методы позволяют анализировать и трансформировать полученные данные в карты электронной плотности · Electron density map reveals the structure of the protein/ такие карты отображают структуру белка X-Ray Diffraction Pattern (raw data) Electron Density Map (protein structure) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Analyzing the data/ анализ полученных данных · Diffraction pattern is raw data generated by X-ray bombardment/ дифракционная картина является первичным результатом облучения. Ее необходимо доработать. · Mathematical methods of Fourier analysis are used to transform the raw data into an electron density map/ математические методы позволяют анализировать и трансформировать полученные данные в карты электронной плотности · Electron density map reveals the structure of the protein/ такие карты отображают структуру белка X-Ray Diffraction Pattern (raw data) Electron Density Map (protein structure) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Advantages and disadvantages of Xray crystallography/ плюсы и минусы · Advantages/ преимущества · Highest resolution experimental technique/ высокопропускная техника · First developed in early 20 th century, hence very mature/ впервые использована в начале 20 века, следовательно – усовершенствована и хорошо известна · Disadvantages/ недостатки · Structures revealed not entirely complete/ не полностью отображает структуру · High-quality crystallized samples are unavailable for many types of proteins / необходима затрата больших усилий приготовлении высококачественного образца, не все белки можно кристаллизировать · Example: membrane-bound proteins/ пример: белки, связанные с мембраной · Crystal structure may not equal in vivo structure/ кристалическая структура не соответствует структуре in vivo © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Advantages and disadvantages of Xray crystallography/ плюсы и минусы · Advantages/ преимущества · Highest resolution experimental technique/ высокопропускная техника · First developed in early 20 th century, hence very mature/ впервые использована в начале 20 века, следовательно – усовершенствована и хорошо известна · Disadvantages/ недостатки · Structures revealed not entirely complete/ не полностью отображает структуру · High-quality crystallized samples are unavailable for many types of proteins / необходима затрата больших усилий приготовлении высококачественного образца, не все белки можно кристаллизировать · Example: membrane-bound proteins/ пример: белки, связанные с мембраной · Crystal structure may not equal in vivo structure/ кристалическая структура не соответствует структуре in vivo © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Principles of NMR/ принцип ЯМР · The spins of certain atomic nuclei generate a magnetic field/ спины некоторых атомных ядер генерируют магнитное поле · A sufficient amount of electromagnetic radiation will cause the spin to flip/ электромагнитная радиация будет влиять на спин · Absorption of this energy can be recorded/ изменение энергии регистрируется energy spin = +1/2 DE 1 DE 2 spin = – 1/2 B 1 B 2 magnetic field © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Principles of NMR/ принцип ЯМР · The spins of certain atomic nuclei generate a magnetic field/ спины некоторых атомных ядер генерируют магнитное поле · A sufficient amount of electromagnetic radiation will cause the spin to flip/ электромагнитная радиация будет влиять на спин · Absorption of this energy can be recorded/ изменение энергии регистрируется energy spin = +1/2 DE 1 DE 2 spin = – 1/2 B 1 B 2 magnetic field © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Preparing a sample for NMR spectroscopy/ изготовление образцов для ЯМР · NMR machines are big and expensive/ машины для ЯМР громоздкие и дорогие · More sensitivity means greater size and expense/ повышение чувствительности такого аппарата коррелирует с его размером и стоимостью · Samples move freely in aqueous solution/ образцы свободно двигаются в водном растворе · No crystallization is necessary / кристаллизация не нужна An NMR machine © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Preparing a sample for NMR spectroscopy/ изготовление образцов для ЯМР · NMR machines are big and expensive/ машины для ЯМР громоздкие и дорогие · More sensitivity means greater size and expense/ повышение чувствительности такого аппарата коррелирует с его размером и стоимостью · Samples move freely in aqueous solution/ образцы свободно двигаются в водном растворе · No crystallization is necessary / кристаллизация не нужна An NMR machine © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
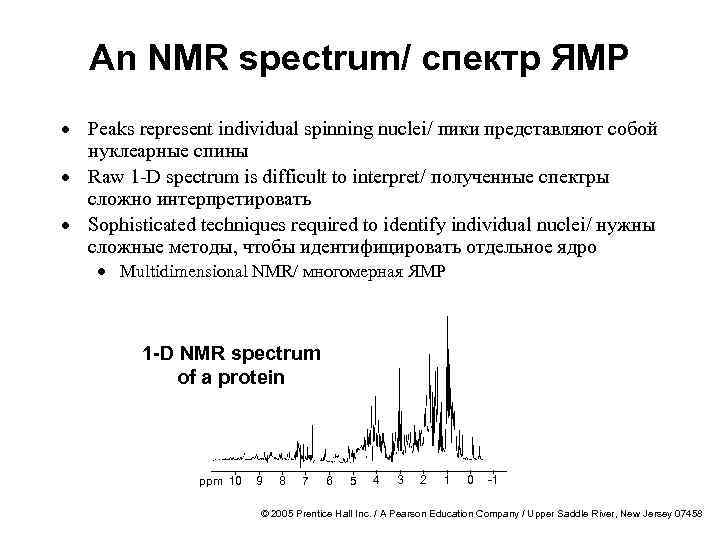 An NMR spectrum/ спектр ЯМР · Peaks represent individual spinning nuclei/ пики представляют собой нуклеарные спины · Raw 1 -D spectrum is difficult to interpret/ полученные спектры сложно интерпретировать · Sophisticated techniques required to identify individual nuclei/ нужны сложные методы, чтобы идентифицировать отдельное ядро · Multidimensional NMR/ многомерная ЯМР 1 -D NMR spectrum of a protein ppm 10 9 8 7 6 5 4 3 2 1 0 -1 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
An NMR spectrum/ спектр ЯМР · Peaks represent individual spinning nuclei/ пики представляют собой нуклеарные спины · Raw 1 -D spectrum is difficult to interpret/ полученные спектры сложно интерпретировать · Sophisticated techniques required to identify individual nuclei/ нужны сложные методы, чтобы идентифицировать отдельное ядро · Multidimensional NMR/ многомерная ЯМР 1 -D NMR spectrum of a protein ppm 10 9 8 7 6 5 4 3 2 1 0 -1 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
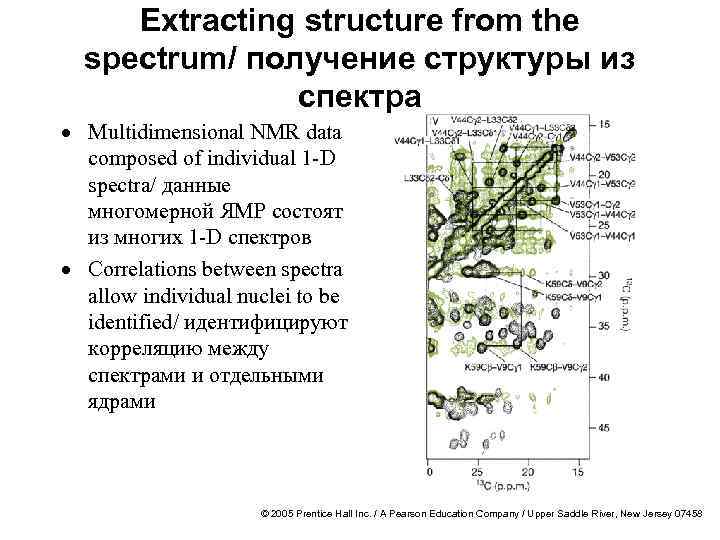 Extracting structure from the spectrum/ получение структуры из спектра · Multidimensional NMR data composed of individual 1 -D spectra/ данные многомерной ЯМР состоят из многих 1 -D спектров · Correlations between spectra allow individual nuclei to be identified/ идентифицируют корреляцию между спектрами и отдельными ядрами © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Extracting structure from the spectrum/ получение структуры из спектра · Multidimensional NMR data composed of individual 1 -D spectra/ данные многомерной ЯМР состоят из многих 1 -D спектров · Correlations between spectra allow individual nuclei to be identified/ идентифицируют корреляцию между спектрами и отдельными ядрами © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
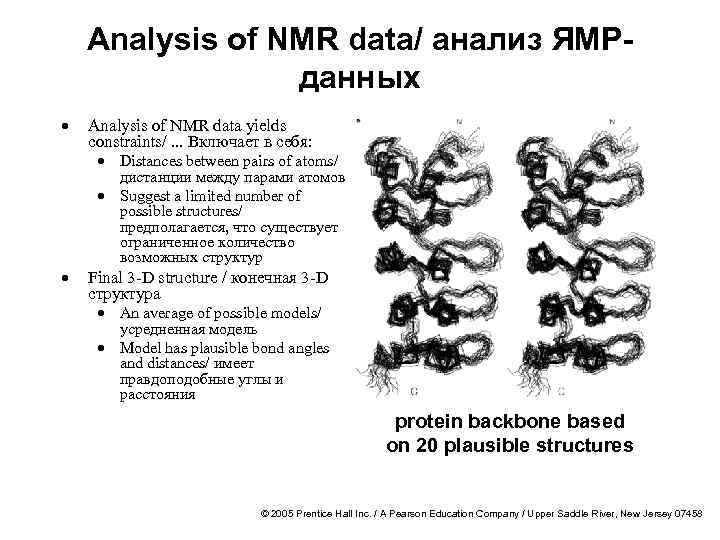 Analysis of NMR data/ анализ ЯМРданных · · Analysis of NMR data yields constraints/. . . Включает в себя: · Distances between pairs of atoms/ дистанции между парами атомов · Suggest a limited number of possible structures/ предполагается, что существует ограниченное количество возможных структур Final 3 -D structure / конечная 3 -D структура · An average of possible models/ усредненная модель · Model has plausible bond angles and distances/ имеет правдоподобные углы и расстояния protein backbone based on 20 plausible structures © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Analysis of NMR data/ анализ ЯМРданных · · Analysis of NMR data yields constraints/. . . Включает в себя: · Distances between pairs of atoms/ дистанции между парами атомов · Suggest a limited number of possible structures/ предполагается, что существует ограниченное количество возможных структур Final 3 -D structure / конечная 3 -D структура · An average of possible models/ усредненная модель · Model has plausible bond angles and distances/ имеет правдоподобные углы и расстояния protein backbone based on 20 plausible structures © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Advantages and disadvantages of NMR spectroscopy/ преимущества и недостатки · Advantages/ преимущества · Does not require crystallized protein/ образец не требует кристаллизации · Can determine protein structure under reasonable physiological conditions: aqueous solution at 30°C/ возможность исследования при физиологических условиях · Good way to test possible protein ligands/ хороший способ, чтобы тестировать возможные белковые лиганды · Allows measurement of protein “foldedness”/ ползволяет отслеживать фолдинг белка · Disadvantages/ недостатки · Requires high protein concentrations/ требует высоких концентраций белка · Much of data analysis is still done manually/ множество данных необходимо получать вручную · Only works well on proteins that are less than 25– 30 k. Da/ хорошо работает для белков меньше 25 -30 к. Да © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Advantages and disadvantages of NMR spectroscopy/ преимущества и недостатки · Advantages/ преимущества · Does not require crystallized protein/ образец не требует кристаллизации · Can determine protein structure under reasonable physiological conditions: aqueous solution at 30°C/ возможность исследования при физиологических условиях · Good way to test possible protein ligands/ хороший способ, чтобы тестировать возможные белковые лиганды · Allows measurement of protein “foldedness”/ ползволяет отслеживать фолдинг белка · Disadvantages/ недостатки · Requires high protein concentrations/ требует высоких концентраций белка · Much of data analysis is still done manually/ множество данных необходимо получать вручную · Only works well on proteins that are less than 25– 30 k. Da/ хорошо работает для белков меньше 25 -30 к. Да © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Cryoelectron microscopy/ криоэлектронная микроскопия · Sample must be frozen in a thin layer of buffer/ Образцы должны быть заморожены в тонком слое буфера · A diffraction pattern is generated using an electron beam/ Дифракционная картина создается с использованием электронного пучка · Trade-off: More electron bombardment means better images, but also damage to specimen/ Компромисс: чем больше электронной бомбардировки, тем лучше изображение, но также и выше повреждение образца · Data analyzed much like X-ray diffraction data/ анализых данных подобен тому, что в рентгеновской кристаллографии © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Cryoelectron microscopy/ криоэлектронная микроскопия · Sample must be frozen in a thin layer of buffer/ Образцы должны быть заморожены в тонком слое буфера · A diffraction pattern is generated using an electron beam/ Дифракционная картина создается с использованием электронного пучка · Trade-off: More electron bombardment means better images, but also damage to specimen/ Компромисс: чем больше электронной бомбардировки, тем лучше изображение, но также и выше повреждение образца · Data analyzed much like X-ray diffraction data/ анализых данных подобен тому, что в рентгеновской кристаллографии © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 An image generated by cryoelectron microscopy/ изображение, полученное путем криоэлектроной микроскопии Capsid protein from Hepatitis B Virus/ белок капсида гепатита Б © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
An image generated by cryoelectron microscopy/ изображение, полученное путем криоэлектроной микроскопии Capsid protein from Hepatitis B Virus/ белок капсида гепатита Б © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Advantages and disadvantages of cryoelectron microscopy/ преимущества и недостатки · Advantages/ преимущество · Proteins do not need to be crystallized/ белки не нуждаются в кристаллизации · Can be applied to membrane-bound proteins/ может быть применено к белкам, связанными с мембраной · Used to determine structure of large protein ensembles/ используется, что опрделения структуры больших белковых комплексов · Disadvantages/ недостатки · Low resolution compared with NMR and X-ray crystallography/ низкая разрешающая способность по сравнению с ЯМР и кристаллографией © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Advantages and disadvantages of cryoelectron microscopy/ преимущества и недостатки · Advantages/ преимущество · Proteins do not need to be crystallized/ белки не нуждаются в кристаллизации · Can be applied to membrane-bound proteins/ может быть применено к белкам, связанными с мембраной · Used to determine structure of large protein ensembles/ используется, что опрделения структуры больших белковых комплексов · Disadvantages/ недостатки · Low resolution compared with NMR and X-ray crystallography/ низкая разрешающая способность по сравнению с ЯМР и кристаллографией © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Site-directed mutagenesis/ сайтнаправленный мутагенез · Goal: To gain insight into a protein’s structure–function relationship by selectively altering the amino acid sequence/ цель: получить представление о структурно-функциональных отношениях белков, избирательно измененяя аминокислотную последовательность · Accomplished using standard molecular biological techniques/ Достигается с помощью стандартных молекулярнобиологических методов · Good technique for understanding proteins that are not amenable to traditional techniques. Хорошая техника для понимания белков, которые не поддаются традиционным методам · Membrane-bound proteins/ Мембраносвязанных белков · Example: the Shaker potassium channel/ Пример: Shaker канал калия © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Site-directed mutagenesis/ сайтнаправленный мутагенез · Goal: To gain insight into a protein’s structure–function relationship by selectively altering the amino acid sequence/ цель: получить представление о структурно-функциональных отношениях белков, избирательно измененяя аминокислотную последовательность · Accomplished using standard molecular biological techniques/ Достигается с помощью стандартных молекулярнобиологических методов · Good technique for understanding proteins that are not amenable to traditional techniques. Хорошая техника для понимания белков, которые не поддаются традиционным методам · Membrane-bound proteins/ Мембраносвязанных белков · Example: the Shaker potassium channel/ Пример: Shaker канал калия © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 The Shaker potassium channel · Shaker potassium channels are ion pores that span the cell membrane of Drosophila neurons/ канал Shaker калия – пора, которая пронизывает мембрану клетки · Allow only potassium ions to pass out of cell/ позволяет лишь ионам калия выходить из клетки · Channel is activated when the voltage across the membrane becomes more positive/ канал активируется, когда напряжение мембраны становится более позитивным · Channel is inactivated after a brief period of time/ канал инактивируется через некоторое время · Channel is known as Shaker because flies with mutated channel shake their legs under ether anesthesia/ канал известен как Shaker, поскольку у мух с мутантным каналом трясутся ноги © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The Shaker potassium channel · Shaker potassium channels are ion pores that span the cell membrane of Drosophila neurons/ канал Shaker калия – пора, которая пронизывает мембрану клетки · Allow only potassium ions to pass out of cell/ позволяет лишь ионам калия выходить из клетки · Channel is activated when the voltage across the membrane becomes more positive/ канал активируется, когда напряжение мембраны становится более позитивным · Channel is inactivated after a brief period of time/ канал инактивируется через некоторое время · Channel is known as Shaker because flies with mutated channel shake their legs under ether anesthesia/ канал известен как Shaker, поскольку у мух с мутантным каналом трясутся ноги © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
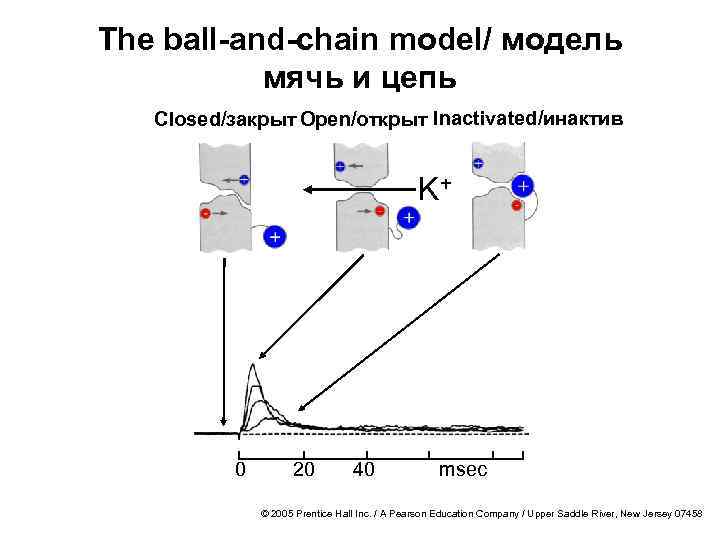 The ball-and-chain model/ модель мячь и цепь Closed/закрыт Open/открыт Inactivated/инактив K+ 0 20 40 msec © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The ball-and-chain model/ модель мячь и цепь Closed/закрыт Open/открыт Inactivated/инактив K+ 0 20 40 msec © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
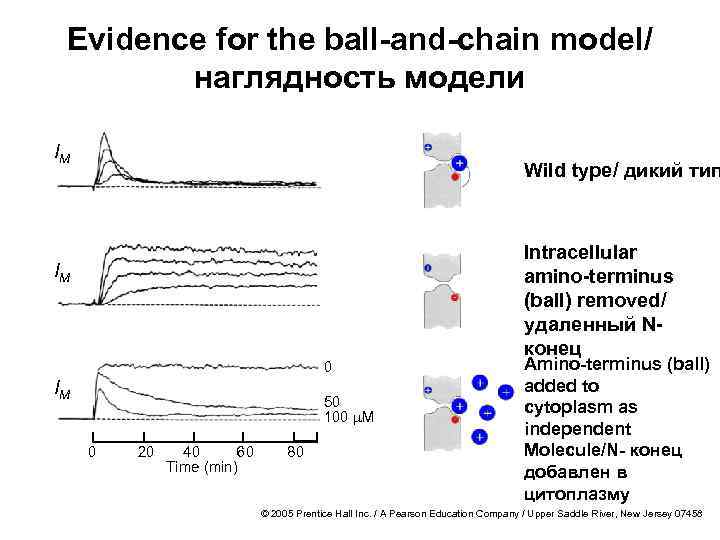 Evidence for the ball-and-chain model/ наглядность модели IM Wild type/ дикий тип IM 0 IM 50 100 m. M 0 20 40 60 Time (min) 80 Intracellular amino-terminus (ball) removed/ удаленный Nконец Amino-terminus (ball) added to cytoplasm as independent Molecule/N- конец добавлен в цитоплазму © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Evidence for the ball-and-chain model/ наглядность модели IM Wild type/ дикий тип IM 0 IM 50 100 m. M 0 20 40 60 Time (min) 80 Intracellular amino-terminus (ball) removed/ удаленный Nконец Amino-terminus (ball) added to cytoplasm as independent Molecule/N- конец добавлен в цитоплазму © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
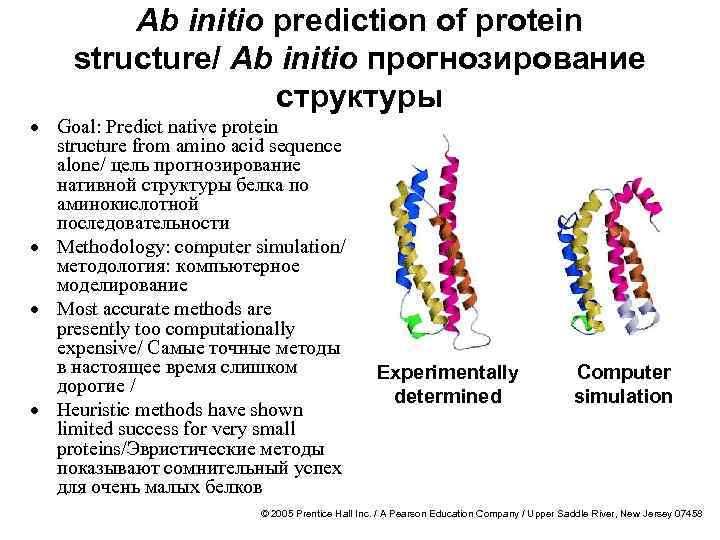 Ab initio prediction of protein structure/ Ab initio прогнозирование структуры · Goal: Predict native protein structure from amino acid sequence alone/ цель прогнозирование нативной структуры белка по аминокислотной последовательности · Methodology: computer simulation/ методология: компьютерное моделирование · Most accurate methods are presently too computationally expensive/ Самые точные методы в настоящее время слишком дорогие / · Heuristic methods have shown limited success for very small proteins/Эвристические методы показывают сомнительный успех для очень малых белков Experimentally determined Computer simulation © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Ab initio prediction of protein structure/ Ab initio прогнозирование структуры · Goal: Predict native protein structure from amino acid sequence alone/ цель прогнозирование нативной структуры белка по аминокислотной последовательности · Methodology: computer simulation/ методология: компьютерное моделирование · Most accurate methods are presently too computationally expensive/ Самые точные методы в настоящее время слишком дорогие / · Heuristic methods have shown limited success for very small proteins/Эвристические методы показывают сомнительный успех для очень малых белков Experimentally determined Computer simulation © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Comparative protein-structure modeling/ моделирование белковой структуры с помощью сравнения · Goal: Construct 3 -D model of a protein of unknown structure (target), based on similarity of sequence to proteins of known structure (templates)/ задача: создать трехмерную модель белка неизвестной структуры (цель), основано на сравнении с белком известной структуры и похожей последовательностью. · Procedure/ процедура: · Fold assignment and template selection/ выбор шаблона · Template–target alignment/ выравнивание с целевым белком · Model building/ построение модели · Model evaluation/ анализ модели © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Comparative protein-structure modeling/ моделирование белковой структуры с помощью сравнения · Goal: Construct 3 -D model of a protein of unknown structure (target), based on similarity of sequence to proteins of known structure (templates)/ задача: создать трехмерную модель белка неизвестной структуры (цель), основано на сравнении с белком известной структуры и похожей последовательностью. · Procedure/ процедура: · Fold assignment and template selection/ выбор шаблона · Template–target alignment/ выравнивание с целевым белком · Model building/ построение модели · Model evaluation/ анализ модели © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
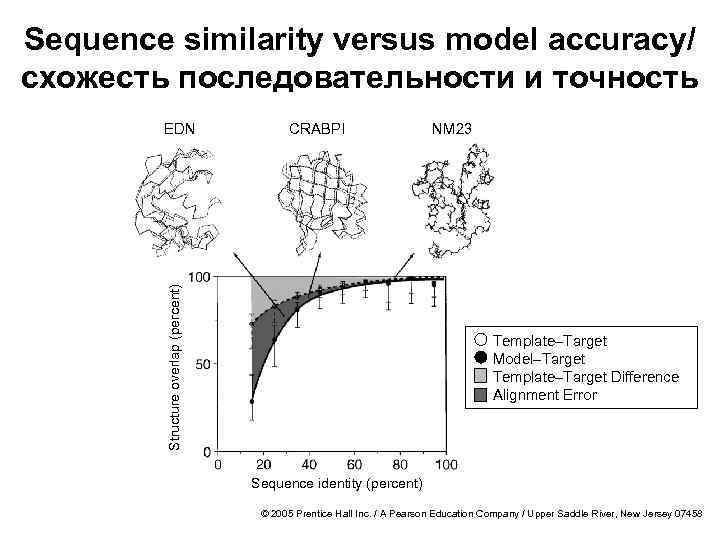 Sequence similarity versus model accuracy/ схожесть последовательности и точность CRABPI Structure overlap (percent) EDN NM 23 Template–Target Model–Target Template–Target Difference Alignment Error Sequence identity (percent) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Sequence similarity versus model accuracy/ схожесть последовательности и точность CRABPI Structure overlap (percent) EDN NM 23 Template–Target Model–Target Template–Target Difference Alignment Error Sequence identity (percent) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Assessment of comparative modeling / Оценка сравнительного моделирования · Presently provides most accurate method for prediction of protein structure/ в настоящее время это наиболее точный метод для предсказания структуры белков · Likely to play large role in structural genomics/ Вероятно, будет играть большую роль в структурной геномике · Model building and evaluation are amenable to automation/ построение и анализ меодли автоматизированы · Predictions are often good enough to predict ligand binding and protein–protein interaction/ Прогнозов зачастую достаточно, чтобы предсказать связывание лиганда и проследить белок-белковые взаимодействия. © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Assessment of comparative modeling / Оценка сравнительного моделирования · Presently provides most accurate method for prediction of protein structure/ в настоящее время это наиболее точный метод для предсказания структуры белков · Likely to play large role in structural genomics/ Вероятно, будет играть большую роль в структурной геномике · Model building and evaluation are amenable to automation/ построение и анализ меодли автоматизированы · Predictions are often good enough to predict ligand binding and protein–protein interaction/ Прогнозов зачастую достаточно, чтобы предсказать связывание лиганда и проследить белок-белковые взаимодействия. © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 The Protein Data Bank/ белковая база данных · The PDB contains 3 -D structural data for proteins, nucleic acids, and carbohydrates/ в банке содержатся данные по трехмерной структуре белков, аминокислот и углеводов · Founded in 1971 with a dozen structures/ основан в 1971 году с десятками структур · As of June 2004, there were 25, 760 structures in the database/ в июне 2004 в базе начислялось 25760 структур · All structure files are reviewed by PDB staff for accuracy and data uniformity/ все файлы проверяются сотрудниками на точность и достоверность · Structural data from the PDB can be freely accessed at: / базу можно свободно просмотреть на: http: //www. rcsb. org/pdb/ © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The Protein Data Bank/ белковая база данных · The PDB contains 3 -D structural data for proteins, nucleic acids, and carbohydrates/ в банке содержатся данные по трехмерной структуре белков, аминокислот и углеводов · Founded in 1971 with a dozen structures/ основан в 1971 году с десятками структур · As of June 2004, there were 25, 760 structures in the database/ в июне 2004 в базе начислялось 25760 структур · All structure files are reviewed by PDB staff for accuracy and data uniformity/ все файлы проверяются сотрудниками на точность и достоверность · Structural data from the PDB can be freely accessed at: / базу можно свободно просмотреть на: http: //www. rcsb. org/pdb/ © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
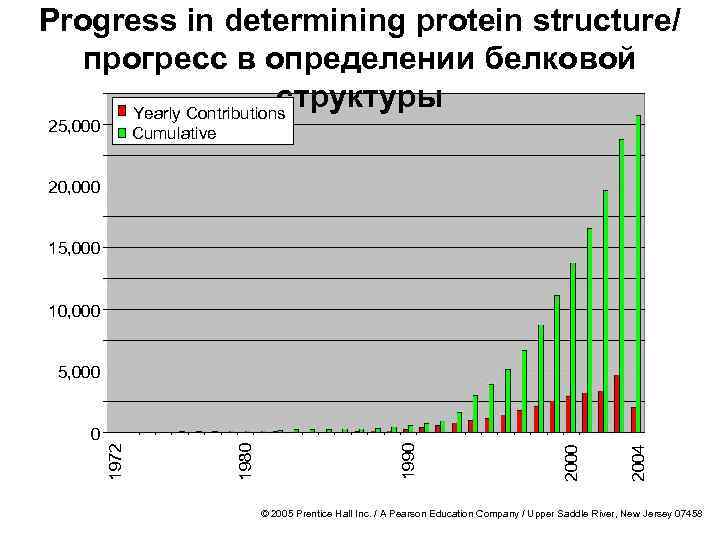 Progress in determining protein structure/ прогресс в определении белковой структуры Yearly Contributions 25, 000 Cumulative 20, 000 15, 000 10, 000 2004 2000 1990 1980 0 1972 5, 000 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Progress in determining protein structure/ прогресс в определении белковой структуры Yearly Contributions 25, 000 Cumulative 20, 000 15, 000 10, 000 2004 2000 1990 1980 0 1972 5, 000 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Success of different methods of determining protein structure/ успех различных методов при определении белковой структуры: · The Protein Data Bank (PDB) is a repository of high-quality 3 -D structural data/ банк является хранилищем информации о трехмерной структуре белков · 80% come from X-ray crystallography/ 80% структур получено с помощью кристаллографии · 16% come from NMR/ 16% - с помощью ЯМР · 2% come from theoretical modeling/ 2% с помощью теоретического моделирования © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Success of different methods of determining protein structure/ успех различных методов при определении белковой структуры: · The Protein Data Bank (PDB) is a repository of high-quality 3 -D structural data/ банк является хранилищем информации о трехмерной структуре белков · 80% come from X-ray crystallography/ 80% структур получено с помощью кристаллографии · 16% come from NMR/ 16% - с помощью ЯМР · 2% come from theoretical modeling/ 2% с помощью теоретического моделирования © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 High-throughput methods/ высокопропускные методы © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
High-throughput methods/ высокопропускные методы © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Automating protein purification / Автоматичная очистка белков · Use multiple promoters on gene of interest to quickly find the best expression system/ использует множественные промоторы гена интереса для того, чтобы быстро найти наилучшую систему экспрессии · Incorporate affinity tags on recombination-based vectors to aid in purification/ использование афинных меток на рекомбинационных векторах для оказания помощи в очистке (белиберда бла) · Use fused green fluorescent protein (GFP) that indicates presence of properly folded proteins in solution/ использование GFP для визуализации процесса фолдинга белка · Concentrate on hyperthermophilic homologs / концентрируется на гипотермофильных гомологах · Very soluble / очень растворим · Stable/ стабильный · Good for ligand-binding studies/хорош для изучения связывания с лигандом © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Automating protein purification / Автоматичная очистка белков · Use multiple promoters on gene of interest to quickly find the best expression system/ использует множественные промоторы гена интереса для того, чтобы быстро найти наилучшую систему экспрессии · Incorporate affinity tags on recombination-based vectors to aid in purification/ использование афинных меток на рекомбинационных векторах для оказания помощи в очистке (белиберда бла) · Use fused green fluorescent protein (GFP) that indicates presence of properly folded proteins in solution/ использование GFP для визуализации процесса фолдинга белка · Concentrate on hyperthermophilic homologs / концентрируется на гипотермофильных гомологах · Very soluble / очень растворим · Stable/ стабильный · Good for ligand-binding studies/хорош для изучения связывания с лигандом © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Automating sample preparation/ автоматическое приготовление образца · Use robots to do thousands of crystallization experiments in parallel/ использование роботов для одновременного проведения тысячи экспериментов кристаллизации · Measure quality of protein crystals / Измерение качества белковых кристаллов · Beamline automation: Use robots instead of humans to mount and center protein crystals for X-ray diffraction/ использование роботов, а не людей, чтобы закрепить и центрировать белок для облучения · New methods for calculating phase in X-ray diffraction measurements/ новые методы рассчета фаз в кристаллографии · MAD · SAD © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Automating sample preparation/ автоматическое приготовление образца · Use robots to do thousands of crystallization experiments in parallel/ использование роботов для одновременного проведения тысячи экспериментов кристаллизации · Measure quality of protein crystals / Измерение качества белковых кристаллов · Beamline automation: Use robots instead of humans to mount and center protein crystals for X-ray diffraction/ использование роботов, а не людей, чтобы закрепить и центрировать белок для облучения · New methods for calculating phase in X-ray diffraction measurements/ новые методы рассчета фаз в кристаллографии · MAD · SAD © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Automating data analysis/ автоматизация анализа данных · Advances in software may eliminate the need for human intervention/ преимущества софта должны исключить потребность в человеческом вмешательстве · Iterative modeling of electron density maps/ моделирование карт электронной плотности · Build multiple models to account for electron density maps that fit amino acid sequence data/ Построение нескольких моделей для сопоставления электронной плотности с аминокислотной последовательностью · Iterative method estimated to be applicable to 50% of proteins currently being analyzed/ Итерационный метод возможно применить к 50% белков, которые в настоящее время анализируются · In the future, the entire process, from sample preparation to 3 -D structural determination, may be fully automated/ В дальнейшем весь процесс, от подготовки образцов до получения 3 -D структуры, может быть полностью автоматизирован © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Automating data analysis/ автоматизация анализа данных · Advances in software may eliminate the need for human intervention/ преимущества софта должны исключить потребность в человеческом вмешательстве · Iterative modeling of electron density maps/ моделирование карт электронной плотности · Build multiple models to account for electron density maps that fit amino acid sequence data/ Построение нескольких моделей для сопоставления электронной плотности с аминокислотной последовательностью · Iterative method estimated to be applicable to 50% of proteins currently being analyzed/ Итерационный метод возможно применить к 50% белков, которые в настоящее время анализируются · In the future, the entire process, from sample preparation to 3 -D structural determination, may be fully automated/ В дальнейшем весь процесс, от подготовки образцов до получения 3 -D структуры, может быть полностью автоматизирован © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Future prospects/ Перспективы · Feasibility of a successful structural genomics program / Возможность успешного развития структурной геномики · Elucidation of protein structure corresponding to 90% of all gene sequences will require at least 16, 000 experimentally determined protein structures / Выяснение структуры белка, соответствующей 90% всех генных последовательностей потребуется по крайней мере 16000 экспериментально определенных белковых структур · Presently too much redundancy in protein-structure community / в настоящее время, слишком много лишнего в структуре белка · However, a coordinated structural-genomics project might take as little as 10 years to complete / Тем не менее, проект структурной геномики может занять 10 лет, чтобы завершиться © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Future prospects/ Перспективы · Feasibility of a successful structural genomics program / Возможность успешного развития структурной геномики · Elucidation of protein structure corresponding to 90% of all gene sequences will require at least 16, 000 experimentally determined protein structures / Выяснение структуры белка, соответствующей 90% всех генных последовательностей потребуется по крайней мере 16000 экспериментально определенных белковых структур · Presently too much redundancy in protein-structure community / в настоящее время, слишком много лишнего в структуре белка · However, a coordinated structural-genomics project might take as little as 10 years to complete / Тем не менее, проект структурной геномики может занять 10 лет, чтобы завершиться © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 The progress of structural genomics/ прогресс структурной геномики · Oldest efforts started in 1997, most since 2000 / поиски белковых структур впервые начали в 1971 году, множество усилий было направлено в 2000. · Success of NESGC is typical/ успех очевиден: · Pursued 5, 187 DNA targets/ найдено 5187 продуктов ДНК · Cloned 1, 675/ клонировано 1675 · Expressed 1, 295 as proteins/ 1295 - белки · Only 773 were water soluble/ лишь 773 растворимы · Crystallized 94/ 94 кристаллизированы · Determined the structure of 50/ определена структура 50 · 22 with X-ray crystallography/ 22 с помощью кристаллографии · 28 with NMR/ 28 – с помощью ЯМР © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The progress of structural genomics/ прогресс структурной геномики · Oldest efforts started in 1997, most since 2000 / поиски белковых структур впервые начали в 1971 году, множество усилий было направлено в 2000. · Success of NESGC is typical/ успех очевиден: · Pursued 5, 187 DNA targets/ найдено 5187 продуктов ДНК · Cloned 1, 675/ клонировано 1675 · Expressed 1, 295 as proteins/ 1295 - белки · Only 773 were water soluble/ лишь 773 растворимы · Crystallized 94/ 94 кристаллизированы · Determined the structure of 50/ определена структура 50 · 22 with X-ray crystallography/ 22 с помощью кристаллографии · 28 with NMR/ 28 – с помощью ЯМР © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Applications of structural genomics / Применение структурной геномики · Structure-based drug design/ создание структурно «правильных» лекарств · Designing drugs that target specific protein domains/ дизайн лекарств, которые имеют специфичные домены для связывания с мишенью · Caveat: Structural-genomics projects presently ignore membrane-bound proteins, which account for ~50% of all drug targets/ Примечание: проэкты структурной геномики в настоящее время обходят мембраносвязанных белков, которые в 50% случаев, являются мишенями для лекарственных средств · Better understanding of human disease/ лучшее понимание человеческих заболеваний · Improvements in the design of industrial enzymes / Улучшения в создании промышленных ферментов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Applications of structural genomics / Применение структурной геномики · Structure-based drug design/ создание структурно «правильных» лекарств · Designing drugs that target specific protein domains/ дизайн лекарств, которые имеют специфичные домены для связывания с мишенью · Caveat: Structural-genomics projects presently ignore membrane-bound proteins, which account for ~50% of all drug targets/ Примечание: проэкты структурной геномики в настоящее время обходят мембраносвязанных белков, которые в 50% случаев, являются мишенями для лекарственных средств · Better understanding of human disease/ лучшее понимание человеческих заболеваний · Improvements in the design of industrial enzymes / Улучшения в создании промышленных ферментов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 RNA / РНК · RNA function/ функции РНК · · Protein synthesis/ синтез белков Targeting / мишень RNA splicing / сплайсинг Chromosome replication / репликация хромосом · Understanding of RNA much less developed than that of proteins/ РНК менее изучена, чем белки · 1982: Discovery of ribozymes—RNA molecules that can act like enzymes/ 1982 год: открытие рибозимов · 1994: First crystal structure of ribozyme/ 1994 год: впервые кристаллизированы © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
RNA / РНК · RNA function/ функции РНК · · Protein synthesis/ синтез белков Targeting / мишень RNA splicing / сплайсинг Chromosome replication / репликация хромосом · Understanding of RNA much less developed than that of proteins/ РНК менее изучена, чем белки · 1982: Discovery of ribozymes—RNA molecules that can act like enzymes/ 1982 год: открытие рибозимов · 1994: First crystal structure of ribozyme/ 1994 год: впервые кристаллизированы © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
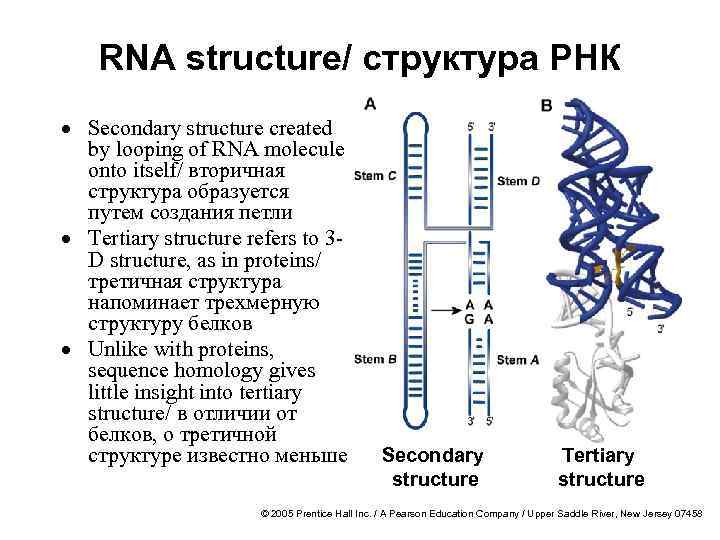 RNA structure/ структура РНК · Secondary structure created by looping of RNA molecule onto itself/ вторичная структура образуется путем создания петли · Tertiary structure refers to 3 D structure, as in proteins/ третичная структура напоминает трехмерную структуру белков · Unlike with proteins, sequence homology gives little insight into tertiary structure/ в отличии от белков, о третичной структуре известно меньше Secondary structure Tertiary structure © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
RNA structure/ структура РНК · Secondary structure created by looping of RNA molecule onto itself/ вторичная структура образуется путем создания петли · Tertiary structure refers to 3 D structure, as in proteins/ третичная структура напоминает трехмерную структуру белков · Unlike with proteins, sequence homology gives little insight into tertiary structure/ в отличии от белков, о третичной структуре известно меньше Secondary structure Tertiary structure © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 RNA–protein complexes/РНКбелковый комплекс © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
RNA–protein complexes/РНКбелковый комплекс © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 The challenges of determining RNA structure/ сложности в определении структуры РНК · Finding RNA-encoding genes/ поиск РНК-кодирующих генов · Members of known families can be found by comparing secondary structure/ члены известного семейства могут быть найдены путем сравнения вторичной структуры · But how to find novel RNA families? / но как найти новые семейства РНК? · RNA structure is highly context dependent/ структура РНК сильно зависит от окружения · Noncanonical base pairings/ неканонические спаривания основ · e. g. , A–A or A–G/ · Substrate–RNA interactions alter the tertiary structure of RNA/ Связи «субстрат-РНК» изменяют третичную структуру РНК © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The challenges of determining RNA structure/ сложности в определении структуры РНК · Finding RNA-encoding genes/ поиск РНК-кодирующих генов · Members of known families can be found by comparing secondary structure/ члены известного семейства могут быть найдены путем сравнения вторичной структуры · But how to find novel RNA families? / но как найти новые семейства РНК? · RNA structure is highly context dependent/ структура РНК сильно зависит от окружения · Noncanonical base pairings/ неканонические спаривания основ · e. g. , A–A or A–G/ · Substrate–RNA interactions alter the tertiary structure of RNA/ Связи «субстрат-РНК» изменяют третичную структуру РНК © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Prospects for RNA structural genomics/ перспективы определения структуры РНК · Techniques for structure determination/ методы для определения структуры: · X-ray crystallography/ кристаллография · NMR/ ЯМР · Cryoelectron microscopy/ криоэлектронная микроскопия · Chemical probing/ химические реакции · High-throughput methods/ высоко-пропускные методы · RNA structure database for comparative modeling/ данные структуры РНК для сравнительного моделирования © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Prospects for RNA structural genomics/ перспективы определения структуры РНК · Techniques for structure determination/ методы для определения структуры: · X-ray crystallography/ кристаллография · NMR/ ЯМР · Cryoelectron microscopy/ криоэлектронная микроскопия · Chemical probing/ химические реакции · High-throughput methods/ высоко-пропускные методы · RNA structure database for comparative modeling/ данные структуры РНК для сравнительного моделирования © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Summary/ выводы · · · Determining protein structure is more difficult than sequencing a genome / Определение структуры белка является более сложным, чем секвенирование генома · Different techniques required for different classes of proteins / Различные методы, необходимые для различных классов белков · Customized approaches make automation difficult / необходимость индивидуальных подходов усложняет автоматизацию процесса Requirements for structural genomics/ Требования к структурной геномике · Determination of structure of 10% of all proteins (a representative sample) / Определение структуры 10% всех белков (репрезентативная выборка) · Development of high-throughput methods / Развитие высокопропускных методов · Reorganizing the efforts of structural biologists to avoid redundancy / Реорганизация усилий структурных биологов, для избегания дублирования RNA structural genomics is important, but undeveloped at this time/ определение РНК является важным процессом, но, увы, недостаточно развито на сегодняшний день. © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Summary/ выводы · · · Determining protein structure is more difficult than sequencing a genome / Определение структуры белка является более сложным, чем секвенирование генома · Different techniques required for different classes of proteins / Различные методы, необходимые для различных классов белков · Customized approaches make automation difficult / необходимость индивидуальных подходов усложняет автоматизацию процесса Requirements for structural genomics/ Требования к структурной геномике · Determination of structure of 10% of all proteins (a representative sample) / Определение структуры 10% всех белков (репрезентативная выборка) · Development of high-throughput methods / Развитие высокопропускных методов · Reorganizing the efforts of structural biologists to avoid redundancy / Реорганизация усилий структурных биологов, для избегания дублирования RNA structural genomics is important, but undeveloped at this time/ определение РНК является важным процессом, но, увы, недостаточно развито на сегодняшний день. © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458


