08_46.ppt
- Количество слайдов: 46
 Chapter 8 Proteomics/ протеомика Using high-throughput methods to identify proteins and to understand their function/ использование высокопропускных методов для идентификации белков и понимания их функции © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Chapter 8 Proteomics/ протеомика Using high-throughput methods to identify proteins and to understand their function/ использование высокопропускных методов для идентификации белков и понимания их функции © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Contents/ содержание · Definition of proteomics/ определение протеомики · Proteomics technologies/ технологии · · 2 -D gel electrophoresis/ двухмерный электрофорез Mass spectrometry/ спектрометрия Protein chips/ белковые чипы Yeast two-hybrid method/ дрожжевой метод «двух гибридов» · Biochemical genomics/ биохимическая геномика · Using proteomics to uncover transcriptional networks/ использование протеомики для понимания транскрипционных сетей © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Contents/ содержание · Definition of proteomics/ определение протеомики · Proteomics technologies/ технологии · · 2 -D gel electrophoresis/ двухмерный электрофорез Mass spectrometry/ спектрометрия Protein chips/ белковые чипы Yeast two-hybrid method/ дрожжевой метод «двух гибридов» · Biochemical genomics/ биохимическая геномика · Using proteomics to uncover transcriptional networks/ использование протеомики для понимания транскрипционных сетей © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 What is proteomics? что такое протеомика · An organism’s proteome/ организменный протеом: · A catalog of all proteins/ набор всех белков · Expressed throughout life/ экспрессирующихся пожизненно · Expressed under all conditions/ экспрессирующихся в определенных условиях · The goals of proteomics/ цели протеомики · To catalog all proteins/ обнаружить все белки · To understand their functions/ понять функции · To understand how they interact with each other/ понять как они взаимодействуют друг с другом © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
What is proteomics? что такое протеомика · An organism’s proteome/ организменный протеом: · A catalog of all proteins/ набор всех белков · Expressed throughout life/ экспрессирующихся пожизненно · Expressed under all conditions/ экспрессирующихся в определенных условиях · The goals of proteomics/ цели протеомики · To catalog all proteins/ обнаружить все белки · To understand their functions/ понять функции · To understand how they interact with each other/ понять как они взаимодействуют друг с другом © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 The challenges of proteomics/ проблемы протеомики · Splice variants create an enormous diversity of proteins/ сплайсинговые варианты создают огромное разнообразие белков · ~25, 000 genes in humans give rise to 200, 000 to 2, 000 different proteins/ 25 000 генов человеческого генома может экспрессировать 200 000 – 2 000 разных белков · Splice variants may have very diverse functions/ сплайсинговые варианты имеют очень разные функции · Proteins expressed in an organism will vary according to age, health, tissue, and environmental stimuli/ белки, экспрессирующиеся в организме, изменяются под влиянием возраста и действия внешних факторов · Proteomics requires a broader range of technologies than genomics/ протеомика нуждается в более широком спектре технологий, чем геномика © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The challenges of proteomics/ проблемы протеомики · Splice variants create an enormous diversity of proteins/ сплайсинговые варианты создают огромное разнообразие белков · ~25, 000 genes in humans give rise to 200, 000 to 2, 000 different proteins/ 25 000 генов человеческого генома может экспрессировать 200 000 – 2 000 разных белков · Splice variants may have very diverse functions/ сплайсинговые варианты имеют очень разные функции · Proteins expressed in an organism will vary according to age, health, tissue, and environmental stimuli/ белки, экспрессирующиеся в организме, изменяются под влиянием возраста и действия внешних факторов · Proteomics requires a broader range of technologies than genomics/ протеомика нуждается в более широком спектре технологий, чем геномика © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Diversity of function in splice variants/ многообразие функций в сплайсинговых вариантах · Example: the calcitonin gene/ пример: ген кальцитонина · Gene variant #1/ 1 вариант · Protein: calcitonin/ белок - кальситонин · Function: increases calcium uptake in bones/ функция – увеличение концентрации кальция в костях · Gene variant #2/ генный вариант 2 · Protein: calcitonin gene-related polypeptide / белок: полипептид, родственный кальцитонину · Function: causes blood vessels to dilate/ фунция: расширение сосудов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Diversity of function in splice variants/ многообразие функций в сплайсинговых вариантах · Example: the calcitonin gene/ пример: ген кальцитонина · Gene variant #1/ 1 вариант · Protein: calcitonin/ белок - кальситонин · Function: increases calcium uptake in bones/ функция – увеличение концентрации кальция в костях · Gene variant #2/ генный вариант 2 · Protein: calcitonin gene-related polypeptide / белок: полипептид, родственный кальцитонину · Function: causes blood vessels to dilate/ фунция: расширение сосудов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Posttranslational modifications/ посттрансляционные модификации · Proteolytic cleavage/ протеолитическое расщепление · Fragmenting protein/ фрагментация белка · Addition of chemical groups/ добавление химической группы © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Posttranslational modifications/ посттрансляционные модификации · Proteolytic cleavage/ протеолитическое расщепление · Fragmenting protein/ фрагментация белка · Addition of chemical groups/ добавление химической группы © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Chemical modifications/ химические модификации · · · · Phosphorylation: activation and inactivation of enzymes/ фосфорилирование: активация/инактивация Acetylation: protein stability, used in histones/ ацетилирование: стабильность белка, используется в гистонах Methylation: regulation of gene expression/ метилирование: регулирование генной экспрессии Acylation: membrane tethering, targeting/ ацилирование: связывание с мемраной, мишенью Glycosylation: cell–cell recognition, signaling/ глигозилирование: узнавание клетками друг-друга, сигналинг GPI anchor: membrane tethering/ гликофосфатидилинозитоловое заякоривание: связывание с мембранной Hydroxyproline: protein stability, ligand interactions/ гидроксипролин: стабильность белка Sulfation: protein–protein and ligand interactions/ сульфирование: распознавание белком белка и лиганда. Disulfide-bond formation: protein stability/ образование дисульфидных связей: стабильность белка Deamidation: protein–protein and ligand interactions/ деамидирование: распознавание белком белка и лиганда. Pyroglutamic acid: protein stability/ пироглютаминовая кислота: стабильность белка Ubiquitination: destruction signal/ убиквитинизирование: деструктивный сигнал Nitration of tyrosine: inflammation/ добавление азота к тирозину: возгорание : D © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Chemical modifications/ химические модификации · · · · Phosphorylation: activation and inactivation of enzymes/ фосфорилирование: активация/инактивация Acetylation: protein stability, used in histones/ ацетилирование: стабильность белка, используется в гистонах Methylation: regulation of gene expression/ метилирование: регулирование генной экспрессии Acylation: membrane tethering, targeting/ ацилирование: связывание с мемраной, мишенью Glycosylation: cell–cell recognition, signaling/ глигозилирование: узнавание клетками друг-друга, сигналинг GPI anchor: membrane tethering/ гликофосфатидилинозитоловое заякоривание: связывание с мембранной Hydroxyproline: protein stability, ligand interactions/ гидроксипролин: стабильность белка Sulfation: protein–protein and ligand interactions/ сульфирование: распознавание белком белка и лиганда. Disulfide-bond formation: protein stability/ образование дисульфидных связей: стабильность белка Deamidation: protein–protein and ligand interactions/ деамидирование: распознавание белком белка и лиганда. Pyroglutamic acid: protein stability/ пироглютаминовая кислота: стабильность белка Ubiquitination: destruction signal/ убиквитинизирование: деструктивный сигнал Nitration of tyrosine: inflammation/ добавление азота к тирозину: возгорание : D © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Practical applications/ практическое применение · Comparison of protein expression in diseased and normal tissues/ сравнение экспрессии белков в нормальных и патологических клетках · Likely to reveal new drug targets/ необходимы для выявления новых мишеней для лекарств · Today ~500 drug targets/ сегодня известно около 500 таких мишеней · Estimates of possible drug targets: 10, 000– 20, 000/ по оценкам, возможных мишеней существует около 20000 · Protein expression signatures associated with drug toxicity/ характерная черта экспрессии белка может быть индикатором токсичности лекарства · To make clinical trials more efficient/ можно усовершенствовать техники лечения · To make drug treatments more effective/ создать более эфективные лекарства © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Practical applications/ практическое применение · Comparison of protein expression in diseased and normal tissues/ сравнение экспрессии белков в нормальных и патологических клетках · Likely to reveal new drug targets/ необходимы для выявления новых мишеней для лекарств · Today ~500 drug targets/ сегодня известно около 500 таких мишеней · Estimates of possible drug targets: 10, 000– 20, 000/ по оценкам, возможных мишеней существует около 20000 · Protein expression signatures associated with drug toxicity/ характерная черта экспрессии белка может быть индикатором токсичности лекарства · To make clinical trials more efficient/ можно усовершенствовать техники лечения · To make drug treatments more effective/ создать более эфективные лекарства © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Technologies for proteomics/ технологии протеомики · 2 -D gel electrophoresis/ двумерный элекстрофорез · Separates proteins in a mixture on the basis of their molecular weight and charge/Разделение белков в смеси на основе их молекулярной массы и заряда · Mass spectrometry/ Масс-спектрометрия · Reveals identity of proteins/ Показывает идентичность белков · Protein chips/ белковые чипы · A wide variety of identification methods/ Широкий спектр методов идентификации · Yeast two-hybrid method/ дрожжевой метод двух белков · Determines how proteins interact with each other/ Определяет, как белки взаимодействуют друг с другом · Biochemical genomics/ биохимическая геномика · Screens gene products for biochemical activity/ тестирует генные продукты на их биохимическую активность © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Technologies for proteomics/ технологии протеомики · 2 -D gel electrophoresis/ двумерный элекстрофорез · Separates proteins in a mixture on the basis of their molecular weight and charge/Разделение белков в смеси на основе их молекулярной массы и заряда · Mass spectrometry/ Масс-спектрометрия · Reveals identity of proteins/ Показывает идентичность белков · Protein chips/ белковые чипы · A wide variety of identification methods/ Широкий спектр методов идентификации · Yeast two-hybrid method/ дрожжевой метод двух белков · Determines how proteins interact with each other/ Определяет, как белки взаимодействуют друг с другом · Biochemical genomics/ биохимическая геномика · Screens gene products for biochemical activity/ тестирует генные продукты на их биохимическую активность © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
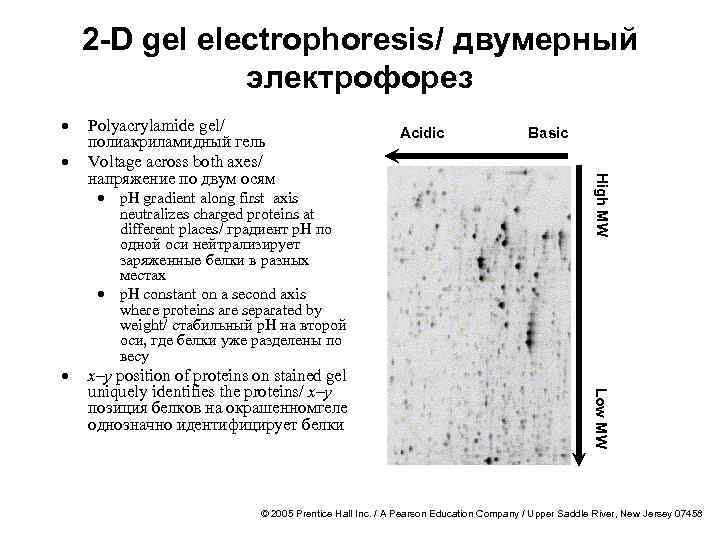 2 -D gel electrophoresis/ двумерный электрофорез · · neutralizes charged proteins at different places/ градиент р. Н по одной оси нейтрализирует заряженные белки в разных местах · p. H constant on a second axis where proteins are separated by weight/ стабильный р. Н на второй оси, где белки уже разделены по весу Basic Low MW x–y position of proteins on stained gel uniquely identifies the proteins/ x–y позиция белков на окрашенномгеле однозначно идентифицирует белки Acidic High MW · Polyacrylamide gel/ полиакриламидный гель Voltage across both axes/ напряжение по двум осям · p. H gradient along first axis © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
2 -D gel electrophoresis/ двумерный электрофорез · · neutralizes charged proteins at different places/ градиент р. Н по одной оси нейтрализирует заряженные белки в разных местах · p. H constant on a second axis where proteins are separated by weight/ стабильный р. Н на второй оси, где белки уже разделены по весу Basic Low MW x–y position of proteins on stained gel uniquely identifies the proteins/ x–y позиция белков на окрашенномгеле однозначно идентифицирует белки Acidic High MW · Polyacrylamide gel/ полиакриламидный гель Voltage across both axes/ напряжение по двум осям · p. H gradient along first axis © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Differential in gel electrophoresis/ разделение в электрофорезном геле · Label protein samples from control and experimental tissues/ белки образца, контроля и эксперимента обозначают with benzoic acid Cy 3 · Fluorescent dye #1 for control/ флюоресцентным красителем № 1 - контроль · Fluorescent dye #2 for experimental sample/ вторым красителем - эксперимент · Mix protein samples together/ смешивают образцы · Identify identical proteins from different samples by dye color/ идентифицируют белки разных образцов по цвету красителя without benzoic acid Cy 5 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Differential in gel electrophoresis/ разделение в электрофорезном геле · Label protein samples from control and experimental tissues/ белки образца, контроля и эксперимента обозначают with benzoic acid Cy 3 · Fluorescent dye #1 for control/ флюоресцентным красителем № 1 - контроль · Fluorescent dye #2 for experimental sample/ вторым красителем - эксперимент · Mix protein samples together/ смешивают образцы · Identify identical proteins from different samples by dye color/ идентифицируют белки разных образцов по цвету красителя without benzoic acid Cy 5 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Caveats associated with 2 -D gels/ примечания к данному методу · Poor performance of 2 -D gels for the following: / этот метод плохо подходит, когда используются: · Very large proteins/ большие белки · Very small proteins/ маленькие белки · Less abundant proteins/ маловстречающиеся белки · Membrane-bound proteins/ белки, связанные с мембраной · Presumably, the most promising drug targets/ возможно, наиболее подходящие мишени для лекарств © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Caveats associated with 2 -D gels/ примечания к данному методу · Poor performance of 2 -D gels for the following: / этот метод плохо подходит, когда используются: · Very large proteins/ большие белки · Very small proteins/ маленькие белки · Less abundant proteins/ маловстречающиеся белки · Membrane-bound proteins/ белки, связанные с мембраной · Presumably, the most promising drug targets/ возможно, наиболее подходящие мишени для лекарств © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Mass spectrometry/ массспектрометрия · Measures mass-to-charge ratio/ соотношение массы и заряда · Components of mass spectrometer/ компоненты спектрофотометра · Ion source/ источник ионов · Mass analyzer/ анализатор масс · Ion detector/ ионный детектор · Data acquisition unit/ Устройство сбора данных A mass spectrometer © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Mass spectrometry/ массспектрометрия · Measures mass-to-charge ratio/ соотношение массы и заряда · Components of mass spectrometer/ компоненты спектрофотометра · Ion source/ источник ионов · Mass analyzer/ анализатор масс · Ion detector/ ионный детектор · Data acquisition unit/ Устройство сбора данных A mass spectrometer © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Ion sources used for proteomics/ ионные источники, используемые в протеомике · · · Proteomics requires specialized ion sources/протеомика нуждается в специальных ионных источниках Electrospray Ionization (ESI)/ электроспрей · With capillary electrophoresis and liquid chromatography/ с помощью капилярного электрофореза и жидкой хроматографии Matrix-assisted laser desorption/ionization (MALDI)/Матрично- ESI MALDI активированная лазерная десорбция/ионизация · Extracts ions from sample surface/ экстрагирует ионы из образца © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Ion sources used for proteomics/ ионные источники, используемые в протеомике · · · Proteomics requires specialized ion sources/протеомика нуждается в специальных ионных источниках Electrospray Ionization (ESI)/ электроспрей · With capillary electrophoresis and liquid chromatography/ с помощью капилярного электрофореза и жидкой хроматографии Matrix-assisted laser desorption/ionization (MALDI)/Матрично- ESI MALDI активированная лазерная десорбция/ионизация · Extracts ions from sample surface/ экстрагирует ионы из образца © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
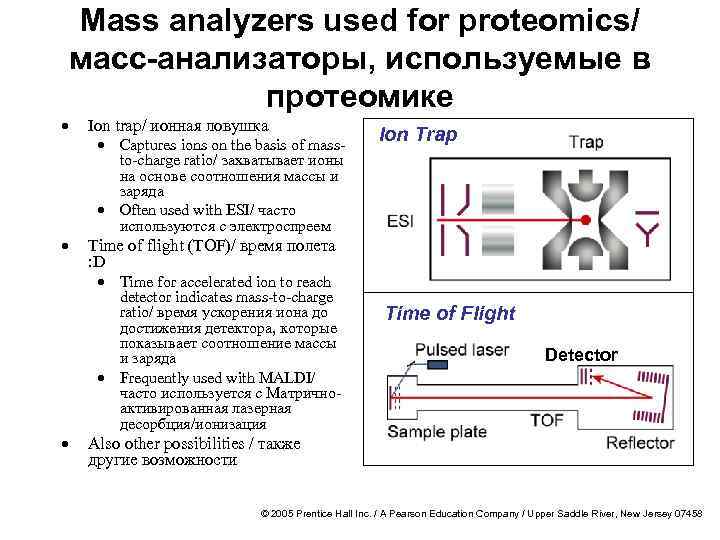 · · · Mass analyzers used for proteomics/ масс-анализаторы, используемые в протеомике Ion trap/ ионная ловушка · Captures ions on the basis of mass- Ion Trap to-charge ratio/ захватывает ионы на основе соотношения массы и заряда · Often used with ESI/ часто используются с электроспреем Time of flight (TOF)/ время полета : D · Time for accelerated ion to reach detector indicates mass-to-charge ratio/ время ускорения иона до достижения детектора, которые показывает соотношение массы и заряда · Frequently used with MALDI/ часто используется с Матричноактивированная лазерная десорбция/ионизация Time of Flight Detector Also other possibilities / также другие возможности © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
· · · Mass analyzers used for proteomics/ масс-анализаторы, используемые в протеомике Ion trap/ ионная ловушка · Captures ions on the basis of mass- Ion Trap to-charge ratio/ захватывает ионы на основе соотношения массы и заряда · Often used with ESI/ часто используются с электроспреем Time of flight (TOF)/ время полета : D · Time for accelerated ion to reach detector indicates mass-to-charge ratio/ время ускорения иона до достижения детектора, которые показывает соотношение массы и заряда · Frequently used with MALDI/ часто используется с Матричноактивированная лазерная десорбция/ионизация Time of Flight Detector Also other possibilities / также другие возможности © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
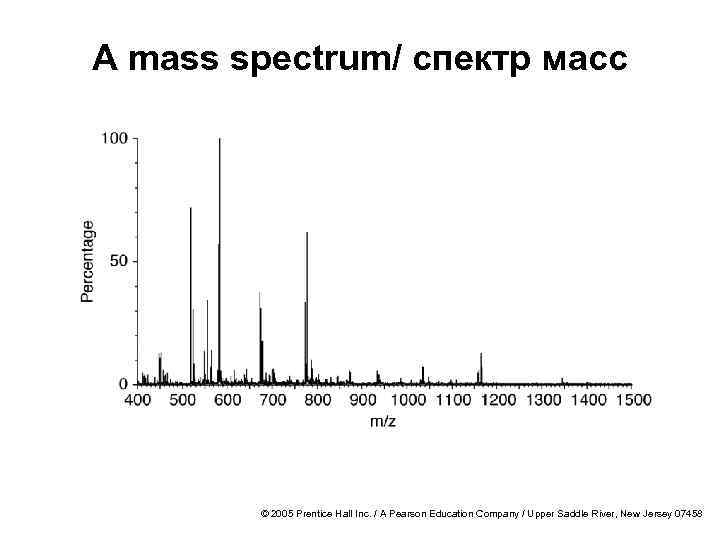 A mass spectrum/ спектр масс © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
A mass spectrum/ спектр масс © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Identifying proteins with mass spectrometry / Идентификация белков с масс-спектрометрией · Preparation of protein sample/ подготовка образца · Extraction from a gel/ экстрагирование из геля · Digestion by proteases — e. g. , trypsin/ действие протеазами (к примеру, трипсином) · Mass spectrometer measures mass-charge ratio of peptide fragments/ спектрометр измеряет зависимость массы от заряда пептидных фрагментов · Identified peptides are compared with database/ идентифицированные белки сравнивают с базой данных · Software used to generate theoretical peptide mass fingerprint (PMF) for all proteins in database/ используются программы для создания теоретического отпечатка массы белка · Match of experimental readout to database PMF allows researchers to identify the protein/ потивопоставляют экспериментальные данные с базой для того, чтобы идентифицировать белок © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Identifying proteins with mass spectrometry / Идентификация белков с масс-спектрометрией · Preparation of protein sample/ подготовка образца · Extraction from a gel/ экстрагирование из геля · Digestion by proteases — e. g. , trypsin/ действие протеазами (к примеру, трипсином) · Mass spectrometer measures mass-charge ratio of peptide fragments/ спектрометр измеряет зависимость массы от заряда пептидных фрагментов · Identified peptides are compared with database/ идентифицированные белки сравнивают с базой данных · Software used to generate theoretical peptide mass fingerprint (PMF) for all proteins in database/ используются программы для создания теоретического отпечатка массы белка · Match of experimental readout to database PMF allows researchers to identify the protein/ потивопоставляют экспериментальные данные с базой для того, чтобы идентифицировать белок © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
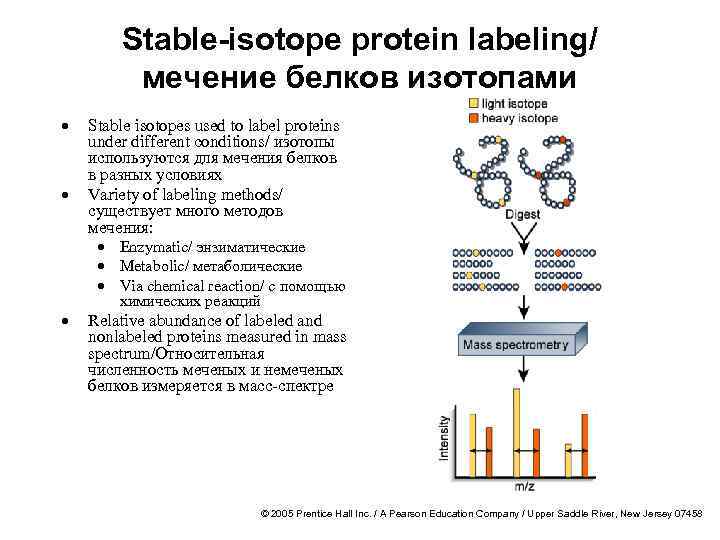 Stable-isotope protein labeling/ мечение белков изотопами · · · Stable isotopes used to label proteins under different conditions/ изотопы используются для мечения белков в разных условиях Variety of labeling methods/ существует много методов мечения: · Enzymatic/ энзиматические · Metabolic/ метаболические · Via chemical reaction/ с помощью химических реакций Relative abundance of labeled and nonlabeled proteins measured in mass spectrum/Относительная численность меченых и немеченых белков измеряется в масс-спектре © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Stable-isotope protein labeling/ мечение белков изотопами · · · Stable isotopes used to label proteins under different conditions/ изотопы используются для мечения белков в разных условиях Variety of labeling methods/ существует много методов мечения: · Enzymatic/ энзиматические · Metabolic/ метаболические · Via chemical reaction/ с помощью химических реакций Relative abundance of labeled and nonlabeled proteins measured in mass spectrum/Относительная численность меченых и немеченых белков измеряется в масс-спектре © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
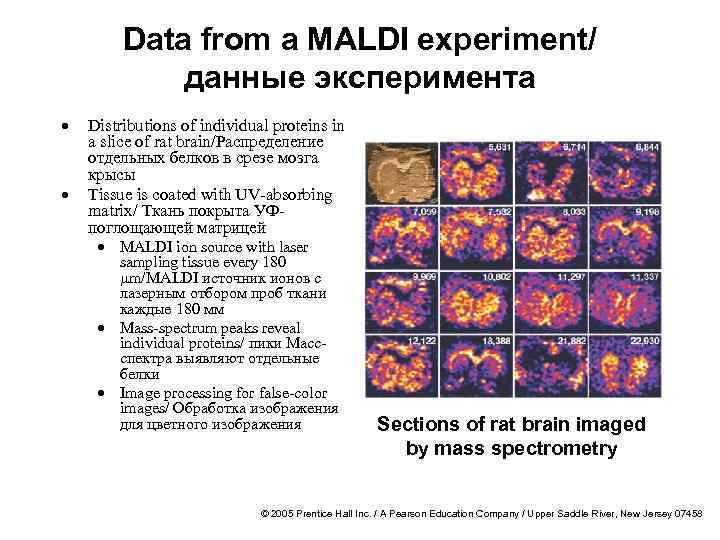 Data from a MALDI experiment/ данные эксперимента · · Distributions of individual proteins in a slice of rat brain/Распределение отдельных белков в срезе мозга крысы Tissue is coated with UV-absorbing matrix/ Ткань покрыта УФпоглощающей матрицей · MALDI ion source with laser sampling tissue every 180 mm/MALDI источник ионов с лазерным отбором проб ткани каждые 180 мм · Mass-spectrum peaks reveal individual proteins/ пики Массспектра выявляют отдельные белки · Image processing for false-color images/ Обработка изображения для цветного изображения Sections of rat brain imaged by mass spectrometry © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Data from a MALDI experiment/ данные эксперимента · · Distributions of individual proteins in a slice of rat brain/Распределение отдельных белков в срезе мозга крысы Tissue is coated with UV-absorbing matrix/ Ткань покрыта УФпоглощающей матрицей · MALDI ion source with laser sampling tissue every 180 mm/MALDI источник ионов с лазерным отбором проб ткани каждые 180 мм · Mass-spectrum peaks reveal individual proteins/ пики Массспектра выявляют отдельные белки · Image processing for false-color images/ Обработка изображения для цветного изображения Sections of rat brain imaged by mass spectrometry © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Limitations of mass spectrometry/ ограничения масс-спектроскопии · Not very good at identifying minute quantities of protein/ не слишком хороша для идентификации незначительных количеств белка · Trouble dealing with phosphorylated proteins/ с фосфорилированными белками · Doesn’t provide concentrations of proteins/ Не обеспечивает концентрацию белков · Improved software eliminating human analysis is necessary for high-throughput projects/ Улучшенное программное обеспечение для элиминации устранения человеческого вмешатльства, также необходимо для высокой пропускной способности © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Limitations of mass spectrometry/ ограничения масс-спектроскопии · Not very good at identifying minute quantities of protein/ не слишком хороша для идентификации незначительных количеств белка · Trouble dealing with phosphorylated proteins/ с фосфорилированными белками · Doesn’t provide concentrations of proteins/ Не обеспечивает концентрацию белков · Improved software eliminating human analysis is necessary for high-throughput projects/ Улучшенное программное обеспечение для элиминации устранения человеческого вмешатльства, также необходимо для высокой пропускной способности © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
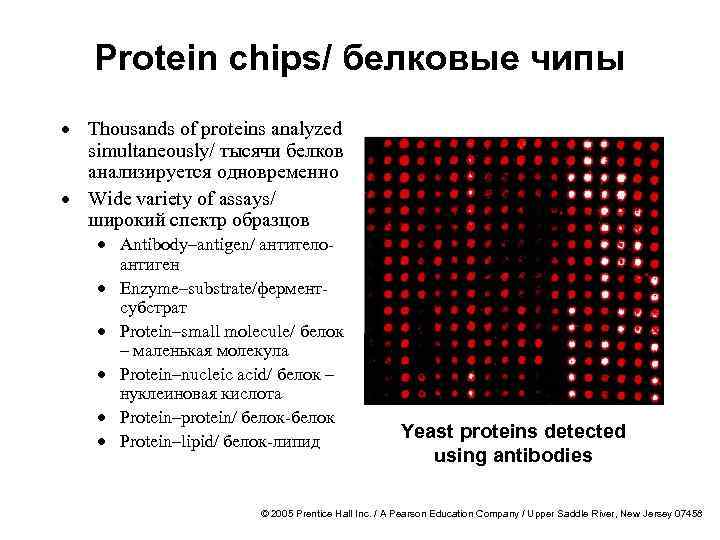 Protein chips/ белковые чипы · Thousands of proteins analyzed simultaneously/ тысячи белков анализируется одновременно · Wide variety of assays/ широкий спектр образцов · Antibody–antigen/ антителоантиген · Enzyme–substrate/ферментсубстрат · Protein–small molecule/ белок – маленькая молекула · Protein–nucleic acid/ белок – нуклеиновая кислота · Protein–protein/ белок-белок · Protein–lipid/ белок-липид Yeast proteins detected using antibodies © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Protein chips/ белковые чипы · Thousands of proteins analyzed simultaneously/ тысячи белков анализируется одновременно · Wide variety of assays/ широкий спектр образцов · Antibody–antigen/ антителоантиген · Enzyme–substrate/ферментсубстрат · Protein–small molecule/ белок – маленькая молекула · Protein–nucleic acid/ белок – нуклеиновая кислота · Protein–protein/ белок-белок · Protein–lipid/ белок-липид Yeast proteins detected using antibodies © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
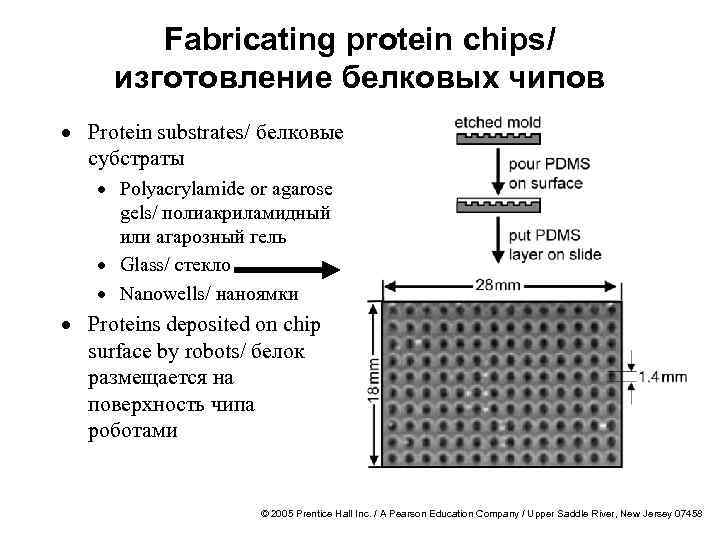 Fabricating protein chips/ изготовление белковых чипов · Protein substrates/ белковые субстраты · Polyacrylamide or agarose gels/ полиакриламидный или агарозный гель · Glass/ стекло · Nanowells/ наноямки · Proteins deposited on chip surface by robots/ белок размещается на поверхность чипа роботами © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Fabricating protein chips/ изготовление белковых чипов · Protein substrates/ белковые субстраты · Polyacrylamide or agarose gels/ полиакриламидный или агарозный гель · Glass/ стекло · Nanowells/ наноямки · Proteins deposited on chip surface by robots/ белок размещается на поверхность чипа роботами © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
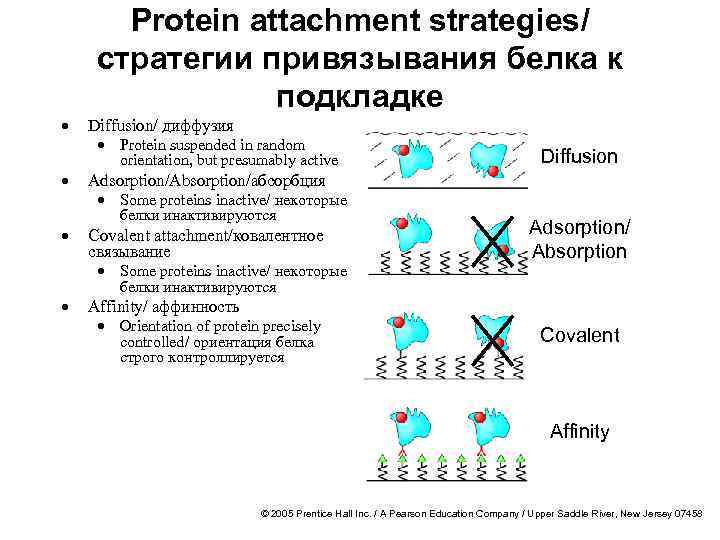 · · Protein attachment strategies/ стратегии привязывания белка к подкладке Diffusion/ диффузия · Protein suspended in random orientation, but presumably active Diffusion Adsorption/Absorption/абсорбция · Some proteins inactive/ некоторые белки инактивируются Covalent attachment/ковалентное связывание · Some proteins inactive/ некоторые Adsorption/ Absorption белки инактивируются Affinity/ аффинность · Orientation of protein precisely controlled/ ориентация белка строго контроллируется Covalent Affinity © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
· · Protein attachment strategies/ стратегии привязывания белка к подкладке Diffusion/ диффузия · Protein suspended in random orientation, but presumably active Diffusion Adsorption/Absorption/абсорбция · Some proteins inactive/ некоторые белки инактивируются Covalent attachment/ковалентное связывание · Some proteins inactive/ некоторые Adsorption/ Absorption белки инактивируются Affinity/ аффинность · Orientation of protein precisely controlled/ ориентация белка строго контроллируется Covalent Affinity © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Classes of capture molecules/ классы поглощаемых молекул · Different capture molecules must be used to study different interactions/ разнообразные поглощаемые молекулы должны быть использованы для изучения различных взаимодействий · Examples/ примеры · Antibodies (or antigens) for detection/ антитела/антигены · Proteins for protein-protein interaction/ белки для белокбелкового взаимодействия · Enzyme-substrate for biochemical function/ фермент -субстрат для биохимических функций Antigen– antibody Protein– protein Aptamers Enzyme– substrate Receptor– ligand © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Classes of capture molecules/ классы поглощаемых молекул · Different capture molecules must be used to study different interactions/ разнообразные поглощаемые молекулы должны быть использованы для изучения различных взаимодействий · Examples/ примеры · Antibodies (or antigens) for detection/ антитела/антигены · Proteins for protein-protein interaction/ белки для белокбелкового взаимодействия · Enzyme-substrate for biochemical function/ фермент -субстрат для биохимических функций Antigen– antibody Protein– protein Aptamers Enzyme– substrate Receptor– ligand © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Reading out results/ выводы · Fluorescence/ флюоресценция · Most common method/ наиболее распространенный метод · Fluorescent probe or tag / флюоресцентная проба или метка · Can be read out using standard nucleic acid microarray technology/ может быть вычислена с помощью стандартных микрочипов нуклеиновых кислот · Surface-enhanced laser desorption/ionization (SELDI)/ поверхностно-усиленная лазерная десорпция/ионизация · Laser ionizes proteins captured by chip/ лазерная ионизация белков улавливается чипом · Mass spectrometer analyzes peptide fragments/ масс-спектрометр анализирует пептидные фрагменты · Atomic-force microscopy/ атомно-силовая микроскопия · Detects changes in chip surface due to captured proteins/ детектирует изменение окраса на поверхности чипа для захвата белка © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Reading out results/ выводы · Fluorescence/ флюоресценция · Most common method/ наиболее распространенный метод · Fluorescent probe or tag / флюоресцентная проба или метка · Can be read out using standard nucleic acid microarray technology/ может быть вычислена с помощью стандартных микрочипов нуклеиновых кислот · Surface-enhanced laser desorption/ionization (SELDI)/ поверхностно-усиленная лазерная десорпция/ионизация · Laser ionizes proteins captured by chip/ лазерная ионизация белков улавливается чипом · Mass spectrometer analyzes peptide fragments/ масс-спектрометр анализирует пептидные фрагменты · Atomic-force microscopy/ атомно-силовая микроскопия · Detects changes in chip surface due to captured proteins/ детектирует изменение окраса на поверхности чипа для захвата белка © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Difficulties in designing protein chips/ проблемы в создании белковых чипов · Unique process is necessary for constructing each probe element/ Уникальный процесс необходим для построения каждого зонда элемент · Challenging to produce and purify each protein on chip/ поместить и очистить каждый белок на чипе · Proteins can be hydrophobic or hydrophilic/Белки могут быть гидрофобными или гидрофильными · Difficult to design a chip that can detect both/ Трудно разработать чип, который может обнаруживать оба © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Difficulties in designing protein chips/ проблемы в создании белковых чипов · Unique process is necessary for constructing each probe element/ Уникальный процесс необходим для построения каждого зонда элемент · Challenging to produce and purify each protein on chip/ поместить и очистить каждый белок на чипе · Proteins can be hydrophobic or hydrophilic/Белки могут быть гидрофобными или гидрофильными · Difficult to design a chip that can detect both/ Трудно разработать чип, который может обнаруживать оба © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Yeast two-hybrid method/ дрожжевой метод «двух гибридов · Goal: Determine how proteins interact with each other/ цель: определить как белки взаимодействуют друг с другом · Method/ метод · Use yeast transcription factors/ используются дрожжевые транскрипционные факторы · Gene expression requires the following: генная экспрессия нуждается в следующем: · A DNA-binding domain: днк-связывающий домен · An activation domain/ активирующий домен · A basic transcription apparatus/ базовый транскрипционный аппарат · Attach protein 1 to DNA-binding domain (bait)/ прикрепить белок 1 к днк -связывающему домену · Attach protein 2 to activation domain (prey)/ привязать белок 2 к активирующему домену · Reporter gene expressed only if protein 1 and protein 2 interact with each other/ репортерный ген экспрессируется лишь если белок 1 и бело 2 взаимодействуют друг с другом © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Yeast two-hybrid method/ дрожжевой метод «двух гибридов · Goal: Determine how proteins interact with each other/ цель: определить как белки взаимодействуют друг с другом · Method/ метод · Use yeast transcription factors/ используются дрожжевые транскрипционные факторы · Gene expression requires the following: генная экспрессия нуждается в следующем: · A DNA-binding domain: днк-связывающий домен · An activation domain/ активирующий домен · A basic transcription apparatus/ базовый транскрипционный аппарат · Attach protein 1 to DNA-binding domain (bait)/ прикрепить белок 1 к днк -связывающему домену · Attach protein 2 to activation domain (prey)/ привязать белок 2 к активирующему домену · Reporter gene expressed only if protein 1 and protein 2 interact with each other/ репортерный ген экспрессируется лишь если белок 1 и бело 2 взаимодействуют друг с другом © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
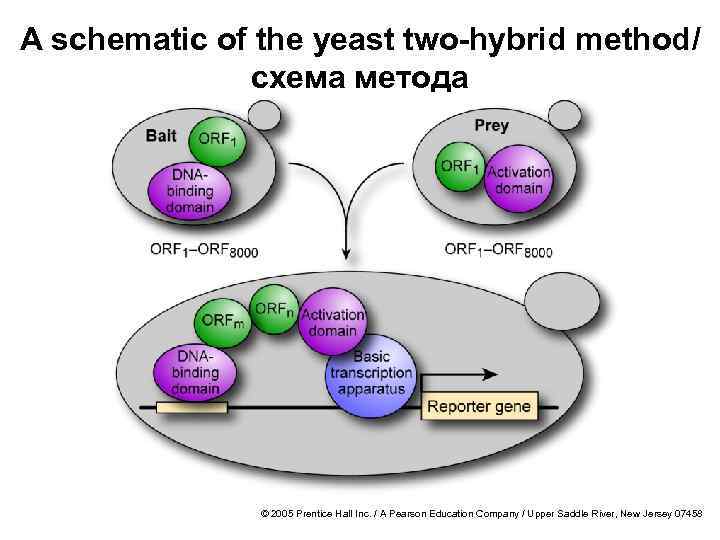 A schematic of the yeast two-hybrid method/ схема метода © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
A schematic of the yeast two-hybrid method/ схема метода © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Results from a yeast two-hybrid experiment/ выводы двугибридного метода на дрожжах · Goal: To characterize protein–protein interactions among 6, 144 yeast ORFs/ цель: охарактеризировать белок-белковіе взаимодействия среди 6144 открытых рамок считывания дрожжей · 5, 345 were successfully cloned into yeast as both bait and prey/ 5345 были успешно клонированы у дрожжей по bait and prey (см слайд 27) · Identity of ORFs determined by DNA sequencing in hybrid yeast/ идентифицировать открытую рамку считывания по днксеквенированию в гибридах дрожжей · 692 protein–protein interaction pairs/692 белок-белкового взаимодействия · Interactions involved 817 ORFs/ в взаимодействия вовлечены 817 открытых рамок считывания © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Results from a yeast two-hybrid experiment/ выводы двугибридного метода на дрожжах · Goal: To characterize protein–protein interactions among 6, 144 yeast ORFs/ цель: охарактеризировать белок-белковіе взаимодействия среди 6144 открытых рамок считывания дрожжей · 5, 345 were successfully cloned into yeast as both bait and prey/ 5345 были успешно клонированы у дрожжей по bait and prey (см слайд 27) · Identity of ORFs determined by DNA sequencing in hybrid yeast/ идентифицировать открытую рамку считывания по днксеквенированию в гибридах дрожжей · 692 protein–protein interaction pairs/692 белок-белкового взаимодействия · Interactions involved 817 ORFs/ в взаимодействия вовлечены 817 открытых рамок считывания © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Caveats associated with the yeast two -hybrid method/ примечания, связанные с дрожжевым методом · There is evidence that other methods may be more sensitive/ очевидно, что другой метод может быть более чувствительным · Some inaccuracy reported when compared against known protein–protein interactions/ возникают некоторые неточности при сравнении с известными белок-белковыми взаимодействиями (далее по тексту –ББВ!!!) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Caveats associated with the yeast two -hybrid method/ примечания, связанные с дрожжевым методом · There is evidence that other methods may be more sensitive/ очевидно, что другой метод может быть более чувствительным · Some inaccuracy reported when compared against known protein–protein interactions/ возникают некоторые неточности при сравнении с известными белок-белковыми взаимодействиями (далее по тексту –ББВ!!!) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Subcellular localization of the yeast proteome/ субклеточная локализация дрожжевого протеома · Complete genome sequences allow each ORF to be precisely tagged with a reporter molecule/ целая геномная последовательность позволяет открытой рамке считывания быть строго помеченой репортерной молекулой · Tagged ORF proteins indicate subcellular localization/ меченые ОРС белков сообщают о локализации · Useful for the following: / используется для следующего: · Correlating to regulatory modules/ коррелирует с регуляторными модулями · Verifying data on protein–protein interactions/ проверка ББВ в базе данных · Annotating genome sequence/ аннотирование геномных последовательностей © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Subcellular localization of the yeast proteome/ субклеточная локализация дрожжевого протеома · Complete genome sequences allow each ORF to be precisely tagged with a reporter molecule/ целая геномная последовательность позволяет открытой рамке считывания быть строго помеченой репортерной молекулой · Tagged ORF proteins indicate subcellular localization/ меченые ОРС белков сообщают о локализации · Useful for the following: / используется для следующего: · Correlating to regulatory modules/ коррелирует с регуляторными модулями · Verifying data on protein–protein interactions/ проверка ББВ в базе данных · Annotating genome sequence/ аннотирование геномных последовательностей © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
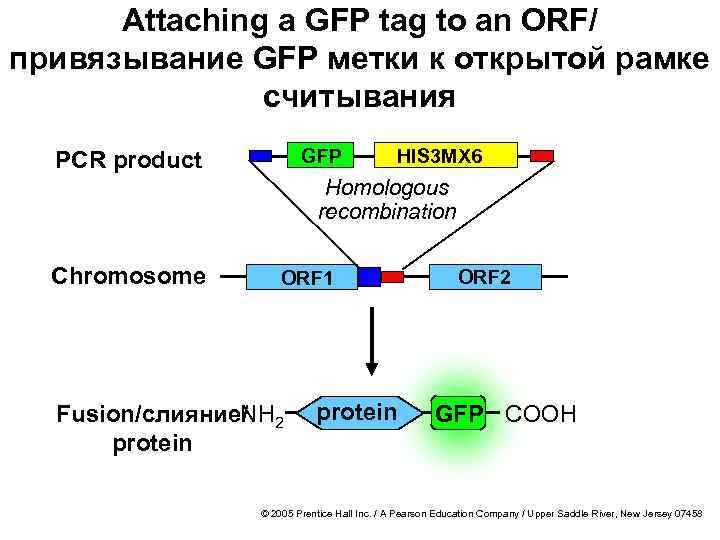 Attaching a GFP tag to an ORF/ привязывание GFP метки к открытой рамке считывания GFP PCR product HIS 3 MX 6 Homologous recombination Chromosome ORF 1 NH Fusion/слияние/ 2 protein ORF 2 GFP COOH © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Attaching a GFP tag to an ORF/ привязывание GFP метки к открытой рамке считывания GFP PCR product HIS 3 MX 6 Homologous recombination Chromosome ORF 1 NH Fusion/слияние/ 2 protein ORF 2 GFP COOH © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Location of proteins revealed/локализация обнаруженных белков · 75% of yeast proteome localized/ 75% дрожжевого протеома локализировано · > 40% of proteins in cytoplasm/ 40% белков в цитоплазме · 67% of proteins were previously unlocalized/ 67% - не имеют постоянного места · Localizations correlate with transcriptional modules/ локализация связана с транскрипционными модулями cytoplasm nucleus A protein localized to the nucleus © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Location of proteins revealed/локализация обнаруженных белков · 75% of yeast proteome localized/ 75% дрожжевого протеома локализировано · > 40% of proteins in cytoplasm/ 40% белков в цитоплазме · 67% of proteins were previously unlocalized/ 67% - не имеют постоянного места · Localizations correlate with transcriptional modules/ локализация связана с транскрипционными модулями cytoplasm nucleus A protein localized to the nucleus © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Pregenomics biochemical assays/ прегеномный биохимический анализ · Methods used to find genes responsible for specific biochemical activity before the inception of genomics/ методы, использованные для поиска генов ответственны за специфическую биохимическую активность · Laboriously purify responsible protein/ трудно очистить белок · Often expensive and time consuming/ часто дорого и занимает много времени · Expression cloning/ клонировано · Introduce c. DNA pools into cells/ помещение к. ДНК в клетки · Look for biochemical activity in those cells/ наблюдени биохимической активности в клетках · Caveat: Often difficult to detect biochemical activity in cell’s biochemical “background”/ примечание: часто сложно детектировать биохимическую активность в клетках © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Pregenomics biochemical assays/ прегеномный биохимический анализ · Methods used to find genes responsible for specific biochemical activity before the inception of genomics/ методы, использованные для поиска генов ответственны за специфическую биохимическую активность · Laboriously purify responsible protein/ трудно очистить белок · Often expensive and time consuming/ часто дорого и занимает много времени · Expression cloning/ клонировано · Introduce c. DNA pools into cells/ помещение к. ДНК в клетки · Look for biochemical activity in those cells/ наблюдени биохимической активности в клетках · Caveat: Often difficult to detect biochemical activity in cell’s biochemical “background”/ примечание: часто сложно детектировать биохимическую активность в клетках © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Biochemical genomics/ биохимическая геномика · Genome of an organism is already known/ используется, когда геном известен · Approach/ подход: · Construct plasmids for all ORFs/ создание плазмиды для всех ОРС · Attach ORFs to sequence that will facilitate purification/соответствие ОРС последовательности, которое облегчит очищение · Transform cells/ трансформация клеток · Isolate ORF products/ изолирование продуктов ОРС · Test for biochemical activity/ тест на биохимическую активность © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Biochemical genomics/ биохимическая геномика · Genome of an organism is already known/ используется, когда геном известен · Approach/ подход: · Construct plasmids for all ORFs/ создание плазмиды для всех ОРС · Attach ORFs to sequence that will facilitate purification/соответствие ОРС последовательности, которое облегчит очищение · Transform cells/ трансформация клеток · Isolate ORF products/ изолирование продуктов ОРС · Test for biochemical activity/ тест на биохимическую активность © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
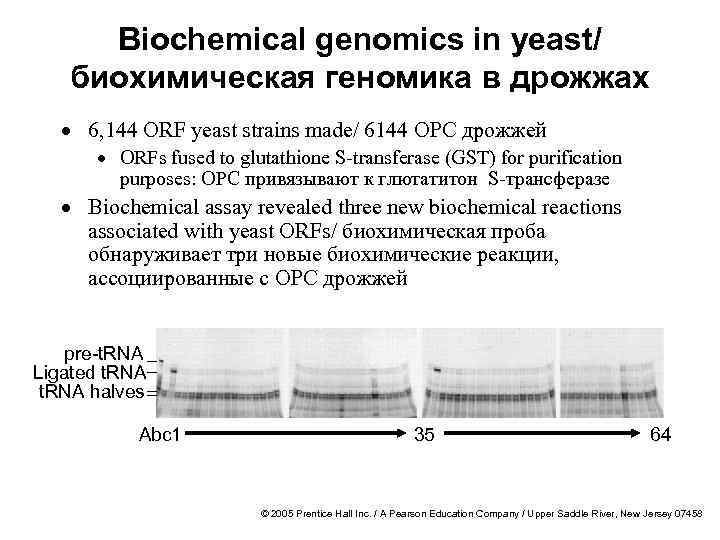 Biochemical genomics in yeast/ биохимическая геномика в дрожжах · 6, 144 ORF yeast strains made/ 6144 ОРС дрожжей · ORFs fused to glutathione S-transferase (GST) for purification purposes: ОРС привязывают к глютатитон S-трансферазе · Biochemical assay revealed three new biochemical reactions associated with yeast ORFs/ биохимическая проба обнаруживает три новые биохимические реакции, ассоциированные с ОРС дрожжей pre-t. RNA Ligated t. RNA halves Abc 1 35 64 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Biochemical genomics in yeast/ биохимическая геномика в дрожжах · 6, 144 ORF yeast strains made/ 6144 ОРС дрожжей · ORFs fused to glutathione S-transferase (GST) for purification purposes: ОРС привязывают к глютатитон S-трансферазе · Biochemical assay revealed three new biochemical reactions associated with yeast ORFs/ биохимическая проба обнаруживает три новые биохимические реакции, ассоциированные с ОРС дрожжей pre-t. RNA Ligated t. RNA halves Abc 1 35 64 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
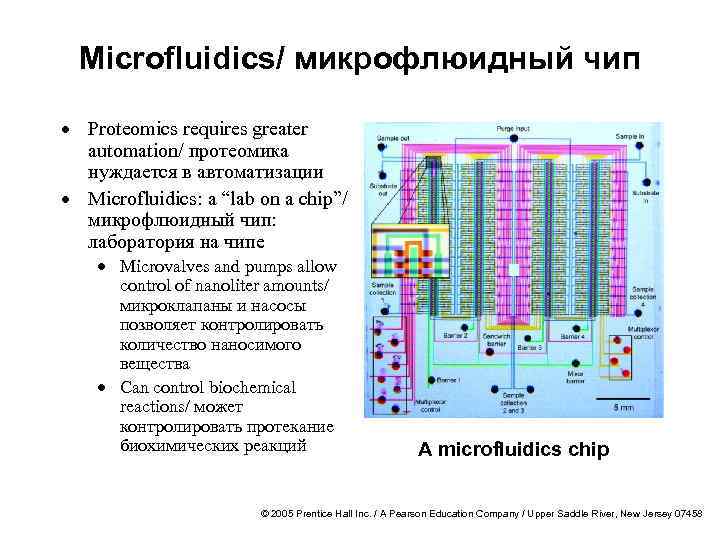 Microfluidics/ микрофлюидный чип · Proteomics requires greater automation/ протеомика нуждается в автоматизации · Microfluidics: a “lab on a chip”/ микрофлюидный чип: лаборатория на чипе · Microvalves and pumps allow control of nanoliter amounts/ микроклапаны и насосы позволяет контролировать количество наносимого вещества · Can control biochemical reactions/ может контролировать протекание биохимических реакций A microfluidics chip © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Microfluidics/ микрофлюидный чип · Proteomics requires greater automation/ протеомика нуждается в автоматизации · Microfluidics: a “lab on a chip”/ микрофлюидный чип: лаборатория на чипе · Microvalves and pumps allow control of nanoliter amounts/ микроклапаны и насосы позволяет контролировать количество наносимого вещества · Can control biochemical reactions/ может контролировать протекание биохимических реакций A microfluidics chip © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Microfluidics in action/ чипы в действии loading mixing compartmentalization purging 500 mm © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Microfluidics in action/ чипы в действии loading mixing compartmentalization purging 500 mm © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
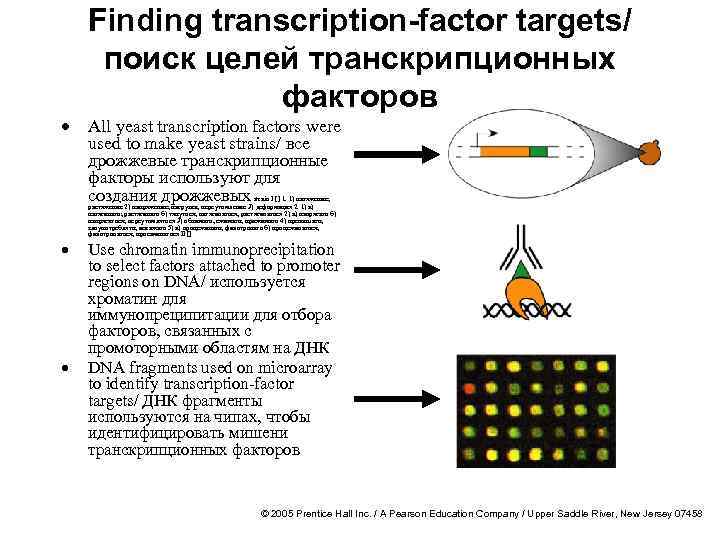 · · · Finding transcription-factor targets/ поиск целей транскрипционных факторов All yeast transcription factors were used to make yeast strains/ все дрожжевые транскрипционные факторы используют для создания дрожжевых strain I [] 1. 1) натяжение; растяжение 2) напряжение; нагрузка; переутомление 3) деформация 2. 1) а) натягивать; растягивать б) тянуться; натягиваться, растягиваться 2) а) напрягать б) напрягаться; переутомляться 3) обнимать, сжимать; прижимать 4) превышать; злоупотреблять; искажать 5) а) процеживать; фильтровать б) процеживаться; фильтроваться; просачиваться II [] Use chromatin immunoprecipitation to select factors attached to promoter regions on DNA/ используется хроматин для иммунопреципитации для отбора факторов, связанных с промоторными областям на ДНК DNA fragments used on microarray to identify transcription-factor targets/ ДНК фрагменты используются на чипах, чтобы идентифицировать мишени транскрипционных факторов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
· · · Finding transcription-factor targets/ поиск целей транскрипционных факторов All yeast transcription factors were used to make yeast strains/ все дрожжевые транскрипционные факторы используют для создания дрожжевых strain I [] 1. 1) натяжение; растяжение 2) напряжение; нагрузка; переутомление 3) деформация 2. 1) а) натягивать; растягивать б) тянуться; натягиваться, растягиваться 2) а) напрягать б) напрягаться; переутомляться 3) обнимать, сжимать; прижимать 4) превышать; злоупотреблять; искажать 5) а) процеживать; фильтровать б) процеживаться; фильтроваться; просачиваться II [] Use chromatin immunoprecipitation to select factors attached to promoter regions on DNA/ используется хроматин для иммунопреципитации для отбора факторов, связанных с промоторными областям на ДНК DNA fragments used on microarray to identify transcription-factor targets/ ДНК фрагменты используются на чипах, чтобы идентифицировать мишени транскрипционных факторов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
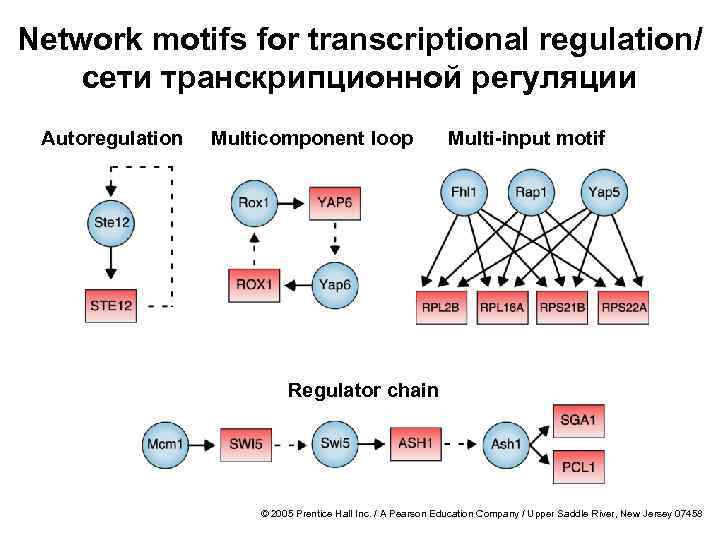 Network motifs for transcriptional regulation/ сети транскрипционной регуляции Autoregulation Multicomponent loop Multi-input motif Regulator chain © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Network motifs for transcriptional regulation/ сети транскрипционной регуляции Autoregulation Multicomponent loop Multi-input motif Regulator chain © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Studying proteins with posttranslational modifications/ изучение белков с посттрансляционными модификациями · Example: tyrosine phosphorylation/ пример: фосфорилированый тирозин · Traditional method/ традиционный метод · Radioactive labeling with 32 P followed by gel electrophoresis or chromatography/ радиоактивная метка фосфором следует за гель-электрофорезом или хроматографией · Problems using mass spectrometry are being overcome to allow high-throughput analysis/ преодоление проблем, появляющихся при масс-спектрофотометрии позволяют повысить пропускную способность анализа · Better purification techniques/ лучшие техники очищения · Mass spectrometers more capable of detecting phosphorylated peptides/ масс-спектрофотометры более способны определить фосфорилированые пептиды © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Studying proteins with posttranslational modifications/ изучение белков с посттрансляционными модификациями · Example: tyrosine phosphorylation/ пример: фосфорилированый тирозин · Traditional method/ традиционный метод · Radioactive labeling with 32 P followed by gel electrophoresis or chromatography/ радиоактивная метка фосфором следует за гель-электрофорезом или хроматографией · Problems using mass spectrometry are being overcome to allow high-throughput analysis/ преодоление проблем, появляющихся при масс-спектрофотометрии позволяют повысить пропускную способность анализа · Better purification techniques/ лучшие техники очищения · Mass spectrometers more capable of detecting phosphorylated peptides/ масс-спектрофотометры более способны определить фосфорилированые пептиды © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 HUPO · Human Proteome Organisation (HUPO) was established in 2002/ организация человеческого протеома была основана в 2002 · Mission/ цель · Consolidate proteomics organizations in different countries into single worldwide body/ консолидировать (Значение · книжн. укреплять (укрепить), объединять (объединить), сплачивать ( сплотить) для усиления деятельности ) протеомные организации в разных странах мира в единственную организацию · Scientific and educational programs to help spread proteomics knowledge and technology/ научные и образовательные программы для распространения технологий и знания о протеоме · Coordination of public proteomics initiatives/ координация публичной инициативы · Examples of current initiatives/ примеры текущих инициативы · Human Liver Proteome Project/ бла · Human Plasma Proteome Project © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
HUPO · Human Proteome Organisation (HUPO) was established in 2002/ организация человеческого протеома была основана в 2002 · Mission/ цель · Consolidate proteomics organizations in different countries into single worldwide body/ консолидировать (Значение · книжн. укреплять (укрепить), объединять (объединить), сплачивать ( сплотить) для усиления деятельности ) протеомные организации в разных странах мира в единственную организацию · Scientific and educational programs to help spread proteomics knowledge and technology/ научные и образовательные программы для распространения технологий и знания о протеоме · Coordination of public proteomics initiatives/ координация публичной инициативы · Examples of current initiatives/ примеры текущих инициативы · Human Liver Proteome Project/ бла · Human Plasma Proteome Project © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Future prospects/ будущие перспективы · The next decade may see the complete deciphering of the proteome of yeast/ в следующие 10 лет ожидается полная расшифровка протеома · More initiatives, like the Human Liver Proteome Project, are underway/ формирование большего кол-ва инициатив · Better understanding of disease/ лучшее понимание болезней © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Future prospects/ будущие перспективы · The next decade may see the complete deciphering of the proteome of yeast/ в следующие 10 лет ожидается полная расшифровка протеома · More initiatives, like the Human Liver Proteome Project, are underway/ формирование большего кол-ва инициатив · Better understanding of disease/ лучшее понимание болезней © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Summary I/ вывод · Goals of proteomics / цели протеомики · Identify and ascribe function to proteins under all biologically plausible conditions/ идентифицировать и приписать функции белкам под всеми возможными биологическими состояниями · Proteomics methods/ методы протеомики · 2 -D gel electrophoresis for separating proteins on the basis of charge and molecular weight/ двумерный электрофорез для разделения белков на основе заряда и молекулярной массы · Mass spectrometry for identifying proteins by measuring the mass-tocharge ratio of their ionized peptide fragments/ масс-спектрофотометрия для идентификации белков, путем измерения соотношения массы к заряду и их ионизируванных фрагментов · Protein chips to identify proteins, to detect protein–protein interactions, to perform biochemical assays, and to study drug–target interactions/ белковые чипы идентификции белков для определения белокбелковых взаимодействий, чтобы представить биохимические пробы, и выучить взаимодествия лекарства-мишени © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Summary I/ вывод · Goals of proteomics / цели протеомики · Identify and ascribe function to proteins under all biologically plausible conditions/ идентифицировать и приписать функции белкам под всеми возможными биологическими состояниями · Proteomics methods/ методы протеомики · 2 -D gel electrophoresis for separating proteins on the basis of charge and molecular weight/ двумерный электрофорез для разделения белков на основе заряда и молекулярной массы · Mass spectrometry for identifying proteins by measuring the mass-tocharge ratio of their ionized peptide fragments/ масс-спектрофотометрия для идентификации белков, путем измерения соотношения массы к заряду и их ионизируванных фрагментов · Protein chips to identify proteins, to detect protein–protein interactions, to perform biochemical assays, and to study drug–target interactions/ белковые чипы идентификции белков для определения белокбелковых взаимодействий, чтобы представить биохимические пробы, и выучить взаимодествия лекарства-мишени © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Summary II · Proteomics methods (continued)/ методы протеомики · Yeast two-hybrid method for studying protein–protein interactions/ для изучения белок-белковых взаимодействий · Biochemical genomics for high-throughput assays/ биохимический высокопропускной геномный анализ · Some accomplishments of proteomics/ некоторые достижения протеомики · Example: yeast/ пример: дрожжи · Yeast two-hybrid method reveals interactome/ двугибридный метод позволил определить интерактом (Інтерактом — повний набір молекулярних взаємодій в клітині або тканині. ) · Transcriptional regulatory networks deduced/ выявлены регуляторные транскрипционные сети · Biochemical genomics uncovers new ORF functions/ Биохимическая геномика раскрывает новые функции ОРС · Subcellular localization of proteins/ субклеточная локализация белков © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Summary II · Proteomics methods (continued)/ методы протеомики · Yeast two-hybrid method for studying protein–protein interactions/ для изучения белок-белковых взаимодействий · Biochemical genomics for high-throughput assays/ биохимический высокопропускной геномный анализ · Some accomplishments of proteomics/ некоторые достижения протеомики · Example: yeast/ пример: дрожжи · Yeast two-hybrid method reveals interactome/ двугибридный метод позволил определить интерактом (Інтерактом — повний набір молекулярних взаємодій в клітині або тканині. ) · Transcriptional regulatory networks deduced/ выявлены регуляторные транскрипционные сети · Biochemical genomics uncovers new ORF functions/ Биохимическая геномика раскрывает новые функции ОРС · Subcellular localization of proteins/ субклеточная локализация белков © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Summary III · Future prospects/перспективы · Better technology for studying posttranslational modifications/ улучшение технологий для изучения поттрансляционных модификаций · ~10 years for completion of yeast proteome? / полная характеристика протеома дрожжей © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Summary III · Future prospects/перспективы · Better technology for studying posttranslational modifications/ улучшение технологий для изучения поттрансляционных модификаций · ~10 years for completion of yeast proteome? / полная характеристика протеома дрожжей © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458


