10_45.ppt
- Количество слайдов: 45
 Chapter 10 Глава 10 Comparative Genomics Сравнительная геномика Insights gained through comparison of genomes from different species Выводы, полученные путем сравнения геномов различных видов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Chapter 10 Глава 10 Comparative Genomics Сравнительная геномика Insights gained through comparison of genomes from different species Выводы, полученные путем сравнения геномов различных видов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Contents · History/ История · Synteny/ Синтения (Прим. ред: расположение нескольких генов в одном положении на разных хромосомах) · Conservation and function/ Консервативность и функции · Sequence-similarity searches/Поиск подобных последовательностей · Gene finding / Нахождение генов · Regulatory-sequence identification/ Идентификация регуляторных последовательностей · Interaction mapping/ Картирование взаимодействий? · Genes and evolution/ Гены и эволюция © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Contents · History/ История · Synteny/ Синтения (Прим. ред: расположение нескольких генов в одном положении на разных хромосомах) · Conservation and function/ Консервативность и функции · Sequence-similarity searches/Поиск подобных последовательностей · Gene finding / Нахождение генов · Regulatory-sequence identification/ Идентификация регуляторных последовательностей · Interaction mapping/ Картирование взаимодействий? · Genes and evolution/ Гены и эволюция © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 History/История · Human Genome Project decided to use smaller genomes as warmup for human genome / В проэкте «Геном человека» было предложено использовать более короткие последовательности генома для того, чтобы подготовить, по сути, основу для сиквенса человеческого генома · Resulted in sequencing the following/ Результаты сиквенса · Many bacteria/ Многих бактерий · Model-organism genomes/модельных организмов · Yeast, C. elegans, Arabidopsis, Drosophila / пивных дрожжей S. cerevisiae, нематоды C. elegans, арабидопсиса Arabidopsis thaliana, и плодовой мушки Drosophila melanogaster · Comparison of these genome sequences provided basis for field of comparative genomics / Сравнение этих геномов положило начало сравнительной геномики © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
History/История · Human Genome Project decided to use smaller genomes as warmup for human genome / В проэкте «Геном человека» было предложено использовать более короткие последовательности генома для того, чтобы подготовить, по сути, основу для сиквенса человеческого генома · Resulted in sequencing the following/ Результаты сиквенса · Many bacteria/ Многих бактерий · Model-organism genomes/модельных организмов · Yeast, C. elegans, Arabidopsis, Drosophila / пивных дрожжей S. cerevisiae, нематоды C. elegans, арабидопсиса Arabidopsis thaliana, и плодовой мушки Drosophila melanogaster · Comparison of these genome sequences provided basis for field of comparative genomics / Сравнение этих геномов положило начало сравнительной геномики © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Early comparative genomics · Comparative genomics prior to obtaining full genome sequence/ Сравнительная геномика позволяет изучить последовательность целого генома, включая · Genome size/ Размер генома · Compared DNA content among species / Сравнение ДНК между видами · Single-copy and repetitive DNA/однокопийную и повторяющуюся ДНК · Used hybridization kinetics/Используя гибридизацию · Found that the amount of repetitive DNA differed greatly among species /дает понимание о большой вариабельности повторяющихся последовательностей между видами © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Early comparative genomics · Comparative genomics prior to obtaining full genome sequence/ Сравнительная геномика позволяет изучить последовательность целого генома, включая · Genome size/ Размер генома · Compared DNA content among species / Сравнение ДНК между видами · Single-copy and repetitive DNA/однокопийную и повторяющуюся ДНК · Used hybridization kinetics/Используя гибридизацию · Found that the amount of repetitive DNA differed greatly among species /дает понимание о большой вариабельности повторяющихся последовательностей между видами © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Synteny/Синтения · Synteny: genes that are in the same relative position on two different chromosomes /Синтения: гены, которые находятся в идентичных позициях на разных хромосомах · Genetic and physical maps compared between species/Генетическое и физическое картирования производятся для сравнения между видами · Or between chromosomes of the same species/Или между хромосомами одного вида · Closely related species generally have similar order of genes on chromosomes/Родственные виды в основном имеют высокий уровень синтении · Synteny can be used to identify genes in one species based on map position in another/Синтения может быть использована для нахождения генов одного вида, зная позицию данного гена у другого гена © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Synteny/Синтения · Synteny: genes that are in the same relative position on two different chromosomes /Синтения: гены, которые находятся в идентичных позициях на разных хромосомах · Genetic and physical maps compared between species/Генетическое и физическое картирования производятся для сравнения между видами · Or between chromosomes of the same species/Или между хромосомами одного вида · Closely related species generally have similar order of genes on chromosomes/Родственные виды в основном имеют высокий уровень синтении · Synteny can be used to identify genes in one species based on map position in another/Синтения может быть использована для нахождения генов одного вида, зная позицию данного гена у другого гена © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Synteny of Grass genomes/Синтения растительных геномов · · Synteny among crop genomes: rice, maize, and wheat / Синтения между рисом, кукурузой и пшеницей Rice is smallest genome–in center/ Рис имеет наименьший геном, следовательно находится на рисунке в центре. Wheat is largest genome–outer circle / Пшеница имеет наибольший геном, следовательно представляет собой внешний круг. Genes found in similar places on chromosomes are indicated / показаны гены, занимающие одинаковую позицию на хромосомах. © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Synteny of Grass genomes/Синтения растительных геномов · · Synteny among crop genomes: rice, maize, and wheat / Синтения между рисом, кукурузой и пшеницей Rice is smallest genome–in center/ Рис имеет наименьший геном, следовательно находится на рисунке в центре. Wheat is largest genome–outer circle / Пшеница имеет наибольший геном, следовательно представляет собой внешний круг. Genes found in similar places on chromosomes are indicated / показаны гены, занимающие одинаковую позицию на хромосомах. © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Synteny of sequenced genomes/ Синтения секвенированых последовательностей · When sequences from mouse and human genomes are compared, we find regions of remarkable synteny/Когда было произведено сравнение геномов мыши и человека, были найдены регионы синтении. · Genes are in almost identical order for long stretches along the chromosome /На длинных участках хромосом гены человека и мыши располагаются в одинаковых участках. Human Chr 14 Mouse Chr 14 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Synteny of sequenced genomes/ Синтения секвенированых последовательностей · When sequences from mouse and human genomes are compared, we find regions of remarkable synteny/Когда было произведено сравнение геномов мыши и человека, были найдены регионы синтении. · Genes are in almost identical order for long stretches along the chromosome /На длинных участках хромосом гены человека и мыши располагаются в одинаковых участках. Human Chr 14 Mouse Chr 14 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
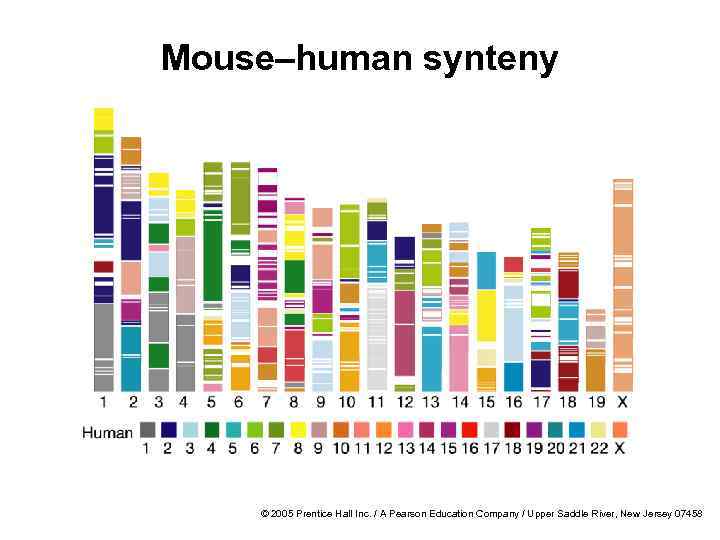 Mouse–human synteny © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Mouse–human synteny © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Comparing sequenced genomes/Сравнение последовательностей геномов · Comparison of genomic sequences from different species can help identify the following: /Сравнение геномных последовательностей разных видов может помочь идентифицировать следующее: · Gene structure/Структуру генов · Gene function/Их функции · Regulatory sequences/Регуляторные последовательности · Interactions between gene products/ Взаимодействия между генными продуктами ? (тупо как-то: ) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Comparing sequenced genomes/Сравнение последовательностей геномов · Comparison of genomic sequences from different species can help identify the following: /Сравнение геномных последовательностей разных видов может помочь идентифицировать следующее: · Gene structure/Структуру генов · Gene function/Их функции · Regulatory sequences/Регуляторные последовательности · Interactions between gene products/ Взаимодействия между генными продуктами ? (тупо как-то: ) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Evolution and sequence conservation/Эволюция и консервативность последовательностей · Genome comparisons based on observation: conservation = function/ Сравнение геномов базируется на основе наблюдений: консервативность=функция · If there are no constraints on DNA sequence, then random mutations will occur/ Если последовательность ДНК не имеет границ– будут иметь место случайные мутации (? Щито это вообще такойе? ) · Over tens of millions of years, these random mutations will make two related sequences different/ Через 10 миллионов лет эти случайные мутации приведут к образованию нового генома © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Evolution and sequence conservation/Эволюция и консервативность последовательностей · Genome comparisons based on observation: conservation = function/ Сравнение геномов базируется на основе наблюдений: консервативность=функция · If there are no constraints on DNA sequence, then random mutations will occur/ Если последовательность ДНК не имеет границ– будут иметь место случайные мутации (? Щито это вообще такойе? ) · Over tens of millions of years, these random mutations will make two related sequences different/ Через 10 миллионов лет эти случайные мутации приведут к образованию нового генома © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Function and sequence conservation/ Фунция и консервативность последовательностей · However, if there are constraints such as the following, then there will be sequence similarity when related sequences are compared: /Однако, если последовательность ограничена, то родственные последовательности при сравнивании покажут сходство. · DNA codes for protein/белок-кодирующая ДНК · Or site for transcription factor binding/или сайт связывания с транскрипционным фактором · Basic rule when comparing two related sequences: /Базовое правило при сравнение двух родственных последовательностей · Sequence conservation = functional importance/ Консервативность последовательности = важная функция © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Function and sequence conservation/ Фунция и консервативность последовательностей · However, if there are constraints such as the following, then there will be sequence similarity when related sequences are compared: /Однако, если последовательность ограничена, то родственные последовательности при сравнивании покажут сходство. · DNA codes for protein/белок-кодирующая ДНК · Or site for transcription factor binding/или сайт связывания с транскрипционным фактором · Basic rule when comparing two related sequences: /Базовое правило при сравнение двух родственных последовательностей · Sequence conservation = functional importance/ Консервативность последовательности = важная функция © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Orthologs and Paralogs I/ортологи и паралоги I (если в пределах одного организма в результате хромосомной мутации произошло удвоение гена, то его копии называют паралогами. ) · When comparing sequence from different genomes, must distinguish between two types of closely related sequences/ При сравнении последовательности двух различных геномов можно четко различить некоторые очень схожие последовательности · Orthologs are genes found in two species that had a common ancestor/ Ортологи – гены, находящиеся у двух видов и имеющие общего «предка» · Paralogs are genes found in the same species that were created through gene duplication events / Паралоги – гены, находящиеся в пределах вида, получены путем дупликаций © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Orthologs and Paralogs I/ортологи и паралоги I (если в пределах одного организма в результате хромосомной мутации произошло удвоение гена, то его копии называют паралогами. ) · When comparing sequence from different genomes, must distinguish between two types of closely related sequences/ При сравнении последовательности двух различных геномов можно четко различить некоторые очень схожие последовательности · Orthologs are genes found in two species that had a common ancestor/ Ортологи – гены, находящиеся у двух видов и имеющие общего «предка» · Paralogs are genes found in the same species that were created through gene duplication events / Паралоги – гены, находящиеся в пределах вида, получены путем дупликаций © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
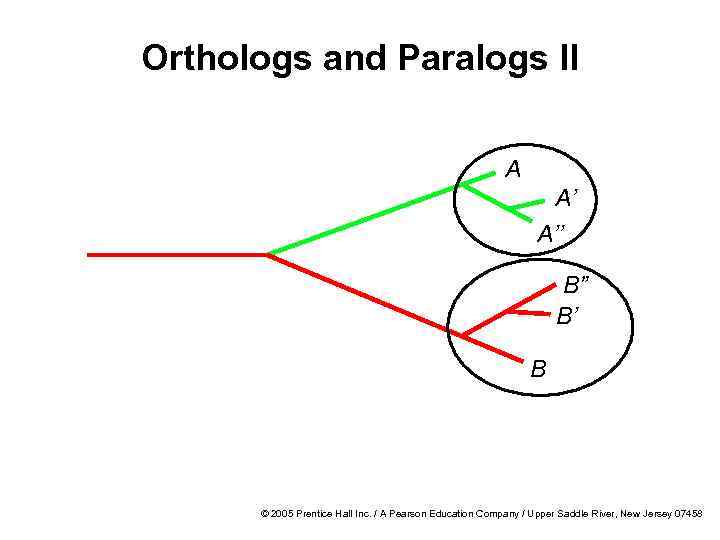 Orthologs and Paralogs II A A’ A’’ B” B’ B © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Orthologs and Paralogs II A A’ A’’ B” B’ B © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Sequence similarity and gene function/ Подобность последовательностей и функция генов · Sequence comparisons that implicate function are widely used to determine whether newly sequenced c. DNA or genomic region encodes gene of known function/Сравнение кодирующих последовательностей широко используется для определения новой последовательности к. ДНК или геномной области закодированных генов, функция которых известна · Search for similar sequence in other species (or in same species)/Поиск похожих последовательностей у других видов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Sequence similarity and gene function/ Подобность последовательностей и функция генов · Sequence comparisons that implicate function are widely used to determine whether newly sequenced c. DNA or genomic region encodes gene of known function/Сравнение кодирующих последовательностей широко используется для определения новой последовательности к. ДНК или геномной области закодированных генов, функция которых известна · Search for similar sequence in other species (or in same species)/Поиск похожих последовательностей у других видов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Homology searches/ Поиск гомологий · Search databases of DNA sequences/Поиск последовательностей ДНК в базах данных · Use computer algorithms to align sequences/использование компьютерных алгоритмов для выравнивания последовательностей · Don’t require perfect matches between sequences/ не требуется идеально сопоставлять последовательности · Allow for insertions, deletions, and base changes/ учитывая инсерции, делеции и изменение оснований · Most commonly used algorithms: /Наиболее используемые алгоритмы: · BLAST · FASTA © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Homology searches/ Поиск гомологий · Search databases of DNA sequences/Поиск последовательностей ДНК в базах данных · Use computer algorithms to align sequences/использование компьютерных алгоритмов для выравнивания последовательностей · Don’t require perfect matches between sequences/ не требуется идеально сопоставлять последовательности · Allow for insertions, deletions, and base changes/ учитывая инсерции, делеции и изменение оснований · Most commonly used algorithms: /Наиболее используемые алгоритмы: · BLAST · FASTA © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Example of homology search/ Пример поиска гомологии · The sea squirt, Ciona intestinalis, makes a coat primarily of cellulose/ Покров губки Ciona intestinalis состоит преимущественно из целлюлозы · A BLAST search was performed on the Ciona genome, using an Arabidopsis endoglucanase gene involved in cellulose synthesis/ Был произведен поиск в базе БЛАСТ по геному этой губки, используя ген ендоглюканазы арабидопсиса, который вовлечен в синтез целлюлозы · Extensive homology was found with a Ciona gene flanked by genes found in Drosophila and human/Высокая гомология была найдет в гене губки, который фланкированный генами, найденными у дрозофилы и человека · It is postulated that the Ciona endoglucanase gene may have arisen by lateral gene transfer /Был сделан вывод, что ген эндоглюканазы губки появился путем транслокаций. © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Example of homology search/ Пример поиска гомологии · The sea squirt, Ciona intestinalis, makes a coat primarily of cellulose/ Покров губки Ciona intestinalis состоит преимущественно из целлюлозы · A BLAST search was performed on the Ciona genome, using an Arabidopsis endoglucanase gene involved in cellulose synthesis/ Был произведен поиск в базе БЛАСТ по геному этой губки, используя ген ендоглюканазы арабидопсиса, который вовлечен в синтез целлюлозы · Extensive homology was found with a Ciona gene flanked by genes found in Drosophila and human/Высокая гомология была найдет в гене губки, который фланкированный генами, найденными у дрозофилы и человека · It is postulated that the Ciona endoglucanase gene may have arisen by lateral gene transfer /Был сделан вывод, что ген эндоглюканазы губки появился путем транслокаций. © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Discovery of endoglucanase gene in sea squirt genome/ Изучение гена ендоглюканазы губки Arabidopsis Korrigan Transporter Endoglucanase Splicing factor C. intestinalis c. DNA C. elegans and Drosophila Human © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Discovery of endoglucanase gene in sea squirt genome/ Изучение гена ендоглюканазы губки Arabidopsis Korrigan Transporter Endoglucanase Splicing factor C. intestinalis c. DNA C. elegans and Drosophila Human © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Homology search for the mouse genome/ Поиск гомологий в геноме мыши · Homology search of all genes in the mouse genome/ Гомологии искали в целом геноме мыши · 27% in other metazoans/ 27% всего генома присутствует у других многоклеточных · 29% in other eukaryotes/ 29% у эукариот · 6% in other chordates/ 6% - у хордовых · 14% in other mammals/14%-у других млекопитающих · Less than 1% rodent specific / и лишь менее 1% является родоспецифичными генами Eukaryote Other Metazoan Chordate Mammal Rodent specific © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Homology search for the mouse genome/ Поиск гомологий в геноме мыши · Homology search of all genes in the mouse genome/ Гомологии искали в целом геноме мыши · 27% in other metazoans/ 27% всего генома присутствует у других многоклеточных · 29% in other eukaryotes/ 29% у эукариот · 6% in other chordates/ 6% - у хордовых · 14% in other mammals/14%-у других млекопитающих · Less than 1% rodent specific / и лишь менее 1% является родоспецифичными генами Eukaryote Other Metazoan Chordate Mammal Rodent specific © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Problems of genome annotation/ Проблемы расшифровки генома · Identifying genes and regulatory regions in sequenced genomes is challenging/затруднительно идентифицировать гены и регуляторные последовательности · Open reading frames (ORFs) are usually good indication of genes / открытые рамки считывания отлично подходят для индикации генов · However, it is difficult to determine which ORFs belong to a gene/ все равно сложно опрелелить к какому гену относится та или иная рамка считывания · Many mammalian genes have small exons and large introns / многие гены млекопитающих имеют маленькие экзоны и большие интроны © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Problems of genome annotation/ Проблемы расшифровки генома · Identifying genes and regulatory regions in sequenced genomes is challenging/затруднительно идентифицировать гены и регуляторные последовательности · Open reading frames (ORFs) are usually good indication of genes / открытые рамки считывания отлично подходят для индикации генов · However, it is difficult to determine which ORFs belong to a gene/ все равно сложно опрелелить к какому гену относится та или иная рамка считывания · Many mammalian genes have small exons and large introns / многие гены млекопитающих имеют маленькие экзоны и большие интроны © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Computational approaches to gene identification/ компьютерные подходы для идентификации генов · Computer programs analyze genomic sequence/Следующие компьютерные программы анализируют геномные последовательности: · GRAIL · Gene. Finder · Look for ORFs, splice sites, poly A addition sites, etc. /Они производят поиск открытых рамок считывания, сайтов сплайсинга, поли. А сайтов и т. д · Predict gene structure/ предусматривают структуру гена · Frequently wrong/ часто ошибаются · Usually miss exons at beginning or end of gene/Часто пропускают экзоны в начале или в конце гена · Sometimes predict exon when one doesn’t really exist/ Иногда находят экзон, где его в самом деле нет © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Computational approaches to gene identification/ компьютерные подходы для идентификации генов · Computer programs analyze genomic sequence/Следующие компьютерные программы анализируют геномные последовательности: · GRAIL · Gene. Finder · Look for ORFs, splice sites, poly A addition sites, etc. /Они производят поиск открытых рамок считывания, сайтов сплайсинга, поли. А сайтов и т. д · Predict gene structure/ предусматривают структуру гена · Frequently wrong/ часто ошибаются · Usually miss exons at beginning or end of gene/Часто пропускают экзоны в начале или в конце гена · Sometimes predict exon when one doesn’t really exist/ Иногда находят экзон, где его в самом деле нет © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 How genome comparisons help / Как может помочь сравнение генома · When comparing genomes of different species, the genes normally have the same exon–intron structure/ При сравнении геномов разных видов, преимущественно гены имеют одинаковые структуры экзонов-интронов · Look for conserved ORFs in both genomes/ Производится поиск консервативных открытых рамок считывания в сравниваемых геномах · Frequently permit accurate identification of genes · Fugu–human comparison found >1, 000 genes/с помощью сопоставления генома рыбы фугу с геномом человек было найдено более 1000 генов · Mouse–human comparison indicates only 25, 000 genes in genome/при сравнении мышь-человек – 25000 генов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
How genome comparisons help / Как может помочь сравнение генома · When comparing genomes of different species, the genes normally have the same exon–intron structure/ При сравнении геномов разных видов, преимущественно гены имеют одинаковые структуры экзонов-интронов · Look for conserved ORFs in both genomes/ Производится поиск консервативных открытых рамок считывания в сравниваемых геномах · Frequently permit accurate identification of genes · Fugu–human comparison found >1, 000 genes/с помощью сопоставления генома рыбы фугу с геномом человек было найдено более 1000 генов · Mouse–human comparison indicates only 25, 000 genes in genome/при сравнении мышь-человек – 25000 генов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
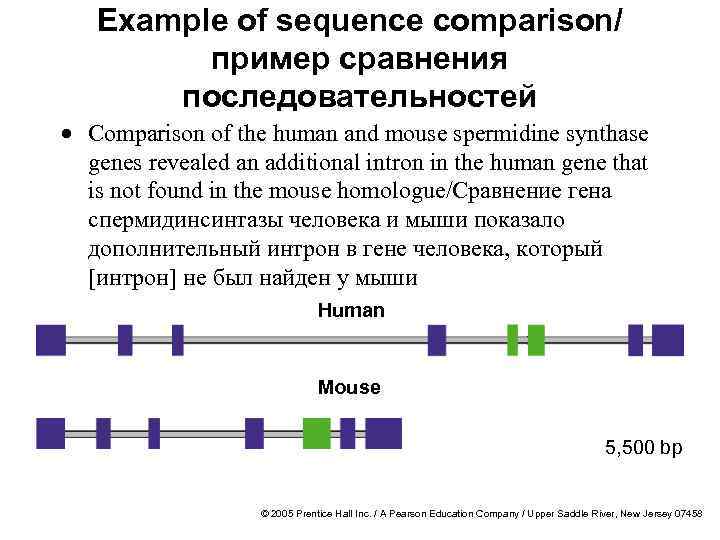 Example of sequence comparison/ пример сравнения последовательностей · Comparison of the human and mouse spermidine synthase genes revealed an additional intron in the human gene that is not found in the mouse homologue/Сравнение гена спермидинсинтазы человека и мыши показало дополнительный интрон в гене человека, который [интрон] не был найден у мыши Human Mouse 5, 500 bp © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Example of sequence comparison/ пример сравнения последовательностей · Comparison of the human and mouse spermidine synthase genes revealed an additional intron in the human gene that is not found in the mouse homologue/Сравнение гена спермидинсинтазы человека и мыши показало дополнительный интрон в гене человека, который [интрон] не был найден у мыши Human Mouse 5, 500 bp © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
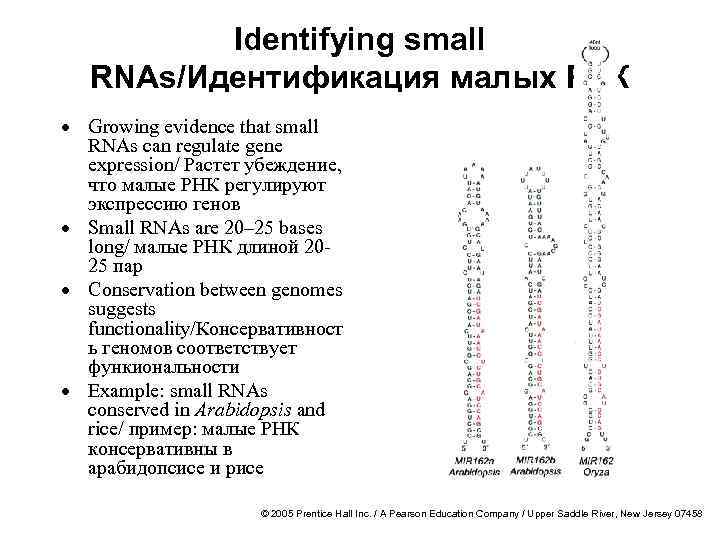 Identifying small RNAs/Идентификация малых РНК · Growing evidence that small RNAs can regulate gene expression/ Растет убеждение, что малые РНК регулируют экспрессию генов · Small RNAs are 20– 25 bases long/ малые РНК длиной 2025 пар · Conservation between genomes suggests functionality/Консервативност ь геномов соответствует функиональности · Example: small RNAs conserved in Arabidopsis and rice/ пример: малые РНК консервативны в арабидопсисе и рисе © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Identifying small RNAs/Идентификация малых РНК · Growing evidence that small RNAs can regulate gene expression/ Растет убеждение, что малые РНК регулируют экспрессию генов · Small RNAs are 20– 25 bases long/ малые РНК длиной 2025 пар · Conservation between genomes suggests functionality/Консервативност ь геномов соответствует функиональности · Example: small RNAs conserved in Arabidopsis and rice/ пример: малые РНК консервативны в арабидопсисе и рисе © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Regulatory-sequence identification/поиск регуляторных последовательностей · A large portion of the genome contains regulatory information/ Большая часть генома содержит регуляторную информацию · Regulatory sequence includes the following: /Регуляторные последовательности включают в себя следующие элементы: · Cis-regulatory elements: tell genes when and where to turn on/Цис-последовательности: определяют место и время работы гена · Basal transcription machinery binding sites / сайты связывания транскрипционного аппарата · Enhancers/ энхансерные области · Can be 5’ of gene, 3’ of gene, or in intron/ 5’ или 3 ’ область, или интрон © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Regulatory-sequence identification/поиск регуляторных последовательностей · A large portion of the genome contains regulatory information/ Большая часть генома содержит регуляторную информацию · Regulatory sequence includes the following: /Регуляторные последовательности включают в себя следующие элементы: · Cis-regulatory elements: tell genes when and where to turn on/Цис-последовательности: определяют место и время работы гена · Basal transcription machinery binding sites / сайты связывания транскрипционного аппарата · Enhancers/ энхансерные области · Can be 5’ of gene, 3’ of gene, or in intron/ 5’ или 3 ’ область, или интрон © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
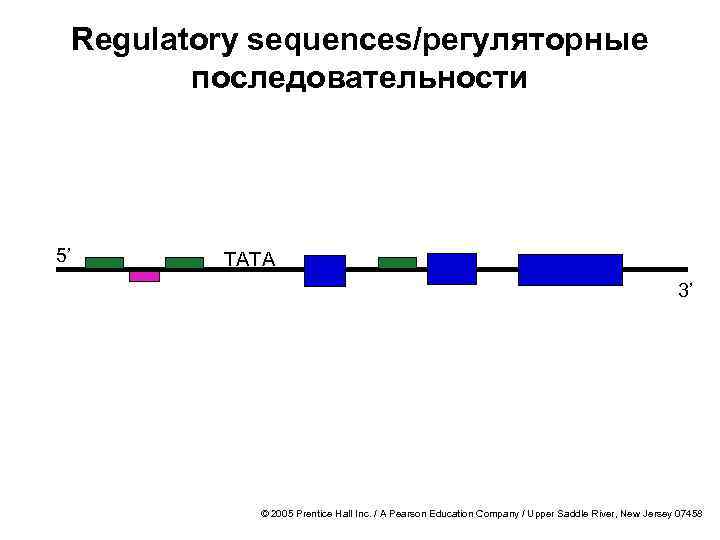 Regulatory sequences/регуляторные последовательности 5’ TATA 3’ © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Regulatory sequences/регуляторные последовательности 5’ TATA 3’ © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Finding regulatory sequences/ нахождение регуляторных последовательностей · Regulatory sequences are difficult to identify using computer programs/регуляторные последовательности сложно идентифицировать, используя компьютерные программы · Problem is that most enhancer sequences have yet to be identified/Проблемным является то, что большинство енхансерных последовательностей уже должны быть определены · They are usually short: 6– 10 base pairs/ в основном они короткие – 6 -10 пар · Those that are known are usually degenerate/ Также известно, что в большинстве случаев они являются вырожденными · They can differ in one or more base pairs/ они могут отличаться на одну и более пару оаснований · Still bind the cognate transcription factor/ или быть связанными с транскрипционными факторами © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Finding regulatory sequences/ нахождение регуляторных последовательностей · Regulatory sequences are difficult to identify using computer programs/регуляторные последовательности сложно идентифицировать, используя компьютерные программы · Problem is that most enhancer sequences have yet to be identified/Проблемным является то, что большинство енхансерных последовательностей уже должны быть определены · They are usually short: 6– 10 base pairs/ в основном они короткие – 6 -10 пар · Those that are known are usually degenerate/ Также известно, что в большинстве случаев они являются вырожденными · They can differ in one or more base pairs/ они могут отличаться на одну и более пару оаснований · Still bind the cognate transcription factor/ или быть связанными с транскрипционными факторами © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 · Comparisons to identify regulatory elements/ сравнение для идентифицирования регуляторных элементов Comparisons of genomes of different species can identify regulatory elements/сравнение геномов различных видов может позволить найти регуляторные элементы · Change in intergenic regions and introns usually more rapid than in coding regions/ изменения в межгенных регионах и интронах происходят чаще, чем в кодирующих последовательностях · Nevertheless, regulatory elements tend to be conserved/Однако, регуляторные последовательности часто являются консервативными · Conserved regions called “phylogenetic footprint”/консервативные регионы называют «филогенетическим отпечатком» » © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
· Comparisons to identify regulatory elements/ сравнение для идентифицирования регуляторных элементов Comparisons of genomes of different species can identify regulatory elements/сравнение геномов различных видов может позволить найти регуляторные элементы · Change in intergenic regions and introns usually more rapid than in coding regions/ изменения в межгенных регионах и интронах происходят чаще, чем в кодирующих последовательностях · Nevertheless, regulatory elements tend to be conserved/Однако, регуляторные последовательности часто являются консервативными · Conserved regions called “phylogenetic footprint”/консервативные регионы называют «филогенетическим отпечатком» » © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Phylogenetic footprint/ филогенетический отпечаток · Identification of conserved regulatory regions usually requires comparing genomes of closely related species/ идентификация консервативных регуляторных регионов обычно нуждается в сравнении геномов родственных видов · If too distantly related, very difficult to find conservation/ если виды не родственные консервативные последовательности обнаружить весьма сложно · Nevertheless, mouse–human sequence comparison has revealed many conserved cis-regulatory elements/однако, в геномных последовательностях мыши и человека было выявлено множество консервативных цис-регуляторных элементов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Phylogenetic footprint/ филогенетический отпечаток · Identification of conserved regulatory regions usually requires comparing genomes of closely related species/ идентификация консервативных регуляторных регионов обычно нуждается в сравнении геномов родственных видов · If too distantly related, very difficult to find conservation/ если виды не родственные консервативные последовательности обнаружить весьма сложно · Nevertheless, mouse–human sequence comparison has revealed many conserved cis-regulatory elements/однако, в геномных последовательностях мыши и человека было выявлено множество консервативных цис-регуляторных элементов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
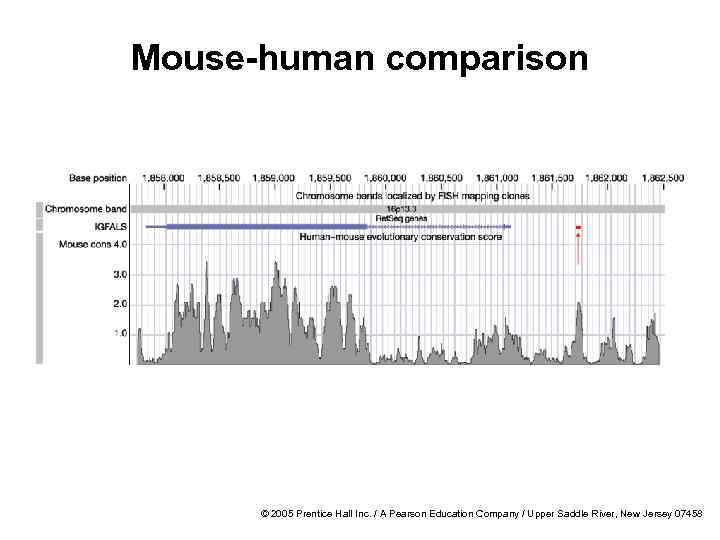 Mouse-human comparison © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Mouse-human comparison © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Using multiple species for phylogenetic footprinting/ использование разных видов для филогенетического отпечатка · The location of regulatory sequences can also be found by comparing several related sequences/местонахождения регуляторных последовательностей может быть найдено, путем сравнения некоторых родственных последоватнльностей · Multiple alignments performed/ выполняют многочисленное выравнивание · Better able to home in on important regions · Conservation alone not enough; need to validate importance of elements/консервативность не является отличительным признаком регуляторных последовательностей, необходимо подтвердить важность элементов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Using multiple species for phylogenetic footprinting/ использование разных видов для филогенетического отпечатка · The location of regulatory sequences can also be found by comparing several related sequences/местонахождения регуляторных последовательностей может быть найдено, путем сравнения некоторых родственных последоватнльностей · Multiple alignments performed/ выполняют многочисленное выравнивание · Better able to home in on important regions · Conservation alone not enough; need to validate importance of elements/консервативность не является отличительным признаком регуляторных последовательностей, необходимо подтвердить важность элементов © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Interaction mapping/ интерактивное картирование (и снова щто-щто? ? ) · Protein–protein interactions include the following: /белок-белковые взаимодействия включают в себя следующее: · The transfer of information in a genetic pathway/ перенос информации по генетическому пути · Scaffolding to tether other proteins/ связывание с другим белком · Enzymatic reactions/ энзиматические реакции · Large molecular machines such as motors/большие молекулятные машины (такие как белки-моторы) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Interaction mapping/ интерактивное картирование (и снова щто-щто? ? ) · Protein–protein interactions include the following: /белок-белковые взаимодействия включают в себя следующее: · The transfer of information in a genetic pathway/ перенос информации по генетическому пути · Scaffolding to tether other proteins/ связывание с другим белком · Enzymatic reactions/ энзиматические реакции · Large molecular machines such as motors/большие молекулятные машины (такие как белки-моторы) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Rosetta stone approach БЕЗ ПЕРЕВОДА · Observation: In some species, interaction proteins encoded by single gene, while in other species the same proteins are encoded in two genes/Наблюдени: у некоторых видов белки взаимодействия кодируются одним геном, в то время как у других видов те же белки кодируются двумя генами. · Systematic search through sequenced genomes for these relationships should identify proteins that interact/ систематический поиск в секвенированных геномах позволяет идентифицировать такие белки © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Rosetta stone approach БЕЗ ПЕРЕВОДА · Observation: In some species, interaction proteins encoded by single gene, while in other species the same proteins are encoded in two genes/Наблюдени: у некоторых видов белки взаимодействия кодируются одним геном, в то время как у других видов те же белки кодируются двумя генами. · Systematic search through sequenced genomes for these relationships should identify proteins that interact/ систематический поиск в секвенированных геномах позволяет идентифицировать такие белки © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
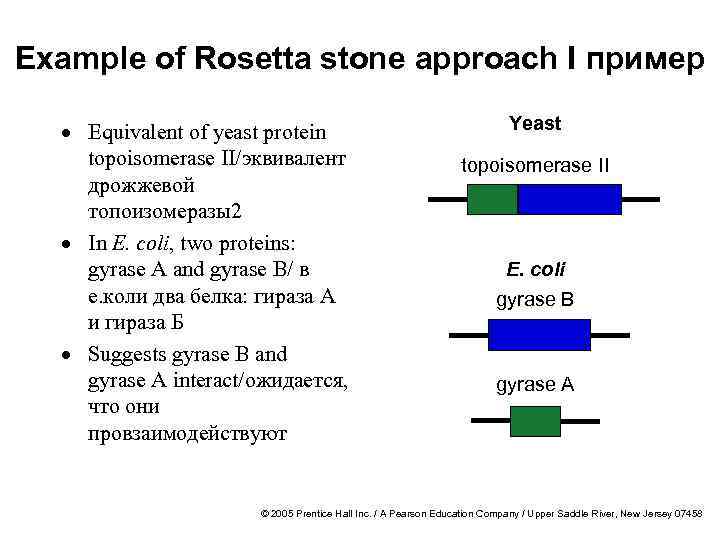 Example of Rosetta stone approach I пример · Equivalent of yeast protein topoisomerase II/эквивалент дрожжевой топоизомеразы2 · In E. coli, two proteins: gyrase A and gyrase B/ в е. коли два белка: гираза А и гираза Б · Suggests gyrase B and gyrase A interact/ожидается, что они провзаимодействуют Yeast topoisomerase II E. coli gyrase B gyrase A © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Example of Rosetta stone approach I пример · Equivalent of yeast protein topoisomerase II/эквивалент дрожжевой топоизомеразы2 · In E. coli, two proteins: gyrase A and gyrase B/ в е. коли два белка: гираза А и гираза Б · Suggests gyrase B and gyrase A interact/ожидается, что они провзаимодействуют Yeast topoisomerase II E. coli gyrase B gyrase A © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
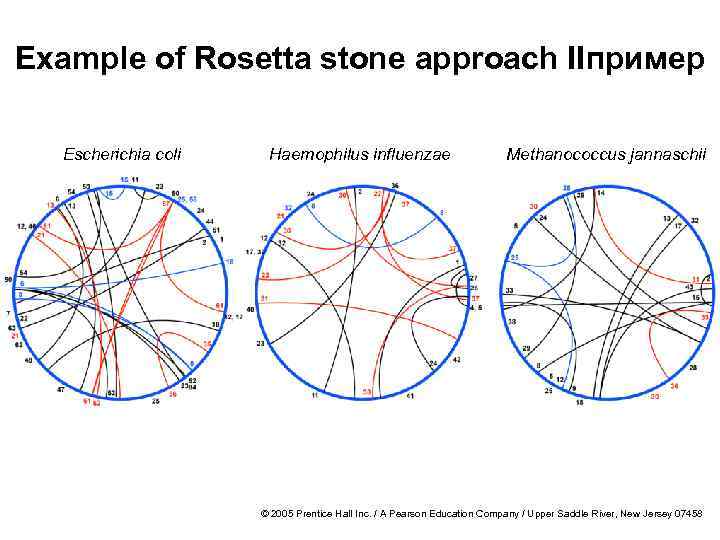 Example of Rosetta stone approach IIпример Escherichia coli Haemophilus influenzae Methanococcus jannaschii © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Example of Rosetta stone approach IIпример Escherichia coli Haemophilus influenzae Methanococcus jannaschii © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Higher level comparisons / высший уровень сравнения · Comparisons between genomes not just to better identify genes and regulatory sequences/сопоставление между геномами используют не только, чтобы найти гены или регуляторные последовательности · Evolution of adaptive traits occurs through the following: /эволюция адаптативных особеностей происходит таким образом: · Evolution of new genes/ эволюция новых генов · Changing when and where genes express/ изменение экспрессии · Thus, comparisons of genes found in genome can provide information about mechanisms of evolution/ Так, сравнение генов может дать информацию об эволюционных процессах © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Higher level comparisons / высший уровень сравнения · Comparisons between genomes not just to better identify genes and regulatory sequences/сопоставление между геномами используют не только, чтобы найти гены или регуляторные последовательности · Evolution of adaptive traits occurs through the following: /эволюция адаптативных особеностей происходит таким образом: · Evolution of new genes/ эволюция новых генов · Changing when and where genes express/ изменение экспрессии · Thus, comparisons of genes found in genome can provide information about mechanisms of evolution/ Так, сравнение генов может дать информацию об эволюционных процессах © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Genes and genomes/ гены и геномы · Comparison of total gene numbers in sequenced genomes: / Сравнение общего количества генов секвенированных геномов · Smaller than originally expected/ меньше, чем ожидалось · Example: Human genome thought to have 100, 000 genes/ пример: предполагалось, что человеческий геном содержит 100 000 генов · Now thought to be closer to 20, 000– 25, 000 genes/теперь считается, что их около 20 000 -25 000 · Suggests that many new functions arise in gene expression/ Ожидалось, что много новых функций являются результатом генной экспрессии · Use old genes in new ways/ используют старые гены новым образом © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Genes and genomes/ гены и геномы · Comparison of total gene numbers in sequenced genomes: / Сравнение общего количества генов секвенированных геномов · Smaller than originally expected/ меньше, чем ожидалось · Example: Human genome thought to have 100, 000 genes/ пример: предполагалось, что человеческий геном содержит 100 000 генов · Now thought to be closer to 20, 000– 25, 000 genes/теперь считается, что их около 20 000 -25 000 · Suggests that many new functions arise in gene expression/ Ожидалось, что много новых функций являются результатом генной экспрессии · Use old genes in new ways/ используют старые гены новым образом © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Selective expansion of genes/ избирательная экспансия генов · Although comparisons show not as much difference in numbers of genes as expected, we still see striking differences in the numbers of some gene families/ также сравнение показывает не такую и большую разницу в количестве генов, насколько ожидалось, сколько поразительные отличия в количестве семейств генов · Examples: / примеры: · Roundworm, C. elegans, has a large number of nuclear receptor genes/ дождевой червь, имеет большое количество генов ядерных рецепторов · Drosophila has a large number of zinc-finger transcription factors/ у дрозофилы имеется множество транскрипционных факторов группы цинковых пальцев. · Plants have no G-protein-coupled receptors/ растения не имеют рецепторов, сопряжённых с G-белком (серпентины) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Selective expansion of genes/ избирательная экспансия генов · Although comparisons show not as much difference in numbers of genes as expected, we still see striking differences in the numbers of some gene families/ также сравнение показывает не такую и большую разницу в количестве генов, насколько ожидалось, сколько поразительные отличия в количестве семейств генов · Examples: / примеры: · Roundworm, C. elegans, has a large number of nuclear receptor genes/ дождевой червь, имеет большое количество генов ядерных рецепторов · Drosophila has a large number of zinc-finger transcription factors/ у дрозофилы имеется множество транскрипционных факторов группы цинковых пальцев. · Plants have no G-protein-coupled receptors/ растения не имеют рецепторов, сопряжённых с G-белком (серпентины) © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 What is the difference between man and ape? /какая разница между человеком и обезьяной · Man and chimpanzee have a genomewide similarity of greater than 95%. / человек и шимпанзе имеют гомологию свыше 95%. · What accounts for differences between species? / что обуславливает разницу? · Recent study suggests that differences between species are due to specific gene expression differences/ изучение показало, что разница между ними происходит из-за специфических различий экспрессии генов · Striking differences found only in brain/ Поразительные различия найдены только в мозге © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
What is the difference between man and ape? /какая разница между человеком и обезьяной · Man and chimpanzee have a genomewide similarity of greater than 95%. / человек и шимпанзе имеют гомологию свыше 95%. · What accounts for differences between species? / что обуславливает разницу? · Recent study suggests that differences between species are due to specific gene expression differences/ изучение показало, что разница между ними происходит из-за специфических различий экспрессии генов · Striking differences found only in brain/ Поразительные различия найдены только в мозге © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Human–ape gene expression comparisons 1. 3 Human 1. 0 Chimp Human 5. 5 Chimp Rhesus © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Human–ape gene expression comparisons 1. 3 Human 1. 0 Chimp Human 5. 5 Chimp Rhesus © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Trait-to-gene approach/ · Methods being developed to identify genes involved in adaptive traits/ метод, разработанный для того, чтобы идентифицировать гены, отвечающие за адаптационные особенности · Underlying reasoning: Organisms that have a particular trait either share related genes or have developed new genes to perform the same function/ причина, лежащая в основе : организмы, которые имеют редкие черты, или распределяют гены или разрабатывают новые гены, чтобы изменить функцию © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Trait-to-gene approach/ · Methods being developed to identify genes involved in adaptive traits/ метод, разработанный для того, чтобы идентифицировать гены, отвечающие за адаптационные особенности · Underlying reasoning: Organisms that have a particular trait either share related genes or have developed new genes to perform the same function/ причина, лежащая в основе : организмы, которые имеют редкие черты, или распределяют гены или разрабатывают новые гены, чтобы изменить функцию © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
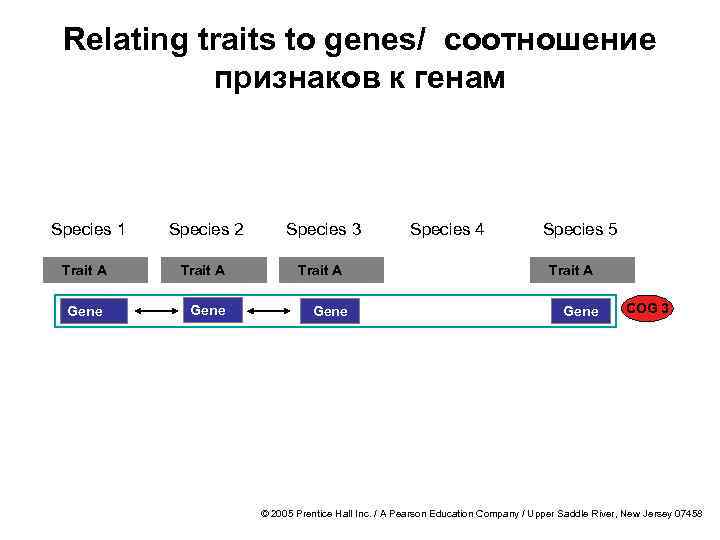 Relating traits to genes/ соотношение признаков к генам Species 1 Species 2 Species 3 Trait A Gene Species 4 Species 5 Trait A Gene COG 3 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Relating traits to genes/ соотношение признаков к генам Species 1 Species 2 Species 3 Trait A Gene Species 4 Species 5 Trait A Gene COG 3 © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Example of trait-to-gene approach I/ пример trait-to-gene метода · Comparisons made of bacterial genomes / сравнение проводилось на бактериальных геномах · Need many genomes / необходимо много геномов · Looked for genes involved in flagellar function / поиск генов, вовлеченных в выполнение функций жгутика · Identified 43 of 45 known genes/ идентифицировано 43 из 45 известных генов · Found 5 additional genes that program said should be involved in flagellar function/ найдено 5 дополнительных генов, которые ВОЗМОЖНО могут быть вовлеченными в выполнение тех функций · Knocked out 3 and found that 2 resulted in bacteria with defective flagella/ выключено 3 гена, и найдено, что 2 из них приводят к созданию бактерии с дефективным джгутиком © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Example of trait-to-gene approach I/ пример trait-to-gene метода · Comparisons made of bacterial genomes / сравнение проводилось на бактериальных геномах · Need many genomes / необходимо много геномов · Looked for genes involved in flagellar function / поиск генов, вовлеченных в выполнение функций жгутика · Identified 43 of 45 known genes/ идентифицировано 43 из 45 известных генов · Found 5 additional genes that program said should be involved in flagellar function/ найдено 5 дополнительных генов, которые ВОЗМОЖНО могут быть вовлеченными в выполнение тех функций · Knocked out 3 and found that 2 resulted in bacteria with defective flagella/ выключено 3 гена, и найдено, что 2 из них приводят к созданию бактерии с дефективным джгутиком © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
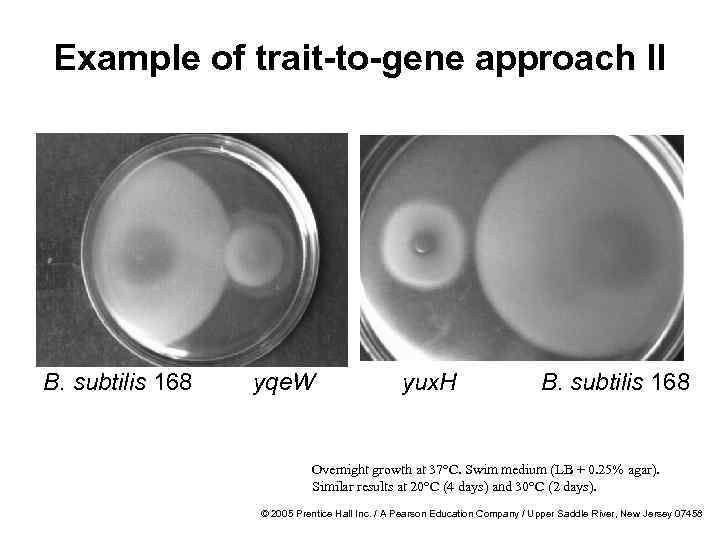 Example of trait-to-gene approach II B. subtilis 168 yqe. W yux. H B. subtilis 168 Overnight growth at 37°C. Swim medium (LB + 0. 25% agar). Similar results at 20°C (4 days) and 30°C (2 days). © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Example of trait-to-gene approach II B. subtilis 168 yqe. W yux. H B. subtilis 168 Overnight growth at 37°C. Swim medium (LB + 0. 25% agar). Similar results at 20°C (4 days) and 30°C (2 days). © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 The goal of comparative genomics/ цель сравнительной геномики © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
The goal of comparative genomics/ цель сравнительной геномики © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
 Summary · Synteny = similar relative positions of genes on chromosomes/ Синтения = аналогичное расположение генов на другой хромосоме · Conservation = function/ Консервативность=функция · Homology searches/ Поиск гомологии · Gene structure prediction/ прогнозирование структуры гена · Regulatory-sequence identification/ идентификация регуляторных последоватнльностей · Interaction mapping/ картирование взаимодействий · Genes and evolution/ гены и эволюция © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458
Summary · Synteny = similar relative positions of genes on chromosomes/ Синтения = аналогичное расположение генов на другой хромосоме · Conservation = function/ Консервативность=функция · Homology searches/ Поиск гомологии · Gene structure prediction/ прогнозирование структуры гена · Regulatory-sequence identification/ идентификация регуляторных последоватнльностей · Interaction mapping/ картирование взаимодействий · Genes and evolution/ гены и эволюция © 2005 Prentice Hall Inc. / A Pearson Education Company / Upper Saddle River, New Jersey 07458


