7-СИНТЕЗ БЕЛКА.ppt
- Количество слайдов: 86

БИОСИНТЕЗ БЕЛКА лекция 1

Рекогниция • Рекогниция - это подготовительный этап трансляции, суть которого в образовании ковалентной связи между t. РНК и соответствующей аминокислотой. • Состоит из двух стадий: • 1. Активирование аминокислоты. • 2. Присоединение аминокислоты к t. РНК аминоацилирование. • Обе стадии рекогниции осуществляются ферментом аминоацил-t. РНК-синтетазой (APC-азой, кодазой). Существует 20 вариантов кодаз (по числу аминокислот). У каждой кодазы 3 центра опознавания. Каждая АРС-аза узнает третичную структуру t. РНК. 2
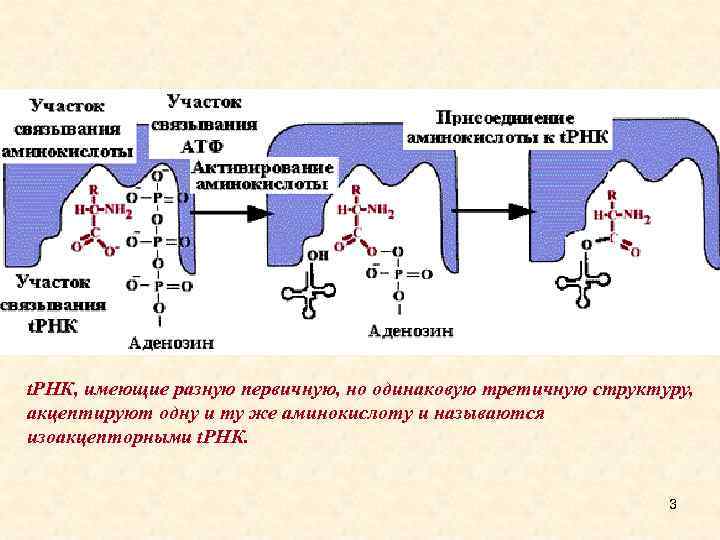
t. РНК, имеющие разную первичную, но одинаковую третичную структуру, акцептируют одну и ту же аминокислоту и называются изоакцепторными t. РНК. 3
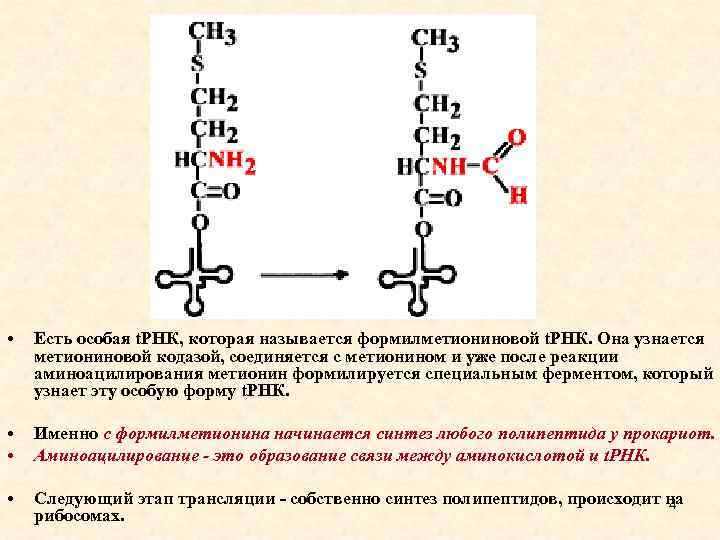
• Есть особая t. РНК, которая называется формилметиониновой t. РНК. Она узнается метиониновой кодазой, соединяется с метионином и уже после реакции аминоацилирования метионин формилируется специальным ферментом, который узнает эту особую форму t. РНК. • • Именно с формилметионина начинается синтез любого полипептида у прокариот. Аминоацилирование - это образование связи между аминокислотой и t. PHК. • Следующий этап трансляции - собственно синтез полипептидов, происходит на 4 рибосомах.
5
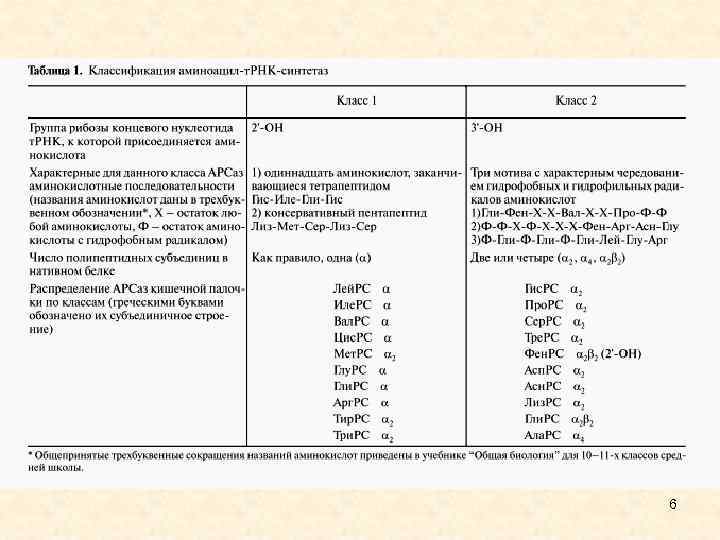
6
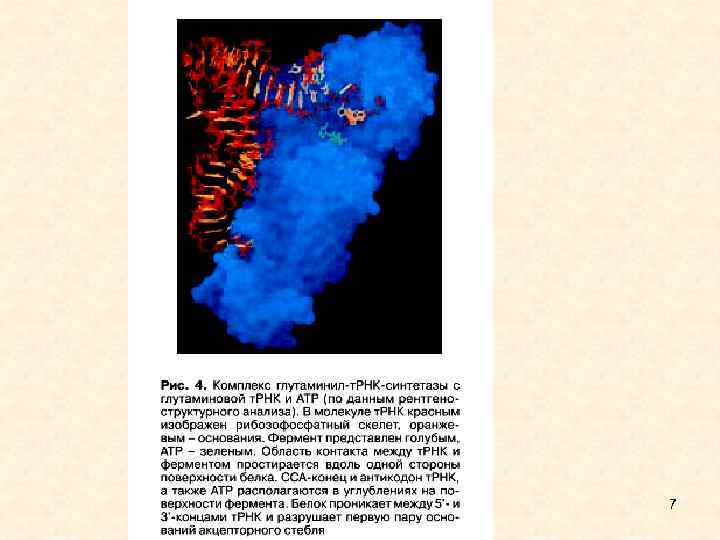
7
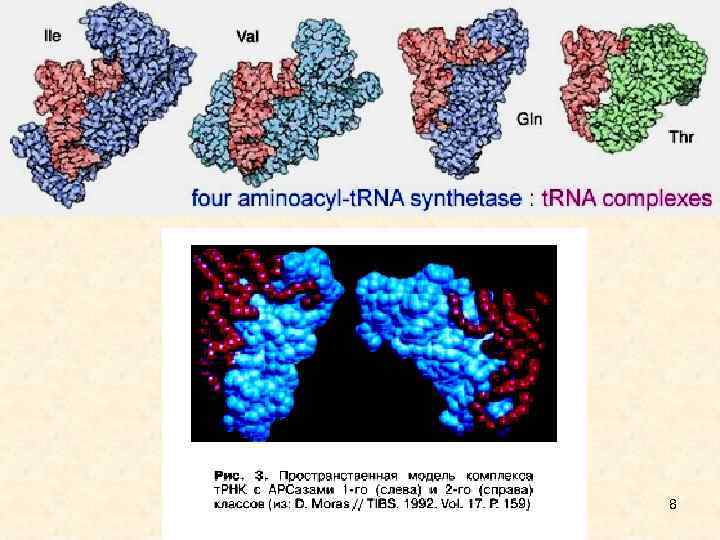
8
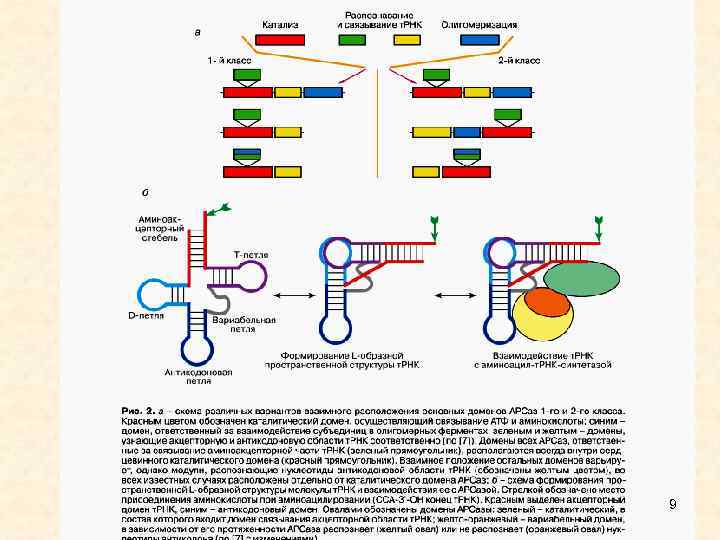
9
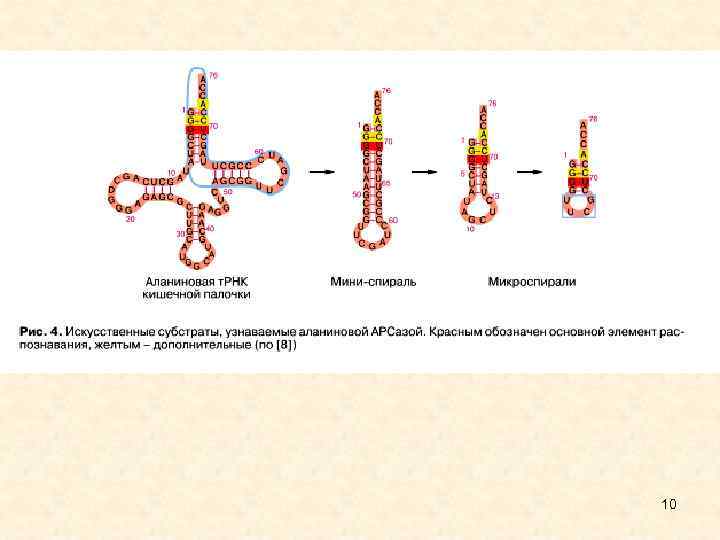
10

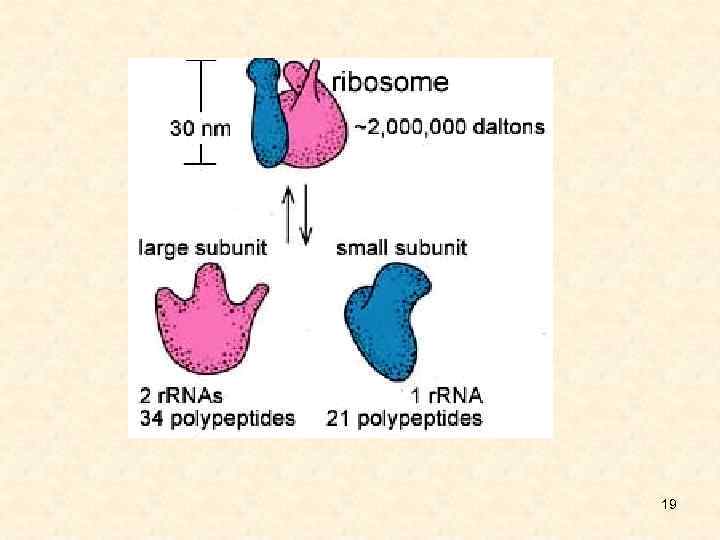
Структура рибосом • Рибосомы - немембранные самые мелкие клеточные органеллы, при этом они едва ли не самые сложные. В клетке E. сoli присутствует около 103 -5 х103 рибосом. Линейные размеры прокариотической рибосомы 210 х 290 Å. У эукариот - 220 х 320 Å. • • • Выделяют четыре класса рибосом: 1. Прокариотические 70 S. 2. Эукариотические 80 S. 3. Рибосомы митохондрий (55 S - у животных, 75 S - у грибов). 4. Рибосомы хлоропластов (70 S у высших растений). • S - коэффициент седиментации или константа Сведберга. Отражает скорость осаждения молекул или их компонентов при центрифугировании, зависящую от конформации и молекулярного веса. • Каждая рибосома состоит из 2 -х субъединиц (большой и малой). 11

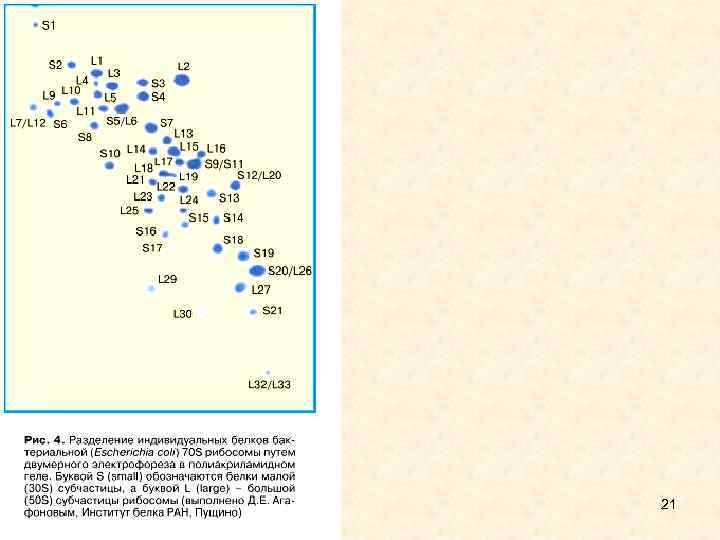
Прокариотическая рибосома Эукариотическая рибосома 70 S 80 S 50 S 5 S r. РНК 23 S r. РНК 34 молекулы белков, из них 31 разные 30 S 16 S r. РНК 21 белок 60 S 5 S r. РНК 5. 8 S r. РНК 28 S r. РНК не менее 50 разных белков 40 S 18 S r. РНК не менее 33 разных белков Сложность объясняется тем, что все элементы рибосом представлены в одном экземпляре, за исключением одного белка, присутствующего в 4 копиях в 50 S субъединице, и не могут быть заменены. меньшей степени - марганца Mn 2+). 12
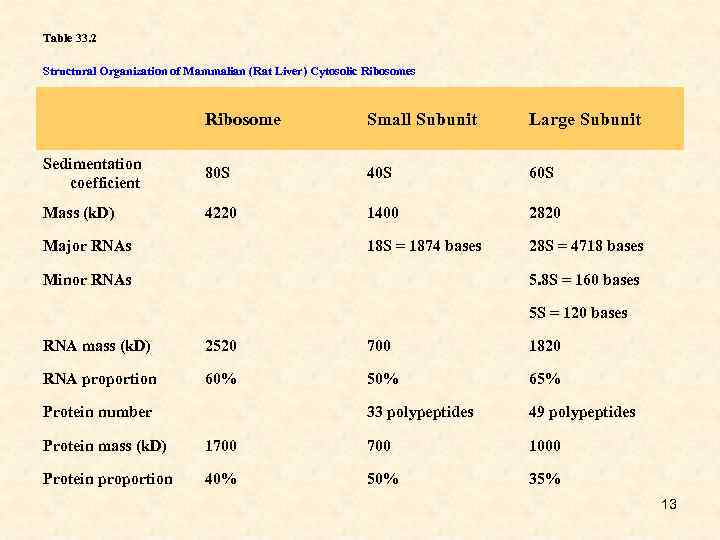
Table 33. 2 Structural Organization of Mammalian (Rat Liver) Cytosolic Ribosomes Ribosome Small Subunit Large Subunit Sedimentation coefficient 80 S 40 S 60 S Mass (k. D) 4220 1400 2820 Major RNAs 18 S = 1874 bases 28 S = 4718 bases Minor RNAs 5. 8 S = 160 bases 5 S = 120 bases RNA mass (k. D) 2520 700 1820 RNA proportion 60% 50% 65% Protein number 33 polypeptides 49 polypeptides Protein mass (k. D) 1700 1000 Protein proportion 40% 50% 35% 13

r. РНК выполняют не только функцию каркасов субъединиц рибосом, но и принимают непосредственное участие в синтезе полипептидов. 23 S r. РНК входит в каталитический пептидилтрансферазный центр, 16 S r. РНК необходима для установки на 30 S субъединице инициирующего кодона m. РНК, 5 S r. РНК - для правильной ориентации аминоацил-t. РНК на рибосоме. Все r. РНК обладают развитой вторичной структурой: около 70% нуклеотидов собрано в шпильки. r. РНК в значительной степени метилированы (СН 3 -группа во втором положении рибозы, а также в азотистых основаниях). Порядок сборки субъединиц из r. РНК и белков строго определен. Субъединицы, не соединенные друг с другом, представляют собой диссоциированные рибосомы. Соединенные - ассоциированные рибосомы. Для ассоциации нужны не только конформационные изменения, но и ионы магния Mg 2+ (до 2 х103 ионов на рибосому). Магний нужен для компенсации отрицательного заряда r. РНК. Все реакции матричного синтеза (репликация, транскрипция и трансляция) связаны с ионами магния Mg 2+ (в меньшей степени - марганца Mn 2+). 14
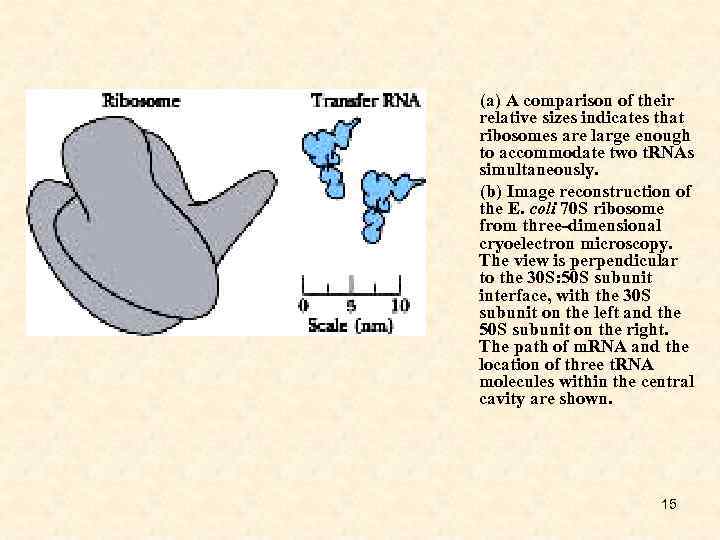
(a) A comparison of their relative sizes indicates that ribosomes are large enough to accommodate two t. RNAs simultaneously. (b) Image reconstruction of the E. coli 70 S ribosome from three-dimensional cryoelectron microscopy. The view is perpendicular to the 30 S: 50 S subunit interface, with the 30 S subunit on the left and the 50 S subunit on the right. The path of m. RNA and the location of three t. RNA molecules within the central cavity are shown. 15
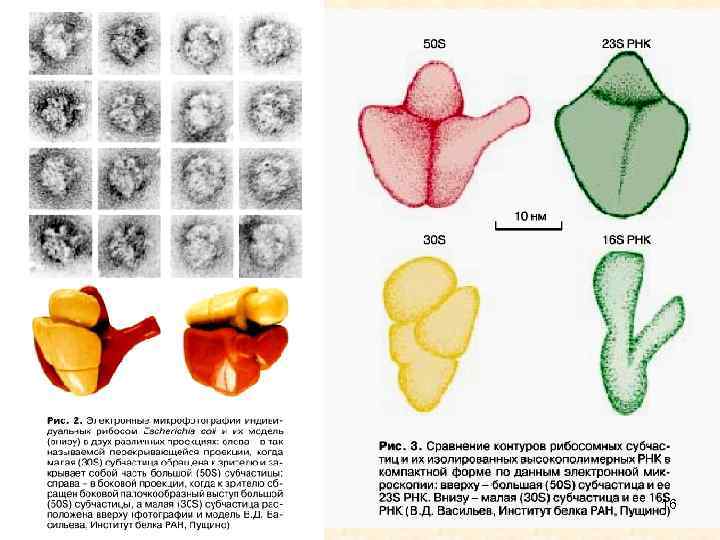
16
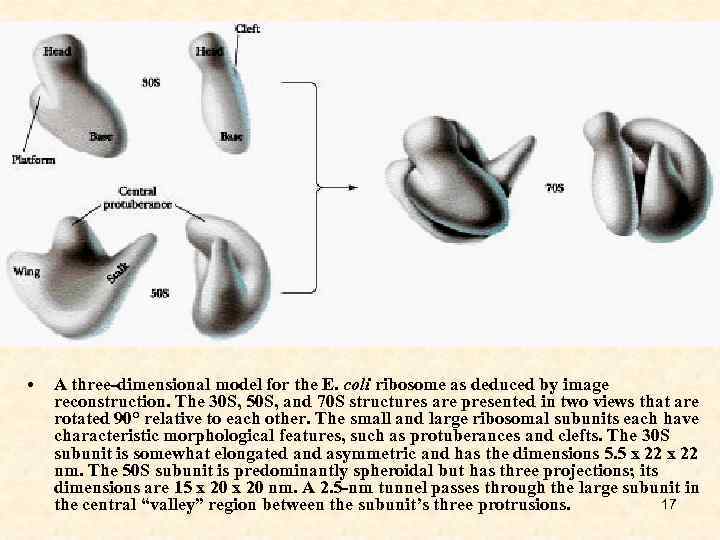
• A three-dimensional model for the E. coli ribosome as deduced by image reconstruction. The 30 S, 50 S, and 70 S structures are presented in two views that are rotated 90° relative to each other. The small and large ribosomal subunits each have characteristic morphological features, such as protuberances and clefts. The 30 S subunit is somewhat elongated and asymmetric and has the dimensions 5. 5 x 22 nm. The 50 S subunit is predominantly spheroidal but has three projections; its dimensions are 15 x 20 nm. A 2. 5 -nm tunnel passes through the large subunit in 17 the central “valley” region between the subunit’s three protrusions.
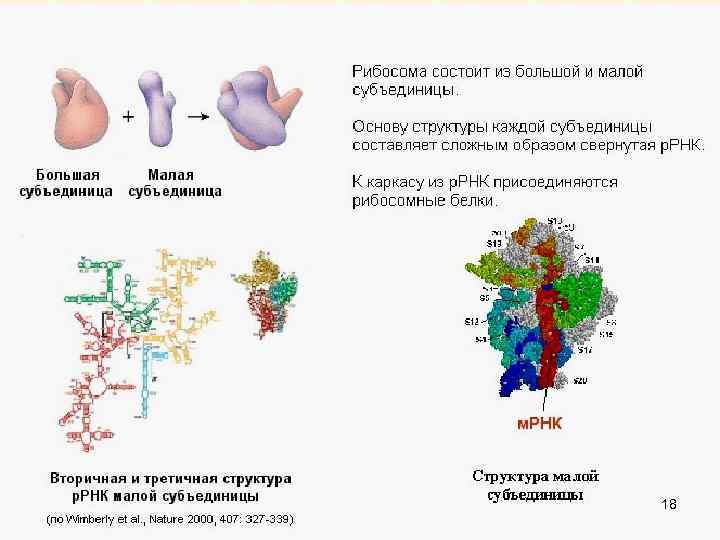
18
19
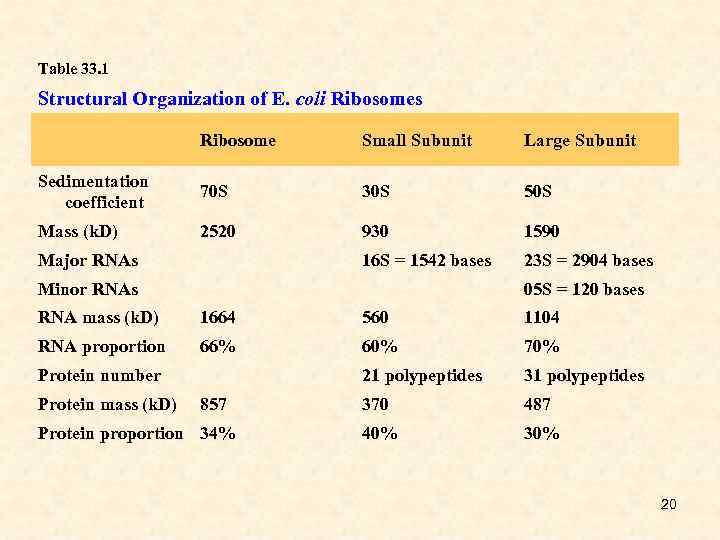
Table 33. 1 Structural Organization of E. coli Ribosomes Ribosome Small Subunit Large Subunit Sedimentation coefficient 70 S 30 S 50 S Mass (k. D) 2520 930 1590 Major RNAs 16 S = 1542 bases 23 S = 2904 bases Minor RNAs 05 S = 120 bases RNA mass (k. D) 1664 560 1104 RNA proportion 66% 60% 70% Protein number 21 polypeptides 31 polypeptides Protein mass (k. D) 857 370 487 40% 30% Protein proportion 34% 20
21

Каталитические центры рибосом • Асп - центр специфического узнавания. Здесь происходит взаимодействие кодон-антикодон. • Р-центр - пептидильный, донорный. Он является донором формилметионина при инициации, или пептидила при элонгации трансляции. • А-центр - аминоацильный, акцепторный. Акцептирует формилметионин в самом начале или пептидил при элонгации трансляции. • К-центр - каталитический (фермент пептидилтрансфераза). В К-центре задействована 23 S r. РНК и несколько белков большой субъединицы. 22
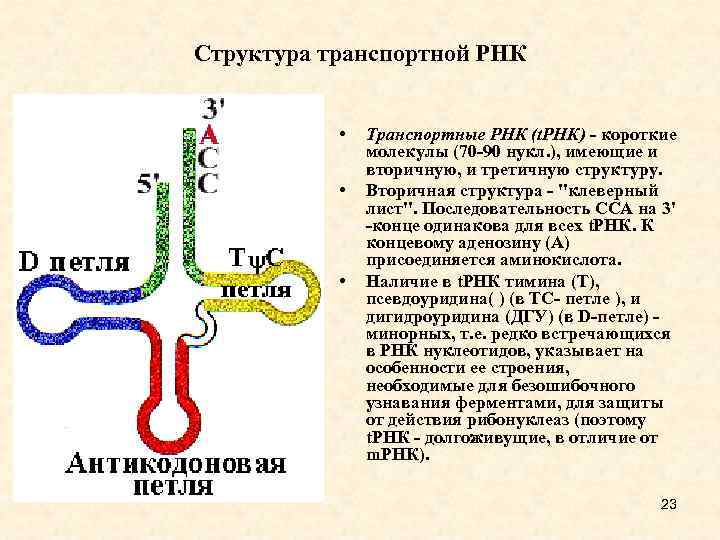
Структура транспортной РНК • • • Транспортные РНК (t. РНК) - короткие молекулы (70 -90 нукл. ), имеющие и вторичную, и третичную структуру. Вторичная структура - "клеверный лист". Последовательность CCA на 3' -конце одинакова для всех t. РНК. К концевому аденозину (А) присоединяется аминокислота. Наличие в t. РНК тимина (T), псевдоуридина( ) (в TC- петле ), и дигидроуридина (ДГУ) (в D-петле) - минорных, т. е. редко встречающихся в РНК нуклеотидов, указывает на особенности ее строения, необходимые для безошибочного узнавания ферментами, для защиты от действия рибонуклеаз (поэтому t. РНК - долгоживущие, в отличие от m. РНК). 23
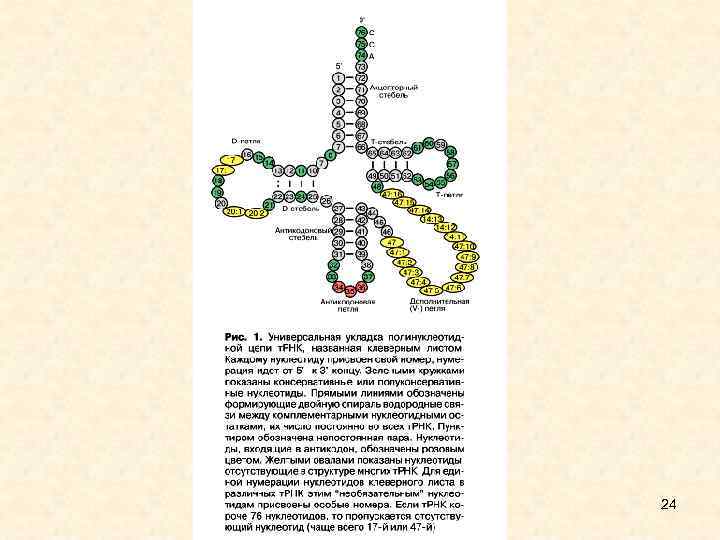
24
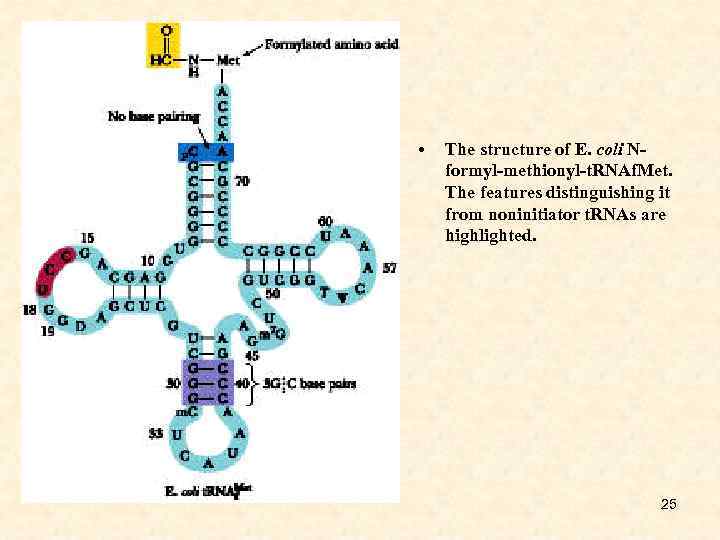
• The structure of E. coli Nformyl-methionyl-t. RNAf. Met. The features distinguishing it from noninitiator t. RNAs are highlighted. 25
26

• • • Третичная структура в проекции на плоскость имеет форму бумеранга. Разнообразие первичных структур t. РНК - 61+1 - по количеству кодонов (соответственно числу антикодонов в t. РНК) + формилметиониновая t. РНК, у которой антикодон такой же, как у метиониновой t. РНК. Разнообразие третичных структур - 20 (по количеству аминокислот). 27
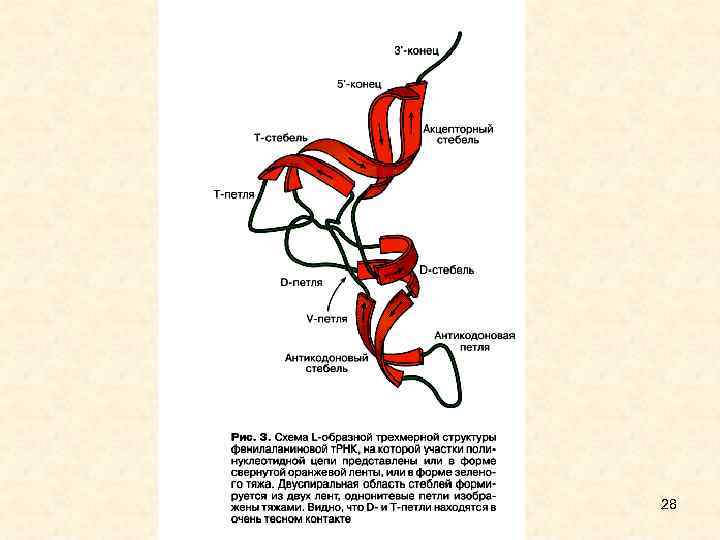
28

Синтез полипептидов на рибосоме • • У прокариот перед каждым геном и соответственно в m. РНК перед копией каждого гена имеется лидерная последовательность. Она может быть разного размера (до 160 нукл. ) и разной первичной структуры, но обязательно содержит полипуриновую последовательность Шайна. Дальгарно, которая комплементарна 3'-концевому участку 16 S r. РНК. Комплементарными могут быть 3 -9 нуклеотидов. • Назначение комплементарного взаимодействия 3'-концевого участка 16 S r. РНК и последовательности Шайна-Дальгарно - правильная установка инициирующего кодона AUG на малой субъединице рибосомы. • Инициирующий кодон находится на растоянии 3 -10 нукл. от последовательности Шайна-Дальгарно. 29
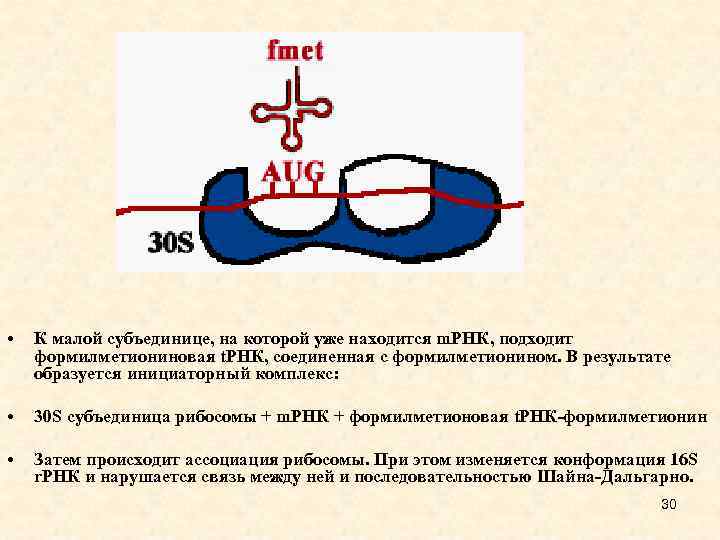
• К малой субъединице, на которой уже находится m. РНК, подходит формилметиониновая t. РНК, соединенная с формилметионином. В результате образуется инициаторный комплекс: • 30 S субъединица рибосомы + m. РНК + формилметионовая t. РНК-формилметионин • Затем происходит ассоциация рибосомы. При этом изменяется конформация 16 S r. РНК и нарушается связь между ней и последовательностью Шайна-Дальгарно. 30
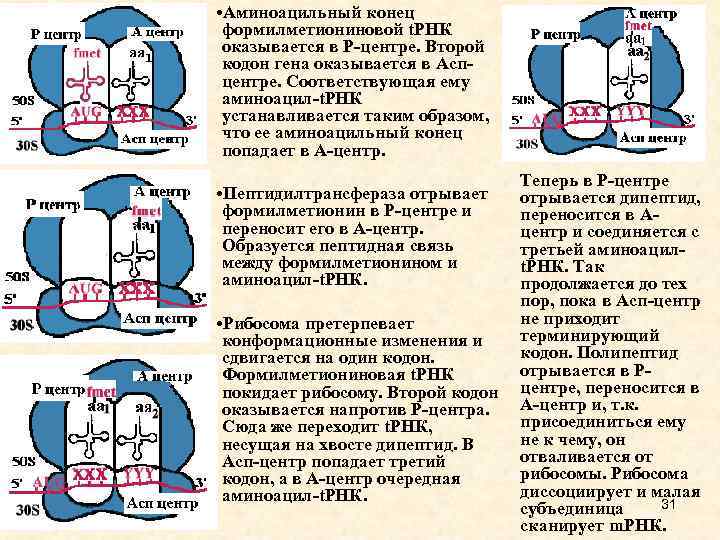
• Аминоацильный конец формилметиониновой t. РНК оказывается в Р-центре. Второй кодон гена оказывается в Аспцентре. Соответствующая ему аминоацил-t. РНК устанавливается таким образом, что ее аминоацильный конец попадает в А-центр. Теперь в Р-центре отрывается дипептид, переносится в Ацентр и соединяется с третьей аминоацилt. РНК. Так продолжается до тех пор, пока в Асп-центр не приходит • Рибосома претерпевает терминирующий конформационные изменения и кодон. Полипептид сдвигается на один кодон. отрывается в РФормилметиониновая t. РНК покидает рибосому. Второй кодон центре, переносится в оказывается напротив Р-центра. А-центр и, т. к. присоединиться ему Сюда же переходит t. РНК, не к чему, он несущая на хвосте дипептид. В отваливается от Асп-центр попадает третий рибосомы. Рибосома кодон, а в А-центр очередная диссоциирует и малая аминоацил-t. РНК. 31 субъединица сканирует m. РНК. • Пептидилтрансфераза отрывает формилметионин в Р-центре и переносит его в А-центр. Образуется пептидная связь между формилметионином и аминоацил-t. РНК.
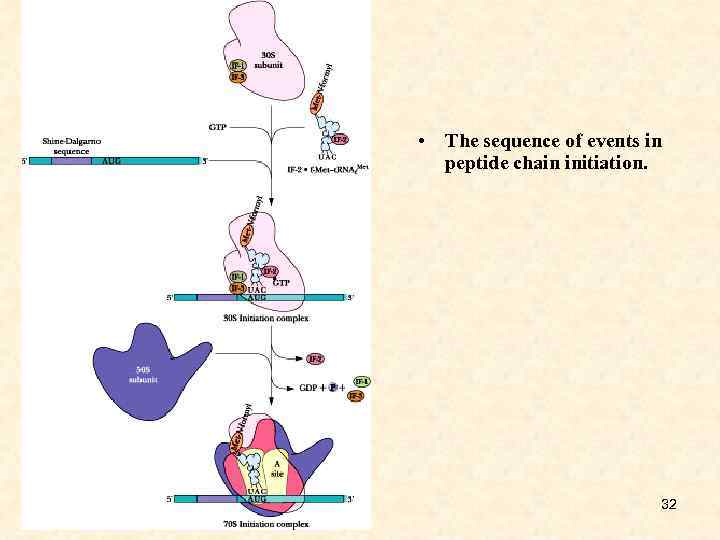
• The sequence of events in peptide chain initiation. 32
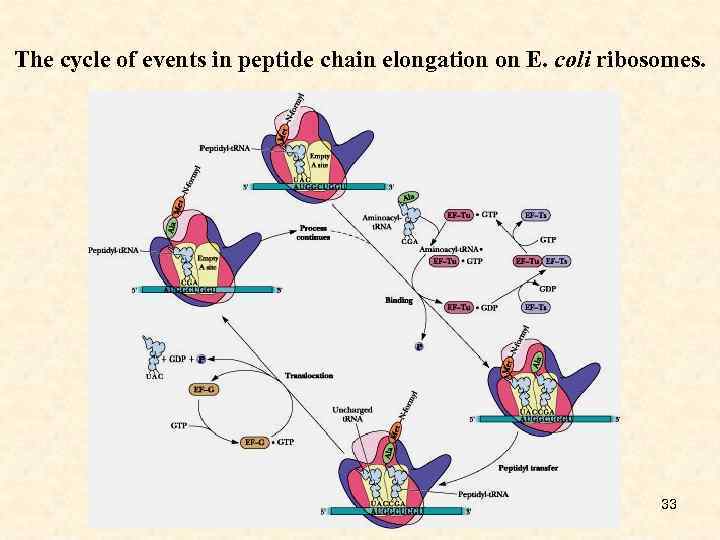
The cycle of events in peptide chain elongation on E. coli ribosomes. 33

• In vivo на каждой стадии (образования инициаторного комплекса, инициации, элонгации и терминации) участвуют различные белковые факторы, которые препятствуют посадке на рибосому деацилированных t. РНК или запрещают посадку формилметиониновой-t. РНК в А-центр. • На всех этапах принимают участие молекулы ГТФ, которые дефосфорилируются. • Смысл гидролиза ГТФ не в отдаче энергии, а в свидетельстве того, что данный этап трансляции пройден. 34
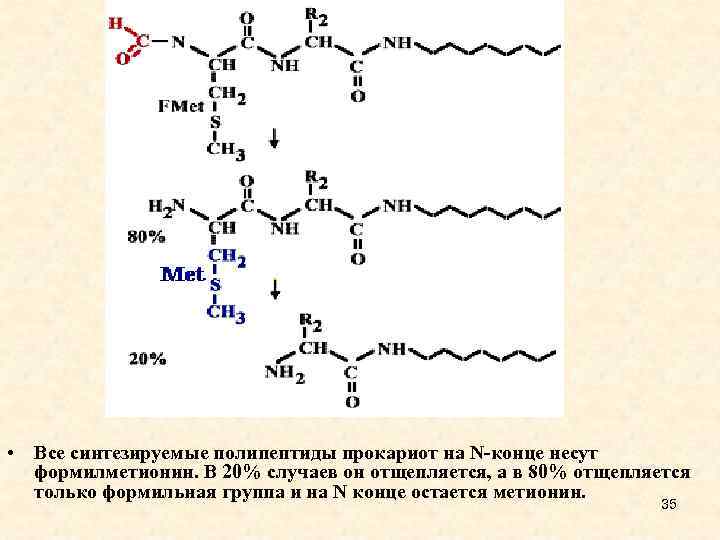
• Все синтезируемые полипептиды прокариот на N-конце несут формилметионин. В 20% случаев он отщепляется, а в 80% отщепляется только формильная группа и на N конце остается метионин. 35

• Регуляция образования рибосомных РНК и белков рибосом E. сoli • Ежеминутно в E. сoli образуется около 500 рибосом. • Имеется 7 оперонов, в которых закодированы r. РНК (всего 3 разных r. РНК х 7 оперонов = 21 ген). В формировании рибосом участвуют 52 различных белка, а значит 52 гена, их кодирующих. В итоге, • 73 гена должны работать координированно, чтобы не было избытка белков или r. РНК. • Вначале образуется про-r. РНК, которая метилируется и процессируется (т. е. "созревает"). • Количество r. РНК регулируется количеством рибосомных оперонов, 36 скоростью их транскрипции и работой ферментов метилаз и эндонуклеаз.
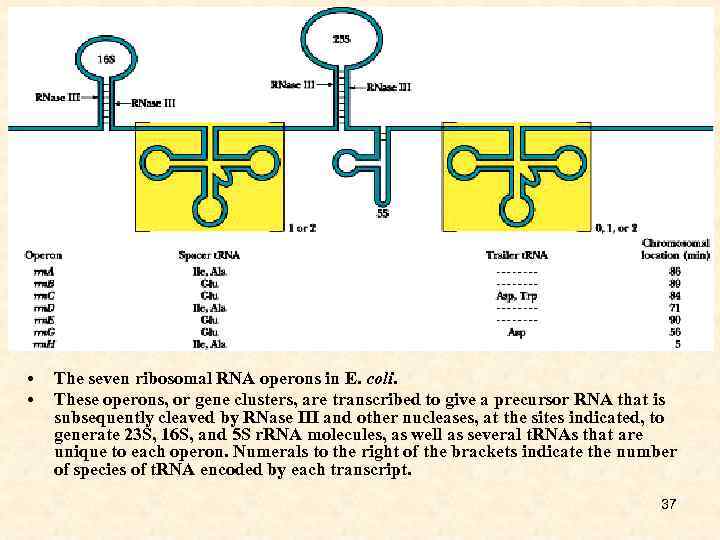
• • The seven ribosomal RNA operons in E. coli. These operons, or gene clusters, are transcribed to give a precursor RNA that is subsequently cleaved by RNase III and other nucleases, at the sites indicated, to generate 23 S, 16 S, and 5 S r. RNA molecules, as well as several t. RNAs that are unique to each operon. Numerals to the right of the brackets indicate the number of species of t. RNA encoded by each transcript. 37
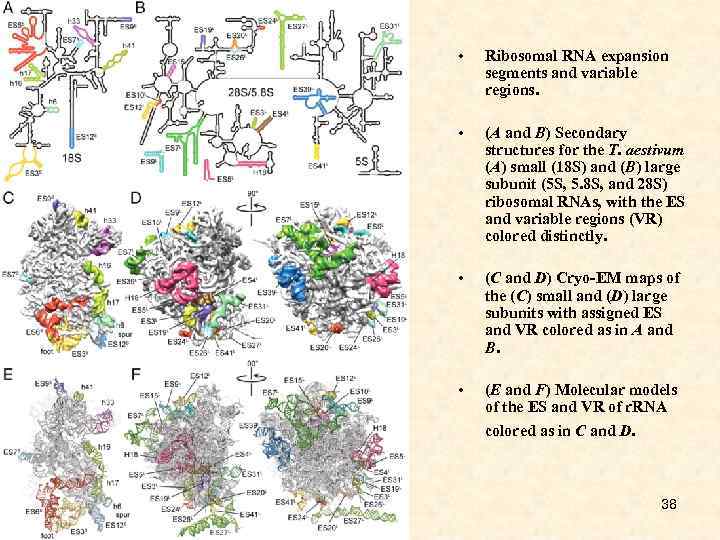
• Ribosomal RNA expansion segments and variable regions. • (A and B) Secondary structures for the T. aestivum (A) small (18 S) and (B) large subunit (5 S, 5. 8 S, and 28 S) ribosomal RNAs, with the ES and variable regions (VR) colored distinctly. • (C and D) Cryo-EM maps of the (C) small and (D) large subunits with assigned ES and VR colored as in A and B. • (E and F) Molecular models of the ES and VR of r. RNA colored as in C and D. 38
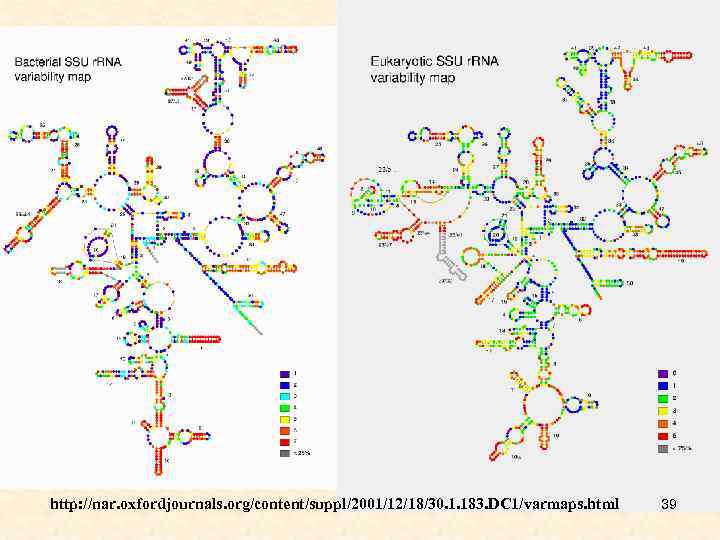
http: //nar. oxfordjournals. org/content/suppl/2001/12/18/30. 1. 183. DC 1/varmaps. html 39
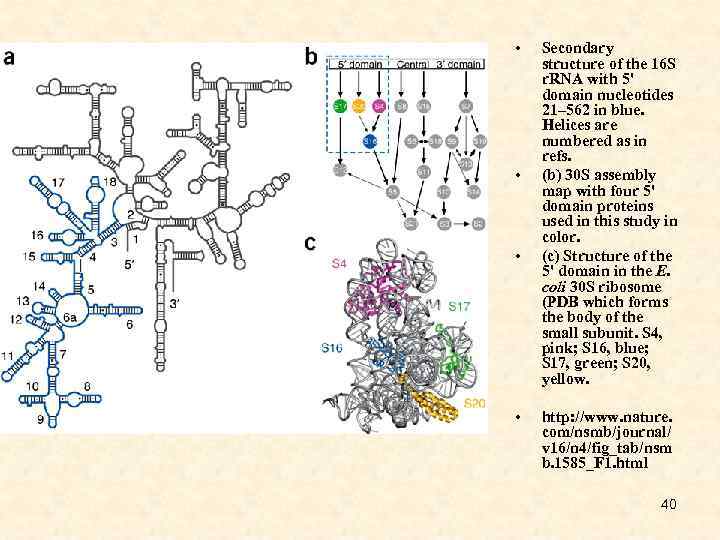
• • Secondary structure of the 16 S r. RNA with 5' domain nucleotides 21– 562 in blue. Helices are numbered as in refs. (b) 30 S assembly map with four 5' domain proteins used in this study in color. (c) Structure of the 5' domain in the E. coli 30 S ribosome (PDB which forms the body of the small subunit. S 4, pink; S 16, blue; S 17, green; S 20, yellow. http: //www. nature. com/nsmb/journal/ v 16/n 4/fig_tab/nsm b. 1585_F 1. html 40

• Conserved Bases in r. RNA Are Clustered in Single-Stranded Regions • Although retention of secondary structure apparently plays a leading role in r. RNA evolution, conservation of r. RNA primary structure at particular places is also a significant feature of these molecules. Comparison of the base sequences of 16 S-like (r. RNAs) (the small subunit r. RNA) from diverse phylogenetic sources reveals that about one-third of the nucleotides are universally conserved. Interestingly, many of these nucleotides are clustered in a few single-stranded regions of the molecule. Several such sequences on the order of 10 to 20 nucleotides long are essentially invariant in all organisms examined. Such conservation strongly suggests that these unpaired stretches play some functional role during protein synthesis. 41

• • • Имеется 7 разных оперонов, в которых закодированы рибосомные белки. Регуляция каждого из них осуществляется отдельно. α-оперон регулируется белком S 4. Если в клетке имеется свободная 16 S r. РНК, то S 4 связывется с ней (1). Если же 16 S r. РНК не хватает, то он связывается с m. РНК, считывающейся с данного оперона (2). Причем связывается в районе лидера и тем самым мешает трансляции. • Таким образом, осуществляется регуляция на уровне трансляции. 42
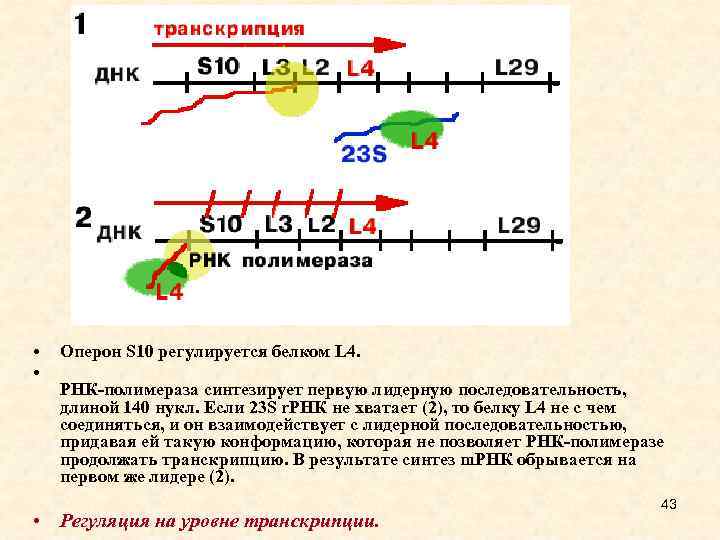
• • Оперон S 10 регулируется белком L 4. РНК-полимераза синтезирует первую лидерную последовательность, длиной 140 нукл. Если 23 S r. РНК не хватает (2), то белку L 4 не с чем соединяться, и он взаимодействует с лидерной последовательностью, придавая ей такую конформацию, которая не позволяет РНК-полимеразе продолжать транскрипцию. В результате синтез m. РНК обрывается на первом же лидере (2). • Регуляция на уровне транскрипции. 43
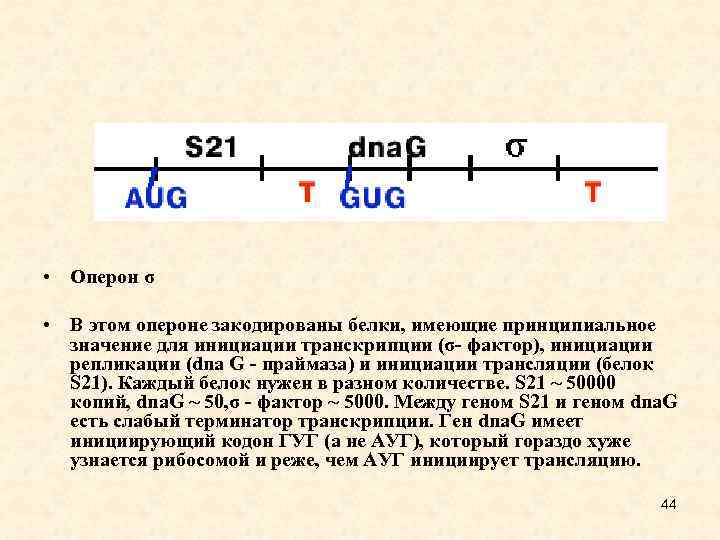
• Оперон σ • В этом опероне закодированы белки, имеющие принципиальное значение для инициации транскрипции (σ- фактор), инициации репликации (dna G - праймаза) и инициации трансляции (белок S 21). Каждый белок нужен в разном количестве. S 21 ~ 50000 копий, dna. G ~ 50, σ - фактор ~ 5000. Между геном S 21 и геном dna. G есть слабый терминатор транскрипции. Ген dna. G имеет инициирующий кодон ГУГ (а не АУГ), который гораздо хуже узнается рибосомой и реже, чем АУГ инициирует трансляцию. 44

Аттенуация (ослабление) • Рассмотрим систему аттенуации на примере триптофанового оперона E. сoli. Этот оперон регулируется по схеме негативной репрессии. При недостатке в клетке триптофана оперон открыт. При увеличении концентрации триптофана РНК-полимераза не доходит даже до первого цистрона. • Между оператором и первым цистроном есть протяженный участок (162 п. н. ), который содержит последовательность Шайна. Дальгарно. Она расположена ближе к цистрону, все остальное же представляет собой аттенуатор. • В этом районе происходит прекращение транскрипции и отсоединение РНК-полимеразы от ДНК. Это сделано для того, чтобы остановить РНК-полимеразу, которая уже в пути, в том случае, если концентрация триптофана в клетке к этому моменту повысилась. 45
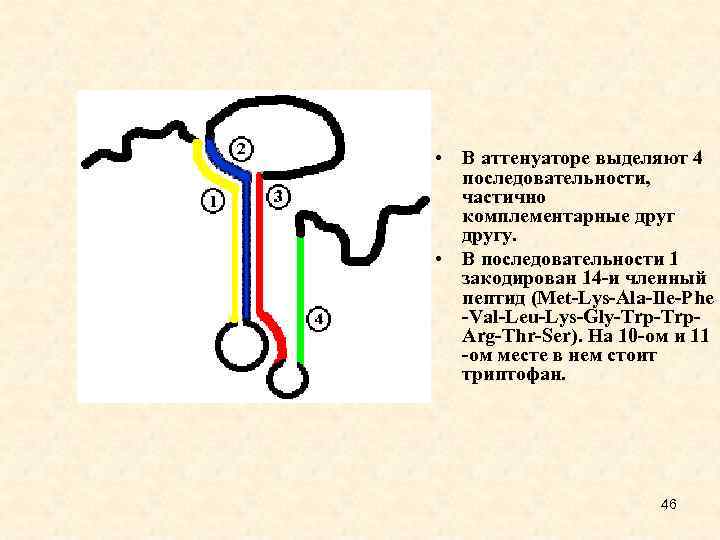
• В аттенуаторе выделяют 4 последовательности, частично комплементарные другу. • В последовательности 1 закодирован 14 -и членный пептид (Met-Lys-Ala-Ile-Phe -Val-Leu-Lys-Gly-Trp. Arg-Thr-Ser). На 10 -ом и 11 -ом месте в нем стоит триптофан. 46
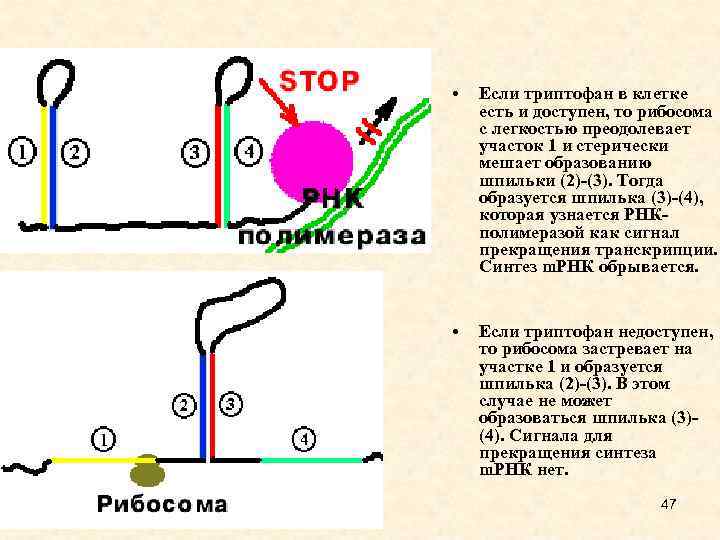
• Если триптофан в клетке есть и доступен, то рибосома с легкостью преодолевает участок 1 и стерически мешает образованию шпильки (2)-(3). Тогда образуется шпилька (3)-(4), которая узнается РНКполимеразой как сигнал прекращения транскрипции. Синтез m. РНК обрывается. • Если триптофан недоступен, то рибосома застревает на участке 1 и образуется шпилька (2)-(3). В этом случае не может образоваться шпилька (3)(4). Сигнала для прекращения синтеза m. РНК нет. 47
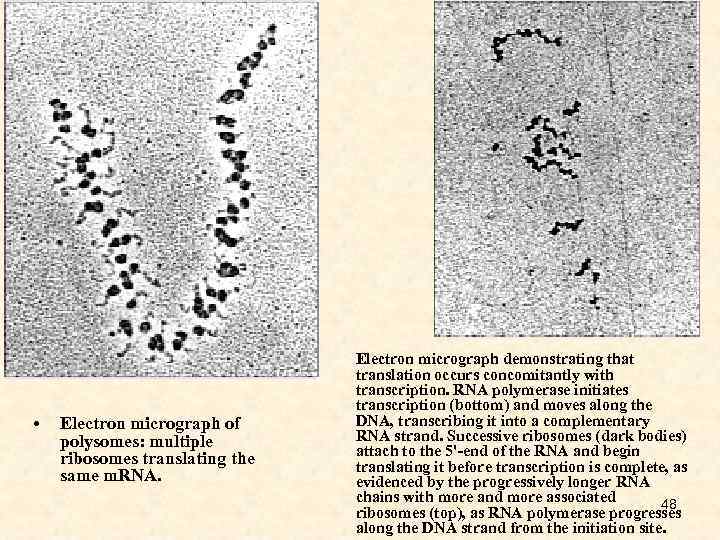
• Electron micrograph of polysomes: multiple ribosomes translating the same m. RNA. Electron micrograph demonstrating that translation occurs concomitantly with transcription. RNA polymerase initiates transcription (bottom) and moves along the DNA, transcribing it into a complementary RNA strand. Successive ribosomes (dark bodies) attach to the 5'-end of the RNA and begin translating it before transcription is complete, as evidenced by the progressively longer RNA chains with more and more associated 48 ribosomes (top), as RNA polymerase progresses along the DNA strand from the initiation site.
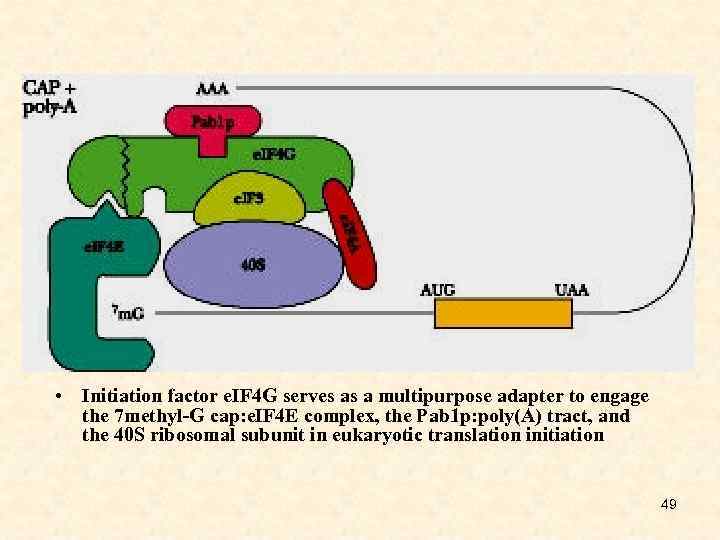
• Initiation factor e. IF 4 G serves as a multipurpose adapter to engage the 7 methyl-G cap: e. IF 4 E complex, the Pab 1 p: poly(A) tract, and the 40 S ribosomal subunit in eukaryotic translation initiation 49
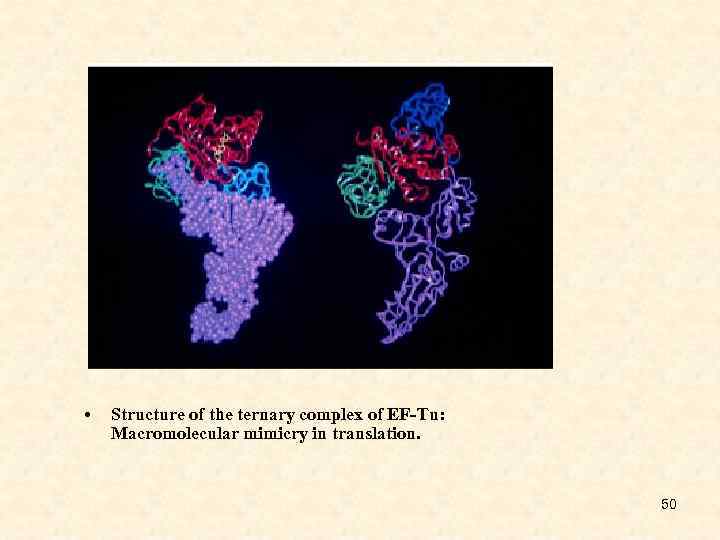
• Structure of the ternary complex of EF-Tu: Macromolecular mimicry in translation. 50
51

Properties of E. coli Elongation Factors Factor Mass (k. D) Molecules/ Function Cell EF-Tu 43 70, 000 Binds aminoacyl-t. RNA in presence of GTP EF-Ts 74 10, 000 Displaces GDP from EF-Tu EF-G 77 20, 000 Binds GTP, promotes translocation of ribosome along m. RNA 52

Properties of Eukaryotic Translation Initiation Factors Factor Subunit Size Function (k. D) e. IF 1 15 Enhances initiation complex formation e. IF 1 A 17 Stabilizes Met-t. RNAi binding to 40 S ribosomes e. IF 2 e. IF 2 B e. IF 2 C 125 36 50 55 270 26 39 58 67 82 94 GTP-dependent Met-t. RNAi binding to 40 S ribosomes Regulated by phosphorylation Binds Met-t. RNAi Binds GTP, Met-t. RNAi Promotes guanine nucleotide exchange on e. IF 2 Binds GTP Binds ATP Regulated by phosphorylation Stabilizes ternary complex in presence of RNA 53
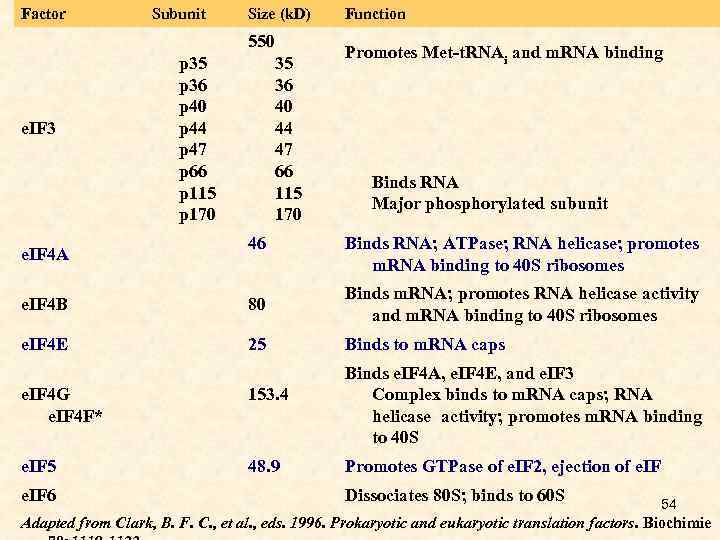
Factor Subunit Size (k. D) 550 p 35 p 36 p 40 p 44 p 47 p 66 p 115 p 170 e. IF 3 35 36 40 44 47 66 115 170 Function Promotes Met-t. RNAi and m. RNA binding Binds RNA Major phosphorylated subunit e. IF 4 A 46 Binds RNA; ATPase; RNA helicase; promotes m. RNA binding to 40 S ribosomes e. IF 4 B 80 Binds m. RNA; promotes RNA helicase activity and m. RNA binding to 40 S ribosomes e. IF 4 E 25 Binds to m. RNA caps e. IF 4 G e. IF 4 F* 153. 4 Binds e. IF 4 A, e. IF 4 E, and e. IF 3 Complex binds to m. RNA caps; RNA helicase activity; promotes m. RNA binding to 40 S e. IF 5 48. 9 Promotes GTPase of e. IF 2, ejection of e. IF 6 Dissociates 80 S; binds to 60 S 54 Adapted from Clark, B. F. C. , et al. , eds. 1996. Prokaryotic and eukaryotic translation factors. Biochimie

Some Protein Synthesis Inhibitor System Inhibited Mode of Action Initiation Aurintricarboxylic acid Prokaryotic Prevents IF binding to 30 S subunit Kasugamycin Prokaryotic Inhibits F-Met-t. RNAf. Met binding Streptomycin Prokaryotic Prevents formation of initiation complexes 55

Some Protein Synthesis Inhibitors Elongation: Aminoacyl- Prokaryotic Inhibits aminoacyl-t. RNA binding at A site Prokaryotic Codon misreading, insertion of improper amino acid Prokaryotic Peptidyl transferase inhibitor Prokaryotic Binds to 50 S subunit, blocks peptidyl transferase activity Erythromycin Prokaryotic Binds to 50 S subunit, blocks peptidyl transferase activity Cycloheximide Eukaryotic Inhibits translocation of 56 peptidyl-t. RNA Binding Tetracycline Streptomycin Elongation: Peptide Bond Formation Sparsomycin Chloramphenicol

Some Protein Synthesis Inhibitors Elongation: Both Inhibits EF-G: GDP dissociation from ribosome Thiostrepton Prokaryotic Inhibits ribosomedependent EF-Tu and EF-G GTPase Diphtheria toxin Eukaryotic Inactivates e. EF 2 through ADP-ribosylation Premature Termination Puromycin Both Aminoacyl-t. RNA analog, which acts as a peptidyl acceptor, aborting further peptide elongation Ribosome Inactivation Ricin Eukaryotic Catalytic inactivation of 28 S r. RNA 57 Translocation Fusidic acid

Table 33. 6 Some Protein Synthesis Inhibitor System Inhibited Mode of Action Initiation Aurintricarboxylic acid Prokaryotic Prevents IF binding to 30 S subunit Kasugamycin Prokaryotic Inhibits F-Met-t. RNAf. Met binding Streptomycin Prokaryotic Prevents formation of initiation complexes Elongation: Aminoacyl-t. RNA Binding Tetracycline Prokaryotic Inhibits aminoacyl-t. RNA binding at A site Streptomycin Prokaryotic Codon misreading, insertion of improper amino acid Elongation: Peptide Bond Formation Sparsomycin Prokaryotic Peptidyl transferase inhibitor Chloramphenicol Prokaryotic Binds to 50 S subunit, blocks peptidyl transferase activity Erythromycin Prokaryotic Binds to 50 S subunit, blocks peptidyl transferase activity Cycloheximide Eukaryotic Inhibits translocation of peptidyl-t. RNA Elongation: Translocation Fusidic acid Both Inhibits EF-G: GDP dissociation from ribosome Thiostrepton Prokaryotic Inhibits ribosome-dependent EF-Tu and EF-G GTPase Diphtheria toxin Eukaryotic Inactivates e. EF 2 through ADP-ribosylation Premature Termination Puromycin Both Aminoacyl-t. RNA analog, which acts as a peptidyl acceptor, aborting further peptide elongation Ribosome Inactivation Ricin Eukaryotic Catalytic inactivation of 28 S r. RNA 58

Properties of E. coli Initiation Factors Factor Mass Molecules/ Function (k. D) Ribosome IF-1 9 0. 15 Assists IF-3 function IF-2 97 Binds initiator t. RNA and GTP IF-3 23 0. 25 Binds to 30 S subunits and directs m. RNA binding 59

Факторы инициации трансляции дрожжей S. cerevisiae Фактор Субъединица Предполагаемая функция e. IF 1 Обеспечивает связывание Met-т. РНК и м. РНК с 40 S субчастицей рибосом и распознавание инициирующего AUG-кодона (у животных) e. IF 1 A Обеспечивает диссоциацию 40 S– 60 S субчастиц рибосом, связывание Met-т. РНК и распознавание инициирующего кодона (у животных) e. IF 2 α, β, γ Участвует в выборе инициирующего кодона e. IF 2 B α, β, γ, δ, ε Oбеспечивает обмен гуанилового нуклеотида на e. IF 2 e. IF 3 α, β, γ, δ, ε, ζ, η, θ Обеспечивает связывание Met-т. РНК и м. РНК с 40 S субчастицей и диссоциацию 40 S– 60 S субчастиц рибосом e. IF 4 A АТРаза, РНК-связывающая хеликаза e. IF 4 B Хеликаза, облегчающая связывание РНК e. IF 4 E Взаимодействует с кэп-группой м. РНК e. IF 4 G G 1(p 150), G 2(p 130) Взаимодействует с e. IF 3, e. IF 4 E и Pab 1 p e. IF 4 H Стимулирует активность e. IF 4 B и компонентов e. IF 4 F e. IF 5 Вызывает диссоциацию факторов инициации e. IF 5 A Функции неизвестны, мутации изменяют стабильность м. РНК e. IF 6 Вызывает диссоциацию 40 S– 60 S субчастиц рибосом 60

Регуляция экспрессии генов на уровне трансляции: В процесс биосинтеза белка рибосомами вовлекается большое количество м. РНК, экипированных разнообразными регуляторными элементами. Даже в случае клеток дрожжей количество транслируемых видов м. РНК превышает 6000. Регуляторные последовательности м. РНК влияют на эффективность трансляции двумя основными путями: 1) изменением активности компонентов системы трансляции, взаимодействующих с регуляторными доменами м. РНК; 2) изменением структуры регуляторных элементов самих м. РНК. Как и в случае транскрипции, механизмы регуляции экспрессии генов на уровне трансляции осуществляют контроль эффективности всех основных этапов синтеза полипептидных цепей: инициации, элонгации и терминации. 61

РНК антисмысловые как регуляторы трансляции Прокариотические антисмысловые РНК длиной 70 -110 нт образуют структуры типа "стебель-петля" , в которых стебель защищает эти РНК от деградации, а петля длиной шесть-восемь нт служит для первоначального взаимодействия с м. РНК-мишенью. После образования комплексов РНК-РНК наблюдали изменение стабильности м. РНК, эффективности процессинга РНК-мишени, терминации транскрипции или инициации их трансляции. Антисмысловые РНК являются мощными природными модуляторами экспрессии генов у прокариот. Данные о возможном участии природных антисмысловых РНК в регуляции трансляции у эукариот противоречивы. 62

Короткие ОРС как регуляторы трансляции Около 10% м. РНК растений содержат в своих 5'-концевых лидерных последовательностях более одного AUG-кодона. Некоторые из них удаляются с помощью альтернативного сплайсинга. Другие возникают в результате использования РНК-полимеразами альтернативных промоторов при инициации транскрипции соответствующих генов. Присутствие коротких ОРС в лидерных последовательностях м. РНК, как правило, сопровождается снижением эффективности трансляции таких матриц. Функционирование этого механизма обнаружено в клетках млекопитающих, растений и дрожжей. 63

• Трансляция у эукариот: Регуляция: Анализ стартов трансляции • Был проведен анализ стартов трансляции Drosophila и позвоночных. Были выведены различающиеся консенсусы для позвоночных (CANCAUG) и Drosophila ((C, A)AA(A, C)AUG). Полученная консенсусная последовательность для позвоночных близка к вышеприведенным консенсусам, предложенным Kozak и Lutcke с соавторами. • Консенсусные последовательности окружения старта трансляции для м. РНК дрожжей были предложены в работах Hamilton с соавторами. Эти консенсусы, полученные в результате анализа инициаторных районов 96 генов (WA(A, C)AAUGUCY) и 131 гена ((A, Y)AWAAUGUCU), оказались близки. • В ряде работ были предприняты попытки поиска в последовательностях эукариотических РНК структур, аналогичных последовательности Шайна-Дальгарно прокариот. Результаты этих исследований в целом согласуются с гипотезой Козак. 64

Трансляция у эукариот: м. РНК-р. РНК взаимодействия Предполагается, что взаимодействие комплементарных участков м. РНК и р. РНК во время трансляции имеет также место и у эукариотических видов, однако это предположение еще нуждается в экспериментальной проверке. О такой возможности свидетельствует, в частности, присутствие в эукариотических клетках стабильных комплексов м. РНК с рибосомной РНК. Стабильные межмолекулярные комплексы 18 S р. РНК и м. РНК, освобожденные от рибосомных белков, были выделены из клеток человека. В последние годы получены свидетельства о взаимодействии м. РНК с 18 S рибосомной РНК человека при инициации трансляции. Аналогичные данные были получены при исследовании гибридизации молекул 5 S р. РНК, 18 S р. РНК и м. РНК человека, а также ингибирования процессов трансляции олигонуклеотидами, комплементарными 18 S р. РНК. Закономерности распределения нуклеотидов в различных позициях кодонов м. РНК, обнаруженные Трифоновым, дают основания полагать, что м. РНК- р. РНК взаимодействия участвуют в механизме контроля рамки считывания , основывающегося на комплементарных контактах между м. РНК и р. РНК. Доводом в пользу этого предположения является существование скрытого периодического мотива 5'(GCU)3' в эукариотической м. РНК, комплементарного фрагменту 18 S р. РНК. 65

Энхансеры трансляционные: Общие сведения Трансляционными энхансерами называются специфические последовательности матричной РНК, располагающиеся в 5' и 3'- нетранслируемых областях в окрестностях последовательности Шайна-Дальгарно и кодона AUG , и способствующие отбору молекул матричных РНК рибосомой. Энхансерные последовательности узнают комплементарные участки 16 S рибосомной РНК , с которой они взаимодействуют на стадии образования инициаторного контакта м. РНК с рибосомой. Предполагается, что это взаимодействие приводит к относительному увеличению концентрации м. РНК в окрестностях 30 S субъединицы рибосомы , повышая таким образом возможности трансляции матричных РНК. Энхансер трансляционный UUAACUUUA Трансляционный энхансер UUAACUUUA, получивший название "эпсилон", был обнаружен в окрестностях последовательности Шайна- Дальгарно в гене 10 фага Т 4. Эта AU- богатая последовательность значительно усиливает экспрессию данного гена, а также эффективно функционирует, будучи помещенной перед другими неродственными генами. Сходный энхансерный элемент (UUAAUUUAC и UUAACU) был выявлен в гене atp Escherichia coli. Последовательность "эпсилон" комплементарна нуклеотидам 458466 16 S рибосомной РНК E. coli, располагающимся на поверхности субъединицы рибосомы. Предполагается, что эти пиримидин-богатые последовательности могут также взаимодействовать с рибосомным белком S 1. 66
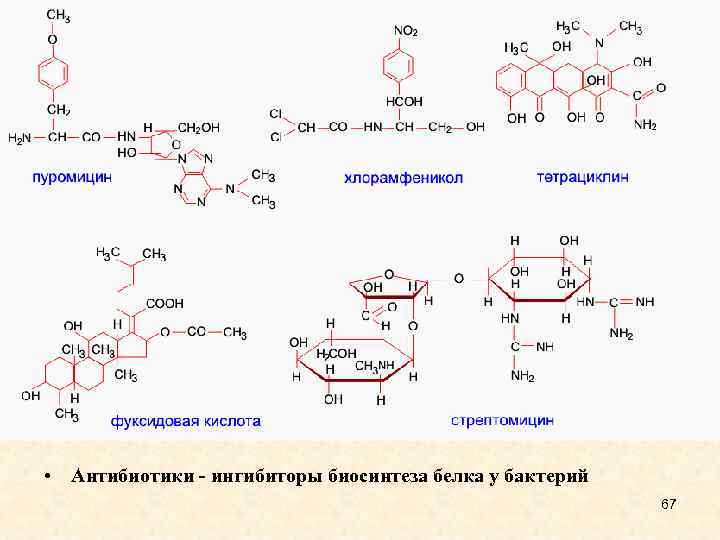
• Антибиотики - ингибиторы биосинтеза белка у бактерий 67
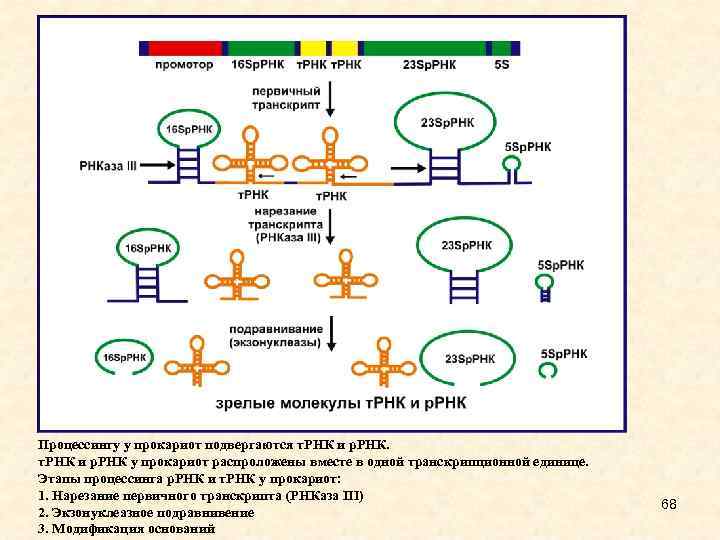
Процессингу у прокариот подвергаются т. РНК и р. РНК у прокариот распроложены вместе в одной транскрипционной единице. Этапы процессинга р. РНК и т. РНК у прокариот: 1. Нарезание первичного транскрипта (РНКаза III) 2. Экзонуклеазное подравнивение 3. Модификация оснований 68

Структура генов т. РНК Гены разных т. РНК сгруппированы в кластеры и расположены в разных хромосомах. У человека на гаплоидный геном приходится 1300 генов т. РНК. Промотор генов т. РНК располагается в внутри кодирующей последовательности и имеет два района (А- и В-боксы). В генах т. РНК есть один интрон размером от 14 до 60 нп. Он располагается через один нуклеотид от 3'-конца антикодона. 69
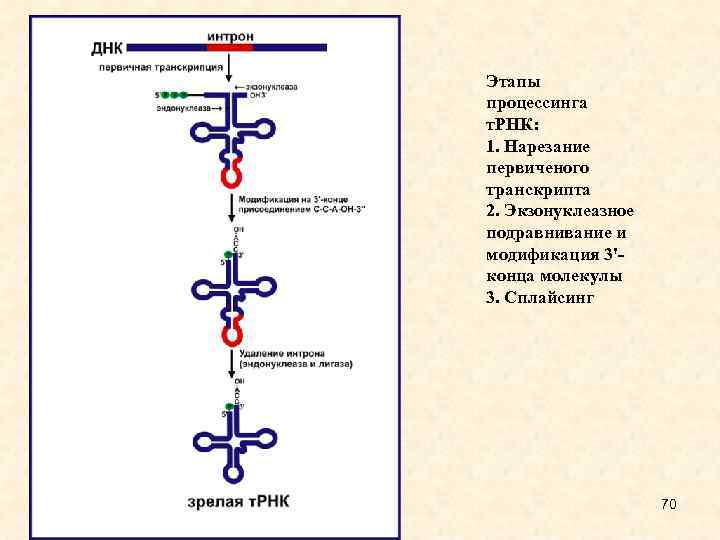
Этапы процессинга т. РНК: 1. Нарезание первиченого транскрипта 2. Экзонуклеазное подравнивание и модификация 3'конца молекулы 3. Сплайсинг 70
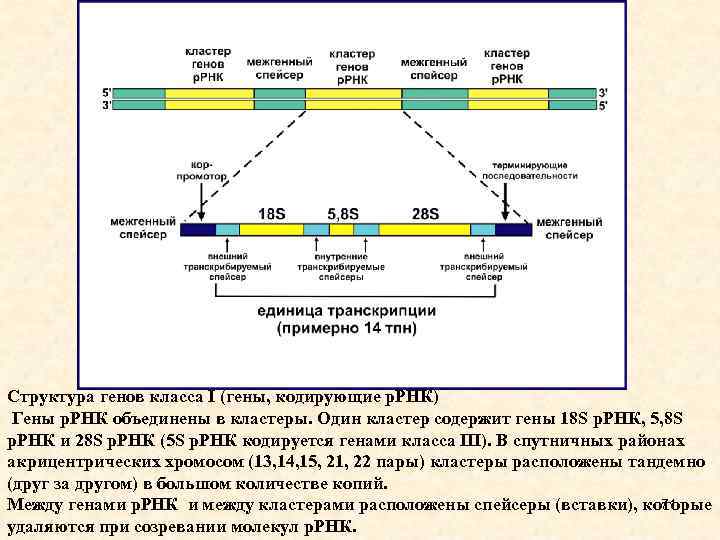
Структура генов класса I (гены, кодирующие р. РНК) Гены р. РНК объединены в кластеры. Один кластер содержит гены 18 S р. РНК, 5, 8 S р. РНК и 28 S р. РНК (5 S р. РНК кодируется генами класса III). В спутничных районах акрицентрических хромосом (13, 14, 15, 21, 22 пары) кластеры расположены тандемно (друг за другом) в большом количестве копий. 71 Между генами р. РНК и между кластерами расположены спейсеры (вставки), которые удаляются при созревании молекул р. РНК.
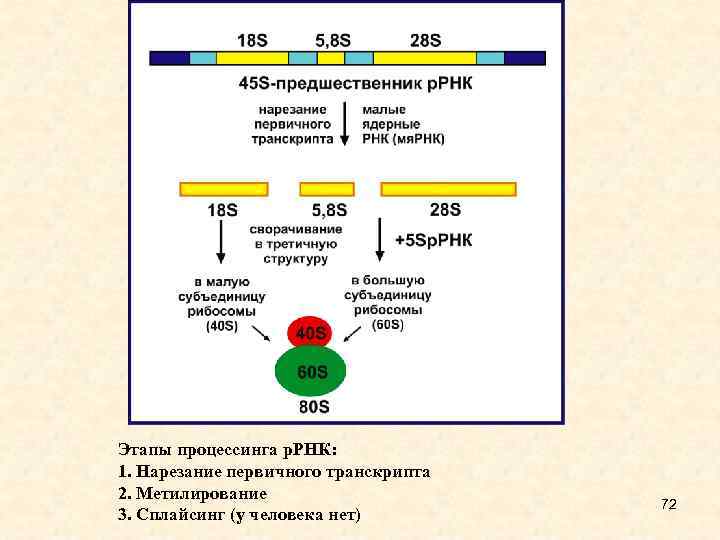
Этапы процессинга р. РНК: 1. Нарезание первичного транскрипта 2. Метилирование 3. Сплайсинг (у человека нет) 72

73
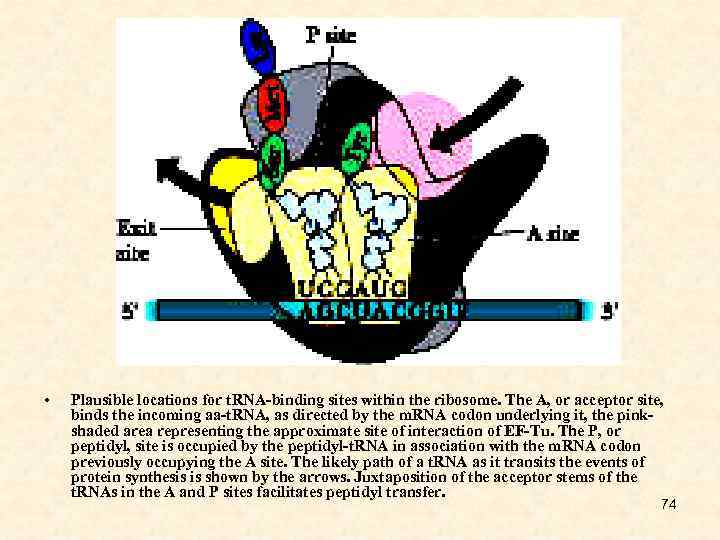
• Plausible locations for t. RNA-binding sites within the ribosome. The A, or acceptor site, binds the incoming aa-t. RNA, as directed by the m. RNA codon underlying it, the pinkshaded area representing the approximate site of interaction of EF-Tu. The P, or peptidyl, site is occupied by the peptidyl-t. RNA in association with the m. RNA codon previously occupying the A site. The likely path of a t. RNA as it transits the events of protein synthesis is shown by the arrows. Juxtaposition of the acceptor stems of the t. RNAs in the A and P sites facilitates peptidyl transfer. 74
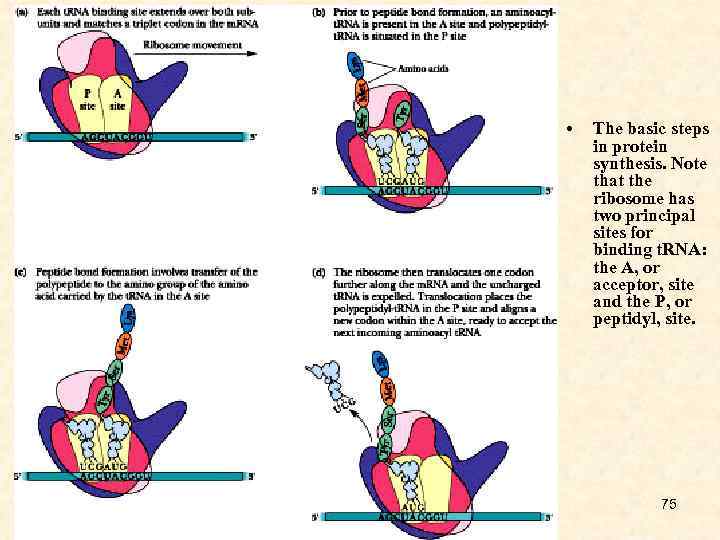
• The basic steps in protein synthesis. Note that the ribosome has two principal sites for binding t. RNA: the A, or acceptor, site and the P, or peptidyl, site. 75
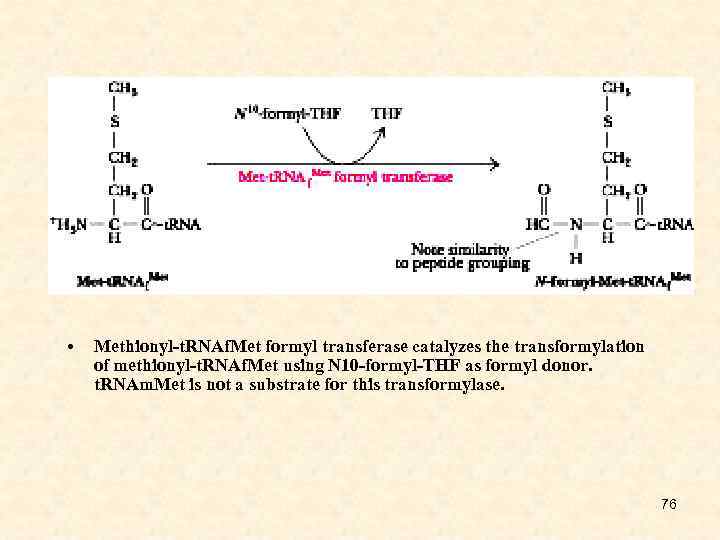
• Methionyl-t. RNAf. Met formyl transferase catalyzes the transformylation of methionyl-t. RNAf. Met using N 10 -formyl-THF as formyl donor. t. RNAm. Met is not a substrate for this transformylase. 76
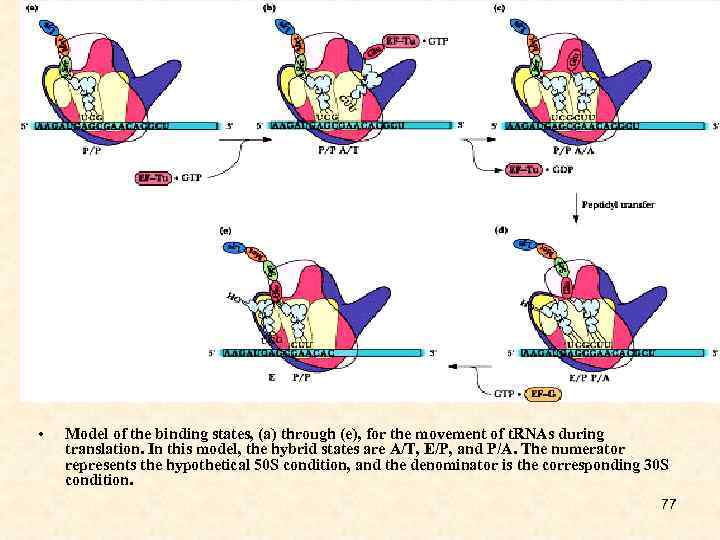
• Model of the binding states, (a) through (e), for the movement of t. RNAs during translation. In this model, the hybrid states are A/T, E/P, and P/A. The numerator represents the hypothetical 50 S condition, and the denominator is the corresponding 30 S condition. 77
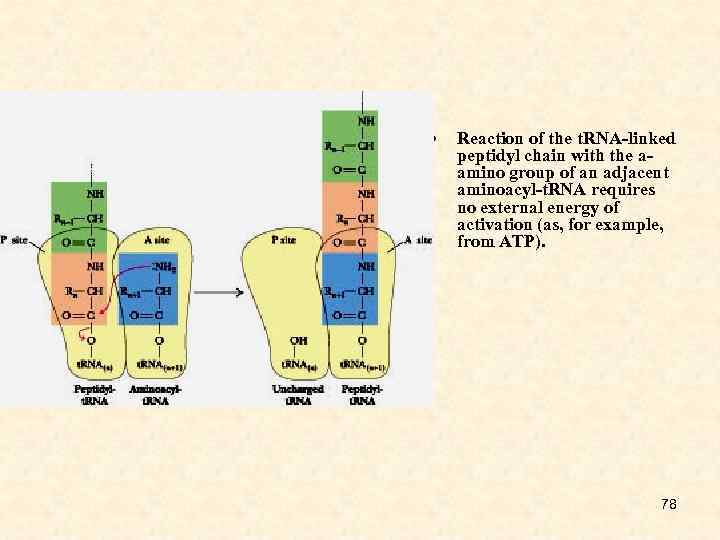
• Reaction of the t. RNA-linked peptidyl chain with the aamino group of an adjacent aminoacyl-t. RNA requires no external energy of activation (as, for example, from ATP). 78
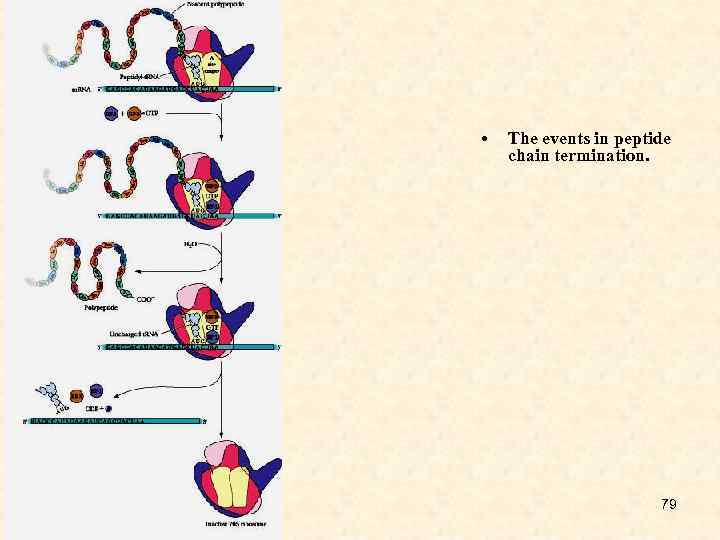
• The events in peptide chain termination. 79
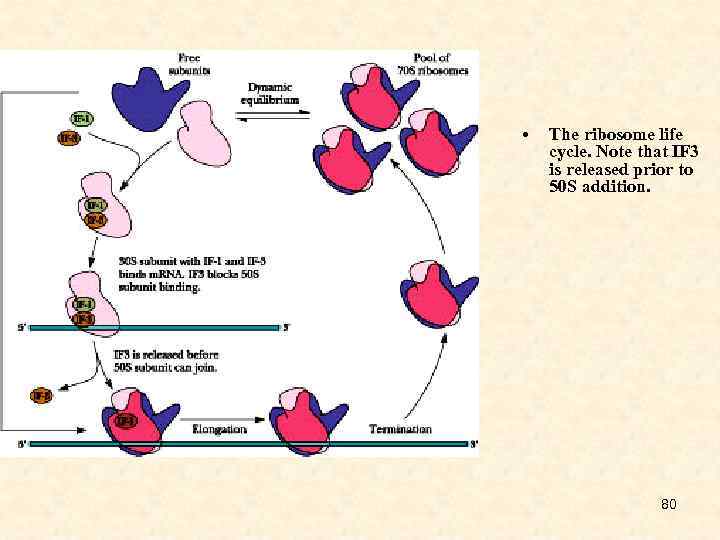
• The ribosome life cycle. Note that IF 3 is released prior to 50 S addition. 80
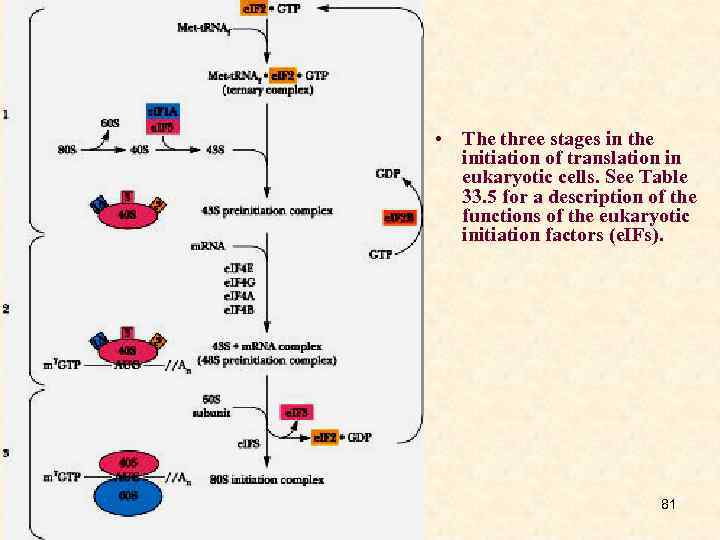
• The three stages in the initiation of translation in eukaryotic cells. See Table 33. 5 for a description of the functions of the eukaryotic initiation factors (e. IFs). 81
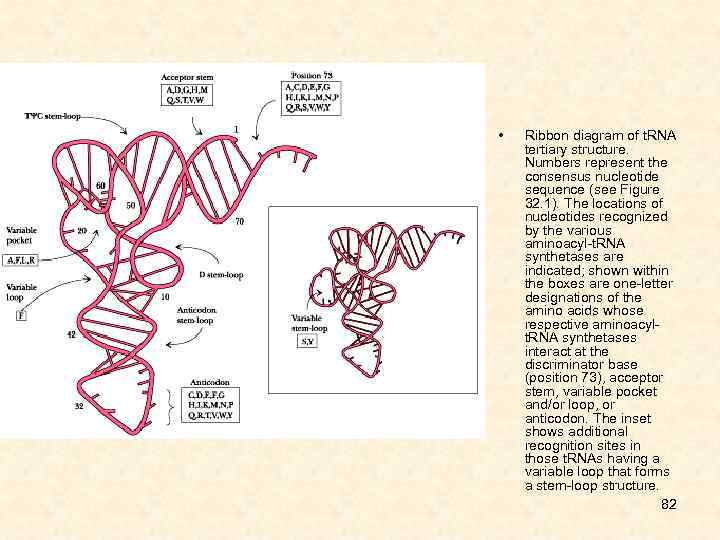
• Ribbon diagram of t. RNA tertiary structure. Numbers represent the consensus nucleotide sequence (see Figure 32. 1). The locations of nucleotides recognized by the various aminoacyl-t. RNA synthetases are indicated; shown within the boxes are one-letter designations of the amino acids whose respective aminoacylt. RNA synthetases interact at the discriminator base (position 73), acceptor stem, variable pocket and/or loop, or anticodon. The inset shows additional recognition sites in those t. RNAs having a variable loop that forms a stem-loop structure. 82
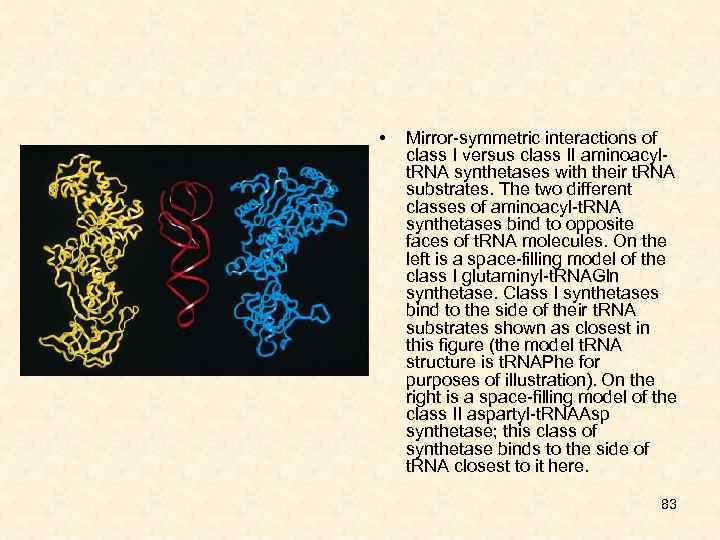
• Mirror-symmetric interactions of class I versus class II aminoacylt. RNA synthetases with their t. RNA substrates. The two different classes of aminoacyl-t. RNA synthetases bind to opposite faces of t. RNA molecules. On the left is a space-filling model of the class I glutaminyl-t. RNAGln synthetase. Class I synthetases bind to the side of their t. RNA substrates shown as closest in this figure (the model t. RNA structure is t. RNAPhe for purposes of illustration). On the right is a space-filling model of the class II aspartyl-t. RNAAsp synthetase; this class of synthetase binds to the side of t. RNA closest to it here. 83
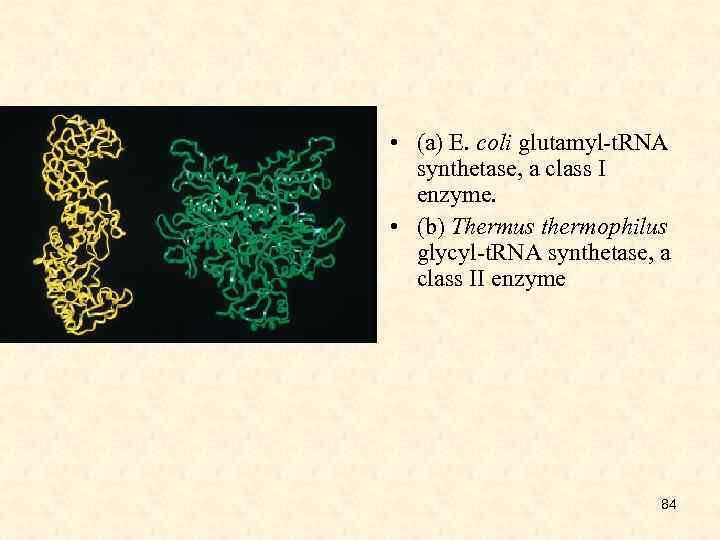
• (a) E. coli glutamyl-t. RNA synthetase, a class I enzyme. • (b) Thermus thermophilus glycyl-t. RNA synthetase, a class II enzyme 84
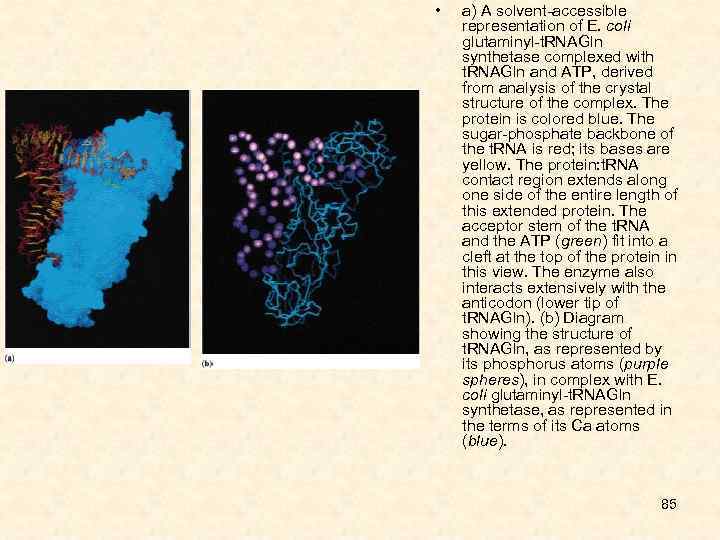
• a) A solvent-accessible representation of E. coli glutaminyl-t. RNAGln synthetase complexed with t. RNAGln and ATP, derived from analysis of the crystal structure of the complex. The protein is colored blue. The sugar-phosphate backbone of the t. RNA is red; its bases are yellow. The protein: t. RNA contact region extends along one side of the entire length of this extended protein. The acceptor stem of the t. RNA and the ATP (green) fit into a cleft at the top of the protein in this view. The enzyme also interacts extensively with the anticodon (lower tip of t. RNAGln). (b) Diagram showing the structure of t. RNAGln, as represented by its phosphorus atoms (purple spheres), in complex with E. coli glutaminyl-t. RNAGln synthetase, as represented in the terms of its Ca atoms (blue). 85
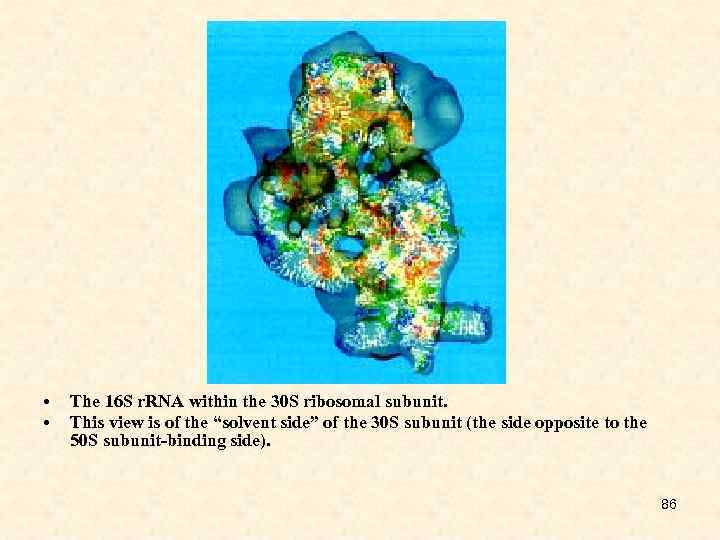
• • The 16 S r. RNA within the 30 S ribosomal subunit. This view is of the “solvent side” of the 30 S subunit (the side opposite to the 50 S subunit-binding side). 86
7-СИНТЕЗ БЕЛКА.ppt