155e7ce7d2b7e86166bef3715765fd53.ppt
- Количество слайдов: 136

BIOLOGIA/MEDICINA A Revolução da Genômica/Bioinformática

A Verdadeira Revolução Início do séc. 20: Mendel e as leis da hereditariedade. 1944: DNA como elemento carreador da informação genética (Avery) 1953: Watson/Crick e aestrutura do DNA. Anos 70 e 80: Biologia Molecular/Biotecnologia Anos 90 e séc. 21: Genômica/Bioinformática

História da Biologia Molecular n n n 1951 1953 1957 1960’s 1967 1972 1973 1977 1978 1980 1983 Fred Sanger, Amino Acid Sequence of Insulin Watson/Crick, Estrutura do DNA Francis Crick, Central Dogma, DNA RNA Protein Nirenberg, Matthaei, The Genetic Code Shapiro and Beckwith, First gene cloned, Lac. Z Paul Berg, First recombinant DNA molecule Cohen/Boyer, First recombinant organism Maxam/Gilbert and Fred Sanger, DNA sequencing Fred Sanger, Complete sequence of phage X 174 David Botstein, Restriction Fragment Length Polymorphisms (RFLP) Kerry Mullis, PCR Lee Hood, First Automated DNA Sequencer

Sequenciamento do DNA Sanger, Gilbert (Nobel 1980)

Sequenciamento Automático Leroy Hood 30 kb por corrida

A era genômica

376 Genomas Concluídos! 251 como rascunho! 342 em andamento 327 Bacterial, 27 Archeal, 22 Eukaryotic n n n 1995, Haemophilus influenzae 1996, Methanococcus jannaschii 1997, Saccharomyces cerevisiae 1997, Escherichia coli 1998, Caenorhabditis elegans 2000, Drosophila melanogaster 2000, Arabidopsis thaliana 2001, Homo sapiens 2002, Schizosaccharomyces pombe 2002, Oryza sativa 2002, Mus musculus 2005, Pan troglodites http: //www. ncbi. nlm. nih. gov 09/07/2006

Um modelo genético

Genoma Humano 2001 n International Consortium n n Grupos acadêmicos Celera Genomics n Companhia Privada

A nova revolução da Genômica 454 ~120 MB de DNA por corrida 12 KB/US$ Solexa - Illumina ~01 GB de DNA por corrida 100 KB/US$ Tecnologia de Capilar = 0. 5 KB/US$ SOLi. D - ABI ~03 GB de DNA por corrida 300 KB/US$

Nova tecnologia Dispensa clonagem dos fragmentos em sistemas bacterianos n Dispensa a preparação de DNA molde para sequenciamento n Reações feitas em paralelo em volume extremamente pequeno - nanotecnologia n

Aplicações n Sequenciamento de Genomas ¨ sequenciamento de novo ¨ re-sequenciamento - variabilidade SNPs e mutações n Sequenciamento de Transcriptomas ¨ variabilidade - splicing, poliadenilação ¨ quantificação de expressão gênica

Sequenciamento de novo

Re-sequenciamento

Transcriptoma

Projeto 454 Participantes: LICR-SP, LICR-NY, Venter Institute Objetivo: Sequenciamento extensivo dos genes expressos na linhagem celular HCC 1954 (tumor de mama) buscando conhecer, com um único set de dados, alterações genéticas e epi-genéticas neste tipo de câncer. Sequenciador: 454
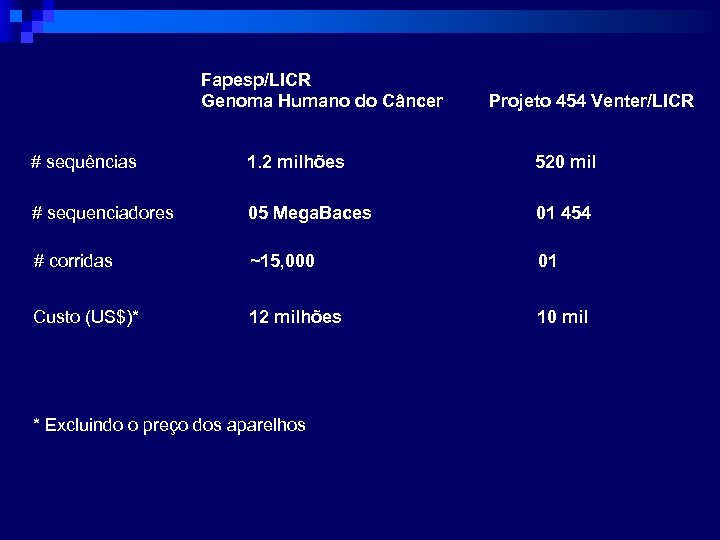
Fapesp/LICR Genoma Humano do Câncer Projeto 454 Venter/LICR # sequências 1. 2 milhões 520 mil # sequenciadores 05 Mega. Baces 01 454 # corridas ~15, 000 01 Custo (US$)* 12 milhões 10 mil * Excluindo o preço dos aparelhos
Um objetivo a curto prazo

Os sequenciadores de nova geração promovem uma mudança no paradigma Geração de dados deixa de ser o fator limitante Com os bilhões de datapoints gerados em horas, o processamento e análise dos dados tornou-se o maior gargalo das pesquisas biomédicas.

Bioinformática Computação n Matemática n Biologia n

O que é Bioinformática?
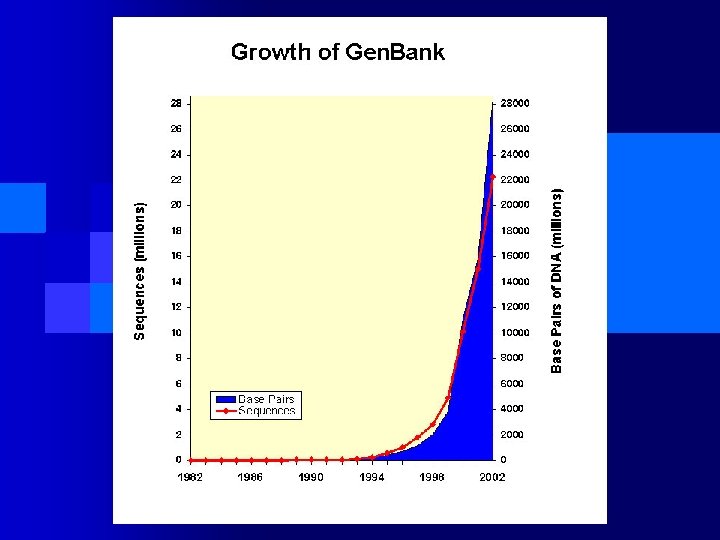


Bioinformática - História n n n n 1970, Needleman/Wunch, Alinhamento Global. 1972, Margaret Dayhoff, Matrizes de Comparação. 1979, Walter Goad, Gen. Bank. 1981, Smith/Waterman, Alinhamento Local. 1989, NHGRI, Projeto Genoma Humano. 1990, Altschul/Gish/Miller/Myers/Lipman, BLAST. 1994, Eddy/Krogh/Durbin, Hidden Markov Models (HMMs).

Bioinformática - Importância n Poucas pessoas adequadamente treinadas em Biologia e Computação. n Biologia em larga-escala. Produção de dados em massa gera uma demanda para análises computacionais. n Economiza tempo e dinheiro.

Bioinformática n n Desenvolvimento de ferramentas. Forma de explorar novos dados. Processamento de dados gerados por projetos em larga-escala. Uma nova forma de se fazer ciência dirigida por hipóteses.

Bioinformática O Bioinformata - Manipula a informação. - Desenvolve ferramentas - Bancos de dados locais. - Local. - Mta programação. - Habilidades de TI. O Usuário - Recursos da Web. - Local ou remoto. - nada de programação. - pouca habilidade de TI.

Cinco websites que todos devem conhecer n NCBI (The National Center for Biotechnology Information; ¨ n EBI (The European Bioinformatics Institute) ¨ n http: //genome. ucsc. edu/ Swiss. Prot/Ex. PASy (Swiss Bioinformatics Resource) ¨ n http: //www. ebi. ac. uk/ The UCSC Genome Browser ¨ n http: //www. ncbi. nlm. nih. gov/ http: //expasy. cbr. nrc. ca/sprot/ PDB (The Protein Databank) ¨ http: //www. rcsb. org/PDB/

NCBI (http: //www. ncbi. nlm. nih. gov/) n Acesso aos bancos de dados via Entrez ¨ Medline/OMIM ¨ Genbank/Genpept/Structures n Servidor de BLAST ¨ Todos os tipos de Blast Portal do Genoma Humano n Muito, muito mais……. . n

EBI (http: //www. ebi. ac. uk/) n Acesso a bancos de dados via SRS ¨ EMBL, Swiss. Prot, …… n Muitas outras ferramentas ¨ Clustal. W, DALI, …

UCSC Genome Browser (http: //genome. ucsc. edu/) n Banco de dados e Browser para genomas de diferentes espécies ¨ Humano, camundongo, rato, zebrafish, etc…. n Muitas outras ferramentas ¨ SNPs, domínios prtéicos, genômica comparativa, etc….

Swiss. Prot (http: //www. expasy. ch/sprot/) n Checagem manual. ¨ O número de entradas errôneas é bastante reduzido. Cross-link extensivo com outros bancos n Swiss. Prot é o ‘gold-standard’ em termos de bancos de dados e é o melhor lugar para se começar uma análise se vc procura info para uma ou poucas n

Protein Data Bank – PDB (http: //www. rcsb. org/pdb/) n Armazena a estrutura tri-dimensional para milhares de proteínas n Acesso a vários serviços relacionados a biologia estrutural

Bancos de Sequência Primários Gen. Bank (USA) http: //www. ncbi. nlm. nih. gov/Genbank EMBL (Europa) http: //www. ebi. ac. uk/embl/ DDBJ (Japão) http: //www. ddbj. nig. ac. jp/

Homologia - Ortologia - Paralogia

Dois conceitos importantes n Paralogia: O evento que originou às duas sequências é um evento de duplicação gênica! FUNÇÕES SIMILARES! n Orthologia: O evento que deu origem às duas sequências é um evento de especiação! FUNÇÕES IDÊNTICAS!

Como definir função? Alinhamento de sequências n Motivos (padrões consensuais) n Blocos, perfis, etc. . n Hidden Markov Models - HMM n
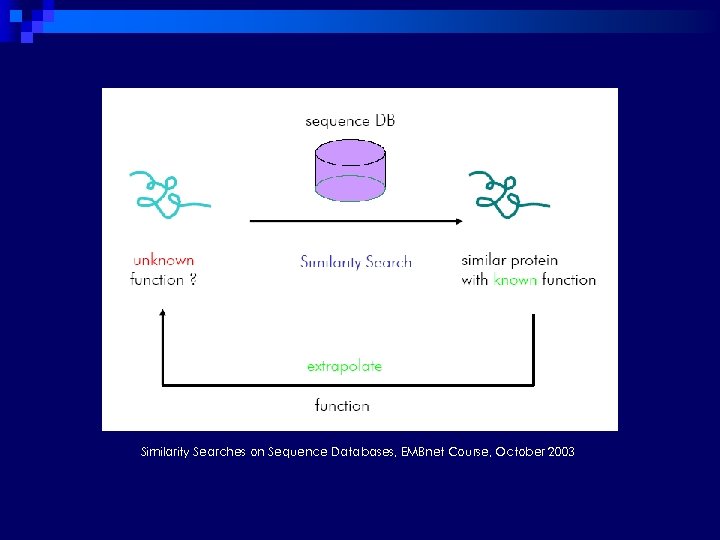
Similarity Searches on Sequence Databases, EMBnet Course, October 2003

Alinhamento 0 1 2 3 4 5 6 7 8 9 10 G A A - G G A T TA G G A T C G G A - -A G n Identidade - MATCH n Semelhança / divergência - MISMATCH n Lacunas - GAPS n Inserção/Deleção - INDELS

Alinhamento Qual é o melhor alinhamento ? Alinhamento 1: Alinhamento 2: A–C–GG–ACT | || A T C G G A T – C T ATCGGATCT | ||| -|| A–CGG–ACT

Pontuação Esquema de pontuação match: +2 mismatch: +1 indel: – 2 Alinhamento 1: (5 *2) + (1*1) + (4*-2) = 10 + 1 – 8 = 3 Alinhamento 2: (6 *2) + (1*1) + (2*-2) = 12 + 1 – 4 = 9 Escore final = soma dos escores para cada posição Favorece os matches, penaliza os gaps

Matriz de Substituição n Tabela de comparação n Reflete a probabilidade ou frequência de determinada substituição em sequências biologicamente relacionadas n p(A B) = p(B A) n Construídas pelo estudo do alinhamento de diversas sequências relacionadas n AA ou nucleotídeos

Percent Accepted Mutation (PAM - Dayhoff) n Margaret Dayhoff (1978) n Probabilidade de substituição de aa em alinhamentos globais de sequências homólogas n Cada matriz reflete as mutações entre sequências que divergiram por determinado período de tempo n Mutações aceitas => não afetam negativamente a viabilidade da proteína n Primeira matriz ¨ 71 grupos de proteínas, 85% de similaridade ¨ 1572 substitutições de aminoácidos n Expansão do número de proteínas => 1991 database

Matrizes PAM n Premissa => cada mutação é independente das mutações anteriores n Consequência => as substituições observadas em curtos períodos podem ser extrapoladas para longos períodos n PAM 1 => sequências com 1% ou menos de divergência =>1 mutação aceita a cada 100 aminoácidos n PAM N mutações = (PAM 1)N n PAM 250 => 250 mutações por 100 aa => 250% mutações em 2500 milhões anos n PAM 250: 20% similar - PAM 120: 40% - PAM 80: 50% - PAM 60: 60%

http: //www. blc. arizona. edu/courses/bioinformatics/dayhoff. html

Blocks Substitution Matrix (BLOSUM) n Kenikoff & Henikoff (1992) n Frequência de substituição de aa em um conjunto de ~2000 padrões (blocos) n Maior número de sequências consideradas => mais de 500 famílias n Alinhamentos locais de sequências relacionadas e não geradas a partir de extrapolações n BLOSUM 62 é o padrão para BLAST 2. 0 => sequências moderadamente distantes ou mais próximas n Sequências Consenso ¨ 60% idênticas: BLOSUM 60 ¨ 80% idênticas : BLOSUM 80
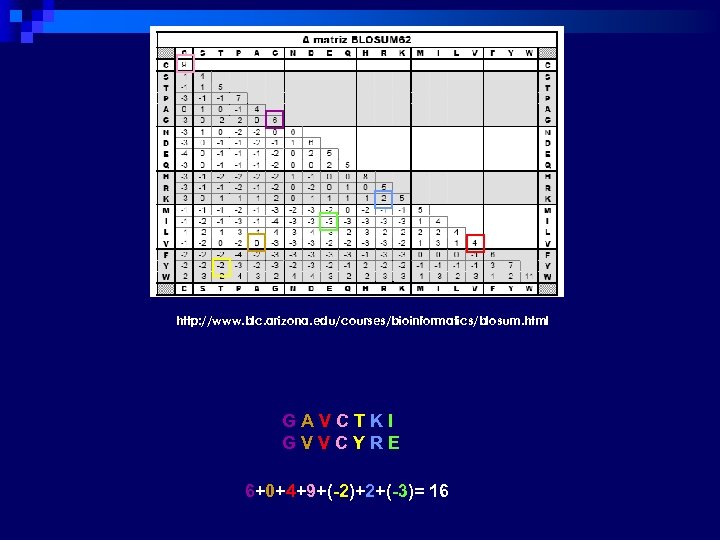
http: //www. blc. arizona. edu/courses/bioinformatics/blosum. html GAVCTKI GVVCYRE 6+0+4+9+(-2)+2+(-3)= 16

Relação ente BLOSUM e PAM http: //www. ncbi. nlm. nih. gov/Education/BLASTinfo/Scoring 2. html PAM => origens evolutivas de proteínas BLOSUM => domínios conservados

Global vs. Local Global n Alinhamento de toda a sequência utilizado o maior número de caracteres possíveis n Sequências similares e de tamanho aproximado Local n Segmentos com o maior número de identidades n Regiões alinhadas e não alinhadas (≠ mismatch) n Sequências similares em algumas regiões, que diferem em tamanho ou que compartilham domínios conservados

Aplicações Global n Deduzir histórias evolutivas entre membros da mesma família n Estabelecer a existência de um ancestral comum (homologia) Local n Inferir funções biológicas n Identificar regiões conservadas e de alta similaridade (sítio ativo, domínios) entre outras pouco conservadas n Reconstruir sequências de DNA a partir de seus fragmentos n Comparar sequências de m. RNA (sem íntrons) à sequência genômica

Métodos de Análise n Diagramas - DOT PLOT n Algoritmo de Programação Dinâmica n Algoritmos Heurísticos - Word-Based ou K-tuples

Dot Plot
Inserções & Deleções
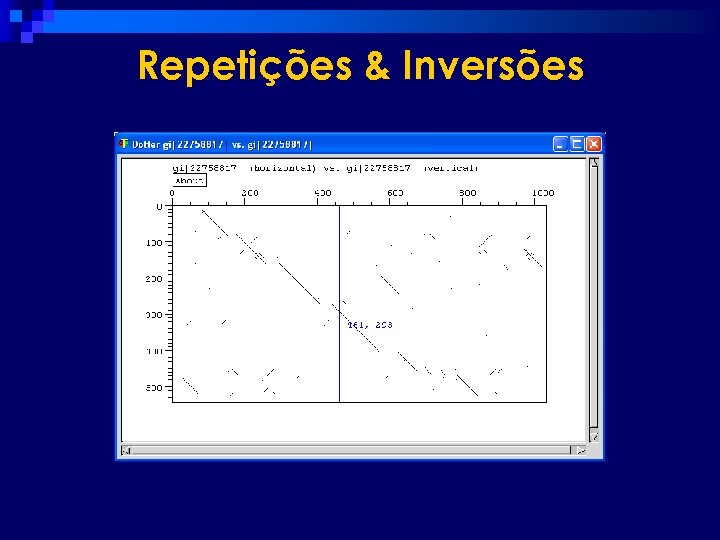
Repetições & Inversões

Programas Disponíveis n Dotter (http: //www. cgr. ki. se/cgr/groups/sonnhammer/Dotter. html) n COMPARE & DOTPLOT (Genetics Computer Group) n PLALIGN (http: //fasta. bioch. virginia. edu/fasta_list. html) n Web browser (http: //www. isrec. isb-sib. ch/java/dotlet/Dotlet. html)

Programação Dinâmica n Needleman & Wunsch (1970) n Compara cada par de caracteres nas duas sequências n Posiciona os gaps de forma a obter o maior número de alinhamentos idênticos ou similares n Gera uma matriz de números que representa todos os possíveis alinhamentos de acordo com um sistema de escore n Alinhamento ótimo => maior escore

Limitações n Computacionalmente lento n Número de alinhamentos cresce exponencialmente com a média dos comprimentos das sequências (n) n Número de cálculos => proporcional a n 2 ou n 3 n Memória => capacidade da ordem de n 2

Needleman-Wunsch As sequências abcdefghajklm abbdhijk São alinhadas e scores são dados a b c d e f g h a | | a b b d. . . h i match 4 4 mismatch -3 -3 gap_open -2 gap_extend -1 -1 -1 Score total de 24 -6 -2 -3 = 13. j | j 4 k l m | k 4

Needleman-Wunsch O alinhamento de maior score entre as duas sequências é considerado o mais provável.
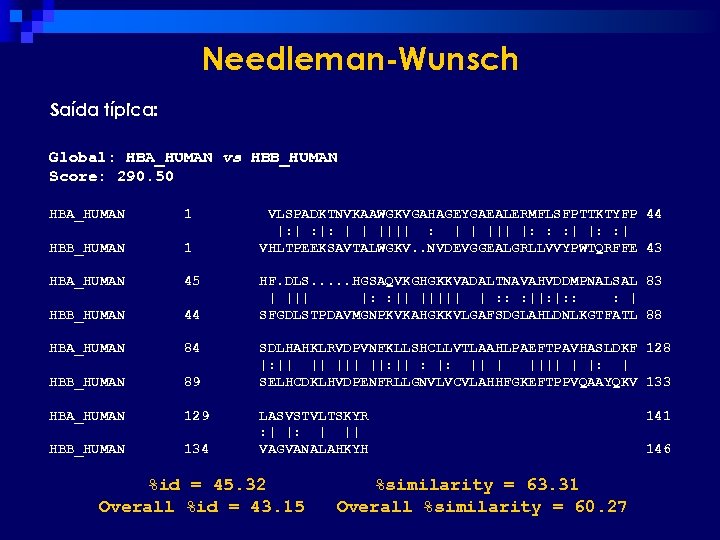
Needleman-Wunsch Saída típica: Global: HBA_HUMAN vs HBB_HUMAN Score: 290. 50 HBA_HUMAN 1 HBB_HUMAN 1 HBA_HUMAN 45 HBB_HUMAN 44 HBA_HUMAN 84 HBB_HUMAN 89 HBA_HUMAN 129 HBB_HUMAN 134 VLSPADKTNVKAAWGKVGAHAGEYGAEALERMFLSFPTTKTYFP 44 |: | : |: | | |||| : | | ||| |: : : | |: : | VHLTPEEKSAVTALWGKV. . NVDEVGGEALGRLLVVYPWTQRFFE 43 HF. DLS. . . HGSAQVKGHGKKVADALTNAVAHVDDMPNALSAL 83 | ||| |: : || ||||| | : : : ||: |: : : | SFGDLSTPDAVMGNPKVKAHGKKVLGAFSDGLAHLDNLKGTFATL 88 SDLHAHKLRVDPVNFKLLSHCLLVTLAAHLPAEFTPAVHASLDKF 128 |: || || ||: || : |: || | |||| | |: | SELHCDKLHVDPENFRLLGNVLVCVLAHHFGKEFTPPVQAAYQKV 133 LASVSTVLTSKYR : | || VAGVANALAHKYH %id = 45. 32 Overall %id = 43. 15 %similarity = 63. 31 Overall %similarity = 60. 27 141 146

Razões para se usar um banco de sequências n Eu acabei de obter uma sequência. O que é sabido à respeito desta sequência? Ela é única? n Eu tenho uma sequência única. Ela tem similaridade com alguma outra sequência de função conhecida? n Eu encontrei uma nova proteína em um determinado organismo. Existe um ortólogo conhecido? n Eu decidi trabalhar com um gene novo. Eu não tenho como obter um clone contendo a sequência deste gene. Eu preciso da sequência do c. DNA para fazer uma PCR.

O que envolve uma busca ? n Algoritmos de busca (BLAST, FASTA) n Matrizes de comparação (PAM vs. BLOSUM) n Banco de dados (nr, db. EST) n Parâmetros de busca (filtros on/off, threshold, etc…)

Basic Local Alignment Search Tool n Método heurístico => método empírico, que utiliza a fórmula ´tentativa e erro´ para encontrar as soluções n Significado estatístico => determina se um alinhamento ocorre aleatoriamente ou não n Vantagem => pelo menos 50 vezes mais rápido que os algoritmos de programação dinâmica e mais apropriados para busca em bancos de dados n Desvantagem => não garante uma solução com um alinhamento ótimo como os algoritmos de programação dinâmica

Aplicações n Identificar sequências ortólogas e parálogas n Descobrir novos genes ou proteínas n Descobrir variantes de genes e proteínas n Investigar Expressed Sequence Tags - ESTs n Explorar a estrutura e função de proteínas

BLAST WEB Pages n BLAST (NCBI – National Center for Biotechnology Information): http: //www. ncbi. nlm. nih. gov/BLAST/ n BLAST 2 (Swiss EMBnet server - European Molecular Biology network? ? ): http: //www. ebi. ac. uk/blastall/ n WU-BLAST (Washington University): http: //blast. wustl. edu/

Blast é Heurístico 1) Tabela de busca com todas as ´palavras´ (words) de comprimento W (3 aa ou 11 nucleot. ) mais as palavras vizinhas semelhantes, que aparecem pelo menos T vezes na sequência query. 2) Busca de sementes (hits, hot spots) na sequência do banco de dados que alinhem com as palavras previamente estabelecidas. 3) Extensão das sementes em ambas as direções, produzindo alinhamentos locais máximos (HSP - high scoring pair) com ou sem lacunas, de acordo com os parâmetros estabelecidos. 4) Registro da informação em um arquivo Seq. Align (ASN. 1). 5) A informação é utilizada para buscar sequências similares. Os resultados podem ser reformatados sem a necessidade de refazer a busca.

Sensibilidade vs. Seletividade Sensibilidade Habilidade de encontrar a maior parte dos membros relacionados à família da sequência query Seletividade Habilidade de não identificar sequências de outras famílias como falsopositivos “Grau de cobertura dos membros da família dado um nível de falso-positivos”

Escores e Estatística Bit Score n Indica quão bom é o alinhamento. Quanto maior o escore, melhor o alinhamento n Considera o número de resíduos idênticos ou similares e a quantidade de gaps n Influenciado pela Matriz de Substituição (padrão: BLOSUM 62 ) n Exceção: blastn and Mega. BLAST n Normalização: bit scores de diferentes alinhamentos podem ser comparados

Escores e Estatística E-value n Significado estatístico do alinhamento n Quanto menor o escore, mais significativo é o alinhamento n E-value = 0. 05. Significa que existem 5 chances em 100 (1 em 20) da similaridade entre as sequências ocorrer aleatoriamente n Influenciado pelo tamanho do banco de dados e o sistema de escore utilizado

Etapas de Busca 1) Selecionar a sequência (query) 2) Selecionar o banco de dados 3) Selecionar o programa 4) Definir os parâmetros
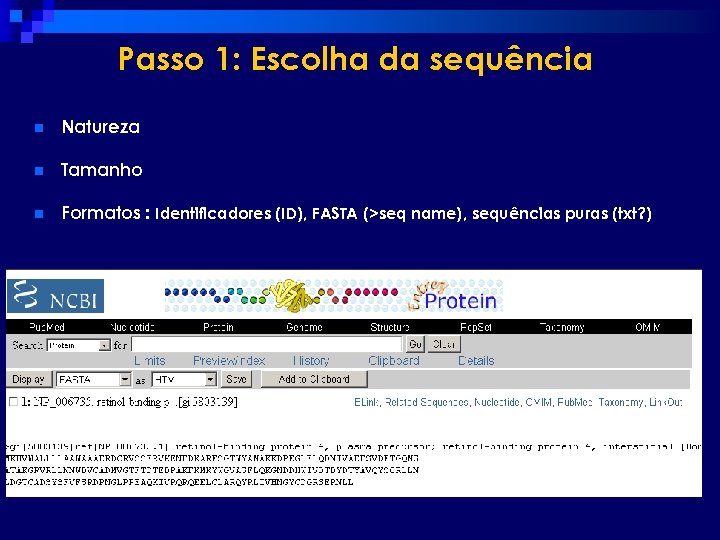
Passo 1: Escolha da sequência n Natureza n Tamanho n Formatos : Identificadores (ID), FASTA (>seq name), sequências puras (txt? )
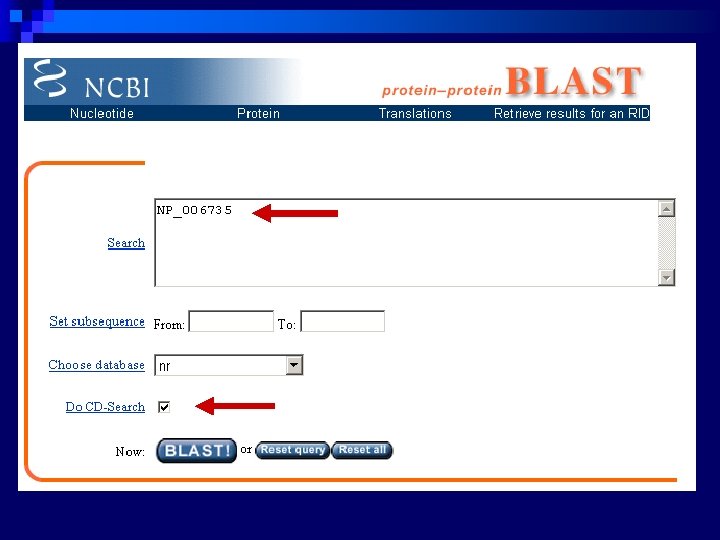


Passo 2: Seleção do Banco de Dados Proteínas Gen. Bank, PDB, SWISSPROT, PIR, REPBASE 68 e BDGP Nucleotídeos Gen. Bank, EMBL, DDBJ, PDB, REPBASE, BDGP, EST 69, STS 70, vetores, sequências de mitocôndrias, GSS 71, sequências HTGS 72 Conteúdo n não-redundância, n periodicidade de atualização n organismos ou espécies n sequências patenteadas n interesse imunológico n elementos repetitivos, etc.
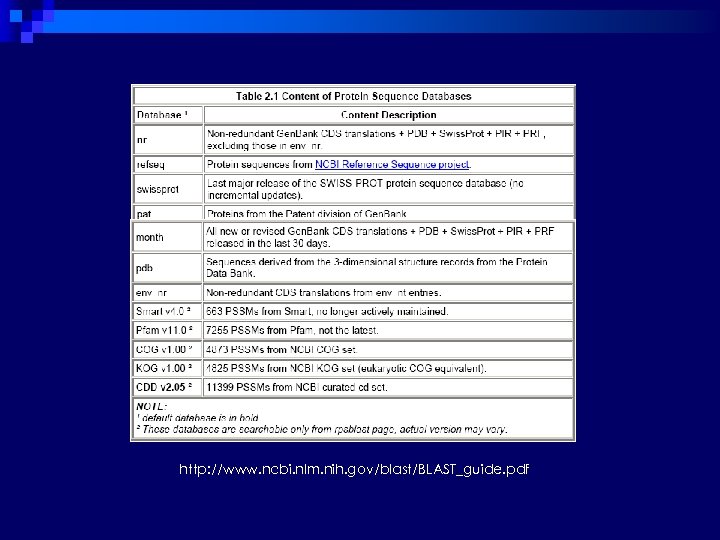
http: //www. ncbi. nlm. nih. gov/blast/BLAST_guide. pdf
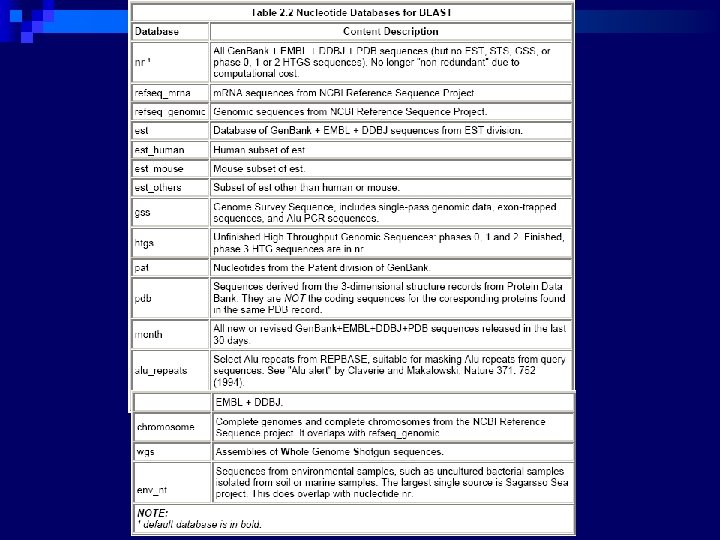

Passo 3: Seleção do Programa n Natureza da sequência n Finalidade da busca n Banco de dados

DNA codifica 6 proteínas potenciais 5’ CAT CAA 5’ ATC AAC 5’ TCA ACT 5’ CATCAACTACAACTCCAAAGACACCCTTACACATCAACAAACCTACCCAC 3’ 3’ GTAGTTGATGTTGAGGTTTCTGTGGGAATGTGTAGTTGTTTGGATGGGTG 5’ 5’ GTG GGT 5’ TGG GTA 5’ GGG TAG

Tipos de Programas programa entrada blastn DNA blastp protein blastx DNA tblastn protein tblastx DNA banco de dados 1 1 6 6 36 DNA protein DNA

Passo 4: Seleção dos parâmetros n n Tamanho da palavra (w-er) Filtros E value Matriz de substituição & penalidades para gap Sensibilidade e velocidade => W, T e X Seletividade => cutoff score
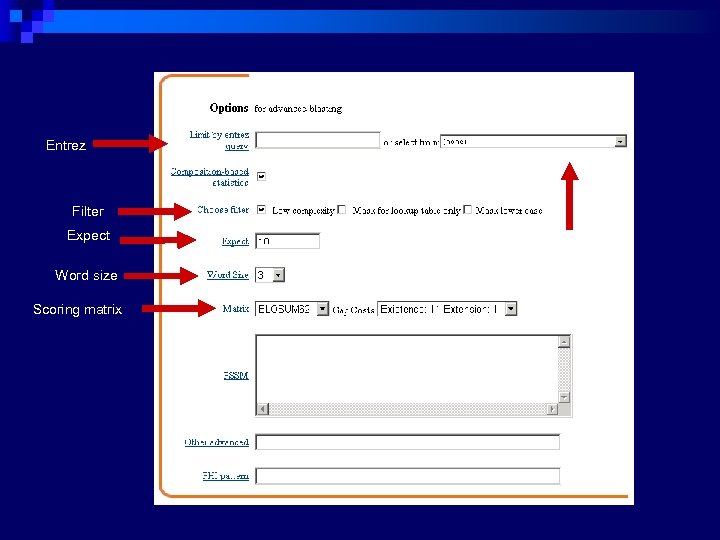
Entrez Filter Expect Word size Scoring matrix organism
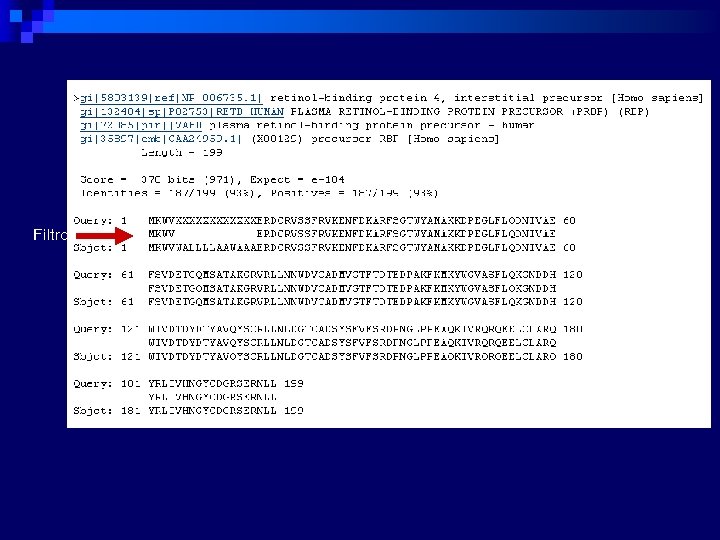
Filtro

Report Header n n n Tipo de programa (BLASTP), versão (2. 2. 1) e data da versão Artigo que descreve o BLAST, request ID (issued by QBLAST), a definição da sequência e resumo do banco de dados Taxonomy reports: mostra o resultado deste BLAST na base de informação do banco de dados Taxonomy
Taxonomy Report

Graphical Overview query database hits n n Quanto mais próximas da query, mais semelhantes Barras em rosa: lower-scoring matches que alinham em 2 regiões (resíduos 3 -60 e 220500) Segmento rachurado: as duas regiões de similaridade estão na mesma proteína mas esta região não alinha Outras barras: lower-scoring matches
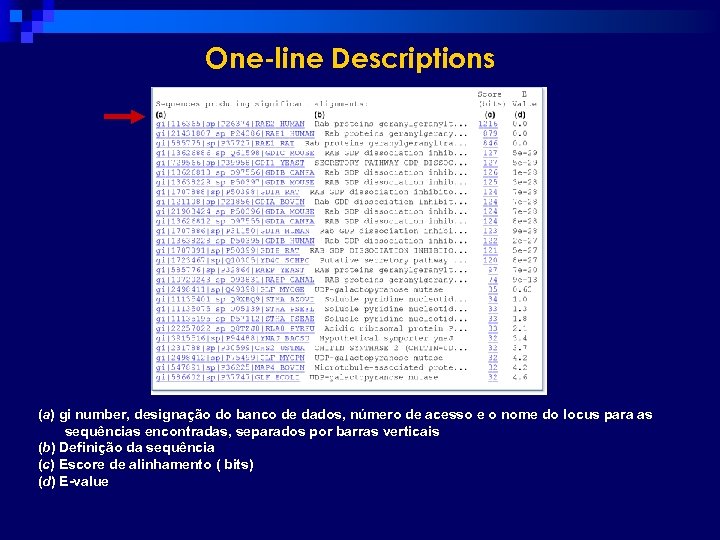
One-line Descriptions (a) gi number, designação do banco de dados, número de acesso e o nome do locus para as sequências encontradas, separados por barras verticais (b) Definição da sequência (c) Escore de alinhamento ( bits) (d) E-value

Pairwise Sequence Alignment


Famílias de Elementos Repetitivos Alu n L 1 n L 2 n Tais sequências podem gerar alinhamentos espúrios. n

Alu Constitutes about 5% of the human genome. n Short interspersed repeats. n Found in primate genomes. n ALU elements often found in 3’ regions or introns. n

Blast usando uma sequência de Alu


Como identificar e remover elementos repetitivos Filter para elementos repetitivos no servidor de Blast do NCBI n Repeat Masker: http: //ftp. genome. washington. edu/cgibin/Repeat. Masker n
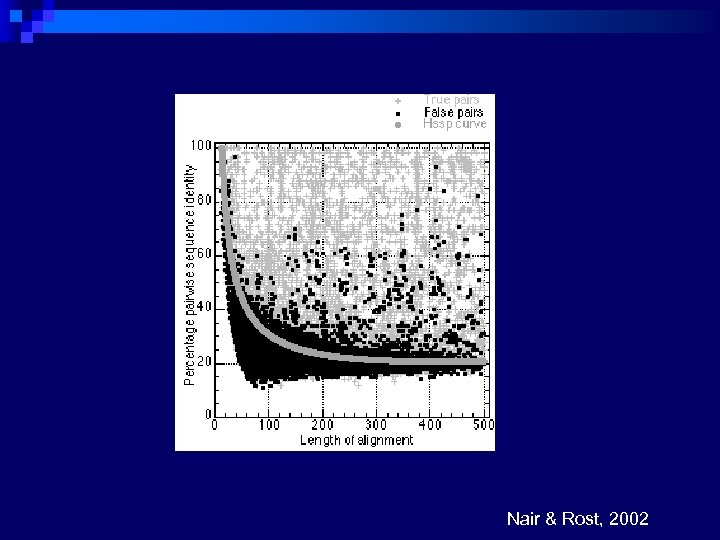
Nair & Rost, 2002

Way out! - psi-Blast - pattern (phi-Blast) - Hidden Markov Models (HMMs)

Position Specific Interactive (PSI)BLAST n Detecta proteínas fracamente relacionadas ou novos membros de uma família protéica (mais sensível) n Utilizado quando o BLAST padrão falha em encontrar hits significativos ou retorna hits com decrições do tipo "hypothetical protein" ou "similar to. . . " n Busca iterativa => comparam-se as sequências de alto escore com a sequência de busca para determinar quais delas são altamente conservadas n Sequências resultantes => construção de um modelo de escore específico por posição (consenso) => Position-Specific Scoring Matrix (PSSM ou profile)

PSI-BLAST - Algoritmo n Busca com BLASTp normal n Construção de um consenso a partir das regiões alinhadas com E values menores que o limite estabelecido (padrão = 0. 005) n Utilizando este consenso, procede a uma nova pesquisa sobre a base de dados n Quaisquer novos hits abaixo do limite são incluídos em um novo PSSM n Fim do processo (convergência) : nenhuma nova sequência é adicionada ao consenso em iterações subsequentes

Pattern-Hit Initiated (PHI)-BLAST n Busca proteínas que contém padrão especificado pelo usuário E é similar à sequência query em relação in the vicinity ao padrão n Reduz o número de hits que contém o padrão no banco de dados mas pode também apresentar nenhuma homologia ao query n Exemplo de sequência query e um padrão no formato Pro. Site: >gi|4758958|ref|NP_004148. 1| Human c. AMP-dependent protein kinase MSHIQIPPGLTELLQGYTVEVLRQQPPDLVEFAVEYFTRLREARAPASVLPAATPRQSLGHPPPEPGPDR VADAKGDSESEEDEDLEVPVPSRFNRRVSVCAETYNPDEEEEDTDPRVIHPKTDEQRCRLQEACKDILLF KNLDQEQLSQVLDAMFERIVKADEHVIDQGDDGDNFYVIERGTYDILVTKDNQTRSVGQYDNRGSFGELA LMYNTPRAATIVATSEGSLWGLDRVTFRRIIVKNNAKKRKMFESFIESVPLLKSLEVSERMKIVDVIGEK IYKDGERIITQGEKADSFYIIESGEVSILIRSRTKSNKDGGNQEVEIARCHKGQYFGELALVTNKPRAAS AYAVGDVKCLVMDVQAFERLLGPCMDIMKRNISHYEEQLVKMFGSSVDLGNLGQ n Padrão encontrado: [LIVMF]-G-E-x-[GAS]-[LIVM]-x(5, 11)-R-[STAQ]-A-x-[LIVMA]-x-[STACV]

Hidden Markov Models n n An approach based on statistical sampling theory Previously used with success for natural language processing Model sequence as a Markov model that is not known (hidden) Observed sequence is a noisy representation of the hidden “true” model
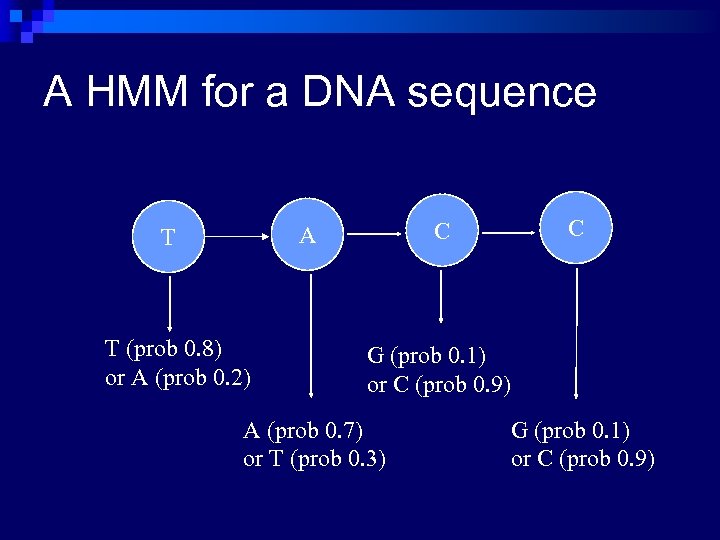
A HMM for a DNA sequence T (prob 0. 8) or A (prob 0. 2) C C A T G (prob 0. 1) or C (prob 0. 9) A (prob 0. 7) or T (prob 0. 3) G (prob 0. 1) or C (prob 0. 9)

Idea of HMM Since multiple alignment of k sequences take O(Nk) time, instead estimate a statistical model of the sequences n Align the multiple sequences to this model n This is equivalent to aligning the sequences to one another n

Protein Family Classification Pfam n large collection of multiple sequence alignments and hidden Markov models n covers many common protein domains and families n ¨ Over 73% of all known protein sequences have at least one match ¨ 5, 193 different protein families

Pfam n Initial multiple alignment of seeds using a program such as Clustal n Alignment hand scrutinized and adjusted

Pfam Links to the Pfam software: n n http: //pfam. wustl. edu/ http: //www. sanger. ac. uk/Software/Pfam/index. shtml n View some examples: ¨ http: //pfam. wustl. edu/

Locating ORFs n Simplest method of predicting coding regions is to search for open reading frames (ORFs) n open reading frames begin with a start (AUG) codon, and ends with one of three stop codons n Six total reading frames

Locating ORFs n Prokaryotes: DNA sequences coding for proteins generally transcribed into m. RNA which is translated into protein with very little modification n Locating an open reading frame from a start codon to a stop codon can give a strong suggestion into protein coding regions n Longer ORFs are more likely to predict protein-coding regions than shorter ORFs.

Locating ORFs n Eukaryotes: m. RNA undergoes processing to remove introns before the protein is translated n ORF corresponding to a gene may contain regions with stop codons found within intronic regions n Posttranscriptional modification makes gene prediction more difficult

Filogenia n Problema de determinação de árvores filogenéticas ¨ Encontrar a árvore que melhor descreve a relação entre um conjunto de objetos (espécies ou táxons) Cenoura Baleia Chimpanzé Humano

Filogenia n Táxons e Complexidade ¨ 3 árvores possíveis para 4 táxons

Filogenia n Táxons e Explosão Combinatorial

Métodos para reconstrução filogenética 03 métodos principais: : ¨ Parsimônia ¨ Métodos baseados em distância ¨ Verossimilhança máxima

Parsimônia n Dá preferência à topologia que requer o menor número de mudanças.

Filogenia n Métodos de Distância ¨ A distância evolutiva é calculada para todos os pares de táxons ¨ Matriz de Distâncias ¨ A árvore filogenética é construída considerando a relação entre esses valores de distâncias

O método de evolução mínima n Para todas as topologias possíveis : n n Calcula o comprimento de todos os ramos, S Mantém a árvore com menos S. Problema: computacionalmente intenso. Não é usado com mais de 25 sequências.

Filogenia n Métodos com Critério de Ótimo ¨ Máxima Verossimilhança n Determina-se a probabilidade de um modelo evolutivo gerar um certo dado ¨ Considera todos os sítios e todas as possibilidades de mutações em todos os nós internos da árvore proposta n Multiplica-se a probabilidade de cada sítio ¨ Probabilidade da árvore n Pode ser utilizado para análises de características e de valores n Mais consistente e com estimativas com menor variância n Não é simples e intuitivo n Computacionalmente intenso

Bootstrap procedure O suporte para cada ramo interno é expresso em termos the % de réplicas.
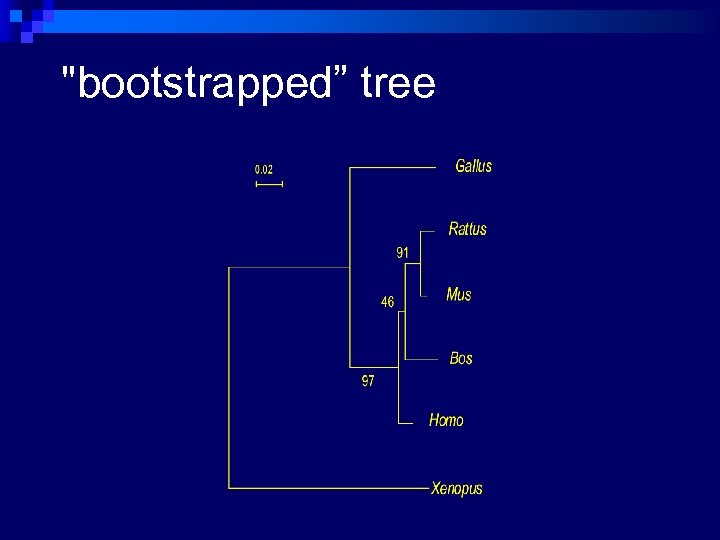
"bootstrapped” tree

Bootstrap Ramos internos suportados por ≥ 90% das réplicas são considerados estatisticamente significativos. n O procedimento de bootstrap não define se um programa é bom. Uma árvore errada pode ter 100% de suporte de bootstrap em seus ramos internos. n

Tempo de processamento para vários programas distance < parsimony ~ PHYML << Bayesian < classical ML NJ DNAPARS PHYML Mr. Bayes fast. DNAml, PAUP

Recursos de Web para filogenia n Compilações ð Uma lista de web sites http: //www. ucmp. berkeley. edu/subway/phylogen. htm l ð Uma lista grande de programas http: //evolution. genetics. washington. edu/ phylip/software. html

Recursos de Web para filogenia n Editor de alinhamento ð SEAVIEW : para windows e unix http: //pbil. univ-lyon 1. fr/software/seaview. html n Programas para filogenia molecular ð PHYLIP : http: //evolution. genetics. washington. edu/phylip. html ð PAUP : http: //paup. csit. fsu. edu/index. html ð PHYLO_WIN : http: //pbil. univ-lyon 1. fr/software/phylowin. html ð Mr. Bayes : ð http: //morphbank. ebc. uu. se/mrbayes/ ð PHYML : http: //www. lirmm. fr/~guindon/phyml. htm

Recursos de Web para filogenia n Desenho de árvores NJPLOT (para todas as plataformas) http: //pbil. univ-lyon 1. fr/software/njplot. html n Aulas de filogenia http: //www. bioinf. org/molsys/lectures. html

sandro@ludwig. org. br

Ontologia Fornecer um vocabulário estruturado e controlado para representar o conhecimento biológico nos bancos de dados. for the

Gene Onthology (GO) n Biological Process ¨ Objetivo n Molecular Function ¨ Função n dentro da célula, tecido… básica ou tarefa Cellular Component ¨Compartimento ou complexo
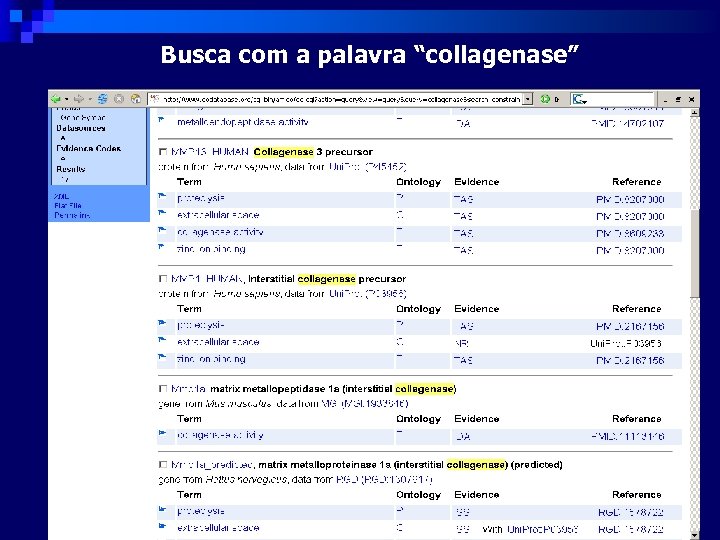
Busca com a palavra “collagenase”

Conteúdo do GO • molecular function • biological process • cellular component 7422 termos 8972 termos 1472 termos • all 17, 866 terms

155e7ce7d2b7e86166bef3715765fd53.ppt