Lesson3_with_comments.ppt
- Количество слайдов: 17

Биофак МГУ, кафедра биофизики Факультатив по программированию на языке Python для биологов Занятие 3 Основы объектноориентированного программирования в Python: работа с классами Преподаватель: Филипп Орехов
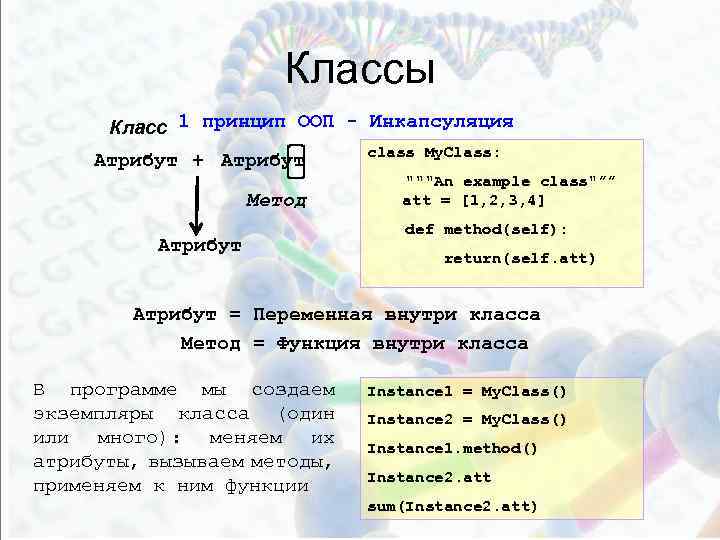
Классы Класс 1 принцип ООП - Инкапсуляция Атрибут + Атрибут Метод Атрибут class My. Class: """An example class"”” att = [1, 2, 3, 4] def method(self): return(self. att) Атрибут = Переменная внутри класса Метод = Функция внутри класса В программе мы создаем экземпляры класса (один или много): меняем их атрибуты, вызываем методы, применяем к ним функции Instance 1 = My. Class() Instance 2 = My. Class() Instance 1. method() Instance 2. att sum(Instance 2. att)

Классы 2 принцип ООП - Наследование class My. Class: att=12345 … class Derived. Class (My. Class): … >> My. Class. att 12345 >> Derived. Class. att 12345 3 принцип ООП – Полиморфизм Методы дочерних классов можно менять

Плюсы ООП С помощью объектного подхода: - экономим время и силы (не нужно писать один и тот же код много раз) - добиваемся удобной и продуманной иерархичности данных и функций в программе
Структуры биомолекул RCSB Protein Data Bank - RCSB PDB http: //www. rcsb. org/
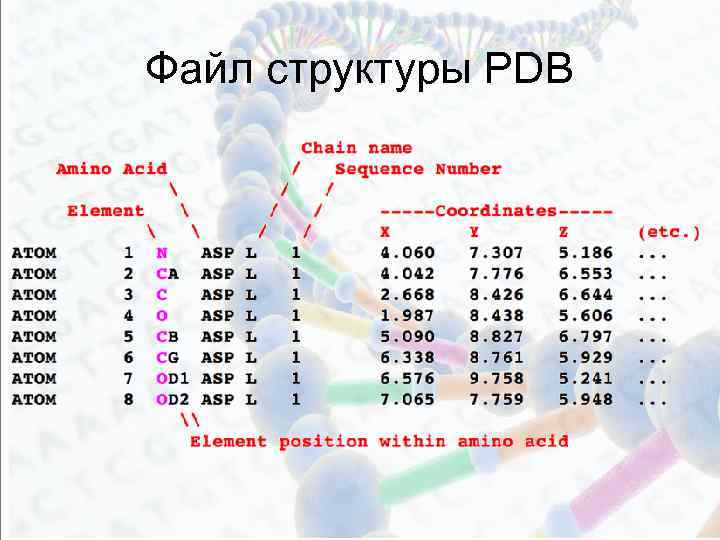
Файл структуры PDB
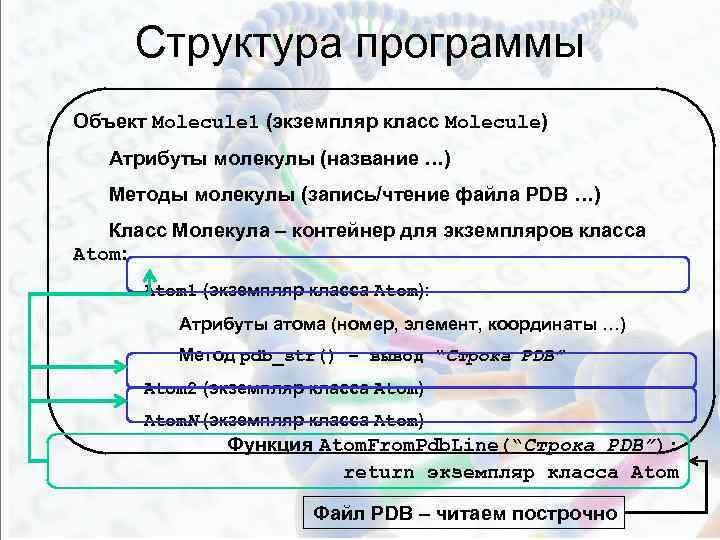
Структура программы Объект Molecule 1 (экземпляр класс Molecule) Атрибуты молекулы (название …) Методы молекулы (запись/чтение файла PDB …) Класс Молекула – контейнер для экземпляров класса Atom: Atom 1 (экземпляр класса Atom): Атрибуты атома (номер, элемент, координаты …) Метод pdb_str() – вывод “Строка PDB” Atom 2 (экземпляр класса Atom) Atom. N (экземпляр класса Atom) Функция Atom. From. Pdb. Line(“Строка PDB”): return экземпляр класса Atom Файл PDB – читаем построчно

Пример PDB В группе В Контакте: Файлы test. pdb и test_shuffled. pdb Листинг занятия - файл class_pdb. py

Создаем класс для атомов class Atom: # имя класса """Class for atoms””” # xyz = [] экземпляров, # описание класса # эта переменная была бы общая для всех def __init__(self): # а здесь перечисляются переменные, создаваемые при создании каждого нового экземпляра класса Atom # self – обращение к самому экземпляру self. xyz = [] # список с координатами атома self. type = ”CA" # тип атома (первая буква - элемент) self. chain_id = “A" # id цепи полипептида self. res_type = ”ALA” # тип аминокислотного остатка self. res_num = 0 self. num = 0 # номер аминокислотного остатка в цепи # номер атома
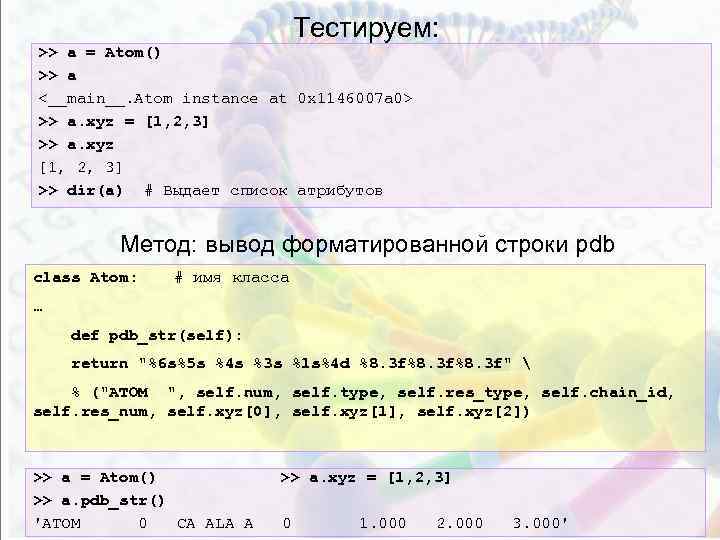
Тестируем: >> a = Atom() >> a <__main__. Atom instance at 0 x 1146007 a 0> >> a. xyz = [1, 2, 3] >> a. xyz [1, 2, 3] >> dir(a) # Выдает список атрибутов Метод: вывод форматированной строки pdb class Atom: # имя класса … def pdb_str(self): return "%6 s%5 s %4 s %3 s %1 s%4 d %8. 3 f%8. 3 f" % ("ATOM ", self. num, self. type, self. res_type, self. chain_id, self. res_num, self. xyz[0], self. xyz[1], self. xyz[2]) >> a = Atom() >> a. pdb_str() 'ATOM 0 CA ALA A >> a. xyz = [1, 2, 3] 0 1. 000 2. 000 3. 000'
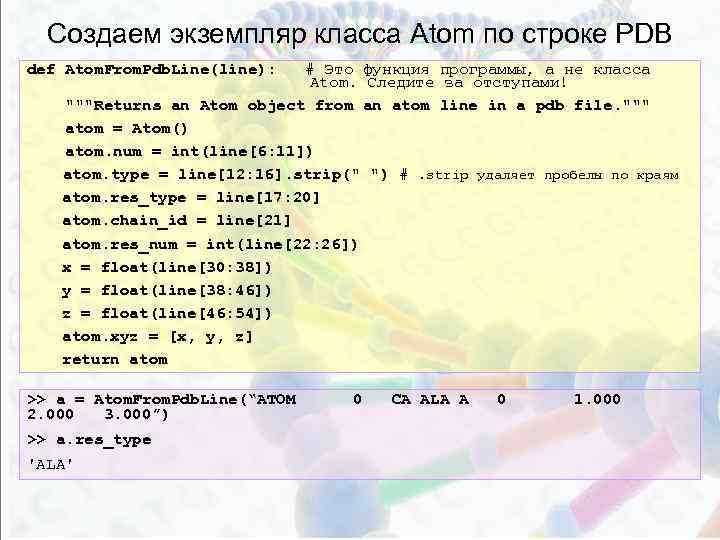
Создаем экземпляр класса Atom по строке PDB def Atom. From. Pdb. Line(line): # Это функция программы, а не класса Atom. Следите за отступами! """Returns an Atom object from an atom line in a pdb file. """ atom = Atom() atom. num = int(line[6: 11]) atom. type = line[12: 16]. strip(" ") #. strip удаляет пробелы по краям atom. res_type = line[17: 20] atom. chain_id = line[21] atom. res_num = int(line[22: 26]) x = float(line[30: 38]) y = float(line[38: 46]) z = float(line[46: 54]) atom. xyz = [x, y, z] return atom >> a = Atom. From. Pdb. Line(“ATOM 2. 000 3. 000”) >> a. res_type 'ALA' 0 CA ALA A 0 1. 000

Создаем класс для молекулы class Molecule: """Class for molecules””” # описание класса def __init__(self, pdb=""): # Функция инициирования может принимать аргументы, здесь – имя файла pdb self. id = '' # название молекулы self. _atoms = [] # в этом списке будем хранить экземпляры атомов if pdb: # если задано имя файла со структурой self. read_pdb(pdb) # то прочитать его с помощью ф-ции, которую мы напишем чуть позже Добавляем в класс полезные методы # количество атомов def n_atom(self): return len(self. _atoms) # возвращает список с атомами def atoms(self): return self. _atoms # вернуть атом с индексом i def atom(self, i): return self. _atoms[i] # удалить все атомы def clear(self): self. _atoms = [] # добавляем атом def insert_atom(self, atom): self. _atoms. append(atom)

Добавляем в класс полезные методы def erase_atom(self, atom_num): # удаляем атом с заданным номером for atom in self. _atoms: # итерируем по списку с атомами if atom. num == atom_num: # если номер атома равен заданному self. _atoms. remove(atom) # удаляем его из списка атомов del atom # и удаляем этот экземпляр класса Atom return # выходим из цикла, т. к. нашли нужный атом def Добавляем метод чтения из файла read_pdb(self, fname): # fname – аргумент с именемpdb файла pdb self. clear() # если молекула не пустая, то очистить ее for line in open(fname, 'r'). readlines(): # читаем построчно if line. startswith("ATOM"): # если первое поле - ATOM atom = Atom. From. Pdb. Line(line); # создаем экземпляр атома self. insert_atom(atom) if len(self. _atoms) == 1: # присваиваем молекуле имя цепи self. id = atom. chain_id
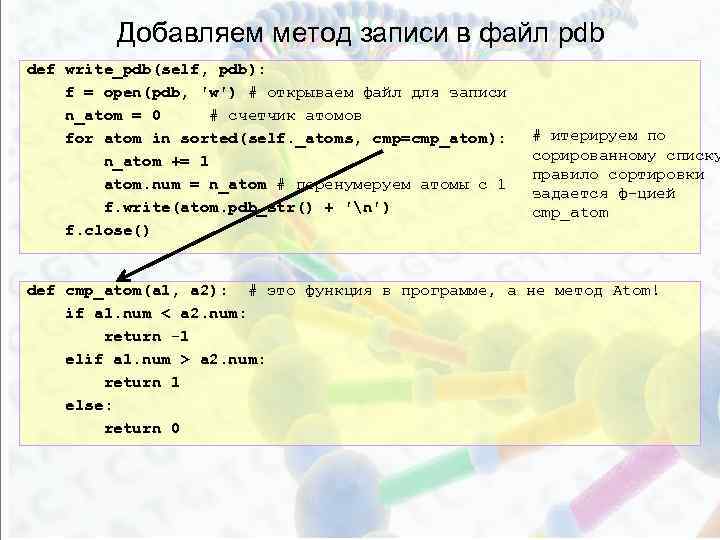
Добавляем метод записи в файл pdb def write_pdb(self, pdb): f = open(pdb, 'w') # открываем файл для записи n_atom = 0 # счетчик атомов for atom in sorted(self. _atoms, cmp=cmp_atom): n_atom += 1 atom. num = n_atom # перенумеруем атомы с 1 f. write(atom. pdb_str() + 'n') f. close() # итерируем по сорированному списку правило сортировки задается ф-цией cmp_atom def cmp_atom(a 1, a 2): # это функция в программе, а не метод Atom! if a 1. num < a 2. num: return -1 elif a 1. num > a 2. num: return 1 else: return 0

Добавляем метод расчета центра молекулы def center(self): # надо найти средние координаты x, y и z # Можно использовать цикл: for i in self. _atoms: # итерируем по списку атомов x_c += i. xyz[0] # суммируем координату x x_c = x_c / self. n_atom() # делим на количество атомов # а можно использовать List comprehensions y_c = sum([i. xyz[1] for i in self. _atoms]) / self. n_atom() # или лямбда-функцию в связке с map() z_c = sum(map(lambda i: i. xyz[2], self. _atoms)) / self. n_atom() return [x_c, y_c, z_c] Map(func, list) – применяет функцию func к каждому элементу списка list
![>> a = Molecule(“test. pdb”) >> a. _atoms[1]. xyz [76. 77, 63. 545, 49. >> a = Molecule(“test. pdb”) >> a. _atoms[1]. xyz [76. 77, 63. 545, 49.](https://present5.com/presentation/362059_335543877/image-16.jpg)
>> a = Molecule(“test. pdb”) >> a. _atoms[1]. xyz [76. 77, 63. 545, 49. 23] >> a. n_atom() 128 >> a. erase_atom(10) >> a. n_atom() 127 >> a. insert_atom(a. atom(5)) >> a_n_atom() 128 >> a. center() [73. 42757812500003, 59. 273328125000006, 48. 424867187499984] >> b = Molecule() >> b. read_pdb(“test_shuffled. pdb”) >> b. write_pdb(“test_ordered. pdb”) (In unix shell: >> diff test. pdb test_ordered. pdb)
Домашнее задание – написать класс для работы с последовательностями нуклеотидов в fasta-формате
Lesson3_with_comments.ppt