774b2e655cf7e8ee9227cc00e42bfc34.ppt
- Количество слайдов: 25
 Анализ аминокислотной последовательности: паттерны, домены, семейства … или что, где и как искать?
Анализ аминокислотной последовательности: паттерны, домены, семейства … или что, где и как искать?
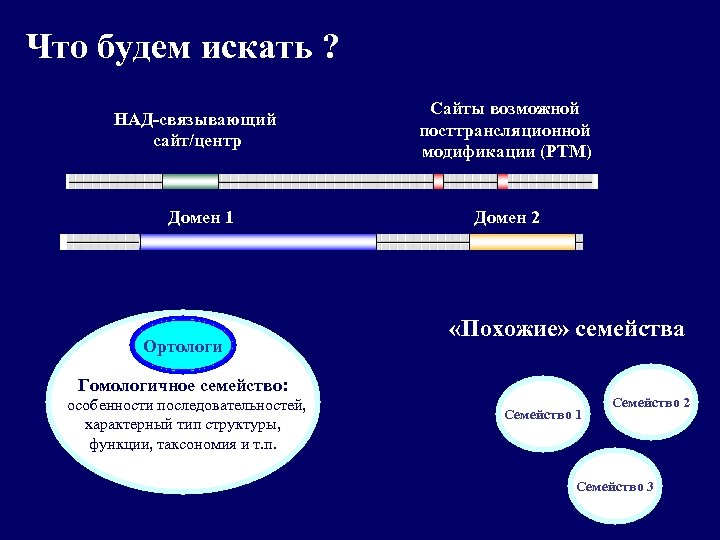 Что будем искать ? НАД-связывающий сайт/центр Домен 1 Ортологи Сайты возможной посттрансляционной модификации (РТМ) Домен 2 «Похожие» семейства Гомологичное семейство: особенности последовательностей, характерный тип структуры, функции, таксономия и т. п. Семейство 1 Семейство 2 Семейство 3
Что будем искать ? НАД-связывающий сайт/центр Домен 1 Ортологи Сайты возможной посттрансляционной модификации (РТМ) Домен 2 «Похожие» семейства Гомологичное семейство: особенности последовательностей, характерный тип структуры, функции, таксономия и т. п. Семейство 1 Семейство 2 Семейство 3
 Основные понятия и термины • Место, сайт(site) • Мотив (motif) – • Домен (domain) – • Семейство – • Суперсемейство - ? • Паттерн (pattern) – • Позиционно специфическая матрица весов (PSSM) – • Профиль–PSSM – • Профиль–HМM • Подпись (signature) – • «Oтпечатки пальцев» (fingerprints) – • Кластер -
Основные понятия и термины • Место, сайт(site) • Мотив (motif) – • Домен (domain) – • Семейство – • Суперсемейство - ? • Паттерн (pattern) – • Позиционно специфическая матрица весов (PSSM) – • Профиль–PSSM – • Профиль–HМM • Подпись (signature) – • «Oтпечатки пальцев» (fingerprints) – • Кластер -
 Домен – единица эволюции, структуры и функции белков. Домен – компактная, относительно независимо сворачивающаяся структура, относительно консервативная в процессе эволюции. Белки могут состоять из одного или многих доменов. nitrogen fixation positive activator protein
Домен – единица эволюции, структуры и функции белков. Домен – компактная, относительно независимо сворачивающаяся структура, относительно консервативная в процессе эволюции. Белки могут состоять из одного или многих доменов. nitrogen fixation positive activator protein
 Мотив ? • Мотив в аминокислотной последовательности - набор консервативных остатков, важных для функции белка и расположенных на определенном (обычно коротком) расстоянии друг от друга в последовательности. • Мотив структуры (структурный мотив) – часто встречающийся в белках элемент пространственной структуры ( -спираль, шпилька, -поворот). В общем случае, структурные мотивы не обязательно соответствуют мотивам в аминокислотным последовательностях. Один домен может содержать один или несколько мотивов в аминокислотной последовательности. Мотив может не входить в домены. Не в любом выравнивании легко найти мотив.
Мотив ? • Мотив в аминокислотной последовательности - набор консервативных остатков, важных для функции белка и расположенных на определенном (обычно коротком) расстоянии друг от друга в последовательности. • Мотив структуры (структурный мотив) – часто встречающийся в белках элемент пространственной структуры ( -спираль, шпилька, -поворот). В общем случае, структурные мотивы не обязательно соответствуют мотивам в аминокислотным последовательностях. Один домен может содержать один или несколько мотивов в аминокислотной последовательности. Мотив может не входить в домены. Не в любом выравнивании легко найти мотив.
 Интуитивно понятно: • Семейство - группа белков, имеющая общее происхождение, их аминокислотные последовательности выравниваются по всей длине со значимым весом и имеют сходную доменную структуру. Мнения расходятся, когда речь идет о критериях: • насколько должны быть похожи белки одного семейства (id>=30%, id>= 50%) ? ? ? должны белки одного семейства выполнять одну и ту же функцию? ? Superfamily Family Subfamily
Интуитивно понятно: • Семейство - группа белков, имеющая общее происхождение, их аминокислотные последовательности выравниваются по всей длине со значимым весом и имеют сходную доменную структуру. Мнения расходятся, когда речь идет о критериях: • насколько должны быть похожи белки одного семейства (id>=30%, id>= 50%) ? ? ? должны белки одного семейства выполнять одну и ту же функцию? ? Superfamily Family Subfamily
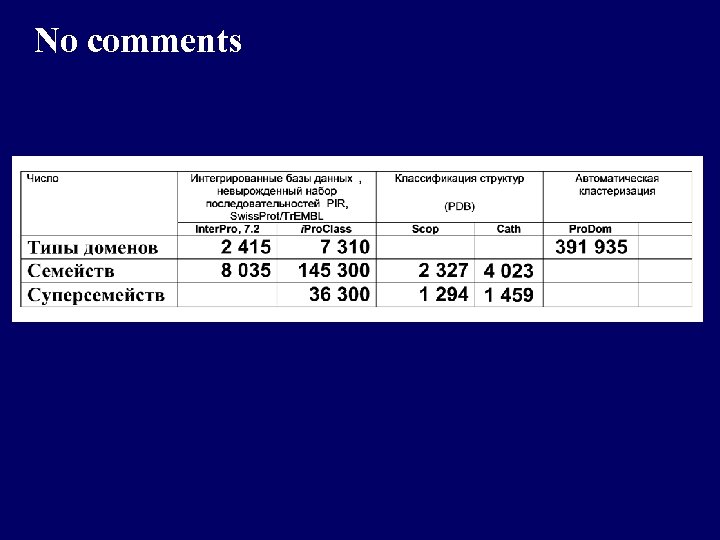 No comments
No comments
 Основные понятия и термины • Место, сайт(site) - • Паттерн (pattern) – • Позиционно специфическая матрица весов (PSSM) – • Профиль–PSSM – • Профиль–HМM • Подпись (signature) – • «Oтпечатки пальцев» (fingerprints) - • Мотив (motif) – • Домен (domain) – • Семейство – • Суперсемейство - ?
Основные понятия и термины • Место, сайт(site) - • Паттерн (pattern) – • Позиционно специфическая матрица весов (PSSM) – • Профиль–PSSM – • Профиль–HМM • Подпись (signature) – • «Oтпечатки пальцев» (fingerprints) - • Мотив (motif) – • Домен (domain) – • Семейство – • Суперсемейство - ?

 Банки белковых семейств и доменов, производные от банков аминокислотных последовательностей Коллекции мотивов Коллекции доменов PROSITE , 1989 BLOCKS PRINTS Pfam SMART Pro. Dom, 1995 SUPERFAMILY Inter. Pro, 1999 (Integrated Resource of Protein Families)
Банки белковых семейств и доменов, производные от банков аминокислотных последовательностей Коллекции мотивов Коллекции доменов PROSITE , 1989 BLOCKS PRINTS Pfam SMART Pro. Dom, 1995 SUPERFAMILY Inter. Pro, 1999 (Integrated Resource of Protein Families)
 PROSITE - биологически значимые сайты, паттерны и профили Выравнивание хорошо изученного семейства http: //www. expasy. ch/prosite/ Функционально важные остатки 4 -5 консервативных остатков Паттерн Поиск в SP Если находим только «правильные» , то ОК Если много лишнего, то увеличиваем паттерн Паттерн – регулярное выражение UNIX’a: [AC]-x-V-x(4)-{ED} Ala или Cys- х-Val- х- х- х - х- (любой, но не Glu или Asp)
PROSITE - биологически значимые сайты, паттерны и профили Выравнивание хорошо изученного семейства http: //www. expasy. ch/prosite/ Функционально важные остатки 4 -5 консервативных остатков Паттерн Поиск в SP Если находим только «правильные» , то ОК Если много лишнего, то увеличиваем паттерн Паттерн – регулярное выражение UNIX’a: [AC]-x-V-x(4)-{ED} Ala или Cys- х-Val- х- х- х - х- (любой, но не Glu или Asp)
 PROSITE - биологически значимые сайты, паттерны и профили
PROSITE - биологически значимые сайты, паттерны и профили
 F F Y F F L F K K P P K E K L A I V V F L L F V V L I L S H C G K A S G Q E A E N E A V C V PROSITE T G Q Профиль или весовая матрица A C D E F G H I K L M N P Q R S T V W Y L M L I I I L -18 -22 -35 -27 60 -30 -13 3 -26 14 3 -22 -30 -32 -18 -22 -10 0 9 34 L F L L A I V -10 -33 0 15 -30 -20 -12 -27 25 -28 -15 -6 24 5 9 -8 -10 -25 -18 V Q G K D Q C -1 -18 -32 -25 12 -28 -25 21 -25 19 10 -24 -26 -25 -22 -16 -6 22 -18 -1 Релиз 18. 25, 14. 04 2004 1257 документов, 1706 разных паттернов, правил и профилей. -8 -18 -33 -26 14 -32 -25 25 -27 27 14 -27 -28 -26 -22 -21 -7 25 -19 1 8 -22 -7 -9 -26 28 -16 -29 -6 -27 -17 1 -14 -9 -10 11 -5 -19 -25 -23 -3 -26 6 23 -29 -14 14 -23 4 -20 -10 8 -10 24 0 2 -8 -26 -27 -12 3 22 -17 -9 -15 -23 -22 -8 -15 -9 -9 -15 -22 -16 -18 -1 2 6 -34 -19 -10 -24 -34 -24 4 -33 -22 33 -27 33 25 -24 -17 -23 -24 -10 19 -20 0 -2 -19 -31 -23 12 -27 -23 19 -26 26 12 -24 -26 -23 -22 -19 -7 16 -17 0 -8 -7 0 -1 -29 -5 -10 -23 0 -21 -11 -4 -18 7 -4 -4 -11 -16 -28 -18
F F Y F F L F K K P P K E K L A I V V F L L F V V L I L S H C G K A S G Q E A E N E A V C V PROSITE T G Q Профиль или весовая матрица A C D E F G H I K L M N P Q R S T V W Y L M L I I I L -18 -22 -35 -27 60 -30 -13 3 -26 14 3 -22 -30 -32 -18 -22 -10 0 9 34 L F L L A I V -10 -33 0 15 -30 -20 -12 -27 25 -28 -15 -6 24 5 9 -8 -10 -25 -18 V Q G K D Q C -1 -18 -32 -25 12 -28 -25 21 -25 19 10 -24 -26 -25 -22 -16 -6 22 -18 -1 Релиз 18. 25, 14. 04 2004 1257 документов, 1706 разных паттернов, правил и профилей. -8 -18 -33 -26 14 -32 -25 25 -27 27 14 -27 -28 -26 -22 -21 -7 25 -19 1 8 -22 -7 -9 -26 28 -16 -29 -6 -27 -17 1 -14 -9 -10 11 -5 -19 -25 -23 -3 -26 6 23 -29 -14 14 -23 4 -20 -10 8 -10 24 0 2 -8 -26 -27 -12 3 22 -17 -9 -15 -23 -22 -8 -15 -9 -9 -15 -22 -16 -18 -1 2 6 -34 -19 -10 -24 -34 -24 4 -33 -22 33 -27 33 25 -24 -17 -23 -24 -10 19 -20 0 -2 -19 -31 -23 12 -27 -23 19 -26 26 12 -24 -26 -23 -22 -19 -7 16 -17 0 -8 -7 0 -1 -29 -5 -10 -23 0 -21 -11 -4 -18 7 -4 -4 -11 -16 -28 -18
 Pfam • http: //www. sanger. ac. uk/Software/Pfam/index. shtml • Большая коллекция множественных выравниваний, доменов, семейств и профилей-HMM для них. • Состоит из 2 -х частей: § Pfam. A – курируемая часть, покрывает 73% SWISS-Prot+Tr. EMBL § Pfam. B – большое число маленьких семейств из автоматически сгенерированной базы доменов Pro. Dom, не вошедших в Pfam. A. • Удобна для анализа доменной структуры белков.
Pfam • http: //www. sanger. ac. uk/Software/Pfam/index. shtml • Большая коллекция множественных выравниваний, доменов, семейств и профилей-HMM для них. • Состоит из 2 -х частей: § Pfam. A – курируемая часть, покрывает 73% SWISS-Prot+Tr. EMBL § Pfam. B – большое число маленьких семейств из автоматически сгенерированной базы доменов Pro. Dom, не вошедших в Pfam. A. • Удобна для анализа доменной структуры белков.
 Pfam 1. Множественное выравнивание (Clustal. X) некоторого семейства или кластера. 2. Экспертиза и корректировка выравниваниязатравки. 3. Построение профиля-НММ для затравки. 4. Поиск в базе данных а. к. последовательностей новых членов данной группы.
Pfam 1. Множественное выравнивание (Clustal. X) некоторого семейства или кластера. 2. Экспертиза и корректировка выравниваниязатравки. 3. Построение профиля-НММ для затравки. 4. Поиск в базе данных а. к. последовательностей новых членов данной группы.
 Pro. Dom • http: //www. toulouse. inra. fr/prodom. html • Рассматриваются все последовательности в SWISSProt+Tr. EMBL. • Автоматическое выделение доменов (программа DOMAINER: сначала локальное попарное выравнивание (blastp) всех против всех, затем кластеризация) • Коллекция доменов - >150 000 семейств. • Некоторые семейства выделены на основе выравниваний из Pfam. A. • Гомогенность семейства оценивается с помощью диаметра (max расстояния между 2 доменами в семействе) и радиуса (ср. кв. расстояние между доменами и консенсусом семейства). Оба параметра измеряются в РАМ
Pro. Dom • http: //www. toulouse. inra. fr/prodom. html • Рассматриваются все последовательности в SWISSProt+Tr. EMBL. • Автоматическое выделение доменов (программа DOMAINER: сначала локальное попарное выравнивание (blastp) всех против всех, затем кластеризация) • Коллекция доменов - >150 000 семейств. • Некоторые семейства выделены на основе выравниваний из Pfam. A. • Гомогенность семейства оценивается с помощью диаметра (max расстояния между 2 доменами в семействе) и радиуса (ср. кв. расстояние между доменами и консенсусом семейства). Оба параметра измеряются в РАМ
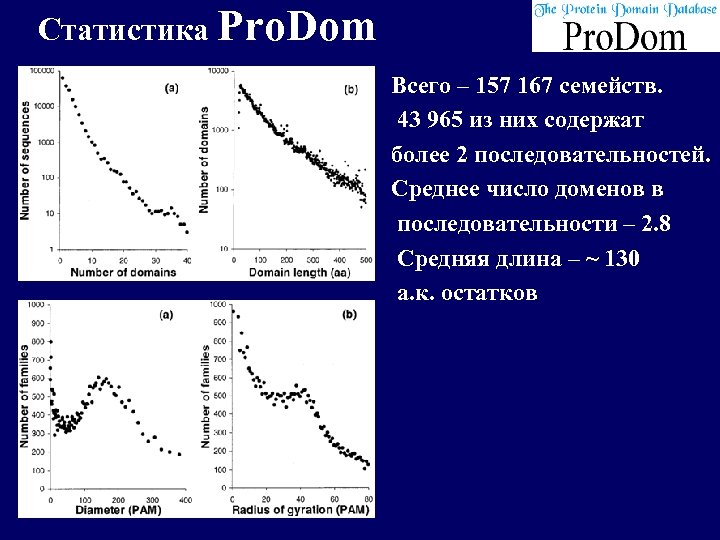 Статистика Pro. Dom Всего – 157 167 семейств. 43 965 из них содержат более 2 последовательностей. Среднее число доменов в последовательности – 2. 8 Средняя длина – ~ 130 а. к. остатков
Статистика Pro. Dom Всего – 157 167 семейств. 43 965 из них содержат более 2 последовательностей. Среднее число доменов в последовательности – 2. 8 Средняя длина – ~ 130 а. к. остатков
 Comparison of protein family databases: an example Pfam Prosite Prints Blocks Smart (Pro. Dom, PIRaln, Pro. Class, Systers, Picasso etc. not shown) Example: ENTK_HUMAN (Enteropeptidase precursor)
Comparison of protein family databases: an example Pfam Prosite Prints Blocks Smart (Pro. Dom, PIRaln, Pro. Class, Systers, Picasso etc. not shown) Example: ENTK_HUMAN (Enteropeptidase precursor)
 Создание интегрированной базы данных Inter. Pro PROSITE PFAM Интегрирование PRINTS Pro. Dom SMART TIGRFAMs PIRSF родственных подписей «вручную» Inter. Pro entries IPR 000001 IPR 011000 SUPERFAMILY Inter. Pro- an integrated resource of protein families, domains and functional sites.
Создание интегрированной базы данных Inter. Pro PROSITE PFAM Интегрирование PRINTS Pro. Dom SMART TIGRFAMs PIRSF родственных подписей «вручную» Inter. Pro entries IPR 000001 IPR 011000 SUPERFAMILY Inter. Pro- an integrated resource of protein families, domains and functional sites.
 Entry types in Inter. Pro • Family - group of evolutionarily related proteins, that share one or more domains/repeats in common. • Domain -independent structural unit which can be found alone or in conjunction with other domains or repeats. • Repeat -region occurring more than once that is not expected to fold into a globular domain on its own. • PTM (post-translational modification) -The sequence motif is defined by the molecular recognition of this region in a cell. • Active site -catalytic pockets of enzymes where the catalytic residues are known. • Binding site –binds compounds but is not necessarily involved in catalysis.
Entry types in Inter. Pro • Family - group of evolutionarily related proteins, that share one or more domains/repeats in common. • Domain -independent structural unit which can be found alone or in conjunction with other domains or repeats. • Repeat -region occurring more than once that is not expected to fold into a globular domain on its own. • PTM (post-translational modification) -The sequence motif is defined by the molecular recognition of this region in a cell. • Active site -catalytic pockets of enzymes where the catalytic residues are known. • Binding site –binds compounds but is not necessarily involved in catalysis.
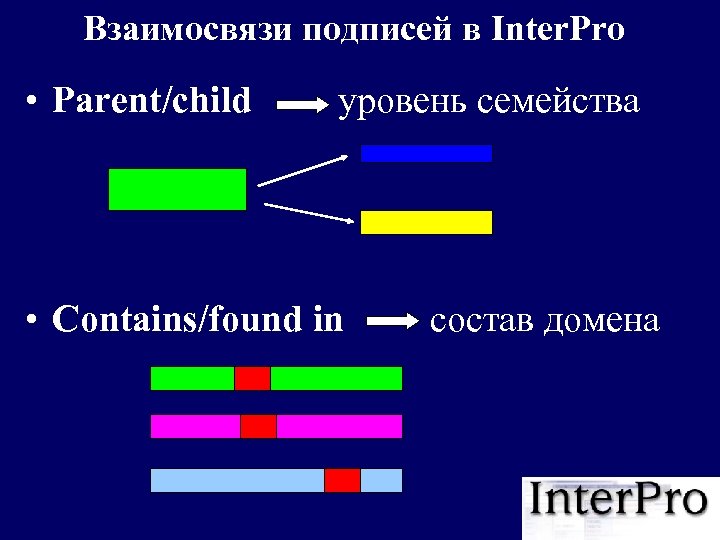 Взаимосвязи подписей в Inter. Pro • Parent/child уровень семейства • Contains/found in состав домена
Взаимосвязи подписей в Inter. Pro • Parent/child уровень семейства • Contains/found in состав домена
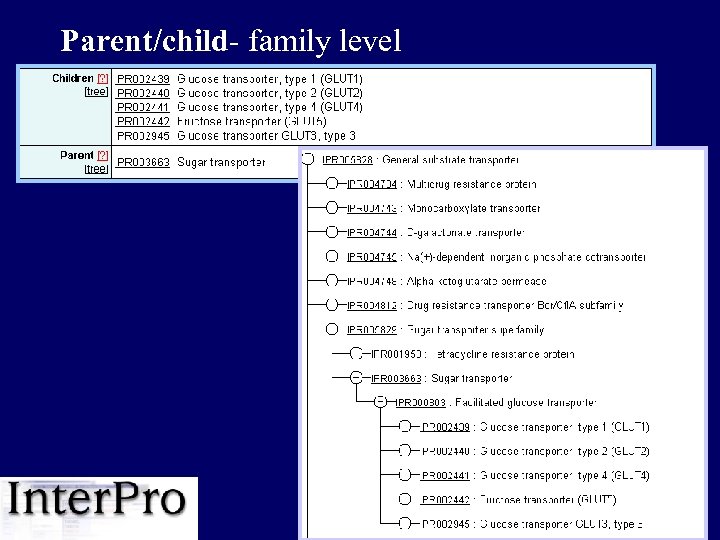 Parent/child- family level
Parent/child- family level
 Contains/found in
Contains/found in
 PROTOMAP • http: //www. protomap. cs. huji. ac. il • Automatic classification of all SWISS-PROT proteins into groups of related proteins (also including Tr. EMBL now) • Based on pairwise similarities • Has hierarchical organisation for sub- and superfamily distinctions • 13 354 clusters, 5869 2 proteins, 1403 10 • Keeps SP annotation eg description, keywords • Can search with a sequence -classify it into existing clusters
PROTOMAP • http: //www. protomap. cs. huji. ac. il • Automatic classification of all SWISS-PROT proteins into groups of related proteins (also including Tr. EMBL now) • Based on pairwise similarities • Has hierarchical organisation for sub- and superfamily distinctions • 13 354 clusters, 5869 2 proteins, 1403 10 • Keeps SP annotation eg description, keywords • Can search with a sequence -classify it into existing clusters



