d01ccedda77476fb2198bb7fa97c0013.ppt
- Количество слайдов: 60

A DNS tulajdonságai hogyan használhatók a manipulációban? Erősen negatív töltésű Lineáris információ Könnyen izolálható, elektroforézis, Összeilleszthető Kettős szálú Megolvasztható, kelálható festékekkel – érzékeny vizualizáció Komplementer szálák Információ megőrizhető, újragenerálható Kémiailag hasítható és ligálható Egy illetve két szálon darabolható, összerakható Anellálható a két szál reverzibilesen Teljes mértékben vagy részlegesen (primer) két azonos vagy hasonló szál anellálható, hibridizációk, chip Könnyen szintetizálható a másik szál alapján RNS-DNS hibridizáció Specifikus szekvenciák Transzformálható tetszőleges sejtekbe Könnyen szintetizálható kémiailag Szabályozó szekvenciák (a sejtben funkcionál) Szekvenálás, mutagenezis, PCR Könyvtárak készíthetők és könnyen szelektálható Restrikciós hasítás, poli. A (c. DNS), hibridizáció, in situ hibridizáció, expresszió specifikus szekvenciák Kihasználható a sejtek hibajavító mechanizmusai, heterológ expresszió Primer szintézis, génszintézis Indukálható heterolóh expresszió, knock-out

A hagyományos miniprep módszer I. • • • • 3 ml E. coli kultúra (médium: LB vagy 2 YT) – 16 -20 óra; 37 °C centrifugálás (2 x) 1. 5 ml-es Eppendorf csőbe – 1 -2 perc; 6000 rpm a tiszta felülúszó eltávolítása +100 l 1. (GTE) oldat → a sejtek szuszpendálása +200 l 2. oldat → lízis NAGYON ÓVATOSAN KEVERNI +150 l 3. oldat → fehérjék, genomi DNS kicsapódik – vortex centrifugálás – 10 perc, 15000 rpm felülúszó tiszta Epp. csőbe + 800 ml 95 %-os etanol → csapadék (10 perc szobahőn) centrifugálás – 10 perc, 15000 rpm – felűlúszó eltávolítva mosás + 1 ml 70 %-os etanol – centrifgálás - felűlúszó eltávolítva száritás feloldani 50 ml TE oldatban

A hagyományos miniprep módszer II. • Kitermelés – nagy kópiaszámú plazmid → 5 -10 g DNS – kis kópiaszámú plazmid → 1 -2 g DNS • A plazmid RNS-sel szennyezett!! – RNáz (DNáz mentes – hőkezelés ) hozzáadása (1 mg/ml) az 1. oldathoz vagy a TE pufferhez • a gazdasejttől függően nukleáz szennyeződés – további enzimes kezelés során aktiválódik – fenol extrakció – a 3. oldat centrifugálása utáni felülúszóhoz azonos térfogatú fenol: kloroform: izo-amilakohol 25: 24: 1 keveréket adunk – vortex – centrifugálás – a tiszta felső (vizes) fázis → tiszta Epp. csőbe – etanolos kicsapás, mosás, szárítás – FIGYELEM: a fenol veszélyes!!! bőrre jutva égési sérülés, lassan gyógyul – a reagens maradéka veszélyes hulladék – külön gyűjteni Phase lock gel
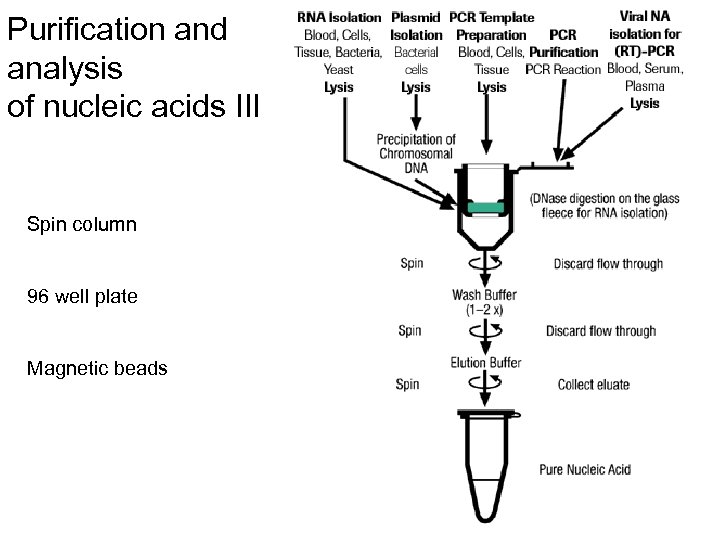
Purification and analysis of nucleic acids III Spin column 96 well plate Magnetic beads 4
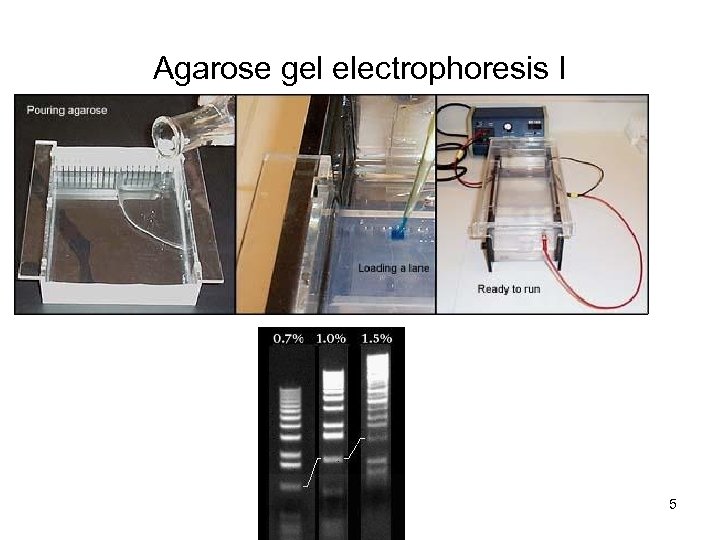
Agarose gel electrophoresis I 5

DNS fragment preparálás restrikciós fragmentek futtatása –Et. Br (SYBRSafe) rövid festés áztatással fragmentek kivágása UV boxon az agar feloldása kaotróp oldószerrel tisztítás szilika filterrel 6
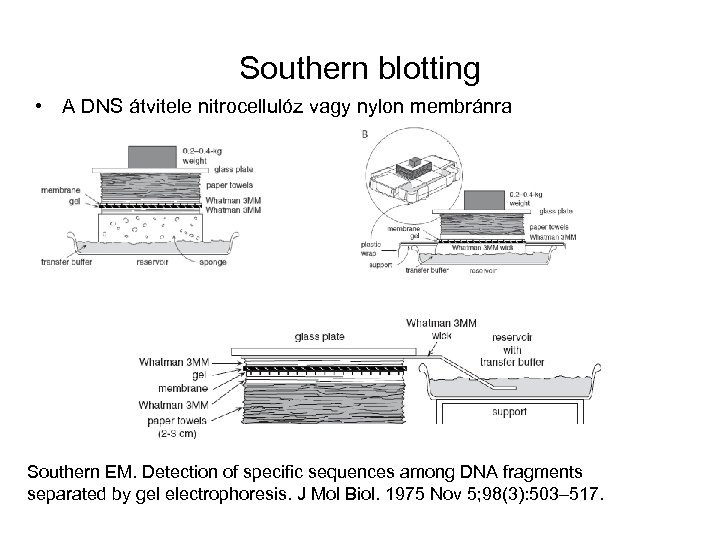
Southern blotting • A DNS átvitele nitrocellulóz vagy nylon membránra Southern EM. Detection of specific sequences among DNA fragments separated by gel electrophoresis. J Mol Biol. 1975 Nov 5; 98(3): 503– 517.

HIBRIDIZÁCIÓS TECHNIKÁK • DNS, RNS immobilizálása nitrocellulóz vagy nylon membránon • a hibridizálás szigorúsága („stringency”): hőmérséklet , • ionerősség • próba anellálási hőmérséklete: • Tm 2(A+T)+4(G+C) (n<18) • Tm=81, 5 -16, 6(lg[Na+])+0, 41(%G+C)-(600/hossz) (n>13) • SOUTHERN-BLOTTING („LENYOMATTECHNIKA”) • DNS fragmentum, inszert azonosítás, RFLP • NORTHERN- BLOTTING • m. RNS méret meghatározás, szemi-kvantitálás • KOLÓNIA- ÉS PLAKK-HIBRIDIZÁCIÓ • klónkeresés („szkrínelés”) génkönyvtárban 8
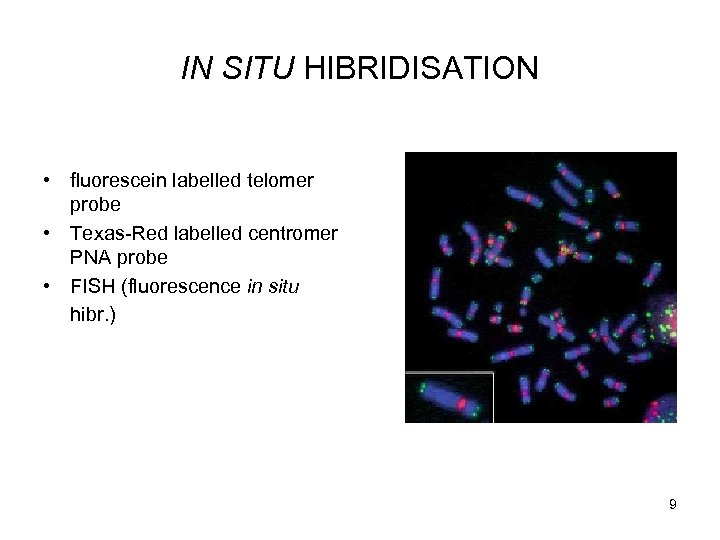
IN SITU HIBRIDISATION • fluorescein labelled telomer probe • Texas-Red labelled centromer PNA probe • FISH (fluorescence in situ hibr. ) 9

A molekuláris klónozás enzimei • • restrikciós endonukleázok (REases) metilázok ligázok polimerázok egyéb nukleázok kinázok foszfatázok 10

II típusú restrikciós endonukleázok • Hasítás a felismert szekvencián belül – középen: • Eco. RV tompa (blunt) GAT ATC – aszimmetrikusan: ragadós (sticky) • 5’ túlnyúló Eco. RI G AATTC • 3’ túlnyúló Kpn. I GGTAC C • A ragadós vég hossza: 1, 2, 3, 4, (5) • A felismert helyek gyakorisága – Ptetra=4 -4 =1/256 – Phexa=4 -6 =1/4096 – Phexa(2)=4 -6 x 4 -2 =1/65536 (pl. CCCGGG) 11

Programmok restrikciós térképezéshez • TACG V 3 http: //athena. bioc. uvic. ca/tacg 3/formt. html • NEBcutter V 2. 0 http: //tools. neb. com/NEBcutter 2/index. php • Plas. Mapper Version 2. 0 http: //wishart. biology. ualberta. ca/Plas. Mapper/ • Vector NTI Viewer 4. 0. 1 letölthető: http: //szilagyl. web. elte. hu/program/ az egészet bemásolni a helyi lemezre, C: VNTI alá 12
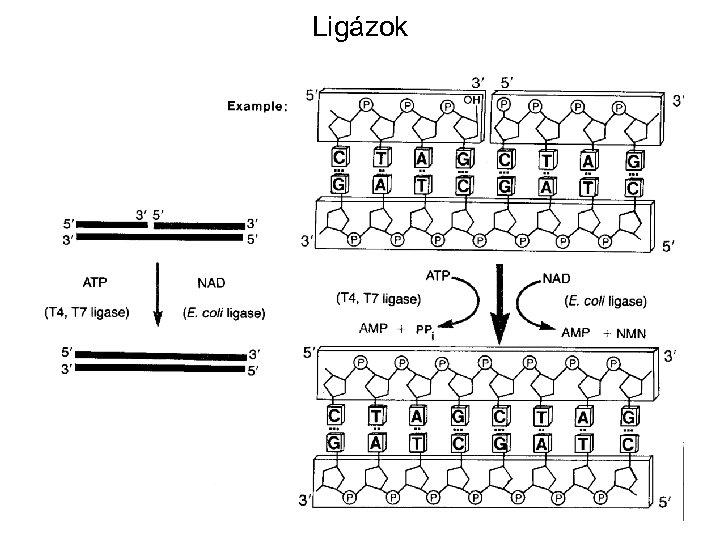
Ligázok 13
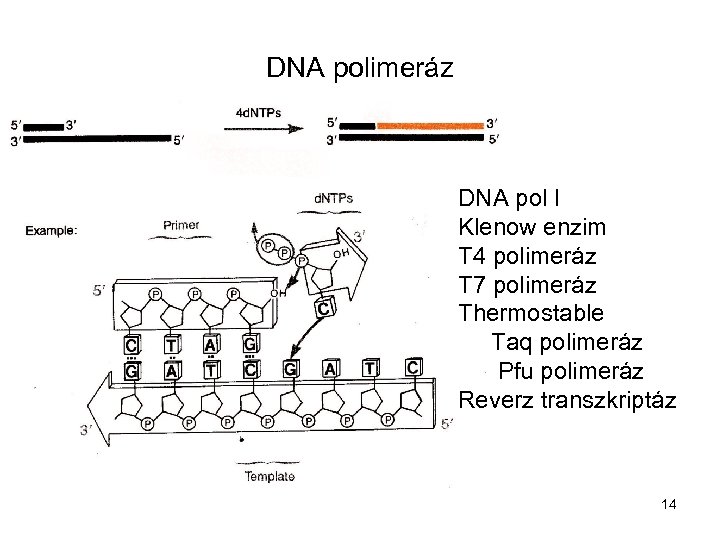
DNA polimeráz DNA pol I Klenow enzim T 4 polimeráz T 7 polimeráz Thermostable Taq polimeráz Pfu polimeráz Reverz transzkriptáz 14
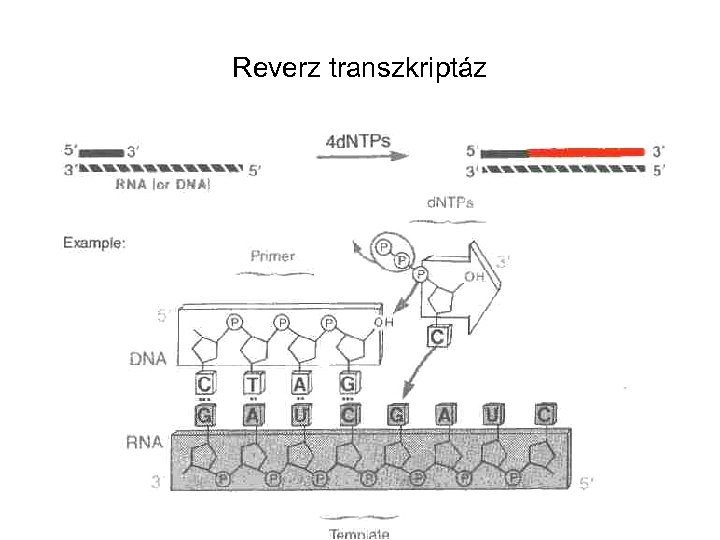
Reverz transzkriptáz 15
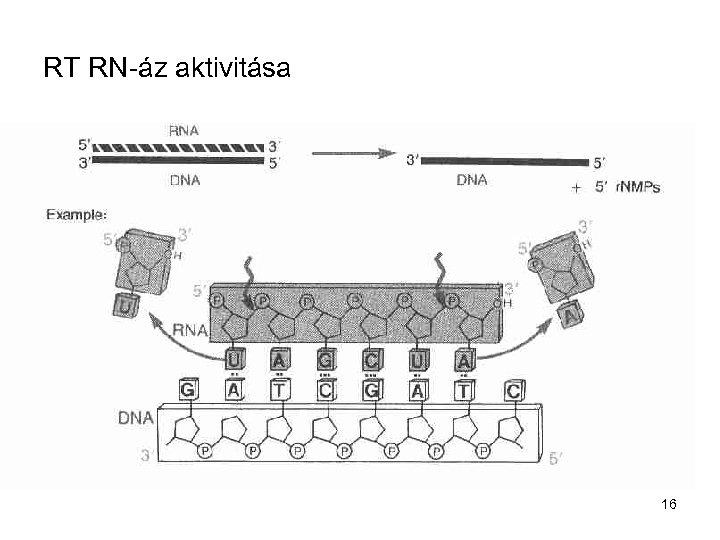
RT RN-áz aktivitása 16

Nukleázok • egy-szálú nukleázok – Bal 31 nukleáz – S 1 nucleáz, Mung bean nukleáz – Exonukleáz VII • nem specifikus két-szálú – DNase I • két-szálú 3’ → 5’ – Exonuclease III • két-szálú 5’ → 3’ – lambda exonukleáz 17
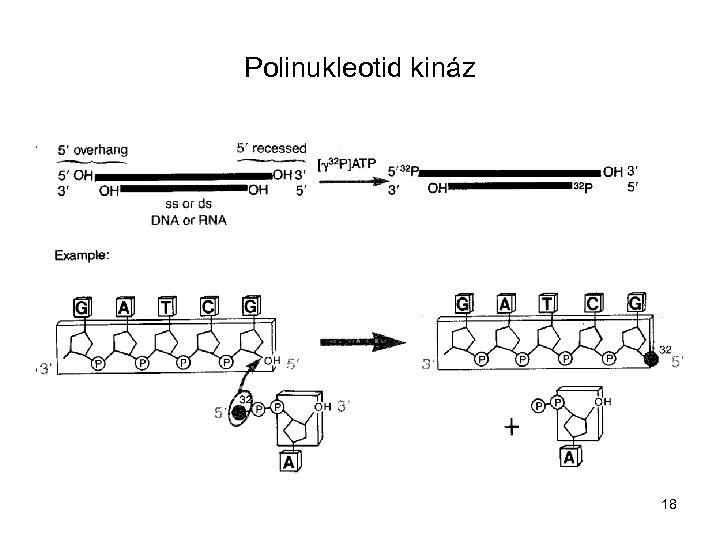
Polinukleotid kináz 18

Foszfatázok az 5’ foszfát csoport eltávolítása • Bacterial alkaline phosphatase (BAP) • Calf intestinal phosphatase (CIP) • Shrimp phosphatase 19

Vektorok

• Cloning vectors • Plasmids Expression – prokaryotic – eukaryotic • Phage – M 13 – lambda • Cosmids • Artificial chromosomes 21
22

Selection I • Selection for the uptake of plasmid – Antibiotic resistance • • • Ampicillin Tertacycline Chloramphenicol Streptomycine Kanamycine 23
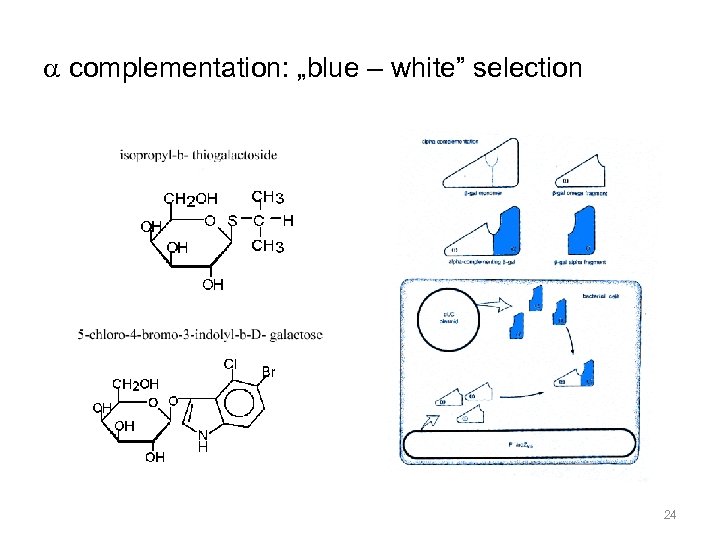
complementation: „blue – white” selection 24
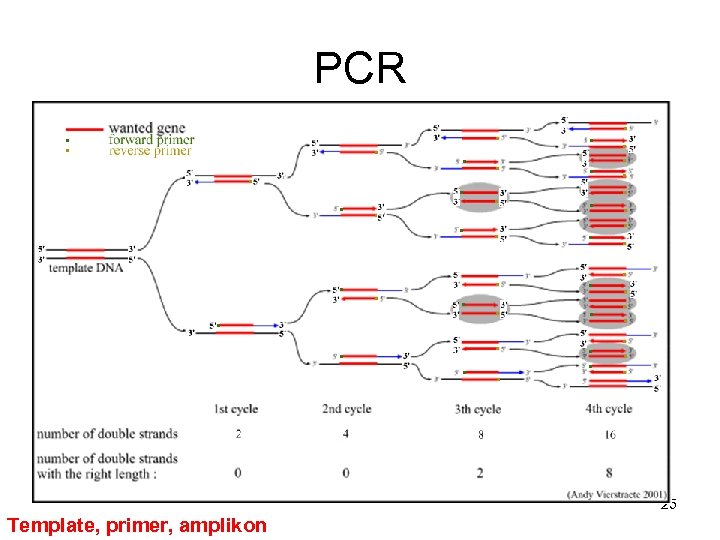
PCR 25 Template, primer, amplikon

Different forms of PCR • • • Asymmertic Degenerate Hot-start In-situ Inverse LAMultiplex Nested RACE Real-time RTTouchdown • • • one oligo in high excess base on a. a. sequence to increase specificity in tissue sections what is outside? long and accurate several oligo pairs 2 oligo pairs rapid amplification of c. DNA ends continuous detection of products coupled to reverse transcription continuous decrease of annaeling temperature 26

Primer design A simple set of rules for primer sequence design 1. primers should be 17 -28 bases in length; 2. base composition should be 50 -60% (G+C); 3. primers should end (3') in a G or C, or CG or GC: this prevents "breathing" of ends and increases efficiency of priming; 4. Tms between 55 -80 o. C are preferred; 5. runs of three or more Cs or Gs at the 3'-ends of primers may promote mispriming at G or C-rich sequences (because of stability of annealing), and should be avoided; 6. 3'-ends of primers should not be complementary (ie. base pair), as otherwise primer dimers will be synthesized preferentially to any other product; 7. primer self-complementarity (ability to form 2 o structures such as hairpins) should be avoided. 27

Rekombináns konstrukciók, klónozás 28

Praktikus tanácsok • Aszimmetrikus ligálás (két, nem kompatibilis enzim) – az inzert méretétől függetlenül 3: 1 inzert: vektor arány – inzert koncentráció < 5 g/ml – ha kevés az inzert – foszfatáz kezelt vektor felesleg • Tompa végű ligálás – mindig foszfatáz kezelt vektor – HC ligáz • A ligálás hőmérséklete – szobahőméréséklet 29

Cloning strategies • • • cohesive and/or blunt ends - compatible enzymes Single enzyme (self closing of the vector – CIP treatment) Two enzymes - directional cloning „tailing” Linkers: p. CGGATCCG Eco. RI Adapters: AATTCGCGGCCGC Eco. RI -Not. I GCGCCGGCGp Filling of 5’ overhang (any polymerase) Cut back of 3’ overhangs – exonuclease –T 4 polymerase • • • Partial filling to generate compatible overhangs AAGCTT Hind. III TCTAGA Xba. I AAG CTAGA TTCGA TCT AAGCTAGA TTCGATCT 30

Könyvtárak, szekvenálás, mutagenezis

• GÉNKÖNYVTÁRAK • GENOMIÁLIS KÖNYVTÁR • (könyvtár rendelésre: pl. Stratagene) vektor: -fág (helyettesítő), kozmid, YAC, PAC, BAC méret: genom méret • N = ln(1 -P)/ln[1 -(I/G)] klónok száma valószínűség inszert (gén) méret N = ln(1 -0, 99)/ln[1 -(2 x 104/3 x 109)] = 690. 000 vektor előkészítés: vektor karok izolálása (stuffer eltáv. ), defoszforilálás, részleges feltöltés inszert előkészítés: genomiális DNS izolálás (>100 kb; proteináz-K), emésztés v. mechanikus hasítás, méret szerint frakcionálás ligálás, in vitro csomagolás v. transzfekció
![c. DNS könyvtárak vektor: -fág (inszerciós), fágemid méret: N = ln(1 -P)/ln[1 -(1/n)] ha c. DNS könyvtárak vektor: -fág (inszerciós), fágemid méret: N = ln(1 -P)/ln[1 -(1/n)] ha](https://present5.com/presentation/d01ccedda77476fb2198bb7fa97c0013/image-33.jpg)
c. DNS könyvtárak vektor: -fág (inszerciós), fágemid méret: N = ln(1 -P)/ln[1 -(1/n)] ha 0, 01% RNS n=10. 000, N=46. 050 RNS, m. RNS izolálás (RNáz veszély! DEPC kezelés) guanidin-izotiocianát; oligo-d. T cellulóz c. DNS szintézis primer: oligo-d. T, oligo-d. T-adapter, random oligó, specifikus oligó reverz-transzkriptáz, RNáz H, DNS pol. áz I, ligáz, T 4 DNS pol. áz linker v. adapter
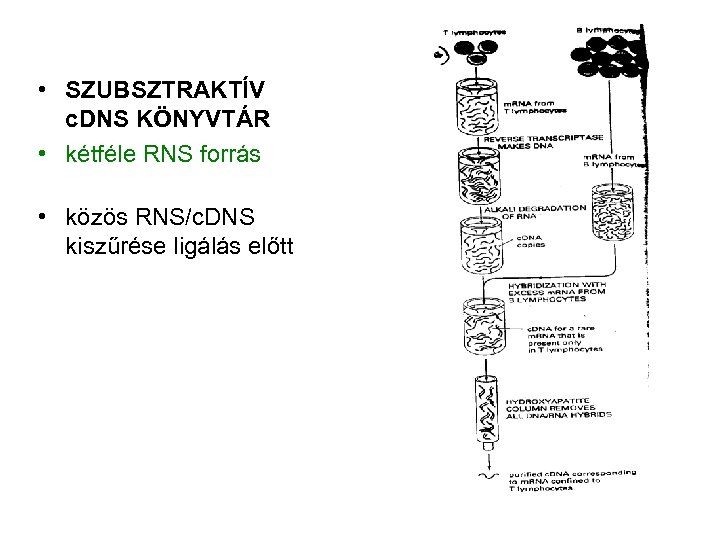
• SZUBSZTRAKTÍV c. DNS KÖNYVTÁR • kétféle RNS forrás • közös RNS/c. DNS kiszűrése ligálás előtt
35
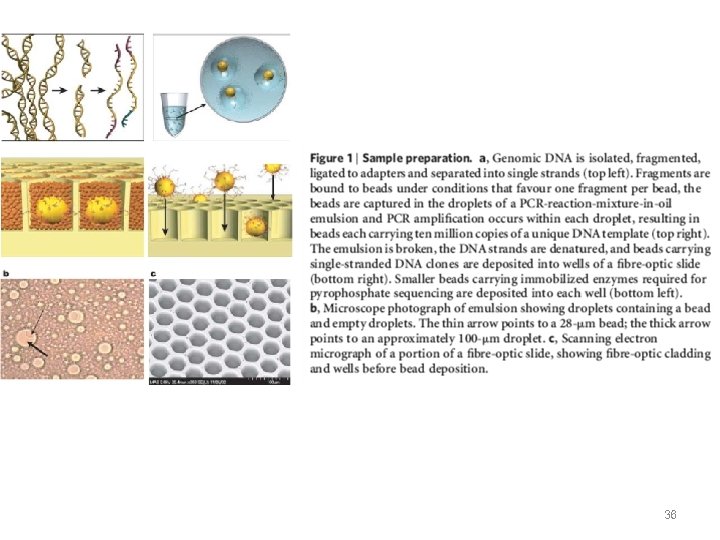
36

• BIOINFORMATIKA • • ADATBÁZISOK DNS adatbázisok: Gen. Bank (NIH, NCBI); http: //www. ncbi. nlm. nih. gov ’ 99 okt. : 4, 9 m szekv. , 3, 8 Gb 50. 000 faj; 54% humán; 25 teljes genom ’ 02 okt. 20197497568 ’ 05 jun. >89 millárd – ~500 teljes genom • • • db. EST („expressed sequence tags”) PCR amplifikált c. DNS (300 -500 bp) 3, 3 millió (1, 6 m. humán) Uni. Gen nem-redundáns humán gének (EST) (75 ezer) db. STS („sequence tagged sites”) rövid genomiális markerek (88 ezer) OMIM („online mendelian inheritance in man”) EMBL (EMBL/EBI; http: //www. ebi. ac. uk/embl International Nucleotide Sequence Database Collaboration Gen. Bank, EMBL, DDBJ Fly. Base (muslinca), SGD (sörélesztő), IMGT (immunológiai), stb. • • • 37

Expressziós rendszerek 38

„TÚLTERMELŐ” RENDSZEREK: általában heterológ rendszer expressziós vektor prokarióta v. eukarióta sejtkultúra, transzgénikus állat cél: könyvtár szkrínelés ( gt 11, -ZAP, p. CDNAI, II) antitest előállítás ORF vizsgálat PROTEIN ENGINEERING 39
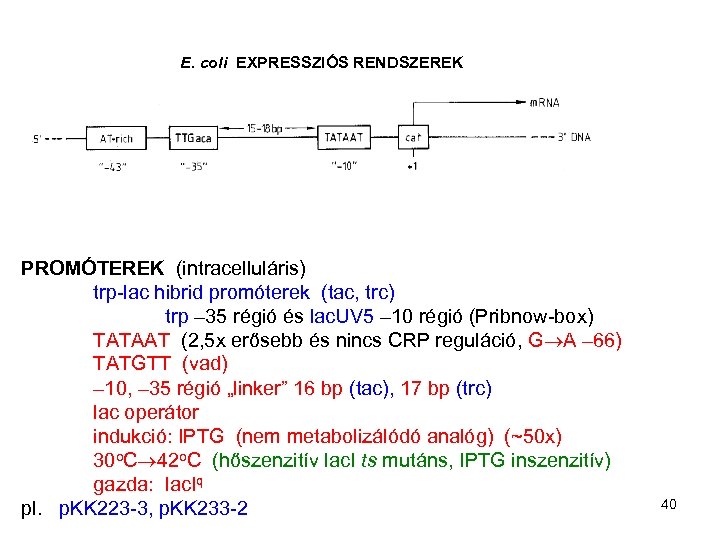
E. coli EXPRESSZIÓS RENDSZEREK PROMÓTEREK (intracelluláris) trp-lac hibrid promóterek (tac, trc) trp – 35 régió és lac. UV 5 – 10 régió (Pribnow-box) TATAAT (2, 5 x erősebb és nincs CRP reguláció, G A – 66) TATGTT (vad) – 10, – 35 régió „linker” 16 bp (tac), 17 bp (trc) lac operátor indukció: IPTG (nem metabolizálódó analóg) (~50 x) 30 o. C 42 o. C (hőszenzitív lac. I ts mutáns, IPTG inszenzitív) gazda: lac. Iq pl. p. KK 223 -3, p. KK 233 -2 40
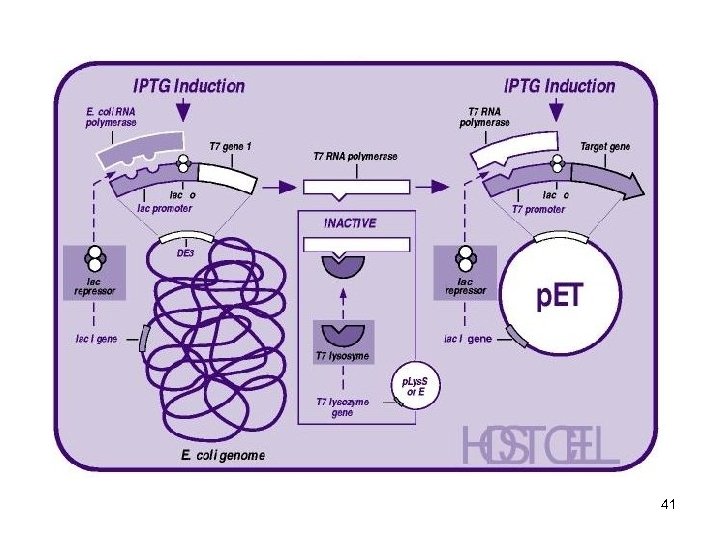
41
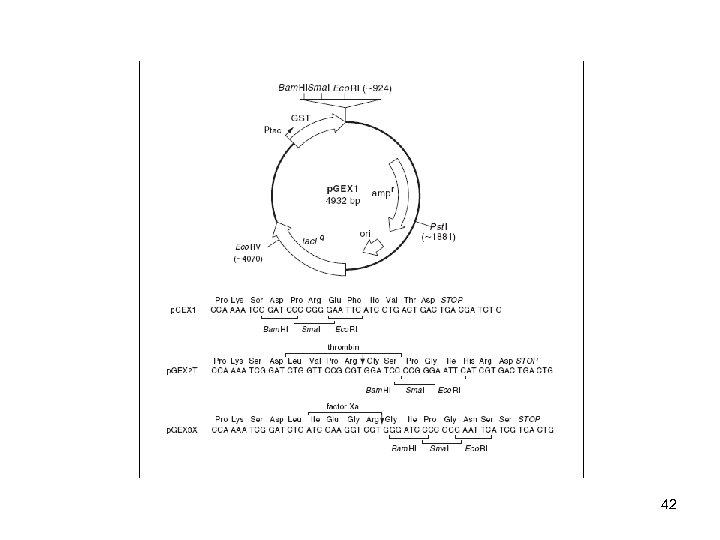
42

AFFINITÁS- CÍMKE („TAG”) FÚZIÓK His-tag N- vagy C-terminális 6 x. His Ni-affinitás gyanta (Ni 2+-nitrilotriecetsav) pl. p. ET 15 (N), p. ET 20 (C) T 7 -tag T 7 gén 1 „leader” szekvencia (11 as. ) transzláció enhancer S-tag ribonukleáz A S-peptid (15 as. ) detektálás, izolálás: biotinált S-protein, S-protein affinitás kvantitálás: S-protein + poli(C); TCA, 260 nm (20 fmol) Strap-tag C-terminális AWRHPQFGG (affinitás streptavidinhez) epitóp-cimkék FLAG-peptid (DYKDDDDK), c-myc-tag V 5 -tag parmyovirus SP 5 43

PROBLÉMÁK, OPTIMALIZÁLÁS inszertben: SD-szekvencia+Met, terminátor, erős másodlagos szerkezet szekvencia analízis, hely-specifikus mutációk kódhasználat: Arg(AGG, AGA) arg. U (t. RNS gén) koexpresszió Rosetta host t. RNS 8 ritka kodonra SD-szekvencia (…AGGA…) és ATG távolsága 5 -11 bp m. RNS stabilizálás (30 sec-20 min) terminátor beépítés, omp. A 5’-UTR transzláció termináció UAA, UAAU 44

fehérje instabilitás: degradáció, rossz folding lon-, opm. T-, clp-, fts- gazdák chaperonin koexpresszió (Dna. K-Dna. J, Gro. EL-Gro. ES) hideg indukció (hideg sokk fehérje promóter; csp. A) fúziós fehérje inklúziós test („inclusion body”): renaturáció de: könnyű izolálás; proteáz védelem periplazma expresszió poszttranszlációs módosítások hiánya iniciáló Met eltávolítás, N-acetilálás, metilálás, glikozilálás stb. koexpresszió eukarióta enzimmel 45

ÉLESZTŐ EXPRESSZIÓS RENDSZEREK S. cerevisiae vektorok („shuttle”): YEp, YCp, YIp szelekciós markerek: LEU 2, TRP 1, URA 3, HIS 3 (auxotróf gazda) promóterek: Erősség szabályozás PGK, GAPD ++++ gyenge indukció glükóz GAL 1 +++ erős indukció galaktóz ADH 2 ++ glükóz represszió PHO 5 ++ foszfát represszió Hibridek, pl. ADH/GAPD terminátorok: PGK, GAPD, CYC 1 transzkripció termináció, processzálás, poliadeniláció 46

Pichia pastoris („szuperexpresszió”, g/l) gyors, egyszerű, olcsó metilotróf élesztő promóter: alkohol-oxidáz (AOX 1) indukció: metanol transzfer vektorok („shuttle”) (Invitrogen) vektor beépítés kromoszómába homológ rekombinációval szelekció: His+, Mut- fenotípus (Me. OH utilization deficient) 47

• DICTYOSTELIUM EXPRESSZIÓS RENDSZER • D. discoideum sejtes nyálkagomba • plazmid replikon (nagy kópiaszám) • promóter: aktin (konstitutív), diszkoidin (induklható) • His-tag, epitóp-tag; neo rezisztencia (G 418 antibiotikum) 48

BAKULOVÍRUS EXPRESSZIÓS RENDSZER rovar specifikus vírusok Ac. NPV: Autographa californica (alfalfa araszoló) nukleáris polihedrózis vírus (50 x 400 m) 128 kb cirkuláris genom (154 gén) bimbózás (10 -12 h) v. okklúziós vírus (20 -24 h) szaporodási ciklus előnyök: egyszerű, relatíve olcsó genomiális DNS is jó (mérethatár nincs) poszttranszlációs módosulások, eukarióta chaperoninok szekréció, kompartmentalizáció gazda: Sf 9 (Spodoptera frugiperda) báb petefészek sejttenyészet v. lárva monolayer v. szuszpenziós kultúra 49
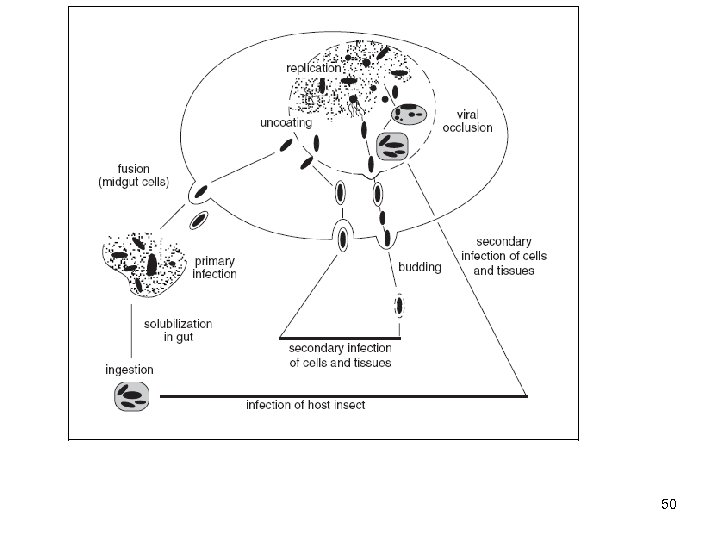
50

Az emlős expressziós vektorok szabályzó elemei vírus genomokból származnak papillomavírus (BPV) bovine papovavírus (szemölcs), sokféle gazda stabil episzóma (20 -50 kópia) Epstein-Barr vírus (EBV) herpesz (170 kb) stabil episzóma: Ori. P origó (1, 8 kb) és EBNA-1 nukleáris antigén (2, 6 kb) humán citomegalovírus (CMV) legerősebb promóter/enhancer (major immediate early, h. CMV-MIE) retrovírus Rouse szarkóma, RSV; murine leukémia, MLV; egér emlőtumor, MMTV LTR („long terminal repeat”): promóter, poliadenilációs és RT szignál permanens expresszió transzfekció: deficiens provírus gazda (in vivo „csomagolás”) infekció: rekombináns vírus kromoszóma integráció több génes vektorok két promóter v. IRES („internal ribosome entry site”) génterápia vektorok 51

INDUKÁLHATÓ RENDSZEREK • szteroid-hormon indukció adenovírus major late promóter + GRE (glucocorticoid response element) gazda: glükokortikoid receptor túltermelő CHO indukció: dexametazon (1000 x) • „lac switch” (Stratagene) 1. vektor: RSV-LTR promóter + lac operátor (3 x) + inszert 2. vektor: CMV promóter + lac. I (NLS szignál, 21 bp) indukció: IPTG • ekdizon-indukció (Invitrogen) 1. vektor: HSP promóter + ekdizon response elem + inszert 2. vektor: CMV és RSV promóter ekdizon receptor heterodimer indukció: muriszteron 52

„tet-off, tet-on” rendszer Tn 10 tetraciklin rezisztencia operon: operátor + tet represszor 1. („response”) vektor: tet operátor + CMV mini promóter + inszert 2. (regulátor) vektor: CMV promóter tet repressor: VP 16 aktivációs domén (kiméra transzaktivátor) indukció: tet (v. dox) elvonás („tet-off”) ill. hozzáadás („tet-on”) (mutáns tet represszor) T-Rex rendszer (Invitrogen) CMV promóter + 2 x tet operátor + inszert indukció: tetraciklin Tet-On® and Tet-Off® Advanced Inducible Gene Expression Systems (Clontech) 53

EXPRESSZIÓ TRANSZGÉNIKUS ÁLLATBAN szövetspecifikus promóter (emlőmirigy) -laktalbumin, - v. -kazein állat: egér, nyúl, kecske, juh, marha génbevitel: megtermékenyített petesejt pronukleusz álvemhes anya transzgénikus utód (azonosítás: genomiális DNS analízis farokból) transzgenikus vonal rekombináns fehérje: tej pl. t. PA: 6 g/l 1 -proteáz inhibitor: 20 g/l pl. vektor: p. BC 1 (Invitrogen) 54

GÉN „TARGETING” Célzott génkilövés („knockout”) / génbevitel („knockin”), mutációk Genom megváltoztatása hely-specifikus rekombinációval transzfekció: ES (egér embrionális „stem”) sejt targeting vektor (inszerció v. helyettesítés) szekvencia homológia (10 -14 kb) szelekciós gén (pl. neo rezisztencia) a konstrukció közepén homológ és nem-homológ rekombináció pozitív (neo rez. , G 418) ÉS negatív szelekció: HSV timidin-kináz a konstrukció szélén ganciklovir (1, 3 -dihidroxi-2 -propoximetil-guanin) 55

Fehérje adatbázisok: Swissprot /http: //www. expasy. ch/sprot/ annotált, nem-redundáns 82 ezer szekv. (29 millió as. ) Tr. EMBL DNS adatbázis transzláció 198 ezer ORF PROSITE fehérje családok, domének, motívumok (1400 féle) Swiss 2 DPAGE 2 -dimenziós PAGE (proteomok) (600) Térszerkezeti adatbázis: PDB (Protein Data Bank) http: //www. rcsb. org/pdb/ kristály (9100) és NMR (1800) és modell szerkezetek (230) atom-koordináták, megjelenítés: Ras. Mol, Chime, Pdb. Viewer NDB (nukleinsav 3 D), RELIBase (receptor-ligand) Publikációk: Pub. Med (NCBI, NLM) http: //www. ncbi. nlm. nih. gov/Pub. Med/ Medline: 3900 folyóirat, 9 millió citáció Molekuláris biológiai szoftverek: Biocatalog 56

IN VITRO MUTAGENEZIS Deléciók: restrikciós fragmentum kivágás exonukleázok kétirányú (Exo. III + S 1; BAL 31) (10 g, 2 kb DNS + 0, 5 U BAL 31=~20 bp/min) egyirányú („nested” deléció) Inszerciók: „fill-in” reakció linkerek, adapterek szintetikus inszert transzpozon mutagenezis Szubsztitúciók kazetta mutagenezis 57

Hely-specifikus mutációk („site-directed mutagenesis”) szintetikus oligonukleotidok 16 -18 mer (több mutáció, hosszabb) (5’-foszforilálás!) egyszálú templát (M 13 v. fágemid) heteroduplex DNS (vad és mutáns szál) in vitro szintézis pl. T 4 DNS pol. áz + T 4 DNS ligáz mutáns azonosítása: restrikciós hely +/(GCG „map/silent” program) szekvenálás denaturáló gradiens gélelektroforézis (DGGE) 58
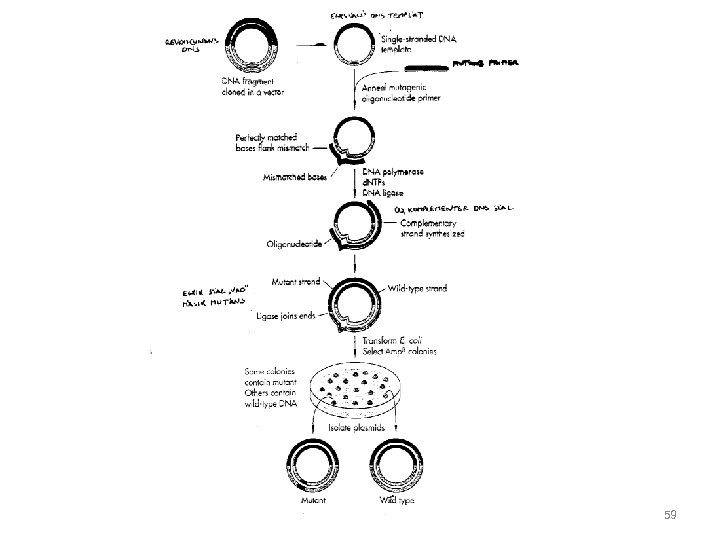
59
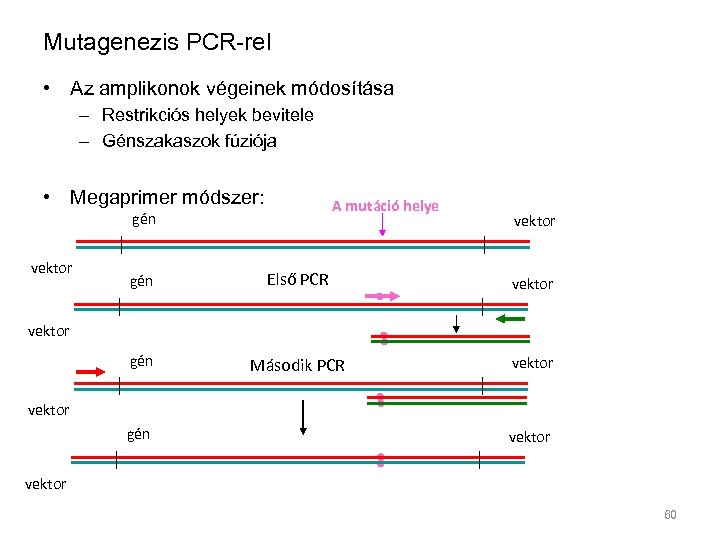
Mutagenezis PCR-rel • Az amplikonok végeinek módosítása – Restrikciós helyek bevitele – Génszakaszok fúziója • Megaprimer módszer: A mutáció helye gén vektor gén Első PCR vektor gén Második PCR vektor gén vektor 60
d01ccedda77476fb2198bb7fa97c0013.ppt