c2c647090e252d4acfa6a8c32dbc5309.ppt
- Количество слайдов: 39

프로테옴 데이터 분석을 위한 바이오인포매틱스 기술 2002. 11. 5. 한국기초과학지원연구원 프로테옴분석팀 권 경 훈 khoon@kbsi. re. kr

목 차 • 프로테오믹스 • 단백질의 질량스펙트럼 • 탄뎀 질량스펙트럼 • 프로테옴 데이터베이스 시스템 • ICAT 방법에 의한 프로테옴 정량분석

프로테옴 (Proteome), 프로테오믹스 (Proteomics) § Protein + ome = Proteome 특정 세포나 특수 상황에서 만들어지고 작용하는 단백질들의 총합 § 프로테오믹스(Proteomics) 환경이 바뀜에 따라서 단백질들이 어떻게 생성되고 변화하며 어떻게 상호작용하는가를 연구
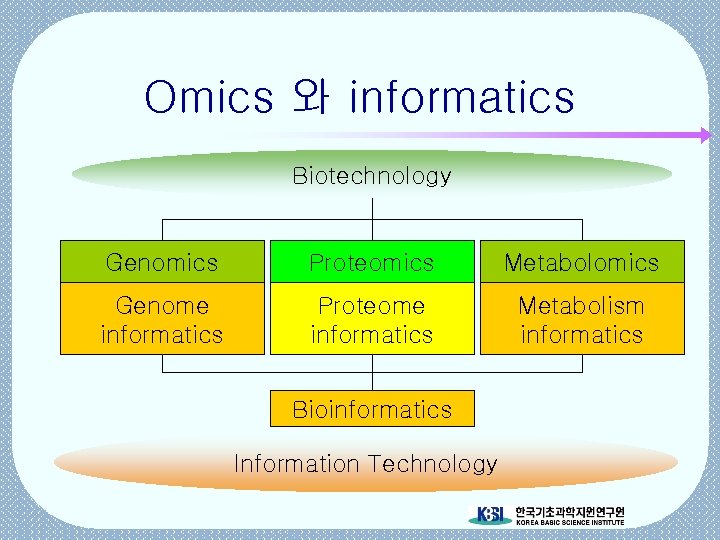
Omics 와 informatics Biotechnology Genomics Proteomics Metabolomics Genome informatics Proteome informatics Metabolism informatics Bioinformatics Information Technology

Omics 의 관계 metabolomics Molecular machinery of cellular processes proteomics Characterization of proteome genomics Genome sequencing 1989 Peptide mass fingerprinting 1986 peptide sequencing by MS/MS

Proteome informatics • 단백질 동정 기술 • 통합 Database 구축 • 단백질 기능, 구조 분석 • System biology

• 프로테오믹스 • 단백질의 질량스펙트럼 • 탄뎀 질량스펙트럼 • 프로테옴 데이터베이스 시스템 • ICAT 방법에 의한 프로테옴 정량분석

프로테옴 데이터 electrophoresis Mass spectrometry Database search 단백질 분리 펩타이드 분자량 측정 단백질 동정
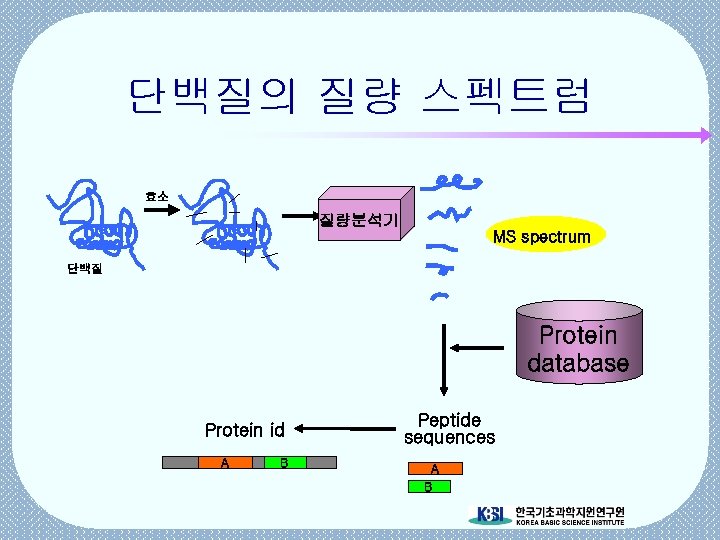
단백질의 질량 스펙트럼 효소 질량분석기 MS spectrum 단백질 Protein database Protein id A B Peptide sequences A B
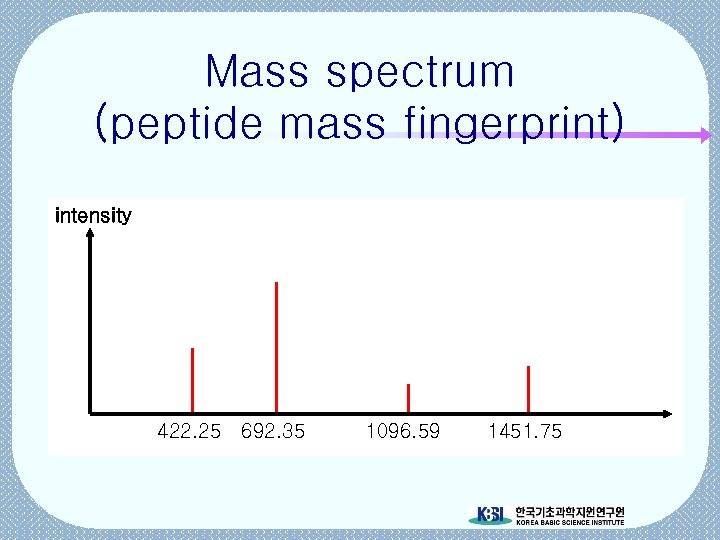
Mass spectrum (peptide mass fingerprint) intensity 422. 25 692. 35 1096. 59 1451. 75
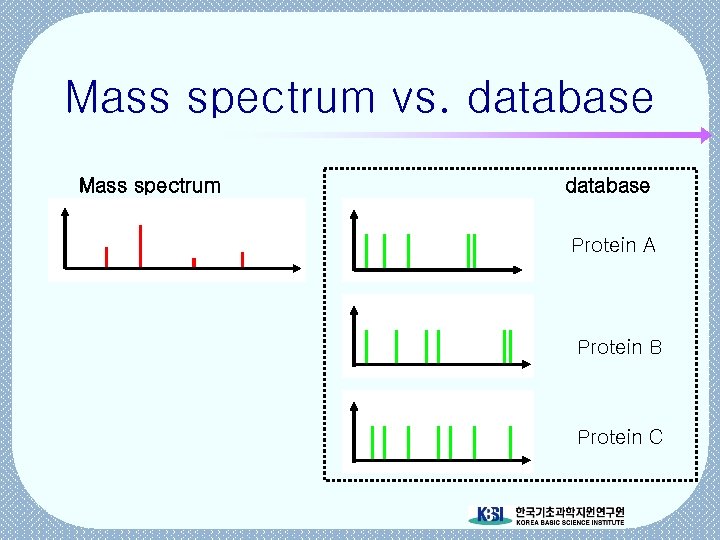
Mass spectrum vs. database Mass spectrum database Protein A Protein B Protein C
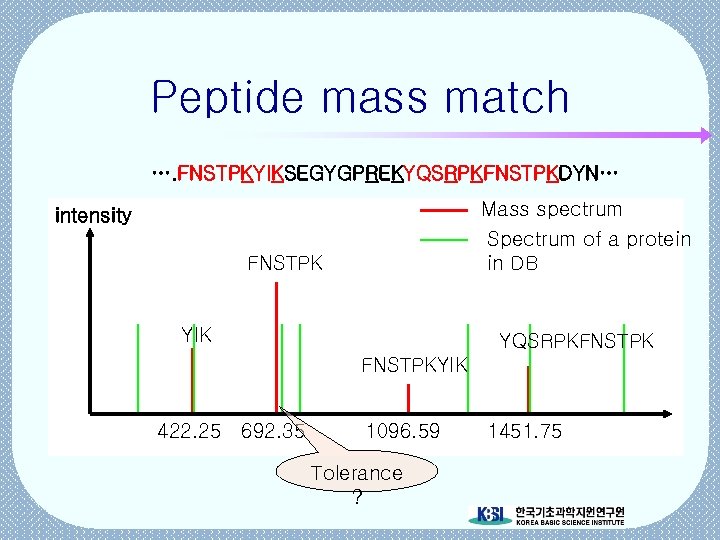
Peptide mass match …. FNSTPKYIKSEGYGPREKYQSRPKFNSTPKDYN… Mass spectrum Spectrum of a protein in DB intensity FNSTPK YIK YQSRPKFNSTPKYIK 422. 25 692. 35 1096. 59 Tolerance ? 1451. 75
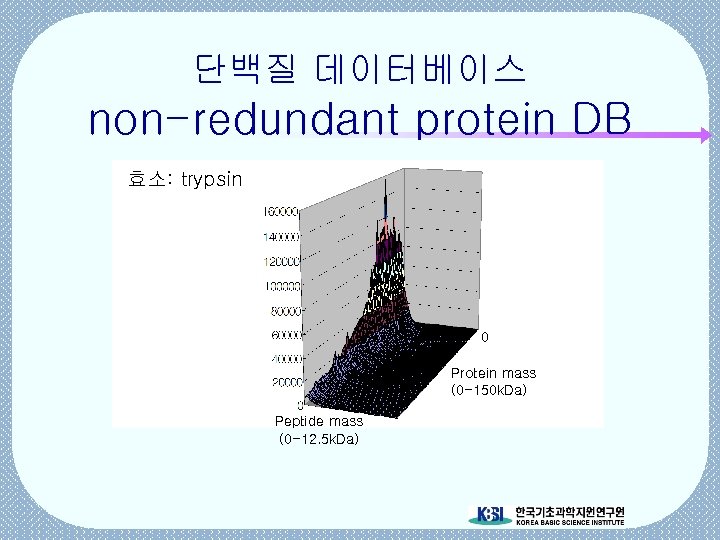
단백질 데이터베이스 non-redundant protein DB 효소: trypsin 0 Protein mass (0 -150 k. Da) Peptide mass (0 -12. 5 k. Da)

PMF search programs MS-Fit http: //prospector. ucsf. edu/ MASCOT http: //www. matrixscience. com/ Pept. Ident http: //www. expasy. org/ Peptide. Search http: //www. mann. embl-heidelberg. de/
MS-Fit
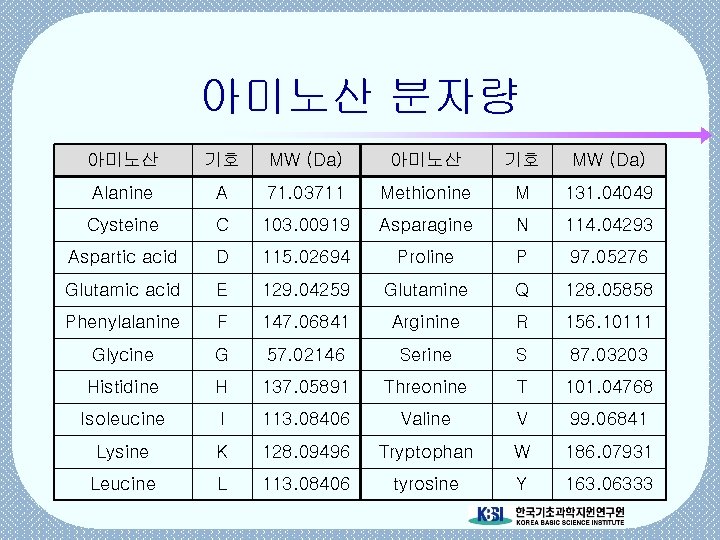
아미노산 분자량 아미노산 기호 MW (Da) Alanine A 71. 03711 Methionine M 131. 04049 Cysteine C 103. 00919 Asparagine N 114. 04293 Aspartic acid D 115. 02694 Proline P 97. 05276 Glutamic acid E 129. 04259 Glutamine Q 128. 05858 Phenylalanine F 147. 06841 Arginine R 156. 10111 Glycine G 57. 02146 Serine S 87. 03203 Histidine H 137. 05891 Threonine T 101. 04768 Isoleucine I 113. 08406 Valine V 99. 06841 Lysine K 128. 09496 Tryptophan W 186. 07931 Leucine L 113. 08406 tyrosine Y 163. 06333

Peptide mass fingerprint 의 한계 • Mass tolerance 내에서 분자량이 일치하는 아미노산 서열을 찾아야 함. modification 문제 • 대부분의 경우 여러 단백질을 검색 결과로 얻으므로 검색결과의 신뢰도가 문제 발생. 확인 과정 필요 • 많은 양의 프로테옴 데이터 검색을 위해서는 batch 작업이 필요함.
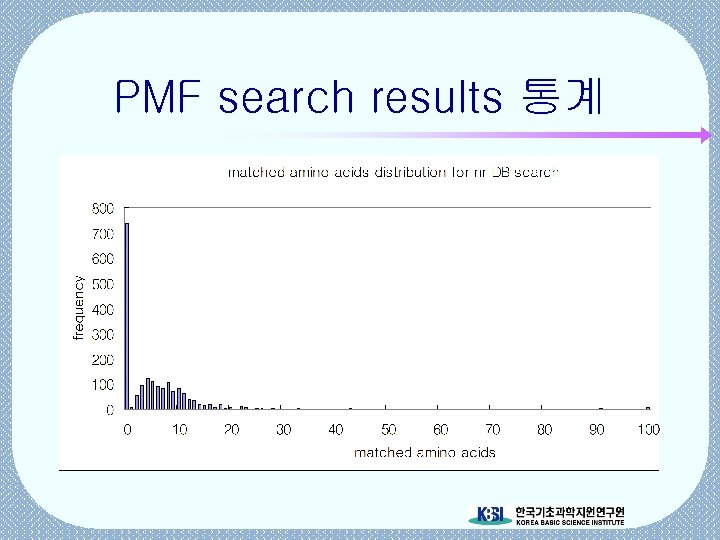
PMF search results 통계

• 프로테오믹스 • 단백질의 질량스펙트럼 • 탄뎀 질량스펙트럼 • 프로테옴 데이터베이스 시스템 • ICAT 방법에 의한 프로테옴 정량분석
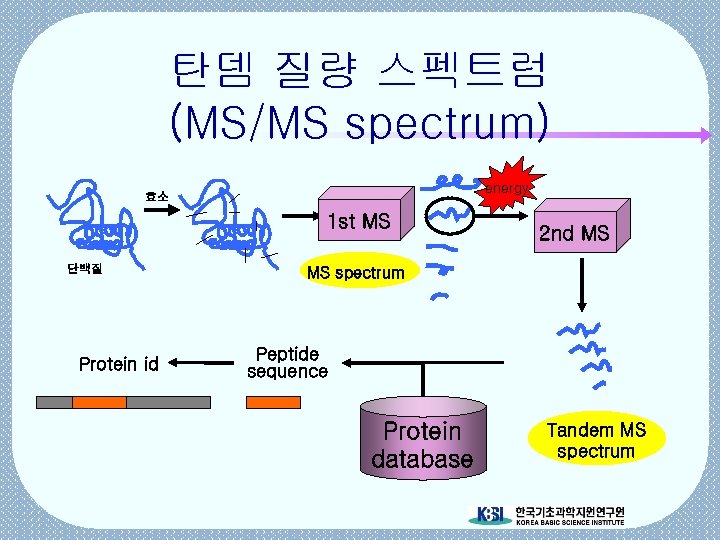
탄뎀 질량 스펙트럼 (MS/MS spectrum) energy 효소 1 st MS 단백질 Protein id 2 nd MS MS spectrum Peptide sequence Protein database Tandem MS spectrum
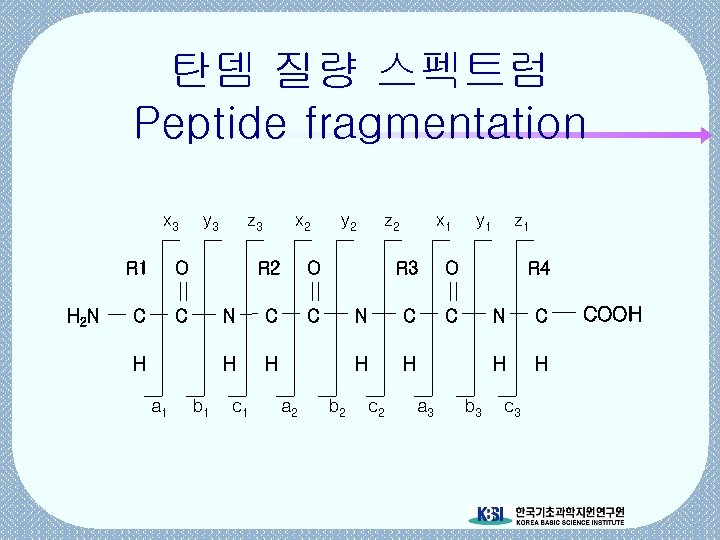
탄뎀 질량 스펙트럼 Peptide fragmentation x 3 R 1 H 2 N C z 3 O C y 3 x 2 R 2 a 1 b 1 C H H C H z 2 O N y 2 c 1 x 1 N b 2 C C H z 1 O H a 2 R 3 y 1 c 2 R 4 b 3 C H a 3 N H c 3 COOH
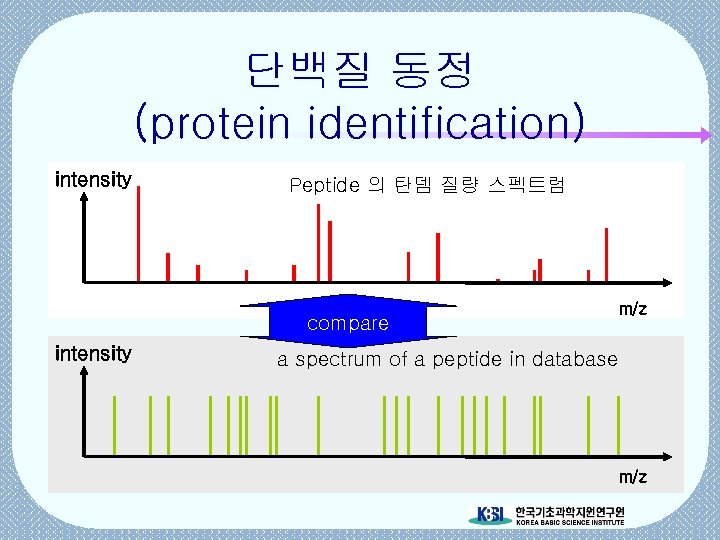
단백질 동정 (protein identification) intensity Peptide 의 탄뎀 질량 스펙트럼 compare intensity m/z a spectrum of a peptide in database m/z
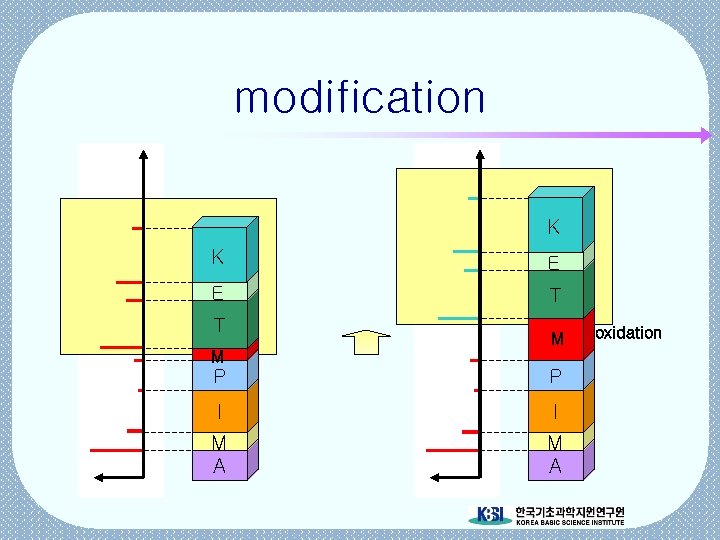
modification K K E E T T M M P P I I M A oxidation

MS/MS Search programs MASCOT http: //www. matrixscience. com/ MS-TAG http: //prospector. ucsf. edu/ SEQUEST http: //fields. scripps. edu/sequest/
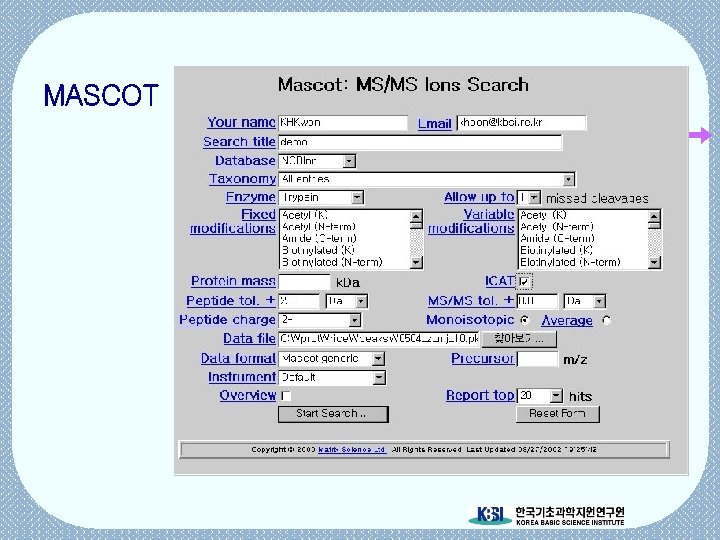
MASCOT
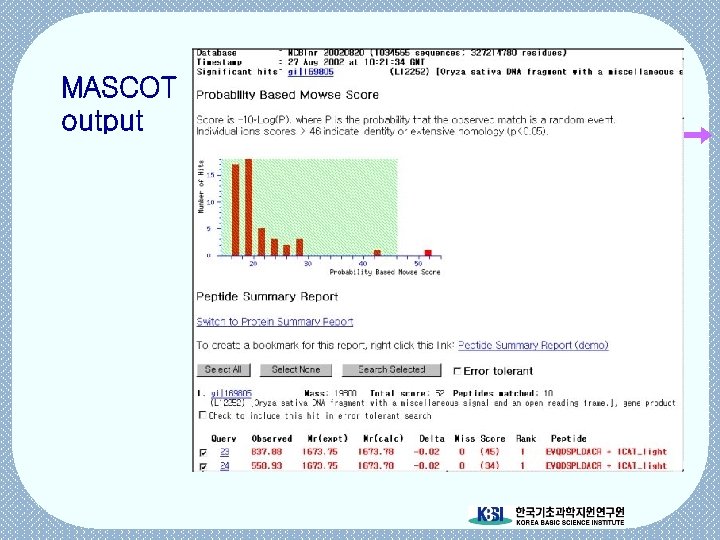
MASCOT output
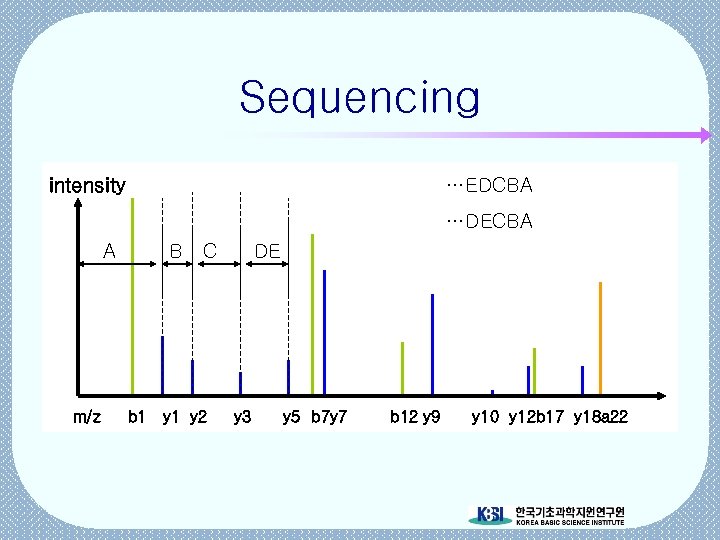
Sequencing intensity …EDCBA …DECBA A m/z B C b 1 y 2 DE y 3 y 5 b 7 y 7 b 12 y 9 y 10 y 12 b 17 y 18 a 22
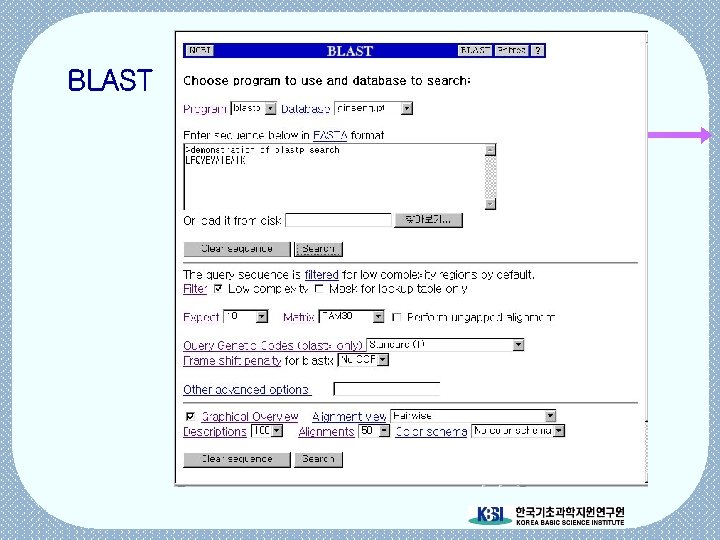
BLAST

Scores Homology search (BLAST) alignment score e-value Peptide Mass Fingerprint Number of matched peptides Coverage MOWSE score Probability based on MOWSE score Tandem mass search Cross-correlation coverage

• 프로테오믹스 • 단백질의 질량스펙트럼 • 탄뎀 질량스펙트럼 • 프로테옴 데이터베이스 시스템 • ICAT 방법에 의한 프로테옴 정량분석

Proteomics Trends 1. High-throughput Proteomics 2. High-throughput automated identification of proteins 3. 2. Quantitation 4. Quantitation of relative protein abundance
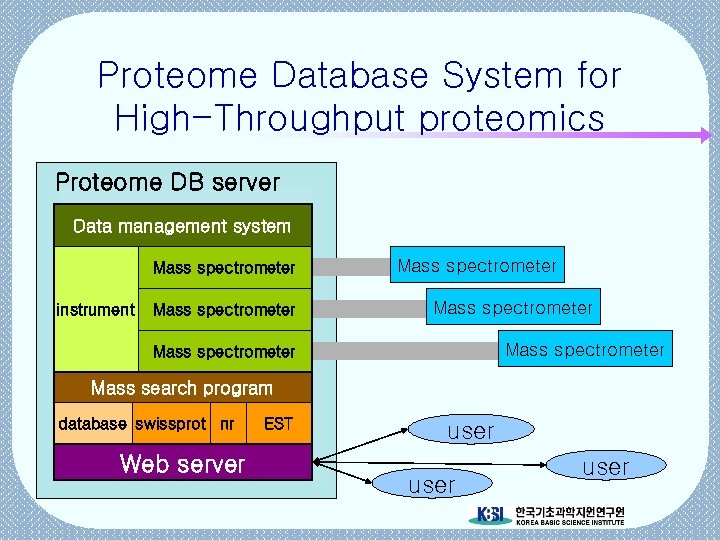
Proteome Database System for High-Throughput proteomics Proteome DB server Data management system Mass spectrometer instrument Mass spectrometer Mass spectrometer Mass search program database swissprot nr Web server EST user

• 프로테오믹스 • 단백질의 질량스펙트럼 • 탄뎀 질량스펙트럼 • 프로테옴 데이터베이스 시스템 • ICAT 방법에 의한 프로테옴 정량분석

Quantitation: ICAT (Isotope-coded Affinity Tag) § 정량적 단백질 발현 분석의 혁신적인 방법 § 서로 다른 환경에서 발현된 단백질들을 비교하는 정량적 분석 방법 § 2 D gel 의 단점 보완 • 실험의 반복성 구현 • 실험 소요시간 단축 (1 week/exp. ) • membrane protein 분석 가능 • 작은 분자량의 단백질 측정
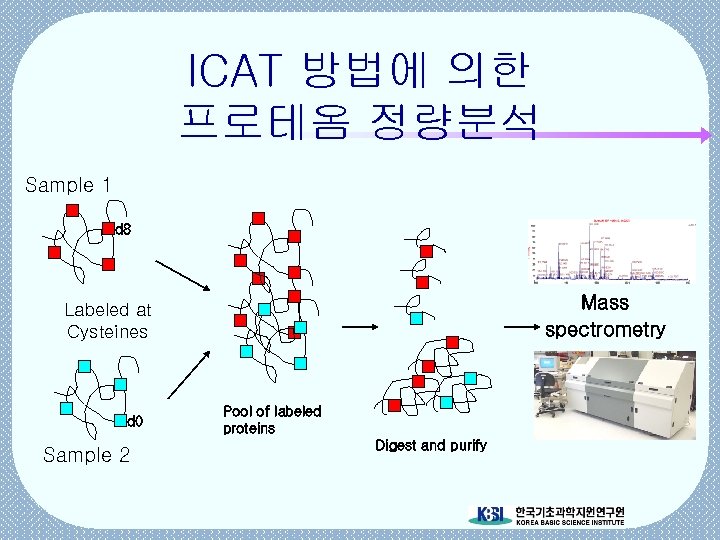
ICAT 방법에 의한 프로테옴 정량분석 Sample 1 d 8 Mass spectrometry Labeled at Cysteines d 0 Sample 2 Pool of labeled proteins Digest and purify
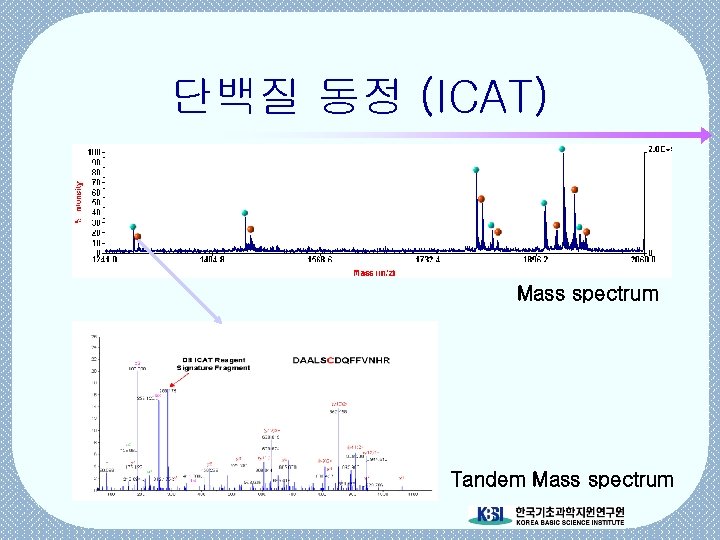
단백질 동정 (ICAT) Mass spectrum Tandem Mass spectrum
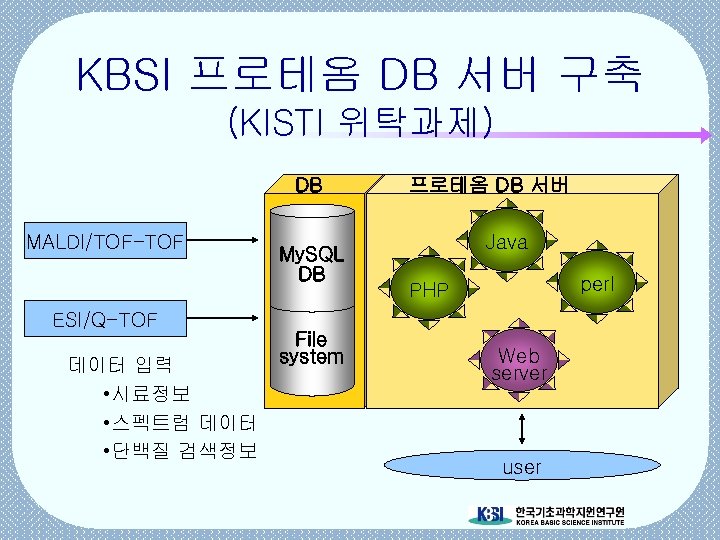
KBSI 프로테옴 DB 서버 구축 (KISTI 위탁과제) DB MALDI/TOF-TOF ESI/Q-TOF 데이터 입력 • 시료정보 • 스펙트럼 데이터 • 단백질 검색정보 My. SQL DB File system 프로테옴 DB 서버 Java perl PHP Web server user

한국기초과학지원연구원 프로테옴분석팀 김승일 박사 : 팀장 : 2 D-gel 박영목 박사 : 프론티어 과제 수행 Post doc. : 안영희, 김영혜, 권수미 유종신 박사 : 선임부장 : 질량분석 김수현 박사 : 탄수화물 분석 정영호 박사 : 형광분석기기 김영환 박사 : MALDI/TOF-TOF 최종순 박사 : DNA sequencing system 김진영 박사 : LC/MS, ESI/Q-TOF 조 건 : Tandem Mass 권경훈, 김미옥 박사 : informatics 위촉연구원 : 김재우, 황수경, 김수정, 박영재, 유용철, 이용주, 조옥기, 이정화, 권금녀, 김은아, 박은정, 이경민, 이은미, 정미영, 전영선, 김경욱, 문윤정, 송승열, 이영재, 조미선,

감사합니다. Proteome Informatics 교육 안내 - 실험실용 Protein Informatics 시스템 개발 교육 12월 3일(화) 오후 1: 00 – 5: 30 - 장소: 한국기초과학지원연구원 (대전본원) ( http: //www. kbsi. re. kr/ )
c2c647090e252d4acfa6a8c32dbc5309.ppt