Неканонические формы ДНК и их биологическое значение Н.Г.

Неканонические формы ДНК и их биологическое значение Н.Г. Долинная



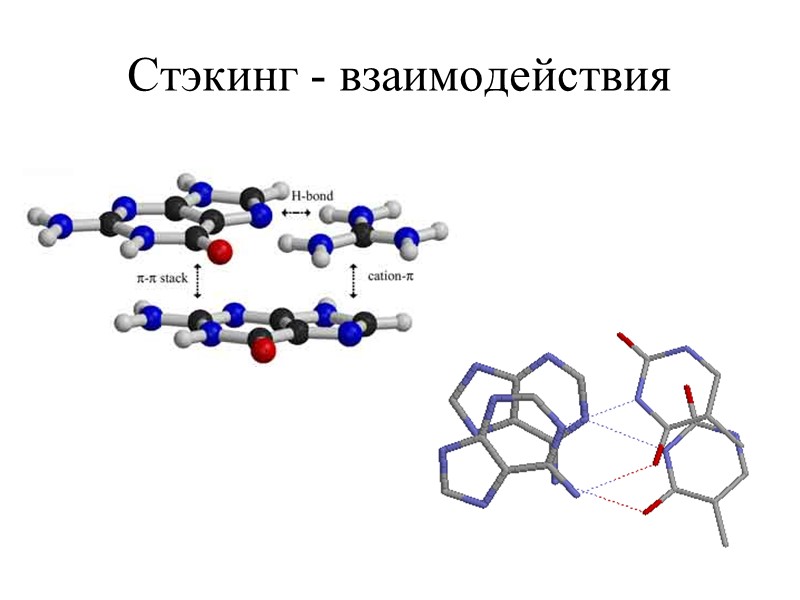
Стэкинг - взаимодействия

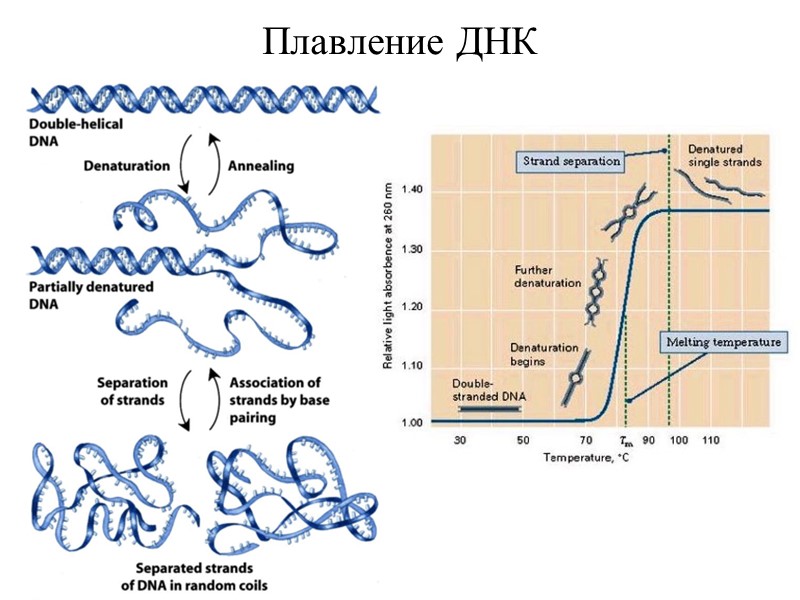
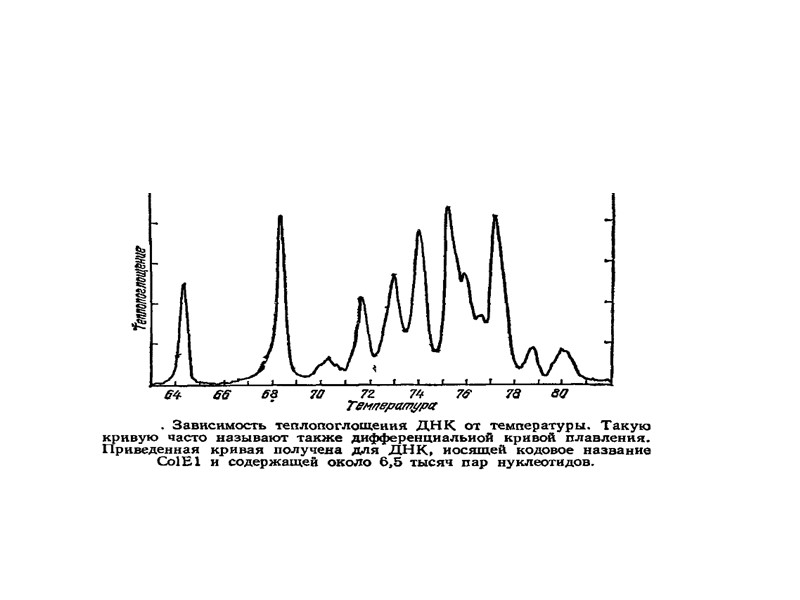
Плавление ДНК


Электронная микроскопия плавления ДНК-дуплекса
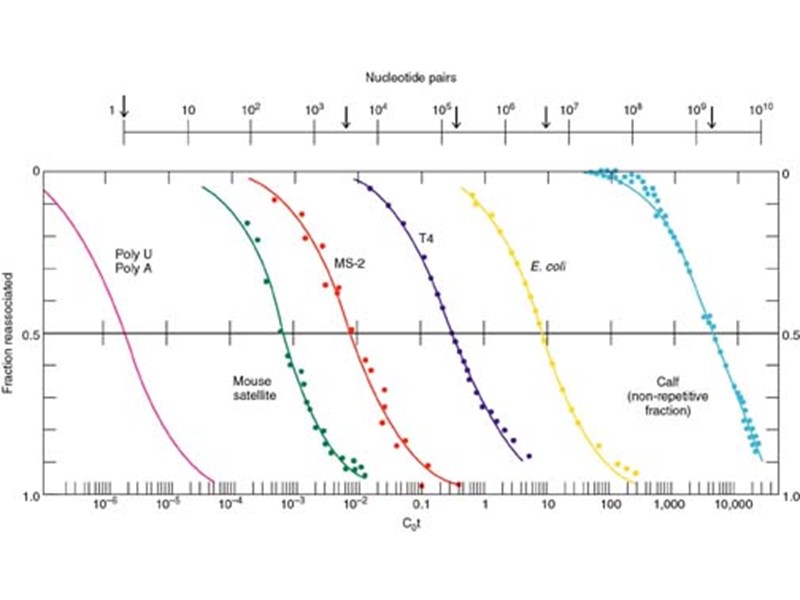
Ренатурация ДНК С – концентрация одноцепочечных участков k – константа скорости второго порядка t – время (сек) С0 - начальная концентрация ДНК


Гибриды ДНК-РНК

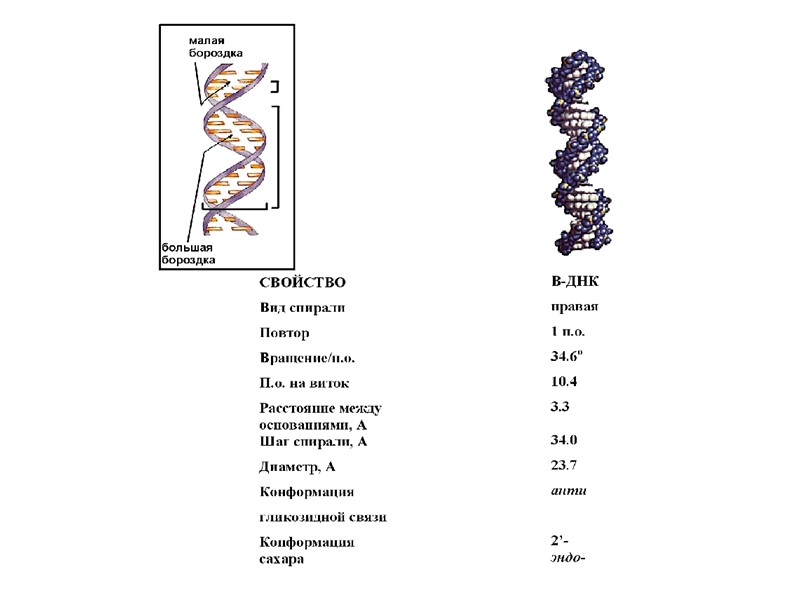
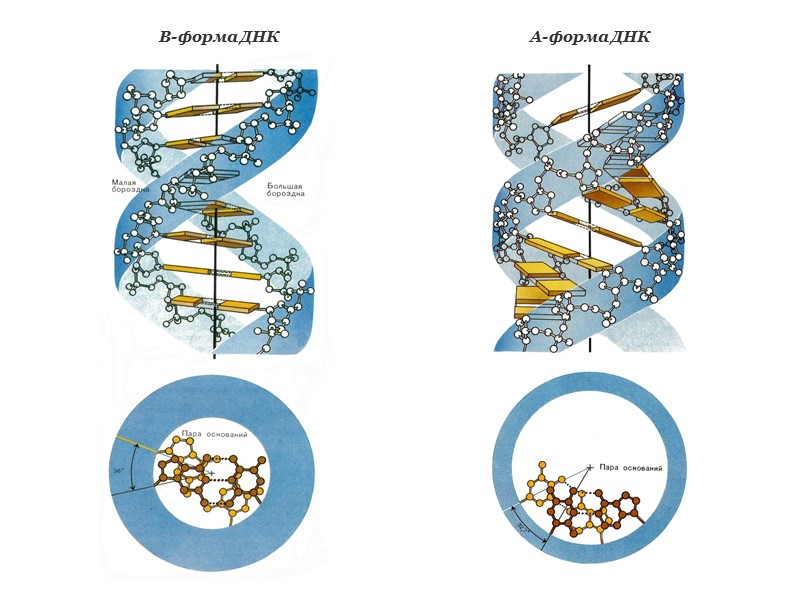
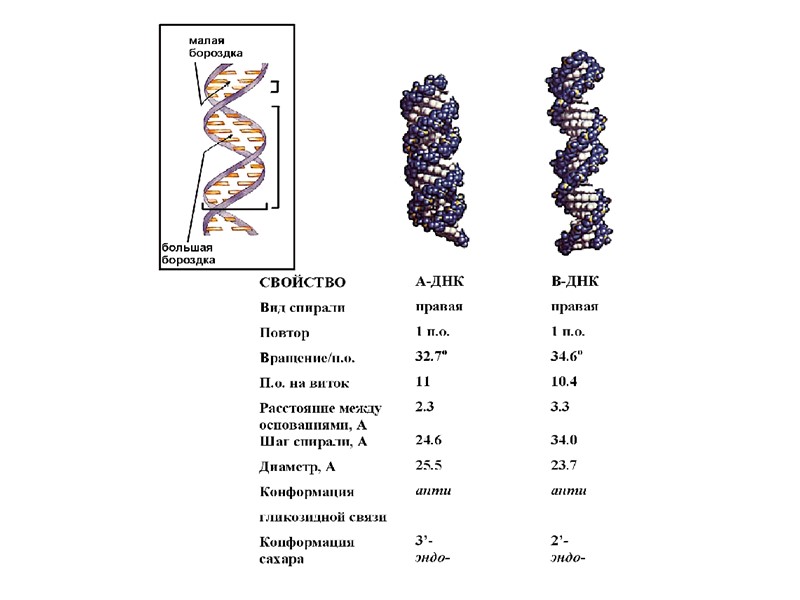
В-форма ДНК А-форма ДНК

Канонические структуры ДНК (А- и В- формы) двойные спирали с антипараллельной ориентацией цепей правозакрученные стабилизированы Уотсон-Криковскими парами оснований (A-T, G-C) плектонимичные дуплексы (для разделения цепей необходимо раскрутить двойную спираль) вторичная структура всей молекулы может быть выведена из конформации отдельной нуклеотидной пары линейные (неразветвленные) спирали
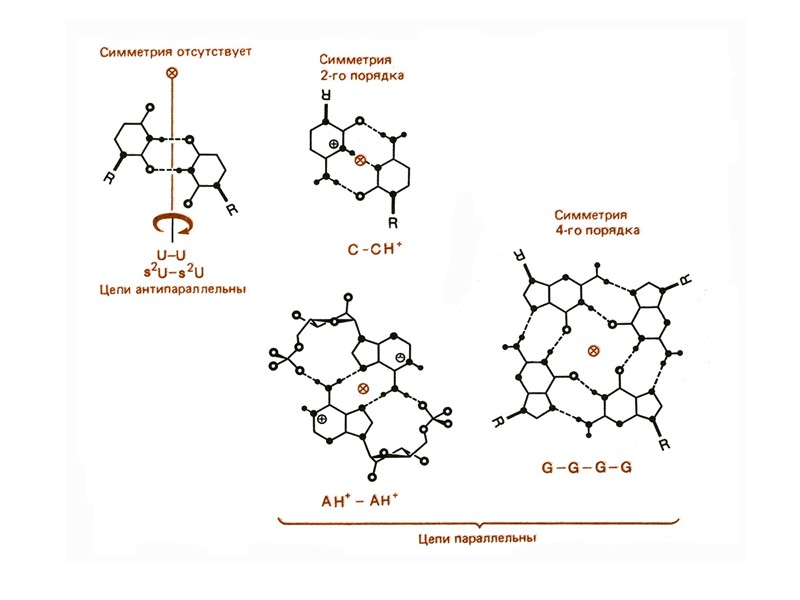
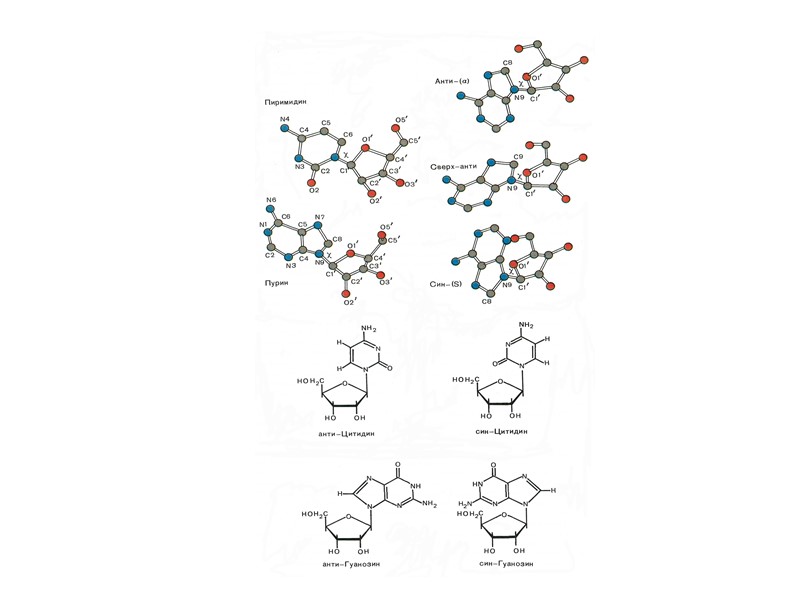
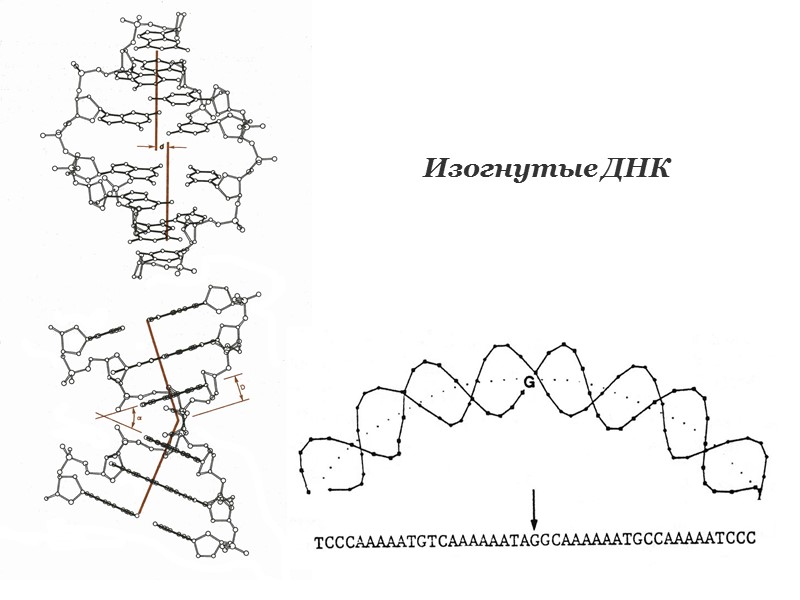
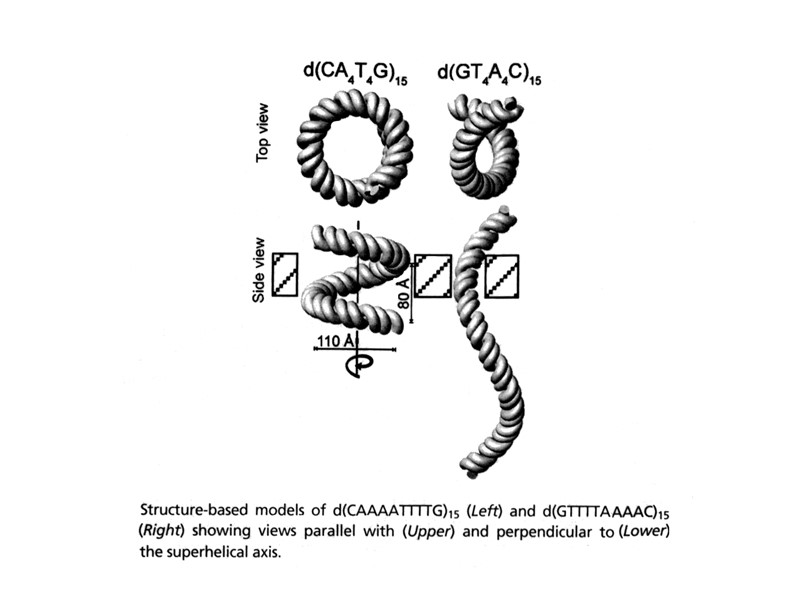
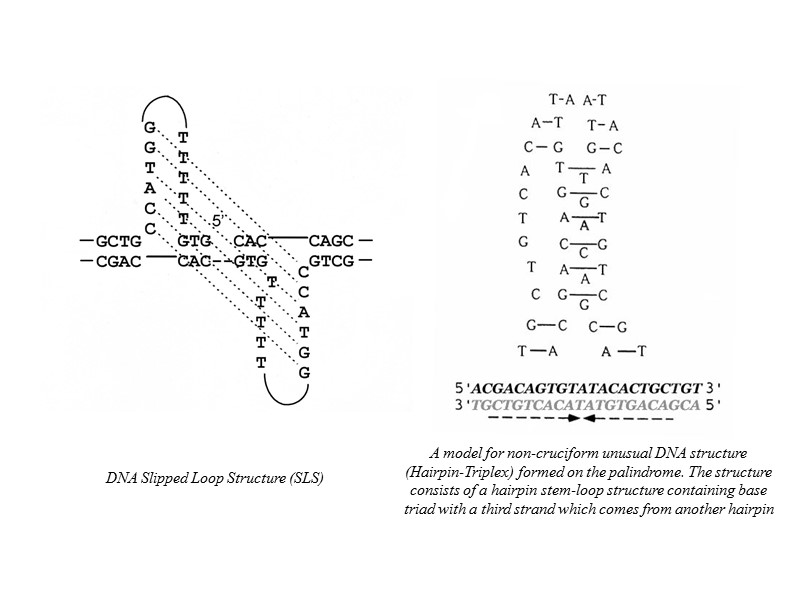
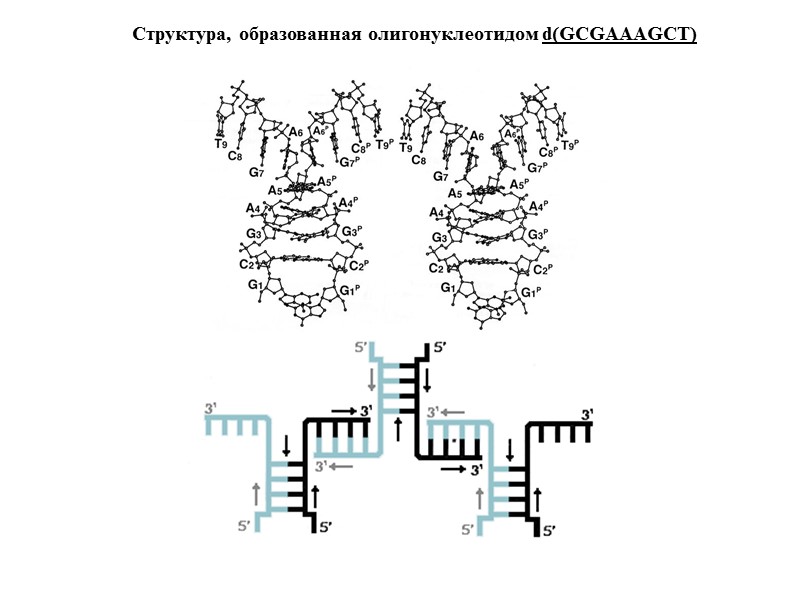
Последовательности ДНК, склонные к образованию неканонических форм повторяющиеся моно-, ди- и тринуклеотидные последовательности, включая пурин-пиримидиновые и пурин-пуриновые повторы гомопуриновые–гомопиримидиновые цепи палиндромные последовательности зеркально-симметричные последовательности двойные спирали, содержащие только А-Т пары oligoA-тракты, oligoG-тракты, oligoC-тракты более сложные типы внутренней симметрии


G-quadruplex structures Quadruplex is stabilized by Na+ and especially K+ ions by a size-selective coordination mechanism. K+ ions are sandwiched between G-quartets to give G8-K+-ion octamer. Oligonucleotide G-quadruplexes differ in their chain number and orientation.


i DNA
Конформационные переходы в ДНК

B-ДНК Бок-о-бок ДНК
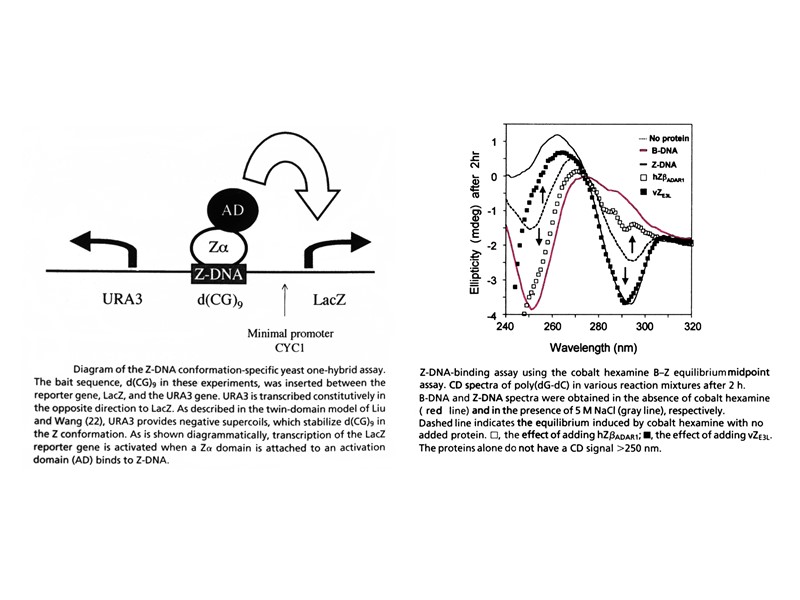
Z form of DNA DNA sequences containing alternating purine and pyrimidine bases have a potential to form the left-handed zigzag Z-DNA, which maintains Watson-Crick base pairing with 12 bp per helical turn d(CG)n / d(CG)n > d(TG)n / d(AC)n > d(TA)n / d(TA)n High ionic strength is required to neutralize the negative charge of phosphate groups which are in close proximity in Z-DNA

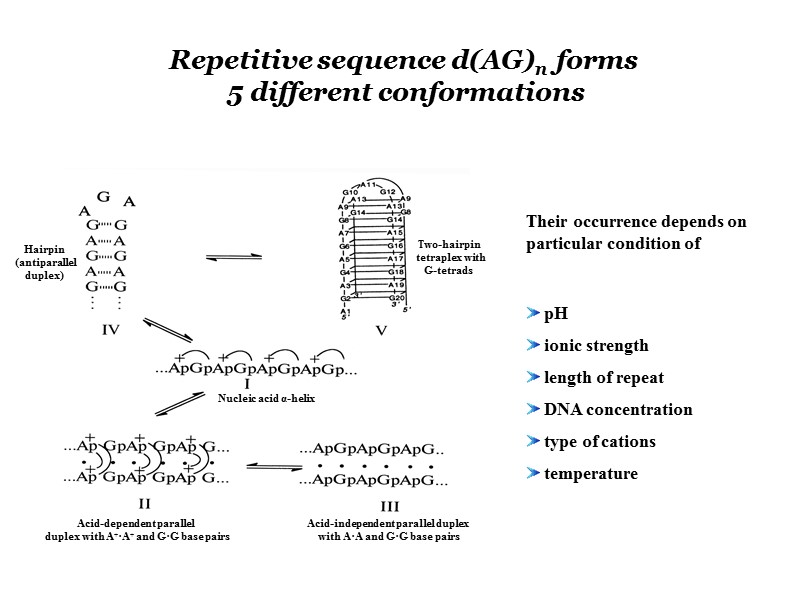
Hairpin (antiparallel duplex) Two-hairpin tetraplex with G-tetrads Nucleic acid α-helix Acid-dependent parallel duplex with A+·A+ and G·G base pairs Acid-independent parallel duplex with A·A and G·G base pairs Repetitive sequence d(AG)n forms 5 different conformations Their occurrence depends on particular condition of pH ionic strength length of repeat DNA concentration type of cations temperature
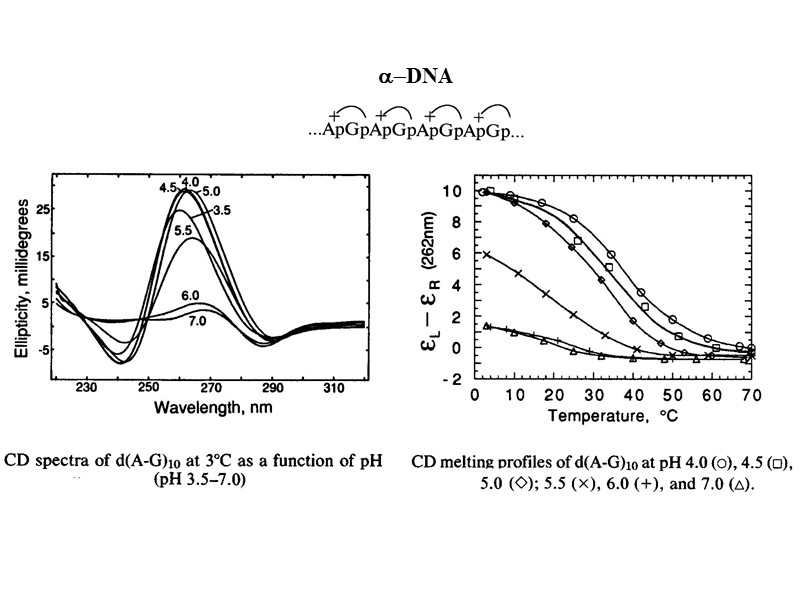
a-DNA
Одноцепочечная спираль d(A-G)10 (alpha-DNA)
UV melting profiles for d(A-G)30 in 0.001 M Na+ between pH 3.5 – 6.0 Acid parallel-stranded DNA-duplex
A+-A+ G-G

S1 – cleavage products of d(A-G)10 at pH 7.4 DNA hairpin tetraplex
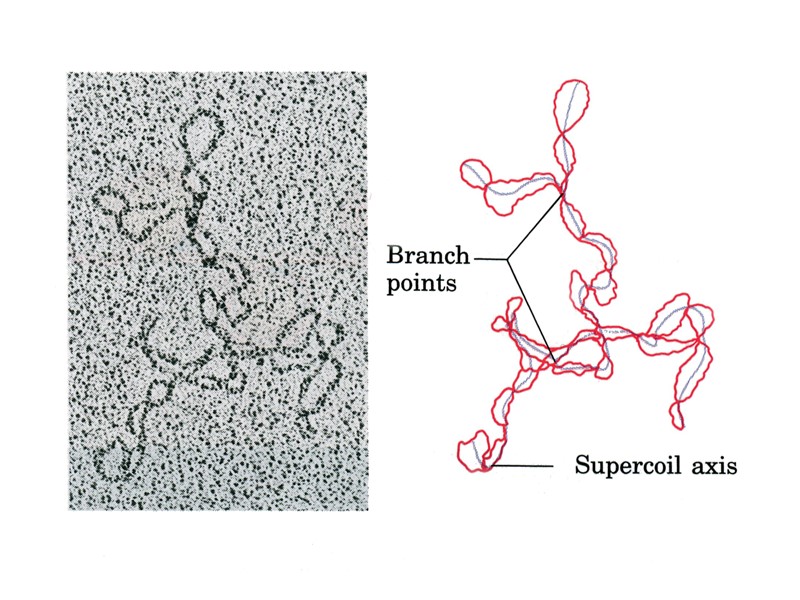
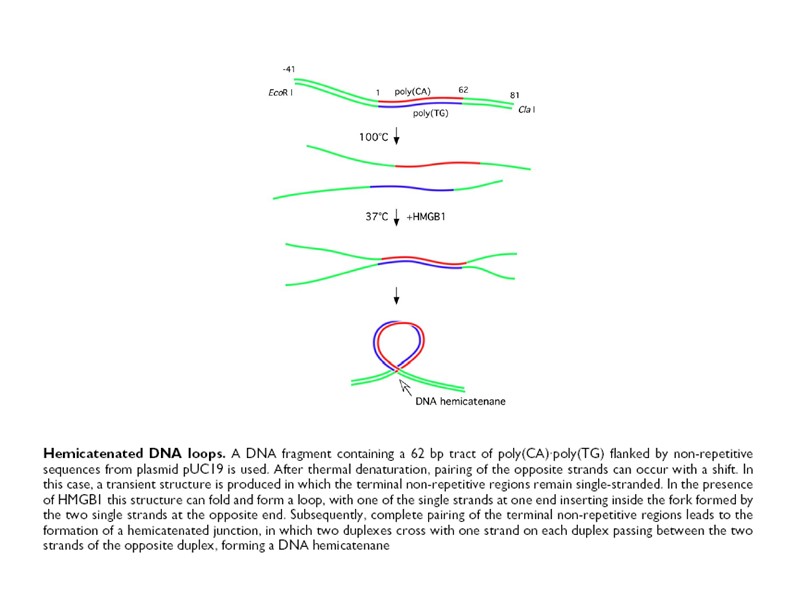
Hemicatenated DNA loops poly(CA)-poly(TG)
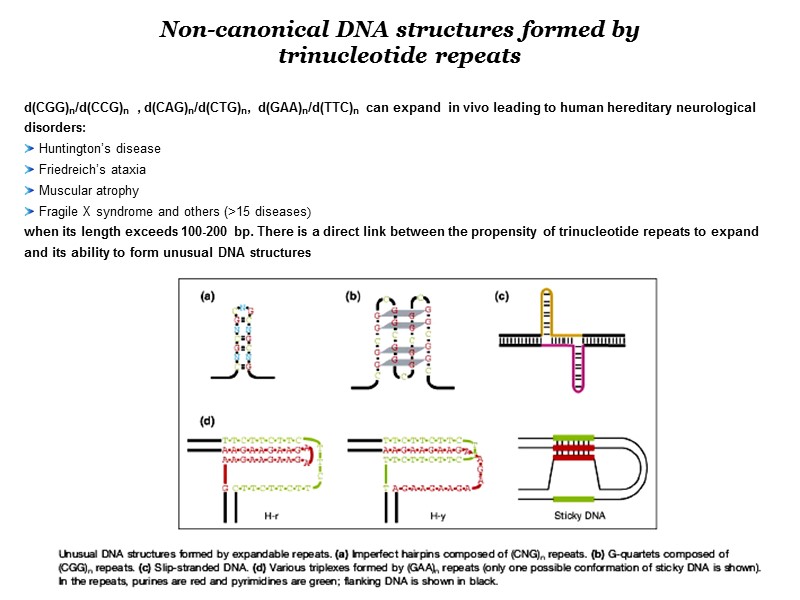
Non-canonical DNA structures formed by trinucleotide repeats d(CGG)n/d(CCG)n , d(CAG)n/d(CTG)n, d(GAA)n/d(TTC)n can expand in vivo leading to human hereditary neurological disorders: Huntington’s disease Friedreich’s ataxia Muscular atrophy Fragile X syndrome and others (>15 diseases) when its length exceeds 100-200 bp. There is a direct link between the propensity of trinucleotide repeats to expand and its ability to form unusual DNA structures

Fragile X syndrome
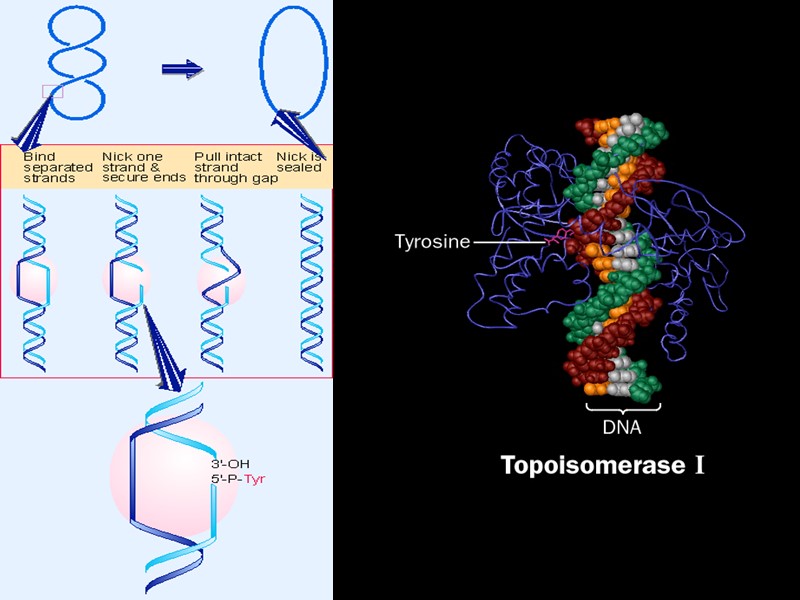
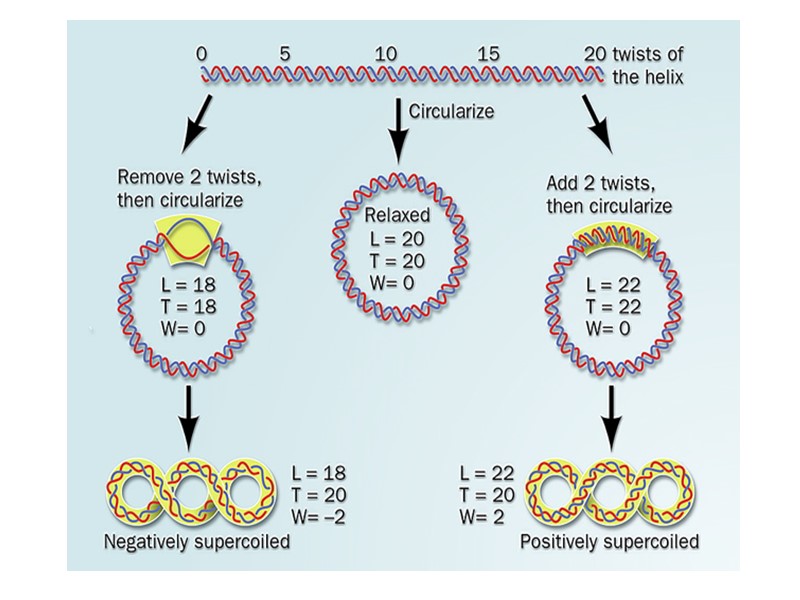

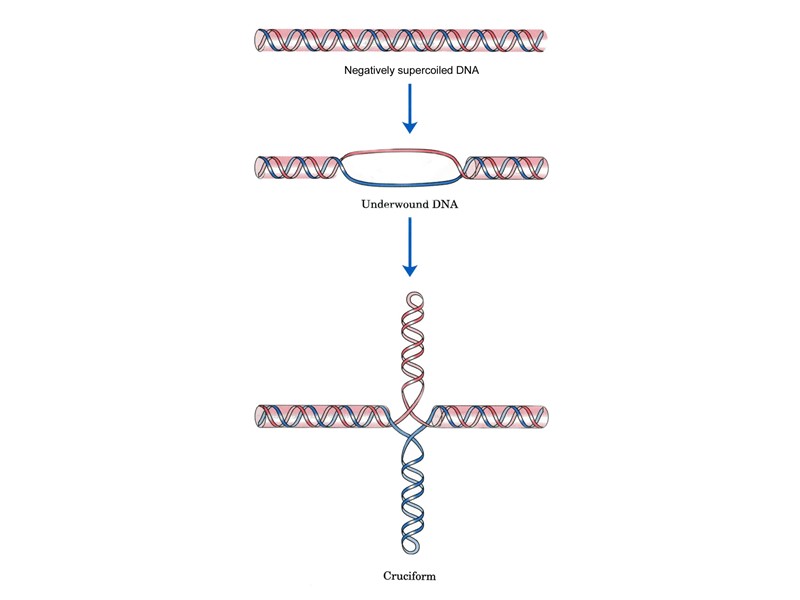
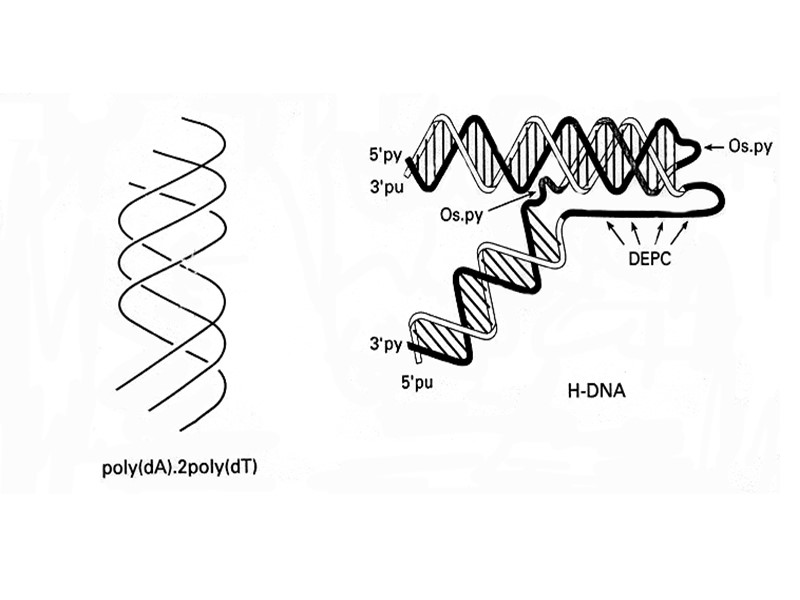
DNA triple-stranded structures Pyrimidine third strands bind parallel to the duplex purine strand trough Hoogsteen bonds. Protonation of the third strand cytosines makes the stability of this system pH - dependent. Watson-Crick duplex target The affinity and specificity of third strand binding is affected by a number of factors: target sequence length and composition presence of purine-pyrimidine inversions in DNA-duplex target solution conditions (mono- and divalent ions concentration)
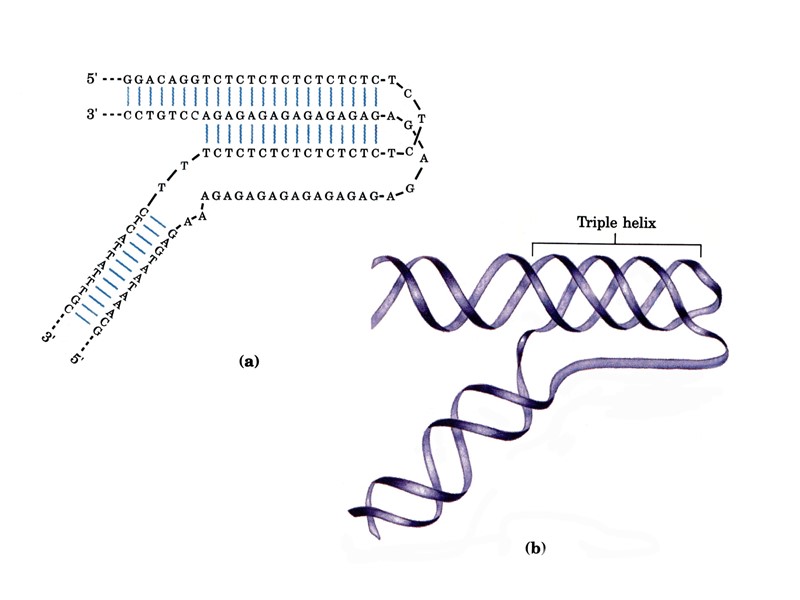
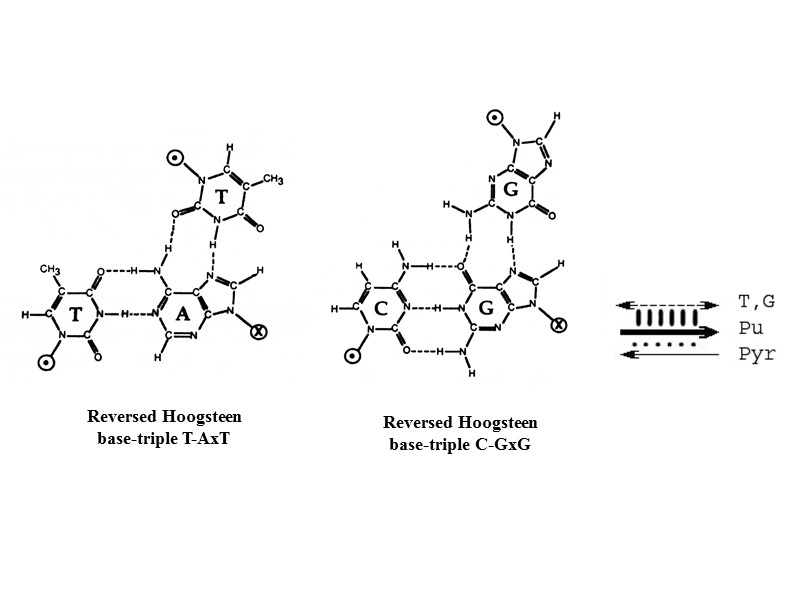
Reversed Hoogsteen base-triple T-AxT Reversed Hoogsteen base-triple C-GxG
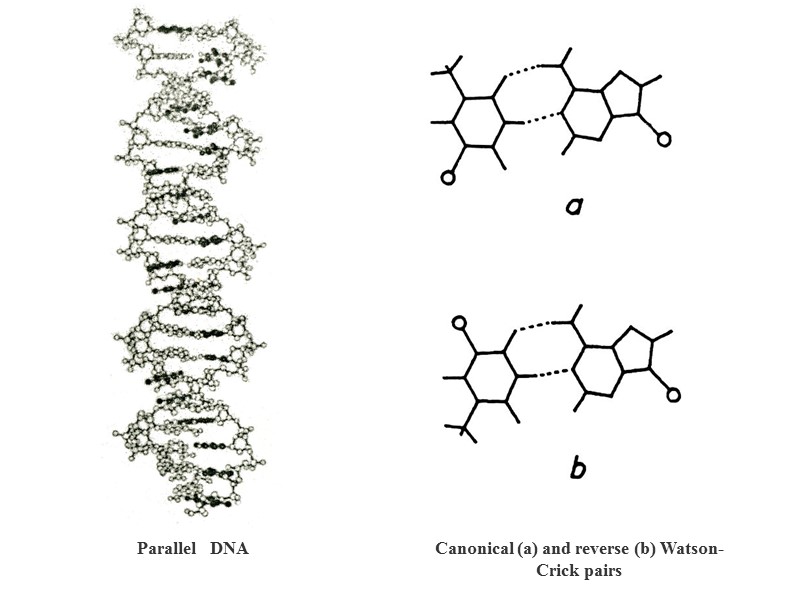
Parallel DNA Canonical (a) and reverse (b) Watson-Crick pairs

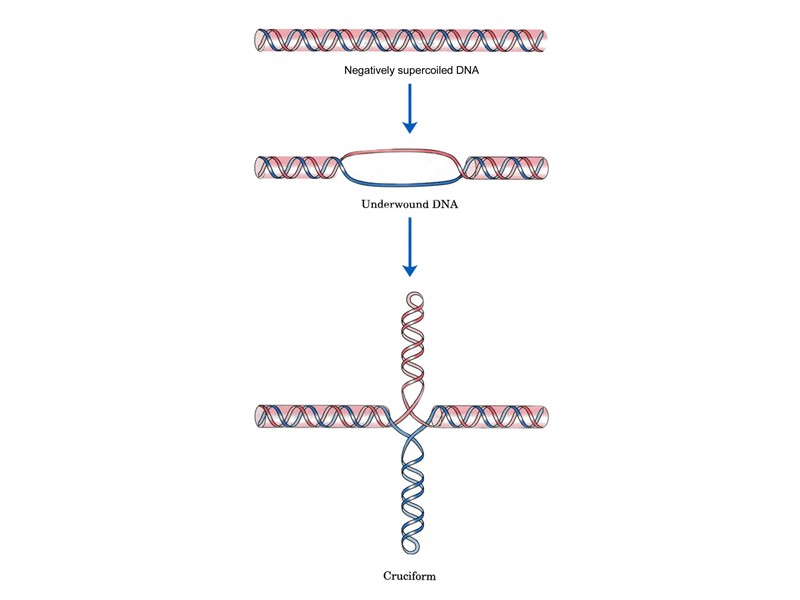
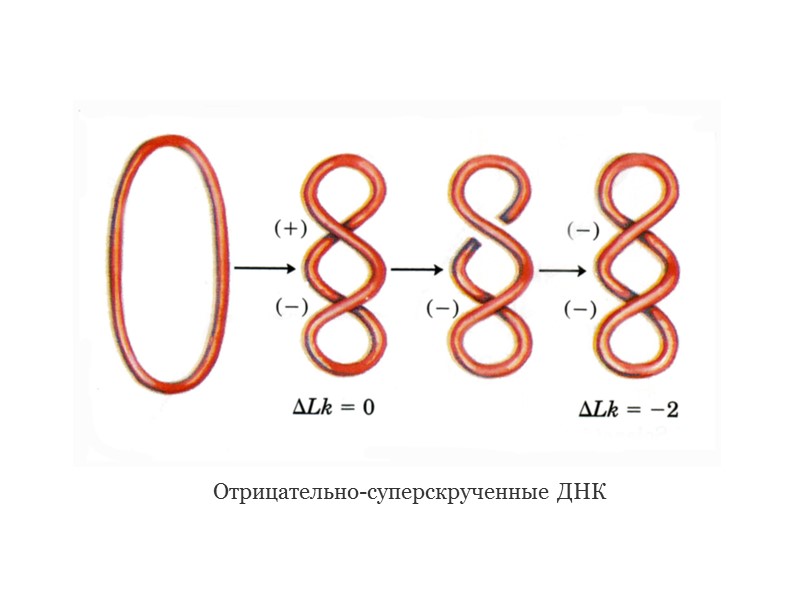
Отрицательно-суперскрученные ДНК



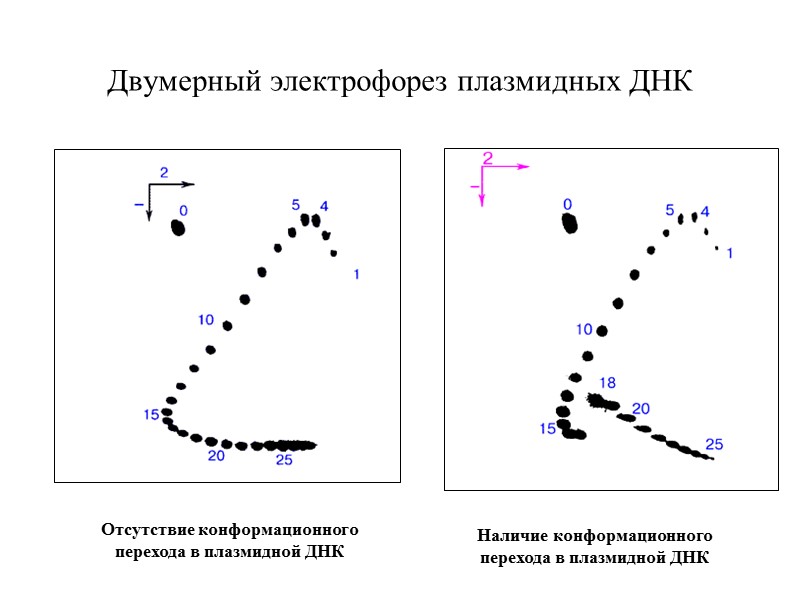
Двумерный электрофорез плазмидных ДНК Отсутствие конформационного перехода в плазмидной ДНК Наличие конформационного перехода в плазмидной ДНК
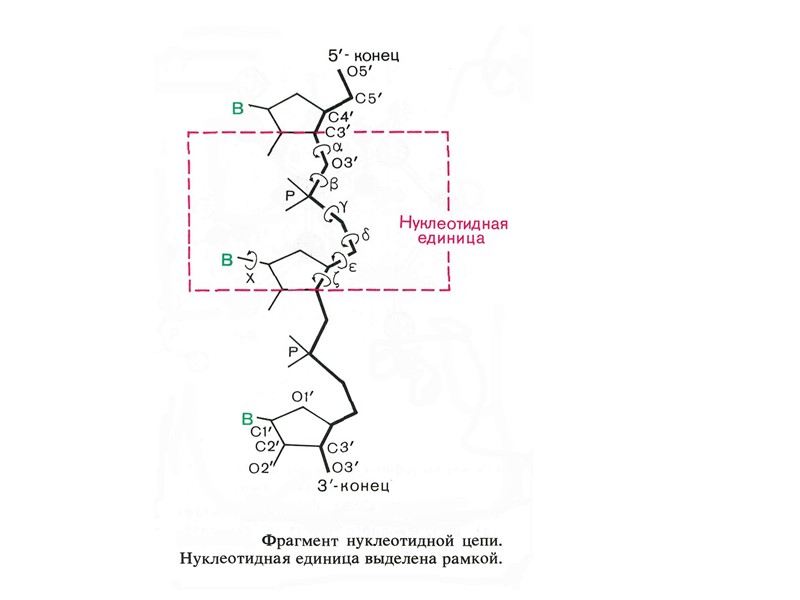
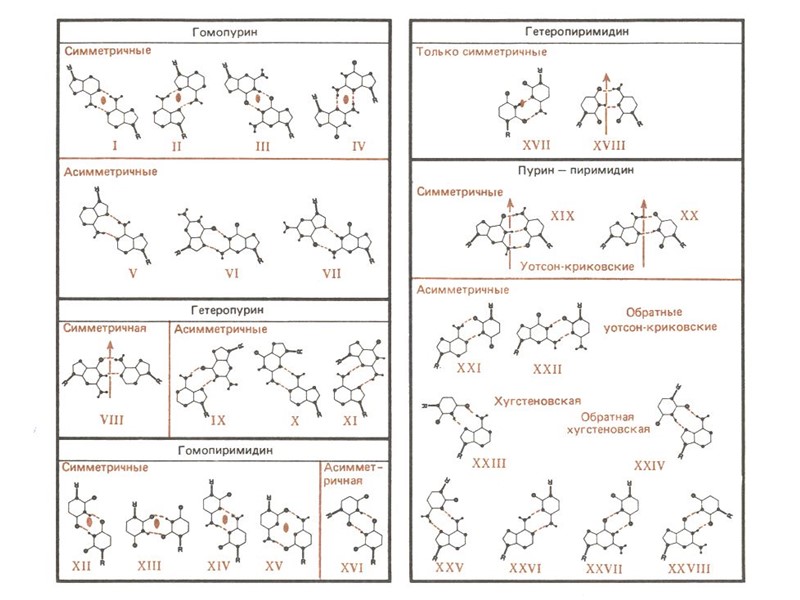
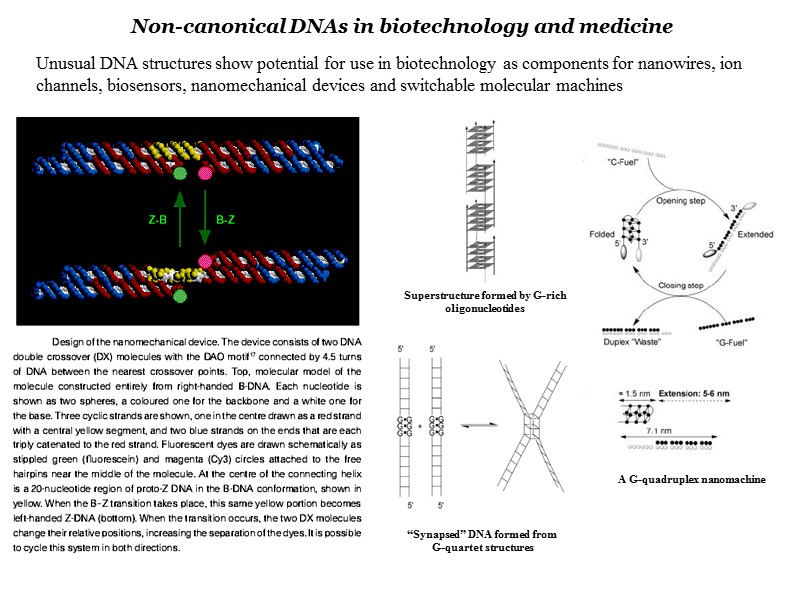
Последовательности, склонные к формированию неканонических форм
Последовательности, образующие неканонические формы ДНК, относительно редки в прокариотах, но широко распространены в эукариотических организмах. Обычно они локализованы в важных участках генома: теломеры хромосом центромерная область горячие точки мутаций промоторные участки генов (часто онкогенов) локусы экспансии тринуклеотидных повторов стык генов легких и тяжелых цепей иммуноглобулина горячие точки рекомбинации
7-dolinnaya.ppt
- Количество слайдов: 67
