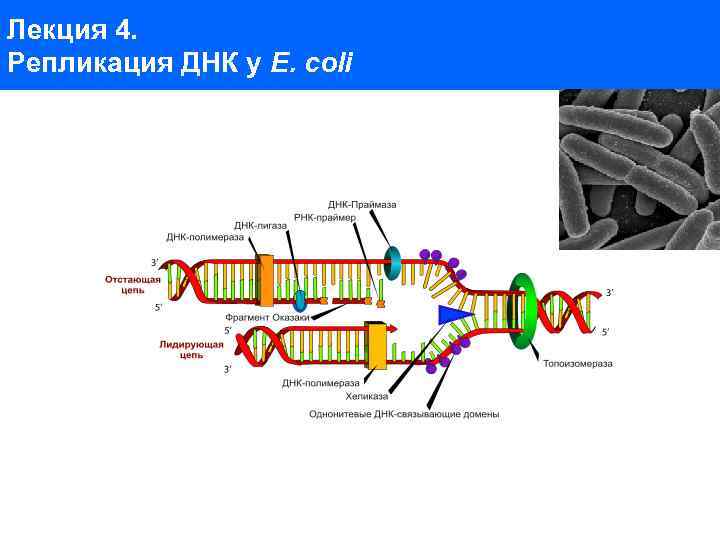
 Лекция 4. Репликация ДНК у E. coli
Лекция 4. Репликация ДНК у E. coli
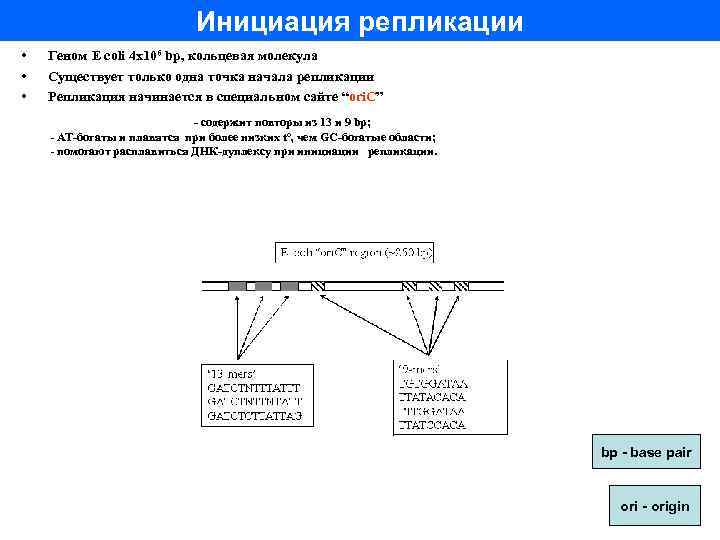 Инициация репликации • • • Геном E coli 4 х106 bp, кольцевая молекула Существует только одна точка начала репликации Репликация начинается в специальном сайте “ori. C” - cодержит повторы из 13 и 9 bp; - АТ-богаты и плавятся при более низких to, чем GC-богатые области; - помогают расплавиться ДНК-дуплексу при инициации репликации. bp - base pair ori - origin
Инициация репликации • • • Геном E coli 4 х106 bp, кольцевая молекула Существует только одна точка начала репликации Репликация начинается в специальном сайте “ori. C” - cодержит повторы из 13 и 9 bp; - АТ-богаты и плавятся при более низких to, чем GC-богатые области; - помогают расплавиться ДНК-дуплексу при инициации репликации. bp - base pair ori - origin
 ori. C Повторы в сайте ori. C: R-боксы – красные прямоугольники R 1 -R 5; голубые стрелки – АТ-богатые 13 - членные последовательности; Seq. A – белок, предотвращающий реинициацию репликации вновь синтезированной ДНК. Серые прямоугольники – сайты метилирования GATC. Зеленые прямоугольники – сайты связывания с комплексом Dna. A-ATP. IHF – Integration Host Factor. В ori. C присутствуют 5 R-боксов, состоящих из 9 пн каждый, и 3 АТ-богатых повторов из 13 пн. Две пары находятся в противоположной ориентации относительно друга. Такой же R-бокс найден в промоторе белка 16 к. Да, ген которого сцеплен с ori. C, и в промоторе гена dna. A, находящегося в том же районе и кодирующего белок Dna. A. Белок 16 к. Да необходим для инициации репликации ДНК и ответствен за ее двунаправленность. Сначала с ori. C связывается инициирующий белок Dna. A, который узнает этот сайт по повторам. Связывание Dna. A с R-боксами стимулирует частичную денатурацию ДНК в районе АТ-богатых повторов из 13 пн.
ori. C Повторы в сайте ori. C: R-боксы – красные прямоугольники R 1 -R 5; голубые стрелки – АТ-богатые 13 - членные последовательности; Seq. A – белок, предотвращающий реинициацию репликации вновь синтезированной ДНК. Серые прямоугольники – сайты метилирования GATC. Зеленые прямоугольники – сайты связывания с комплексом Dna. A-ATP. IHF – Integration Host Factor. В ori. C присутствуют 5 R-боксов, состоящих из 9 пн каждый, и 3 АТ-богатых повторов из 13 пн. Две пары находятся в противоположной ориентации относительно друга. Такой же R-бокс найден в промоторе белка 16 к. Да, ген которого сцеплен с ori. C, и в промоторе гена dna. A, находящегося в том же районе и кодирующего белок Dna. A. Белок 16 к. Да необходим для инициации репликации ДНК и ответствен за ее двунаправленность. Сначала с ori. C связывается инициирующий белок Dna. A, который узнает этот сайт по повторам. Связывание Dna. A с R-боксами стимулирует частичную денатурацию ДНК в районе АТ-богатых повторов из 13 пн.
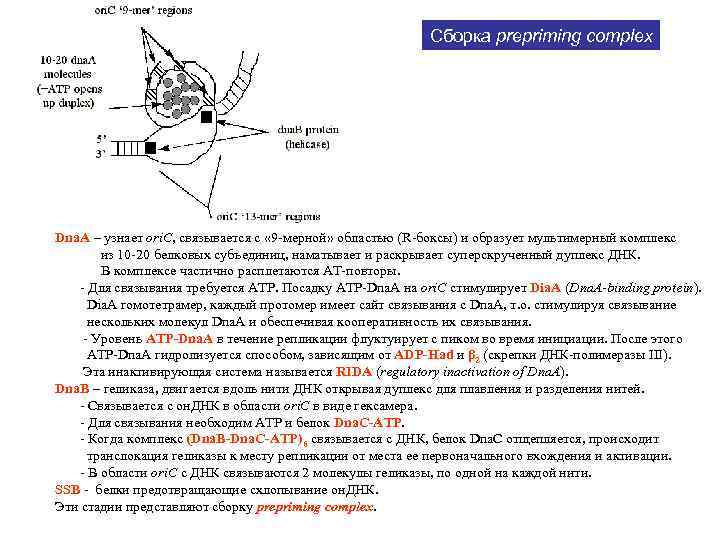 Сборка prepriming complex Dna. A – узнает ori. C, связывается с « 9 -мерной» областью (R-боксы) и образует мультимерный комплекс из 10 -20 белковых субъединиц, наматывает и раскрывает суперскрученный дуплекс ДНК. В комплексе частично расплетаются АТ-повторы. - Для связывания требуется АТР. Посадку ATP-Dna. A на ori. C стимулирует Dia. A (Dna. A-binding protein). Dia. A гомотетрамер, каждый протомер имеет сайт связывания с Dna. A, т. о. стимулируя связывание нескольких молекул Dna. A и обеспечивая кооперативность их связывания. - Уровень ATP-Dna. A в течение репликации флуктуирует с пиком во время инициации. После этого ATP-Dna. A гидролизуется способом, зависящим от ADP-Had и β 2 (скрепки ДНК-полимеразы III). Эта инактивирующая система называется RIDA (regulatory inactivation of Dna. A). Dna. B – геликаза, двигается вдоль нити ДНК открывая дуплекс для плавления и разделения нитей. - Связывается с он. ДНК в области ori. C в виде гексамера. - Для связывания необходим АТР и белок Dna. C-ATP. - Когда комплекс (Dna. B-Dna. C-ATP)6 связывается с ДНК, белок Dna. C отщепляется, происходит транслокация геликазы к месту репликации от места ее первоначального вхождения и активации. - В области ori. C с ДНК связываются 2 молекулы геликазы, по одной на каждой нити. SSB - белки предотвращающие схлопывание он. ДНК. Эти стадии представляют сборку prepriming complex.
Сборка prepriming complex Dna. A – узнает ori. C, связывается с « 9 -мерной» областью (R-боксы) и образует мультимерный комплекс из 10 -20 белковых субъединиц, наматывает и раскрывает суперскрученный дуплекс ДНК. В комплексе частично расплетаются АТ-повторы. - Для связывания требуется АТР. Посадку ATP-Dna. A на ori. C стимулирует Dia. A (Dna. A-binding protein). Dia. A гомотетрамер, каждый протомер имеет сайт связывания с Dna. A, т. о. стимулируя связывание нескольких молекул Dna. A и обеспечивая кооперативность их связывания. - Уровень ATP-Dna. A в течение репликации флуктуирует с пиком во время инициации. После этого ATP-Dna. A гидролизуется способом, зависящим от ADP-Had и β 2 (скрепки ДНК-полимеразы III). Эта инактивирующая система называется RIDA (regulatory inactivation of Dna. A). Dna. B – геликаза, двигается вдоль нити ДНК открывая дуплекс для плавления и разделения нитей. - Связывается с он. ДНК в области ori. C в виде гексамера. - Для связывания необходим АТР и белок Dna. C-ATP. - Когда комплекс (Dna. B-Dna. C-ATP)6 связывается с ДНК, белок Dna. C отщепляется, происходит транслокация геликазы к месту репликации от места ее первоначального вхождения и активации. - В области ori. C с ДНК связываются 2 молекулы геликазы, по одной на каждой нити. SSB - белки предотвращающие схлопывание он. ДНК. Эти стадии представляют сборку prepriming complex.
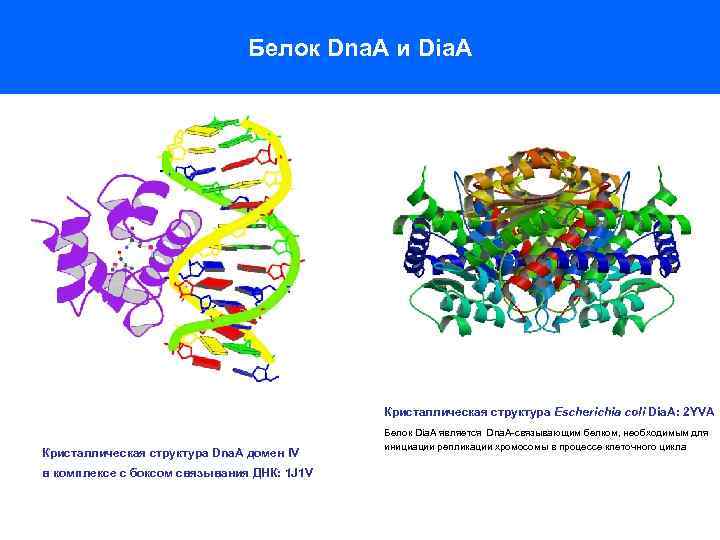 Белок Dna. A и Dia. A Кристаллическая структура Escherichia coli Dia. A: 2 YVA Кристаллическая структура Dna. A домен IV в комплексе с боксом связывания ДНК: 1 J 1 V Белок Dia. A является Dna. A-связывающим белком, необходимым для инициации репликации хромосомы в процессе клеточного цикла
Белок Dna. A и Dia. A Кристаллическая структура Escherichia coli Dia. A: 2 YVA Кристаллическая структура Dna. A домен IV в комплексе с боксом связывания ДНК: 1 J 1 V Белок Dia. A является Dna. A-связывающим белком, необходимым для инициации репликации хромосомы в процессе клеточного цикла
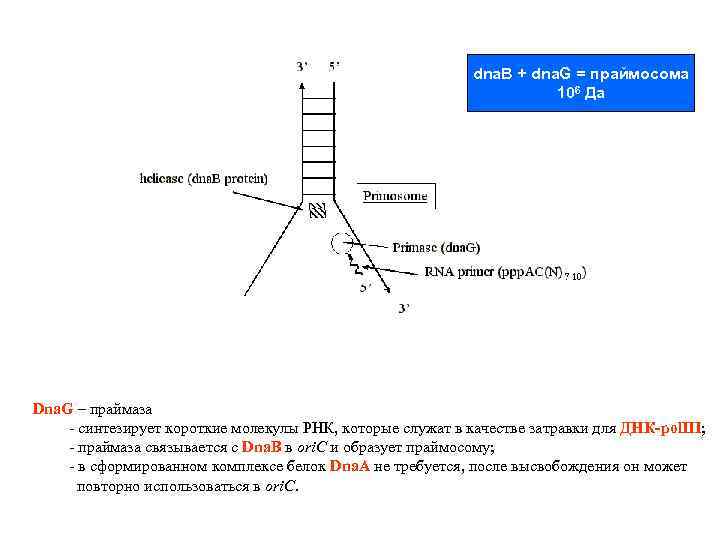 dna. B + dna. G = праймосома 106 Да Dna. G – праймаза - синтезирует короткие молекулы РНК, которые служат в качестве затравки для ДНК-pol. III; - праймаза связывается с Dna. B в ori. C и образует праймосому; - в сформированном комплексе белок Dna. A не требуется, после высвобождения он может повторно использоваться в ori. C.
dna. B + dna. G = праймосома 106 Да Dna. G – праймаза - синтезирует короткие молекулы РНК, которые служат в качестве затравки для ДНК-pol. III; - праймаза связывается с Dna. B в ori. C и образует праймосому; - в сформированном комплексе белок Dna. A не требуется, после высвобождения он может повторно использоваться в ori. C.
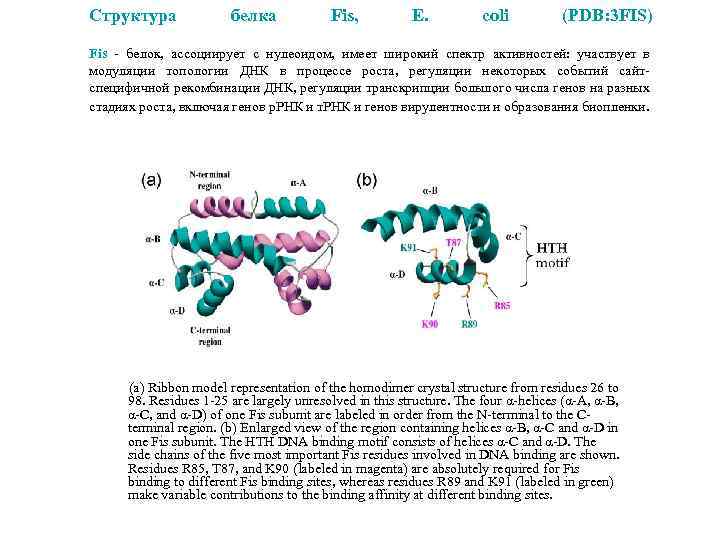 Структура белка Fis, E. coli (PDB: 3 FIS) Fis - белок, ассоциирует с нулеоидом, имеет широкий спектр активностей: участвует в модуляции топологии ДНК в процессе роста, регуляции некоторых событий сайтспецифичной рекомбинации ДНК, регуляции транскрипции большого числа генов на разных стадиях роста, включая генов р. РНК и т. РНК и генов вирулентности и образования биопленки. (a) Ribbon model representation of the homodimer crystal structure from residues 26 to 98. Residues 1 -25 are largely unresolved in this structure. The four α-helices (α-A, α-B, α-C, and α-D) of one Fis subunit are labeled in order from the N-terminal to the Cterminal region. (b) Enlarged view of the region containing helices α-B, α-C and α-D in one Fis subunit. The HTH DNA binding motif consists of helices α-C and α-D. The side chains of the five most important Fis residues involved in DNA binding are shown. Residues R 85, T 87, and K 90 (labeled in magenta) are absolutely required for Fis binding to different Fis binding sites, whereas residues R 89 and K 91 (labeled in green) make variable contributions to the binding affinity at different binding sites.
Структура белка Fis, E. coli (PDB: 3 FIS) Fis - белок, ассоциирует с нулеоидом, имеет широкий спектр активностей: участвует в модуляции топологии ДНК в процессе роста, регуляции некоторых событий сайтспецифичной рекомбинации ДНК, регуляции транскрипции большого числа генов на разных стадиях роста, включая генов р. РНК и т. РНК и генов вирулентности и образования биопленки. (a) Ribbon model representation of the homodimer crystal structure from residues 26 to 98. Residues 1 -25 are largely unresolved in this structure. The four α-helices (α-A, α-B, α-C, and α-D) of one Fis subunit are labeled in order from the N-terminal to the Cterminal region. (b) Enlarged view of the region containing helices α-B, α-C and α-D in one Fis subunit. The HTH DNA binding motif consists of helices α-C and α-D. The side chains of the five most important Fis residues involved in DNA binding are shown. Residues R 85, T 87, and K 90 (labeled in magenta) are absolutely required for Fis binding to different Fis binding sites, whereas residues R 89 and K 91 (labeled in green) make variable contributions to the binding affinity at different binding sites.
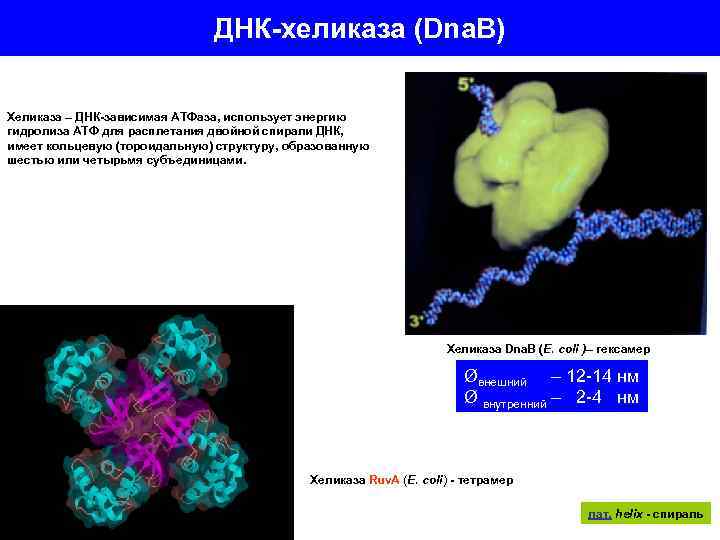 ДНК-хеликаза (Dna. B) Хеликаза – ДНК-зависимая АТФаза, использует энергию гидролиза АТФ для расплетания двойной спирали ДНК, имеет кольцевую (тороидальную) структуру, образованную шестью или четырьмя субъединицами. Хеликаза Dna. B (E. coli )– гексамер Øвнешний – 12 -14 нм Ø внутренний – 2 -4 нм Хеликаза Ruv. A (E. coli) - тетрамер лат. helix - спираль
ДНК-хеликаза (Dna. B) Хеликаза – ДНК-зависимая АТФаза, использует энергию гидролиза АТФ для расплетания двойной спирали ДНК, имеет кольцевую (тороидальную) структуру, образованную шестью или четырьмя субъединицами. Хеликаза Dna. B (E. coli )– гексамер Øвнешний – 12 -14 нм Ø внутренний – 2 -4 нм Хеликаза Ruv. A (E. coli) - тетрамер лат. helix - спираль
 N-terminal domain of E. coli Dna. B helicase, 1 JWE Dna. C, 2 VYF Dna. G, 1 DDE Dna. C-ss. DNA, 2 VYE
N-terminal domain of E. coli Dna. B helicase, 1 JWE Dna. C, 2 VYF Dna. G, 1 DDE Dna. C-ss. DNA, 2 VYE
 ssb- белки ssb-белки состоят из четырех идентичных субъединиц (тетрамер, 88 к. Да) -кооперативно связываются с он. ДНК; - стабилизируют он. ДНК; - обеспечивают условия для синтеза ДНК; -удаляют возможные элементы вторичной структуры ДНК; - стимулируют работу ДНК-полимеразы; - повышают точность работы ДНК-полимеразы. PDB: 1 SRU, ssb- proteins, E. coli ssb - single strand binding proteins – белки, связывающиеся с одноцепочечной ДНК
ssb- белки ssb-белки состоят из четырех идентичных субъединиц (тетрамер, 88 к. Да) -кооперативно связываются с он. ДНК; - стабилизируют он. ДНК; - обеспечивают условия для синтеза ДНК; -удаляют возможные элементы вторичной структуры ДНК; - стимулируют работу ДНК-полимеразы; - повышают точность работы ДНК-полимеразы. PDB: 1 SRU, ssb- proteins, E. coli ssb - single strand binding proteins – белки, связывающиеся с одноцепочечной ДНК
 Элонгация репликации + Праймосома + ДНК-гираза = реплисома + ДНK-pol. III - После синтеза фрагмента размером 9 -12 bp, ДНК - pol. III холофермент поступает в репликативную вилку и использует РНК в качестве праймера для синтеза ДНК в лидирующей нити. - При образовании реплисомы происходит АТР-зависимое формирование димерного комплекса холофермента ДНК-pol. III, связанного с 3’-концом праймера.
Элонгация репликации + Праймосома + ДНК-гираза = реплисома + ДНK-pol. III - После синтеза фрагмента размером 9 -12 bp, ДНК - pol. III холофермент поступает в репликативную вилку и использует РНК в качестве праймера для синтеза ДНК в лидирующей нити. - При образовании реплисомы происходит АТР-зависимое формирование димерного комплекса холофермента ДНК-pol. III, связанного с 3’-концом праймера.
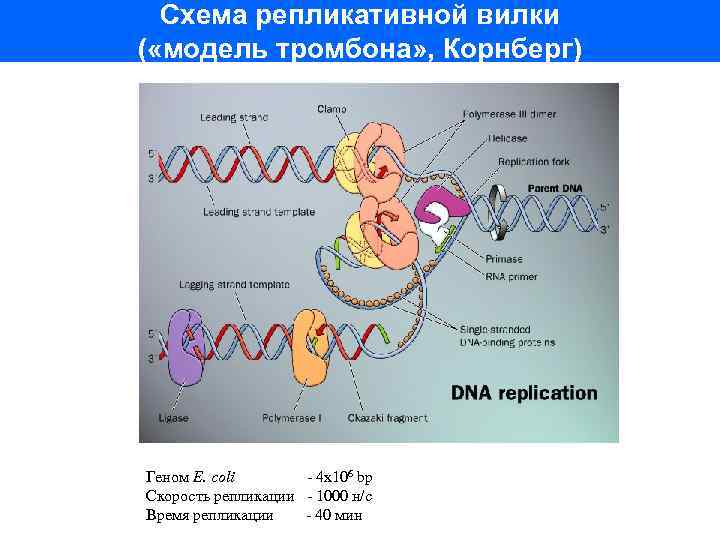 Схема репликативной вилки ( «модель тромбона» , Корнберг) Геном E. coli - 4 х106 bp Скорость репликации - 1000 н/с Время репликации - 40 мин
Схема репликативной вилки ( «модель тромбона» , Корнберг) Геном E. coli - 4 х106 bp Скорость репликации - 1000 н/с Время репликации - 40 мин
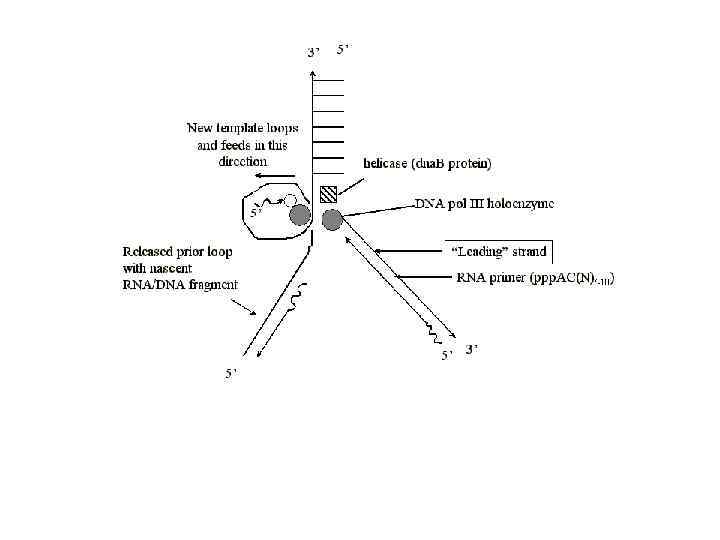
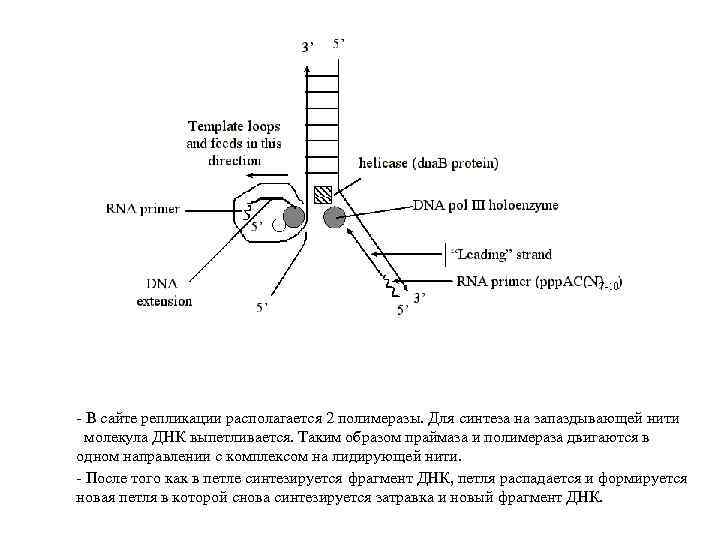 - В сайте репликации располагается 2 полимеразы. Для синтеза на запаздывающей нити молекула ДНК выпетливается. Таким образом праймаза и полимераза двигаются в одном направлении с комплексом на лидирующей нити. - После того как в петле синтезируется фрагмент ДНК, петля распадается и формируется новая петля в которой снова синтезируется затравка и новый фрагмент ДНК.
- В сайте репликации располагается 2 полимеразы. Для синтеза на запаздывающей нити молекула ДНК выпетливается. Таким образом праймаза и полимераза двигаются в одном направлении с комплексом на лидирующей нити. - После того как в петле синтезируется фрагмент ДНК, петля распадается и формируется новая петля в которой снова синтезируется затравка и новый фрагмент ДНК.
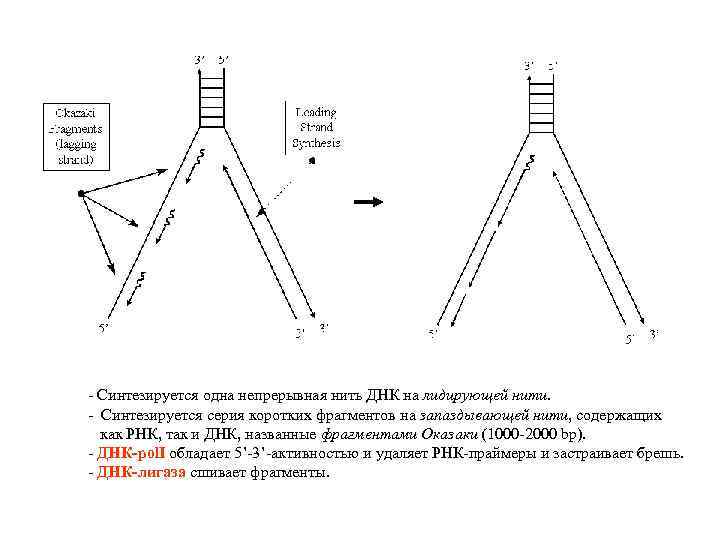 - Синтезируется одна непрерывная нить ДНК на лидирующей нити. - Синтезируется серия коротких фрагментов на запаздывающей нити, содержащих как РНК, так и ДНК, названные фрагментами Оказаки (1000 -2000 bp). - ДНК-pol. I обладает 5’-3’-активностью и удаляет РНК-праймеры и застраивает брешь. - ДНК-лигаза сшивает фрагменты.
- Синтезируется одна непрерывная нить ДНК на лидирующей нити. - Синтезируется серия коротких фрагментов на запаздывающей нити, содержащих как РНК, так и ДНК, названные фрагментами Оказаки (1000 -2000 bp). - ДНК-pol. I обладает 5’-3’-активностью и удаляет РНК-праймеры и застраивает брешь. - ДНК-лигаза сшивает фрагменты.
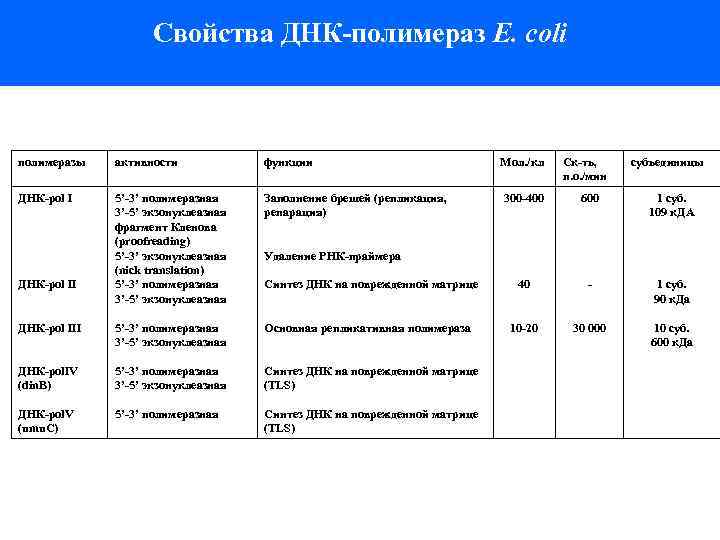 Cвойства ДНК-полимераз E. coli полимеразы активности функции ДНК-pol I 5’-3’ полимеразная 3’-5’ экзонуклеазная фрагмент Кленова (proofreading) 5’-3’ экзонуклеазная (nick translation) 5’-3’ полимеразная 3’-5’ экзонуклеазная Заполнение брешей (репликация, репарация) ДНК-рol III Мол. /кл Ск-ть, п. о. /мин субъединицы 300 -400 600 1 суб. 109 к. ДА Синтез ДНК на поврежденной матрице 40 - 1 суб. 90 к. Да 5’-3’ полимеразная 3’-5’ экзонуклеазная Основная репликативная полимераза 10 -20 30 000 10 суб. 600 к. Да ДНК-pol. IV (din. B) 5’-3’ полимеразная 3’-5’ экзонуклеазная Синтез ДНК на поврежденной матрице (TLS) ДНК-pol. V (umu. C) 5’-3’ полимеразная Синтез ДНК на поврежденной матрице (TLS) ДНК-pol II Удаление РНК-праймера
Cвойства ДНК-полимераз E. coli полимеразы активности функции ДНК-pol I 5’-3’ полимеразная 3’-5’ экзонуклеазная фрагмент Кленова (proofreading) 5’-3’ экзонуклеазная (nick translation) 5’-3’ полимеразная 3’-5’ экзонуклеазная Заполнение брешей (репликация, репарация) ДНК-рol III Мол. /кл Ск-ть, п. о. /мин субъединицы 300 -400 600 1 суб. 109 к. ДА Синтез ДНК на поврежденной матрице 40 - 1 суб. 90 к. Да 5’-3’ полимеразная 3’-5’ экзонуклеазная Основная репликативная полимераза 10 -20 30 000 10 суб. 600 к. Да ДНК-pol. IV (din. B) 5’-3’ полимеразная 3’-5’ экзонуклеазная Синтез ДНК на поврежденной матрице (TLS) ДНК-pol. V (umu. C) 5’-3’ полимеразная Синтез ДНК на поврежденной матрице (TLS) ДНК-pol II Удаление РНК-праймера
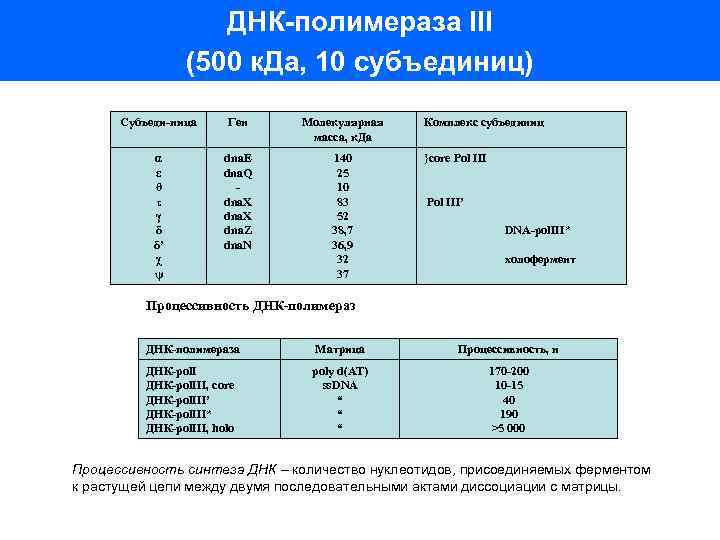 ДНК-полимераза III (500 к. Да, 10 субъединиц) Субъеди-ница Ген Молекулярная масса, к. Да α ε θ τ γ δ δ’ χ ψ dna. E dna. Q dna. X dna. Z dna. N 140 25 10 83 52 38, 7 36, 9 32 37 Комплекс субъединиц }core Pol III’ DNA-pol. III* холофермент Процессивность ДНК-полимераза Матрица Процессивность, н ДНК-pol. III, core ДНК-pol. III’ ДНК-pol. III* ДНК-pol. III, holo poly d(AT) ss. DNA “ “ “ 170 -200 10 -15 40 190 >5 000 Процессивность синтеза ДНК – количество нуклеотидов, присоединяемых ферментом к растущей цепи между двумя последовательными актами диссоциации с матрицы.
ДНК-полимераза III (500 к. Да, 10 субъединиц) Субъеди-ница Ген Молекулярная масса, к. Да α ε θ τ γ δ δ’ χ ψ dna. E dna. Q dna. X dna. Z dna. N 140 25 10 83 52 38, 7 36, 9 32 37 Комплекс субъединиц }core Pol III’ DNA-pol. III* холофермент Процессивность ДНК-полимераза Матрица Процессивность, н ДНК-pol. III, core ДНК-pol. III’ ДНК-pol. III* ДНК-pol. III, holo poly d(AT) ss. DNA “ “ “ 170 -200 10 -15 40 190 >5 000 Процессивность синтеза ДНК – количество нуклеотидов, присоединяемых ферментом к растущей цепи между двумя последовательными актами диссоциации с матрицы.
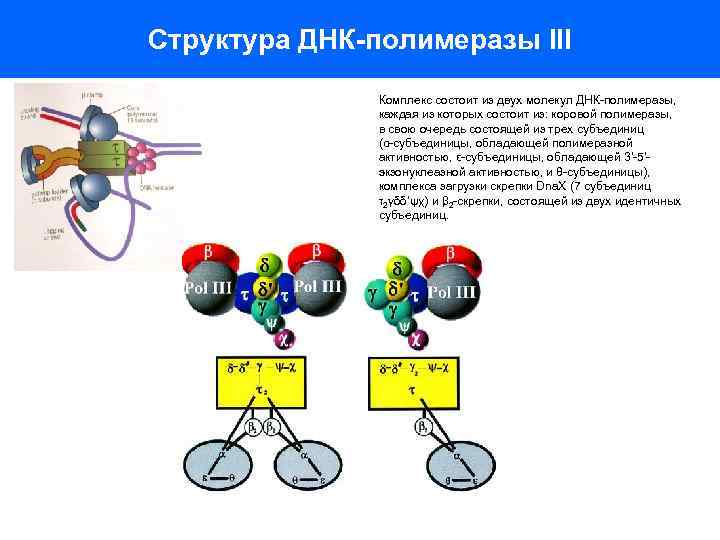 Структура ДНК-полимеразы III Комплекс состоит из двух молекул ДНК-полимеразы, каждая из которых состоит из: коровой полимеразы, в свою очередь состоящей из трех субъединиц (α-субъединицы, обладающей полимеразной активностью, ε-субъединицы, обладающей 3’-5’экзонуклеазной активностью, и θ-субъединицы), комплекса загрузки скрепки Dna. X (7 субъединиц τ2γδδ’ψχ) и β 2 -скрепки, состоящей из двух идентичных субъединиц.
Структура ДНК-полимеразы III Комплекс состоит из двух молекул ДНК-полимеразы, каждая из которых состоит из: коровой полимеразы, в свою очередь состоящей из трех субъединиц (α-субъединицы, обладающей полимеразной активностью, ε-субъединицы, обладающей 3’-5’экзонуклеазной активностью, и θ-субъединицы), комплекса загрузки скрепки Dna. X (7 субъединиц τ2γδδ’ψχ) и β 2 -скрепки, состоящей из двух идентичных субъединиц.
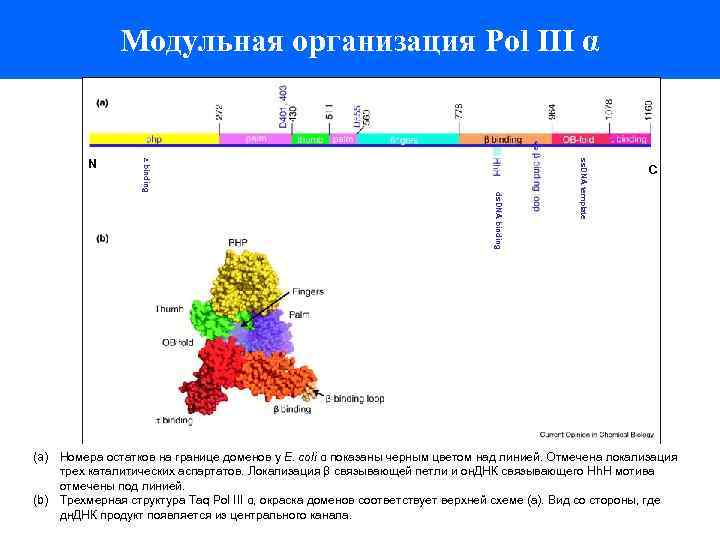 Модульная организация Pol III α ss. DNA template ds. DNA binding ε binding N C (a) Номера остатков на границе доменов у E. coli α показаны черным цветом над линией. Отмечена локализация трех каталитических аспартатов. Локализация β связывающей петли и он. ДНК связывающего Hh. H мотива отмечены под линией. (b) Трехмерная структура Taq Pol III α, окраска доменов соответствует верхней схеме (а). Вид со стороны, где дн. ДНК продукт появляется из центрального канала.
Модульная организация Pol III α ss. DNA template ds. DNA binding ε binding N C (a) Номера остатков на границе доменов у E. coli α показаны черным цветом над линией. Отмечена локализация трех каталитических аспартатов. Локализация β связывающей петли и он. ДНК связывающего Hh. H мотива отмечены под линией. (b) Трехмерная структура Taq Pol III α, окраска доменов соответствует верхней схеме (а). Вид со стороны, где дн. ДНК продукт появляется из центрального канала.
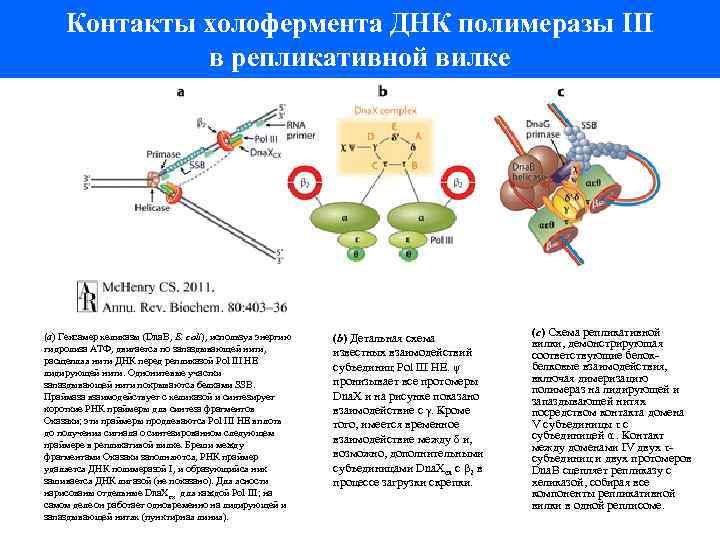 Контакты холофермента ДНК полимеразы III в репликативной вилке (a) Гексамер хеликазы (Dna. B, E. coli), используя энергию гидролиза АТФ, двигается по запаздывающей нити, расщепляя нити ДНК перед репликазой Pol III HE лидирующей нити. Однонитевые участки запаздывающей нити покрываются белками SSB. Праймаза взаимодействует с хеликазой и синтезирует короткие РНК праймеры для синтеза фрагментов Оказаки; эти праймеры продлеваются Pol III HE вплоть до получения сигнала о синтезированном следующем праймере в репликативой вилке. Бреши между фрагментами Оказаки заполняются, РНК праймер удаляется ДНК полимеразой I, и образующийся ник зашивается ДНК лигазой (не показано). Для ясности нарисованы отдельные Dna. X cx для каждой Pol III; на самом деле он работает одновременно на лидирующей и запаздывающей нитях (пунктирная линия). (b) Детальная схема известных взаимодействий субъединиц Pol III HE. ψ пронизывает все протомеры Dna. X и на рисунке показано взаимодействие с γ. Кроме того, имеется временное взаимодействие между δ и, возможно, дополнительными субъединицами Dna. Xcx с β 2 в процессе загрузки скрепки. (c) Схема репликативной вилки, демонстрирующая соответствующие белокбелковые взаимодействия, включая димеризацию полимераз на лидирующей и запаздывающей нитях посредством контакта домена V субъединицы τ с субъединицей α. Контакт между доменами IV двух τсубъединиц и двух протомеров Dna. B сцепляет репликазу с хеликазой, собирая все компоненты репликативной вилки в одной реплисоме.
Контакты холофермента ДНК полимеразы III в репликативной вилке (a) Гексамер хеликазы (Dna. B, E. coli), используя энергию гидролиза АТФ, двигается по запаздывающей нити, расщепляя нити ДНК перед репликазой Pol III HE лидирующей нити. Однонитевые участки запаздывающей нити покрываются белками SSB. Праймаза взаимодействует с хеликазой и синтезирует короткие РНК праймеры для синтеза фрагментов Оказаки; эти праймеры продлеваются Pol III HE вплоть до получения сигнала о синтезированном следующем праймере в репликативой вилке. Бреши между фрагментами Оказаки заполняются, РНК праймер удаляется ДНК полимеразой I, и образующийся ник зашивается ДНК лигазой (не показано). Для ясности нарисованы отдельные Dna. X cx для каждой Pol III; на самом деле он работает одновременно на лидирующей и запаздывающей нитях (пунктирная линия). (b) Детальная схема известных взаимодействий субъединиц Pol III HE. ψ пронизывает все протомеры Dna. X и на рисунке показано взаимодействие с γ. Кроме того, имеется временное взаимодействие между δ и, возможно, дополнительными субъединицами Dna. Xcx с β 2 в процессе загрузки скрепки. (c) Схема репликативной вилки, демонстрирующая соответствующие белокбелковые взаимодействия, включая димеризацию полимераз на лидирующей и запаздывающей нитях посредством контакта домена V субъединицы τ с субъединицей α. Контакт между доменами IV двух τсубъединиц и двух протомеров Dna. B сцепляет репликазу с хеликазой, собирая все компоненты репликативной вилки в одной реплисоме.
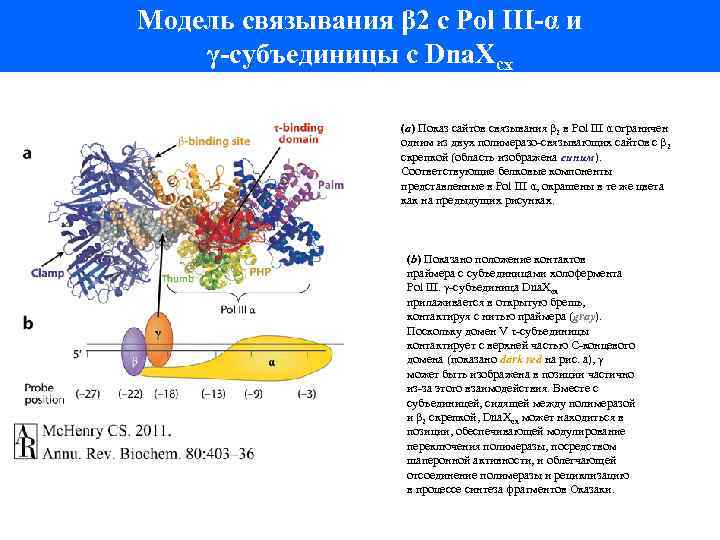 Модель связывания β 2 с Pol III-α и γ-субъединицы с Dna. Xcx (a) Показ сайтов связывания β 2 в Pol III α ограничен одним из двух полимеразо-связывающих сайтов с β 2 скрепкой (область изображена синим). Соответствующие белковые компоненты представленные в Pol III α, окрашены в те же цвета как на предыдущих рисунках. (b) Показано положение контактов праймера с субъединицами холофермента Pol III. γ-субъединица Dna. Xcx прилаживается в открытую брешь, контактируя с нитью праймера (gray). Поскольку домен V τ-субъединицы контактирует с верхней частью C-концевого домена (показано dark red на рис. а), γ может быть изображена в позиции частично из-за этого взаимодействия. Вместе с субъединицей, сидящей между полимеразой и β 2 скрепкой, Dna. Xcx может находиться в позиции, обеспечивающей модулирование переключения полимеразы, посредством шаперонной активности, и облегчающей отсоединение полимеразы и рециклизацию в процессе синтеза фрагментов Оказаки.
Модель связывания β 2 с Pol III-α и γ-субъединицы с Dna. Xcx (a) Показ сайтов связывания β 2 в Pol III α ограничен одним из двух полимеразо-связывающих сайтов с β 2 скрепкой (область изображена синим). Соответствующие белковые компоненты представленные в Pol III α, окрашены в те же цвета как на предыдущих рисунках. (b) Показано положение контактов праймера с субъединицами холофермента Pol III. γ-субъединица Dna. Xcx прилаживается в открытую брешь, контактируя с нитью праймера (gray). Поскольку домен V τ-субъединицы контактирует с верхней частью C-концевого домена (показано dark red на рис. а), γ может быть изображена в позиции частично из-за этого взаимодействия. Вместе с субъединицей, сидящей между полимеразой и β 2 скрепкой, Dna. Xcx может находиться в позиции, обеспечивающей модулирование переключения полимеразы, посредством шаперонной активности, и облегчающей отсоединение полимеразы и рециклизацию в процессе синтеза фрагментов Оказаки.
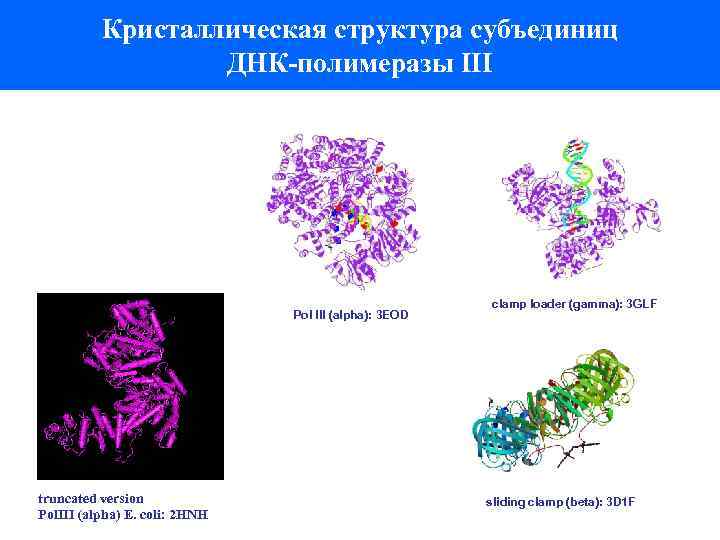 Кристаллическая структура субъединиц ДНК-полимеразы III Pol III (alpha): 3 EOD truncated version Pol. III (alpha) E. coli: 2 HNH clamp loader (gamma): 3 GLF sliding clamp (beta): 3 D 1 F
Кристаллическая структура субъединиц ДНК-полимеразы III Pol III (alpha): 3 EOD truncated version Pol. III (alpha) E. coli: 2 HNH clamp loader (gamma): 3 GLF sliding clamp (beta): 3 D 1 F
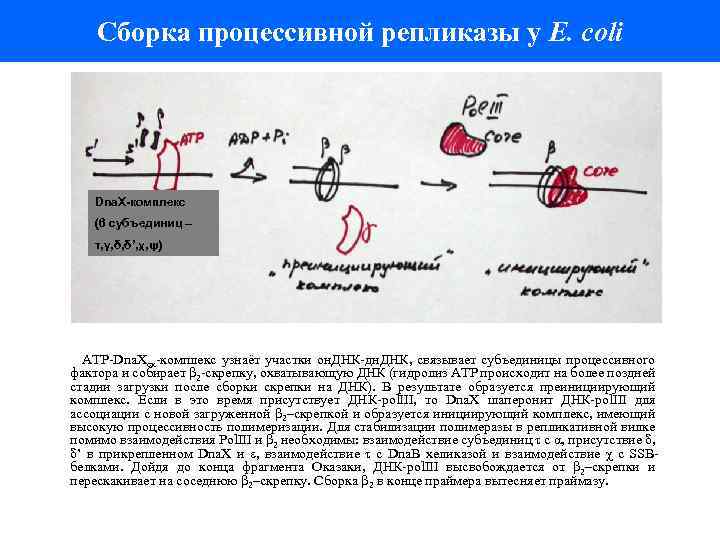 Сборка процессивной репликазы у E. coli Dna. X-комплекс (6 субъединиц – τ, γ, δ, δ’, χ, ψ) АТР-Dna. Xcx-комплекс узнаёт участки он. ДНК-дн. ДНК, связывает субъединицы процессивного фактора и собирает β 2 -скрепку, охватывающую ДНК (гидролиз АТР происходит на более поздней стадии загрузки после сборки скрепки на ДНК). В результате образуется преинициирующий комплекс. Если в это время присутствует ДНК-pol. III, то Dna. X шаперонит ДНК-pol. III для ассоциации с новой загруженной β 2–скрепкой и образуется инициирующий комплекс, имеющий высокую процессивность полимеризации. Для стабилизации полимеразы в репликативной вилке помимо взаимодействия Pol. III и β 2 необходимы: взаимодействие субъединиц τ c α, присутствие δ, δ’ в прикрепленном Dna. X и ε, взаимодействие τ с Dna. B хеликазой и взаимодействие χ с SSBбелками. Дойдя до конца фрагмента Оказаки, ДНК-pol. III высвобождается от β 2–скрепки и перескакивает на соседнюю β 2–скрепку. Сборка β 2 в конце праймера вытесняет праймазу.
Сборка процессивной репликазы у E. coli Dna. X-комплекс (6 субъединиц – τ, γ, δ, δ’, χ, ψ) АТР-Dna. Xcx-комплекс узнаёт участки он. ДНК-дн. ДНК, связывает субъединицы процессивного фактора и собирает β 2 -скрепку, охватывающую ДНК (гидролиз АТР происходит на более поздней стадии загрузки после сборки скрепки на ДНК). В результате образуется преинициирующий комплекс. Если в это время присутствует ДНК-pol. III, то Dna. X шаперонит ДНК-pol. III для ассоциации с новой загруженной β 2–скрепкой и образуется инициирующий комплекс, имеющий высокую процессивность полимеризации. Для стабилизации полимеразы в репликативной вилке помимо взаимодействия Pol. III и β 2 необходимы: взаимодействие субъединиц τ c α, присутствие δ, δ’ в прикрепленном Dna. X и ε, взаимодействие τ с Dna. B хеликазой и взаимодействие χ с SSBбелками. Дойдя до конца фрагмента Оказаки, ДНК-pol. III высвобождается от β 2–скрепки и перескакивает на соседнюю β 2–скрепку. Сборка β 2 в конце праймера вытесняет праймазу.
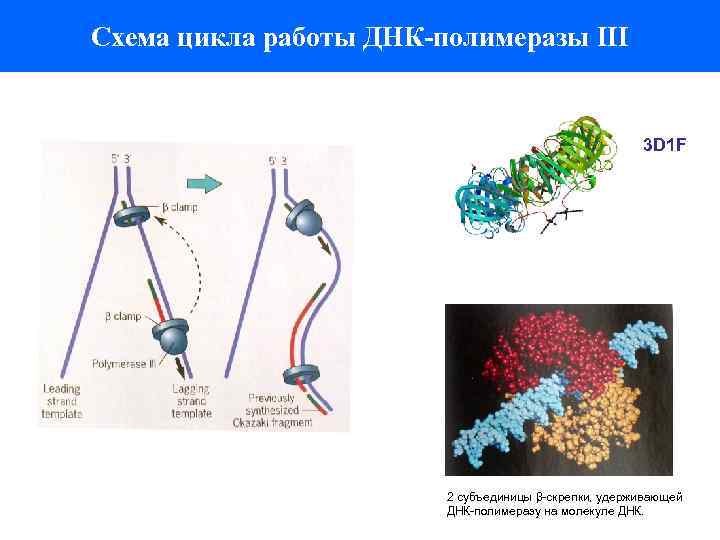 Схема цикла работы ДНК-полимеразы III 3 D 1 F 2 субъединицы β-скрепки, удерживающей ДНК-полимеразу на молекуле ДНК.
Схема цикла работы ДНК-полимеразы III 3 D 1 F 2 субъединицы β-скрепки, удерживающей ДНК-полимеразу на молекуле ДНК.
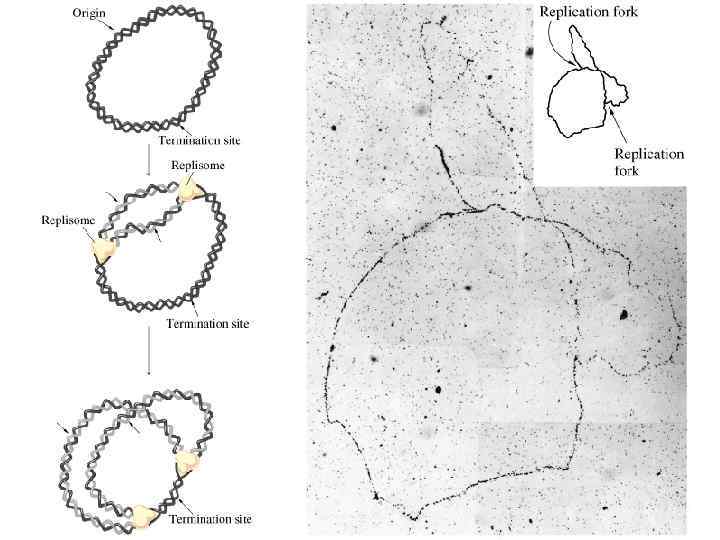
 Терминация репликации ter. C 4 ter. C 5 ter. C 1 • • ter. C 3 ter. C 2 • вилка ori. C вилка • • • Существует специальный сайт окончания репликации; Сайт содержит несколько повторов ter. C-последовательности (~23 bp). У E. coli имеется несколько сайтов ter. C, которые выглядят как 5’-A/T TTAGTTACAACAT A/C CT A/T A/T T-3’: ter. C 1 блокирует вилку репликации, движущуюся против часовой стрелки, а ter. C 2 и ter. C 3 - идущую по часовой стрелке. У многих видов бактерий в хромосомах и плазмидах сайты ter. C гомологичны (последовательности консервативны) и представлены в виде инвертированных повторов. Терминация включает связывание tus-белка с ter. Cпоследовательностями; tus, по-видимому, предотвращают раскручивание ДНК геликазой; Продуктами репликации являются два сцепленных кольца; Разъединение колец осуществляет топоизомераза II типа (топоизомераза IV).
Терминация репликации ter. C 4 ter. C 5 ter. C 1 • • ter. C 3 ter. C 2 • вилка ori. C вилка • • • Существует специальный сайт окончания репликации; Сайт содержит несколько повторов ter. C-последовательности (~23 bp). У E. coli имеется несколько сайтов ter. C, которые выглядят как 5’-A/T TTAGTTACAACAT A/C CT A/T A/T T-3’: ter. C 1 блокирует вилку репликации, движущуюся против часовой стрелки, а ter. C 2 и ter. C 3 - идущую по часовой стрелке. У многих видов бактерий в хромосомах и плазмидах сайты ter. C гомологичны (последовательности консервативны) и представлены в виде инвертированных повторов. Терминация включает связывание tus-белка с ter. Cпоследовательностями; tus, по-видимому, предотвращают раскручивание ДНК геликазой; Продуктами репликации являются два сцепленных кольца; Разъединение колец осуществляет топоизомераза II типа (топоизомераза IV).
 Сегрегация бактериальных репликонов 1 — ДНК; 2 — прикрепление хромосомы к ЦПМ: 3 — ЦПМ; 4 — клеточная стенка: 5 — синтезированный участок ЦПМ; 6 — новый материал клеточной стенки. Механизм распределения бактериальных хромосом: А — бактериальная клетка содержит частично реплицированную хромосому, прикрепленную к мембране в точке (или точках) репликации; Б — репликация хромосомы завершена. В бактериальной клетке две дочерние хромосомы, каждая из которых прикреплена к цитоплазматической мембране. Показан синтез клеточной стенки и цитоплазматической мембраны; В — продолжающийся синтез мембраны и клеточной стенки приводит к разделению дочерних хромосом. Показано начало деления клетки путем образования поперечной перегородки.
Сегрегация бактериальных репликонов 1 — ДНК; 2 — прикрепление хромосомы к ЦПМ: 3 — ЦПМ; 4 — клеточная стенка: 5 — синтезированный участок ЦПМ; 6 — новый материал клеточной стенки. Механизм распределения бактериальных хромосом: А — бактериальная клетка содержит частично реплицированную хромосому, прикрепленную к мембране в точке (или точках) репликации; Б — репликация хромосомы завершена. В бактериальной клетке две дочерние хромосомы, каждая из которых прикреплена к цитоплазматической мембране. Показан синтез клеточной стенки и цитоплазматической мембраны; В — продолжающийся синтез мембраны и клеточной стенки приводит к разделению дочерних хромосом. Показано начало деления клетки путем образования поперечной перегородки.
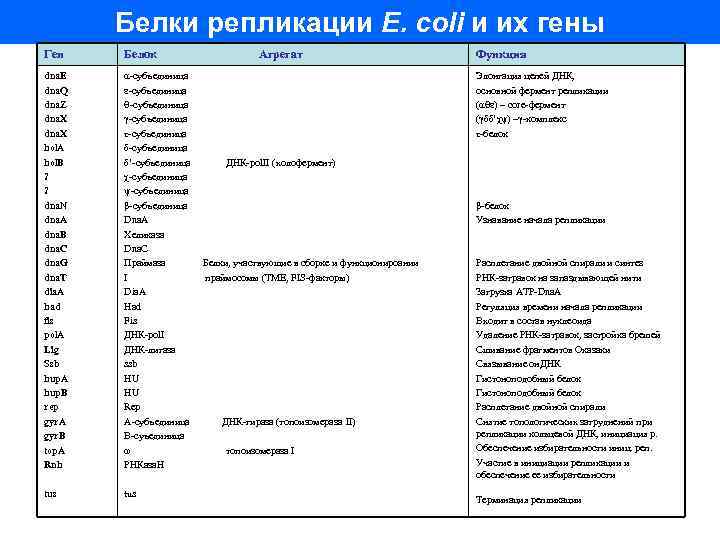 Белки репликации E. coli и их гены Ген Белок dna. E dna. Q dna. Z dna. X hol. A hol. B ? ? dna. N dna. A dna. B dna. C dna. G dna. T dia. A had fis pol. A Lig Ssb hup. A hup. B rep gyr. A gyr. B top. A Rnh α-субъединица ε-субъединица θ-субъединица γ-субъединица τ-субъединица δ’-субъединица χ-субъединица ψ-субъединица β-субъединица Dna. A Хеликаза Dna. C Праймаза I Dia. A Had Fis ДНК-pol. I ДНК-лигаза ssb HU HU Rep А-субъединица В-суъединица ω РНКаза. Н tus Агрегат Функция Элонгация цепей ДНК, основной фермент репликации (αθε) – core-фермент (γδδ’χψ) –γ-комплекс τ-белок ДНК-pol. II (холофермент) β-белок Узнавание начала репликации Белки, участвующие в сборке и функционироании праймосомы (TME, FIS-факторы) ДНК-гираза (топоизомераза II) топоизомераза I Расплетание двойной спирали и синтез РНК-затравок на запаздывающей нити Загрузка ATP-Dna. A Регуляция времени начала репликации Входит в состав нуклеоида Удаление РНК-затравок, застройка брешей Сшивание фрагментов Оказаки Связывание он. ДНК Гистоноподобный белок Расплетание двойной спирали Снятие топологических затруднений при репликации кольцевой ДНК, инициация р. Обеспечение избирательности иниц. реп. Участие в инициации репликации и обеспечение ее избирательности Терминация репликации
Белки репликации E. coli и их гены Ген Белок dna. E dna. Q dna. Z dna. X hol. A hol. B ? ? dna. N dna. A dna. B dna. C dna. G dna. T dia. A had fis pol. A Lig Ssb hup. A hup. B rep gyr. A gyr. B top. A Rnh α-субъединица ε-субъединица θ-субъединица γ-субъединица τ-субъединица δ’-субъединица χ-субъединица ψ-субъединица β-субъединица Dna. A Хеликаза Dna. C Праймаза I Dia. A Had Fis ДНК-pol. I ДНК-лигаза ssb HU HU Rep А-субъединица В-суъединица ω РНКаза. Н tus Агрегат Функция Элонгация цепей ДНК, основной фермент репликации (αθε) – core-фермент (γδδ’χψ) –γ-комплекс τ-белок ДНК-pol. II (холофермент) β-белок Узнавание начала репликации Белки, участвующие в сборке и функционироании праймосомы (TME, FIS-факторы) ДНК-гираза (топоизомераза II) топоизомераза I Расплетание двойной спирали и синтез РНК-затравок на запаздывающей нити Загрузка ATP-Dna. A Регуляция времени начала репликации Входит в состав нуклеоида Удаление РНК-затравок, застройка брешей Сшивание фрагментов Оказаки Связывание он. ДНК Гистоноподобный белок Расплетание двойной спирали Снятие топологических затруднений при репликации кольцевой ДНК, инициация р. Обеспечение избирательности иниц. реп. Участие в инициации репликации и обеспечение ее избирательности Терминация репликации


